Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détection MicroRNA dans les tumeurs prostatiques par quantitative PCR en temps réel (qPCR)
Dans cet article
Résumé
Quantitative de réaction en chaîne polymérase en temps réel (qPCR) est une méthode rapide et sensible pour enquêter sur les niveaux d'expression de microARN (miARN différents) des molécules dans des échantillons tumoraux. En utilisant cette expression méthode de centaines de molécules différentes miARN peuvent être amplifiés, quantifiés, analysés et partir du même modèle d'ADNc.
Résumé
Les microARN (miARN) sont simple brin, 18-24 nucléotides de long, non codantes des molécules d'ARN. Ils sont impliqués dans pratiquement tous les processus cellulaires, y compris le développement 1, l'apoptose 2, et la régulation du cycle cellulaire 3. MiARN sont estimés à réguler l'expression de 30% à 90% des gènes humains 4 en se liant à leurs ARN messagers (ARNm cibles) 5. Dérèglement généralisé des miARN a été rapporté dans diverses maladies et sous-types de cancer 6. En raison de leur prévalence et de la structure unique, ces petites molécules sont susceptibles d'être la prochaine génération de biomarqueurs, des agents thérapeutiques et / ou des cibles.
Les méthodes utilisées pour enquêter sur l'expression du miARN comprennent colorant SYBR Green I-base ainsi que Taqman-sonde qPCR base. Si miARN sont pour être utilisés efficacement dans le contexte clinique, il est impératif que leur détection dans de nouvelles et / ou archivés des échantillons cliniques être fiables, reproductibles, et spS PROPRES. qPCR a été largement utilisé pour valider l'expression des miARN dans les analyses du génome entier, comme les études microarray 7. Les échantillons utilisés dans ce protocole étaient des patients ayant subi une prostatectomie radicale pour cancer de la prostate cliniquement localisé, mais d'autres tissus et des lignées cellulaires peuvent être substitués po spécimens de la prostate étaient composant logiciel enfichable congelés dans l'azote liquide après résection. Les variables cliniques et de suivi d'information pour chaque patient ont été recueillies pour une analyse ultérieure 8.
Quantification des niveaux miRNA dans des échantillons de tumeurs de la prostate. Les principales étapes de l'analyse qPCR des tumeurs sont: Total extraction de l'ARN, la synthèse d'ADNc, et la détection des produits qPCR utilisant des amorces spécifiques de miARN. L'ARN total, qui comprend l'ARNm, miRNA, et d'autres petits ARN ont été extraits à partir d'échantillons à l'aide du réactif Trizol. Système de Qiagen miScript a été utilisé pour synthétiser l'ADNc et d'effectuer qPCR (figure 1). MiARN endogènes ne sont pas polyadenylated, donc pendant le processus de transcription inverse, un poly (A) polymérase polyadenylates miRNA. Le miARN est utilisé comme modèle pour synthétiser l'ADNc en utilisant l'oligo-dT et la transcriptase inverse. Séquence d'étiquette universel sur l'extrémité 5 'de l'oligo-dT amorces facilite l'amplification de l'ADNc dans l'étape de PCR. D'amplification du produit de PCR est détectée par le niveau de fluorescence émise par SYBR Green, un colorant qui s'intercale dans l'ADN double brin. Amorces spécifiques miRNA, ainsi que d'une amorce universelle qui se lie à la séquence de balises universel amplifier des séquences spécifiques miRNA.
Les dosages d'amorces miScript sont disponibles pour plus d'un millier spécifiques à l'homme miARN et des centaines de souris spécifiques miARN. Méthode de quantification relative a été utilisée ici pour quantifier l'expression des miARN. À corriger des variations entre les différents échantillons, les niveaux d'expression d'un miARN cible est normalisée à des niveaux d'expression d'un gène de référence. Le choix d'un gesur lequel ne pour normaliser l'expression de cibles est critique dans Procédé quantification relative de l'analyse. Des exemples de gènes de référence généralement utilisés dans cette capacité sont les RNU6B petits ARN, RNU44, et RNU48 car ils sont considérés être exprimé de façon stable dans la plupart des échantillons. Dans ce protocole, RNU6B est utilisé en tant que gène de référence.
Protocole
1. Prélèvement d'échantillons de la prostate
- Recueillir des échantillons de la prostate au moment de la prostatectomie. L'échantillon est orienté à l'aide des repères anatomiques. La prostate et les vésicules séminales sont peints comme suit: vert à droite, bleu à gauche.
- Un tour de taille aléatoire transversal de la prostate est prélevée perpendiculairement à la surface rectale, congelés dans l'azote liquide, et conservés à -80 ° C 9.
- Tranches en banque de spécimens sont photocopiées, orientée (antérieure, postérieure, droite et gauche), quadrisécable. Les articles sont coupés à l'aide du cryostat.
- Les sections sont colorées avec H & E et examiné par un pathologiste de déterminer et de délimiter les zones tumorales par rapport normale sur les lames colorées et une image correspondante. Les zones marquées sont utilisées en tant que guide pour indiquer les zones à partir de laquelle le tissu tumoral extraire à partir de laquelle l'ARN est extraite dans les étapes ultérieures.
2. Isolation de l'ARN total, y compris les miARN, dans des échantillons
- Placer les échantillons de la prostate congelés sur glace sèche et se référant à l'délimité photocopie, découper une petite partie de la tumeur de la prostate (entre 50 et 100 mg).
- Homogénéiser le tissu tumoral de la prostate dans 1 ml de réactif Trizol. Les quantités dans les étapes suivantes sont basées sur l'utilisation de 1 ml de réactif Trizol.
Note: Ici nous avons utilisé réactif Trizol pour l'ARN extraction, cependant d'autres kits qui isolent les petits ARN contenant l'ARN total peut également être utilisé.
- Incuber les échantillons homogénéisés pour 5 minutes à température ambiante.
- Ajouter 0,2 ml de chloroforme pour les échantillons et agiter vigoureusement pendant 15 secondes. Incuber les échantillons pendant 3 minutes à température ambiante, puis centrifuger à 12 000 xg pendant 15 minutes à 4 ° C.
- Transférer la phase aqueuse supérieure incolore à tubes fraîches, et ajoutez 0,5 ml d'alcool isopropylique.Incuber les échantillons pendant 10 minutes à température ambiante, puis centrifuger à 12 000 xg pendant 10 minutes à 4 ° C.
- Aspirer soigneusement le surnageant sans déranger le culot contenant l'ARN. Laver le culot d'ARN avec 1 ml d'éthanol à 75%. Vortex de l'échantillon et re-sédiments par centrifugation pendant 5 minutes à 7500 xg à 4 ° C.
- Aspirer soigneusement le surnageant et sécher le culot d'ARN pendant 5-10 minutes, en s'assurant que le culot d'ARN n'est pas complètement sèche. Re-dissoudre dans une eau sans nucléase appropriée à la taille des granulés. Mesurer la concentration de l'ARN en utilisant le spectrophotomètre NanoDrop 1000 (absorbance mesure à 260 nm et 280 nm).
- Vérifier la qualité et l'intégrité des échantillons d'ARN à l'aide Agilent Bioanalyzer.
3. Transcription inverse de l'ARN
- La transcription inverse de l'ARN a été réalisée à l'aide miScript Kit de transcription inverse selon les instructions du fabricant (Qiagen). Ce kit comprend untranscriptase inverse et un poly (A) polymérase. Le tampon miScript RT comprend Mg 2 +, dNTP, d'oligo-dT amorces, et des amorces aléatoires.
- Utilisez entre 10 pg et 1 ug d'ARN pour synthétiser l'ADNc. Si vous utilisez plus de 1 ug d'ARN, intensifier la réaction linéairement au volume approprié.
- Préparer un mélange maître qui contient 5X miScript RT Buffer (4 pi), Mix miScript Reverse Transcription (1 pi), et l'eau sans RNase pour amener les réactions à volume final de 20 ul. Comprennent également la matrice ARN (jusqu'à 1 mg) dans le mélange maître.
- Incuber les échantillons pendant 60 minutes à 37 ° C, suivie immédiatement par une incubation de 5 minutes à 95 ° C. Cette étape peut être réalisée dans une machine à PCR, du bain chauffant bloc, ou de l'eau. Thermocycleurs sont la méthode la plus adaptée et précise. Stocker l'ADNc sur la glace pour le court terme, et -20 ° C pour le stockage à long terme.
4. Génération d'une courbe standard
- Avant expertiseiment avec miRNAs cibles, une courbe d'étalonnage est générée en utilisant des concentrations connues ADNc de contre leurs points de croisement (CP) (figure 2).
- Préparer une série de dilutions de 2 fois, 10 fois, 50 fois, 250 fois, et 1250 fois l'ADNc d'origine d'un échantillon qui est connu pour avoir une expression importante de votre gène d'intérêt.
- Exécutez le PCR comme spécifié dans la section 5 "PCR en temps réel pour la détection de miARN", avec la modification que l'ADNc est en dilutions en série n'est pas une dilution statique 40x.
- Effectuer une analyse utilisant un logiciel RelQuant (Roche) pour générer votre courbe standard.
Remarque: une nouvelle courbe standard doit être généré pour chaque gène d'intérêt.
5. PCR en temps réel pour la détection de miRNA
- PCR en temps réel pour les miARN a été réalisée à l'aide miScript SYBR Green PCR Kit de dosage et de Primer miScript selon les instructions du fabricant (Qiagen). Préparer un mélange maître contenant2x QuantiTect SYBR Green PCR Master Mix, 10x miScript Universal Primer, 10x Assay Primer miScript, et l'eau sans RNase. Préparer un mélange maître pour une réaction un volume de 20 ul.
- Le dosage apprêt est spécifique à l'miARN d'intérêt. Pour reconstituer miScript Primer dosage 10x, centrifuger brièvement le flacon, et ajouter 550 ul de tampon TE, pH 8,0. Vortex brièvement le flacon pour mélanger, amorces aliquotes à des volumes plus petits, et conserver à -20 ° C. Deux amorces sont nécessaires:. Amorces pour le gène cible et le gène de référence RNU6B est utilisé tel quel le gène de référence.
- Diluer les ADNc 40x et entreposer des portions supplémentaires à -20 ° C.
- ADNc sert de modèle pour la PCR. Utilisez 2 pi de 40x ADNc dilué et de dispenser à la 20 pi Light Cycler capillaires (Roche).
- Ajouter 18 ul du mélange maître à chaque capillaire, et centrifuger à l'aide d'un adaptateur capillaire.
- Placez les capillaires dans un capillaire en temps réel basé cycleur, comme LightCycler 3.5 Real-Time System PCR avec un format carrousel 32-capillaire.
- Exécutez le programme du cyclisme sur la PCR comme suit:
Pour activer la polymérase HotStarTaq qui est dans le 2x QuantiTect SYBR Green PCR Master Mix, pré-incuber à 95 ° C pendant 15 minutes.
Suivi par 50 cycles de:
Dénaturation, 15 s, 94 ° C;
Recuit, 30 s, 55 ° C;
Extension, 30 s, 70 ° C. - Sélectionner un échantillon d'être le calibrateur, et définir sa valeur cible normalisée à 1. Comparer l'expression relative de la miARN dans tous les autres échantillons à l'étalonneur.
Remarque: Dans une étude, le même échantillon de calibrage doit être utilisé pour maintenir la cohérence des résultats.
6. Analyse des données
- Courbes d'amplification pour les réactions de PCR sont représentées graphiquement et numériquement par la version du logiciel biochimiques moléculaire LightCycler 3.5 (Roche). Quantifier les réactions dans le "Quantification" onglet, une exporter les données vers un fichier texte.
- Importez les données vers le logiciel d'analyse RelQuant (Roche) pour générer des résultats de quantification. Importez des fichiers séparés pour le gène cible, gène de référence, et les données de la courbe standard.
- Spécifiez la position de l'étalon à la fois pour cible et gène de référence. Précisez également la position des échantillons. Les données sont exprimées en tant que cible pour référencer ratio de différents échantillons, divisé par le ratio de référence cible du calibrateur. La courbe standard généré précédemment pour un miRNA particulier et gène de ménage est utilisé comme norme de référence pour l'extrapolation des données quantitatives pour cibles des miRNA de concentrations inconnues.
- Trois répliques d'échantillons sont analysés en tant que groupe et les concentrations moyennes et les écarts types de la triple est calculé. Si l'un des triplets est incompatible avec le reste de l'ensemble, il sera exclu par le programme.
7. Les résultats représentatifs
Un exemple d'analyse qPCR sur des échantillons de la prostate est illustré à la figure 3. Les résultats sont représentés numériquement, ainsi que sous forme graphique. Les graphiques montrant les niveaux d'expression du gène de référence, U6, commencer une amplification exponentielle à environ cycle de 20, tandis que l'expression du gène cible, miR-98, a montré l'amplification retardé à peu près au cycle de 25. Les données de cette expérience a été exportés dans un fichier texte et analysées par un logiciel d'analyse RelQuant. Positions des capillaires contenant le calibrateur et des échantillons sont précisées. La figure 4 illustre la façon dont le calibrateur est fixé à 1, et l'expression d'autres échantillons par rapport à l'étalon.
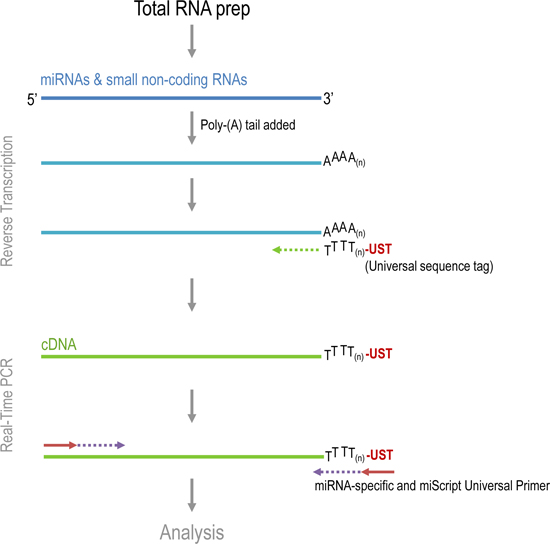
Figure 1. Diverses mesures dans miScript transcription inverse et PCR en temps réel.

Figure 2. Une courbe standard est générée en utilisant une série de dilutions de 2 fois, 10 fois, 50 fois, 250 fois, et 1250 fois l'échantillon initial d'ADNc.

Figure 3. Roche LightCycler Software moléculaire biochimiques montre toute l'information de l'expérience graphique et par le texte. Quantitative en temps réel des parcelles d'amplification par PCR indiquent une augmentation de la fluorescence à partir d'échantillons différents.

Les données Figure 4. Ont été quantifiés à l'aide des logiciels d'analyse LightCycler RelQuant. Habituellement, trois répétitions des échantillons sont analysés en tant que groupe et les échantillons qui donnent des résultats manifestement incompatible sont exclus et les concentrations moyennes et les écarts types de la triple est calculé.
Access restricted. Please log in or start a trial to view this content.
Discussion
Expressions aberrantes de certains miARN ont été constamment trouvé dans des tumeurs de la prostate par rapport au tissu normal 10, et quelques-uns de ces miARN ont été nommés comme nouveaux agents thérapeutiques potentiels contre le cancer de la prostate 11. Par conséquent, les niveaux d'expression aberrants de miARN peuvent être utiles biomarqueurs diagnostiques et / ou pronostique. La méthodologie qPCR en temps réel vous est présenté ici un essai pour la quantification précise ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Ce travail a été financé par le Société canadienne du cancer Institut de recherche, accorder aucune. 019038.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | |
| Réactif TRIzol | Invitrogen | 15596 | |
| Kit miScript Reverse Transcription | Qiagen | 218061 | |
| Les dosages d'amorces miScript | Qiagen | Expérience spécifique | |
| miScript SYBR Green PCR Kit | Qiagen | 218073 | |
| LightCycler 3.5 Real-Time System PCR | Roche | ||
| Light Cycler capillaires | Roche | 04929292001 | |
| NanoDrop 1000 spectrophotomètre | Thermo Scientific | 2538 | |
| Agilent 2100 Bioanalyzer | G2943CA |
Références
- Reinhart, B. J. MicroRNAs in plants. Genes Dev. 16, 1616-1616 (2002).
- Xu, P. The Drosophila microRNA Mir-14 suppresses cell death and is required for normal fat metabolism. Curr. Biol. 13, 790(2003).
- Bueno, M. J., Perez, deC. astro, I,, Malumbres, M. Control of cell proliferation pathways by microRNAs. Cell Cycle. 7, 3143-3143 (2008).
- Friedman, R. C. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19, 92(2009).
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116, 281-281 (2004).
- Croce, C. M. Causes and consequences of microRNA dysregulation in cancer. Nat. Rev. Genet. 10, 704(2009).
- Coppola, V., De, M. R., Bonci, D. MicroRNAs and prostate cancer. Endocr. Relat. Cancer. 17, 1-1 (2010).
- Gordanpour, A. miR-221 Is down-regulated in TMPRSS2:ERG fusion-positive prostate cancer. Anticancer Res. 31, 403-403 (2011).
- Nam, R. K. Expression of the TMPRSS2:ERG fusion gene predicts cancer recurrence after surgery for localised prostate cancer. Br. J. Cancer. 97, 1690(2007).
- Ambs, S. Genomic profiling of microRNA and messenger RNA reveals deregulated microRNA expression in prostate cancer. Cancer Res. 68, 6162(2008).
- Liu, C. The microRNA miR-34a inhibits prostate cancer stem cells and metastasis by directly repressing CD44. Nat. Med. 17, 211(2011).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon