JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
정량 실시간 PCR (qPCR)에 의한 전립선 종양에 MicroRNA 감지
요약
양적 실시간 중합 효소 사슬 반응 (qPCR)는 종양 샘플에서 여러 microRNA의 표현 수준 (miRNA) 분자를 조사하기 위해 신속하고 민감한 방법입니다. 다른 miRNA 분자의 수백이 메서드 표현식을 사용하면, 증폭 계량, 같은 cDNA 템플릿에서 분석할 수 있습니다.
초록
MicroRNAs (miRNAs)는 단일 좌초된, 18-24 염기 길이 비 코딩 RNA 분자입니다. 그들은 개발 1, apoptosis 2 및 세포주기 조절 3를 포함해 거의 모든 세포 과정에 관여하고 있습니다. MiRNAs들은 목표 메신저 RNAs (mRNAs) 5에 바인딩하여 4 인간 유전자의 90 %에 30 %의 표현을 조절하는 것으로 추정된다. miRNAs의 전역 dysregulation는 다양한 질환 및 암 subtypes 6에서보고되었습니다. 그들의 유행과 독특한 구조로 인해,이 작은 분자 생체 치료, 대리인 및 / 또는 타겟의 차세대 될 것 같다.
miRNA 발현을 조사하는 데 사용되는 방법은 SYBR 녹색 나는 염료 기반뿐만 아니라 Taqman-탐침 기반 qPCR을 포함합니다. miRNAs 효과적으로 임상 환경에서 사용할 수있다면 그것은 신선 및 / 또는 보관된 임상 시료에서의 검색이 정확하고 재현성 및 SP가 될 게 급선무다ecific. qPCR 널리 같은 microarray 연구 7과 전체 게놈 분석의 miRNAs의 표현 검증에 사용되고 있습니다. 이 프로토콜에 사용되는 샘플은 임상화된 전립선암에 대한 급진적인 prostatectomy을 받았습니다 환자에서 있었다, 그러나 다른 조직과 세포 라인 스냅인 냉동는 절제술 후 액체 질소에 있던 전립선 표본 들여 대체하실 수 있습니다. 임상 변수와 각각의 환자에 대한 후속 정보는 이후의 분석 8시 수집되었다.
전립선 종양 샘플에 miRNA 수준의 부량. 종양의 qPCR 분석의 주요 단계는 다음과 같습니다 miRNA 특정 primers를 사용하여 총 RNA 추출, cDNA 합성 및 qPCR 제품의 탐지. mRNA, miRNA 및 기타 작은 RNAs를 포함 총 RNA는 TRIzol 시약을 사용하여 표본에서 추출되었다. Qiagen의 miScript 시스템은 cDNA 합성 및 qPCR를 (그림 1)를 수행하는 데 사용되었다. 내생 miRNAs는 PO 없습니다역방향 전송 과정에서 따라서 lyadenylated, 폴리 (A) 중합 효소는 miRNA를 polyadenylates. miRNA는 oligo-DT 및 역방향 Transcriptase를 사용하여 cDNA 합성하는 템플릿으로 사용됩니다. oligo-DT primers의 5 '끝에 보편 태그 시퀀스가 PCR 단계에서 cDNA의 증폭을 용이하게합니다. PCR 제품의 증폭은 SYBR 녹색, 이중 좌초된 DNA에 intercalates 염료에 의해 방출되는 형광의 수준에 의해 감지된다. 유니버설 태그 시퀀스에 바인딩 범용 프라이머와 함께 특정 miRNA의 primers는 특정 miRNA 시퀀스를 증폭합니다.
miScript 프라이머의 Assays는 만 이상의 사람이 특정 miRNAs와 murine 특정 miRNAs 수백 수 있습니다. 상대 부량 방법은 miRNAs의 표현을 수량 여기에 사용되었습니다. 다른 샘플 사이 다양성에 대한 해결하려면 대상 miRNA의 발현 수준은 참조 유전자의 발현 수준으로 정규화됩니다. GE의 선택NE 분석의 상대적인 양을 정함 방식의 중요한 목표의 표현을 정상화하는하는입니다. 일반적으로이 용량에 사용된 참조 유전자의 예로는 그들이 안정적으로 대부분의 샘플을 통해 표현되는 것으로 간주되는대로 작은 RNAs의 RNU6B, RNU44 및 RNU48 있습니다. 이 프로토콜에서는 RNU6B는 참조 유전자로 사용됩니다.
프로토콜
1. 전립선 샘플 모음집
- prostatectomy시 전립선 샘플을 수집합니다. 시료는 해부 랜드마크를 사용하여 지향하고 있습니다. 전립선과 정액 vesicles은 다음과 같이 색칠하고 있습니다 : 오른쪽 녹색, 왼쪽 파란색.
- 전립선의 임의의 가로 midsection은 직장 표면에 수직 이동 액화 질소에 냉동 및 -80 ° C ~ 9시 저장됩니다.
- 표본의 틀었 조각은 (후부, 앞부분은 오른쪽과 왼쪽) 지향, photocopied quadrisected 있습니다. 섹션은 그라 이오 스탯을 사용하여 절단하고 있습니다.
- 섹션은 H & E 물들일과 스테인드 슬라이드와 해당 이미지에 종양이 대 정상적인 영역을 결정하고 윤곽을 그리다하는 병리학자에 의해 검토됩니다. 표시된 영역은 RNA가 후속 단계에서 추출됩니다있는 종양 조직을 추출할 수있는 영역을 표시하기위한 가이드로 사용됩니다.
2. 샘플에서 miRNA 포함하여 총 RNA를, 분리
- 드라이 아이스와 delineated 복사를 참조에 대한 장소 냉동 전립선 샘플, 전립선 종양의 일부를 (50-100 밀리그램 사이) 그만.
- TRIzol 시약 1 ML에서 전립선 종양 조직을 Homogenize. 다음 단계에서 수량은 TRIzol 시약 1 ML의 사용을 기반으로합니다.
참고 : 다음은 소규모 고립 그러나 다른 키트 RNA를 포함하는 총 RNA도 사용할 수 추출 RNA에 대한 TRIzol 시약을 사용 하였다.
- 상온에서 5 분간 균질 견본을 품어.
- 샘플로 클로로포름의 0.2 ML을 추가하고 15 초 동안 풍성히 악수. 실온에서 3 분간 샘플을 부화 후 4에서 15 분 동안 12,000 XG에 원심 분리기 ° C.
- 새로운 튜브로 무색 위 수성 위상을 전송하고, 이소 프로필 알코올 0.5 ML을 추가합니다.상온에서 10 분 샘플을 부화 후 4에서 10 분 동안 12,000 XG에 원심 분리기 ° C.
- RNA를 포함하는 펠렛을 방해하지 않고 뜨는을 자세히 기음. 75 % 에탄올 1 ML과 함께 RNA 펠릿 씻으십시오. 와동 4에 7500의 XG에서 5 분간 원심 분리하여 샘플을 다시 침전 ° C.
- 조심스럽게 RNA 펠릿가 완전히 건조되지 못하게하기 위해 5-10 분 동안 표면에 뜨는 건조 RNA 펠렛을 기음. 펠렛의 크기에 맞는 Nuclease없는 물에 다시 디졸브. NanoDrop 1000 분광 광도계 (260 nm의 및 280 nm의 흡광도에서 측정)를 사용하여 RNA의 농도를 측정합니다.
- 애질런트 Bioanalyzer를 사용하여 RNA 샘플의 품질과 무결성을 확인합니다.
3. RNA의 역방향 스크립트 작성
- RNA의 역방향 전사는 제조 업체의 지침 (Qiagen)에 따라 miScript 역방향 전송 키트를 사용하여 수행되었다. 이 키트를 포함transcriptase와 폴리 (A) 중합 효소를 반대. miScript RT 버퍼는 MG 2를 포함 +, dNTPs, oligo-DT primers, 그리고 무작위 primers.
- 10 PG 및 cDNA 합성하는 RNA 1 μg 사이에 사용하십시오. RNA의 1 이상 μg를 사용하는 경우 해당 볼륨에 선형적으로 반응을 확장할.
- 20 μl의 최종 볼륨에 대한 반응을 가져올 수있는 5 배 miScript RT 버퍼 (4 μl), miScript 역방향 전사 믹스 (1 μl)와 RNase없는 물이 들어있는 마스터 믹스를 준비합니다. 또한 마스터 믹스의 템플릿 RNA가 (최대 1 μg)가 있습니다.
- 37시 60 분 샘플을 품어 ° C ~ 95 ° C.에서 5 분간 배양하여 바로 다음에 이 단계 블록, 또는 물에 목욕을 가열, PCR 기계에서 수행할 수 있습니다. Thermocyclers는 가장 적합하고 정확한 방법입니다. 단기 얼음 cDNA를 저장하고, -20 ° C에서 장기 보관을위한.
4. 표준 곡선을 생성
- exper 이전대상 miRNAs와 iment, 표준 곡선들은 건너 포인트 (CP) (그림 2)에 대해 알려진 농도의 cDNAs를 사용하여 생성됩니다.
- 2 배, 10 배, 50 배, 250 배, 그리고 관심의 유전자의 실질적인 표현을하는 것으로 알려져 샘플의 1,250 배 원래 cDNA의 dilutions 일련의를 준비합니다.
- 5 항 "miRNA의 탐지를위한 실시간 PCR"에 명시된 바와 같이 cDNA 시리얼 dilutions없는 정적 40x 희석에있는 것을 수정하여, PCR을 실행합니다.
- 당신의 표준 곡선을 생성하는 RelQuant 소프트웨어 (로슈)를 사용하여 분석을 수행합니다.
참고 : 새로운 표준 곡선은 관심있는 각각의 유전자가 생성되어야합니다.
5. miRNA의 탐지를위한 실시간 PCR
- miRNAs를위한 실시간 PCR은 제조 업체의 지침 (Qiagen)에 따라 miScript SYBR 녹색 PCR Kit 및 miScript의 프라이머 분석을 사용하여 수행되었다. 포함하는 마스터 믹스를 준비2X QuantiTect SYBR 녹색 PCR 마스터 믹스, 10X miScript 유니버셜 프라이머, 10X miScript 프라이머 분석 및 RNase없는 물. 20 μl 볼륨 반응을위한 마스터 믹스를 준비합니다.
- 프라이머 분석은 관심 miRNA에만 적용됩니다. reconstitute 10X miScript의 프라이머 분석하려면, 간단히 병을 원심 분리기와 550 μl TE 버퍼, 산도 8.0을 추가합니다. -20 ° C.의 소용돌이 섞어 짧게 유리병, 작은 볼륨으로 나누어지는의 primers, 및 매장 두 primers이 필요합니다. 대상 유전자와 기준 유전자에 대한 primers RNU6B은 레퍼런스 유전자 usedas입니다.
- -20 ° C.의 cDNA 40x 및 매장 추가 aliquots를 희석
- cDNA는 PCR을위한 템플릿 역할을합니다. 40x 희석 cDNA 2 μl를 사용하여 20 μl 가벼운 자전거 타는 사람 모세 혈관 (로슈)로 분배.
- 각각의 모세관에 마스터 믹스 18 μl를 추가하고, 모세관 어댑터를 사용하여 원심 분리기.
- 같은 LightCycler 세와 같은 모세관 기반의 리얼 타임 자전거 타는 사람에 모세 혈관을 놓습니다.32 모세 회전 목마 형식 5 리얼 타임 PCR 시스템.
- 다음과 같이 PCR 사이클 프로그램을 실행합니다 :
2X QuantiTect SYBR 녹색 PCR 마스터 믹스에 HotStarTaq 효소를 활성화하기 위해, 95의 사전 품어 15 분 동안 ° C에서.
50주기를 얹는 :
변성, 15 초, 94 ° C;
어닐링, 30의, 55 ° C;
확장, 30 초, 70 ° C. - 캘리 브 레이터로 샘플을 선택하고 1의 표준 목표 금액을 설정합니다. 캘리 브 레이터에 대한 모든 다른 샘플에 miRNA의 상대적 표현을 비교합니다.
참고 : 공부 내에서 동일한 보정 샘플 결과의 일관성을 유지하는 데 사용해야합니다.
6. 데이터 분석
- PCR 반응을위한 증폭 곡선 분자 Biochemicals의 LightCycler 소프트웨어 버전 3.5 (로슈)의 그래픽과 수치로 표시됩니다. , "부량"탭에 반응을 수량ND는 텍스트 파일로 데이터를 내보낼 수 있습니다.
- 부량 결과를 생성하는 RelQuant 분석 소프트웨어 (로슈)로 데이터를 가져옵니다. 대상 유전자 참조 유전자와 표준 곡선 데이터를위한 별도의 파일을 가져옵니다.
- 대상 및 참조 유전자 모두 캘리 브 레이터의 위치를 지정합니다. 또한 샘플의 위치를 지정합니다. 데이터는 캘리 브 레이터의 기준 비율을 대상으로 나누어 서로 다른 샘플 비율을 참조하는 대상으로 표시됩니다. 이전에 특정 miRNA와 가사 유전자에 대해 생성된 표준 곡선은 미지 농도의 miRNA 목표에 대한 양적 데이터를 바탕위한 참조 표준으로 사용됩니다.
- 샘플 그룹으로 분석 농도와 세중의 표준 편차를 계산 의미하고 세 복제합니다. triplicates 중 하나가 집합의 나머지 부분과 일관성이있다면, 그것은 프로그램에 의해 제외됩니다.
7. 대표 결과
전립선 샘플에 qPCR 분석의 예제는 그림 3에 표시됩니다. 결과뿐만 아니라 그래픽, 수치 표시됩니다. 대상 유전자 미르-98, 표현 사이클 25 정도 지연될 증폭을 보여주면서 레퍼런스 유전자, U6의 표현 수준을 보여주는 그래프, 대한 사이클 20 지수 증폭을 시작합니다. 본 실험에서 데이터를 텍스트 파일로 내보낸 및 RelQuant 분석 소프트웨어에 의해 분석되었다. 캘리 브 레이터 및 샘플을 포함하는 모세 혈관의 위치가 지정됩니다. 그림 4는 캘리 브 레이터가 1로 설정하고, 캘리 브 레이터에 상대적으로 다른 샘플을 표현하는 방법을 보여줍니다.
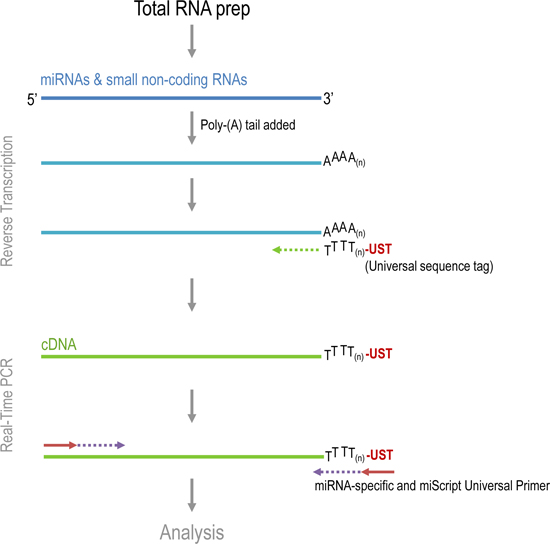
그림 1. miScript 리버스 녹음 및 실시간 PCR의 여러 단계.

그림 2. 표준 곡선은 2 배, 10 배, 50 배, 250 배, 그리고 1250 배 원래 cDNA 샘플의 dilutions 일련를 사용하여 생성됩니다.

그림 3. 로슈 분자 Biochemicals의 LightCycler 소프트웨어는 그래픽의 실험과 텍스트에 의한 전체 정보를 보여줍니다. 정량 실시간 PCR의 증폭 플롯 다른 샘플에서 형광 증가 보여줍니다.

그림 4. 데이터 RelQuant LightCycler 분석 소프트웨어를 사용하여 계량했다. 보통 세 그룹과 분명히 일관성없는 결과는 제외됩니다 생산 및 농도와 세중의 표준 편차를 계산 뜻 샘플로 분석되는 샘플 복제합니다.
Access restricted. Please log in or start a trial to view this content.
토론
일부 miRNAs의 탈선 표현은 일관되게 정상 조직 10, 그리고 이러한 miRNAs 중 일부는 전립선 암의 11에 대한 잠재적인 새로운 치료 대리인으로 명명되었습니다에 비해 전립선 종양에서 발견되었습니다. 따라서 miRNAs의 탈선 표현 수준은 유용한 진단 및 / 또는 전조 생체 자료가 될 수 있습니다. 여기서 제시 실시간 qPCR 방법론은 전립선 종양 조직에서 miRNA 수준의 정확한 양을 정함 ?...
Access restricted. Please log in or start a trial to view this content.
공개
관심의 어떠한 충돌 선언 없습니다.
감사의 말
이 작품은 캐나다 암 학회 연구원 추진하는 사업, 아무도 부여 없습니다. 019,038.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 시약의 이름 | 회사 | 카탈로그 번호 | |
| TRIzol 시약 | Invitrogen | 15,596 | |
| miScript 역방향 전송 키트 | Qiagen | 218,061 | |
| miScript 프라이머의 Assays | Qiagen | 구체적인 실험 | |
| miScript SYBR 녹색 PCR 키트 | Qiagen | 218,073 | |
| LightCycler 3.5 리얼 타임 PCR 시스템 | 로슈 | ||
| 라이트 자전거 타는 사람 모세 혈관 | 로슈 | 04929292001 | |
| NanoDrop 1000 분광 광도계 | 써모 과학 | 2538 | |
| 애질런트 2100 Bioanalyzer | G2943CA |
참고문헌
- Reinhart, B. J. MicroRNAs in plants. Genes Dev. 16, 1616-1616 (2002).
- Xu, P. The Drosophila microRNA Mir-14 suppresses cell death and is required for normal fat metabolism. Curr. Biol. 13, 790(2003).
- Bueno, M. J., Perez, deC. astro, I,, Malumbres, M. Control of cell proliferation pathways by microRNAs. Cell Cycle. 7, 3143-3143 (2008).
- Friedman, R. C. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19, 92(2009).
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116, 281-281 (2004).
- Croce, C. M. Causes and consequences of microRNA dysregulation in cancer. Nat. Rev. Genet. 10, 704(2009).
- Coppola, V., De, M. R., Bonci, D. MicroRNAs and prostate cancer. Endocr. Relat. Cancer. 17, 1-1 (2010).
- Gordanpour, A. miR-221 Is down-regulated in TMPRSS2:ERG fusion-positive prostate cancer. Anticancer Res. 31, 403-403 (2011).
- Nam, R. K. Expression of the TMPRSS2:ERG fusion gene predicts cancer recurrence after surgery for localised prostate cancer. Br. J. Cancer. 97, 1690(2007).
- Ambs, S. Genomic profiling of microRNA and messenger RNA reveals deregulated microRNA expression in prostate cancer. Cancer Res. 68, 6162(2008).
- Liu, C. The microRNA miR-34a inhibits prostate cancer stem cells and metastasis by directly repressing CD44. Nat. Med. 17, 211(2011).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유