Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Photopatterning protéines et des cellules dans une solution aqueuse Environnement Utilisation de TiO
Dans cet article
Résumé
We describe a protocol for modifying cell affinity of a scaffold surface in aqueous environment. The method takes advantage of titanium dioxide photocatalysis to decompose organic film in the photo-irradiated region. We show that it can be used to create microdomains of scaffolding proteins, both ex situ and in situ.
Résumé
Les contaminants organiques adsorbés à la surface du dioxyde de titane (TiO 2) peuvent être décomposés par photocatalyse sous rayonnement ultraviolet (UV). Nous décrivons ici un nouveau protocole employant la photocatalyse du TiO 2 à modifier localement affinité des cellules de la surface du substrat. Pour cette expérience, un film mince de TiO 2 est revêtu par pulvérisation sur une lamelle de verre, et la surface de TiO 2 a ensuite été modifié par une monocouche organosilane dérivé de l'octadécyltrichlorosilane (OTS), qui inhibe l'adhérence cellulaire. L'échantillon a été immergé dans un milieu de culture cellulaire, et concentre la lumière UV a été irradié à une région de forme octogonale. Quand un neuronales cellules PC12 lignée de cellules ont été étalées sur l'échantillon, les cellules adhèrent seulement sur la zone irradiée aux UV. On montre en outre que cette modification de surface peut également être réalisée in situ, autrement dit, même lorsque les cellules sont en croissance sur le substrat. Modification appropriée de la surface nécessaire une matrice extracellulaire protein collagène d'être présent dans le milieu au moment de l'irradiation UV. La technique présentée ici peut éventuellement être utilisé dans plusieurs types de cellules pour la construction de structuration des systèmes de co-culture ou de manipuler arbitrairement cellules dans la culture.
Introduction
Semiconductor processus de lithographie et de ses dérivés tels que la photolithographie - 1,2, la lithographie par faisceau d'électrons 3-6 et microcontact impression 7-10 - sont devenus un outil créé en biologie cellulaire pour cultiver des cellules vivantes dans une position définie et la géométrie. Procédé de formation de motifs repose sur l'utilisation de substrats micro-usinés, comprenant des micro-île de revêtement permissive de cellules dans un arrière-plan non permissive. Un tel substrat sert de matrice à motif cellules. Ces technologies nous ont fourni les nouvelles méthodes pour modifier des cellules et leur fonction à un niveau mono et multi-cellulaire, pour extraire les propriétés intrinsèques des cellules, et d'augmenter le débit de base de cellules criblage de médicaments 11.
Le degré de liberté dans un patron de cellules augmenterait considérablement si la géométrie du motif de modèle pourrait être modifié in situ, à savoir, tandis que les cellules sont cultivées sur des sta tête. Les procédés classiques de formation de motif ne peut pas être directement appliquées ici, étant donné qu'ils traitent les échantillons dans une atmosphère ou sous vide. Par conséquent diverses nouvelles techniques de modification de surface ont été proposées, qui sont basées, par exemple, sur des composés photo-réactifs 12,13 ou ablation laser 5,14, juste pour en nommer quelques-uns. Les méthodes proposées ont été bien examiné par Robertus et al. 15, et plus récemment par Choi et al. 16 et 17 par Nakanishi.
Ici, dans cet article, nous décrivons un nouveau protocole de modification in situ surface, qui tire parti de la dégradation photocatalytique de molécules organiques sur un dioxyde de titane (TiO 2) de surface 18,19. Dans ce procédé, un film de TiO 2 est insérée entre le substrat de verre et le film organique qui assure l'interface des cellules, et le film organique est décomposé in situ en irradiant localement ultraviolet (UV)la lumière pour une région d'intérêt (λ <388 nm). Nous montrons que le nouveau protocole peut être utilisé pour créer micropatterns de protéines de la matrice extracellulaire et des cellules vivantes ex situ et in situ. TiO 2 est biocompatible, chimiquement stable, et optiquement transparents, caractéristiques de ce qui le rend sympathique à introduire dans des expériences de culture cellulaire. Ce protocole fournit une alternative de matériaux à base scientifique pour modifier les échafaudages de culture cellulaire en milieu de culture cellulaire.
Protocole
1. Préparation de TiO 2 revêtues de verre Coverslip
- Nombre les lamelles à l'aide d'une pointe à tracer de diamant. Cela permet non seulement de garder une trace de chaque lamelles, mais aussi pour veiller à ce que le bon côté de l'échantillon est orientée vers le haut. Nettoyer les lamelles couvre-objet, d'abord sous exécutant ddH 2 O, puis en les immergeant dans une solution de piranha (H 2 SO 4: H 2 O 2 = 4: 1). Après 10 minutes, rincez soigneusement les lamelles, 8 fois en ddH 2 O. Sécher les lamelles sous N2 flux.
- Ensemble de TiO 2 dans la cible de radiofréquence (RF) de système de pulvérisation. Fixez les lamelles sur un support de l'équipement de pulvérisation à l'aide d'un ruban de polyimide de l'échantillon. Placez le porte-échantillon dans la chambre de pulvérisation. Évacuer la chambre jusqu'à ce que la pression atteint 2,0 × 10 -4 Pa.
- Introduire du gaz Ar dans la chambre et régler la pression de dépôt à 4,0 mTorr. Tout en gardant l'obturateur fermé, augmenter progressivementla puissance RF à 70 W.
- Ouvrir le volet et par pulvérisation cathodique pendant 15 min pour obtenir un film d'une épaisseur de 120 à 150 nm (Figure 1: Etape A). Taux de croissance du film doit être dérivé pour chaque machine.
2. Revêtement de surface avec Cell-répulsif Film
- Rendre hydrophile la surface de TiO 2 par traitement de l'échantillon avec du plasma O 2, en suivant les instructions fournies par le fabricant du réacteur à plasma. Nous traitons l'échantillon pendant 5 min à 200 W avec O 2 débit de 100 sccm. Immerger l'échantillon dans le trou DDH 2 O et de confirmer que la surface est superhydrophile. Séchez soigneusement la surface sous le N 2 flux.
- Préparez 1 mM octadécyltrichlorosilane (OTS) solution par addition de 39,6 OTS pi à 100 ml de toluène. Immerger l'échantillon dans la solution pendant 1 heure à température ambiante. Effectuer cette étape l'intérieur d'un sac à gants 2 -filled N (Figure 1: Étape B).
- Pour éliminer les molécules physisorbées, sonicate l'échantillon dans le toluène, l'acétone, l'éthanol, et ddH 2 O pendant 5 minutes chacune, dans cet ordre. Rincer l'échantillon quatre fois en frais ddH 2 O et sécher la surface sous le N 2 flux. La surface doit être hydrophobe avec un angle de 100-110 ° de contact.
3. Ex-Situ structuration de surface
- Travailler dans une hotte à flux laminaire, tirer plusieurs traces de rayures avec un abonné de diamant sur la surface. Les marques aident à garder la trace des régions traitées et également mettre au point des microscopes. Stériliser le TiO 2 revêtu OTS en plongeant l'échantillon dans de l'éthanol 70% pendant 5 min. Puis rincer l'échantillon deux fois dans stérilisée ddH 2 O.
- Placer l'échantillon dans un plat de 35 mm, et ajouter 2 ml de milieu de croissance de PC12 (voir l'étape 4.2). Incuber pendant plus de 3 heures dans un incubateur à CO 2 (37 ° C). Cette procédure est destinée à laisser sérumalbumines absorbent sur la surface. Albumines adsorbées inhibent l'adsorption ultérieure d'autres proteins et des cellules (Figure 1: l'étape c).
- En attendant, mettre en place le microscope inversé à fluorescence.
- Allumez la lampe à arc, insérez le cube de filtre UV, et définir l'objectif de 20X.
- Mesurer l'intensité lumineuse I (W cm -2) au moyen d'un compteur d'intensité UV, et de calculer la durée d'irradiation t (en secondes) pour une dose de d (en cm -2 J) en tant que: t = d / I.
Par exemple, pour irradier à une dose de 200 J cm -2 utilisant une source lumineuse de 600 mW cm -2, 333 secondes d'irradiation est nécessaire. - Utilisez un micromètre et fermer le diaphragme de champ pour définir la taille de la région à irradier, par exemple, 200 um.
- Après l'incubation de trois heures, de compléter le milieu avec 200 ul de 3,0 mg ml-1 de type IV collagène (Col-IV; concentration finale de 300 ug ml-1).
- Transférer le plat de 35 mm de la platine du microscope. Trouver le scratch marche, focaliser le microscope sur la surface de l'échantillon, et irradier la lumière UV à une dose de 200 J cm -2 (Figure 1: Etape D). La zone d'irradiation UV peut être modifiée, soit en ajustant l'ouverture du diaphragme de champ ou par le remplacement du diaphragme de champ avec un masque métallique de géométrie d'arbitrage.
- Remplacez le milieu avec un milieu de croissance frais (sans Col-IV), et placer l'échantillon dans l'incubateur.
4. Culture Cellulaire
- Culture de routine de cellules PC12 est effectuée sur une boîte en matière plastique revêtue de collagène.
- Pour revêtir un plat de culture de tissu de 60 mm avec Col-IV, mouiller d'abord la surface avec le trou DDH 2 O et aspirer tous ddH 2 O.
- Préparer 300 ng ml -1 Col-IV en diluant la solution initiale (3 mg ml -1) 10x avec ddH 2 O.
- Ajouter 200 pi de 300 pg ml -1 Col-IV par 60 mm plat. Laissez la solution se propager à travers la surface.
- À sec til plat dans une hotte à flux laminaire pendant environ 1 heure.
- Rincer la surface deux fois avec 4 ml de tampon phosphate salin de Dulbecco la (D-PBS). Lorsqu'ils ne sont pas immédiatement d'utiliser le plat enduit, rincez le plat une fois avec 4 ml de ddH 2 O. Après aspiration ddH 2 O, stocker le plat dans l'incubateur. Essayez de ne pas le garder stocké pendant plus de deux semaines.
- Les cellules PC12 sont cultivées dans un milieu de croissance qui se compose de: moyen (faible taux de glucose) de Eagle modifié par Dulbecco + sérum solution à 1% de pénicilline-streptomycine + 10% de sérum de veau foetal 5% + cheval. Incuber les cellules dans un incubateur à CO 2 (37 ° C, 5% CO 2), et remplacer la moitié du milieu tous les deux jours. Passage cellules avant qu'elles atteignent la confluence.
- Aspirer le milieu de croissance, et ajouter 2 ml de PBS + (D-PBS + 10 mg ml -1 d'albumine de sérum bovin (BSA) + 10 mM d'EDTA) préchauffé à 37 ° C. Incuber pendant 5 min à 37 ° C. Tapoter doucement le plat de détacher tous les CELLS.
- Recueillir les cellules dans un tube conique de 15 ml. Rincer le plat avec 3 ml de frais D-PBS, préchauffée à 37 ° C.
- Centrifuger le tube à 150 g pendant 4 min.
- Aspirer le surnageant et ajouter 1 ml de milieu de croissance.
- Pour la culture de routine, diviser les cellules 1: 3 à 1: 5.
- Pour étaler les cellules sur l'échantillon modifié-UV OTS / TiO 2, comptez densité cellulaire et ajouter 3,0 × 10 5 cellules dans une boîte de 35 mm (Figure 1: Étape E). Incuber le plat au incubateur humidifié (37 ° C, 5% CO 2) pendant 1-2 jours. Les deux facteurs de croissance et naïf nerf (NGF) cellules PC12 -differentiated peuvent être utilisées pour les expériences de mise en forme. Pour la différenciation NGF, 100 ng ml -1 de 7S-NGF est ajouté à la croissance moyenne plusieurs jours avant l'étalement des cellules sur l'échantillon. NGF-différenciation semble augmenter le adhesibility des cellules PC12 et facilite la manipulation, en particulier dans le modelage in situ eXperiments.
5. In-Situ structuration de surface
- Après 1-2 jours de culture sur la surface modifiée ex-situ, confirment que les cellules attachent croissant et seulement sur la région irradiée aux UV. Mettre en place le microscope, comme décrit dans l'étape 3.4.
- Transférer l'échantillon dans une nouvelle boîte de culture tissulaire de 35 mm contenant du milieu de croissance, 100 ng ml-1 NGF (dans le cas de l'utilisation de cellules différenciées NGF), et 100 ug ml-1 Col-IV.
- Placez le plat sur la platine du microscope. Trouver la position appropriée, et irradier la lumière UV à une dose de 200 J cm -2 (Figure 1: Etape F, G). La région permissive nouvellement créé est indiqué par un astérisque dans la figure 1: Etape G.
- Essayer de terminer le traitement d'un seul échantillon dans les 30 min. Après irradiation, transférer l'échantillon de nouveau dans le milieu sans Col-IV.
- Soyez extrêmement prudent lors de la réalisation du plat avec cultured cellules et le transfert de l'échantillon de plat à plat pour éviter le détachement des cellules motifs.
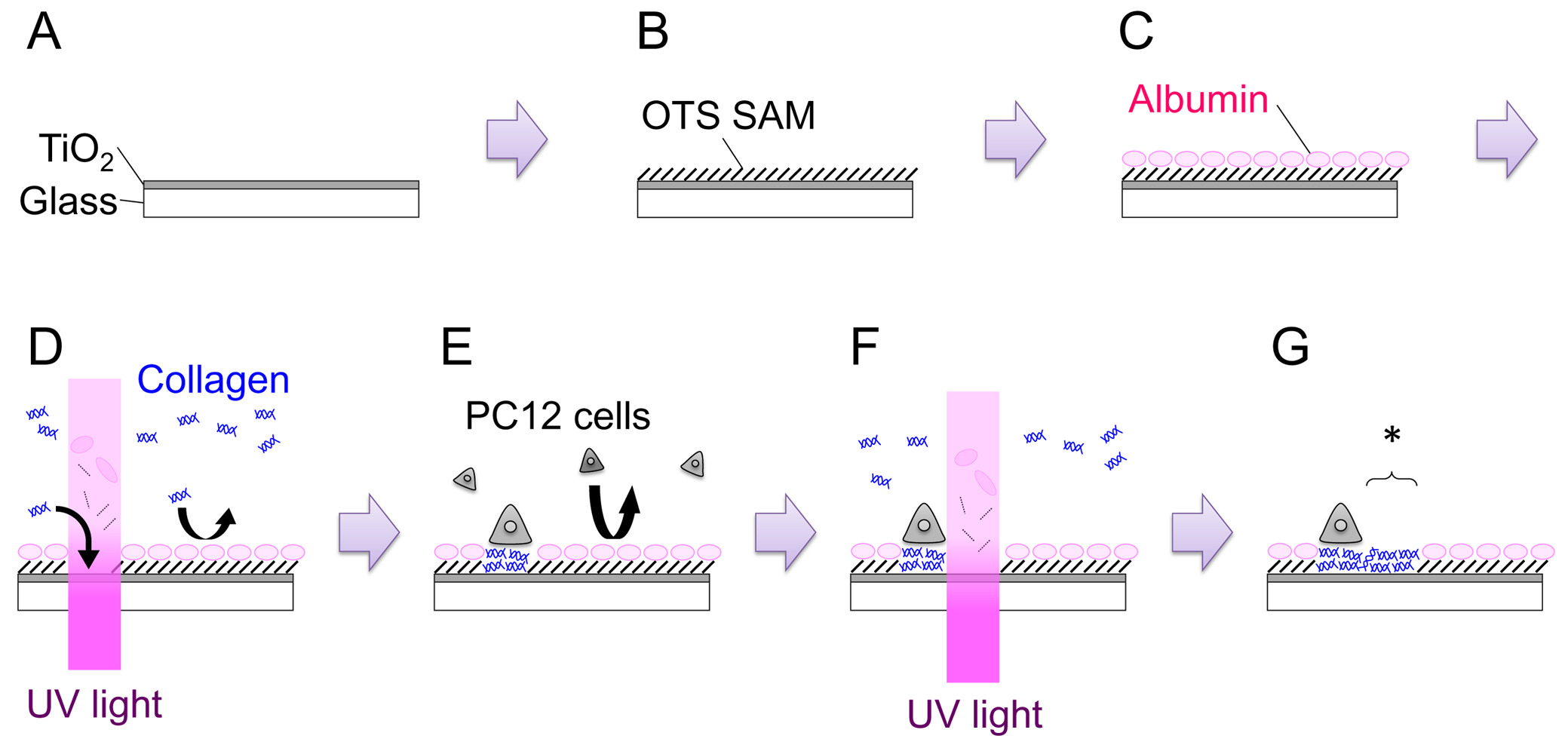
Figure 1. Représentation schématique de l'ensemble du processus. Voir le texte pour plus de détails. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Résultats
La figure 2A montre une image au microscope électronique à balayage en coupe transversale (SEM) de la pellicule de TiO 2 déposée par pulvérisation cathodique. De l'observation, l'épaisseur du film a été estimée à environ 150 nm. Notable ici est la planéité de la couche de TiO 2 déposée. Une analyse plus poussée par microscopie à force atomique (AFM) a révélé que la rugosité racine carrée moyenne (RMS) de la surface était de 0,2 nm (figure
Discussion
Dans notre protocole actuel, TiO 2 film a été formé par RF-pulvérisation magnétron. Nous privilégions cette méthode de dépôt, car il nous permet de préparer de façon reproductible un film photocatalytique de TiO 2 avec une rugosité sous-nm. Bien que les processus de dépôt par pulvérisation sont familiers aux spécialistes des matériaux et des ingénieurs en électronique, il peut ne pas être tout à fait accessible aux biologistes. Dans ce cas, TiO 2 film spin-enduit ser...
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
Authors thank Mr. Kotaro Okubo for the kind assistance with SEM imaging. This work was supported by the Japan Society for the Promotion of Science Grant-in-Aid for Basic Research (B) (20310069), Grant-in-Aid for Research Activity Start-up (25880021), and by research grants from the Kurata Memorial Hitachi Science and Technology Foundation and the Nippon Sheet Glass Foundation for Materials Science and Engineering.
matériels
| Name | Company | Catalog Number | Comments |
| Glass coverslip | Warner Instruments | CS-15R15 | 15 mm diameter, #1.5 thickness |
| Diamond scriber | Ogura Jewel Industry | D-Point Pen | |
| RF sputtering system | ANELVA | SPC350 | |
| TiO2 sputtering target | Kojundo Chemical Lab | Titanium (IV) oxide, target | Purity, 99.9% |
| Plasma reactor | Yamato | PR301 | |
| n-octadecyltrichlorosilane (OTS) | Aldrich | 104817 | |
| Toluene | Wako | 204-01866 | |
| Tissue-culture dish (35 mm) | Greiner | 627160 | |
| Tissue-culture dish (60 mm) | BD Falcon | 353002 | |
| Type-IV collagen | Nitta Gelatin | Cellmatrix Type IV | |
| D-PBS | Gibco | 14190-144 | |
| Dulbecco's modified Eagle's medium (DMEM) | Gibco | 11885-084 | |
| Fetal bovine serum | Gibco | 12483-020 | Heat-inactivate and pass through a 0.22 μm filter before use |
| Horse serum | Gibco | 26050-088 | Pass through a 0.22 μm filter before use |
| Penicillin-streptomycin (100x) | Nacalai tesque | 26253-84 | |
| 7S nerve growth factor (NGF) | Alomone Labs | N-130 | |
| Bovine serum albumin (BSA) | Sigma | A2153 | |
| EDTA | Dojindo | N001 | Stock solution in 0.5 M |
| TiO2 nanoparticle | Tayca | TKD-701 |
Références
- Hughes, M. A., Brennan, P. M., Bunting, A. S., Shipston, M. J., Murray, A. F. Cell Patterning on Photolithographically Defined Parylene-C: SiO2 Substrates. J. Vis. Exp. (85), e50929 (2014).
- Kleinfeld, D., Kahler, K. H., Hockberger, P. E. Controlled Outgrowth of Dissociated Neurons on Patterned Substrates. J. Neurosci. 8, 4098-4120 (1988).
- Pensen, D., Heinz, W. F., Werbin, J. L., Hoh, J. H., Haviland, D. B. Electron Beam Patterning of Fibronectin Nanodots that Support Focal Adhesion Formation. Soft Matter. 3, 1280-1284 (2007).
- Tanii, T., et al. Application of Organosilane Monolayer Template to Quantitative Evaluation of Cancer Cell Adhesive Ability. Jpn. J. Appl. Phys. 50, 06GL01 (2011).
- Yamamoto, H., et al. In-Situ Guidance of Individual Neuronal Processes by Wet Femtosecond Laser Processing of Self-Assembled Monolayers. Appl. Phys. Lett. 99, 163701-1610 (2011).
- Yamamoto, H., et al. Differential Neurite Outgrowth is Required for Axon Specification by Cultured Hippocampal Neurons. J. Neurochem. 123, 904-910 (2012).
- Shen, K., Qi, J., Kam, L. C. Microcontact Printing of Proteins for Cell Biology. J. Vis. Exp. (22), e1065 (2008).
- Johnson, D. M., LaFranzo, N. A., Maurer, J. A. Creating Two-Dimensional Patterned Substrates for Protein and Cell Confinement. J. Vis. Exp. (55), e3164 (2011).
- Singhvi, R., et al. Engineering Cell Shape and Function. Science. 264, 696-698 (1126).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric Control of Cell Life and Death. Science. 276, 1425-1428 (1997).
- Degot, S., et al. Improved Visualization and Quantitative Analysis of Drug Effects using Micropatterned Cells. J. Vis. Exp. (46), e2514 (2010).
- Nakanishi, J., et al. Photoactivation of a Substrate for Cell Adhesion under Standard Fluorescence Microscopes. J. Am. Chem. Soc. 126, 16314-16315 (2004).
- Kim, M., et al. Addressable Micropatterning of Multiple Proteins and Cells by Microscope Projection Photolithography Based on a Protein Friendly Photoresist. Langmuir. 26, 12112-12118 (2010).
- Deka, G., Okano, K., Kao, F. -. J. Dynamic Photopatterning of Cells In Situ by Q-Switched Neodymium-Doped Yttrium Ortho-Vanadate. Laser. J. Biomed. Opt. 19, 011012 (2014).
- Robertus, J., Browne, W. R., Feringa, B. L. Dynamic Control over Cell Adhesive Properties using Molecular-Based Surface Engineering Strategies. Chem. Soc. Rev. 39, 354-378 (2010).
- Choi, I., Yeo, W. -. S. Self-Assembled Monolayers with Dynamicity Stemming from (Bio)chemical Conversions: From Construction to Application. ChemPhysChem. 14, 55-69 (2013).
- Nakanishi, J. Switchable Substrates for Analyzing and Engineering Cellular Functions. Chem. Asian J. 9, 406-417 (2014).
- Yamamoto, H., et al. In Situ Modification of Cell-Culture Scaffolds by Photocatalytic Decomposition of Organosilane Monolayers. Biofabrication. 6, 035021 (2014).
- Sekine, K., Yamamoto, H., Kono, S., Ikeda, T., Kuroda, A., Tanii, T. Surface Modification of Cell Scaffold in Aqueous Solution using TiO2 Photocatalysis and Linker Protein L2 for Patterning Primary Neurons. e-J. Surf. Sci. Nanotech. 13, 213-218 (2015).
- Arima, Y., Iwata, H. Effects of Surface Functional Groups on Protein Adsorption and Subsequent Cell Adhesion using Self-Assembled Monolayers. J. Mater. Chem. 17, 4079-4087 (2007).
- Fujishima, A., Zhang, X., Tryk, D. A. TiO2 Photocatalysis and Related Surface Phenomena. Surf. Sci. Rep. 63, 515-582 (2008).
- Sigal, G. B., Mrksich, M., Whitesides, G. M. Effect of Surface Wettability on the Adsorption of Proteins and Detergents. J. Am. Chem. Soc. 120, 3464-3473 (1998).
- Zhang, X., et al. A Transparent and Photo-Patternable Superhydrophobic Film. Chem. Commun. 2007, 4949-4951 (1039).
- Kaech, S., Banker, G. Culturing Hippocampal Neurons. Nat. Protoc. 1, 2406-2415 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon