Method Article
Multiplexage automatisé de l’échantillon à l’aide de l’étiquetage isotopique combiné des précurseurs et du marquage isobarique (cPILOT)
Dans cet article
Résumé
L’étiquetage isotopique combiné des précurseurs et le marquage isobarique (cPILOT) est une stratégie améliorée de multiplexage d’échantillons qui est capable d’augmenter le nombre d’échantillons qui peuvent être analysés simultanément avec les étiquettes isobariques disponibles. L’incorporation d’une plate-forme robotique a considérablement augmenté le débit expérimental, la reproductibilité et la précision quantitative.
Résumé
Nous avons introduit un flux de travail protéomique quantitatif à haut débit, un étiquetage isotopique combiné précurseur et un marquage isobarique (cPILOT) capable de multiplexer jusqu’à 22 ou 24 échantillons avec des étiquettes de masse tandem ou n isobarique, des étiquettes isobariques de leucine de N-dimethyl, respectivement, dans une seule expérience. Ce multiplexage d’échantillons amélioré réduit considérablement les temps d’acquisition de spectrométrie de masse et augmente l’utilité des reagents isobariques commerciaux coûteux. Toutefois, le processus manuel des étapes de manipulation et de pipetage de l’échantillon dans la stratégie peut être laborieux, long, et introduire la perte d’échantillon et l’erreur quantitative. Ces limitations peuvent être surmontées par l’incorporation de l’automatisation. Ici, nous avons transféré le protocole cPILOT manuel à un dispositif automatisé de manutention des liquides qui peut préparer de grands nombres d’échantillons (c.-à-d. 96 échantillons) en parallèle. Dans l’ensemble, l’automatisation augmente la faisabilité et la reproductibilité du cPILOT et permet une large utilisation par d’autres chercheurs avec des dispositifs d’automatisation comparables.
Introduction
La protéomique basée sur la spectrométrie de masse (SP) est un outil de recherche indispensable pour identifier les biomarqueurs spécifiques à la maladie, comprendre la progression de la maladie et créer des pistes de développement thérapeutique. Ceci peut être réalisé à partir d’une gamme d’échantillons cliniques liés à la maladie tels que le sérum sanguin/plasma, les fluides proximaux, et lestissus 1,2. La découverte et la validation des biomarqueurs protéomiques ont récemment été prises en considération en raison de la puissance des stratégies de multiplexagede l’échantillon 3,4. Le multiplexage d’échantillon est une technique qui permet la comparaison et la quantification simultanées de deux conditions d’échantillon ou plus dans une seule injectionde SP 5,6. Le multiplexage de l’échantillon est réalisé en codant à barres des peptides ou des protéines à partir de plusieurs échantillons avec des étiquettes chimiques, enzymatiques ou métaboliques et en obtenant de l’information sur la SP à partir de tous les échantillons dans le cas d’une seule expérience de SP ou de SP/SP. Parmi les étiquettes isobariques disponibles figurent des reagents de marquage isobarique (iTRAQ), des étiquettes de masse en tandem commercial (TMT) et des réaccéléments isobariques synthétisés maison N, N-dimethyl leucine (DiLeu) avec des capacités jusqu’à 16-plex7 et 21-plex8,respectivement.
L’étiquetage isotopique combiné des précurseurs et le marquage isobarique (cPILOT) est une technologie améliorée de multiplexage d’échantillons. cPILOT combine l’étiquetage isotopique du peptide N-termini avec des isotopes légers [−(CH3)2]et lourds [−(13C2H3)2] isotopes à faible pH (∼2,5), ce qui maintient les résidus de lysine disponibles pour l’étiquetage isobarique élevé subséquent (8,5) à l’aide de TMT, DiLeu, ou iTRAQ marquage3,9,10,11,12,13,14. Le système d’étiquetage double de la stratégie cPILOT est représenté dans la figure supplémentaire 1 avec deux échantillons utilisant un exemple de peptide. La précision et la précision de la quantification basée sur le TMT au niveau MS2 peuvent être compromises en raison de la présence d’ions co-isolés et co fragmentés appelé l’effet d’interférence15. Cette limitation des ratios d’ion de reporter inexacts peut être surmontée à l’aide de spectromètres de masse Tribrid Orbitrap. Par exemple, l’effet d’interférence peut être surmonté en isolant un pic dans une paire dimethylated au niveau MS1 dans le spectromètre de masse, en soumettant le pic léger ou lourd à la fragmentation de MS2 dans le piège à ions linéaire, puis en soumettant le fragment ms2 le plus intense pour HCD-MS3 pour obtenir des informations quantitatives. Afin d’augmenter les chances de sélection des peptides sans lysine amines disponibles pour générer des ions reporter, une acquisition sélective ms3 basée sur le fragment y-1 peut également être utilisé et est une approche qui peut entraîner un pourcentage plus élevé de peptides quantifiables avec cPILOT9. La combinaison de l’étiquetage léger et lourd augmente les capacités de multiplexage de l’échantillon par un facteur de 2x à celui obtenu avec les étiquettes isobariques individuelles. Nous avons récemment utilisé cPILOT pour combiner jusqu’à 24 échantillons en une seule expérience avec les reagents DiLeu16. En outre, cPILOT a été utilisé pour étudier les modifications oxydatives post-translationnelles14, y compris la nitrationprotéique 17, d’autres protéomesmondiaux 9, et a démontré des applications à travers plusieurs échantillons de tissus dans un modèle de souris de la maladie d’Alzheimer11.
La préparation robuste de l’échantillon est une étape critique d’une expérience cPILOT et peut prendre beaucoup de temps, être laborieuse et étendue. Le multiplexage amélioré d’échantillon exige le pipetting étendu et le personnel de laboratoire hautement qualifié, et il y a plusieurs facteurs qui peuvent fortement influencer la reproductibilité de l’expérience. Par exemple, une manipulation minutieuse des échantillons est nécessaire pour assurer des temps de réaction similaires pour tous les échantillons et pour maintenir un pH tampon approprié pour les échantillons légers et lourds dimethylated. En outre, la préparation manuelle de dizaines à des centaines d’échantillons peut introduire une erreur expérimentale élevée. Par conséquent, afin de réduire la variabilité de la préparation des échantillons, d’améliorer la précision quantitative et d’augmenter le débit expérimental, nous avons mis au point un flux de travail automatisé du CPILOT. L’automatisation se fait à l’aide d’un dispositif robotique de manipulation des liquides qui peut compléter de nombreux aspects du flux de travail( Figure 1). La préparation de l’échantillon, de la quantification des protéines à l’étiquetage peptidique, a été effectuée sur un gestionnaire de liquide automatisé. Le gestionnaire de liquide automatisé est intégré à un appareil à pression positive (PPA) pour les échanges tampons entre les plaques d’extraction à phase solide (SPE), le shaker orbital et un dispositif de chauffage/refroidissement. La plate-forme robotique contient 28 emplacements de pont pour accueillir des plaques et des tampons. Il y a deux gousses avec une pince pour transférer les plaques à l’intérieur des emplacements du pont : une tête de pipetage à volume fixe à 96 canaux (5-1100 μL) et 8 sondes à volume variable de canal (1-1000 μL). La plate-forme robotique est contrôlée à l’aide d’un logiciel. L’utilisateur doit être formé professionnellement avant d’utiliser le gestionnaire de liquide robotique. La présente étude se concentre sur l’automatisation du flux de travail manuel cPILOT, qui peut être à forte intensité de main-d’œuvre pour le traitement de plus de 12 échantillons en un seul lot. Afin d’augmenter le débit de l’approche cPILOT11, nous avons transféré le protocole cPILOT à un gestionnaire de liquide robotique pour traiter plus de 10 échantillons en parallèle. L’automatisation permet également des réactions similaires pour chaque échantillon en parallèle au cours des différentes étapes du processus de préparation de l’échantillon, ce qui a nécessité des utilisateurs hautement qualifiés à réaliser pendant le cPILOT manuel. Ce protocole se concentre sur la mise en œuvre du dispositif automatisé de manutention des liquides pour effectuer cPILOT. La présente étude décrit le protocole d’utilisation de ce système automatisé et démontre ses performances à l’aide d’une analyse « preuve de concept » de 22 plex des homogénéisations hépatiques de souris.
Protocole
Tous les protocoles sur les animaux ont été approuvés par le Comité institutionnel de protection et d’utilisation des animaux de l’Université de Pittsburgh. Une souris de contrôle mâle (C57/BLJ) a été achetée commercialement et logée dans la Division des ressources animales de laboratoire de l’Université de Pittsburgh. Des souris ont été alimentées le chow ad libitum standard de laboratoire de rongeur et maintenues dans un cycle clair/foncé de 12 h. Le tissu hépatique a été récolté et stocké à −80 °C.
1. Extraction de protéines
REMARQUE : Ces étapes sont exécutées manuellement.
- Laver le foie de souris (100 mg) avec de la solution saline et homogénéiser avec 500 μL d’urée de 8 M à l’aide d’un homogénéiseur mécanique à l’aide d’une matrice de lysage Perles A pour 4 m/s pendant 20 s.
REMARQUE : Dans cette étude, des inhibiteurs de la protéase ou de la phosphatase n’ont pas été ajoutés, mais peuvent être ajoutés au tampon si nécessaire, sur la base de l’expérience. En outre, les étapes d’extraction des protéines peuvent être ajustées en conséquence pour divers types d’échantillons. - Transférer l’homogénéité tissulaire dans un nouveau tube de microcentrifugeuse, rincer et combiner les tubes de lysing avec 100-500 μL de PBS avec 8 M d’urée.
- Centrifugez les tissus homogénéisés (12 800 x g,4 °C et 15 min) et recueillez le supernatant.
REMARQUE : Le reste des étapes sont effectuées sur le gestionnaire de liquide robotique disponible dans le laboratoire de l’utilisateur. Le gestionnaire liquide doit être capable d’aspirer et de distribuer des tampons à partir et à l’ANCI des types spécifiés de plaque, avec une participation minimale de l’opérateur. - Déterminer la concentration de protéines à l’aide d’un essai d’acide bicinchoninique (BCA) selon les instructions du fabricant.
- Allumez le PPA, le dispositif de chauffage/refroidissement, et les pompes à vide et connectez tous les accessoires avec le gestionnaire liquide. Le gestionnaire liquide affiche une lumière bleue une fois qu’il est connecté à l’ordinateur et prêt à fonctionner.
- Pour reagent plaque 1 (96 plaque de puits), ajouter 30 μL de 25 mM 1,4-dithiothreitol (DTT), 25 mM 30 μL de cystéine et 25 mM 30 μL d’iodoacetamide (IAA) aux rangées 1, 2 et 3, respectivement.
- Ajouter 8 M d’urée et 20 mM de Tris avec 10 mM CaCl2 (pH 8,2) à une plaque de réservoir. Ajouter 500 μL d’acide formique à 2 mL de plaque de puits profond, 20 μL de trypsine pour ramer 1 de plaque de trypsine et la placer dans l’emplacement du pont tel que spécifié par la méthode.
REMARQUE : L’IAA et la trypsine ont été ajoutées à la plaque de puits 96 avant d’ajouter à l’échantillon. - Aliquot 300 μL du foie homogénéiser dans un tube de 500 μL et placer sur une plaque de puits de 2 mL de profondeur et placer à 4 °C jusqu’au début du protocole. Pour cette expérience, 22 aliquots ont été générés à partir de l’homogénéisation hépatique unique.
REMARQUE : Ajouter une norme interne de contrôle de la qualité (p. ex., caséine alpha bovine ou autre protéine exogène) avec le rapport 1 μg standard : échantillon de protéines de 100 μg. - Définissez le volume de tampons à ajouter aux échantillons par noms et valeurs variables selon le tableau 1. Basé sur le BCA, entrez le volume de tampon d’échantillon et de normalisation (8 M d’urée) sur une feuille de calcul et attachez-le au logiciel (tableau 2).
REMARQUE : Une dilution supplémentaire avec tampon peut être nécessaire si la concentration en protéines est trop élevée. Les concentrations résultantes pour l’échantillon du tissu hépatique étaient de 10 μg/μL. Le volume des tampons a été optimisé pour 100 μg de protéines. - Retirer les tubes d’échantillon de 4 °C, les placer sur l’emplacement de pont spécifié du gestionnaire de liquide automatisé et laisser chauffer pendant 10 min.
- Ouvrez la méthode et suivez les instructions pour placer les conseils requis (1070, 90 et 230 μL), et labware dans les positions désirées. Une fois que tous les logiciels de laboratoire et les conseils sont en place, recoupez avec la disposition du pont final et cliquez sur Suivant pour continuer le protocole.
REMARQUE : La mise en place guidée informe l’utilisateur d’ajouter un volume de tampon requis au labware spécifique en fonction du protocole et de l’emplacement du deck à garder.
2. Réduction de l’échantillon, alkylation et digestion
- Chargez 230 bouts de μL et aspirez 90 μL de 8 M d’urée du réservoir et distribuez à la rangée 1 de la plaque noire de puits de 2 mL de profondeur. Déchargez les pourboires. Répétez cette étape jusqu’à ce que 8 M d’urée soient ajoutés à tous les puits correspondant aux 22 échantillons.
REMARQUE : Le tampon de dénaturation déroule la structure tridimensionnelle des protéines pour produire la structure primaire afin que la trypsine puisse agir et briser efficacement la protéine. La pipette à 8 canaux peut aspirer différents volumes pour les 8 canaux et peut distribuer à différents endroits dans le labware. Dans cette étape, tous les canaux aspiraient au même volume que défini dans le logiciel. - Après avoir ajouté le tampon de dénaturation, chargez les pointes de 90 μL et aspirez 10 μL (correspond à 100 μg) d’homogénéité du foie de souris à l’aide de la pipette à 8 canaux à la plaque noire de puits de 2 mL de profondeur. Déchargez les pourboires. Répétez cette étape jusqu’à ce que tous les 22 échantillons soient transférés.
- Après chaque transfert, effectuez une étape de mélange pour aspirer et distribuer 50 μL du contenu du puits trois fois pour assurer le mélange de la protéine avec le tampon pour montrer un rapport de 1:1.
REMARQUE : Retirer l’échantillon homogénéiser le bouillon et le placer en -80 °C. - Pour réduire les protéines dénaturées, chargez une rangée de 90 pointes de μL et aspirez 3 μL de TNT de la plaque de reagent 1. Distribuez la TNT aux rangées 1 et 2 de la plaque noire de puits de 2 mL de profondeur et déchargez les pointes.
REMARQUE : La protéine : Le rapport molaire de TNT est maintenu à 1:40 pour la réduction des liaisons de disulfide. Le calcul du rapport molaire est basé sur la masse protéique de l’albumine bovine de sérum (BSA), qui est de 66,5 x 103 g/mol. - Sceller la plaque d’échantillon avec du papier d’aluminium et l’incuber à 37 °C pendant 600 s à 300 rpm.
- Ajouter 30 μL de 0,25 M D’IAM à la rangée 3 de la plaque 1 du reagent juste avant utilisation et placer sur le pont. Déséalez la plaque d’échantillon et retournez sur le pont.
REMARQUE : IAM est sensible à la lumière. - Chargez une rangée de 90 bouts de μL, aspirez 6 μL d’IAM de la plaque de reagent 1 à la rangée 3, et distribuez à la rangée 1 de la plaque d’échantillon. Déchargez les pourboires.
REMARQUE : La protéine : Le rapport molaire IAM est maintenu à 1:80 pour chaque échantillon. Cette réaction doit se faire dans l’obscurité. - Sceller la plaque d’échantillon et incuber à 4 °C pendant 30 min à 300 rpm sur le dispositif de chauffage/refroidissement.
REMARQUE : L’étanchéité de la plaque est effectuée pour empêcher l’échantillon de s’allumer, de s’évaporer et de se déverser du puits. - Déséliser la plaque d’échantillon et charger une rangée de 90 pointes de μL et aspirer 5 μL de cystéine de la plaque de réagenage 1 à la rangée 2. Distribuez aux rangées 1 et 2 de la plaque d’échantillon et déchargez les pointes. Incuber à température ambiante pendant 30 min.
NOTE : La protéine : Le rapport molaire de L-cysteine est maintenu à 1:40. - Placez la plaque d’échantillon sur le shaker orbital pour effectuer une secousse à temps de 1800 rpm pendant 30 min.
- Ajouter 800 μL de tampon Tris de 20 mM avec 10 mM CaCl2 (pH 8,2) à chaque puits de la plaque d’échantillon pour diluer la concentration d’urée à 2 M. Décharger les pointes.
REMARQUE : L’activité de la trypsine est entravée à des concentrations d’urée plus élevées et doit donc être réduite à moins de 2 M. Cette étude a été réalisée avec un rapport protéine-trypsine molaire de 50: 1 pour 14 h. - Ajouter 20 μL de trypsine à la rangée 1, colonne 1-12 d’une plaque de puits de 96 et placer à un endroit déterminé sur le pont.
- Chargez une rangée de 90 bouts de μL, aspirez 2 μL de trypsine de la rangée de plaque de trypsine 1, et distribuez aux rangées 1 et 2 de la plaque d’échantillon. Déchargez les pourboires.
- Sceller la plaque et l’incuber pendant 14 h à 37 °C à 600 rpm sur le dispositif de chauffage/refroidissement.
- Après l’incubation, déséchez la plaque d’échantillon et ajoutez 5 % d’acide formique à la rangée 3, colonne 1- 12 d’une plaque de collecte de puits profond et placez-la sur le pont spécifié.
- Arrêtez la digestion en ajoutant 150 μL d’acide formique de 5% de la rangée 3 de la plaque acide formique et distribuez à la plaque d’échantillon aux rangées 1 et 2. Déchargez les pourboires.
3. Desalting étape 1
- Desalt les peptides avec plaque SPE contenant 20 mg de matériau Targa C-18. Fixez le volume pour chaque échange tampon comme 600 μL et fixez la pression à 100 mbar.
- Chargez 1070 pointes de μL et aspirez 600 μL d’acétyltrile et distribuez aux rangées 1 et 2 de la plaque SPE. Placez la plaque SPE sur PPA et appliquez une pression.
- À l’aide des mêmes pointes, aspirer 600 μL d’acétyl et distribuer aux rangées 1 et 2 de la plaque SPE. Placez la plaque SPE sur PPA et appliquez une pression.
REMARQUE : Le débit peut être drainé à l’aide d’une pompe d’aspiration. - Chargez 1070 pointes de μL et aspirez 600 μL de tampon A (100 % d’eau dans 0,1 % d’acide formique) et distribuez aux rangées 1 et 2 de la plaque SPE. Placez la plaque SPE sur PPA et appliquez une pression.
REMARQUE : Si le volume du tampon ne réduit pas de façon significative, essayez d’augmenter la pression ou le temps. - Chargez 2 rangées de 1070 pointes de μL, aspirez 534 μL d’échantillons digérés et distribuez aux rangées 1 et 2 de la plaque SPE. Placez la plaque SPE sur PPA et appliquez une pression. Répétez cette étape jusqu’à ce que tous les échantillons soient chargés.
REMARQUE : Étant donné que le volume total après l’ajout d’acide formique sera de 1068 μL et que les échantillons ont été ajoutés en deux passages. - Chargez 2 rangées de pointes μL usées de 1070, aspirez 600 μL de tampon A et distribuez aux rangées 1 et 2 de la plaque SPE. Placez la plaque SPE sur PPA et appliquez une pression.
- Répétez l’étape ci-dessus une fois pour nettoyer l’échantillon.
- Chargez 1 rangée de 1070 pointes de μL, aspirez 600 μL d’ACN : Eau (60:40) et distribuez aux rangées 1 et 2 de plaque SPE. Placez la plaque SPE sur une plaque de collecte pour élucider les peptides à l’aide de PPA et appliquer de la pression.
- Répétez l’étape ci-dessus pour eluter les peptides dans la plaque de collecte. Sécher la plaque de collecte à la sécheresse et la conserver à -80°C jusqu’à ce qu’elle soit encore en traitement.
4. Étiquetage de diméthylation (peptide N-termini)
- Placez les conseils requis (1070, 90 et 230 μL) et les logiciels de laboratoire dans les positions souhaitées dans le logiciel. Une fois que tous les logiciels de laboratoire et les conseils sont en place, recoupez avec la disposition du pont final et cliquez sur Suivant pour continuer le protocole.
- Pour reagent plaque 2, ajouter 450 μL d’acide acétique de 1%, 50 μL de formaldéhyde léger (CH2O), 50 μL de formaldéhyde lourd(13CD2O), et 150 μL d’acide formique aux rangées 1, 2, 3 et 4 respectivement.
- Pour reagent plaque 3 ajouter 50 μL de CB léger, 50 μL de CB lourd, aux rangées 1 et 2 et 50 μL d’ammoniac à la rangée 3 et 4.
- Chargez 2 rangées de 230 pointes de μL et aspirez 100 μL d’acide acétique de 1 % de la rangée 1 de la plaque de réagent 2 et distribuez-les aux peptides séchés dans la plaque d’échantillon 2 (plaque de collecte de l’étape de désaltage) rangées 1 et 2 et effectuez une secousse à temps pendant 5 min à 18 h.
- Chargez 90 pointes de μL et aspirez 16 μL de 60 mM (4 %) CH2O (37% wt/v) de la rangée 2 de la plaque de réaccente 2 et distribuer à la rangée 1 de la plaque d’échantillon. Déchargez les pourboires.
- Chargez 1 rangée de 90 pointes de μL et aspirez 16 μL de 60 mM (4 %) 13 Ans CD2O (20% wt/v) de la rangée 3 de la plaque de reagent 2 et distribuer à la rangée 2 de la plaque d’échantillon. Déchargez les pourboires.
REMARQUE : Dans cette ligne d’étude, la première ligne correspond à la lumière et la rangée 2 correspond à de lourds échantillons d diméthylés (voir la figure 1). - Chargez 2 rangées de 90 bouts de μL et aspirez 16 μL de 24 mM NaBH3CN et 24 mM NaBD3CN à partir des rangées 1 et 2 de la plaque de reagent 3 et distribuez aux rangées 1 et 2, respectivement de la plaque d’échantillon.
- Déchargez les pointes et effectuez une secousse minuterie pendant 15 min à 18 h à l’aide du shaker orbital.
REMARQUE : L’ajout de cyanoborohydride de sodium déclenche la réaction afin de réduire la variabilité entre les expériences cette étape est effectuée une fois par lot. - Chargez 2 rangées de pointes de 90 μL et aspirez 32 μL d’ammoniac de 1 % (~28-30% v/v) des rangées 3 et 4 de la plaque de réagenage 3 et distribuez aux rangées 1 et 2 respectivement de la plaque d’échantillon 2. Déchargez les pourboires.
REMARQUE : L’ajout d’ammoniac arrête la réaction afin de réduire la variabilité entre les expériences cette étape est effectuée une fois par lot. - Combinez des volumes égaux de peptides légers et lourds (1:1) dimethylated à une nouvelle plaque de puits de 2 mL de profondeur pour le desalage.
REMARQUE : Dans cette étude, 90 μL du contenu du puits de la rangée 1 ont été combinés avec 90 μL de la rangée 2 de la plaque d’échantillon 2 à une nouvelle plaque de collecte de 2 mL (plaque d’échantillon 3). Le rapport de lumière : le mélange lourd dépend du protocole expérimental, dans cette étude un rapport de 1:1 a été utilisé. - Chargez 2 rangées de 230 pointes de μL et aspirez 32 μL d’acide formique de 5 % aux échantillons combinés et effectuez une secousse à temps pendant 30 s à 1800 rpm.
REMARQUE : L’efficacité de la diméthylation dépend du pH du mélange de réaction et tout changement dans le pH tampon entraînera une étiquetage incomplète du peptide N-termini. L’efficacité de la diméthylation devrait être supérieure à 97% lorsqu’elle est recherchée comme modification dynamique au peptide N-termini. Dans cette étude, l’efficacité d’étiquetage des peptides légers et lourds était de 99,7 % et de 99,5 % respectivement.
5. Desalting étape 2
- Effectuez des désaltations d’échantillons semblables à l’étape 1 du desalt pour les échantillons combinés.
- Séchez les échantillons dans la plaque d’échantillon à l’aide d’un aspirateur à vitesse et conservez-les à -80 °C jusqu’à ce qu’ils soient traités plus avant.
6. Marquage isobarique (résidus de Lys)
- Suivez la configuration guidée des logiciels de laboratoire et placez les conseils requis (1070, 90 et 230 μL), avec des tampons appropriés. Une fois que tous les logiciels de laboratoire et les conseils sont en place, recoupez avec la disposition du pont final et cliquez sur Suivant pour continuer le protocole.
- Ajouter 250 μL de bicarbonate d’ammonium triethyle de 100 mM (TEAB), 30 μL d’hydroxylamine (10 % w/v) aux rangées 1 et 2 de la plaque de réagencé 4. Placez les tubes TMT sur la plaque de puits profond de 2 mL selon la feuille de calcul du tableau 3.
- Gardez un tube vide de 1,5 mL dans un support de support de tube pour mettre en commun les peptides marqués. Placer la plaque d’échantillon séchée à la plaque de traitement P9 et TMT au P14.
- Pour reconstituer les peptides, chargez 230 pointes de μL et aspirez 100 μL de bicarbonate d’ammonium triethyle de 100 mM (TEAB) tampon (pH ~8,5) de la rangée 1 à la plaque TEAB et distribuez aux peptides séchés à la rangée 3 de la plaque d’échantillon 3. Placez la plaque sur le shaker orbital pendant 30 s à 1800 rpm.
REMARQUE : Reconstituer 100 μg de peptides diméthylés à une concentration de 1 μg/μL. - Chargez 90 pointes de μL et aspirez 10 μL d’acétylonitrile anhydre du H12 de la plaque TMT et distribuez-les dans chacun des tubes TMT séchés.
- Retirez les tubes TMT, vortex et faites tourner rapidement les tubes et retournez sur le pont dans une plaque de puits profonde.
- Chargez 1 rangée de pointes de 90 μL et aspirez 12,5 μL des peptides d diméthylés combinés et distribuez-les à la rangée 1 de la plaque de traitement TMT. Déchargez les pourboires.
NOTE: Le TMT: peptide ratio a été maintenu à 1:8 pour cette expérience. - Chargez 1 rangée de 90 pointes de μL et aspirez 10 μL de TMT et distribuez à la rangée 1 de la plaque de traitement TMT. Déchargez les pointes, effectuez un shake à l’heure pendant 1 heure à 1800 rpm.
- Chargez 1 rangée de pointes de 90 μL et aspirez 8 μL d’hydroxylamine (10 % w/v) de la rangée 2 à la plaque TEAB et distribuez à la rangée 1 de la plaque de traitement TMT. Déchargez les pointes, effectuez une secousse minuter pendant 15 min à 1800 rpm.
- Mélanger 30,5 μL des peptides étiquetés TMT de la plaque de traitement TMT au tube de 1,5 mL. Déchargez les pourboires après chaque transfert.
- Retirer le tube avec les échantillons mis en commun et sécher pour évaporer l’acétyltrile. Reconstituer les peptides dans 0,2 mL d’eau dans 0,1 % d’acide formique et retourner sur le pont.
REMARQUE : L’efficacité d’étiquetage du marquage isobarique est également spécifique au pH et l’efficacité de l’étiquetage doit être supérieure à 98 % par instruction du fabricant. Dans cette étude, l’efficacité d’étiquetage TMT des peptides légers et lourds terminés par lysine était de 99,4 % et de 99,5 % respectivement.
7. Étape de desalting
- Effectuez des désaltations d’échantillons semblables au desalte 1 pour un échantillon.
- Sécher les échantillons et les conserver en -80 °C jusqu’à l’analyse.
8. Chromatographie liquide–Spectrométrie de masse tandem (LC-MS/MS) et MS3
- Reconstituer les peptides dans l’eau de qualité SP avec 0,1 % de FA pour obtenir une concentration d’environ 1 μg/μL. Filtrer les échantillons avec des tubes de microcentrifugeuse contenant un filtre de 0,65 μm. Peptides centrifugeuses à 12.000 x g pendant 3 min et placer le flux à travers dans un flacon auto-échantillonneur.
REMARQUE : La concentration de peptide peut être confirmée à ce stade si désiré. Les peptides devraient être reconstitués dans de l’eau de qualité LC-MS et soumis à un essai de peptide BCA. Dans cette étude, l’essai de BCA de peptide n’a pas été exécuté et toutes les quantités de peptide ont été basées sur l’essai initial de BCA de protéine. - Préparez les tampons de phase mobile comme suit : 100 % (v/v) d’eau avec 0,1 % de FA (A) et 100 % d’ACN avec 0,1 % de FA (B).
- Injecter 1 μL d’échantillon sur une colonne de piège remplie de 2 cm de matériau C18 (3 μm, 100 Å pore size).
REMARQUE : Le nettoyage de l’échantillon sur le piège est le suivant : 10 min, 100 % A; 2 μL/min à l’aide d’un système de chromatographie liquide 2D. - Exécutez la méthode de séparation analytique. Utilisez une colonne capillaire en silice fondue laser de 100 μm i.d. x 26 cm emballée avec du matériau C18 (2,5 μm, 100 Å). Le gradient est: 0-10 min, 10% B; 10-30 min, 10-15% B; 30-75 min, 15-30% B; 75-88 min, 30-60% B; 88-92 min, 60-90% B; 92-99 min, 90% B; 99-100 min, 90-10% B, 100-120 min, 10% B; 300 nL/min, 120 min.
- Exécutez l’acquisition de données pour le spectromètre de masse pendant que la méthode de séparation analytique est en cours d’exécution.
- Utilisez les paramètres suivants pour l’analyse de l’enquête sur la SP : 375-1 500 m/z,résolution de 120 000, temps de cycle 3 s (vitesse de pointe), cible de contrôle automatique du gain (AGC) 4,0e5, temps d’injection maximal 50 ms.
- Utilisez les paramètres suivants pour cid-MS/MS avec piège à ions : analyses d’acquisition dépendantes des données (DDA) par résultat, largeur d’isolement de 2 m/z, énergie de collision normalisée de 35 % (RCE), 0,25 q d’activation, temps d’activation de 10 ms, 1,0e4 AGC, temps d’injection maximal de 100 ms.
- Utilisez les paramètres suivants pour HCD-MS3 SPS 10: Plage de balayage 100-400 m/z, nombre de scans dépendants 10, AGC 5.0e4, temps d’injection maximum 118 ms, énergie de collision HCD 55%, fenêtred’isolement MS 2 2.
9. Analyse des données
- La recherche a généré des fichiers RAW pour la liste des protéines et peptides à l’aide d’un logiciel d’analyse des protéines contre une base de données appropriée.
REMARQUE : Les fichiers RAW générés dans cette étude ont été recherchés contre une base de données Uniprot de souris. - Puisqu’il y a l’étiquetage léger et lourd dans un fichier RAW, recherchez chaque fichier avec deux flux de travail pour les peptides dimethylated légers et lourds.
- Rechercher les fichiers RAW contre la base de données Uniprot souris (07/13/2019) avec 53035 séquence avec les paramètres suivants: clivage trypsine avec maximum deux clivages manqués, peptide avec une longueur minimale de 6 acides aminés, tolérance de masse parente de 15 ppm, 1 tolérance à la fragmentation Da Modifications statiques : diméthylation légère (+28.031, peptide N-terminus) ou diméthylation lourde (+36.076 Da, peptide N-terminus), carbamidomethyl (+57.021 Da, C), Modifications dynamiques : oxydation (+15.995 Da, M), étiquette isobarique 11-plex (229.163 Da, K), 1% FDR, quantification d’ion de reporter avec tolérance d’intégration maximale de 30 ppm, centroïde le plus confiant pour la méthode d’intégration.
REMARQUE: Le pic miméthylated lourd comprend également une autre modification qui est ~ 7 Da (+35.069 Da, peptide N-terminus) de la lumière dimethylated pic et donc un autre nœud de recherche devrait être incorporé pour inclure cette modification aussi. - Effectuer la quantification d’ions de journaliste basée sur l’intensité, 65% d’allumette de masse de SPS, le rapport moyen de S/N 10, la correction isotopique, la normalisation et l’échelle n’ont pas été exécutées.
REMARQUE : La normalisation et l’échelle peuvent être effectuées en fonction de la quantité totale de peptide ou d’une protéine spécifique ajoutée à l’échantillon. Les échantillons de QC peuvent également être inclus dans les canaux de normalisation inter-lots ou intra-lots sur la base d’une mise à l’échelle interne àdeux queues 18. Les contaminations isotopiques des différents canaux TMT n’ont pas été fournies à la recherche, les utilisateurs sont invités à ajouter la contamination isotopique de différents ions reporter.
Résultats
La figure 2 montre les données représentatives sur la SP d’un peptide identifié dans les 22 canaux iion des reporters à partir d’une expérience cPILOT de 22 plex, y compris les répliques de flux de travail. La figure 2 (en haut) représente une paire de pointe doublement chargée séparée par un espacement de 4 Da m/z indiquant un seul groupe de diméthyle incorporé dans le peptide. Les paires de pointe miméthylées légères et lourdes ont été isolées et fragmentées indépendamment pour donner la séquence du peptide. La séquence du peptide est G(diméthyle)AAELMQQK (TMT-11plex) et correspond à la protéine Betaine-homocysteine S-méthyl transferase. Les ions fragmentaires les plus intenses pour les pics de diméthylated légers et lourds(non montrés)ont été encore isolés pour la fragmentation de MS3 et les ions reporter(m/z 126-131) sont indiqués dans la figure 2 (en bas). Les intensités d’ion de journaliste sont directement proportionnées à l’abondance de peptide dans l’échantillon. L’abondance peptidique des échantillons implique que la capacité de pipetage de la plate-forme robotique est assez uniforme sur les 22 échantillons. Dans l’ensemble, cette expérience cPILOT de 22 plex a donné lieu à 1 326 identifications de protéines (1209-light/1181-heavy) résultant de 3098 peptides (6137-light/5872-heavy)(tableau 4). La figure 3 montre l’intrigue de la boîte de l’abondance log10 par rapport à l’intensité totale des ion de reporter sur l’ensemble des 22 canaux montrant une variabilité moindre entre les puits et entre les échantillons. L’évaluation de l’automatisation totale a été effectuée en examinant l’erreur dans l’abondance des ion des journalistes dans chaque protéine dans les 22 échantillons. La figure 4 montre que le traitement de l’échantillon avec la plate-forme robotique a donné lieu à de très faibles valeurs de CV. Plus précisément, dans les 3098 peptides identifiés, le CV moyen dans l’abondance des ions des journalistes était de 12,36 % et de 15,03 % pour les peptides diméthylés légers et lourds, respectivement. Parmi ces peptides 2032 de ces peptides ont eu le signal d’ion de journaliste au-dessus du seuil minimum et ont été jugés quantifiables.
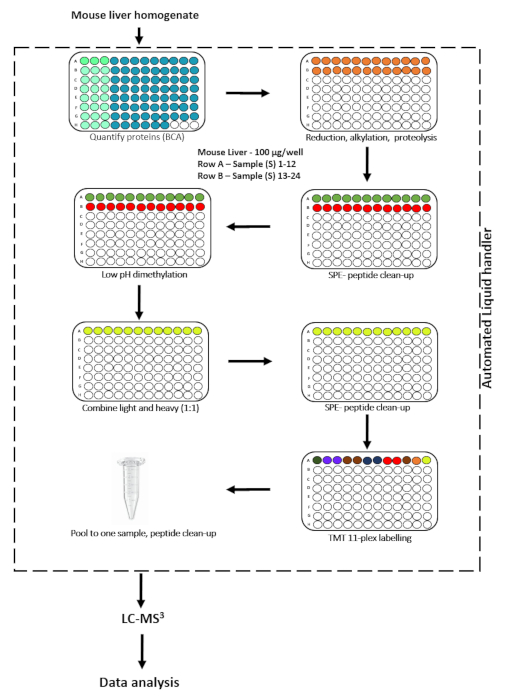
Figure 1. Flux de travail expérimental pour traiter 22 échantillons en parallèle avec un protocole cPILOT automatisé. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
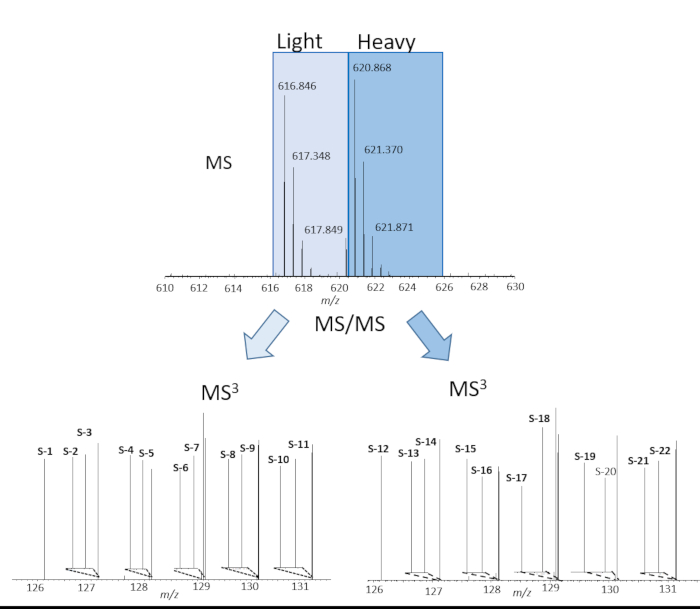
Figure 2. Quantification des peptides sur 22 échantillons. Exemple de spectres ms (en haut) et MS3 (en bas) du peptide G(diméthyle)AAELMQQK (TMT-11plex) quantifiés dans l’expérience cPILOT automatisée de 22 plex pour les pics légers d diméthylés (en bas à gauche) et lourds diméthylés (en bas à droite). S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
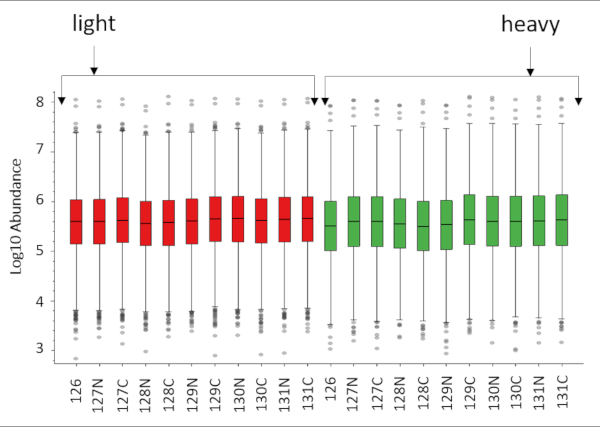
Figure 3. Parcelle de boîte d’intensités totales d’ion de reporter par rapport à l’abondance de log10 de 22 échantillons utilisant le découvreur de protéome 2.3. Le fichier RAW a été recherché deux fois pour les peptides légers et lourds, les protéines ID séparément avec TMT comme modification dynamique, la lumière (+28.031 Da) et lourd (+36.076 & +35.069 Da) diméthylation au peptide N-termini comme modification statique. Une recherche combinée avec toutes les modifications ci-dessus a été exécuté en utilisant Proteome Discover 2.3 pour obtenir le Journal 10 Abondance d’intensités peptidiques sur tous les canaux. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
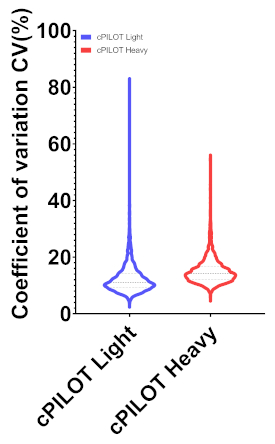
Figure 4. Parcelles de violon de co-efficacité de la variation de l’abondance peptidique à partir des intensités d’ion reporter résumé à travers les canaux 126-131 m/z. Le peptide a été quantifié avec une valeur moyenne de CV de 12,36 et 15,03 pour les peptides légers (2373) et lourds (2533) quantifiables. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Figure supplémentaire 1. Illustration du cPILOT avec un peptide simple. Montrant l’étiquetage isotopique de deux échantillons différents et le marquage isobarique avec TMT126, le mélange résultant a été injecté à MS pour LC-MS3. S’il vous plaît cliquez ici pour télécharger ce fichier.
| Nom variable | Valeur | Description |
| DesaltSamp1 | 1065 | Volume à utiliser pour desalt étape 1 |
| DesaltSamp2 | 392 | Volume à utiliser pour desalt étape 2 |
| DesaltSamp3 | 100 | Volume à utiliser pour desalt étape 3 |
| DevMode (En) | Faux | Faux va réduire les temps d’incubation à 30sec- Vrai suivra le moment d’incubation dans le protocole. |
| DTTVol (en) | 3 | Volume de TNT |
| FilterPlate | Targa | Plaque utilisée pour le desalage |
| FilterPlateVol | 600 | Volume pour le desalting |
| HAWaterWashes | Faux | Nombre de lavages d’eau sur la plaque SPE |
| IAMVol (IAMVol) | 2 | Volume d’iodoacetamide |
| PeptideTMTVol | 12.5 | Volume de peptide pour l’étiquetage TMT |
| Pression | 100 | pression mbar chez PPA |
| TempOffSet ( TempOffSet ) | 1 | Changement de température |
| TMTVol (en) | 10 | Volume d’étiquettes isobariques à ajouter |
| TrisVol (TrisVol) | 800 | Volume pour diluer l’échantillon avant la digestion |
| TrypsinVol TrypsinVol | 2 | Volume de trypsine |
| UsePopTimer (usePopTimer) | Vrai | True affiche les options pour appliquer une pression sur la plaque si nécessaire |
Tableau 1. Liste des variables utilisées dans le protocole cPILOT automatisé.
| Source Dil | DilWell ( DilWell ) | Dest | DestWell ( DestWell ) | DilVolume DilVolume | StockSource | Stockwell | SampleVol (en) | SampleID (en) |
| 8M_Urea | 1 | Échantillons | A1 (en) | 90 | Stock_Samples | A1 (en) | 10 | 1 |
| 8M_Urea | 1 | Échantillons | A2 (en) | 90 | Stock_Samples | A1 (en) | 10 | 2 |
| 8M_Urea | 1 | Échantillons | A3 (en) | 90 | Stock_Samples | A1 (en) | 10 | 3 |
| 8M_Urea | 1 | Échantillons | A4 (en) | 90 | Stock_Samples | A1 (en) | 10 | 4 |
| 8M_Urea | 1 | Échantillons | A5 (en) | 90 | Stock_Samples | A1 (en) | 10 | 5 |
| 8M_Urea | 1 | Échantillons | A6 (en) | 90 | Stock_Samples | A1 (en) | 10 | 6 |
| 8M_Urea | 1 | Échantillons | A7 (en) | 90 | Stock_Samples | A1 (en) | 10 | 7 |
| 8M_Urea | 1 | Échantillons | A8 (en) | 90 | Stock_Samples | A1 (en) | 10 | 8 |
| 8M_Urea | 1 | Échantillons | A9 (en) | 90 | Stock_Samples | A1 (en) | 10 | 9 |
| 8M_Urea | 1 | Échantillons | A10 (en) | 90 | Stock_Samples | A1 (en) | 10 | 10 |
| 8M_Urea | 1 | Échantillons | A11 (en) | 90 | Stock_Samples | A1 (en) | 10 | 11 |
| 8M_Urea | 1 | Échantillons | A12 (en) | 90 | Stock_Samples | A1 (en) | 10 | 12 |
| 8M_Urea | 1 | Échantillons | B1 (en) | 90 | Stock_Samples | A1 (en) | 10 | 13 |
| 8M_Urea | 1 | Échantillons | B2 | 90 | Stock_Samples | A1 (en) | 10 | 14 |
| 8M_Urea | 1 | Échantillons | B3 (en) | 90 | Stock_Samples | A1 (en) | 10 | 15 |
| 8M_Urea | 1 | Échantillons | B4 (en) | 90 | Stock_Samples | A1 (en) | 10 | 16 |
| 8M_Urea | 1 | Échantillons | B5 (en) | 90 | Stock_Samples | A1 (en) | 10 | 17 |
| 8M_Urea | 1 | Échantillons | B6 (en) | 90 | Stock_Samples | A1 (en) | 10 | 18 |
| 8M_Urea | 1 | Échantillons | B7 (en) | 90 | Stock_Samples | A1 (en) | 10 | 19 |
| 8M_Urea | 1 | Échantillons | B8 (en) | 90 | Stock_Samples | A1 (en) | 10 | 20 |
| 8M_Urea | 1 | Échantillons | B9 (en) | 90 | Stock_Samples | A1 (en) | 10 | 21 |
| 8M_Urea | 1 | Échantillons | B10 (en) | 90 | Stock_Samples | A1 (en) | 10 | 22 |
| 8M_Urea | 1 | Échantillons | B11 (en) | 90 | Stock_Samples | A1 (en) | 10 | 23 |
| 8M_Urea | 1 | Échantillons | B12 (en) | 90 | Stock_Samples | A1 (en) | 10 | 24 |
Tableau 2. Volume d’homogénéité du foie de souris et 8 M d’urée.
| SourceWell (en) | SourceWell2 (en) | Reporter Ion | DestWell1 ( DestWell1 ) | DestWell2 ( DestWell2 ) | Volume | SampleID (en) |
| A1 (en) | C1 (C1) | 126 | A1 (en) | E1 (e1) | 10 | 1 |
| A3 (en) | C3 (C3) | 127N (127N) | A2 (en) | E2 (e2) | 10 | 2 |
| A5 (en) | C5 (en) | 127C (127C) | A3 (en) | E3 (E3) | 10 | 3 |
| A7 (en) | C7 (en) | 128N (128N) | A4 (en) | E4 (e4) | 10 | 4 |
| A9 (en) | C9 (en) | 128C (128C) | A5 (en) | E5 | 10 | 5 |
| A11 (en) | C11 (C11) | 129N (129N) | A6 (en) | E6 (e6) | 10 | 6 |
| B2 | D2 (D2) | 129C (129C) | A7 (en) | E7 (e7) | 10 | 7 |
| B4 (en) | D4 (en) | 130N (130N) | A8 (en) | E8 (e8) | 10 | 8 |
| B6 (en) | D6 (en) | 130c | A9 (en) | E9 (E9) | 10 | 9 |
| B8 (en) | D8 (en) | 131N (131N) | A10 (en) | E10 | 10 | 10 |
| B10 (en) | D10 (D10) | 131C (131C) | A11 (en) | E11 (E11) | 10 | 11 |
Tableau 3. Nombre total de peptides, protéines et peptides spectrals correspondances (MSP).
| CPILOT automatisé | ||
| Lumière | Lourd | |
| Protéines | 1209 | 1181 |
| Peptides | 6137 | 5872 |
| MSP | 14948 | 16762 |
Tableau 4. Codage à barres des étiquettes isobariques avec les échantillons étiquetés légers et lourds.
Discussion
cPILOT est une stratégie de multiplexage améliorée qui peut analyser jusqu’à 24 échantillons en une seule expérience. La capacité de multiplexage dépend du nombre de combinaisons de marquage isotopiques et isobariques disponibles. L’introduction du TMTpro7, qui est capable d’étiqueter 16 échantillons en une seule expérience, peut repousser les limites du cPILOT à 32-plex. cPILOT se compose de plusieurs étapes de pipetage et nécessite des soins approfondis et des compétences de l’utilisateur pour effectuer la préparation de l’échantillon. Même avec un utilisateur expert, les erreurs manuelles sont inévitables, ce qui invite l’utilisation de plates-formes robotiques pour traiter des échantillons dans la stratégie cPILOT. Étant donné que cPILOT utilise le marquage dépendant du pH des peptides, le pH doit être maintenu pour la lumière et l’ensemble lourd d’échantillons dimethylated. Le pH légèrement acide-basique peut avoir comme conséquence la diméthylation des résidus de N-termini et de lysine. Un avantage du cPILOT est qu’il ne nécessite que la moitié des étiquettes isobariques puisque le peptide N-termini sont occupés avec les groupes de diméthyle. Cela permet un plus grand nombre d’échantillons d’être étiquetés à la moitié du coût. La manipulation de plus grands nombres d’échantillons exige que les temps d’exposition des reagents soient similaires pour le premier et le dernier échantillon d’un lot. Un distributeur de pipette pouvant accueillir jusqu’à 32 échantillons en parallèle peut être réalisé avec l’utilisation de dispositifs robotiques de manutention des liquides.
Afin de traiter plusieurs échantillons par cPILOT, le workflow manuel a été modifié pour intégrer l’automatisation. Le gestionnaire de liquide robotique utilisé dans cette étude a deux gousses avec des capacités de pipetage à 96 canaux et à 8 canaux, avec une pince pour placer les plaques dans les 28 emplacements de pont disponibles. Le gestionnaire liquide est intégré à un appareil à pression positive, shaker orbital, et un dispositif pour chauffer / refroidir les échantillons dans la plaque de puits 96. L’appareil à pression positive aide à effectuer des échanges tampons dans les plaques SPE pendant le nettoyage, tandis que le shaker orbital aide à vortex / mélanger les échantillons. La plate-forme robotique a été programmée pour aspirer et distribuer des tampons et des échantillons à des plaques de 96 puits, à l’incubation, à des échantillons de vortex et à des plaques de transfert. Les liquides avec des viscosités différentes, telles que l’acétylonitrile et l’eau, nécessitent des considérations spécifiques de pipetting qui peuvent également être programmées dans la méthode.
Le flux de travail cPILOT, à partir de la quantification des protéines par BCA à l’étiquetage des peptides à l’aide d’étiquettes isobariques (c.-à-d. TMT), a été effectué sur le système de manutention liquide. Le protocole complet a été mis à l’échelle pour utiliser 96 plaques de puits profonds qui peuvent contenir 2 mL par puits. Les tampons ont été préparés avant le début de l’expérience et ajoutés à la plaque de puits 96 afin de permettre le traitement parallèle de l’échantillon. Dans la présente étude, 22 répliques de flux de travail de l’homogénéité du foie de souris ont été ajoutées aux plaques profondes du puits et prises par le protocole cPILOT. Enfin, un seul échantillon composé des peptides marqués par le foie de souris équimolar de 22 plex a été injecté au spectromètre de masse. Les intensités d’ion de reporter correspondant à l’abondance de peptide dans les échantillons ont démontré que les échantillons traités avec le gestionnaire liquide ont des CV inférieurs au protocole manuel(données non montrées). La plate-forme robotique a également grandement amélioré la reproductibilité du traitement des échantillons. La reproductibilité et la robustesse sont des facteurs très importants lors du traitement d’un grand nombre d’échantillons. Les erreurs de pipetage peuvent conduire à une mauvaise interprétation complète des données et ici la plate-forme robotique a fourni une faible variation inter-échantillon. L’utilisation de la plate-forme robotique pour le cPILOT a également réduit le temps nécessaire à la préparation des échantillons. Par exemple, après avoir développé la méthode automatisée, il a fallu 2,5 h pour traiter 22 échantillons contre 7,5 h pour le cPILOT manuel. Des expériences sont en cours dans notre laboratoire pour évaluer davantage les comparaisons des workflows manuels et automatisés du CPILOT. D’après les rapports précédents de notre laboratoire, les taux d’intensité des ions de reporter de protéines dans le cPILOT manuel étaient en moyenne de 20 %, certaines valeurs aberrantes dépassantcette valeur 12.
cPILOT est une stratégie de dérivation chimique au niveau du peptide, qui peut être utilisée pour n’importe quel type d’échantillon comme les cellules, les tissus et les liquides organiques. cPILOT offre un multiplexage d’échantillons amélioré et avec l’incorporation de l’automatisation peut faciliter le multiplexage d’échantillons à haut débit dans la protéomique. Ce débit est nécessaire pour faire progresser la compréhension de la maladie et de la biologique et la découverte de biomarqueurs.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs reconnaissent les fonds de démarrage de l’Université Vanderbilt et le prix NIH (R01GM117191) à RASR.
matériels
| Name | Company | Catalog Number | Comments |
| 0.6 mL eppendorf tubes, 500 pk | Fisher Scientific | 05-408-120 | Any brand of 0.6 mL eppendorf tubes are sufficient |
| 0.65 µm Ultrafree MC DV centrifugal filter units | EMD Millipore | UFC30DV00 | |
| 1.5 mL eppendorf tubes, 500 pk | Fisher Scientific | 05-408-129 | Any brand of 1.5 mL eppendorf tubes are sufficient |
| 2 ml black deep well plate | Analytical Sales and Services, Inc. | 59623-23BKGC | Any brand of black 96-well plate is sufficient |
| 2 ml clear deep well plate | VWR | 75870-796 | |
| Acetic Acid | J.T. Baker | 9508-01 | |
| Acetonitrile - MS Grade | Fisher Scientific | A955-4 | 4 L quantity is not necessary |
| Agilent 500µL plate | Agilent | 203942-100 | Reagent plate for adding buffers |
| Ammonium formate | Acros Organics | 208-753-9 | |
| Ammonium hydroxide solution (28 - 30%) | Sigma Aldrich | 320145-500ML | |
| Analytical balance | Mettler Toledo | AL54 | |
| BCA protein assay kit | Pierce Thermo Fisher Scientific | 23227 | |
| Biomek i7 hybrid | Beckmann | Any liquid handling device with ability to use positive pressure, heating/cooling and Vortex the samples. | |
| C18 packing material (2.5 µm, 100 Å) | Bruker | This item is no longer available from Bruker. Alternative packing material with listed specifications will be sufficient | |
| Centrifuge with plate rotor | Thermo Scientific | 69720 | |
| Micro 21R Centrifuge | Sorval | 5437 | |
| Dionex 3000 UHPLC | Thermo Scientific | This model is no longer available. Any nano LC with an autosampler is sufficient. | |
| Dithiothreiotol (DTT) | Fisher Scientific | BP172-5 | |
| Formaldehyde (13CD2O) solution; 20 wt % in D2O, 98 atom % D, 99 atom % 13C | Sigma Aldrich, Chemistry | 596388-1G | |
| Formaldehyde (CH2O) solution; 36.5 - 38% in H2O | Sigma Aldrich, Life Science | F8775-25ML | |
| Formic Acid | Fluka Analytical | 94318-250ML-F | |
| Fusion Lumos Mass Spectrometer | Thermo Scientific | This model is no longer available. Other high resolution instruments (e.g. Orbitrap Elite, Orbitrap Fusion, or Orbitrap Fusion Lumos) can be used. | |
| Hydroxylamine hydrochloride | Sigma Aldrich, Chemistry | 255580-100G | |
| Iodoacetamide (IAM) | Acros Organics | 144-48-9 | |
| Isobaric Tagging Kit (TMT 11-plex) | Thermo Fisher Scientific | 90061 | |
| L-1-tosylamido-2 phenylethyl cholormethyl ketone (TPCK)-treated Trypsin from bovine pancreas | Sigma Aldrich, Life Science | T1426-100MG | |
| L-Cysteine | Sigma Aldrich, Chemistry | 168149-25G | |
| Mechanical Homogenizer (i.e. FastPrep-24 5G) | MP Biomedicals | 116005500 | |
| pH 10 buffer | Fisher Scientific | 06-664-261 | Any brand of pH buffer 10 is sufficient |
| pH 7 buffer | Fisher Scientific | 06-664-260 | Any brand pH buffer 7 is sufficient |
| pH meter (Tris compatiable) | Fisher Scientific (Accumet) | 13-620-183 | Any brand of a pH meter is sufficient |
| Protein software (e.g. Proteome Discoverer) | Thermo Scientific | ||
| Reservior plate 200ml | Agilent | 204017-100 | |
| Sodium Cyanoborodeuteride; 96 atom % D, 98% CP | Sigma Aldrich, Chemistry | 190020-1G | |
| Sodium Cyanoborohydride; reagent grade, 95% | Sigma Aldrich | 156159-10G | |
| Speed-vac | Thermo Scientific | SPD1010 | any brand of speed vac that can accommodate a deep well plate is sufficient |
| Stir plate | VWR | 12365-382 | Any brand of stir plates are sufficient |
| Targa 20 mg SPE plates | Nest Group, Inc. | HNS S18V | These are C18 cartridges |
| Triethyl ammonium bicarbonate (TEAB) buffer | Sigma Aldrich, Life Science | T7408-100ML | |
| Tris | Biorad | 161-0716 | |
| Biomek 24-Place Tube Rack Holder | Beckmann | 373661 | |
| Urea | Biorad | 161-0731 | |
| Water - MS Grade | Fisher Scientific | W6-4 | 4 L quantity is not necessary |
Références
- Diamandis, E. P. Mass spectrometry as a diagnostic and a cancer biomarker discovery tool: opportunities and potential limitations. Molecular and Cell Proteomics. 3 (4), 367-378 (2004).
- Qian, W. -J., Jacobs, J. M., Liu, T., Camp, D. G., Smith, R. D. Advances and challenges in liquid chromatography-mass spectrometry-based proteomics profiling for clinical applications. Molecular and Cellular Proteomics. 5 (10), 1727-1744 (2006).
- Arul, A. B., Robinson, R. A. S. Sample Multiplexing Strategies in Quantitative Proteomics. Analytical Chemistry. 91 (1), 178-189 (2019).
- Everley, R. A., Kunz, R. C., McAllister, F. E., Gygi, S. P. Increasing Throughput in Targeted Proteomics Assays: 54-Plex Quantitation in a Single Mass Spectrometry Run. Analytical Chemistry. 85 (11), 5340-5346 (2013).
- Shiio, Y., Aebersold, R. Quantitative proteome analysis using isotope-coded affinity tags and mass spectrometry. Nature Protocol. 1 (1), 139-145 (2006).
- Gygi, S. P., et al. Quantitative analysis of complex protein mixtures using isotope-coded affinity tags. Nature Biotechnology. 17 (10), 994-999 (1999).
- Thompson, A., et al. TMTpro: Design, Synthesis, and Initial Evaluation of a Proline-Based Isobaric 16-Plex Tandem Mass Tag Reagent Set. Analytical Chemistry. 91 (24), 15941-15950 (2019).
- Frost, D. C., Feng, Y., Li, L. 21-plex DiLue Isobaric Tags for High-Throughput Quantitative Proteomics. Anal. Chem. 92 (12), 8228-8234 (2020).
- Evans, A. R., Robinson, R. A. Global combined precursor isotopic labeling and isobaric tagging (cPILOT) approach with selective MS(3) acquisition. Proteomics. 13 (22), 3267-3272 (2013).
- Robinson, R. A., Evans, A. R. Enhanced sample multiplexing for nitrotyrosine-modified proteins using combined precursor isotopic labeling and isobaric tagging. Analytical Chemistry. 84 (11), 4677-4686 (2012).
- King, C. D., Dudenhoeffer, J. D., Gu, L., Evans, A. R., Robinson, R. A. S. Enhanced Sample Multiplexing of Tissues Using Combined Precursor Isotopic Labeling and Isobaric Tagging (cPILOT). Journal of Visual Experiments. (123), e55406(2017).
- King, C. D., Robinson, R. A. S. Evaluating Combined Precursor Isotopic Labeling and Isobaric Tagging Performance on Orbitraps To Study the Peripheral Proteome of Alzheimer's Disease. Analytical Chemistry. , (2020).
- Evans, A. R., Gu, L., Guerrero, R., Robinson, R. A. Global cPILOT analysis of the APP/PS-1 mouse liver proteome. Proteomics & Clinical Applications. 9 (9-10), 872-884 (2015).
- Gu, L., Evans, A. R., Robinson, R. A. Sample multiplexing with cysteine-selective approaches: cysDML and cPILOT. Journal of American Society of Mass Spectrometer. 26 (4), 615-630 (2015).
- Ting, L., Rad, R., Gygi, S. P., Haas, W. MS3 eliminates ratio distortion in isobaric multiplexed quantitative proteomics. Nature Methods. 8 (11), 937-940 (2011).
- Frost, D. C., Rust, C. J., Robinson, R. A. S., Li, L. Increased N,N-Dimethyl Leucine Isobaric Tag Multiplexing by a Combined Precursor Isotopic Labeling and Isobaric Tagging Approach. Analytical Chemistry. 90 (18), 10664-10669 (2018).
- Gu, L., Robinson, R. A. High-throughput endogenous measurement of S-nitrosylation in Alzheimer's disease using oxidized cysteine-selective cPILOT. Analyst. 141 (12), 3904-3915 (2016).
- Amin, B., Ford, K. I., Robinson, R. A. S. Quantitative proteomics to study aging in rabbit liver. Mechanisms of Ageing and Development. 187, 111227(2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon