Method Article
Enclos de graphène de cellules de mammifères chimiquement fixes pour microscopie électronique en phase liquide
Dans cet article
Résumé
Présenté ici est un protocole pour l’étiquetage des protéines membranaires dans les cellules de mammifères et le revêtement de l’échantillon avec du graphène pour la microscopie électronique de transmission de balayage de phase liquide. La stabilité des échantillons contre les dommages causés par le rayonnement peut également être étudiée avec ce protocole.
Résumé
Un protocole est décrit pour étudier le récepteur du facteur de croissance épidermique humain 2 (HER2) dans la membrane plasmatique intacte des cellules cancéreuses du sein à l’aide de la microscopie électronique à transmission à balayage (STEM). Les cellules de la lignée cellulaire du cancer du sein des mammifères SKBR3 ont été cultivées sur des micropuces de silicium avec des fenêtres de nitrure de silicium (SiN). Les cellules ont été chimiquement fixées, et les protéines HER2 ont été étiquetées avec des nanoparticules quantiques de point (QDs), utilisant un protocole de liaison biotin-streptavidine en deux étapes. Les cellules ont été recouvertes de graphène multicouche pour maintenir un état hydraté, et pour les protéger des dommages causés par le faisceau d’électrons pendant les STEM. Pour examiner la stabilité des échantillons sous irradiation de faisceau d’électrons, une expérience de série de doses a été effectuée. Des échantillons recouverts de graphène et non enduits ont été comparés. Des dommages causés par le faisceau, sous forme d’artefacts lumineux, sont apparus pour certains échantillons non enduits à la dose d’électrons accrue D,tandis qu’aucun artefact n’est apparu sur des échantillons enduits.
Introduction
L’analyse de la fonction protéique membranaire est essentielle pour la recherche biologique cellulaire et pour le développement de médicaments. Une classe d’expériences importantes implique l’examen des positions des protéines membranaires dans les cellules. Cette information peut être utilisée pour déduire des conclusions sur l’assemblage des protéines dans les complexes protéiques et leurs emplacements spécifiques dans la membrane plasmatique, qui, par l’assemblage dynamique et le démontage, conduit une grande variété de fonctions cellulaires. Entre autres techniques, la microscopie légère (LM) et la microscopie électronique (EM) sont utilisées pour étudier les fonctions protéiques dans les cellules. LM permet l’analyse des cellules entières dans le liquide; toutefois, la résolution est limitée à 200-300 nm pour les classiques et jusqu’à 20 nm pour la microscopie de fluorescence de super résolution dans des conditions pratiques1,2. EM fournit environ 1 Å résolutions3, mais la préparation conventionnelle de l’échantillon nécessite la déshydratation, la coloration des métaux pour améliorer le contraste de l’image, et l’incorporation dans une substance de montage, comme la résine, pour la microscopie électronique de transmission (TEM)4. Pour préserver les échantillons biologiques dans un environnement plus natif, les techniques cryo-EM peuvent être utilisées5,,6. Les échantillons sont rapidement congelés dans de la glace amorphe et, au besoin, sectionnés. Une autre option est la fracturation de gel EM7.
Des techniques em pour étudier les protéines membranaires dans les cellules intactes dans leur état naturel et liquide ont émergé au cours de la dernière décennie8,9,10,11. Une résolution spatiale de 2 nm a été obtenue sur les protéines membranaires étiquetées point quantique (QD) dans des cellules entières cultivées sur une membrane SiN et enfermées par une couche de graphène9.
Ici, les détails d’un protocole pour l’étiquetage des protéines et le revêtement de graphène9,12 sont décrits. Le but de ce protocole est d’analyser la distribution spatiale de HER2 dans la membrane des cellules entières fixes, tout en préservant les cellules dans un état hydraté. Le revêtement au graphène empêche le séchage des cellules dans le vide, et réduit également les dommages de rayonnement13. Cette méthode fournit des informations sur les protéines membranaires étiquetées dans la membrane plasmatique intacte, mais la méthode n’est pas utile pour étudier l’ultrastructure cellulaire comme c’est habituellement fait avec EM.
Le graphène est le nanomatériau le plus fin connu, et se compose d’un seul atome de carbone épaisse feuille cristalline disposée dans un treillis nid d’abeille14. Il a des propriétés uniques, y compris une grande flexibilité et la résistance mécanique. Des recherches récentes ont montré que le graphène sans défaut est imperméable aux gaz et aux liquides, mais les défauts permettent la perméation de l’hydrogène15. Cette fuite peut être réduite en utilisant le graphène multicouche tel qu’il est utilisé ici. Le graphène de bilayer s’est récemment avéré utile comme support pour les échantillons de cryo-EM, améliorant l’homogénéité de la fine couche de glace par rapport à l’oxyde de graphène où seules les couches non uniformes peuvent être formées16. Il a également été démontré que le graphène réduisait les dommages causés par les faisceaux d’échantillons biologiques lors de la microscopie électronique à transmission en phase liquide13,17. Comme une expérience exemplaire, HER2 exprimé dans la lignée de cellules cancéreuses du sein mammifères SKBR3 a été étiqueté avec QDs18 et sa distribution spatiale enregistrée à l’aide de STEM. Les cellules ont été ensemencées sur une micropuce Si avec une membrane SiN transparented’électrons 19. Les micropuces ont été choisies comme support car elles sont robustes, compatibles avec LM et EM, et l’ensemble de la procédure d’étiquetage peut être effectuée directement sur la puce19. Après l’attachement de la cellule, HER2 a été étiqueté avec un protocole d’étiquetage en deux étapes20. Tout d’abord, un composé mimétique d’anticorps anti-HER2 biotinylated21 a été attaché à HER2. Les cellules ont ensuite été fixées chimiquement pour prévenir le regroupement des récepteurs induits par l’étiquette et pour augmenter la stabilité de l’ultrastructure cellulaire. Les QDs streptavidin-enduits ont été plus tard liés au complexe mimétique d’HER2-anticorps. Le signal de fluorescence lumineux et le noyau dense en électrons des QDs ont permis la fluorescence corrélative et la microscopie électronique (CLEM)20. CLEM est particulièrement utile parce que les régions cellulaires d’intérêt pour l’analyse STEM peuvent être sélectionnées à partir des images de microscopie de fluorescence de vue d’ensemble soulignant la localisation de HER2 sur les cellules. Les cellules ont été analysées par microscopie de fluorescence pour identifier les régions cellulaires avec des niveaux élevés de HER2. Par la suite, une feuille de graphène épaisse de 3-5 couches a été transférée sur les cellules pour le revêtement9,22. Par la suite, l’échantillon a été monté dans un porte-spécimens EM. Des données STEM ont été acquises à l’aide du détecteur de champ sombre annulaire (ADF), fournissant des informations sur la distribution spatiale de HER2 à la surface de la cellule par rapport à l’emplacement de la surface cellulaire, mais ne donnant aucune information sur l’ultrastructure de la cellule. Pour déterminer la stabilité de l’échantillon sous irradiation de faisceau d’électrons, les échantillons ont été examinés à dose croissante (D) dans une série d’images. La différence entre les échantillons recouverts de graphène et les échantillons non enduits a été étudiée. Plusieurs types de dommages causés par les radiations ont été évalués.
Le protocole décrit ici utilise la ligne de cellules de cancer du sein des mammifères HER2 surexprimante SKBR3 comme un système modèle pour cibler HER223. Le protocole comprend la préparation d’un échantillon recouvert de graphène et d’un échantillon similaire, mais sans revêtement de graphène à des fins de comparaison. L’expérience est préparée en double puisque la fenêtre SiN peut se briser une fois de temps, et pour obtenir un duplicata expérimental dans la plupart des cas. Le rendement global de la méthode est élevé, ce qui signifie que les micropuces avec des cellules recouvertes de graphène sont généralement obtenues avec une erreur exceptionnelle, même si pas toute la fenêtre SiN peut être recouverte de graphène dans tous les cas. Les doublons ne sont pas décrits dans le protocole.
Le protocole d’étiquetage (étapes 1 à 5) est comparable au protocole d’étiquetage du récepteur du facteur de croissance épidermique dans les cellules fibroblastiques COS7 publiée précédemment24; les détails de ce document sont mentionnés concernant la manipulation des micropuces et l’utilisation des plaques de puits. Le protocole suivant est optimisé pour l’étiquetage HER2, le revêtementde graphène 9et l’examen de la tolérance au rayonnement de l’échantillon.
Protocole
1. Nettoyage des micropuces et du revêtement avec poly-L-lysine (PLL) et protéine de fibronectine (FLP)
- Placer deux micropuces avec une membrane SiN (2,0 x 2,6 mm) dans 50 mL d’acétone. Manipuler soigneusement les micropuces avec le côté plat orienté vers le haut. Évitez de casser les bords à l’aide d’une pince à bec plat. Évitez de toucher la surface supérieure des micropuces lorsque vous manipulez avec une pince à épiler pour éviter la rupture de la fenêtre SiN.
REMARQUE : On peut également utiliser des pinces à épiler recouvertes de polytétrafluoroéthylène ou de carbone pour prévenir les dommages causés par les micropuces. - Laver les copeaux pendant 2 min en secouant soigneusement le bécher, en regardant les micropuces ne pas retourner.
- Transférer les micropuces à 50 mL d’éthanol et laver pendant 2 min en secouant soigneusement le bécher. Assurez-vous que le transfert est rapide afin que l’acétone excédentaire ne sèche pas.
- Laver les micropuces avec 50 mL d’eau pendant 10 min.
- Tremper les micropuces dans un bécher fraîchement préparé de 20 mL d’éthanol.
- Placez les micropuces sur un tissu propre pour le séchage.
- Le plasma nettoie les micropuces avec 11,5 sccm O2 et 35 sccm Ar à 70 mTorr et la radiofréquence (RF) -cible de 50 W pendant 5 min.
- Placez les micropuces dans un capot d’écoulement laminaire pour le travail des cellules stériles.
- Préparer une solution de 0,01% PLL dans l’eau. Préparer une solution de 15 μg/mL FLP dans la solution saline tamponnée par le phosphate (PBS).
- Préparer une plaque de 24 puits sous le capot d’écoulement laminaire et remplir 5 puits individuellement avec 1 mL des solutions dans l’ordre suivant: bien 1 – PLL; puits 2 – eau; puits 3 – eau; bien 4 – FLP; bien 5 – PBS et puits 6 – PBS.
REMARQUE : Effectuez des étapes de lavage en trempant une puce dans le puits indiqué 24 ou 96 pendant quelques secondes à l’aide de pinces à épiler. Effectuer des étapes d’incubation en incuber les micropuces dans le puits 24 ou 96 dans la solution indiquée pour le temps et la température indiqués. Transférer les micropuces à un autre puits en quelques secondes. - Incuber les micropuces dans la solution PLL pendant 5 min. Ensuite, laver les micropuces dans l’eau deux fois.
- Incuber les micropuces en FLP pendant 5 min. Ensuite, lavez les micropuces dans PBS deux fois.
- Transférer les deux micropuces dans les puits (un pour chaque micropuce) d’une nouvelle plaque de puits de 96 préremplie avec 50 μL de milieu sans sérum pour l’ensemencement cellulaire.
- Incuber les micropuces à 37 °C et 5 % de CO2 jusqu’à ce que la suspension cellulaire soit préparée.
2. Cellules d’ensemencement sur micropuces membranaires SiN
- Installez toutes les fournitures et équipements dans un capot d’écoulement laminaire pour assurer le fonctionnement stérile.
- Laver la lignée cellulaire du cancer du sein, SKBR3, dans une fiole de culture cellulaire avec un milieu de croissance une fois. Utilisez le milieu aigle modifié (DMEM) de Dulbecco contenant 10 % de sérum fœtal de veau (SFC) et 1 % d’acides aminés non essentiels (NEA) comme milieu de croissance.
- Incuber les cellules avec 1 ml de la solution de détachement cellulaire jusqu’à ce que les cellules se détachent de la fiole.
- Ajouter 5 ml de milieu de croissance aux cellules détachées de la fiole. Transférer cette suspension dans un tube de centrifugeuse.
- Pipette 20 μL de la suspension cellulaire dans un hémocytomètre pour obtenir la concentration cellulaire. Utilisez la formule suivante.
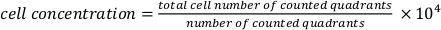 .
. - Préparer une suspension cellulaire dispersée de 2,5 x 105 cellules/mL. Calculer la quantité nécessaire de la suspension de cellule préparée en :
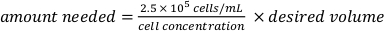
et remplissez-le de milieu de croissance au volume souhaité. - Ajoutez 100 μL de suspension cellulaire aux deux puits d’une plaque de 96 puits contenant les micropuces enduites de PLL et de FLP avec la membrane SiN orientée vers le haut et 50 μL de milieu sans sérum de sorte que chaque puits contient 25 000 cellules.
- Incuber la plaque à 37 °C et 5 % de CO2 pendant 5 min pour attendre que les cellules s’attachent à la micropuce.
REMARQUE : À ce stade, les cellules peuvent se détacher de la micropuce parce qu’elles n’ont pas encore adhéré. - Vérifiez la densité des cellules de la micropuce à l’un microscope inversé. Assurez-vous que les cellules couvrent la fenêtre avec suffisamment d’espace pour s’aplatir et adhérer (voir la figure 1A).
REMARQUE : D’autres cellules peuvent être ajoutées à ce stade si nécessaire. - Transférer les micropuces dans de nouveaux puits contenant 200 μL de milieu de croissance et incuber à 37 °C et 5 % de CO2 pendant la nuit.
- Dans l’après-midi du lendemain, transférer les deux micropuces à un milieu sans sérum (milieu de famine sérique) si les cellules se sont aplaties et ont adhéré à une confluence inspectée visuellement (c.-à-d. la fraction de la surface de la fenêtre couverte de cellules) d’environ 2/3 (voir la figure 1B). Changement au milieu sans sérum au besoin pour amener les cellules dans une condition de départ définie au besoin pour la comparaison entre les différentes expériences25.
- Incuber à 37 °C et 5 % de CO2 pendant la nuit.
REMARQUE : Sachez que les quantités cellulaires peuvent différer selon les taux de croissance et la morphologie cellulaire pour d’autres lignées cellulaires.
3. Étiquetage et fixation HER2
- Préparer les solutions décrites dans le tableau supplémentaire 1.
- Travailler sous le capot d’écoulement laminaire pour assurer le fonctionnement stérile. Utilisez une plaque de 96 puits appelée plaque d’étiquetage I et remplissez 6 puits par micropuce de 200 μL des solutions de la plaque d’étiquetage I : PBS/BSA, PBS/BSA/GS, mimétique anticorps, PBS/BSA, PBS/BSA, PBS/BSA. Utilisez une ligne (une lettre par ligne, p. ex., A1 à A6) de la plaque de puits par micropuce. Chauffer la plaque d’étiquetage à 37 °C.
- Sous une hotte de fumée, préparer une plaque de 24 puits appelée plaque de fixation et remplir 8 puits par micropuce de 500 μL des solutions de plaque de fixation : CB, FA, CB, PBS, PBS, PBS, PBS/glycine, PBS/BSA.
ATTENTION : Le CB est extrêmement toxique par inhalation ou ingestion orale et est dangereux pour les eaux. Travailler sous le capot de fumée avec une protection appropriée et disposer CB selon la fiche de données de sécurité (SDS). Fa est corrosif et nocif pour la peau et la santé. Travaillez sous le capot de fumée et reportez-vous à la SDD pour obtenir des renseignements sur la manipulation et l’élimination. - Préparer une plaque de 96 puits appelée plaque d’étiquetage II et remplir 4 puits par micropuce de 200 μL des solutions de la plaque d’étiquetage II : QDs, PBS/BSA, PBS/BSA, PBS/BSA.
REMARQUE : Ici, une plaque de 24 puits est utilisée pour mieux voir les micropuces dans les puits. Pour utiliser moins d’anticorps mimétiques (étape 3.2) et QDs (étape 3.4), utilisez 96 plaques de puits pour ces étapes.
- Étiquette HER2 dans les cellules.
- Une fois que ces plaques sont prêtes, commencez à étiqueter en plaçant les micropuces dans les puits de la première rangée de la plaque d’étiquetage 1.
- Laver les micropuces avec PBS/BSA dans la plaque de 96 puits marquée comme plaque d’étiquetage I.
- Bloquer les sites non spécifiques pour prévenir la liaison non spécifique de l’anticorps mimétique en cousant avec PBS/BSA/GS pendant 5 min à 37 °C et 5 % de CO2.
- Incuber avec 200 nM anticorps mimétique pendant 10 min à 37 °C et 5% CO2.
- Laver la micropuce trois fois dans PBS/BSA.
- Réparez les cellules.
- Transférer les micropuces à la plaque de fixation de 24 puits dans le capot de fumée.
NOTE: Aucun travail stérile n’est nécessaire à partir d’ici. - Laver une fois avec cb pendant quelques secondes.
- Fixer les cellules avec 3% FA pendant 10 min.
- Laver une fois avec CB et trois fois avec PBS.
- Bloquer les groupes d’aldéhyde libres de FA en cousant avec pbs-glycine pendant 2 min.
- Laver les micropuces avec PBS-BSA une fois.
- Transférer les micropuces à la plaque de fixation de 24 puits dans le capot de fumée.
- Attachez les QD.
- Déplacez les micropuces à la plaque d’étiquetage 96 puits II.
- Incuber avec 20 QDs nM pendant 12 min.
- Laver les micropuces avec PBS/BSA deux fois.
- Conservez les micropuces dans un puits contenant pbs/bsa.
4. Microscopie légère des cellules fixes
- Préparer un plat en verre de 3,5 cm de diamètre avec 2 mL de solution PBS/BSA.
- Prenez les premières micropuces, placez-les à l’envers (cellules orientées vers le bas) dans le plat en verre et placez le plat dans le microscope à fluorescence. Baissez lentement la micropuce dans le liquide pour éviter les dommages sur les cellules.
- Acquérir des images de contraste différentiel (DIC) et de fluorescence de chaque micropuce avec un objectif 40x et le canal de fluorescence approprié.
REMARQUE : Ici, une longueur d’onde d’excitation de 540-580 nm et une longueur d’onde émettrice de 607-683 nm est utilisée pour détecter QD655. - Répétez la procédure pour la deuxième puce.
5. Post-fixation
- Effectuez toutes les étapes sous le capot de fumée.
- Remplir 6 puits par micropuce d’une plaque de puits de 96 avec 200 μL des solutions post-fixation :
CB, GA, CB, PBS, PBS, PBS.
ATTENTION : L’AG est dangereuse pour les eaux, nocive pour la peau, le système respiratoire et les yeux. Travaillez sous le capot de fumée et reportez-vous à la SDD pour obtenir des renseignements sur la manipulation et l’élimination. - Placez les deux micropuces dans leurs puits respectifs avec les cellules orientées vers le haut.
- Laver une fois avec CB.
- Fixer les cellules avec 2% GA pendant 10 min.
- Laver une fois avec CB.
- Laver trois fois avec PBS/BSA.
REMARQUE : La première étape de fixation avec FA fixe déjà la structure biologique, mais un niveau réduit de diffusion des protéines membranaires peut encore se produire, ce qui peut entraîner un regroupement induit par l’étiquette en raison de la présence de streptavidins multiples par QD. Gardez le temps des étapes expérimentales de la fixation FA à l’AG, donc, aussi court que possible pour minimiser la diffusion des protéines dans la membrane. - Conserver dans pbs/bsa pour prévenir les chocs osmotiques et 0,02% d’azide de sodium (NaN3)pour prévenir la croissance bactérienne à 4 °C jusqu’à ce que le revêtement de graphène soit dans une nouvelle plaque de puits. Sceller la plaque de puits avec le film de paraffine pour empêcher le séchage. Les micropuces et les cellules sont stables jusqu’à 2 semaines lorsqu’elles sont stockées à 4 °C.
ATTENTION : Le NaN3 est dangereux pour les eaux et extrêmement toxique par ingestion orale, pour la peau et le système respiratoire. Travaillez sous le capot de fumée et reportez-vous à la SDD pour obtenir des renseignements sur la manipulation et l’élimination.
6. Nettoyage et transfert de graphène sur des cristaux de sel
- Retirer le poly(méthyl méthacrylate) (PMMA)-graphène du PMMA-graphène-sur-polymère (figure 2A).
- Pipette quelques gouttelettes d’eau sur le polymère autour du PMMA-graphène.
REMARQUE : Assurez-vous que le PMMA-graphène se détache facilement du polymère de soutien pendant qu’il flotte sur la surface de l’eau. - Plongez le PMMA-graphène-sur-polymère dans l’eau avec un angle de 30-45° pour libérer le PMMA-graphène.
REMARQUE : Le polymère ne doit être que légèrement mouillé. Le canalisation de trop d’eau sur le polymère soulèvera et éventuellement pliera le PMMA-graphène.
- Pipette quelques gouttelettes d’eau sur le polymère autour du PMMA-graphène.
- Contaminants à base de cuivre d’Etch à l’aide d’une solution de persulfate de sodium (figure 2B).
REMARQUE : Le graphène commercial cultivé sur du papier d’aluminium de cuivre contient souvent des résidus de cuivre sous-micromètre, qui peuvent être enlevés avec une solution d’etchant en cuivre22.- Préparer une solution de 50 mL de 0,42 M de persulfate de sodium dans l’eau.
- Transférer le PMMA-graphène dans la solution de persulfate de sodium avec le côté graphène vers le bas à l’aide d’une lame de verre standard. Le PMMA-graphène flottera sur la solution.
- Laissez le PMMA-graphène dans la solution de persulfate de sodium pendant la nuit.
- Retirez le PMMA-graphène de la solution de persulfate de sodium et placez-le sur l’eau propre à l’aide d’une lame de verre. Laissez-le flotter sur l’eau pendant une demi-heure.
- Répétez l’étape précédente un total de trois fois pour enlever tous les résidus de persulfate de sodium du PMMA-graphène.
- Transférer le PMMA-graphène sur un cristal de chlorure de sodium (NaCl).
- Préparer une solution saturée de NaCl dans l’eau dans une boîte de Pétri.
- Transférer le PMMA-graphène sur la solution NaCl avec le côté graphène vers le bas à l’aide d’une lame de verre.
- Tenez un cristal De NaCl avec des pinces à épiler et prenez le PMMA-graphène flottant.
REMARQUE : La taille du cristal NaCl doit être légèrement supérieure à la taille du PMMA-graphène pour éviter de plier le graphène saillant au bord du sel ou de contacter le graphène avec des pincettes. Dans ces expériences, le cristal nacl de 12 mm x 12 mm x 0,5 mm est utilisé pour ramasser une feuille de graphène de 10 mm x 10 mm et la soutenir par la suite. - Maintenez le PMMA-graphène sur sel verticalement pendant 2 min pour laisser couler l’excès d’eau.
- Laisser sécher le PMMA-graphène sur sel à température ambiante pendant 30 min et cuire au four à 100 °C pendant 20 min pour éliminer complètement l’eau.
- Retirez le PMMA à l’aide d’un lavage d’acétone (figure 2C).
- Préchauffer l’acétone dans une boîte de Pétri en verre à ~50 °C sur une plaque chauffante dans le capot de fumée. Surveillez attentivement la température pour éviter le feu.
- Plongez le PMMA-graphène-sur-sel dans la boîte de Pétri remplie d’acétone et laissez-le dissoudre le PMMA pendant 30 min.
- Répétez l’étape précédente un total de trois fois avec de l’acétone nouvelle et propre.
- Laisser sécher soigneusement l’air du graphène sur sel avant de l’utiliser pour la préparation de l’échantillon.
7. Revêtement de graphène
REMARQUE : La procédure de revêtement de graphène est schématiquement indiquée à la figure 3A.
- Laver une micropuce préparée avec des cellules fixes et étiquetée HER2 dans de l’eau pure pour enlever les résidus de sel du tampon. Placez la puce sur un papier filtre. Les cellules sont visibles sous forme de taches sombres (Figure 3B).
- Coupez le graphène multicouche sur le cristal NaCl en une pièce qui s’adapte à la fenêtre SiN d’une micropuce à l’aide d’une lame de rasoir.
- Préparer un bécher d’eau pure et retirer le graphène du cristal de NaCl en inclinant le cristal d’environ 45° par rapport à la surface de l’eau et en touchant l’eau. Le graphène flottera sur la surface de l’eau (figure 3C).
- Attraper le graphène avec une boucle métallique de la surface de l’eau. Le graphène flottera dans la gouttelette sous la boucle (Figure 3D).
- Touchez la surface supérieure de la puce avec la surface inférieure de la boucle. La puce s’en tiendra à la boucle métallique. Le graphène peut être vu sur le dessus de la puce (Figure 3E).
- Sous un stéréomicroscope, utilisez du papier filtre pour enlever l’eau restante de la micropuce, de sorte que le graphène couvre toutes les cellules de la fenêtre SiN.
REMARQUE : Le graphène se déplace lorsqu’il est effacé. Assurez-vous que le graphène reste sur le dessus de la fenêtre en touchant la puce juste avec le bord du papier filtre. - Utilisez des pinces à épiler pour retirer la puce de la boucle métallique et placez-la sur un papier filtre. Le graphène est visible sous forme de miroitement violet sur la puce (Figure 3F).
- Transférer la puce du papier dans un compartiment Petri.
- Pipette une goutte d’eau dans l’un des compartiments libres et fermez le couvercle pour fournir une atmosphère saturée d’eau.
- Sceller le plat du compartiment avec du film de paraffine et conserver au réfrigérateur à 4 °C si nécessaire pour d’autres mesures.
8. STEM
- Ajuster le STEM à 200 kV d’énergie de faisceau à l’aide d’un échantillon d’alignement/essai pour une taille de sonde d’au moins 0,2 nm en ajustant les lentilles condenseur, un courant de sonde I = 180 pA (des informations sur le courant de la sonde pour différents paramètres de microscope sont fournies par le fabricant dans un délai de 5% de précision) et un semi-angle de faisceau de convergence de 13,2 mrad en insérant une ouverture. Réglez la plage de semi-angle d’ouverture du détecteur STEM ADF (intérieur et extérieur) à 68-280 mrad en ajustant les paramètres de l’objectif du projecteur. Définissez la taille de l’image STEM à 2048 x 2048 pixels, et le temps de séjour du pixel t = 6 μs.
- Chargez la micropuce avec des cellules recouvertes de graphène dans un support de spécimen standard pour TEM de telle sorte que les cellules sont orientées vers le haut.
- Chargez le support dans le microscope électronique.
- Acquérir une image de vue d’ensemble au grossissement (M) = 800x (figure 4) en utilisant les paramètres à l’étape 8.1.
- Identifier une région d’intérêt sur une cellule.
- Imagez les QDs avec un détecteur ADF à M = 80 000x à la taille d = 1,3 nm ( Figure4) à l’aide des paramètres à l’étape 8.1.
- Acquérir une image de la zone après exposition avec un grossissement inférieur (ici, M = 50 000x) pour montrer la zone exposée (Figure 4).
- Calculer la dose d’électrons
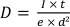
où e est la charge élémentaire. Avec les paramètres donnés ci-dessus,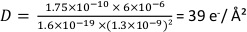
et une erreur de 5%. - Acquérir 20 images pour une série de doses avec les mêmes paramètres, mais avec t = 60 μs résultant en une dose accumulée de
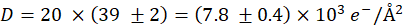 .
. - Pour confirmer la présence de graphène, passez le microscope à TEM à M = 1 200x, sélectionnez une région près d’une cellule et passez en mode diffraction. Enregistrer un modèle de diffraction à un moment d’exposition de 0,5 s, 2048 x 2048 x 3 pixels, et une ouverture de zone sélectionnée de 50 μm (figure 4).
- À la fin de la séance, retirer l’échantillon du microscope, remettre la puce dans le plat du compartiment, sceller le plat avec du film de paraffine et le conserver au réfrigérateur à 4 °C si nécessaire pour d’autres mesures.
- Sélectionnez la deuxième puce préparée de la même manière que la première micropuce, mais sans revêtement de graphène.
- Répétez les étapes 8.2-8.11 mais maintenant pour cet échantillon. Enregistrer un modèle de diffraction avec les mêmes paramètres que dans ce qui précède, comme comparaison (Figure 4).
9. Analyse
REMARQUE : Pour la détection automatisée des positions QD dans une image STEM, l’analyse utilise un plugin de conception locale pour ImageJ (NIH), tel que décrit ailleurs20. Le plugin est disponible sur demande.
- Le logiciel applique automatiquement les étapes suivantes pour détecter les particules d’une image STEM :
- Appliquez un filtre gaussien pour réduire le bruit des pixels.
- Appliquez un filtre à bande Fast Fourier Transform (FFT) pour obtenir uniquement des nanoparticules.
- Définissez un seuil pour binariser l’image.
- Utilisez un diamètre de particules de 10 nm avec un facteur de tolérance de 2 pour la détection des particules.
- Détecter les positions des particules.
- Mesurez la distance de centre-centre entre dix paires de QD par série. Ici, 10 distances de taille variable ont été mesurées par série.
- Calculer le changement relatif de la distance des particules en comparant chaque distance de particules à la distance de la première image à l’aide de :
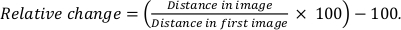 .
.
Résultats
La figure 1A montre les cellules ensemencées de telle sorte que la fenêtre soit couverte mais avec suffisamment d’espace pour leur permettre de s’aplatir et d’adhérer, ce qui conduit à une confluence d’environ 2/3rd (figure 1B). Dans le cas où trop de cellules sont ensemencées sur une micropuce (figure 1C),il n’y a pas suffisamment d’espace pour que toutes les cellules adhèrent à la micropuce. La figure 1D montre la même micropuce après 24 h. Plus de la moitié des cellules ne se sont pas aplaties. D’autre part, si trop peu de cellules sont ensemencées (figure 1E),la fenêtre SiN se retrouvera avec un grand espace vide après 24 h comme on le voit dans la figure 1F. La figure 1G montre l’image DIC des cellules de la figure 1A et de la figure 1B. La figure 1H montre l’image de fluorescence correspondante fausse couleur en jaune indiquant l’étiquetage réussi de HER2.
Les données STEM représentatives sont indiquées à la figure 4. Les cellules SKBR3 recouvertes de graphène (colonne de gauche) et non enduites (colonne de droite) ont été étudiées. Figure 4A,B montrent des images de vue d’ensemble M = 800x des cellules de la fenêtre. Les zones indiquées sous forme d’encarts ont été représentées à M = 80 000x au cours de la série de doses, voir figure 4C,D. Les QDs sont visibles comme des points lumineux ici. La figure 4E et la figure 4F montrent des images m = 50 000x agrandies acquises à l’emplacement des rectangles de la figure 4C,D. Les deux images ont été enregistrées après l’acquisition d’une série de doses avec D = (7,8 ± 0,4) x 103 e-/Å2. La série de doses a été enregistrée à M = 80 000x. Les zones exposées peuvent être reconnues comme des rectangles, auquel cas le rectangle était clairement visible pour l’échantillon non enduit (figure 4F).
Pour vérifier la présence de graphène, des modèles de diffraction des zones sans cellules, mais avec ou sans graphène sur la fenêtre SiN ont été acquis. La structure hexagonale du graphène a été observée dans le modèle de diffraction de l’échantillon recouvert de graphène (figure 4G), alors qu’il était absent pour l’échantillon non enduit (figure 4H). Le modèle de diffraction du graphène à cristaux simples aura une symétrie six fois due à la structure hexagonale fortement ordonnée du graphène. Ainsi, la structure hexagonale indique la présence de graphène sur les échantillons.
Pour étudier l’effet de l’éclairage du faisceau d’électrons sur l’échantillon, des images STEM ont été acquises dans une série d’images avec une dose d’électrons qui s’accumule. Les résultats représentatifs des échantillons non enduits et recouverts de graphène sont indiqués respectivement à la figureA-D et à la figure 5E-G. Figure 5 Toutes les données ont été acquises au bord de la cellule, où la cellule est la plus plate, et la structure observée est donc la plus proche de la membrane SiN. L’exposition d’échantillons non enduits a conduit à l’apparition de structures lumineuses sur les surfaces cellulaires à D = (1,9 ± 0,1) x 103 e-/Å2 (figure 5B). Ces structures sont devenues plus grandes avec des doses plus élevées, de sorte qu’elles étaient clairement visibles dans la dernière image de la série à D = (7,8 ± 0,4) x 103 e-/Å2 (Figure 5C,D). Ces taches n’apparaissaient sur aucun des échantillons recouverts de graphène (figure 5E-G).
D’autres micropuces ont été préparées et examinées à l’aide du protocole décrit ci-dessus. Six échantillons enduits et sept échantillons non enduits ont été étudiés au total. Deux des sept échantillons non enduits ont montré ces artefacts. Aucun des échantillons enduits n’a montré de points lumineux supplémentaires.
Comme une autre mesure des dommages causés par les radiations, la distance entre les QDs a été examinée. Si des dommages structurels devaient se produire, on s’attendrait à ce que les distances entre les QD changent. Les changements dans les distances ont été mesurés pour différentes paires de QDs avec accumulation de D pour une gamme de distances de paire. La figure 5H montre que le changement relatif des particules pour les échantillons non enduits est resté inférieur à 1,3 % en moyenne, tandis que la distance relative moyenne est demeurée inférieure à 0,8 % pour les échantillons enduits. On peut donc conclure que le revêtement de graphène a stabilisé l’échantillon, mais que les échantillons séchés sans revêtement de graphène étaient également remarquablement stables.

Figure 1 : Ensemencement cellulaire sur une fenêtre SiN d’une puce en silicium et étiquetée HER2. (A) Image exemplaire d’une région de fenêtre SiN avec des cellules SKBR3 5 min après l’ensemencement sur la puce. (B) Les mêmes cellules se propagent sur la même fenêtre SiN après 24 h. (C) cellules SKBR3 sur une micropuce Si 5 min après l’ensemencement. (D) Mêmes cellules qu’à (C) après 24 h. Les cellules ne s’aplatissaient pas correctement car il y avait trop de cellules sur les copeaux lors de l’ensemencement. (E) Cellules sur une micropuce 5 min après l’ensemencement. (F) Seules peu de cellules étaient visibles sur la fenêtre parce que trop peu de cellules étaient ensemencées sur la micropuce. (G) Image DIC des mêmes cellules SKBR3 après l’étiquetage QD de HER2. (H) Superposition d’image de DIC et image de fluorescence correspondante des cellules SKBR3 étiquetées avec HER2-QD655 (fausse couleur en jaune). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
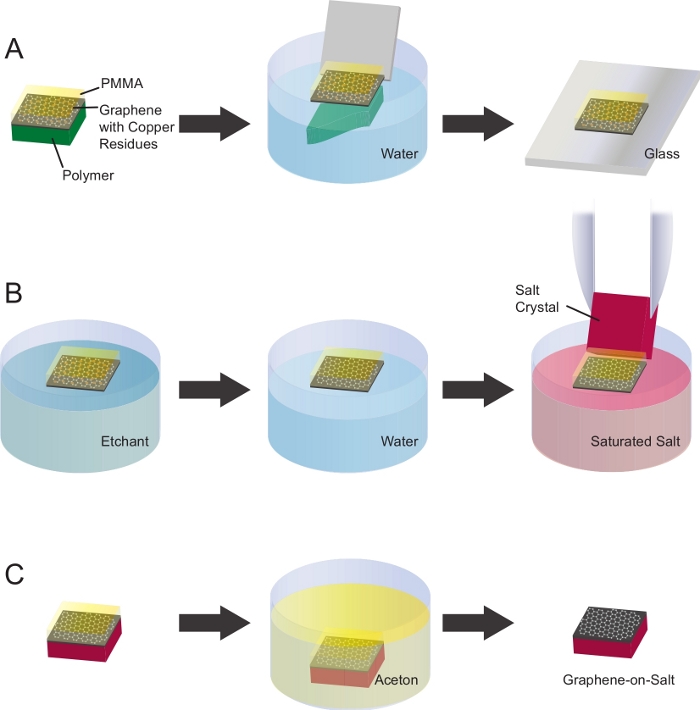
Figure 2 : Nettoyage et transfert du graphène sur les cristaux de NaCl. (A) PMMA-graphène-sur-polymère est immergé dans l’eau pure pour libérer le graphène de PMMA. Le PMMA-graphène peut être attrapé avec une lame de verre. (B) Les contaminations à base de cuivre ont été gravées à l’aide d’une solution de persulfate de sodium. Pour nettoyer le graphène, il a été transféré dans un bécher contenant de l’eau déionisée. Ces étapes ont été répétées 3 fois. Le PMMA-graphène a ensuite été transféré à la solution saturée de NaCl dans l’eau pure. Un cristal de NaCl a été utilisé pour ramasser le graphène de la solution de sel. (C) Le PMMA-graphène sur le cristal de NaCl a été séché pendant 30 min à température ambiante. PMMA a été supprimé en incuber le bloc en acétone pendant 30 min. Veuillez cliquer ici pour voir une version plus grande de cette figure.
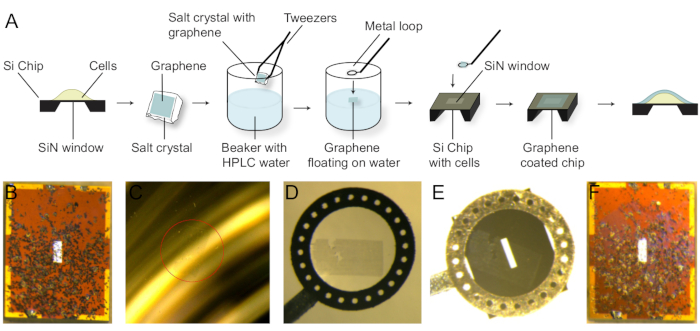
Figure 3 : Revêtement en graphène des cellules ensemencées sur une micropuce Si. (A) Procédure de revêtement de graphène. Le graphène sur le cristal nacl est libéré sur la surface de l’eau. La pièce de graphène est ensuite capturée avec une boucle métallique et transférée sur la micropuce Si. (B) Micropuce (2,0 x 2,6 mm) avec une fenêtre SiN de dimensions 400 x 160 μm sans graphène. Les cellules SKBR3 étaient visibles sous forme de taches sombres. (C) Graphène (cercle rouge) flottant à la surface d’un bécher rempli d’eau. (D) Graphène pris avec une boucle métallique. (E) La puce attachée à la gouttelette d’eau de sorte que le graphène était au-dessus de la fenêtre SiN. (F) Micropuce après revêtement de graphène. Le graphène était visible comme un scintillement violet. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : STEM des cellules SKBR3 recouvertes de graphène et non enduites sur la fenêtre SiN. (A) Image STEM d’un échantillon recouvert de graphène acquis à un M = 800x. (B) Image STEM d’un échantillon non enduit acquis à M = 800x. (C) M = 80 000x image enregistrée à la position du rectangle bleu dans A. Lignes jaunes représente des exemples de distances mesurées à l’intérieur de la particule. (D) M = Image 80 000x enregistrée à la position du rectangle bleu dans B. La ligne jaune représente des exemples de distances mesurées à l’intérieur des particules. (E) M = image 50 000x de la même région que C exposée à D = (7,8±0,4) x 103 e-/Å2. (F) M = 50 000x image de la même région que D et exposée à D = (7,8±0,4) x 103 e-/Å2. La zone exposée a été clairement vue. (G) Modèle de diffraction d’un échantillon recouvert de graphène provenant d’une zone sans cellules. La symétrie six fois du graphène est visible sous forme de points lumineux. (H) Modèle de diffraction d’un échantillon sans graphène ne montrant pas de taches lumineuses 6 fois. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
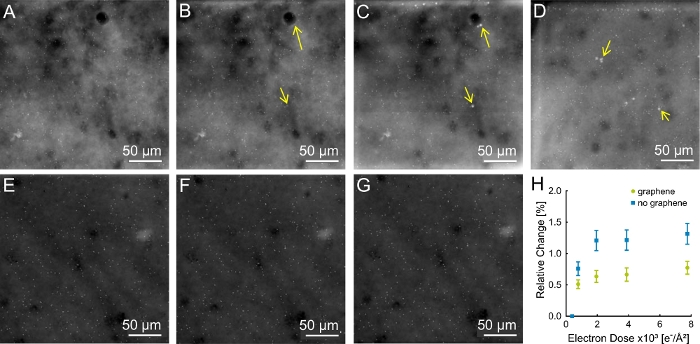
Figure 5 : Artefacts sur les échantillons sans revêtement de graphène. (A) Premières images des régions sur des échantillons sans revêtement de graphène acquis à M = 80 000x, et exposés à D = (0,39 ± 0,02) x 102 e-/Å2. (B) Images avec les premiers artefacts surgissant à D = (1,94 ± 0,1) x 103 e-/Å2 (flèches jaunes). (C, D) Dernière image de la série acquise avec D = (7,8 ± 0,4) x 103 e-/Å2. Les artefacts étaient visibles comme des points lumineux. (E-G) Échantillons enduits de graphène sans artefacts. EE) D = (0,39 ± 0,02) x 102 e-/Å2 (F) D = (1,94 ± 0,1) x 103 e-/Å2 (G) D = (7,8 ± 0,4) x 103 e-/Å2. (H) Variation relative des distances de particules pour les échantillons enduits de graphène et non enduits. Deux des échantillons non enduits montrant des artefacts, dont l’un est indiqué dans (A-C), et la dernière image du deuxième échantillon en D, ainsi que trois échantillons enduits ont été analysés (un est montré dans E-G). Au total, dix paires de QD ont été examinées par échantillon avec des distances variant entre 250 nm et 3 μm. Le changement relatif reflète la moyenne de toutes les mesures dans un groupe. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Tableau supplémentaire 1 : Recettes de solutions et tampons. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Pour mieux comprendre la fonction protéique, il est important d’obtenir des informations sur les emplacements des protéines dans la membrane plasmatique des cellules intactes. Les méthodes d’obtention de ces informations incluent la microscopie de fluorescencesuper-résolution 1,2. Bien que la microscopie à super résolution se soit développée au cours des dernières années, sa résolution est encore limitée à environ 20 nm pour les conditions pratiques des expériences cellulaires, tandis que les protéines de récepteur typiques ont des tailles de l’ordre de 1-10 nm. L’imagerie des protéines sur le niveau unicellulaire et de molécule unique avec une résolution suffisante pour visualiser les protéines est possible avec EM. Mais en raison de la section, les méthodes conventionnelles em ne laissent généralement pas la cellule intacte26, ce qui conduit à la perte d’informations importantes sur le contexte et la distribution spatiale des protéines dans la membrane plasmatique. Méthodes pour les cellules entières avec cryo-TEM ont été développés6, il est possible de combiner l’étiquetage des protéines avec cryo-EM27, également cryo-STEM a été démontré28. Cependant, les flux de travail cryo-EM sont optimisés pour l’étude de l’ultrastructure cellulaire et de la structure protéique, et pas tellement pour analyser les distributions spatiales des protéines membranaires. Le séchage critique de point est une autre méthode de préparation de cellules entières, mais les échantillons sont soumis à plusieurs étapes de séchage, et la technique est très longue29. Les protéines membranaires ont également été examinées par l’intermédiaire de la fracture de congélation7. Dans cette méthode, les cellules sont fixes, congelées et fracturées. Les parties fracturées sont répliquées par des couches de carbone et de platine, et l’échantillon biologique est retiré. Les répliques peuvent ensuite être analysées avec EM30. L’analyse des cellules entières est impossible avec la fraction de gel parce que l’information sur la distribution des protéines dans la membrane dans le contexte de la cellule entière est perdue.
La méthode présentée ici permet d’étudier la membrane cellulaire sans avoir besoin de trancher finement le spécimen9,31. Les cellules sont conservées intactes de sorte que la localisation des protéines membranaires est visible à partir des images de fluorescence, qui sont corrélées avec les images EM. L’étude des protéines au niveau des cellules simples et des molécules uniques dans des cellules intactes à l’état hydraté s’est avérée possible avec une résolution de 2 nm à l’aide de PROTÉINES étiquetées QD à l’aide de cette méthoded’enclos de graphène 9. Il est crucial de maintenir les cellules dans leur état d’origine, car elle préserve la distribution spatiale des protéines membranaires de telle sorte que des analyses soient possibles au niveau des cellules uniques et des molécules uniques, ce qui est important pour comprendre les fonctions protéiques, et développer de nouveaux médicaments pour les approches thérapeutiques.
Un autre aspect critique de l’imagerie des échantillons biologiques avec EM est les dommages de rayonnement des échantillons causés par le faisceau d’électrons. Les solutions incluent souvent la réduction de la dose d’électrons autant que possible ou diverses méthodes de revêtement, telles que l’encapsulation du spécimen entre de fines couches de carbone32. Notre méthode montre que le revêtement de graphène réduit les artefacts induits par le faisceau qui émergent sur la surface cellulaire pour les échantillons non enduits. L’examen des échantillons biologiques chimiquement fixes et recouverts de graphène est possible sous l’irradiation du faisceau d’électrons à 200 keV d’énergie du faisceau jusqu’à D = (7,8±0,4) x 103 e-/Å2 sans dommages causés par les radiations, tels que des taches lumineuses, apparaissant sur l’échantillon. Par rapport à d’autres méthodes em qui impliquent la préparation élaborée d’échantillon, par exemple, la coloration, l’incorporation, (cryo-) sectionnement, la fracturation, etc., la méthode décrite ici prend moins de temps. L’étiquetage des protéines est effectué en quelques heures, et le revêtement de graphène ne nécessite qu’environ 15 minutes pour les chercheurs formés. La préparation de l’échantillon est comparable aux procédures nécessaires pour la microscopie par fluorescence.
Le protocole peut être modifié en quelques étapes. Le graphène sur la micropuce peut également être séché à l’air pour s’assurer que le graphène ne bouge pas lorsqu’il est effacé avec un papier filtre. Si le graphène est contaminé par du sel, il est possible de le laisser flotter à la surface de l’eau pendant environ une heure pour dissoudre le sel et ainsi minimiser la contamination. La contamination par le cuivre ou l’AMPM sur le graphène peut être réduite en prolongeant les étapes de gravure correspondantes du protocole. D’autres méthodes de revêtement de graphène ont été décrites où, par exemple, le graphène-PMMA a été directement déposé sur les cellules, et le PMMA a été enlevé par lavage dans l’acétone par la suite33. Dans notre méthode, PMMA a été enlevé avant le revêtement pour éviter tout dommage possible aux cellules causées par des étapes supplémentaires de lavage de l’acétone. NaCl a été choisi comme un substrat ici parce qu’il est plat, de sorte qu’il ne rides pas le graphène, et il se dissout dans l’eau pour libérer le graphène9. En outre, il peut être coupé dans la taille désirée et aucun résidu de substrat ne sont laissés sur le graphène. Mais en tenant compte de ces critères, d’autres substrats comme le chlorure de potassium peuvent éventuellement être utilisés aussi.
Pour réduire le risque de regroupement potentiel induit par l’étiquette, l’étape de fixation de l’AG peut être mise en œuvre directement après la fixation de la FA, après quoi toutes les protéines membranaires sont immobilisées. La première étape de fixation avec FA fixe déjà la structure biologique, mais un niveau réduit de diffusion des protéines membranaires peut encore se produire34, peut-être conduisant à l’étiquetage induit clustering en raison de la présence de Streptavidin multiple par QD. La fixation avec l’AG peut conduire à un signal d’autofluorescence pendant LM, et est, par conséquent, fait après LM dans le protocole décrit, mais peut être réduite comme décrit ailleurs34. Tampon cacodylate est tout à fait toxique, et d’autres fixatifs peuvent être utilisés ainsi, mais le cacodylate est utilisé ici car il est un tampon couramment utilisé pour les protocoles EM, évite les précipités, empêche la croissance des bactéries et des champignons, et est compatible avec les ions de calcium qui sont nécessaires pour préserver l’intégrité ultrastructurale des membranes lipidiques35. Si nécessaire, le tétroxide d’osmium peut être utilisé comme fixation supplémentaire pour stabiliser les lipides. Cela permettrait d’améliorer le contraste de la structure cellulaire, mais aussi ajouter un autre métal au système et de réduire le contraste obtenu sur les QDs.
Le protocole décrit ici contient de nombreuses étapes qui nécessitent une bonne instruction. Une certaine formation est nécessaire avant de manipuler des micropuces pour éviter de gratter la surface SiN des micropuces et pour éviter la rupture. Comme mentionné précédemment, il est recommandé de préparer des puces dans les doublons que la fenêtre SiN peut se briser de temps en temps. L’obtention du nombre requis de cellules sur une puce nécessite également une certaine expérience. Le revêtement des cellules avec du graphène a besoin d’une certaine formation car il peut être difficile de trouver le bon angle d’inclinaison pour flotter le graphène sur l’eau. Lorsque vous attrapez le graphène de l’eau, il peut également être difficile de voir le graphène mince. Dès que le graphène est sur la puce, l’excès d’eau doit être effacé avec un papier filtre. Cela ne doit être fait qu’avec la pointe d’un papier filtre afin d’éviter de retirer le graphène de la micropuce.
Le revêtement de graphène a empêché les artefacts d’apparaître sur l’échantillon. Mais pour D < 4 x 102 e-/Å2 également aucun artefact n’a émergé pour l’échantillon non enduit, et les artefacts sont apparus pour 2 échantillons non enduits seulement. Ainsi, les examens des cellules non enduites semblent également possibles, bien qu’il serait préférable d’utiliser le graphène et d’éviter le risque de formation d’artefacts. La composition de ces artefacts peut être analysée à l’avenir pour donner des conseils sur la façon d’empêcher leur formation. En ce qui concerne la stabilité structurelle des cellules, seule une amélioration mineure du revêtement de graphène a été observée. Les cellules fixes ont apparemment été stabilisées dans les zones minces examinées, où leur structure se trouvait à proximité de la membrane SiN. Ce que nous n’avons pas examiné ici, cependant, étaient le séchage des artefacts qui sont connus pour se produire pour les échantillons cellulaires lorsqu’ils sont exposés au vide4. Le séchage des cellules conduirait à un rétrécissement des cellules de sorte que les distances QD changeraient aussi en conséquence. Pour la dose d’électrons utilisée ici, la distance des QD des échantillons recouverts de graphène et non enduits est restée stable. D’autres études sont nécessaires pour examiner l’effet du revêtement de graphène sur les cellules pour em.
Une limitation de cette méthode est que la fixation chimique des cellules est nécessaire; par conséquent, aucune expérience cellulaire vivante ne peut être effectuée. Mais dans le cas où l’étiquetage n’est pas nécessaire et les cellules avec une stabilité structurelle plus élevée sont utilisées, par exemple les bactéries, puis les cellules non fixées peuvent être enfermés dans le graphène pour EM36, mais avec une tolérance à la dose d’électrons différente. En outre, les protéines ne sont pas directement détectables, de sorte QDs sont nécessaires pour visualiser les protéines. La méthode bénéficierait de petites étiquettes. Un point de discussion est de savoir s’il est bon ou mauvais que l’ultrastructure n’est pas clairement visible. Notre méthode est similaire à celle de la microscopie de fluorescence où seules les protéines sélectionnées sont visibles37. Augmenter la visibilité de l’ultrastructure ajouterait également beaucoup plus d’informations à l’image, puis à un moment donné empêcher la détection des positions individuelles de l’étiquette. En outre, la méthode décrite ici est pour une espèce de protéine, et des ajouts du protocole sont nécessaires pour être en mesure d’étiqueter plusieurs protéines. Enfin, la méthode fonctionne lorsqu’une petite molécule de liaison à haute affinité, comme l’anticorps mimétique21 ou lenanocorps 38, est disponible. Les anticorps couramment utilisés sont beaucoup plus grands et empêcheraient la détection de l’état fonctionnel des sous-unités protéiques en oligomères.
Notre méthode est utile pour étudier la fonction protéique sur des cellules entières en utilisant em tout en gardant les cellules à l’état hydraté. Il est facilement possible d’examiner des séries de cellules. D’autres types de cellules et de protéines peuvent également être étudiés. Si l’étiquetage des protéines n’est pas nécessaire, un sous-ensemble du protocole peut être utilisé pour le revêtement en graphène d’une grande variété de spécimens biologiques. La capacité d’étudier les cellules entières est pertinente dans la recherche cellulaire pour comprendre les corrélations de la fonction protéique membranaire au niveau moléculaire.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions D. B. Peckys pour son aide dans le protocole de culture cellulaire, F. Weinberg d’avoir examiné le manuscrit, T. Trampert pour son aide dans les expériences et les personnages, S. Smolka pour son aide avec les personnages, et E. Arzt pour son soutien par l’intermédiaire de l’INM. Cette recherche est financée par Else Kröner-Fresenius-Stiftung.
matériels
| Name | Company | Catalog Number | Comments |
| Acetone for HPLC (min. 99.8 %) | Th. Geyer GmbH + Co. KG, Renningen, Germany | 2626-1L | |
| AL54 analytical balance | Mettler-Toledo GmbH, Giessen, Germany | 30029077 | |
| Albumin Fraction V, biotin-free, ≥ 98 %, for molecular biology | Carl Roth GmbH + Co KG, Karlsruhe, Germany | 0163.2 | |
| Anti-HER2 Affibody Molecule, biotin conjugated 20 µM | Affibody, Solna, Sweden | 10.0817.02.0001 | |
| atomic resolution analytical microscope JEM-ARM200F | JEOL (Germany) GmbH, Freising, Germany | ||
| Boric Acid | Sigma-Aldrich, Merck, Darmstadt, Germany | B6768-500G | |
| Cacodylic acid sodium salt trihydrate ≥ 98 % | Carl Roth GmbH + Co KG, Karlsruhe, Germany | 5169.1 | |
| Cell Culture Flasks, PS, treated, sterile, Filter screw cap 50ml | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 7696781 | |
| Cell Culture Flasks, PS, treated, sterile, Filter screw cap 250ml | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 7696782 | |
| Cell Culture Multiwell plates, treated, 24-well | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 7696792 | |
| Cell Culture Multiwell plates, treated, 96-well | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 7696794 | |
| CELLSTAR Cell Culture Flasks TC treated, 250 ml | Greiner Bio-One GmbH, Frickenhausen, Germany | 658170 | |
| CELLSTAR Cell Culture Multiwell Plates 96-well | Greiner Bio-One GmbH, Frickenhausen, Germany | 655180 | |
| CELLSTAR Centrifuge Tubes 15 ml sterile | Greiner Bio-One GmbH, Frickenhausen, Germany | 188271 | |
| CELLview cell culture dish, PS, 35/10mm, glass bottom, 1 compartment | Greiner Bio-One GmbH, Frickenhausen, Germany | 627861 | |
| Centrifuge 5418 | Eppendorf, Wesseling-Berzdorf, Germany | 5401000010 | |
| Centrifuge Tubes 50 ml sterile | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 7696705 | |
| Corning CellStripper non-enzymatic cell dissociation solution | Corning, NY, USA | 25-056-CI | |
| D(+)-Saccharose min. 99,7 %, powdered | Th. Geyer GmbH + Co. KG, Renningen, Germany | 9286.1 | |
| Disposable Hemocytometer C-Chip Neubauer improved | Carl Roth GmbH + Co KG, Karlsruhe, Germany | PK36.1 | |
| Easypet | Eppendorf, Wesseling-Berzdorf, Germany | 4430000018 | |
| Eppendorf Research plus pipettes variable, 0.1 - 2.5 µl | Eppendorf, Wesseling-Berzdorf, Germany | 3123000012 | |
| Eppendorf Research plus pipette variable, 2 -20 µl | Eppendorf, Wesseling-Berzdorf, Germany | 3123000039 | |
| Eppendorf Research plus pipette variable, 10 - 100 µl | Eppendorf, Wesseling-Berzdorf, Germany | 3123000047 | |
| Eppendorf Research plus pipette variable, 100 - 1000 µl | Eppendorf, Wesseling-Berzdorf, Germany | 3123000063 | |
| Ethanol absolute for HPLC (min. 99.9 %) | Th. Geyer GmbH + Co. KG, Renningen, Germany | 2222-1L | |
| Fibronectin-like engineered protein*poly mer-plus | Th. Geyer GmbH + Co. KG, Renningen, Germany | F8141 | |
| Fluorescence Microscope Leica DMI6000 | Leica Microsystems GmbH, Wetzlar, Germany | ||
| Gibco Dulbecco's Modified Eagle Medium (DMEM), high glucose, GlutaMAX Supplement, pyruvate | FisherScientific, Hampton, NH, USA | 31966021 | |
| Gibco Fetal Calf Serum (FCS) | FisherScientific, Hampton, NH, USA | 10099141 | lot number: 1751893 |
| Gibco Goat Serum, New Zealand origin | FisherScientific, Hampton, NH, USA | 16210064 | lot number: 1788320 |
| Gibco Phosphate buffered saline (PBS), pH 7.4 | ThermoFisher Scientific, Waltham, MA, USA | 10010015 | |
| Galaxy 48R CO2 Incubator | New Brunswick, Eppendorf, Wesseling-Berzdorf, Germany | CO48310001 | |
| Gatan Model 950 Solarus— Plasma Cleaner | Gatan GmbH, München, Germany | ||
| Glutaraldehyde solution Grade I, 25% in H2O, specially purified for use as an electron microscopy fixative | Sigma-Aldrich, Merck, Darmstadt, Germany | G5882 | |
| Glycine ≥ 98.5 % Ph.Eur., USP, BP | Carl Roth GmbH + Co KG, Karlsruhe, Germany | T873.1 | |
| Inverted Laboratory Microscope Leica DM IL LED | Leica Microsystems GmbH, Wetzlar, Germany | DM IL LED | |
| Hot plate | Heidolph Instruments GmbH & CO. KG, Schwabach, Germany | MR 3002 | |
| MEM non-essential Aminoacids (NEAAs) 100x W/O L-Glutamine | Biowest, Nuaillé, France | X0557-100 | |
| Microscope glass slides 76 mm x 26 mm | DWK Life Sciences GmbH, Wertheim/Main | ||
| micro tubes (1.5 ml, 2 ml) non-sterile | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 1.5 ml: 7696751, 2 ml: 7696752 | |
| MSc-Advantage— Class II Biological Safety Cabinet | ThermoFisher Scientific, Waltham, USA | ||
| Ocean Microchips SiN window 400x150 µm, 200 nm spacer | DENSsolutions, Delft, The Netherlands | ||
| Omnifix Single-use syringe Luer Lock Solo (10 ml, 20 ml, 50 ml) | B. Braun Melsungen AG, Melsungen, Germany | 10 mL: C542.1 20 ml: T550.1 | purchased via Carl Roth GmbH&Co. KG, Karlsruhe |
| Oven | Memmert GmbH + Co. KG, Schwabach, Germany | VO 200 | |
| Parafilm | VWR, Darmstadt, Germany | #291-0057 | |
| Paraformaldehyde 16 % solution, EM grade | Electron Microscopy Sciences, Hatfield, PA, USA | 15710 | |
| Perfekt-Kescher with handle | Plano GmbH, Wetzlar, Germany | T5112 | |
| Pipette tips with aerosol barrier in racks, non-sterile | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 2-200µl: 7695892 | |
| Pipette tips with aerosol barrier in racks, sterile | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 0.1-10 µl: 7695880 2-100 µl: 7695883 100-1000 µl: 7695886 1250 µl XL: 7695887 | |
| Poly-L-Lysine 0.01 % solution mol.WT.70.000 - 150.000 | Th. Geyer GmbH + Co. KG, Renningen, Germany | P4707 | |
| Qdot 655 Streptavidin Conjugate 1 µM | Invitrogen, ThermoFisher Scientific, Waltham, MA, USA | Q10121MP | |
| Razor blade, Gem Uncoated 3 Facet, Steel Back, Degreased | Personna, AccuTec Blades, Verona, VA, USA | 94-0451 | |
| round filter paper, ashless, Grade 589/3 blue ribbon, diam. 150mm | Schleicher & Schuell, Dassel, Germany | 300212 | |
| Routine stereo Microscope Leica M60 | Leica Microsystems GmbH, Wetzlar, Germany | M60 | |
| Serological ROTILABO pipettes, sterile (1 ml, 2 ml, 10 ml) | Th. Geyer GmbH + Co. KG, Renningen, Germany | 1 ml: N231.1 2 ml: N236.1 10 ml: ET30.1 | |
| Serological pipettes, sterile, (1 ml, 2 ml, 10ml) | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 1ml: 7695550 2ml: 7695551 10ml: 7695553 | |
| Serological pipettes, sterile, (5 ml, 25 ml, 50 ml) | Labsolute, Th. Geyer GmbH + Co. KG, Renningen, Germany | 5 mL: 7695552 25ml: 7695554 50ml: 7695555 | |
| Shaking water bath SW22 | Julabo GmbH, Seelbach, Germany | 9550322 | |
| Sodium chloride | Carl Roth GmbH + Co KG, Karlsruhe, Germany | HN00.1 | |
| Sodium chloride crystals, cleaved 12 mm x 12 mm x 0.5-1 mm | International Crystal Laboratories, Garfield, NJ, USA | 9750 | |
| Sodium tetraborate decahydrate, ACS reagent, ≥ 99.5 % | Sigma-Aldrich, Merck, Darmstadt, Germany | S9640-500G | |
| Sodium persulfate | carl Roth GmbH + Co KG, Karlsruhe, Germany | 4365.2 | |
| syringe filters ROTILABO PES, sterile 0.22 µm | Carl Roth GmbH + Co KG, Karlsruhe, Germany | P668.1 | |
| Trivial Transfer Graphene 3-5layers, 1 cm x 1 cm | ACS material, Pasadena, CA, USA | TTG30011 | |
| Tweezers Dumoxel 03 | Manufactures D’Outils Dumont SA, Montignez, Switzerland | 0103-7-PO | |
| Tweezers Inox 02 | Manufactures D’Outils Dumont SA, Montignez, Switzerland | 0102-7-PO | |
| Tweezers, plastic replaceable tip | Ideal-tek SA, Balerna, Switzerland | 5SVR.SA | |
| Water ROTISOLV HPLC gradient grade | Carl Roth GmbH + Co KG, Karlsruhe, Germany | A511.2 |
Références
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Sigal, Y. M., Zhou, R., Zhuang, X. Visualizing and discovering cellular structures with super-resolution microscopy. Science. 361 (6405), 880-887 (2018).
- Williams, D. B., Carter, C. B. . The Transmission Electron Microscope. , (2009).
- Bozzola, J. J., Russell, L. D. . Electron Microscopy Principles and Techniques for Biologists. , (1999).
- Thompson, R. F., Walker, M., Siebert, C. A., Muench, S. P., Ranson, N. A. An introduction to sample preparation and imaging by cryo-electron microscopy for structural biology. Methods. 100, 3-15 (2016).
- Lucic, V., Leis, A., Baumeister, W. Cryo-electron tomography of cells: connecting structure and function. Histochemistry and Cell Biology. 130 (2), 185-196 (2008).
- Carson, J. L. Fundamental technical elements of freeze-fracture/freeze-etch in biological electron microscopy. Journal of Visualized Experiments. (91), e51694 (2014).
- Nishiyama, H., et al. Atmospheric scanning electron microscope observes cells and tissues in open medium through silicon nitride film. Journal of Structural Biology. 169 (3), 438-449 (2010).
- Dahmke, I. N., et al. Graphene Liquid Enclosure for Single-Molecule Analysis of Membrane Proteins in Whole Cells Using Electron Microscopy. ACS Nano. 11 (11), 11108-11117 (2017).
- Maruyama, Y., Ebihara, T., Nishiyama, H., Suga, M., Sato, C. Immuno EM-OM correlative microscopy in solution by atmospheric scanning electron microscopy (ASEM). Journal of Structural Biology. 180 (2), 259-270 (2012).
- Kinoshita, T., et al. Immuno-electron microscopy of primary cell cultures from genetically modified animals in liquid by atmospheric scanning electron microscopy. Microscopy and Microanalysis. 20 (2), 469-483 (2014).
- Hauwiller, M. R., Ondry, J. C., Alivisatos, A. P. Using graphene liquid cell transmission electron microscopy to study in situ nanocrystal etching. Journal of Visualized Experiments. (135), e57665 (2018).
- Cho, H., et al. The Use of Graphene and Its Derivatives for Liquid-Phase Transmission Electron Microscopy of Radiation-Sensitive Specimens. Nano Letters. 17 (1), 414-420 (2017).
- Meng, F., et al. Graphene-Based Fibers: A Review. Advanced Materials. 27 (35), 5113-5131 (2015).
- Sun, P. Z., et al. Limits on gas impermeability of graphene. Nature. 579 (7798), 229-232 (2020).
- Kato, R., et al. High-precision thickness control of ice layer on CVD grown bilayer graphene for cryo-TEM. Carbon. 160, 107-112 (2020).
- Keskin, S., de Jonge, N. Reduced Radiation Damage in Transmission Electron Microscopy of Proteins in Graphene Liquid Cells. Nano Letters. 18 (12), 7435-7440 (2018).
- Giepmans, B. N., Deerinck, T. J., Smarr, B. L., Jones, Y. Z., Ellisman, M. H. Correlated light and electron microscopic imaging of multiple endogenous proteins using Quantum dots. Nature Methods. 2, 743-749 (2005).
- Ring, E. A., Peckys, D. B., Dukes, M. J., Baudoin, J. P., de Jonge, N. Silicon nitride windows for electron microscopy of whole cells. Journal of Microscopy. 243 (3), 273-283 (2011).
- Peckys, D. B., Korf, U., de Jonge, N. Local variations of HER2 dimerization in breast cancer cells discovered by correlative fluorescence and liquid electron microscopy. Science Advances. 1 (6), 1500165 (2015).
- Eigenbrot, C., Ultsch, M., Dubnovitsky, A., Abrahmsen, L., Hard, T. Structural basis for high-affinity HER2 receptor binding by an engineered protein. Proceedings of the National Academy of Sciences of the United States of America. 107 (34), 15039-15044 (2010).
- Textor, M., de Jonge, N. Strategies for Preparing Graphene Liquid Cells for Transmission Electron Microscopy. Nano Letters. 18, 3313-3321 (2018).
- Henjes, F., et al. Strong EGFR signaling in cell line models of ERBB2-amplified breast cancer attenuates response towards ERBB2-targeting drugs. Oncogenesis. 1, 16 (2012).
- Peckys, D. B., de Jonge, N. Studying the Stoichiometry of Epidermal Growth Factor Receptor in Intact Cells using Correlative Microscopy. Journal of Visualized Experiments. (103), e53186 (2015).
- Pirkmajer, S., Chibalin, A. V. Serum starvation: caveat emptor. American Journal of Physiology. Cell Physiology. 301 (2), 272-279 (2011).
- Kohl, H., Reimer, L. . Transmission electron microscopy: physics of image formation. , (2008).
- Yi, H., et al. Native immunogold labeling of cell surface proteins and viral glycoproteins for cryo-electron microscopy and cryo-electron tomography applications. Journal of Histochemistry and Cytochemistry. 63 (10), 780-792 (2015).
- Wolf, S. G., Houben, L., Elbaum, M. Cryo-scanning transmission electron tomography of vitrified cells. Nature Methods. 11 (4), 423-428 (2014).
- Dukes, M. J., Ramachandra, R., Baudoin, J. P., Jerome, W. G., de Jonge, N. Three-dimensional locations of gold-labeled proteins in a whole mount eukaryotic cell obtained with 3 nm precision using aberration-corrected scanning transmission electron microscopy. Journal of Structural Biology. 174, 552-562 (2011).
- Meier, C., Beckmann, A. Freeze fracture: new avenues for the ultrastructural analysis of cells in vitro. Histochemistry and Cell Biology. 149 (1), 3-13 (2018).
- Peckys, D. B., de Jonge, N. Liquid scanning transmission electron microscopy: imaging protein complexes in their native environment in whole eukaryotic cells. Microscopy and Microanalysis. 20 (2), 346-365 (2014).
- Egerton, R. F. Control of radiation damage in the TEM. Ultramicroscopy. 127, 100-108 (2013).
- Wojcik, M., Hauser, M., Li, W., Moon, S., Xu, K. Graphene-enabled electron microscopy and correlated super-resolution microscopy of wet cells. Nature Communications. 6, 7384 (2015).
- Huebinger, J., Spindler, J., Holl, K. J., Koos, B. Quantification of protein mobility and associated reshuffling of cytoplasm during chemical fixation. Scientific Reports. 8 (1), 17756 (2018).
- Glauert, A. M., Lewis, P. R. . Biological specimen preparation for transmission electron microscopy. , (2014).
- Koo, K., Dae, K. S., Hahn, Y. K., Yuk, J. M. Live Cell Electron Microscopy Using Graphene Veils. Nano Letters. 20 (6), 4708-4713 (2020).
- Pawley, J. B. . Handbook of biological confocal microscopy, 2 edn. , (1995).
- Chung, I., et al. Spatial control of EGF receptor activation by reversible dimerization on living cells. Nature. 464 (7289), 783-787 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon