Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Cartographie du transfert d’énergie par résonance de Förster : une nouvelle méthodologie pour élucider les caractéristiques structurelles mondiales
Dans cet article
Résumé
L’étude détaille la méthodologie de la cartographie FRET, y compris la sélection des sites d’étiquetage, le choix des colorants, l’acquisition et l’analyse des données. Cette méthodologie est efficace pour déterminer les sites de liaison, les changements conformationnels et les mouvements dynamiques dans les systèmes protéiques et est plus utile si elle est réalisée en conjonction avec les informations structurelles 3D existantes.
Résumé
Le transfert d’énergie par résonance de Förster (FRET) est une méthode établie basée sur la fluorescence utilisée pour mesurer avec succès les distances dans et entre les biomolécules in vitro ainsi qu’à l’intérieur des cellules. Dans FRET, l’efficacité du transfert d’énergie, mesurée par les changements d’intensité de fluorescence ou de durée de vie, est liée à la distance entre deux molécules ou étiquettes fluorescentes. La détermination de la dynamique et des changements conformationnels à partir des distances ne sont que quelques exemples d’applications de cette méthode aux systèmes biologiques. Dans certaines conditions, cette méthodologie peut ajouter et améliorer les structures cristallines de rayons X existantes en fournissant des informations sur la dynamique, la flexibilité et l’adaptation aux surfaces de liaison. Nous décrivons l’utilisation du FRET et des déterminations de distance associées pour élucider les propriétés structurelles, par l’identification d’un site de liaison ou les orientations des sous-unités dimères. Grâce à un choix judicieux des sites de marquage et souvent à l’utilisation de plusieurs stratégies de marquage, nous avons appliqué avec succès ces méthodes de cartographie pour déterminer les propriétés structurelles globales d’un complexe protéine-ADN et du système de translocation de protéines SecA-SecYEG. Dans le système SecA-SecYEG, nous avons utilisé des méthodes de cartographie FRET pour identifier le site de liaison des préprotéines et déterminer la conformation locale de la région de séquence de signaux liés. Cette étude décrit les étapes à suivre pour effectuer des études de cartographie FRET, y compris l’identification des sites d’étiquetage appropriés, la discussion des étiquettes possibles, y compris les résidus d’acides aminés non natifs, les procédures d’étiquetage, la façon d’effectuer des mesures et l’interprétation des données.
Introduction
Pour les protéines, l’élucidation de la dynamique ainsi que les connaissances structurelles en 3 dimensions (3D) conduisent à une meilleure compréhension des relations structure-fonction des systèmes biomoléculaires. Les méthodes structurelles, telles que la cristallographie aux rayons X et la microscopie électronique cryogénique, capturent une structure statique et nécessitent souvent la détermination de plusieurs structures pour élucider les aspects de la liaison et de la dynamique des biomolécules1. Cet article décrit une méthode basée sur une solution pour mapper des éléments structurels globaux, tels que des sites de liaison ou des interactions de liaison, qui sont potentiellement plus transitoires et moins facilement capturés par des méthodes statiques. Les systèmes candidats solides pour cette méthodologie sont ceux dans lesquels une structure 3D a déjà été déterminée par cristallographie aux rayons X, spectroscopie RMN ou d’autres méthodes structurelles. Dans ce cas, nous tirons parti de la structure cristalline des rayons X du complexe SecA-SecYEG, un acteur central de la voie sécrétoire générale des protéines, pour cartographier l’emplacement d’un site de liaison peptidique signal à l’aide du transfert d’énergie par résonance de Förster (FRET) avant le transport de la préprotéine à travers la membrane2. La manipulation du système biologique par des modifications génétiques couplée à notre connaissance de la structure 3D a permis de déterminer la conformation de la séquence de signaux et de la région mature précoce immédiatement avant l’insertion dans le canal 3.
FRET implique le transfert d’énergie sans rayonnement d’une molécule (donneur) à une autre (accepteur) d’une manière dépendante de la distance qui traverse l’espace 4,5. L’efficacité de ce transfert est surveillée soit par une diminution du donneur, soit par une augmentation de l’intensité de fluorescence de l’accepteur. L’efficacité du transfert d’énergie peut être décrite comme suit :
E = R06/(R06 + R6)
dans laquelle la valeur R0 est la distance à laquelle le transfert est efficace à 50 %6. La technique a déjà été décrite comme une règle moléculaire et est efficace pour déterminer des distances comprises entre 2,5 et 12 nm, en fonction de l’identité des colorants donneur-accepteur 4,7,8,9. Les intensités de fluorescence du donneur et les durées de vie avec ou sans accepteur permettent de déterminer les efficacités de transfert et, par conséquent, les distances 5,8. En raison de la disponibilité de la technologie, de la sensibilité de la méthode et de la facilité d’utilisation, FRET a également trouvé une large application dans des domaines tels que la spectroscopie de fluorescence à molécule unique et la microscopie confocale6. L’avènement de protéines fluorescentes telles que la protéine fluorescente verte a rendu l’observation de la dynamique intracellulaire et de l’imagerie des cellules vivantes relativement facile10,11. De nombreuses applications FRET telles que celles-ci sont abordées en détail dans ce numéro virtuel.
Dans cette étude, nous nous concentrons particulièrement sur l’utilisation des mesures FRET pour produire des valeurs de distance afin de déterminer les détails structurels. Auparavant, les mesures FRET ont été utilisées efficacement pour déterminer la conformation des molécules d’ADN lorsqu’elles sont liées à la protéine 12,13,14, la dynamique interne des protéines et les interactions de liaison aux protéines 15,16,17. Les avantages de cette méthode résident dans la capacité de déterminer des éléments structurels flexibles et dynamiques dans une solution avec des quantités relativement faibles de matériau. De manière significative, cette méthode est particulièrement efficace lorsqu’elle est utilisée conjointement avec des informations structurelles existantes et ne peut pas être utilisée comme moyen de détermination de la structure 3D. La méthode fournit le meilleur aperçu et le meilleur raffinement de la structure si le travail s’appuie sur des informations structurelles existantes souvent couplées à une simulation informatique18,19. Ici, l’utilisation des distances obtenues à partir de mesures FRET à l’état d’équilibre et résolues dans le temps est décrite pour cartographier un site de liaison, dont l’emplacement n’était pas connu, sur une structure cristallographique existante du complexe SecA-SecYEG, des protéines majeures de la voie sécrétoire générale3.
La voie sécrétoire générale, un système hautement conservé des procaryotes aux eucaryotes en passant par les archées, médie le transport des protéines à travers ou dans la membrane jusqu’à leur emplacement fonctionnel dans la cellule. Pour les bactéries à Gram négatif, telles que E. coli, l’organisme utilisé dans notre étude, les protéines sont insérées ou transloquées à travers la membrane interne vers le périplasme. Le complexe de canaux secY bactériens (appelé translocon) se coordonne avec d’autres protéines pour translocaliser la protéine nouvellement synthétisée, qui est dirigée vers son emplacement correct dans la cellule par une séquence de signaux généralement située à la terminaison N20,21. Pour les protéines liées au périplasme, la protéine ATPase SecA s’associe au tunnel de sortie du ribosome, et à la préprotéine après qu’environ 100 résidus ont été traduits22. Avec la protéine chaperonne SecB, il maintient la préprotéine dans un état déplié. SecA se lie au translocon SecYEG et, à travers de nombreux cycles d’hydrolyse de l’ATP, facilite le transport des protéines à travers la membrane23,24.
SecA est une protéine multi-domaine qui existe sous des formes cytosoliques et membranaires. Une protéine homodimérique dans le cytosol, SecA se compose d’un domaine de liaison ou de réticulation préprotéique25, de deux domaines de liaison aux nucléotides, d’un domaine alaire hélicoïdal, d’un domaine d’échafaudage hélicoïdal et des deux doigts hélicoïdaux (THF)26,27,28,29 (Figure 1). Dans des études cristallographiques antérieures du complexe SecA-SecYEG, l’emplacement du THF a suggéré qu’il était activement impliqué dans la translocation des protéines et les expériences de réticulation ultérieures avec le peptide signal ont établi l’importance de cette région dans la translocation des protéines30,31. Des études antérieures, utilisant la méthodologie de cartographie FRET, ont démontré que les peptides de signal exogènes se lient à cette région de SecA 2,32. Pour bien comprendre la conformation et l’emplacement de la séquence de signaux et de la région mature précoce de la préprotéine avant son insertion dans le canal SecYEG, une chimère protéique dans laquelle la séquence de signal et les résidus de la région mature précoce ont été attachés à SecA par l’intermédiaire d’un liant Ser-Gly a été créée (Figure 1). En utilisant cette construction biologiquement viable, il a été démontré que la séquence de signaux et la région mature précoce de la préprotéine se lient au THF de manière parallèle2. Par la suite, la méthodologie de cartographie FRET a été utilisée pour élucider la conformation et l’emplacement de la séquence de signaux et de la région mature précoce en présence de SecYEG, comme décrit ci-dessous3.
La connaissance de la structure 3D du complexe SecA-SecYEG 33,34,35 et de l’emplacement possible du site de liaison nous a permis de placer judicieusement des étiquettes donneur-accepteur dans des endroits où l’intersection des distances FRET individuelles identifie l’emplacement du site de liaison. Ces mesures de cartographie FRET ont révélé que la séquence de signaux et la région mature précoce de la préprotéine forment une épingle à cheveux avec la pointe située à l’embouchure du canal SecYEG, démontrant que la structure en épingle à cheveux est modélisée avant l’insertion du canal.
Protocole
1. Sélection des sites d’étiquetage
- Identifier au moins trois sites de marquage potentiels pour trianguler le site de liaison putative sur les structures protéiques existantes. Dans ce cas, SecA, SecYEG et préprotéine attachée à SecA par fusion génétique ont été identifiés2.
- Choisissez des sites d’étiquetage à moins de 25-75 Å du site de liaison putative et dans des régions relativement statiques de la protéine, la distance déterminera la paire de colorants FRET spécifique à utiliser36. Localisez les sites de marquage dans des régions protéiques relativement distinctes les unes des autres, de sorte que les sites décrivent les sommets d’un triangle avec le site de liaison putatif situé au centre (Figure 1A-D).
- Introduire ou identifier les résidus de cystéine (Cys) aux sites d’étiquetage d’intérêt dans une protéine qui n’a pas d’autres résidus de Cys, afin d’améliorer l’efficacité de marquage du site spécifique37,38.
- Introduire des acides aminés non naturels, par exemple la p-azidophénylalanine pour le marquage avec la chimie du clic, afin de marquer efficacement une protéine à deux positions distinctes avec différents colorants39,40.
- Testez les mutants de Cys pour la perte de fonction. Vérifier l’activité du mutant sans Cys et du mutant d’acide aminé non naturel à l’aide d’un test d’activité approprié. Dans ce cas, l’activité a été vérifiée avec un test de croissance suivi d’un test IN VITRO de malachite verte ATPase 32,41,42
2. Marquage de la protéine
- Purifier la ou les protéines d’intérêt à une pureté d’au moins 95% pour un marquage précis. Purifier les protéines SecA et SecYEG en suivant les protocoles détaillés dans la référence3. Assurez-vous d’avoir au moins 5 μg de protéines purifiées pour cette étape, car certaines protéines seront perdues pendant le processus d’étiquetage.
- Choisissez deux colorants pour les mesures FRET en fonction de leur valeur R0 et des distances prévues entre les sites marqués. Estimer les valeurs R0 et observer le chevauchement de l’émission du donneur et de l’absorbance de l’accepteur à l’aide des informations de la base de données sur les protéines fluorescentes, qui donne également des spectres pour les colorants couramment utilisés (https://www.fpbase.org/spectra/)36.
REMARQUE: R0 est défini comme la distance à laquelle l’efficacité de transfert est de 50% pour une paire de colorants donnée. Pour les expériences de cartographie, les distances prévues doivent être proches de la valeur R0 de la paire de colorants pour s’assurer que les distances peuvent être mesurées avec précision. - Étiquetez les positions identifiées à l’étape 1.1.1. avec la paire de colorant donneur-accepteur.
- Étiquetez la protéine conformément aux instructions du fabricant en accordant une attention particulière aux paramètres tels que la concentration optimale en protéines, la température, le pH, la durée et le tampon pour les colorants spécifiques utilisés43,44.
- Préparer la protéine à une concentration de 1-2 mg/mL ou environ 10 μM dans un tampon Tris-HCl (pH 7,5), 25 mM KCl, 1 mM EDTA (TKE). Dissoudre le colorant dans le diméthylformamide (DMF) ou le diméthylsulfoxyde (DMSO) jusqu’à une concentration finale de 1 mM. Ajouter le colorant goutte à goutte à la solution en remuant pour obtenir un rapport molaire colorant:protéine de 5:1 (colorant 50 μM : protéine 10 μM).
- Laisser la réaction se poursuivre pendant 4 h à température ambiante (RT) dans un flacon en verre avec un balancement doux ou pendant la nuit à 4 °C. Arrêtez la réaction en ajoutant du β-mercaptoéthanol.
REMARQUE: Si la protéine n’est pas compatible avec le tampon Tris, des tampons phosphate ou HEPES peuvent être utilisés. Maintenir le pH dans la plage de 7,0 à 7,5. Si la protéine a des liaisons disulfures, ajoutez un agent réducteur tel que DTT ou TCEP avant le marquage. Retirer la TNT par dialyse ou filtration sur gel avant d’ajouter du colorant.
- Pour des mesures FRET précises, retirez le colorant libre à l’aide d’un concentrateur centrifuge avec un seuil de poids moléculaire approprié (MWCO) pour laisser le colorant libre circuler tout en conservant la protéine marquée.
- Préparer la membrane du concentrateur en plaçant ~ 1 mL d’eau dans la partie supérieure d’un concentrateur de 3 mL, puis centrifuger l’eau à travers la membrane (au moins 10 min à 4 300 x g).
- Retirer le colorant libre en centrifugant l’échantillon marqué dans un concentrateur (20 min à 4 300 x g). Répétez 3-4 fois et jetez le flux à travers.
- Vérifiez l’efficacité d’étiquetage de la protéine marquée à l’aide de la spectroscopie d’absorption UV-Vis.
REMARQUE: Les mesures FRET nécessitent des rendements d’étiquetage de 50% ou plus. Une efficacité d’étiquetage plus faible réduit le signal FRET et peut entraîner des inexactitudes dans la mesure. - Obtenir un spectre UV-Vis de la protéine marquée avec une plage de 250-700 nm pour observer à la fois la bande d’absorption des protéines et la bande d’absorption maximale du colorant. Mesurer l’absorbance au pic d’absorption du colorant et à 280 nm pour les protéines.
- Déterminer la concentration de protéines et corriger toute contribution du colorant en utilisant le facteur de correction, CF, et les équations suivantes45,46:
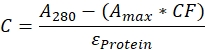
où C est la concentration de la protéine (M), A280 est l’absorbance de l’échantillon à 280 nm, Amax est l’absorbance au maximum d’absorption du colorant, ε protéine est le coefficient d’extinction de la protéine à 280 nm et CF est le facteur de correction, A'280/A’max, où A'280 est l’absorbance à 280 nm et A’max est l’absorbance au maximum maximal pour le colorant uniquement. - Déterminez l’efficacité de l’étiquetage à l’aide de l’équation suivante :
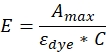
où εcolorant est le coefficient d’extinction molaire du colorant, C est la concentration de la protéine telle que déterminée à l’étape 2.4.5, et E est l’efficacité de marquage. Répétez l’étape 2.4.2 jusqu’à ce que la valeur d’efficacité de l’étiquetage ait plafonné et soit inférieure à 100 %.
3. Déterminez les valeurs R 0
- Mesurez les valeurs R0 in situ. Préparer deux échantillons de protéines à la même concentration de protéines totales, 4 μM, un avec la protéine marquée avec le colorant donneur seulement et un avec la protéine marquée avec le colorant accepteur seulement. Pour SecA, une concentration protéique de 4 μM SecA monomère fonctionne bien pour ces mesures.
- Préparer des volumes d’échantillons de 2,5 mL pour une cuvette de 1 cm x 1 cm, de 600 μL pour une cuvette de 5 mm x 5 mm ou de 200 μL pour une cuvette de 3 mm x 3 mm.
- Allumez le fluoromètre et ouvrez le programme d’acquisition et d’analyse spectrale dans le logiciel de fluorescence si vous utilisez un spectrofluoromètre. Cliquez sur le M rouge pour connecter l’ordinateur à l’instrument (Figure 2A) et choisissez Spectres d’émission.
- Entrez les paramètres de balayage tels que la longueur d’onde d’excitation, la plage de balayage d’émission, la température et la position du changeur d’échantillon à l’aide de l’élément de menu Expérience de collecte (Figure 2B).
- Cliquez sur RTC et optimisez les réglages de l’instrument (par exemple, les fentes spectrales) en surveillant l’émission de fluorescence au pic à l’aide d’une longueur d’onde d’excitation réglée au maximum d’absorption du colorant. Pour SecA, définissez les paramètres suivants : passe-bande sur 1 nm ; les fentes d’excitation et d’émission sont respectivement de 1 et 1,5 mm, avec une température de 25 °C et une vitesse d’agitation de 250 tr/min.
REMARQUE: Ne dépassez pas la capacité de comptage par seconde (cps) de l’instrument (généralement 2 x 106 cps).
- Placez l’échantillon de protéine marqué par le donneur dans le porte-échantillon et cliquez sur Exécuter pour générer un balayage d’émission de la protéine étiquetée avec le colorant du donneur uniquement (protéine du donneur uniquement) en excitant l’échantillon au maximum d’absorption du colorant (par exemple, 488 nm pour AF488) et en balayant le pic d’émission (505-750 nm pour la protéine SecA du donneur uniquement marquée avec AF488).
- Établissez une ligne de base pour l’analyse en étendant l’analyse de 25 à 50 nm au-delà de la fin du pic. Mesurer le rendement quantique de la protéine du donneur uniquement, en effectuant des mesures d’absorption et de fluorescence sur des échantillons de différentes concentrations comme décrit47. Conservez les mêmes réglages de fente pour ces mesures.
- Utilisez un colorant donneur libre comme référence pour le rendement quantique. Obtenez au moins quatre mesures de la protéine du donneur seulement et du colorant libre à différentes concentrations pour une détermination précise.
- Tracer l’intensité de fluorescence ou la zone intégrée par rapport à l’absorbance de la protéine donneuse uniquement et du colorant libre ou de la référence. Déterminer les pentes de la protéine donneuse seulement (penteD) et la référence (penteR).
- Le rendement quantique (Φ) est calculé à l’aide de l’équation suivante :
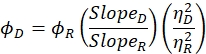
ici ΦD est le rendement quantique de la protéine donneuse uniquement, ΦR est le rendement quantique du colorant libre (cela peut généralement être obtenu auprès du fabricant), la penteD et la penteR sont les pentes déterminées à l’étape 3.4.2 pour la protéine et la référence du donneur uniquement, respectivement et ηD et ηR représentent l’indice de réfraction de la protéine donneuse uniquement et des solutions colorantes sans référence, respectivement 47.
- Obtenez un spectre d’absorption de votre protéine accepteur uniquement à l’aide d’une cellule de longueur de trajet de 1 cm. Générez un spectre de coefficient d’extinction de votre protéine accepteur uniquement en divisant le spectre d’absorption par la concentration en colorant.
- Générer l’intégrale de chevauchement spectral, J (λ) à l’aide d’un programme d’analyse graphique. Un programme de feuille de calcul standard (p. ex., feuille de calcul) peut également être utilisé pour ce processus.
- Multipliez le spectre d’émission de fluorescence de la protéine donneuse uniquement (étape 3.4) par le spectre du coefficient d’extinction de la protéine accepteur uniquement pour générer le spectre de chevauchement.
- Multipliez le spectre de chevauchement résultant par λ4.
- Déterminez l’aire sous la courbe en intégrant la région de chevauchement. La région de chevauchement est définie comme la zone où le spectre d’émission du donneur multiplié par le spectre du coefficient d’extinction de l’accepteur donne des valeurs positives. L’intégrale du chevauchement spectral est définie comme suit :
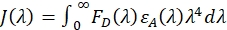
où FD (λ) est le spectre d’émission de la protéine donneuse seule (obtenue à l’étape 3.4) et εA(λ) est le spectre du coefficient d’extinction de la protéine accepteur uniquement et a des unités de M-1 cm-1 (obtenues à l’étape 3.5). L’intégrale de chevauchement spectral résultant devrait avoir des unités de M-1 cm-1nm4. - Normaliser l’intégrale du chevauchement spectral. Divisez l’intégrale de chevauchement par l’aire intégrée du spectre protéique du donneur uniquement sur la même gamme spectrale :
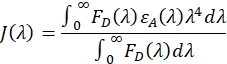
- Calculez la valeur R0 en Å à l’aide de l’équation suivante :
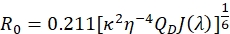
où κ2 est le facteur d’orientation, généralement pris comme 2/3 pour les colorants en rotation libre, η est l’indice de réfraction et peut être approximé comme 1,33 pour les solutions aqueuses diluées, QD est le rendement quantique du donneur (étape 3.4) et J(λ) est l’intégrale de chevauchement spectrale comme déterminé à l’étape 3.6.35. REMARQUE: Si les colorants ne tournent pas librement, des corrections peuvent être introduites comme décrit par Ivanov 48 et mis en œuvre par Auclair49 et Zhang 2,3.
4. Effectuer des mesures spectrales FRET
- Préparer des échantillons de protéines du donneur seulement, de protéines à accepteur seulement et de protéines de donneur-accepteur à la même concentration; une concentration de 4 μM est recommandée. Utiliser 200 μL de solution, si vous utilisez une cuvette de 3 mm x 3 mm, 600 μL si vous utilisez une cuvette de 5 mm x 5 mm ou 2,5 mL si vous utilisez une cuvette de 1 cm x 1 cm.
- Préparez l’échantillon de protéine donneur-accepteur en utilisant des quantités molaires égales de protéines du donneur uniquement et de protéines accepteurs uniquement.
- Conserver la même quantité d’échantillon marqué dans le donneur témoin seulement et accepter uniquement les échantillons de protéines grâce à l’introduction de protéines non marquées en quantité molaire égale aux échantillons du donneur seulement ou de l’accepteur seulement. Par exemple, pour des solutions de même concentration, chaque échantillon FRET de donneur seulement contiendrait 100 μL de protéines de donneur seulement et 100 μL de protéines non marquées pour un volume de 200 μL.
- Générez des spectres d’émission de fluorescence du donneur uniquement, de l’accepteur uniquement et des échantillons donneur-accepteur. Optimisez le signal comme décrit à l’étape 3.2. Une fois optimisé, conservez les mêmes paramètres pour tous les échantillons.
- Obtenez l’analyse du donneur uniquement comme décrit à l’étape 3.3. Excitez la solution au maximum de l’absorption du colorant du donneur et balayez les pics d’émission du donneur et de l’accepteur (attendu).
- Échangez l’échantillon contre la protéine accepteur uniquement ou remplacez la position du changeur d’échantillon par la cuvette contenant la protéine accepteur uniquement.
- Obtenir un scan d’émission de la protéine marquée avec un colorant accepteur uniquement (protéine accepteur uniquement) en utilisant les mêmes paramètres qu’à l’étape 4.2.1. Excitez l’échantillon à la longueur d’onde d’excitation du donneur.
REMARQUE: Ce spectre fournit une correction pour la quantité d’accepteur excité à la longueur d’onde du donneur (FA à l’étape 5.1.2) - Échangez l’échantillon contre l’échantillon de protéine donneur-accepteur ou remplacez la position du changeur d’échantillon par la cuvette contenant la protéine marquée donneur-accepteur.
- Obtenir un scan d’émission de l’échantillon de protéine donneur-accepteur en utilisant les mêmes réglages que dans les étapes 4.2.1 et 4.2.3.
- Pour tous les spectres, corrigez la fluorescence de fond en soustrayant les comptes de fond mesurés à la fin du balayage.
- Mesurer la durée de vie du donneur uniquement et les échantillons donneur-accepteur préparés comme décrit à l’étape 4.1.2. Utilisez un instrument de fluorescence à photon unique corrélé dans le temps capable de mesurer et de résoudre les désintégrations fluorescentes dans la plage de temps de la nanoseconde (10-9 s).
REMARQUE: Pour les paires de colorants FRET, faites correspondre la source lumineuse d’excitation au maximum d’absorption du colorant donneur.- Allumez l’instrument. Ouvrez le logiciel d’acquisition, utilisez le logiciel de contrôle de l’instrument pour l’acquisition de données avec le spectromètre de fluorescence.
- Pour l’acquisition, sélectionnez TCSPC Decay, avec une plage de temps de 55 ns, un gain de 1 et 4096 canaux.
- Obtenir une fonction de réponse de l’instrument (IRF) à l’aide d’une solution de crémeuse non laitière ou d’une solution de diffusion commerciale et surveiller la diffusion à 490 nm. Ajustez le réglage de la fente et utilisez des filtres à densité neutre au besoin pour maintenir un taux de comptage suffisamment faible pour éviter l’empilementd’impulsions 5. Cliquez sur Accepter , puis sur Démarrer. Cela lancera l’acquisition.
REMARQUE: Un taux de comptage maximal de 4000 cps est utilisé pour un taux de répétition de 180 kHz. - Collectez l’IRF à 490 nm jusqu’à ce que le canal de crête ait un maximum de 20 000 comptes. Recueillir un IRF avant et après avoir mesuré chaque désintégration de fluorescence.
- Obtenir les désintégrations de fluorescence du donneur uniquement et des échantillons donneur-accepteur en surveillant l’émission de fluorescence à la longueur d’onde d’émission du donneur, 520 nm.
- Ajustez les paramètres de fente pour un taux de comptage maximal de 4000 cps ou moins. Les réglages de fente sont généralement de 15 à 20 nm pour les échantillons de protéines. Collectez la désintégration jusqu’à ce que 20 000 comptes soient obtenus dans le canal de pointe.
- Analyser la désintégration ou l’intensité de fluorescence (I) en fonction du temps (t) pour la durée de vie de la fluorescence (τ). Ajuster la désintégration à une somme d’exponentielles avec l’équation suivante :
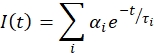
où αI est le facteur préexponentiel de la ième composante et τI est la durée de vie. L’ajustement est reconvolved avec l’IRF pour correspondre à la désintégration de fluorescence. Jugez la qualité de l’ajustement à partir des paramètres réduits Χ2.
5. Analyse des données FRET
- Calculer l’efficacité FRET à partir de la diminution de l’intensité du donneur de l’échantillon donneur-accepteur par rapport au donneur uniquement avec l’équation suivante.
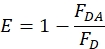
où FDA est l’intensité de fluorescence de l’échantillon donneur-accepteur et FD est l’intensité de fluorescence de l’échantillon donneur seulement au sommet de la fluorescence du donneur. Utilisez les zones intégrées des pics si les données sont bruyantes.- Corriger toute différence d’étiquetage entre les échantillons donneur seulement et donneur-accepteur. Calculez les corrections en fonction du degré d’étiquetage du donneur comme suit.
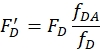
où fDA est l’efficacité d’étiquetage du donneur dans l’échantillon donneur-accepteur et fD est l’efficacité d’étiquetage dans l’échantillon donneur seulement. - Corriger toute contribution de la fluorescence de l’accepteur au spectre excité par le donneur par soustraction du spectre de la protéine accepteur seulement (étape 4.2) du spectre de la protéine donneur-accepteur.
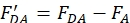
- Corriger les différences dans l’efficacité d’étiquetage de la protéine accepteur uniquement par rapport à l’échantillon de protéine donneur-accepteur, ce qui donne l’équation suivante pour le calcul de l’efficacité:
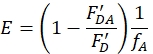
où fA désigne la quantité fractionnaire de l’étiquetage de l’accepteur. Cette équation comprend toutes les corrections dues à l’étiquetage des colorants et à la fluorescence des accepteurs. - Calculez les distances FRET à partir des rendements à l’aide de l’équation suivante :
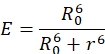
en utilisant la valeur R0 obtenue à l’étape 3.6.5. - Calculer l’efficacité FRET en utilisant les durées de vie de fluorescence du donneur uniquement et des échantillons donneur-accepteur mesurés à l’étape 4.3.3-4.3.5 :
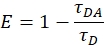
- Utilisez la durée de vie pondérée en fonction de l’amplitude pour calculer les rendements FRET et comparer avec les résultats à l’état d’équilibre5.
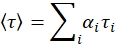
- Calculer la distance comme à l’étape 5.1.4 à partir des rendements déterminés par la durée de vie de la fluorescence. Comparez les valeurs à l’état d’équilibre et les valeurs résolues dans le temps pour les efficacités et les distances FRET et assurez-vous qu’elles sont à l’écart les unes des autres.
- Corriger toute différence d’étiquetage entre les échantillons donneur seulement et donneur-accepteur. Calculez les corrections en fonction du degré d’étiquetage du donneur comme suit.
6. Cartographie des distances
- Utilisez les distances calculées pour mapper le site de liaison sur la structure tridimensionnelle. Calculer les distances et les erreurs pour toutes les paires de colorants et les emplacements examinés à l’aide de l’équation donnée à l’étape 5.1.4 et des valeurs R0 obtenues à l’étape 3.6.5 pour chaque paire FRET.
- Utilisez un programme de visualisation graphique 3D tel que PyMOL50 pour mapper les distances sur la structure (script donné dans le fichier supplémentaire). Les commandes du script peuvent être saisies directement dans la fenêtre de commande avec les informations de distance appropriées.
- Générez un shell pour chaque distance mesurée et l’erreur associée (Figure 3, Figure 4, Figures supplémentaires 1-3).
- Cartographiez la position à travers l’intersection des différentes coquilles (figures supplémentaires 1-3). Le site de liaison du peptide signal a été cartographié à travers les trois emplacements différents sur SecA et SecYEG et quatre emplacements différents sur le peptide signal (Figure 1).
Résultats
Cette étude s’est concentrée sur la détermination de l’emplacement du site de liaison des préprotéines sur SecA avant l’insertion de la préprotéine dans le canal SecYEG. Pour cartographier le site de liaison, des expériences FRET ont été réalisées entre différentes régions de la préprotéine et trois emplacements distincts sur les protéines SecA et SecYEG (Figure 1A-D). À partir des distances obtenues et des structures tridimensionnell...
Discussion
Grâce à l’utilisation de la méthodologie de cartographie FRET, nous avons identifié le site de liaison de la séquence de signaux sur la protéine SecA. Il est important de noter que la présence d’une structure cristalline 3D du complexe a grandement facilité notre étude. La force de cette méthodologie de cartographie réside dans la capacité d’utiliser une structure existante pour identifier les emplacements à étiqueter. Cette méthodologie ne peut pas être utilisée pour déterminer une structure 3D; ...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par la subvention R15GM135904 des National Institutes of Health (attribuée à IM) et la subvention GM110552 des National Institutes of Health (attribuée à DBO).
matériels
| Name | Company | Catalog Number | Comments |
| 490 nm LED laser | Horiba | 1684-LED | |
| Alexa Fluor 647 C2 Maleimide//DIBO Alkyne | Life Technologies | A20347 | |
| Agar | Difco | DF0812 | |
| Alexa Fluor 488 C5 Maleimide/DIBO Alkyne | Life Technologies | A10254 | |
| Alexa Fluor 488 DIBO Alkyne | Life Technologies | S10904 | |
| Alexa Fluor 647 DIBO Alkyne | Life Technologies | S10906 | |
| Amicon Ultra4 Centrifugal filter (50kDa MWCO) | Sigma | UFC805008 | |
| Dodecylmaltoside (DDM) | Anatrace | D310 | |
| E. coli alkaline phosphatase signal peptide SP22 | Biomolecules Midwest | N/A | Synthesized custom item |
| extended signal peptide SP41 | Biomolecules Midwest | N/A | Synthesized custom item |
| FluorEssence | Horiba | version 2.4 | spectral acquisition program for Fluoromax4 spectrofluorometer |
| Fluoromax 4 spectrofluorometer | Horiba | N/A | |
| GlobalsWE | Laboratory for Fluorescence Dynamics, University of California, Irvine | spectral analysis program for time-resolved decays | |
| H4AzidoPheOH | BACHEM | 4020250.0001 | |
| LB (Miller) Broth | Fisher Scientific | BP9723 | |
| Ludox HS-40 colloidal silica (40 wt.% suspension in H2O) | Sigma-Aldrich | 420816 | dilution is needed to make a proper scattering solution |
| PTI Felix GX | Horiba | version 4.1.0.4096 | spectral acquisition program for PTI Time Master Instrument |
| PTI Time Master Instrument | Horiba | NA | |
| Pymol Molecular Graphics Program | Schrodinger | version 2.4 | |
| Water bath | Thermo Scientific | NESLAB RTE 10 |
Références
- Thompson, M. C., Yeates, T. O., Rodriguez, J. A. Advances in methods for atomic resolution macromolecular structure determination. F1000Research. 9, (2020).
- Zhang, Q., Li, Y., Olson, R., Mukerji, I., Oliver, D. Conserved SecA signal peptide-binding site revealed by engineered protein chimeras and Forester resonance energy transfer. Biochemistry. 55 (9), 1291-1300 (2016).
- Zhang, Q., et al. Alignment of the protein substrate hairpin along the SecA two-helix finger primes protein transport in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 114 (35), 9343-9348 (2017).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47, 819-846 (1978).
- Lakowicz, J. R. . Principles of Fluorescence Spectroscopy. , (2006).
- Algar, W. R., Hildebrandt, N., Vogel, S. S., Medintz, I. L. FRET as a biomolecular research tool - understanding its potential while avoiding pitfalls. Nature Methods. 16 (9), 815-829 (2019).
- Magde, D., Wong, R., Seybold, P. G. Fluorescence quantum yields and their relation to lifetimes of rhodamine 6G and fluorescein in nine solvents: improved absolute standards for quantum yields. Photochemistry and Photobiology. 75 (4), 327-334 (2002).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in Enzymology. 211, 353-388 (1992).
- . R0 Values from Some Alexa Fluor Dyes - Table 1.6 Available from: https://www.thermofisher.com/us/en/home/references/molecular-probes-the-handbook/tables/r0-values-for-some-alexa-fluor-dyes.html (2021)
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A Guide to Fluorescent Protein FRET Pairs. Sensors. 16 (9), 1488 (2016).
- Day, R. N., Davidson, M. W. Fluorescent proteins for FRET microscopy: monitoring protein interactions in living cells. BioEssays : News and Reviews in Molecular, Cellular, and Developmental Biology. 34 (5), 341-350 (2012).
- Lee, S. J., Syed, S., Ha, T. Single-Molecule FRET Analysis of Replicative Helicases. Methods in Molecular Biology. 1805, 233-250 (2018).
- Uhm, H., Hohng, S. Single-Molecule FRET Assay for Studying Cotranscriptional RNA Folding. Methods in Molecular Biology. 2106, 271-282 (2020).
- Globyte, V., Joo, C. Single-molecule FRET studies of Cas9 endonuclease. Methods in Enzymology. 616, 313-335 (2019).
- Qiao, Y., Luo, Y., Long, N., Xing, Y., Tu, J. Single-Molecular Förster Resonance Energy Transfer Measurement on Structures and Interactions of Biomolecules. Micromachines. 12 (5), 492 (2021).
- Catipovic, M. A., Bauer, B. W., Loparo, J. J., Rapoport, T. A. Protein translocation by the SecA ATPase occurs by a power-stroke mechanism. The EMBO Journal. 38 (9), 101140 (2019).
- Seinen, A. B., Spakman, D., van Oijen, A. M., Driessen, A. J. M. Cellular dynamics of the SecA ATPase at the single molecule level. Scientific Reports. 11 (1), 1433 (2021).
- Dimura, M., et al. Quantitative FRET studies and integrative modeling unravel the structure and dynamics of biomolecular systems. Current Opinion in Structural Biology. 40, 163-185 (2016).
- Kalinin, S., et al. A toolkit and benchmark study for FRET-restrained high-precision structural modeling. Nature Methods. 9 (12), 1218-1225 (2012).
- Paetzel, M. Structure and mechanism of Escherichia coli type I signal peptidase. Biochimica et Biophysica Acta. 1843 (8), 1497-1508 (2014).
- Ng, D., Brown, J., Walter, P. Signal sequences specify the targeting route to the endoplasmic reticulum membrane. The Journal of Cell Biology. 134 (2), 269-278 (1996).
- Huber, D., et al. SecA Cotranslationally Interacts with Nascent Substrate Proteins In Vivo. Journal of Bacteriology. 199 (2), 00622 (2017).
- Lill, R., et al. SecA protein hydrolyzes ATP and is an essential component of the protein translocation ATPase of Escherichia coli. The EMBO Journal. 8 (3), 961-966 (1989).
- Lill, R., Dowhan, W., Wickner, W. The ATPase activity of SecA is regulated by acidic phospholipids, SecY, and the leader and mature domains of precursor proteins. Cell. 60 (2), 271-280 (1990).
- Kimura, E., Akita, M., Matsuyama, S., Mizushima, S. Determination of a region of SecA that interacts with presecretory proteins in Escherichia coli. The Journal of Biological Chemistry. 266 (10), 6600-6606 (1991).
- Hunt, J. F., et al. Nucleotide control of interdomain interactions in the conformational reaction cycle of SecA. Science. 297 (5589), 2018-2026 (2002).
- Sharma, V., et al. Crystal structure of Mycobacterium tuberculosis SecA, a preprotein tranlsocating ATPase. Proceedings of the National Academy of Sciences of the United States of America. 100 (5), 2243-2248 (2003).
- Vassylyev, D., et al. Crystal structure of the translocation ATPase SecA from Thermus thermophilus reveals a parallel, head-to-head dimer. Journal of Molecular Biology. 364 (3), 248-258 (2006).
- Zimmer, J., Li, W., Rapoport, T. A. A novel dimer interface and conformational changes revealed by an X-ray structure of B. subtilis SecA. Journal of Molecular Biology. 364 (3), 259-265 (2006).
- Zimmer, J., Rapoport, T. A. Conformational flexibility and peptide interaction of the translocation ATPase SecA. Journal of Molecular Biology. 394 (4), 606-612 (2009).
- Bauer, B. W., Rapoport, T. A. Mapping polypeptide interactions of the SecA ATPase during translocation. Proceedings of the National Academy of Sciences of the United States of America. 106 (49), 20800-20805 (2009).
- Auclair, S., et al. Mapping of the signal peptide-binding domain of Escherichia coli SecA using Förster resonance energy transfer. Biochemistry. 49 (4), 782-792 (2010).
- Zimmer, J., Nam, Y., Rapoport, T. A. Structure of a complex of the ATPase SecA and the protein-translocation channel. Nature. 455 (7215), 936-943 (2008).
- Li, L., et al. Crystal structure of a substrate-engaged SecY protein-translocation channel. Nature. 531 (7594), 395-399 (2016).
- Ma, C., et al. Structure of the substrate-engaged SecA-SecY protein translocation machine. Nature Communications. 10 (1), 2872 (2019).
- Lambert, T. J. FPbase: a community-editable fluorescent protein database. Nature Methods. 16 (4), 277-278 (2019).
- Jilaveanu, L. B., Oliver, D. In vivo membrane topology of Escherichia coli SecA ATPase reveals extensive periplasmic exposure of multiple functionally important domains clustering on one face of SecA. The Journal of Biological Chemistry. 282 (7), 4661-4668 (2007).
- Ramamurthy, V., Oliver, D. Topology of the integral-membrane form of Escherichiacoli SecA protein. The Journal of Biological Chemistry. 272 (37), 23239-23246 (1997).
- Chin, J., et al. Addition of p-Azido-L-phenylalanine to the genetic code of Escherichia coli. Journal of the American Chemical Society. 124 (31), 9026-9027 (2002).
- Deiters, A., et al. Adding amino acids with novel reactivity to the genetic code of Saccharomyces cerevisiae. Journal of the American Chemical Society. 125 (39), 11782-11783 (2003).
- Lanzetta, P. A., Alvarez, L. J., Reinach, P. S., Candia, O. A. An improved assay for nanomole amounts of inorganic phosphate. Analytical Biochemistry. 100 (1), 95-97 (1979).
- Mitchell, C., Oliver, D. B. Two distinct ATP-binding domains are needed to promote protein export by Escherichia coli SecA ATPase. Molecular Microbiology. 10 (3), 483-497 (1993).
- Thiol-reactive Probe Labeling Protocol. Thermo Fischer Scientific Available from: https://www.thermofisher.com/us/en/home/references/protocols/cell-and-tissue-analysis/labeling-chemistry-protocols/thiol-reactive-probe-labeling-protocol.html (2021)
- Click Chemistry - Section 3.1. Thermo Fischer Scientific Available from: https://www.thermofisher.com/us/en/home/references/molecular-probes-the-handbook/reagents-for-modifying-groups-other-than-thiols-or-amines/click-chemistry.html (2021)
- Correction Factor. AAT Bioquest Available from: https://www.aatbio.com/resources/correction-factor/ (2019)
- Calculate dye:protein (F/P) molar ratios. Thermo Fischer Scientific Available from: https://tools.thermofischer.com/content/sfs/brochures/TR0031-Calc-FP-rations.pdf (2011)
- A Guide to Recording Fluorescence Quantum Yields. Horiba Scientific Available from: https://static.horiba.com/fileadmin/Horiba/Application/Materials/Material_Research/Quantum_Dots/quantumyieldstrad.pdf (2011)
- Ivanov, V., Li, M., Mizuuchi, K. Impact of emission anisotropy on fluorescence stectroscopy and FRET distance measurements. Biophysical Journal. 97 (3), 922-929 (2009).
- Auclair, S., Oliver, D., Mukerji, I. Defining the solution state dimer structure of Escherichia coli SecA using Forster resonance energy transfer. Biochemistry. 52 (14), 2388-2401 (2013).
- . The PyMOL Molecular Graphics System, Version 2.4 Available from: https://pymol.org/2/ (2021)
- Musial-Siwek, M., Rusch, S. L., Kendall, D. A. Selective photoaffinity labeling identifies the signal peptide binding domain on SecA. Journal of Molecular Biology. 365 (3), 637-648 (2007).
- Miller, A., Wang, L., Kendall, D. A. Synthetic signal peptides specifically recognize SecA and stimulate ATPase activity in the absence of preprotein. The Journal of Biological Chemistry. 273 (19), 11409-11412 (1998).
- Gelis, I., et al. Structural basis for signal-sequence recognition by the translocase motor SecA as determined by NMR. Cell. 131 (4), 756-769 (2007).
- Erlandson, K. J., et al. A role for the two-helix finger of the SecA ATPase in protein translocation. Nature. 455 (7215), 984-988 (2008).
- Das, S., Oliver, D. Mapping of the SecA-SecY and SecA-SecG interfaces by site-directed in vivo photocross-linking. The Journal of Biological Chemistry. 286 (14), 12371-12380 (2011).
- Wheatley, E. G., Pieniazek, S. N., Vitoc, I., Mukerji, I., Beveridge, D. L. Molecular Dynamics Structure Prediction of a Novel Protein-DNA Complex: Two HU Proteins with a DNA Four-way Junction. Innovations in Biomolecular Modeling and Simulations: Volume 2. , 111-128 (2012).
- Vitoc, C. I., Mukerji, I. HU binding to a DNA four-way junction probed by Förster resonance energy transfer. Biochemistry. 50 (9), 1432-1441 (2011).
- Hellman, L. M., Fried, M. G. Electrophoretic mobility shift assay (EMSA) for detecting protein-nucleic acid interactions. Nature Protocols. 2 (8), 1849-1861 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon