このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
フェルスター共鳴エネルギー伝達マッピング:全球構造特徴を解明する新しい方法論
要約
この研究は、標識部位の選択、色素の選択、取得、およびデータ分析を含むFRETマッピングの方法論を詳述している。この方法論は、タンパク質系における結合部位、立体構造変化、および動的運動を決定するのに有効であり、既存の3D構造情報と組み合わせて実行される場合に最も有用である。
要約
Förster共鳴エネルギー移動(FRET)は、 インビトロ および細胞内の生体分子間および生体分子間の距離を首尾よく測定するために使用される確立された蛍光ベースの方法です。FRETにおいて、蛍光強度または寿命の変化によって測定されるエネルギー移動の効率は、2つの蛍光分子または標識間の距離に関する。距離からのダイナミクスと立体構造変化の決定は、この方法の生物学的システムへの応用の一例にすぎません。特定の条件下では、この方法論は、ダイナミクス、柔軟性、および結合表面への適応に関する情報を提供することにより、既存のX線結晶構造に追加し、強化することができる。我々は、結合部位または二量体サブユニットの配向の同定を通じて、構造特性を解明するためのFRETおよび関連する距離決定の使用について記述する。標識部位の賢明な選択、および多くの場合、複数の標識戦略の採用を通じて、タンパク質-DNA複合体およびSecA-SecEGタンパク質転座システムにおけるグローバルな構造特性を決定するために、これらのマッピング法を首尾よく適用しました。SecA-SecYEGシステムでは、FRETマッピング法を使用して、プレタンパク質結合部位を同定し、結合したシグナル配列領域の局所立体構造を決定しました。この研究は、適切な標識部位の同定、非天然アミノ酸残基を含む可能な標識の議論、標識手順、測定の実施方法、およびデータの解釈を含む、FRETマッピング研究を実施するためのステップを概説する。
概要
タンパク質については、3次元(3次元)構造知識とともにダイナミクスを解明することで、生体分子系の構造と機能の関係の理解を深めることができます。X線結晶学や極低温電子顕微鏡などの構造的方法は、静的構造を捉え、生体分子結合やダイナミクスの側面を解明するために複数の構造の決定を必要とすることが多い1。この記事では、結合部位や結合相互作用など、静的な方法ではキャプチャしにくい可能性のあるグローバル構造要素をマッピングするためのソリューションベースの方法について説明します。この方法論の有力な候補系は、X線結晶学、NMR分光法、または他の構造法によって3次元構造が以前に決定されたものである。この場合、タンパク質一般分泌経路の中心プレーヤーであるSecA-SecYEG複合体のX線結晶構造を利用して、膜2を横切るプレタンパク質の輸送の前にフェルスター共鳴エネルギー移動(FRET)を用いてシグナルペプチド結合部位の位置をマッピングする。遺伝子改変による生体系の操作と3次元構造に関する我々の知識を組み合わせることで、チャネル 3への挿入直前のシグナル配列と早期成熟領域の立体構造の決定が可能となった。
FRETは、空間4,5を通る距離依存的な方法で、ある分子(ドナー)から別の分子(アクセプター)へのエネルギーの放射線のない移動を含む。この転送の効率は、ドナーの減少またはアクセプター蛍光強度の増加のいずれかによってモニターされます。エネルギー移動の効率は、次のように記述できます。
E = R 0 6/(R0 6 + R 6)
ここで、R0値は、転送が50%効率的である距離である6。この技術は、以前に分子定規として記載されており、ドナー - アクセプター色素4,7,8,9の同一性に応じて、2.5〜12nmの範囲の距離を決定するのに有効である。ドナーの蛍光強度および寿命は、アクセプターの有無にかかわらず、転写効率の決定を可能にし、その結果、距離5,8である。この技術の利用可能性、方法の感度、および使いやすさにより、FRETは、単一分子蛍光分光法および共焦点顕微鏡6などの分野でも幅広い用途を見出しました。緑色蛍光タンパク質などの蛍光タンパク質の出現により、細胞内動態の観察および生細胞イメージングが比較的容易になった10,11。このような多くのFRETアプリケーションについては、この仮想問題で詳しく説明します。
この研究では、構造の詳細を決定するための距離値を得るためにFRET測定を使用することに特に焦点を合わせています。これまで、FRET測定は、タンパク質12、13、14に結合したときのDNA分子の立体構造、タンパク質の内部動態、およびタンパク質結合相互作用15、16、17を決定するために効果的に使用されてきた。この方法の利点は、比較的少量の材料を含むソリューション中で柔軟で動的な構造要素を決定する能力にあります。重要なことに、この方法は、既存の構造情報と組み合わせて使用する場合に特に効果的であり、3次元構造決定の手段として使用することはできません。この方法は、作業がしばしば計算シミュレーションと組み合わされた既存の構造情報に基づいて構築されている場合、構造の最良の洞察と洗練を提供する18,19。ここでは、定常状態および時間分解FRET測定から得られた距離を使用して、SecA-SecYEG複合体の既存の結晶学的構造上の結合部位を、一般分泌経路3の主要なタンパク質上に位置が知られていなかった。
一般分泌経路は、原核生物から真核生物、古細菌まで高度に保存された系であり、細胞内の機能的位置への膜を横切ってまたは膜内へのタンパク質の輸送を媒介する。我々の研究で使用された生物である大腸菌などのグラム陰性菌の場合、タンパク質は内膜を横切ってペリプラズムに挿入または転座する。細菌のSecYチャネル複合体(トランスロコンと呼ばれる)は、他のタンパク質と協調して、新たに合成されたタンパク質を転座させ、これは、典型的にはN末端に位置するシグナル配列を介して細胞内のその正しい位置に向けられる20,21。ペリプラズムに結合したタンパク質の場合、ATPase SecAタンパク質はリボソームの出口トンネルと会合し、約100残基が翻訳された後のプレタンパク質と会合する22。SecBシャペロンタンパク質とともに、プレタンパク質を折り畳まれていない状態に維持する。SecAは、SecYEGトランスロコンに結合し、そしてATP加水分解の多くのサイクルを経て、膜を横切るタンパク質輸送を促進する23、24。
SecAは、細胞質ゾルおよび膜結合形態で存在するマルチドメインタンパク質である。細胞質ゾル中のホモ二量体タンパク質であるSecAは、プレタンパク質結合または架橋ドメイン25、2つのヌクレオチド結合ドメイン、ヘリカルウィングドメイン、ヘリカルスキャフォールドドメイン、および2つのヘリックスフィンガー(THF)26、27、28、29からなる(図1)。SecA-SecYEG複合体の以前の結晶学的研究において、THFの位置は、それがタンパク質転座に積極的に関与していることを示唆し、その後のシグナルペプチドとの架橋実験は、タンパク質転座におけるこの領域の重要性をさらに確立した30,31。FRETマッピング方法論を用いた以前の研究では、外因性シグナルペプチドがSecA2,32のこの領域に結合することが実証された。SecYEGチャネルへの挿入前のプレタンパク質のシグナル配列と初期成熟領域の立体構造と位置を完全に理解するために、初期成熟領域のシグナル配列と残基がSer-Glyリンカーを介してSecAに結合したタンパク質キメラを作成した(図1)。この生物学的に生存可能な構築物を用いて、プレタンパク質のシグナル配列および早期成熟領域が並行してTHFに結合することをさらに実証した2。続いて、FRETマッピング方法論を用いて、下記3のようにSecYEG存在下におけるシグナル配列および初期成熟領域の立体構造および位置を解明した。
SecA-SecYEG複合体33,34,35の3次元構造と結合部位の可能な位置に関する知識により、個々のFRET距離の交点が結合部位の位置を特定する場所にドナー-アクセプター標識を慎重に配置することができました。これらのFRETマッピング測定は、プレタンパク質のシグナル配列および早期成熟領域が、先端がSecEGチャネルの口に位置するヘアピンを形成することを明らかにし、ヘアピン構造がチャネル挿入前にテンプレート化されることを実証した。
プロトコル
1. ラベリングサイトの選択
- 既存のタンパク質構造上の推定結合部位を三角測量するための少なくとも3つの潜在的な標識部位を同定する。このとき、遺伝子融合によりSecA、SecYEG、およびSecAに結合したプレタンパク質が同定された2。
- 推定結合部位の25〜75Å以内およびタンパク質の比較的静的な領域における標識部位を選択し、その距離は、使用する特定のFRET色素対を決定するであろう36。互いに比較的異なるタンパク質領域内の標識部位の位置を特定し、それらの部位が三角形の頂点を記述し、推定結合部位が中央に位置するようにします(図1A-D)。
- 他のCys残基を有さないタンパク質における目的の標識部位にシステイン(Cys)残基を導入または同定し、特定部位の標識効率を向上させる37、38。
- 非天然アミノ酸、例えばp−アジドフェニルアラニンを、異なる色素で2つの異なる位置で1つのタンパク質を効果的に標識するために、クリックケミストリーで標識するために導入する39、40。
- Cys変異体の機能喪失について試験する。Cysレス変異体および非天然アミノ酸変異体の活性を、適切な活性アッセイを用いて検証する。この場合、活性は、増殖アッセイとそれに続くインビトロマラカイトグリーンATPaseアッセイ32、41、42で検証した。
2. タンパク質の標識
- 正確な標識のために、目的のタンパク質を少なくとも95%の純度に精製します。SecAおよびSecYEGタンパク質を、参考文献3に詳述されているプロトコールに従って精製する。標識プロセス中に一部のタンパク質が失われるため、このステップで少なくとも5 μgの精製タンパク質があることを確認してください。
- FRET測定には、R0値と標識部位間の予測距離に応じて2つの色素を選択します。R0値を推定し、蛍光タンパク質データベースからの情報を使用してドナー発光とアクセプター吸光度が重なり合うのを観察し、これは一般的に使用される色素のスペクトルも与える(https://www.fpbase.org/spectra/)36。
注:R0は、所定の色素対の転写効率が50 %になる距離として定義されます。マッピング実験では、予測距離を色素ペアのR0 値に近づけて、距離を正確に測定できるようにする必要があります。 - ステップ 1.1.1 で識別した位置にラベルを付けます。ドナー - アクセプター色素対を有する。
- 最適なタンパク質濃度、温度、pH、時間の長さ、および使用される特定の色素の緩衝液などのパラメータに特に注意を払って、製造業者の指示に従ってタンパク質を標識する43,44.
- 25 mM トリス塩酸 (pH 7.5)、25 mM KCl、1 mM EDTA(TKE) バッファー中で、1 ~ 2 mg/mL または約 10 μM の濃度でタンパク質を調製します。色素をジメチルホルムアミド(DMF)またはジメチルスルホキシド(DMSO)に終濃度1mMまで溶解する。攪拌しながら溶液に色素を滴下し、色素:タンパク質のモル比が5:1(50 μM色素:10 μMタンパク質)に達するまで加える。
- 穏やかに揺動させるガラスバイアル中で室温(RT)で4時間、または4°Cで一晩反応を進行させる。 β-メルカプトエタノールを加えて反応を停止する。
注:タンパク質がトリス緩衝液と適合しない場合は、リン酸またはHEPES緩衝液を使用することができる。pHを7.0〜7.5の範囲に維持してください。タンパク質がジスルフィド結合を有する場合、標識の前にDTTまたはTCEPなどの還元剤を添加する。色素を添加する前に透析またはゲルろ過によってDTTを除去する。
- 正確なFRET測定のために、適切な分子量カットオフ(MWCO)を備えた遠心濃縮器で遊離色素を除去し、標識タンパク質を保持したまま遊離色素を通過させます。
- 3 mL の濃縮器の上部に ~ 1 mL の水を入れて濃縮器膜を作製し、膜を通して水を遠心分離します (4,300 x g で少なくとも 10 分間)。
- 標識サンプルを濃縮器で遠心分離して遊離色素を除去します(4,300 x gで20分)。3〜4回繰り返し、流れを処分します。
- UV-Vis吸収分光法を用いて標識タンパク質の標識効率を確認してください。
注:FRET測定には、50%以上のラベリング効率が必要です。ラベリング効率が低いとFRET信号が減少し、測定の不正確さにつながる可能性があります。 - 250〜700nmの範囲で標識タンパク質のUV-Visスペクトルを取得し、タンパク質吸収バンドと色素極大吸収バンドの両方を観察する。色素の吸収ピークおよびタンパク質の280nmにおける吸光度を測定する。
- タンパク質の濃度を決定し、補正係数CF、および以下の式45、46を使用して色素からの寄与を補正する:
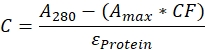
ここで、Cはタンパク質(M)の濃度、A280は280nmにおけるサンプル吸光度、Amaxは色素吸収極大における吸光度、εタンパク質は280nmにおけるタンパク質の吸光係数、CFは補正係数A'280/A'max、A'280は280nmにおける吸光度、A'maxは色素のみのピーク極大における吸光度である。 - 次の式を使用してラベリング効率を決定します。
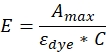
ここで 、ε色素 は色素のモル吸光係数、Cはステップ2.4.5で決定されたタンパク質の濃度、Eは標識効率です。ラベリング効率の値が頭打ちになり、100% 未満になるまで、手順 2.4.2 を繰り返します。
3. R 0 値を決定する
- R0 値をその場で測定します。同じ濃度の全タンパク質(4 μM)で2つのタンパク質サンプルを調製し、1つはドナー色素のみで標識されたタンパク質で、もう1つはアクセプター色素のみで標識されたタンパク質で調製します。SecAの場合、4μM SecAモノマーのタンパク質濃度は、これらの測定に適しています。
- 1 cm x 1 cm のキュベットの場合は 2.5 mL、5 mm x 5 mm のキュベットの場合は 600 μL、3 mm x 3 mm のキュベットの場合は 200 μL のサンプル容量を準備します。
- 分光蛍光光度計を使用している場合は、蛍光計をオンにし、蛍光ソフトウェアでスペクトル取得および分析プログラムを開きます。赤い M をクリックしてコンピュータを計測器に接続し(図2A)、 発光スペクトルを選択します。
- 励起波長、発光スキャンの範囲、温度、サンプルチェンジャー位置などのスキャンパラメータを、[実験の収集]メニュー項目を使用して入力します(図2B)。
- RTCをクリックし、色素の吸収極大に設定された励起波長を使用してピーク時の蛍光発光を監視することにより、機器の設定(スペクトルスリットなど)を最適化します。SecA の場合は、バンドパスを 1 nm に設定します。励起スリットおよび発光スリットをそれぞれ1および1.5mmとして、温度を25°Cおよび攪拌速度を250rpmとした。
メモ:計測器の1秒あたりのカウント数(cps)容量(通常は2 x 106 cps)を超えないようにしてください。
- ドナー標識タンパク質サンプルをサンプルホルダーに入れ、「 実行 」をクリックして、色素吸収極大(例えば、AF488の場合は488 nm)でサンプルを励起し、発光ピーク(AF488で標識されたドナーのみのSecAタンパク質の場合は505-750 nm)をスキャンすることにより、ドナー色素のみ(ドナーのみのタンパク質)で標識されたタンパク質の発光スキャンを生成します。
- ピークの終わりを越えてスキャンを25〜50nm延長することによって、スキャンのベースラインを確立します。ドナーのみのタンパク質の量子収率を測定し、47に記載の異なる濃度のサンプルに対して吸収および蛍光測定を行うことによって。これらの測定では、同じスリット設定を維持します。
- 量子収率の基準として遊離ドナー色素を使用する。正確な測定のために、ドナー専用タンパク質および遊離色素の少なくとも4つの測定値を異なる濃度で取得する。
- ドナー専用タンパク質および遊離色素または基準についての吸光度に対する蛍光強度または積分面積をプロットする。ドナー専用タンパク質の傾き(傾きD)と基準(傾きR)を決定します。
- 量子収率(Φ)は、次の式を使用して計算されます。
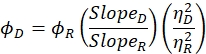
ここでΦDはドナーのみのタンパク質の量子収率であり、ΦRは遊離色素の量子収率であり(これは通常製造業者から入手できる)、スロープDおよびスロープRはドナーのみのタンパク質についてステップ3.4.2で求めた傾きをそれぞれ参照し、ηDおよびη Rとする。 ドナー専用タンパク質およびリファレンスフリー色素溶液の屈折の指標をそれぞれ表す47。
- アクセプターのみのタンパク質の吸収スペクトルを、1cmの経路長の細胞を用いて取得する。アクセプターのみのタンパク質の吸光係数スペクトルを生成するには、吸収スペクトルを色素濃度で除算します。
- スペクトル重なり積分を生成し、 J(λ) をグラフィカル解析プログラムを用いて行う。標準的なワークシートプログラム(スプレッドシートなど)もこのプロセスに使用できます。
- ドナー専用タンパク質の蛍光発光スペクトルにアクセプター専用タンパク質の吸光係数スペクトルを乗算し(ステップ3.4)、オーバーラップスペクトルを生成する。
- 得られたオーバーラップスペクトルにλ4を掛けます。
- 重なり領域の積分によって曲線の下の領域を決定する。オーバーラップ領域は、ドナー発光スペクトルにアクセプター吸光係数スペクトルを掛けた値が正の値をもたらす領域として定義される。スペクトルオーバーラップ積分は次のように定義されます。
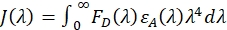
ここで、FD(λ)はドナー専用タンパク質(ステップ3.4で取得)の発光スペクトルであり、εA(λ)はアクセプターのみタンパク質の吸光係数スペクトルであり、M-1 cm-1(ステップ3.5で取得)の単位を有する。結果として得られるスペクトル重なり積分は、M-1 cm-1nm4の単位を有するべきである。 - スペクトル重なり積分を正規化します。重なり積分を、同じスペクトル範囲にわたるドナーのみのタンパク質スペクトルの積分領域で除算する:
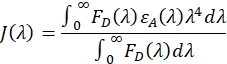
- 次の式を使用して、Å の R0 値を計算します。
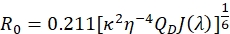
ここで、κ2は配向係数であり、典型的には自由に回転する染料の2/3として取られ、ηは屈折率であり、希薄水溶液の場合は1.33として近似することができ、QDはドナーの量子収率(ステップ3.4)、J(λ)はステップ3.6.35で決定されたスペクトル重なり積分である。注:染料が自由に回転していない場合は、Ivanov 48によって記述され、Auclair49およびZhang 2,3によって実装されるように補正を導入することができます。
4. FRETスペクトル測定の実行
- ドナーのみのタンパク質、アクセプターのみのタンパク質、およびドナー - アクセプタータンパク質のサンプルを同じ濃度で調製する。4 μM の濃度を推奨します。3 mm x 3 mm のキュベットを使用する場合は 200 μL、5 mm x 5 mm のキュベットを使用する場合は 600 μL、1 cm x 1 cm のキュベットを使用する場合は 2.5 mL の溶液を使用します。
- ドナー・アクセプタータンパク質サンプルは、ドナーのみとアクセプターのみの等モル量を用いて調製する。
- ドナーのみまたはアクセプターのみのサンプルのいずれかに等モル量の非標識タンパク質を導入することを通して、対照ドナーのみおよびアクセプターのみのタンパク質サンプルにおいて同じ量の標識サンプルを維持する。例えば、同じ濃度の溶液の場合、各ドナー専用FRETサンプルは、200μLの容量に対して100μLのドナー専用タンパク質および100μLの非標識タンパク質を含むであろう。
- ドナーのみ、アクセプターのみ、およびドナー-アクセプターサンプルの蛍光発光スペクトルを生成します。ステップ 3.2 の説明に従って信号を最適化します。最適化が完了したら、すべてのサンプルに対して同じ設定を維持します。
- ステップ 3.3 の説明に従ってドナー専用スキャンを取得します。ドナー色素吸収極大で溶液を励起し、ドナーおよび(予想される)アクセプター発光ピークにわたってスキャンする。
- サンプルをアクセプターのみのタンパク質に交換するか、サンプルチェンジャーの位置をアクセプターのみのタンパク質を含むキュベットに変更します。
- アクセプター色素のみで標識したタンパク質(アクセプターのみタンパク質)の発光スキャンをステップ4.2.1と同じ設定で取得します。ドナー励起波長で試料を励起する。
注:このスペクトルは、ドナー波長で励起されるアクセプタの量を補正します(ステップ5.1.2のFA ) - サンプルをドナーアクセプタータンパク質サンプルに交換するか、またはドナーアクセプター標識タンパク質を含むキュベットにサンプルチェンジャー位置を変更します。
- ステップ4.2.1および4.2.3と同じ設定を使用して、ドナー - アクセプタータンパク質サンプルの発光スキャンを取得します。
- すべてのスペクトルについて、スキャンの終了時に測定されたバックグラウンドカウントを差し引いてバックグラウンド蛍光を補正します。
- ドナーのみのドナー寿命を測定し、ステップ4.1.2で説明したように調製したドナー-アクセプターサンプルを測定します。ナノ秒(10-9 秒)の時間範囲で蛍光減衰を測定および解決できる時間相関単一光子計数蛍光装置を使用します。
メモ: FRET色素対の場合、励起光源をドナー色素の吸収極大に合わせます。- 楽器の電源を入れます。集録ソフトウェアを開き、蛍光分光器でデータ集録用の機器制御ソフトウェアを使用します。
- 集録には、時間範囲が55ns、ゲインが1~4096チャネルの TCSPCディケイを選択します。
- 非乳製品クリーマーまたは市販の散乱溶液の溶液を用いて機器応答関数(IRF)を取得し、490nmでの散乱をモニターする。スリット設定を調整し、必要に応じてニュートラル密度フィルタを使用して、パルスパイルアップ5を回避するのに十分な低いカウントレートを維持します。 [同意する] をクリックし、[ 開始] をクリックします。これにより、買収が開始されます。
メモ: 180 kHz の繰り返しレートには、4000 cps の最大カウント レートが使用されます。 - ピークチャネルが最大20,000カウントになるまで、IRFを490nmで収集します。各蛍光減衰を測定する前後にIRFを採取する。
- ドナー発光波長、520nmにおける蛍光発光をモニターすることによりドナーのみおよびドナー・アクセプターサンプルの蛍光減衰を得る。
- スリット設定を調整して、最大カウントレートを 4000 cps 以下にします。スリット設定は、通常、タンパク質サンプルの15~20 nmバンドパスです。ピークチャネルで20,000カウントが得られるまで減衰を収集します。
- 蛍光寿命(τ)に対する時間(t)の関数として減衰または蛍光強度(I)を分析する。次の式で減衰を指数の合計に当てはめます。
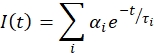
ここで、α I は i番目の成分の事前指数係数であり、τI は寿命である。フィット感は、蛍光減衰と一致するようにIRFと再畳み込まれます。減少したΧ2パラメータから適合の質を判断します。
5. FRETデータの解析
- ドナーに対するドナー・アクセプター試料のドナー強度の低下からFRET効率を次式のみで算出する。
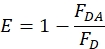
ここで、FDA はドナー・アクセプター試料の蛍光強度であり、 FDはドナー蛍光のピークにおけるドナーのみ試料の蛍光強度である。データがノイズが多い場合は、ピークの統合領域を使用します。- ドナーのみとドナー-アクセプターサンプルの間の標識の違いを訂正してください。ドナーのラベリングの程度に基づいて、次のように補正を計算します。
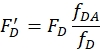
ここで、fDAはドナー-アクセプターサンプルにおけるドナー標識効率であり、fDはドナーのみサンプルにおける標識効率である。 - ドナー-アクセプタータンパク質スペクトルからアクセプターのみのタンパク質スペクトルを減算することによって(ステップ4.2)、ドナー励起スペクトルへのアクセプター蛍光の寄与を補正します。
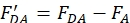
- ドナー-アクセプタータンパク質サンプルに対するアクセプターのみのタンパク質の標識効率の違いを補正し、効率を計算するための次の式を生成します。
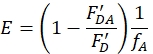
ここで 、fA は アクセプター標識の小数部数を示します。この式には、色素標識およびアクセプター蛍光によるすべての補正が含まれます。 - 次の式を使用して、効率からのFRET距離を計算します。
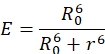
ステップ3.6.5で取得したR0値を使用する。 - ドナーのみの蛍光寿命と、ステップ4.3.3-4.3.5で測定されたドナー-アクセプターサンプルを使用してFRET効率を計算します。
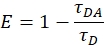
- 振幅加重寿命を使用してFRET効率を計算し、定常状態の結果と比較します5。
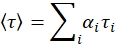
- ステップ5.1.4のように、蛍光寿命によって決定される効率から距離を計算します。FRETの効率と距離について定常状態と時間分解値を比較し、それらが互いの誤差内にあることを確認します。
- ドナーのみとドナー-アクセプターサンプルの間の標識の違いを訂正してください。ドナーのラベリングの程度に基づいて、次のように補正を計算します。
6. 距離のマッピング
- 計算された距離を使用して、結合部位を 3 次元構造にマッピングします。ステップ5.1.4で与えられた式と、各FRETペアのステップ3.6.5で得られたR0 値を使用して、調べたすべての色素ペアと位置の距離と誤差を計算します。
- PyMOL50 などの3Dグラフィカル表示プログラムを使用して、距離を構造にマップします( 補足ファイルに記載されているスクリプト)。スクリプトからのコマンドは、適切な距離情報とともにコマンドウィンドウに直接入力できます。
- 測定された距離と関連する誤差ごとにシェルを生成します(図3、図4、補足図1-3)。
- 異なるシェルの交点を通る位置をマッピングします (補足図 1-3)。シグナルペプチド結合部位は、SecAおよびSecYEG上の3つの異なる位置およびシグナルペプチド上の4つの異なる位置を介してマッピングされた(図1)。
結果
この研究は、SecYEGチャネルへのプレタンパク質の挿入前に、SecA上のプレタンパク質結合部位の位置を決定することに焦点を当てた。結合部位をマッピングするために、プレタンパク質の異なる領域と、SecAおよびSecYEGタンパク質上の3つの異なる位置との間でFRET実験を行った(図1A−D)。得られた距離とSecA、SecYEG、およびプレタンパク質の三次元?...
ディスカッション
FRETマッピング方法論を用いて、我々はSecAタンパク質上のシグナル配列結合部位を同定した。重要なことに、複合体の3次元結晶構造の存在は、我々の研究を大いに促進した。このマッピング手法の強みは、既存の構造を使用してラベル付けする場所を特定できることにあります。この方法論を使用して 3-D 構造を決定することはできません。しかしながら、構造要素56の決定...
開示事項
著者らには開示するものは何もありません。
謝辞
この研究は、米国国立衛生研究所助成金R15GM135904(IMに授与)および国立衛生研究所助成金GM110552(DBOに授与)によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 490 nm LED laser | Horiba | 1684-LED | |
| Alexa Fluor 647 C2 Maleimide//DIBO Alkyne | Life Technologies | A20347 | |
| Agar | Difco | DF0812 | |
| Alexa Fluor 488 C5 Maleimide/DIBO Alkyne | Life Technologies | A10254 | |
| Alexa Fluor 488 DIBO Alkyne | Life Technologies | S10904 | |
| Alexa Fluor 647 DIBO Alkyne | Life Technologies | S10906 | |
| Amicon Ultra4 Centrifugal filter (50kDa MWCO) | Sigma | UFC805008 | |
| Dodecylmaltoside (DDM) | Anatrace | D310 | |
| E. coli alkaline phosphatase signal peptide SP22 | Biomolecules Midwest | N/A | Synthesized custom item |
| extended signal peptide SP41 | Biomolecules Midwest | N/A | Synthesized custom item |
| FluorEssence | Horiba | version 2.4 | spectral acquisition program for Fluoromax4 spectrofluorometer |
| Fluoromax 4 spectrofluorometer | Horiba | N/A | |
| GlobalsWE | Laboratory for Fluorescence Dynamics, University of California, Irvine | spectral analysis program for time-resolved decays | |
| H4AzidoPheOH | BACHEM | 4020250.0001 | |
| LB (Miller) Broth | Fisher Scientific | BP9723 | |
| Ludox HS-40 colloidal silica (40 wt.% suspension in H2O) | Sigma-Aldrich | 420816 | dilution is needed to make a proper scattering solution |
| PTI Felix GX | Horiba | version 4.1.0.4096 | spectral acquisition program for PTI Time Master Instrument |
| PTI Time Master Instrument | Horiba | NA | |
| Pymol Molecular Graphics Program | Schrodinger | version 2.4 | |
| Water bath | Thermo Scientific | NESLAB RTE 10 |
参考文献
- Thompson, M. C., Yeates, T. O., Rodriguez, J. A. Advances in methods for atomic resolution macromolecular structure determination. F1000Research. 9, (2020).
- Zhang, Q., Li, Y., Olson, R., Mukerji, I., Oliver, D. Conserved SecA signal peptide-binding site revealed by engineered protein chimeras and Forester resonance energy transfer. Biochemistry. 55 (9), 1291-1300 (2016).
- Zhang, Q., et al. Alignment of the protein substrate hairpin along the SecA two-helix finger primes protein transport in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 114 (35), 9343-9348 (2017).
- Stryer, L. Fluorescence energy transfer as a spectroscopic ruler. Annual Review of Biochemistry. 47, 819-846 (1978).
- Lakowicz, J. R. . Principles of Fluorescence Spectroscopy. , (2006).
- Algar, W. R., Hildebrandt, N., Vogel, S. S., Medintz, I. L. FRET as a biomolecular research tool - understanding its potential while avoiding pitfalls. Nature Methods. 16 (9), 815-829 (2019).
- Magde, D., Wong, R., Seybold, P. G. Fluorescence quantum yields and their relation to lifetimes of rhodamine 6G and fluorescein in nine solvents: improved absolute standards for quantum yields. Photochemistry and Photobiology. 75 (4), 327-334 (2002).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in Enzymology. 211, 353-388 (1992).
- . R0 Values from Some Alexa Fluor Dyes - Table 1.6 Available from: https://www.thermofisher.com/us/en/home/references/molecular-probes-the-handbook/tables/r0-values-for-some-alexa-fluor-dyes.html (2021)
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A Guide to Fluorescent Protein FRET Pairs. Sensors. 16 (9), 1488 (2016).
- Day, R. N., Davidson, M. W. Fluorescent proteins for FRET microscopy: monitoring protein interactions in living cells. BioEssays : News and Reviews in Molecular, Cellular, and Developmental Biology. 34 (5), 341-350 (2012).
- Lee, S. J., Syed, S., Ha, T. Single-Molecule FRET Analysis of Replicative Helicases. Methods in Molecular Biology. 1805, 233-250 (2018).
- Uhm, H., Hohng, S. Single-Molecule FRET Assay for Studying Cotranscriptional RNA Folding. Methods in Molecular Biology. 2106, 271-282 (2020).
- Globyte, V., Joo, C. Single-molecule FRET studies of Cas9 endonuclease. Methods in Enzymology. 616, 313-335 (2019).
- Qiao, Y., Luo, Y., Long, N., Xing, Y., Tu, J. Single-Molecular Förster Resonance Energy Transfer Measurement on Structures and Interactions of Biomolecules. Micromachines. 12 (5), 492 (2021).
- Catipovic, M. A., Bauer, B. W., Loparo, J. J., Rapoport, T. A. Protein translocation by the SecA ATPase occurs by a power-stroke mechanism. The EMBO Journal. 38 (9), 101140 (2019).
- Seinen, A. B., Spakman, D., van Oijen, A. M., Driessen, A. J. M. Cellular dynamics of the SecA ATPase at the single molecule level. Scientific Reports. 11 (1), 1433 (2021).
- Dimura, M., et al. Quantitative FRET studies and integrative modeling unravel the structure and dynamics of biomolecular systems. Current Opinion in Structural Biology. 40, 163-185 (2016).
- Kalinin, S., et al. A toolkit and benchmark study for FRET-restrained high-precision structural modeling. Nature Methods. 9 (12), 1218-1225 (2012).
- Paetzel, M. Structure and mechanism of Escherichia coli type I signal peptidase. Biochimica et Biophysica Acta. 1843 (8), 1497-1508 (2014).
- Ng, D., Brown, J., Walter, P. Signal sequences specify the targeting route to the endoplasmic reticulum membrane. The Journal of Cell Biology. 134 (2), 269-278 (1996).
- Huber, D., et al. SecA Cotranslationally Interacts with Nascent Substrate Proteins In Vivo. Journal of Bacteriology. 199 (2), 00622 (2017).
- Lill, R., et al. SecA protein hydrolyzes ATP and is an essential component of the protein translocation ATPase of Escherichia coli. The EMBO Journal. 8 (3), 961-966 (1989).
- Lill, R., Dowhan, W., Wickner, W. The ATPase activity of SecA is regulated by acidic phospholipids, SecY, and the leader and mature domains of precursor proteins. Cell. 60 (2), 271-280 (1990).
- Kimura, E., Akita, M., Matsuyama, S., Mizushima, S. Determination of a region of SecA that interacts with presecretory proteins in Escherichia coli. The Journal of Biological Chemistry. 266 (10), 6600-6606 (1991).
- Hunt, J. F., et al. Nucleotide control of interdomain interactions in the conformational reaction cycle of SecA. Science. 297 (5589), 2018-2026 (2002).
- Sharma, V., et al. Crystal structure of Mycobacterium tuberculosis SecA, a preprotein tranlsocating ATPase. Proceedings of the National Academy of Sciences of the United States of America. 100 (5), 2243-2248 (2003).
- Vassylyev, D., et al. Crystal structure of the translocation ATPase SecA from Thermus thermophilus reveals a parallel, head-to-head dimer. Journal of Molecular Biology. 364 (3), 248-258 (2006).
- Zimmer, J., Li, W., Rapoport, T. A. A novel dimer interface and conformational changes revealed by an X-ray structure of B. subtilis SecA. Journal of Molecular Biology. 364 (3), 259-265 (2006).
- Zimmer, J., Rapoport, T. A. Conformational flexibility and peptide interaction of the translocation ATPase SecA. Journal of Molecular Biology. 394 (4), 606-612 (2009).
- Bauer, B. W., Rapoport, T. A. Mapping polypeptide interactions of the SecA ATPase during translocation. Proceedings of the National Academy of Sciences of the United States of America. 106 (49), 20800-20805 (2009).
- Auclair, S., et al. Mapping of the signal peptide-binding domain of Escherichia coli SecA using Förster resonance energy transfer. Biochemistry. 49 (4), 782-792 (2010).
- Zimmer, J., Nam, Y., Rapoport, T. A. Structure of a complex of the ATPase SecA and the protein-translocation channel. Nature. 455 (7215), 936-943 (2008).
- Li, L., et al. Crystal structure of a substrate-engaged SecY protein-translocation channel. Nature. 531 (7594), 395-399 (2016).
- Ma, C., et al. Structure of the substrate-engaged SecA-SecY protein translocation machine. Nature Communications. 10 (1), 2872 (2019).
- Lambert, T. J. FPbase: a community-editable fluorescent protein database. Nature Methods. 16 (4), 277-278 (2019).
- Jilaveanu, L. B., Oliver, D. In vivo membrane topology of Escherichia coli SecA ATPase reveals extensive periplasmic exposure of multiple functionally important domains clustering on one face of SecA. The Journal of Biological Chemistry. 282 (7), 4661-4668 (2007).
- Ramamurthy, V., Oliver, D. Topology of the integral-membrane form of Escherichiacoli SecA protein. The Journal of Biological Chemistry. 272 (37), 23239-23246 (1997).
- Chin, J., et al. Addition of p-Azido-L-phenylalanine to the genetic code of Escherichia coli. Journal of the American Chemical Society. 124 (31), 9026-9027 (2002).
- Deiters, A., et al. Adding amino acids with novel reactivity to the genetic code of Saccharomyces cerevisiae. Journal of the American Chemical Society. 125 (39), 11782-11783 (2003).
- Lanzetta, P. A., Alvarez, L. J., Reinach, P. S., Candia, O. A. An improved assay for nanomole amounts of inorganic phosphate. Analytical Biochemistry. 100 (1), 95-97 (1979).
- Mitchell, C., Oliver, D. B. Two distinct ATP-binding domains are needed to promote protein export by Escherichia coli SecA ATPase. Molecular Microbiology. 10 (3), 483-497 (1993).
- Thiol-reactive Probe Labeling Protocol. Thermo Fischer Scientific Available from: https://www.thermofisher.com/us/en/home/references/protocols/cell-and-tissue-analysis/labeling-chemistry-protocols/thiol-reactive-probe-labeling-protocol.html (2021)
- Click Chemistry - Section 3.1. Thermo Fischer Scientific Available from: https://www.thermofisher.com/us/en/home/references/molecular-probes-the-handbook/reagents-for-modifying-groups-other-than-thiols-or-amines/click-chemistry.html (2021)
- Correction Factor. AAT Bioquest Available from: https://www.aatbio.com/resources/correction-factor/ (2019)
- Calculate dye:protein (F/P) molar ratios. Thermo Fischer Scientific Available from: https://tools.thermofischer.com/content/sfs/brochures/TR0031-Calc-FP-rations.pdf (2011)
- A Guide to Recording Fluorescence Quantum Yields. Horiba Scientific Available from: https://static.horiba.com/fileadmin/Horiba/Application/Materials/Material_Research/Quantum_Dots/quantumyieldstrad.pdf (2011)
- Ivanov, V., Li, M., Mizuuchi, K. Impact of emission anisotropy on fluorescence stectroscopy and FRET distance measurements. Biophysical Journal. 97 (3), 922-929 (2009).
- Auclair, S., Oliver, D., Mukerji, I. Defining the solution state dimer structure of Escherichia coli SecA using Forster resonance energy transfer. Biochemistry. 52 (14), 2388-2401 (2013).
- . The PyMOL Molecular Graphics System, Version 2.4 Available from: https://pymol.org/2/ (2021)
- Musial-Siwek, M., Rusch, S. L., Kendall, D. A. Selective photoaffinity labeling identifies the signal peptide binding domain on SecA. Journal of Molecular Biology. 365 (3), 637-648 (2007).
- Miller, A., Wang, L., Kendall, D. A. Synthetic signal peptides specifically recognize SecA and stimulate ATPase activity in the absence of preprotein. The Journal of Biological Chemistry. 273 (19), 11409-11412 (1998).
- Gelis, I., et al. Structural basis for signal-sequence recognition by the translocase motor SecA as determined by NMR. Cell. 131 (4), 756-769 (2007).
- Erlandson, K. J., et al. A role for the two-helix finger of the SecA ATPase in protein translocation. Nature. 455 (7215), 984-988 (2008).
- Das, S., Oliver, D. Mapping of the SecA-SecY and SecA-SecG interfaces by site-directed in vivo photocross-linking. The Journal of Biological Chemistry. 286 (14), 12371-12380 (2011).
- Wheatley, E. G., Pieniazek, S. N., Vitoc, I., Mukerji, I., Beveridge, D. L. Molecular Dynamics Structure Prediction of a Novel Protein-DNA Complex: Two HU Proteins with a DNA Four-way Junction. Innovations in Biomolecular Modeling and Simulations: Volume 2. , 111-128 (2012).
- Vitoc, C. I., Mukerji, I. HU binding to a DNA four-way junction probed by Förster resonance energy transfer. Biochemistry. 50 (9), 1432-1441 (2011).
- Hellman, L. M., Fried, M. G. Electrophoretic mobility shift assay (EMSA) for detecting protein-nucleic acid interactions. Nature Protocols. 2 (8), 1849-1861 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved