Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse quantitative des propriétés viscoélastiques des globules rouges à l’aide de pinces optiques et de microscopie à défocalisation
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici, un protocole intégré basé sur des pinces optiques et une microscopie de défocalisation est décrit pour mesurer les propriétés rhéologiques des cellules. Ce protocole a une large applicabilité dans l’étude des propriétés viscoélastiques des érythrocytes dans des conditions physiopathologiques variables.
Résumé
Les propriétés viscoélastiques des érythrocytes ont été étudiées par une gamme de techniques. Cependant, les données expérimentales rapportées varient. Ceci n’est pas seulement attribué à la variabilité normale des cellules, mais aussi aux différences dans les méthodes et les modèles de réponse cellulaire. Ici, un protocole intégré utilisant des pinces optiques et une microscopie défocalisante est utilisé pour obtenir les caractéristiques rhéologiques des globules rouges dans la gamme de fréquences de 1 Hz à 35 Hz. Alors que des pincettes optiques sont utilisées pour mesurer la constante élastique du complexe érythrocytaire, la microscopie de défocalisation est capable d’obtenir le profil de hauteur de la cellule, le volume et son facteur de forme, un paramètre qui permet la conversion de la constante élastique complexe en module de cisaillement complexe. De plus, en appliquant un modèle de rhéologie vitreuse douce, l’exposant de mise à l’échelle pour les deux modules peut être obtenu. La méthodologie développée permet d’explorer le comportement mécanique des globules rouges, en caractérisant leurs paramètres viscoélastiques, obtenus dans des conditions expérimentales bien définies, pour plusieurs conditions physiologiques et pathologiques.
Introduction
Les globules rouges matures (GR), également appelés érythrocytes, sont capables de s’étendre plus de deux fois leur taille lorsqu’ils traversent les capillaires les plus étroits du corps humain1. Cette capacité est attribuée à leur capacité unique à se déformer lorsqu’ils sont soumis à des charges externes.
Au cours des dernières années, différentes études ont caractérisé cette caractéristique dans les surfaces de globules rouges 2,3. Le domaine de la physique qui décrit les réponses élastiques et visqueuses des matériaux dues aux charges externes est appelé rhéologie. En général, lorsqu’une force externe est appliquée, la déformation résultante dépend des propriétés du matériau et peut être divisée en déformations élastiques, qui stockent de l’énergie, ou déformations visqueuses, qui dissipent l’énergie4. Toutes les cellules, y compris les globules rouges, présentent un comportement viscoélastique; En d’autres termes, l’énergie est à la fois stockée et dissipée. La réponse viscoélastique d’une cellule peut ainsi être caractérisée par son module de cisaillement complexe G*(ω) = G'(ω) + iG"(ω), où G'(ω) est le module de stockage, lié au comportement élastique, et G » (ω) est le module de perte, lié à sa viscosité4. De plus, des modèles phénoménologiques ont été utilisés pour décrire les réponses cellulaires, l’un des plus utilisés est appelé le modèle de rhéologie vitreusedouce 5, caractérisé par une dépendance de la loi de puissance du module de cisaillement complexe avec la fréquence de charge.
Des méthodes à cellule unique ont été utilisées pour caractériser les propriétés viscoélastiques des globules rouges, en appliquant une force et en mesurant le déplacement en fonction de la charge imposée 2,3. Cependant, pour le module de cisaillement complexe, peu de résultats peuvent être trouvés dans la littérature. En utilisant la diffusion dynamique de la lumière, les valeurs des modules de stockage et de perte de globules rouges ont été rapportées variant de 0,01 à 1 Pa, dans la gamme de fréquences de 1 à 100 Hz6. En utilisant la cytométrie optique de torsion magnétique, un module d’élasticité complexe apparent a été obtenu7, et à des fins de comparaison, un facteur multiplicatif a été revendiqué pour éventuellement clarifier les écarts.
Plus récemment, une nouvelle méthodologie basée sur les pinces optiques (OT) ainsi que la microscopie de défocalisation (DM), en tant qu’outil intégré pour cartographier quantitativement le stockage et la perte des modules de cisaillement des érythrocytes humains sur des charges dépendantes du temps, a été établie 8,9. De plus, un modèle de rhéologie vitreuse souple a été utilisé pour ajuster les résultats et obtenir un coefficient de loi de puissance qui caractérise les globules rouges 8,9.
Dans l’ensemble, la méthodologie développée8,9, dont le protocole est décrit en détail ci-dessous, clarifie les divergences précédentes en utilisant les valeurs mesurées pour le facteur de forme, Ff, qui relie les forces et les déformations aux contraintes et aux déformations dans la surface des globules rouges et peut être utilisée comme nouvelle méthode de diagnostic capable de déterminer quantitativement les paramètres viscoélastiques et les caractéristiques vitreuses molles des globules rouges obtenus à partir de personnes ayant du sang différent. Pathologies. Une telle caractérisation, à l’aide du protocole décrit ci-dessous, peut ouvrir de nouvelles possibilités pour comprendre le comportement des globules rouges d’un point de vue mécanobiologique.
Access restricted. Please log in or start a trial to view this content.
Protocole
Des échantillons de sang humain ont été fournis par des hommes et des femmes adultes volontaires selon les protocoles approuvés par le Comité d’éthique de la recherche de l’Université fédérale de Rio de Janeiro (Protocole 2.889.952) et enregistrés sur la plate-forme brésilienne sous le numéro CAAE 88140418.5.0000.5699. Un formulaire écrit de consentement a été délivré à tous les bénévoles et recueilli auprès d’eux. Les personnes atteintes d’hémoglobinopathie et/ou prenant des médicaments contrôlés ont été exclues. L’ensemble du processus a suivi les lignes directrices approuvées par le comité d’éthique de l’institut.
1. Préparation des porte-échantillons
- Acquérir deux lamelles de recouvrement (24 mm x 60 mm et 24 mm x 32 mm; épaisseur = 0,13-0,17 mm) et une bague en caoutchouc (diamètre = 10 mm; épaisseur = 2 mm) pour chaque porte-échantillon.
- Versez de la graisse de silicone sur la surface de l’anneau en caoutchouc de manière à couvrir tout le périmètre.
- Placez l’anneau en caoutchouc sur le couvercle avec le côté gras face au couvercle. Attendez 5 minutes pour une fixation correcte, les porteurs d’échantillons sont alors prêts à recevoir la culture cellulaire.
REMARQUE: En outre, des plats à fond en verre commerciaux ou faits maison peuvent également être utilisés, comme décrit précédemment10.
2. Culture cellulaire
REMARQUE: Les étapes ci-dessous décrivent comment obtenir des globules rouges sains à partir du sang humain. Il est important que les échantillons soient fraîchement préparés avant chaque expérience.
- Diluer 20 μL de sang dans 250 μL de 1x solution saline tampon phosphate (PBS) contenant 137 mM NaCl, 2,7 mM KCl, 10 mMNa2HPO4, 1,8 mM KH2PO4, 10 mM de glucose et supplémenter avec 1 mg/mL d’albumine sérique bovine (BSA).
- Après centrifugation à 200 x g pendant 2 min à température ambiante, aspirer le surnageant à l’aide d’une pipette et remettre en suspension la pastille de cellule dans 1 ml de 1x solution PBS/BSA. Lavez les cellules 2x dans le tampon.
- Calculer la densité cellulaire à l’aide d’un hémocytomètre et ensemencer 50 000 à 100 000 cellules dans le porte-échantillon préparé à l’étape 1. Attendez 10-15 min pour la fixation de cellule non spécifique à la lame-couverture; Le temps d’attente n’affecte pas les cellules.
- Ajouter à l’échantillon 0,2 μL d’une solution sphérique de polystyrène à 10 % v/v (rayon = 1,52 ± 0,02 μm) pour d’autres expériences d’OT. Confirmer le bon mélange en regardant les échantillons au microscope.
- Après l’ensemencement des cellules, placez simplement le deuxième couvercle au-dessus de l’anneau en caoutchouc (il n’est pas nécessaire d’ajouter de la graisse pour la fixation), fermez la configuration et terminez la préparation de l’échantillon. Les échantillons sont prêts pour l’analyse et la manipulation microscopiques.
3. Configuration du microscope à pince optique
REMARQUE: Les OT sont des outils qui utilisent un faisceau laser hautement focalisé pour piéger des objets microscopiques et mesurer des forces dans la gamme piconewton et des déplacements à l’échelle nanométrique. Le laser OT utilisé (longueur d’onde 1064 nm) doit être correctement aligné, comme décrit précédemment10.
- Brièvement, à l’aide d’au moins deux miroirs séparés par une distance de quelques centimètres (10-20 cm au moins), dirigez un faisceau laser polarisé linéairement vers l’entrée arrière d’un microscope inversé. Alignez précisément le faisceau laser pour entrer dans le microscope en ligne droite (Figure 1).
- Ensuite, réfléchissez le faisceau laser à l’aide d’un miroir dichroïque, installé au microscope, pour procéder parallèlement à l’axe de la lentille de l’objectif et entrer dans la lentille près du centre de son entrée arrière. Cela focalisera le laser pour créer le piège optique (Figure 1).
- Ensuite, pour mesurer les forces avec OT, calibrez le système pour obtenir la rigidité du piège (κOT). Voir10 pour une description plus détaillée de la procédure d’étalonnage OT. Une fois κ OT trouvé, le système OT est prêt pour les expériences de rhéologie.
4. Configuration du DM
REMARQUE: DM est une technique de microscopie optique à fond clair qui permet à des objets transparents de devenir visibles si le microscope est légèrement défocalisé11,12. Une telle technique a été appliquée pour obtenir la forme RBC13. Le même microscope utilisé pour le système OT peut être utilisé pour DM, afin d’obtenir un profil de hauteur grâce à des reconstructions 3D.
- Ajustez le système d’éclairage du microscope en effectuant l’éclairage Köhler14 et, pour une meilleure résolution, ouvrez complètement le condensateur-diaphragme pour effectuer les expériences.
- Utilisez un système de positionnement piézoélectrique pour déplacer l’échantillon dans toutes les coordonnées, avec une précision nanométrique sur l’axe z. Effectuer l’auto-étalonnage du système piézoélectrique sur tous les axes. Une fois toutes les procédures effectuées, le système de microscope est prêt pour les expériences de DM.
5. Expérience et analyse de rhéologie basée sur l’OT
NOTE: L’expérience rhéologique consiste à observer les réponses de la cellule à de petites oscillations de fréquences variables.
- Expérimentation
- À l’aide du système OT, piéger la sphère avec le laser OT, puis l’attacher à un globule rouge en pressant la sphère contre la surface de la cellule près de la surface supérieure et près du bord de la cellule. Utilisez le microscope pour cette étape. Ensuite, trapez une autre sphère et répétez la même procédure de fixation, mais attachez-la maintenant à la lamelle de couverture, près de la cellule. La sphère fixée à la lamelle de couverture est le cordon de référence (Figure 2), qui est nécessaire pour suivre le déplacement piézo et comparer avec la sphère RBC.
- Assurez-vous que la cellule choisie est bien fixée à la lamelle de couverture et que les sphères de référence et de référence ont adhéré à la surface et à la lamelle de couverture, respectivement, avant de commencer la mesure. Identifiez visuellement les cellules non adhérées au fur et à mesure qu’elles se déplaceront au fil du temps, malgré le taux d’adhésion élevé (environ 80% à 90%).
- Ajouter une fonction sinusoïdale d’amplitude, ξ 0 = 0,500 ± 0,001 μm et des fréquences variables de 1 Hz, 7 Hz, 14 Hz, 21 Hz, 28 Hz et 35 Hz, avec des fréquences angulaires respectives, ω, de 6,3 rad/s, 169 rad/s, 88 rad/s, 132 rad/s, 176 rad/s et 220 rad/s, au logiciel de la scène piézoélectrique, à l’aide du logiciel piézoélectrique, comme démontré précédemment 8,9.
- À l’aide de l’étage piézoélectrique, appuyez sur le bouton de démarrage pour permettre le déplacement piézoélectrique et maintenir la sphère RBC dans le piège, soumettez l’échantillon à un cycle de mouvements en utilisant la fonction sinusoïdale précédemment définie. Utilisez une caméra capable de produire des images à 790 images/s ou plus pour enregistrer le mouvement de l’échantillon. Un schéma de l’expérience est présenté à la figure 2.
- Pendant que l’échantillon est soumis à des mouvements sinusoïdaux, activez l’OT pour piéger la sphère attachée à la surface des globules rouges. Quelle que soit la température choisie pour réaliser les expériences, température ambiante, 37 °C, ou une autre température, surveillez attentivement la température pour éviter les variations lors des mesures. Le laser infrarouge (1064 nm) utilisé pour créer l’OT ne provoque presque aucun dommage ou chauffage des cellules.
- Analyse
- Analysez les images obtenues lors des mouvements sinusoïdaux à l’aide d’ImageJ pour trouver la position du centre de masse de chacune des sphères au fil du temps.
NOTE: Ces données permettent de générer des graphiques capables de montrer les différences de phase et d’amplitude entre les deux sphères. Ces informations sont cruciales pour obtenir la réponse viscoélastique des globules rouges. - Pour obtenir le centre de masse de chacune des sphères, ouvrez le logiciel ImageJ. Importez le film entier obtenu lors des mouvements sinusoïdaux.
- Dans l’onglet Image , cliquez sur Ajuster, puis sélectionnez Seuil. La fenêtre de seuil s’ouvre. Sélectionnez N&B. Cela rendra l’arrière-plan blanc et les sphères noires.
- Ajustez le seuil avec les deux barres de défilement sous l’histogramme afin que les deux sphères apparaissent avec le nombre maximal de pixels.
- Sélectionnez la sphère de référence en cliquant sur Fichier > Rectangle. Dessinez un rectangle pour sélectionner la sphère. Après avoir sélectionné la sphère de référence dans une image, assurez-vous que le rectangle sélectionne également correctement la même sphère dans toutes les autres images du film.
- Ensuite, dans l’onglet Analyser , cliquez sur Définir les mesures et sélectionnez l’option Centre de masse .
- Cliquez à nouveau sur l’onglet Analyser et sélectionnez Analyser les particules. Une nouvelle fenêtre s’ouvrira. Définissez la taille et la circularité (en fonction du rayon de la sphère). Cochez les cases suivantes Afficher les résultats et Effacer les résultats. Enfin, cliquez sur OK pour traiter toutes les images.
- Une nouvelle fenêtre contenant un tableau avec les coordonnées xy pour le centre de la masse apparaîtra. Enregistrez ces valeurs de coordonnées en tant que fichier .txt. Répétez la procédure pour l’autre sphère, fixée à la surface RBC.
- Pour obtenir l’amplitude et les différences de phase pour les deux sphères, ouvrez le logiciel d’analyse. Importez les fichiers .txt précédemment obtenus.
- Créez une table à trois colonnes. Dans la première colonne (c0), ajouter le nombre d’images, dans la deuxième colonne (c1) ajouter les coordonnées x pour la sphère de référence, et dans la troisième colonne (c2), les coordonnées x pour la sphère attachée à la surface RBC.
Remarque : Dans cet exemple, comme les mouvements sinusoïdaux ont été effectués uniquement sur l’axe des abscisses, il est seulement nécessaire d’utiliser les coordonnées x pour les deux sphères. - Ensuite, corrélez les images avec le temps. Cliquez sur Windows > Formula Entry. Une nouvelle fenêtre appelée entrée de formule s’ouvre. Dans cette fenêtre, il est possible de mettre en place huit équations différentes, chacune dans une clé spécifique (de F1 à F8).
- Sélectionnez F1, tapez la formule suivante :
C 3 = C0 / (Caméra FPS)
Cliquez sur Exécuter. Cela créera une nouvelle colonne pendant un certain temps dans la table (colonne 4). - Soustrayez la valeur de coordonnées x de chaque image par sa valeur moyenne respective. Pour ce faire, désignez deux clés quelconques sur l’entrée de formule et tapez les équations suivantes pour chaque clé :
C 4 = (C1 - moyenne(c 1)) et c 5 = (c 2 - moyenne(c2))
Cliquez sur le bouton Exécuter . Les résultats apparaîtront dans les colonnes 5 et 6 pour les sphères de référence et les globules rouges, respectivement. - Convertissez les valeurs du centre de masse des pixels en micromètres. Pour cela, utilisez une autre clé sur l’entrée de formule et tapez l’équation suivante :
C 4 = C 4 / Numéro de conversion
Répétez le même processus pour la colonne 5.
REMARQUE: La conversion est acquise à l’aide d’une échelle / règle micrométrique et obtenir son image avec la même configuration de microscope utilisée pour les mesures (y compris la même lentille d’objectif). Cette procédure peut être effectuée pendant l’étalonnage du microscope. Une relation pixel/micromètre est ainsi obtenue. - Générez un tracé avec les centres de masse des deux sphères sur l’axe des y et le temps sur l’axe des x. Pour cela, cliquez sur Galerie > Linéaire et Disperser. Une nouvelle fenêtre s’ouvrira. Sélectionnez la colonne de temps pour l’axe des x et dans l’axe des y, sélectionnez les colonnes du centre de masse en micromètres pour les sphères de référence et de RBC.
- Dans le graphique, sélectionnez uniquement les données relatives à la première fréquence angulaire (6,3 rad/s). Utilisez les outils indiqués à la figure 3.
- Définissez l’équation qui ajuste la courbe de données pour la sphère de référence. Pour cela, cliquez sur Curve Fit > General et Fit1, cochez la case de données liées à la position de la sphère de référence, puis cliquez sur Définir. Une nouvelle fenêtre s’ouvrira pour définir l’équation. La position ξ(t) de la sphère de référence est décrite par:
ξ(t) = ξ0cos(ωt)
où ξ est le mouvement sinusoïdal de l’échantillon, ω est la fréquence angulaire et t est le temps en s. En transposant cette équation au logiciel d’analyse, elle se présentera comme suit :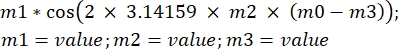 , où m1 est ξ, m2 est f, m0 est le temps t, et m3 est la phase de la fonction cosinus pour t = 0.
, où m1 est ξ, m2 est f, m0 est le temps t, et m3 est la phase de la fonction cosinus pour t = 0. - Estimez les valeurs pour m1, m2 et m3 à partir du graphique. Après avoir défini l’équation, cliquez sur OK. Les données seront ajustées en fonction de l’équation et une courbe accompagnée d’un petit carré apparaîtra dans le graphique avec les valeurs de m1, m2 et m3.
- Définissez l’équation qui ajuste la courbe de données pour la sphère RBC. Pour cela, cliquez sur Ajustement de la courbe > Général et définissez l’équation qui ajustera la courbe pour les données. La position ρ(t) de la sphère RBC est donnée par:
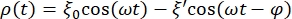
où ξ' est l’amplitude hors phase, et φ est l’angle hors phase. En transposant cette formule au logiciel d’analyse, elle se présentera comme suit :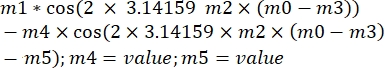 , où m1, m2 et m3 sont les valeurs obtenues dans l’ajustement de la courbe de la sphère de référence. m4 est ξ' et m5 est φ.
, où m1, m2 et m3 sont les valeurs obtenues dans l’ajustement de la courbe de la sphère de référence. m4 est ξ' et m5 est φ. - Estimez les valeurs pour m4 et m5 à partir de la placette. Après avoir défini la formule, cliquez sur OK. Les données seront ajustées en fonction de l’équation, et une courbe avec un petit carré apparaîtra dans le graphique avec les valeurs de m4 et m5.
- Ensuite, créez une nouvelle table pour ajouter les données obtenues à partir de l’ajustement de la courbe dans leurs colonnes respectives. Définissez cinq colonnes différentes pour les paramètres suivants : fréquence angulaire, amplitude (sphère de référence), temps initial, amplitude (sphère RBC) et angle hors phase. Effectuez la même procédure pour toutes les autres fréquences.
- Utilisez les équations suivantes pour trouver les constantes de stockage (K') et de perte (K ») :
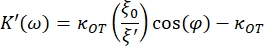
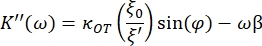
où κOT est la constante élastique OT et β est le coefficient de traînée de Stokes. En transposant les équations au logiciel d’analyse, il se présentera comme suit :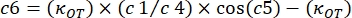 et
et
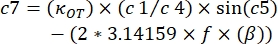 , où β et κOT doivent être remplacés par les valeurs trouvées dans le système.
, où β et κOT doivent être remplacés par les valeurs trouvées dans le système. - Tracez les résultats sur un graphique, en utilisant l’axe des x pour K » et l’axe des y pour K' (Figure 4).
- Analysez les images obtenues lors des mouvements sinusoïdaux à l’aide d’ImageJ pour trouver la position du centre de masse de chacune des sphères au fil du temps.
6. Expérience et analyse du DM pour obtenir le facteur de forme cellulaire global
- Acquisition vidéo
- Déplacez l’étage piézoélectrique dans la direction xy à l’aide du logiciel pour rechercher une cellule isolée attachée à la lame. Pièger et fixer une sphère de polystyrène de diamètre connu à la surface des globules rouges. À l’aide de l’étage piézoélectrique, déplacez légèrement la perle piégée, également fixée à la surface des globules rouges, afin de déformer la cellule, puis fixez la perle à la lamelle de couverture.
REMARQUE: Il est également possible d’utiliser la même cellule à partir des mesures OT. - Modifiez la position de l’axe z pour rechercher l’image focalisée, où le plan de mise au point se trouve au milieu de la cellule choisie. Cette image présente le plus petit contraste avec le niveau de gris au centre de la cellule égal au niveau de gris à l’extérieur de la cellule (arrière-plan).
- Lorsque la position est fixe, utilisez le logiciel de l’appareil photo pour créer un film de la cellule entière avec environ 5 000 images à 8 bits et 256 pixels x 256 pixels, à une fréquence d’images de 25 ips. Ensuite, déplacez la position de l’axe z de 2 μm vers le bas ou vers le haut pour obtenir une image défocalisée pour la cellule choisie. Répétez les paramètres pour créer un film pour cette situation.
- Enfin, sans modifier la position de l’axe z, recherchez une région sans cellules pour répéter la même procédure et créer un film de l’arrière-plan de l’image.
- Déplacez l’étage piézoélectrique dans la direction xy à l’aide du logiciel pour rechercher une cellule isolée attachée à la lame. Pièger et fixer une sphère de polystyrène de diamètre connu à la surface des globules rouges. À l’aide de l’étage piézoélectrique, déplacez légèrement la perle piégée, également fixée à la surface des globules rouges, afin de déformer la cellule, puis fixez la perle à la lamelle de couverture.
- Acquisition d’images de contraste
- Convertissez chacun des trois films en trois images moyennes. À l’aide d’ImageJ, sélectionnez l’un des films, cliquez sur Image > Stacks > Z Project et choisissez l’option Intensité moyenne . Répétez cette procédure pour que les autres films obtiennent leurs images respectives.
- Changez toutes les images obtenues de 8 bits à virgule flottante 32 bits. À l’aide d’ImageJ, cliquez sur Image > Type > 32 bits. Ensuite, cliquez sur Analyser > définir les mesures et choisissez l’option Valeur de gris moyenne . Enfin, cliquez à nouveau sur Analyser > mesurer.
- Ensuite, cliquez sur Traiter > calculatrice d’image et divisez l’image focalisée par l’image d’arrière-plan. Pour ce résultat, multipliez la valeur moyenne du niveau de gris de l’image focalisée. Obtenez la valeur moyenne en cliquant sur Traiter > Math > Multiplier.
- Pour obtenir la valeur moyenne, choisissez l’image focalisée, puis cliquez sur Analyser > mesurer. Pour l’image représentative, la valeur moyenne du niveau de gris est de 69 199. Répétez les procédures ci-dessus pour l’image défocalisée. Dans ce cas, la valeur moyenne du niveau de gris est 69 231. La figure 5 montre les images focalisées et défocalisées avant et après la procédure.
- Les images peuvent perdre le contraste visuel pendant les opérations. Pour mieux visualiser les images, cliquez sur Image > Ajuster > luminosité / contraste et sélectionnez l’option Auto .
- Ensuite, pour trouver le contraste de l’image, utilisez la relation suivante :
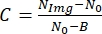 , où N Img est le niveau de gris de la cellule, N0 est le niveau de gris à l’extérieur de la cellule, et est un paramètre constant correspondant au niveau de gris pour une intensité lumineuse nulle qui dépend de la caméra.
, où N Img est le niveau de gris de la cellule, N0 est le niveau de gris à l’extérieur de la cellule, et est un paramètre constant correspondant au niveau de gris pour une intensité lumineuse nulle qui dépend de la caméra. - Pour trouver la valeur B, utilisez un capteur de puissance pour mesurer l’intensité lumineuse. Pour chaque valeur d’intensité lumineuse, une vidéo doit être enregistrée; Ainsi, différentes valeurs d’intensité peuvent être liées à différents niveaux de gris. Enfin, obtenez un ajustement linéaire et reliez B à l’intensité lumineuse nulle15.
- Trouvez la valeur de N0, cliquez sur l’icône Sélection de polygone et dessinez un polygone tel que celui de la figure 6. Ensuite, cliquez sur l’onglet Analyser et sélectionnez Mesurer pour trouver le niveau de gris moyen pour la zone sélectionnée. Chaque image est formée par un ensemble de pixels et chaque pixel a un certain niveau de gris. L’ensemble de tous les niveaux de gris dus à tous les pixels qui composent l’image correspond à NImg.
- Utilisez l’équation de contraste pour déterminer N Img - No et exécutez-la en sélectionnant Processus > Math > Soustraire. Diviser le résultat par N0 - B. Enfin, trouvez le contraste pour l’image focalisée (C 0) et défocalisée (C1).
- Obtention du profil de hauteur
- Pour obtenir le profil de hauteur, utilisez la méthode13 déjà décrite. En bref, utilisez la transformée de Hartley (FHT) pour obtenir l’épaisseur RBC. Dans ImageJ, cliquez sur Traiter > FFT > Options FFT, puis choisissez FHT.
- Soustrayez les images C0 et C1 dans ImageJ à l’aide de Process > Math > Soustrayez pour obtenir l’image pour la procédure suivante, C= C0 - C1.
- Pour l’image C, cliquez sur Processus > FFT > Options FFT, puis choisissez FHT pour effectuer la transformation de Hartley de l’image. Ensuite, divisez par la fréquence spatiale q2 à l’aide du plugin personnalisé DivideQ2. Cliquez sur Plugin > DivideQ2.
Remarque : Le fichier DivideQ2.class doit être copié dans le répertoire plugins où ImageJ est installé. Le plugin est fourni sous forme de fichier .class (fichier supplémentaire 1) à inclure dans le dossier du plugin d’ImageJ. - Ensuite, effectuez la transformation inverse FHT en utilisant Process > FFT > FFT pour obtenir une image avec un niveau de gris proportionnel à la hauteur de la cellule.
- Enfin, cliquez sur Traiter > mathématique > Multiplier pour multiplier l’image résultante à l’aide de la constante suivante:
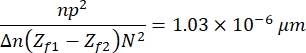
qui est défini en fonction des caractéristiques des images, de l’échantillon et de la configuration expérimentale13. Ici, n = 1,51 est l’indice de réfraction de l’huile, p = 0,0721 μm est la relation entre les micromètres et les pixels des images, Δ n = 0,058 est la différence entre les indices de réfraction du milieu RBC et aqueux, la distance entre les images de focalisation et de défocalisation (Z f1 - Z f2) = 2μm, et la taille des images, N 2 = 256 pxl2. - Utilisez l’image résultante pour obtenir le profil de hauteur (Figure 7). L’image résultante est utilisée dans ImageJ pour observer différents profils de hauteur. Cela dépend de l’endroit dans la cellule où la ligne verticale jaune est placée, par exemple, dans la figure 7 un profil de hauteur limité par la ligne verticale est obtenu, observez-le en appuyant sur Ctrl + K.
- Facteur de forme
- Après avoir trouvé l’image qui contient le profil de hauteur RBC, utilisez le contraste flou de 2 μm pour créer un ensemble de deux images dans ImageJ. Cliquez sur Image > Stacks, puis sélectionnez l’option Images to Stack. Pour trouver le facteur de forme, utilisez une macro personnalisée ImageJ pour analyser la pile. La macro personnalisée ImageJ peut être téléchargée en tant que fichier supplémentaire 2.
Remarque : Le programme utilise l’image de défocalisation pour déterminer les bords de cellule. Il détermine ensuite le périmètre, à l’aide de l’image qui contient le profil de hauteur, pour chaque position horizontale. En plus des arêtes, il détermine le périmètre, ainsi que l’inverse du périmètre. La somme des valeurs de périmètre inverses, multipliée par l’épaisseur de pixels, correspond à l’inverse du facteur de forme. - Insérez la relation pixel/micromètre dans le programme. Obtenez cette valeur à partir de l’expérience d’étalonnage objectif au microscope. Dans l’exemple utilisé, la valeur est 13,87 pixels/μm.
- Choisissez la position de départ horizontale pour placer la ligne jaune dans la première image de la pile. Commencez la ligne avant le début de la cellule et dessinez-la au-delà des limites verticales de la cellule. Dans l’exemple, la ligne jaune a une longueur de 153 pixels et la position initiale est comprise entre i = 70, y1 = 80 et i = 70, et y2 = 195. Ensuite, déplacez la ligne jaune horizontalement jusqu’à ce que la position finale soit f = 245, y1 = 80 et f = 245, et y2 = 195.
- Enfin, pour rechercher les bords de la cellule, le périmètre et l’inverse du périmètre, sélectionnez l’onglet Macro et cliquez sur Exécuter la macro. La macro fournira un tableau avec la position des arêtes, le périmètre et l’inverse du périmètre, ainsi qu’une image de la cellule analysée. Vérifiez si les bords de cette image sont similaires à ceux de la figure 7, sinon répétez la procédure.
- Utilisez la somme de l’inverse du périmètre pour trouver le facteur de forme8.
- Après avoir trouvé l’image qui contient le profil de hauteur RBC, utilisez le contraste flou de 2 μm pour créer un ensemble de deux images dans ImageJ. Cliquez sur Image > Stacks, puis sélectionnez l’option Images to Stack. Pour trouver le facteur de forme, utilisez une macro personnalisée ImageJ pour analyser la pile. La macro personnalisée ImageJ peut être téléchargée en tant que fichier supplémentaire 2.
7. Modèle de rhéologie vitreuse souple et analyse expérimentale
- Organisation des données expérimentales dans un tableau
- Créez une nouvelle table dans le logiciel d’analyse en cliquant sur l’onglet Fichier . Déterminez 10 colonnes différentes (de c0 à c9) pour les paramètres suivants : fréquences angulaires utilisées (rad/s) : c0; Valeurs K' (pN/μm) obtenues pour chaque fréquence angulaire: c1; ErrK': c2; K » (pN/μm) valeurs obtenues pour chaque fréquence angulaire: c3; ErrK »: c4; Valeurs G'(Pa) obtenues pour chaque fréquence angulaire: c5; ErrG': c6; G » (Pa) valeurs obtenues pour chaque fréquence angulaire: c7; ErrG »: c8 et Ff avec son erreur respective : c9 (Figure 8). Utilisez la formule suivante pour remplir les colonnes suivantes :
c5 = (3 x c1) / (8 x 1,28 x 0,087)
C6 = (3 / (8 x 0,087 x 1,28)) x sqrt (C2^2 + (C1 x 0,01 / 1,28)^2 + (C1 x 0,008 / 0,087) ^ 2)
c7 = (3 x c3) / (8 x 1,28 x 0,087)
C8 = (3 / (8 x 0,087 x C4)) x Sqrt (C4^2 + (C3 x 0,01 / 1,28)^2 + (C3 x 0,008 / 0,087)^2)
- Créez une nouvelle table dans le logiciel d’analyse en cliquant sur l’onglet Fichier . Déterminez 10 colonnes différentes (de c0 à c9) pour les paramètres suivants : fréquences angulaires utilisées (rad/s) : c0; Valeurs K' (pN/μm) obtenues pour chaque fréquence angulaire: c1; ErrK': c2; K » (pN/μm) valeurs obtenues pour chaque fréquence angulaire: c3; ErrK »: c4; Valeurs G'(Pa) obtenues pour chaque fréquence angulaire: c5; ErrG': c6; G » (Pa) valeurs obtenues pour chaque fréquence angulaire: c7; ErrG »: c8 et Ff avec son erreur respective : c9 (Figure 8). Utilisez la formule suivante pour remplir les colonnes suivantes :
- Tracer la courbe G' (ω) par rapport à G » (ω)
- Pour générer la courbe G' (ω) versus G » (ω) dans le logiciel d’analyse, utilisez les données du tableau précédent. Cliquez sur Galerie > Linéaire et choisissez le format Nuage de points.
- Une nouvelle fenêtre s’ouvrira. Sélectionnez la colonne G » comme axe des x et la colonne G' comme axe des y. Enfin, cliquez sur le bouton Tracer pour obtenir le graphique.
- Pour ajouter les barres d’erreur dans le tracé, cliquez sur la fenêtre Tracé , puis cliquez sur Tracer > Barres d’erreur. Une nouvelle fenêtre s’ouvrira. Tout d’abord, marquez l’option Erreur Y . Une autre fenêtre, appelée Paramètres de la barre d’erreur , s’ouvrira.
- Cliquez sur % de la valeur, sélectionnez Colonne de données, puis cliquez sur la colonne ErrG'. Enfin, cliquez sur OK > Plot, les barres d’erreur pour les valeurs y apparaîtront. Répétez la même procédure pour les valeurs de l’axe des x en sélectionnant maintenant le carré X Err et la colonne correcte pour l’ErrG ». Le graphique final sera similaire à celui de la figure 9.
- Ajustement des paramètres au modèle de rhéologie vitreuse douce
NOTE: L’analyse des données est divisée en deux parties: 1) courbe ajustant le diagramme G' (ω) versus G » (ω) pour obtenir les paramètres Γ et Gm; 2) ajustement de courbe G' (ω) et G » (ω) en fonction de la fréquence ω, pour obtenir l’exposant de la loi de puissance α.- Courbe ajustant le diagramme G' (ω) versus G » (ω) pour obtenir les paramètres Γ et Gm.
- Cliquez sur l’onglet Ajustement de la courbe , sélectionnez Fit1. Une nouvelle fenêtre s’ouvrira. Sélectionnez le carré et cliquez sur le bouton Définir . Une fenêtre appelée définition générale de l’ajustement de la courbe apparaîtra. Tapez l’équation suivante :
m1 + m0/m2
où m1 = 61,576; m2 = 1, avec une erreur admissible de 1 x 10-5. Ici, m0 représente G », m1 représente Gm et m2 représente Γ.
NOTE: Pour m1 et m2, il est nécessaire d’estimer leurs valeurs respectives lors de la définition de l’ajustement de la courbe. Les estimations ci-dessus étaient basées sur l’exemple d’expérience montré. Dans les expériences, estimer les nombres en fonction des valeurs observées dans la parcelle. - Cliquez sur le bouton OK dans les deux fenêtres et le raccord apparaîtra, comme illustré à la figure 10 - une courbe noire. Vérifiez les valeurs correctes pour m1 et m2, répertoriées dans le tableau d’ajustement de la courbe qui apparaissait avec le tracé.
- Cliquez sur l’onglet Ajustement de la courbe , sélectionnez Fit1. Une nouvelle fenêtre s’ouvrira. Sélectionnez le carré et cliquez sur le bouton Définir . Une fenêtre appelée définition générale de l’ajustement de la courbe apparaîtra. Tapez l’équation suivante :
- Raccord de courbe G ' et G » en fonction de la fréquence angulaire ω
- Ensuite, créez deux autres tracés, à savoir, G' en fonction de ω et G' en fonction de ω. Placez les barres d’erreur uniquement sur l’axe y, comme illustré précédemment.
- Répétez la procédure d’ajustement de courbe, mais maintenant dans l’option Ajustement de courbe, sélectionnez G », puis dans Ajustement général > Définition de courbe, écrivez l’équation suivante :
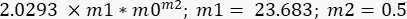
Dans ce cas, m1 a été estimé comme étant la valeur de G » (ω) = 23,683 Pa, lorsque ω = 6,3 rad/s; m2 a été estimé à 0,5 (rappelez-vous que m2 est l’exposant α et varie entre 0 et 1). Insérer la valeur pour Γ = 2,0293, selon le résultat de m2 dans la figure 10. - Cochez l’option Données de poids. Après toutes ces procédures, cliquez sur OK, et un ajustement de courbe similaire à celui de la figure 11 - une courbe bleue apparaîtra. Les valeurs pour α, α = 0,63 ± 0,02 et G 0, G 0 = (3,7 ± 0,3)Pa apparaîtront. Utilisez ces valeurs pour ajuster la courbe suivante, G' (ω).
- Cliquez sur la courbe suivante, répétez la procédure d’ajustement de courbe, mais maintenant dans la définition générale de la courbe d’ajustement, écrivez l’équation suivante :
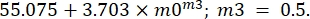
Dans ce cas, m3 n’est qu’une valeur estimée de α pour confirmer la valeur obtenue précédemment. Utilisez les valeurs pour G 0 = 3,703 et G' (ω) = 23,683 Pa lorsque ω = 6,3 rad/s. - Encore une fois, ajoutez 1 x 10-5 comme erreur admissible et marquez l’option Données de poids. Après toutes ces procédures, cliquez sur OK, et un ajustement de courbe similaire à celui de la figure 11 - une courbe verte apparaîtra.
- Courbe ajustant le diagramme G' (ω) versus G » (ω) pour obtenir les paramètres Γ et Gm.
Access restricted. Please log in or start a trial to view this content.
Résultats
La figure 1 représente les schémas du système OT utilisé pour les mesures rhéologiques. La figure 2 montre les schémas de l’expérience de microrhéologie avec les deux sphères et une CRE représentative est également montrée. La figure 3 montre une courbe typique des amplitudes des deux sphères en fonction du temps où les mouvements sinusoïdaux sont produits par l’étage piézoélectrique. Alors que la sphère de r?...
Access restricted. Please log in or start a trial to view this content.
Discussion
Dans ce protocole, une méthode intégrée basée sur des pinces optiques et une microscopie à défocalisation est présentée pour cartographier quantitativement les propriétés viscoélastiques des globules rouges. Les résultats pour les modules de cisaillement de stockage et de perte, ainsi que l’exposant de mise à l’échelle qui caractérise la rhéologie vitreuse molle des globules rouges sont déterminés. L’application de ce protocole pour différentes conditions expérimentales, telles que dans la situa...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun intérêt financier dans les produits décrits dans ce manuscrit et n’ont rien d’autre à divulguer.
Remerciements
Les auteurs tiennent à remercier tous les membres de l’installation de microscopie avancée CENABIO pour leur aide précieuse. Ce travail a été soutenu par les agences brésiliennes Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES) - Code financier 001, Fundação de Amparo à Pesquisa do Estado do Rio de Janeiro (FAPERJ) et Instituto Nacional de Ciência e Tecnologia de Fluidos Complexos (INCT-FCx) ainsi que Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP). B.P. a été soutenu par une subvention JCNE de FAPERJ.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 35mm culture dishes | Corning | 430165 | |
| Bovine serum albumin | Sigma-Aldrich | A9418 | |
| Coverslips | Knittel Glass | VD12460Y1A.01 and VD12432Y1A.01 | |
| Glass-bottom dishes | MatTek Life Sciences | P35G-0-10-C | |
| Glucose | Sigma-Aldrich | G7021 | |
| ImageJ | NIH | https://imagej.nih.gov/ij/ | |
| Immersion oil | Nikon | MXA22165 | |
| Inverted microscope | Nikon | Eclipse TE300 | |
| KaleidaGraph | Synergy Software | https://www.synergy.com/ | |
| KCl | Sigma-Aldrich | P5405 | |
| KH2PO4 | Sigma-Aldrich | P5655 | |
| Microscope camera | Hamamatsu | C11440-10C | |
| Na2HPO4 | Sigma-Aldrich | S5136 | |
| NaCl | Sigma-Aldrich | S5886 | |
| Neubauer chamber | Sigma-Aldrich | BR717805-1EA | |
| Objective lens | Nikon | PLAN APO 100X 1.4 NA DIC H; PLAN APO 60x 1.4 NA DIC H and Plan APO 10x XXNA PH2 | |
| Optical table | Thorlabs | T1020CK | |
| OT laser | IPG Photonics | YLR-5-1064-LP | |
| Polystyrene microspheres | Polysciences | 17134-15 | |
| rubber ring | Forever Seals | NBR O-Ring | |
| Silicone grease | Dow Corning | Z273554 | |
| Stage positioning | PI | P-545.3R8S | |
| Pipette | Gilson | P1000 |
Références
- Fowler, V. M. The human erythrocyte plasma membrane: a Rosetta Stone for decoding membrane-cytoskeleton structure. Current Topics in Membranes. 72, 39-88 (2013).
- Tomaiuolo, G. Biomechanical properties of red blood cells in health and disease towards microfluidics. Biomicrofluidics. 8 (5), 051501(2014).
- Depond, M., Henry, B., Buffet, P., Ndour, P. A. Methods to investigate the deformability of RBC during malaria. Frontiers in Physiology. 10, 1613(2019).
- Boal, D. Mechanics of the Cell. 2 edn. , Cambridge University Press. (2012).
- Balland, M., et al. Power laws in microrheology experiments on living cells: Comparative analysis and modeling. Physical Review E. 74 (2), 021911(2006).
- Amin, M. S., et al. Microrheology of red blood cell membranes using dynamic scattering microscopy. Optics Express. 15 (25), 17001-17009 (2007).
- Puig-de-Morales-Marinkovic, M., Turner, K. T., Butler, J. P., Fredberg, J. J., Suresh, S. Viscoelasticity of the human red blood cell. American Journal of Physiology Cell Physiology. 293 (2), 597-605 (2007).
- Gomez, F., et al. Effect of cell geometry in the evaluation of erythrocyte viscoelastic properties. Physical Review E. 101 (6-1), 062403(2020).
- Gomez, F., et al. Plasmodium falciparum maturation across the intra-erythrocytic cycle shifts the soft glassy viscoelastic properties of red blood cells from a liquid-like towards a solid-like behavior. Experimental Cell Research. 397 (2), 112370(2020).
- Pompeu, P., et al. Protocol to measure the membrane tension and bending modulus of cells using optical tweezers and scanning electron microscopy. STAR Protocols. 2 (1), 100283(2021).
- Agero, U., Mesquita, L. G., Neves, B. R., Gazzinelli, R. T., Mesquita, O. N. Defocusing microscopy. Microscopy Research and Technique. 65 (3), 159-165 (2004).
- Agero, U., Monken, C. H., Ropert, C., Gazzinelli, R. T., Mesquita, O. N. Cell surface fluctuations studied with defocusing microscopy. Physical Review E. 67 (5), 051904(2003).
- Roma, P. M. S., Siman, L., Amaral, F. T., Agero, U., Mesquita, O. N. Total three-dimensional imaging of phase objects using defocusing microscopy: Application to red blood cells. Applied Physics Letters. 104 (25), 251107(2014).
- Köhler, A. New method of illumination for photomicrographical purposes. Journal of the Royal Microscopical Society. 14, 261-262 (1894).
- Nans, A., Mohandas, N., Stokes, D. L. Native ultrastructure of the red cell cytoskeleton by cryo-electron tomography. Biophysical Journal. 101 (10), 2341-2350 (2011).
- Ayala, Y. A., et al. Rheological properties of cells measured by optical tweezers. BMC Biophysics. 9, 5(2016).
- Ayala, Y. A., et al. Effects of cytoskeletal drugs on actin cortex elasticity. Experimental Cell Research. 351 (2), 173-181 (2017).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon