É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise quantitativa das propriedades viscoelásticas das hemácias utilizando pinças ópticas e microscopia desfocagem
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, um protocolo integrado baseado em pinças ópticas e microscopia de desfocagem é descrito para medir as propriedades reológicas das células. Este protocolo tem ampla aplicabilidade no estudo das propriedades viscoelásticas de eritrócitos sob condições fisiopatológicas variáveis.
Resumo
As propriedades viscoelásticas dos eritrócitos têm sido investigadas por uma variedade de técnicas. No entanto, os dados experimentais relatados variam. Isso não é atribuído apenas à variabilidade normal das células, mas também às diferenças nos métodos e modelos de resposta celular. Aqui, um protocolo integrado utilizando pinças ópticas e microscopia desfocagem é empregado para obter as características reológicas das hemácias na faixa de frequência de 1 Hz a 35 Hz. Enquanto pinças ópticas são utilizadas para medir a constante elástica complexo eritrocito, a microscopia de desfocalização é capaz de obter o perfil de altura celular, volume e seu fator de forma um parâmetro que permite a conversão de constante elástica complexa em módulo de cisalhamento complexo. Além disso, aplicando um modelo de reologia vítrea macia, o expoente de escala para ambos os módulos pode ser obtido. A metodologia desenvolvida permite explorar o comportamento mecânico das hemácias, caracterizando seus parâmetros viscoelásticos, obtidos em condições experimentais bem definidas, para diversas condições fisiológicas e patológicas.
Introdução
As hemácias maduras, também conhecidas como eritrócitos, são capazes de se estender por mais que o dobro de seu tamanho ao passar pelos capilares mais estreitos do corpo humano1. Tal capacidade é atribuída à sua capacidade única de deformar quando submetidos a cargas externas.
Nos últimos anos, diferentes estudos têm caracterizado essa característica na superfície das hemácias2,3. A área da física que descreve as respostas elásticas e viscosas dos materiais devido a cargas externas é chamada de reologia. Em geral, quando uma força externa é aplicada, a deformação resultante depende das propriedades do material e pode ser dividida em deformações elásticas, que armazenam energia, ou deformações viscosas, que dissipam energia4. Todas as células, incluindo as hemácias, exibem um comportamento viscoelástico; Em outras palavras, a energia é armazenada e dissipada. A resposta viscoelástica de uma célula pode, assim, ser caracterizada pelo seu módulo de cisalhamento complexo G*(ω) = G'(ω) + iG"(ω), onde G' (ω) é o módulo de armazenamento, relacionado ao comportamento elástico, e G" (ω) é o módulo de perda, relacionado à sua viscosidade4. Além disso, modelos fenomenológicos têm sido utilizados para descrever as respostas celulares, um dos mais utilizados é o chamado modelo de reologia vítreamole5, caracterizado por uma dependência da lei de potência do módulo de cisalhamento complexo com a frequência de carga.
Métodos unicelulares têm sido empregados para caracterizar as propriedades viscoelásticas das hemácias, aplicando força e medindo o deslocamento em função da carga imposta 2,3. Entretanto, para o módulo de cisalhamento complexo, poucos resultados podem ser encontrados na literatura. Usando espalhamento dinâmico de luz, foram relatados valores de armazenamento e perda de hemácias variando de 0,01-1 Pa, na faixa de frequência de 1-100 Hz6. Utilizando-se a citometria de torção magnética óptica, obteve-se módulo elástico complexo aparente7 e, para fins de comparação, um fator multiplicativo foi reivindicado para possivelmente esclarecer as discrepâncias.
Mais recentemente, uma nova metodologia baseada em pinças ópticas (OT) em conjunto com a microscopia de desfocalização (DM), como uma ferramenta integrada para mapear quantitativamente o armazenamento e a perda de módulos de cisalhamento de eritrócitos humanos ao longo de cargas tempo-dependentes, foi estabelecida 8,9. Além disso, um modelo de reologia vítrea mole foi utilizado para ajustar os resultados e obter um coeficiente de lei de potência que caracteriza as hemácias 8,9.
De modo geral, a metodologia desenvolvida8,9, cujo protocolo é descrito em detalhes a seguir, esclarece discrepâncias prévias ao utilizar os valores medidos para o fator de forma, Ff, que relaciona forças e deformações a tensões e deformações na superfície das hemácias e pode ser utilizado como um novo método diagnóstico capaz de determinar quantitativamente os parâmetros viscoelásticos e as características vítreas moles de hemácias obtidas de indivíduos com sangue diferente Patologias. Tal caracterização, utilizando o protocolo descrito a seguir, pode abrir novas possibilidades para a compreensão do comportamento das hemácias do ponto de vista mecanobiológico.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Amostras de sangue humano foram fornecidas por voluntários adultos, de ambos os sexos, de acordo com protocolos aprovados pelo Comitê de Ética em Pesquisa da Universidade Federal do Rio de Janeiro (Protocolo 2.889.952) e registrados na Plataforma Brasil sob o número CAAE 88140418.5.0000.5699. Um termo de consentimento por escrito foi emitido e coletado de todos os voluntários. Foram excluídos aqueles com qualquer hemoglobinopatia e/ou em uso de medicação controlada. Todo o processo seguiu as diretrizes aprovadas pelo comitê de ética do instituto.
1. Preparação dos porta-amostras
- Adquirir duas lamínulas (24 mm x 60 mm e 24 mm x 32 mm; espessura = 0,13-0,17 mm) e um anel de borracha (diâmetro = 10 mm; espessura = 2 mm) para cada porta-amostras.
- Despeje graxa de silicone sobre a superfície do anel de borracha de uma forma que cubra todo o perímetro.
- Coloque o anel de borracha na lamínula com o lado da graxa voltado para a lamínula. Aguarde 5 min para a fixação adequada, os suportes de amostra estão então prontos para receber a cultura celular.
NOTA: Além disso, pratos com fundo de vidro comerciais ou caseiros também podem ser usados, conforme descrito anteriormente10.
2. Cultura celular
NOTA: As etapas abaixo descrevem como obter hemácias saudáveis do sangue humano. É importante que as amostras sejam preparadas na hora antes de cada experimento.
- Diluir 20 μL de sangue em 250 μL de solução salina tampão fosfato (PBS) 1x contendo 137 mM de NaCl, 2,7 mM de KCl, 10 mM Na 2 HPO 4, 1,8 mM KH2PO4, 10 mM de glicose e suplementado com 1 mg/mL de albumina de soro bovino (BSA).
- Após centrifugação a 200 x g por 2 min à temperatura ambiente, aspirar o sobrenadante com pipeta e ressuspender o pellet de células em 1mL de solução de PBS/BSA 1x. Lave as células 2x no tampão.
- Calcular a densidade celular utilizando um hemocitómetro e semear 50 000 a 100 000 células no suporte da amostra preparado na etapa 1. Aguarde de 10 a 15 min para fixação inespecífica da célula na lamínula; O tempo de espera não afeta as células.
- Adicionar à amostra 0,2 μL de uma solução de esfera de poliestireno a 10% v/v (raio = 1,52 ± 0,02 μm) para novos experimentos de OT. Confirme a mistura adequada observando as amostras sob o microscópio.
- Após a semeadura das células, basta colocar a segunda tampa acima do anel de borracha (não é necessário adicionar graxa para fixação), fechar o setup e finalizar o preparo da amostra. As amostras estão prontas para análise e manipulação de microscopia.
3. Configuração do microscópio de pinça óptica
NOTA: OT são ferramentas que usam um feixe de laser altamente focado para aprisionar objetos microscópicos e medir forças na faixa de piconewton e deslocamentos na escala nanométrica. O laser OT utilizado (comprimento de onda de 1064 nm) deve estar devidamente alinhado, como descrito anteriormente10.
- Resumidamente, usando pelo menos dois espelhos separados por uma distância de alguns centímetros (10-20 cm pelo menos), direcione um feixe de laser linearmente polarizado para a entrada traseira de um microscópio invertido. Alinhe precisamente o feixe de laser para entrar no microscópio em linha reta (Figura 1).
- Em seguida, reflita o feixe de laser usando um espelho dicroico, instalado no microscópio, para prosseguir paralelamente ao eixo da lente objetiva e entrar na lente perto do centro de sua entrada posterior. Isso focalizará o laser para criar a armadilha óptica (Figura 1).
- Em seguida, para medir forças com OT, calibrar o sistema para obter a rigidez do trap (κOT). Consulte10 para obter uma descrição mais detalhada do procedimento de calibração OT. Uma vez que κ OT é encontrado, o sistema OT está pronto para os experimentos de reologia.
4. Configuração do DM
NOTA: O DM é uma técnica de microscopia óptica baseada em campo claro que permite que objetos transparentes se tornem visíveis se o microscópio estiver levemente desfocado11,12. Esta técnica tem sido aplicada para a obtenção da forma eritrocitária13. O mesmo microscópio empregado para o sistema OT pode ser usado para MS, para obter um perfil de altura através de reconstruções 3D.
- Ajustar o sistema de iluminação do microscópio realizando a iluminação Köhler14 e, para melhor resolução, abrir totalmente o condensador-diafragma para realizar os experimentos.
- Utilizar um sistema de posicionamento piezoelétrico para deslocar a amostra em todas as coordenadas, com precisão nanométrica no eixo z. Realizar a autocalibração do sistema piezoelétrico em todos os eixos. Uma vez que todos os procedimentos são realizados, o sistema de microscópio está pronto para experimentos de DM.
5. Experimento e análise de reologia baseada em OT
OBS: O experimento de reologia consiste em observar as respostas da célula a pequenas oscilações de frequências variadas.
- Experimentação
- Usando o sistema OT, prenda a esfera com o laser OT e, em seguida, conecte-a a um eritrócito pressionando a esfera contra a superfície da célula perto da superfície superior e perto da borda da célula. Use o microscópio para esta etapa. Em seguida, prenda outra esfera e repita o mesmo procedimento de fixação, mas agora prenda-a à tampa, perto da célula. A esfera presa à lamínula é o cordão de referência (Figura 2), que é necessário para acompanhar o deslocamento do piezo e comparar com a esfera do CH.
- Certifique-se de que a célula escolhida esteja bem presa à lamínula e que a hemácia e as esferas de referência tenham aderido à superfície e à lamínula do eritrócito, respectivamente, antes de iniciar a medição. Identificar visualmente as células não aderidas à medida que elas se moverão ao longo do tempo, apesar da alta taxa de adesão (em torno de 80%-90%).
- Adicione uma função senoidal de amplitude, ξ 0 = 0,500 ± 0,001 μm e frequências variáveis de 1 Hz, 7 Hz, 14 Hz, 21 Hz, 28 Hz e 35 Hz, com respectivas frequências angulares, ω, de 6,3 rad/s, 169 rad/s, 88 rad/s, 132 rad/s, 176 rad/s e 220 rad/s, ao software do estágio piezoelétrico, utilizando o software piezelétrico, como demonstrado anteriormente 8,9.
- Usando o estágio piezelétrico, pressione o botão de partida para permitir o deslocamento piezelétrico e mantenha a esfera de hemácias na armadilha, submetendo a amostra a um ciclo de movimentos usando a função senoidal previamente definida. Use uma câmera capaz de produzir imagens a 790 quadros / s ou mais para registrar o movimento da amostra. Um esquema do experimento é mostrado na Figura 2.
- Enquanto a amostra é submetida a movimentos senoidais, ative o OT para aprisionar a esfera aderida à superfície da hemácia. Independentemente da temperatura escolhida para realizar os experimentos, temperatura ambiente, 37 °C, ou outra temperatura, monitorar cuidadosamente a temperatura para evitar variações durante as medições. O laser infravermelho (1064 nm) usado para criar o OT quase não causa danos ou aquecimento das células.
- Análise
- Analise as imagens obtidas durante os movimentos senoidais usando o ImageJ para encontrar a posição do centro de massa de cada uma das esferas ao longo do tempo.
OBS: Estes dados permitem gerar gráficos capazes de mostrar diferenças de fase e amplitude entre as duas esferas. Tais informações são cruciais para a obtenção da resposta viscoelástica das hemácias. - Para obter o centro de massa de cada uma das esferas, abra o software ImageJ. Importe todo o filme obtido durante os movimentos senoidais.
- Na guia Imagem , clique em Ajustar e selecione Limite. A janela de limite será aberta. Selecione P&B. Isso tornará o fundo branco e as esferas pretas.
- Ajuste o limite com ambas as barras de rolagem sob o histograma para que ambas as esferas apareçam com a quantidade máxima de pixels.
- Selecione a esfera de referência clicando em Arquivo > Retângulo. Desenhe um retângulo para selecionar a esfera. Depois de selecionar a esfera de referência em uma imagem, verifique se o retângulo também seleciona corretamente a mesma esfera em todas as outras imagens do filme.
- Em seguida, na aba Analisar , clique em Definir Medidas e selecione a opção Centro de Massa .
- Clique novamente na guia Analisar e selecione Analisar partículas. Uma nova janela será aberta. Defina o tamanho e a circularidade (dependendo do raio da esfera). Marque as caixas a seguir Exibir Resultados e Limpar Resultados. Finalmente, clique em OK para processar todas as imagens.
- Uma nova janela contendo uma tabela com coordenadas xy para o centro da massa aparecerá. Salve esses valores de coordenadas como um arquivo .txt. Repita o procedimento para a outra esfera, fixada à superfície do eritrócito.
- Para obter a amplitude e as diferenças de fase para ambas as esferas, abra o software de análise. Importe os arquivos .txt obtidos anteriormente.
- Crie uma nova tabela com três colunas. Na primeira coluna (c0) adicione o número de quadros, na segunda coluna (c1) adicione as coordenadas x para a esfera de referência, e na terceira coluna (c2), as coordenadas x para a esfera fixada à superfície RBC.
NOTA: Neste exemplo, como os movimentos senoidais foram realizados apenas no eixo x, é necessário usar apenas as coordenadas x para ambas as esferas. - Em seguida, correlacione os quadros com o tempo. Clique em Windows > Formula Entry. Uma nova janela chamada entrada de fórmula será aberta. Nesta janela, é possível configurar oito equações diferentes, cada uma delas em uma chave específica (de F1 a F8).
- Selecione F1, digite a seguinte fórmula:
c 3 = c0 / (fps da câmera)
Clique em Executar. Isso criará uma nova coluna por um tempo na tabela (coluna 4). - Subtraia o valor da coordenada x de cada quadro pelo seu respectivo valor médio. Para fazer isso, designe quaisquer duas chaves na entrada da fórmula e digite as seguintes equações para cada chave:
c 4 = (c 1 - média(c 1)) e c 5 = (c 2 - média(c2))
Clique no botão Executar . Os resultados aparecerão nas colunas 5 e 6 para as esferas de referência e hemácias, respectivamente. - Converta os valores do centro de massa de pixels em micrômetros. Para isso, use outra chave na entrada da fórmula e digite a seguinte equação:
c 4 = c 4 / número de conversão
Repita o mesmo processo para a coluna 5.
NOTA: A conversão é adquirida usando uma balança/régua micrométrica e obtendo sua imagem com o mesmo arranjo microscópico usado para as medições (incluindo a mesma lente objetiva). Este procedimento pode ser realizado durante a calibração do microscópio. Obtém-se, assim, uma relação pixel/micrômetro. - Gere um gráfico com os centros de massa de ambas as esferas no eixo y e o tempo no eixo x. Para isso, clique em Galeria > Linear e Dispersão. Uma nova janela será aberta. Selecione a coluna de tempo para o eixo x e no eixo y selecione as colunas do centro de massa em micrômetros para esferas de referência e hemácias.
- No gráfico, selecione apenas os dados relacionados à primeira frequência angular (6,3 rad/s). Use as ferramentas indicadas na Figura 3.
- Defina a equação que ajusta a curva de dados para a esfera de referência. Para isso, clique em Curve Fit > General e Fit1, selecione a caixa de dados relacionados à posição da esfera de referência e, em seguida, clique em Definir. Uma nova janela será aberta para definir a equação. A posição ξ(t) da esfera de referência é descrita por:
ξ(t) = ξ0cos(ωt)
onde ξ é o movimento senoidal da amostra, ω é a frequência angular e t é o tempo em s. Transpondo essa equação para o software de análise, ela ficará da seguinte forma: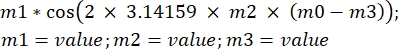 , onde m1 é ξ, m2 é f, m0 é o tempo t e m3 é a fase da função cosseno para t = 0.
, onde m1 é ξ, m2 é f, m0 é o tempo t e m3 é a fase da função cosseno para t = 0. - Estimar os valores de m1, m2 e m3 a partir da parcela. Após definir a equação, clique em OK. Os dados serão ajustados com base na equação e uma curva juntamente com um pequeno quadrado aparecerá no gráfico com os valores de m1, m2 e m3.
- Defina a equação que ajusta a curva de dados para a esfera RBC. Para isso, clique em Ajuste de Curva > Geral e defina a equação que ajustará a curva para os dados. A posição ρ(t) da esfera RBC é dada por:
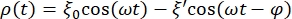
onde ξ' é o ângulo fora da amplitude de fase, e φ é o ângulo fora de fase. Transpondo esta fórmula para o software de análise, ela ficará da seguinte forma: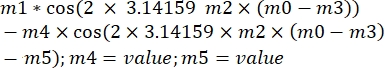 , onde m1, m2 e m3 são os valores obtidos no ajuste da curva da esfera de referência. m4 é ξ' e m5 é φ.
, onde m1, m2 e m3 são os valores obtidos no ajuste da curva da esfera de referência. m4 é ξ' e m5 é φ. - Estimar os valores de m4 e m5 a partir da parcela. Após definir a fórmula, clique em OK. Os dados serão ajustados com base na equação, e uma curva juntamente com um pequeno quadrado aparecerá no gráfico com os valores de m4 e m5.
- Em seguida, crie uma nova tabela para adicionar os dados obtidos do ajuste da curva em suas respectivas colunas. Defina cinco colunas diferentes para os seguintes parâmetros: frequência angular, amplitude (esfera de referência), tempo inicial, amplitude (esfera RBC) e ângulo fora de fase. Execute o mesmo procedimento para todas as outras frequências.
- Use as seguintes equações para localizar as constantes de armazenamento (K') e perda (K"):
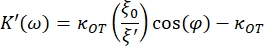
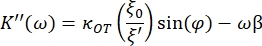
onde κ OT é a constante elástica OT e β é o coeficiente de arrasto de Stokes. Transpondo as equações para o software de análise, ficará da seguinte forma: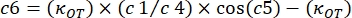 e
e
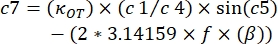 , onde β e κOT precisam ser substituídos pelos valores encontrados no sistema.
, onde β e κOT precisam ser substituídos pelos valores encontrados no sistema. - Plotar os resultados em um gráfico, usando o eixo x para K " e o eixo y para K' (Figura 4).
- Analise as imagens obtidas durante os movimentos senoidais usando o ImageJ para encontrar a posição do centro de massa de cada uma das esferas ao longo do tempo.
6. Experimento e análise do DM para obtenção do fator de forma celular global
- Aquisição de vídeo
- Mova o estágio piezoelétrico na direção xy usando o software para procurar uma célula isolada presa à lamínula. Aprisionar e fixar uma esfera de poliestireno de diâmetro conhecido à superfície do eritrócito. Usando o estágio piezoelétrico, mova levemente o talão aprisionado, também preso à superfície do eritrócito, a fim de deformar a célula e, em seguida, prenda o talão à lamínula.
NOTA: Também é possível usar a mesma célula a partir das medições OT. - Altere a posição do eixo z para localizar a imagem focalizada, onde o plano de foco está no meio da célula escolhida. Esta imagem apresenta o contraste menor com o nível de cinza no centro da célula igual ao nível de cinza fora da célula (fundo).
- Quando a posição for fixa, use o software da câmera para criar um filme de toda a célula com cerca de 5.000 imagens em 8 bits e 256 pixels x 256 pixels, a uma taxa de quadros de 25 fps. Em seguida, mova a posição do eixo z 2 μm para baixo ou para cima para obter uma imagem desfocada para a célula escolhida. Repita os parâmetros para criar um filme para essa situação.
- Finalmente, sem alterar a posição do eixo z, procure uma região sem células para repetir o mesmo procedimento e criar um filme do fundo da imagem.
- Mova o estágio piezoelétrico na direção xy usando o software para procurar uma célula isolada presa à lamínula. Aprisionar e fixar uma esfera de poliestireno de diâmetro conhecido à superfície do eritrócito. Usando o estágio piezoelétrico, mova levemente o talão aprisionado, também preso à superfície do eritrócito, a fim de deformar a célula e, em seguida, prenda o talão à lamínula.
- Aquisição de imagens de contraste
- Converta cada um dos três filmes em três imagens médias. Usando o ImageJ, selecione um dos filmes, clique em Image > Stacks > Z Project e escolha a opção Average Intensity (Intensidade média ). Repita este procedimento para os outros filmes para obter suas respectivas imagens.
- Altere todas as imagens obtidas de 8 bits para ponto flutuante de 32 bits. Usando o ImageJ, clique em Image > Type > 32-bits. Em seguida, clique em Analisar > Definir Medidas e escolha a opção Valor Cinza Médio . Por fim, clique novamente em Analisar > Medir.
- Em seguida, clique em Process > Image Calculator e divida a imagem focada pela imagem de fundo. Para esse resultado, multiplique o valor médio do nível de cinza da imagem focalizada. Obtenha o valor médio clicando em Processar > Matemática > Multiplicar.
- Para obter o valor médio, escolha a imagem focalizada e clique em Analisar > Medir. Para a imagem representativa, o valor médio do nível de cinza é 69.199. Repita os procedimentos acima para a imagem desfocada. Neste caso, o valor médio do nível de cinza é 69.231. A Figura 5 mostra as imagens focadas e desfocadas antes e após o procedimento.
- As imagens podem perder o contraste visual durante as operações. Para visualizar melhor as imagens, clique em Imagem > Ajustar > Brilho/Contraste e selecione a opção Automático .
- Em seguida, para localizar o contraste da imagem, use a seguinte relação:
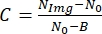 , onde N Img é o nível de cinza da célula, N0 é o nível de cinza fora da célula, e é um parâmetro constante correspondente ao nível de cinza para intensidade de luz zero que depende da câmera.
, onde N Img é o nível de cinza da célula, N0 é o nível de cinza fora da célula, e é um parâmetro constante correspondente ao nível de cinza para intensidade de luz zero que depende da câmera. - Para encontrar o valor B, use um medidor de potência para medir a intensidade da luz. Para cada valor de intensidade luminosa, um vídeo deve ser gravado; Assim, diferentes valores de intensidade podem estar relacionados a diferentes níveis de cinza. Finalmente, obter um ajuste linear e relacionar B com a intensidade luminosa zero15.
- Encontre o valor de N0, clique no ícone Polygon Selection e desenhe um polígono como o da Figura 6. Em seguida, clique na guia Analisar e selecione Medir para encontrar o nível médio de cinza para a área selecionada. Cada imagem é formada por um conjunto de pixels e cada pixel tem um certo nível de cinza. O conjunto de todos os níveis de cinza devido a todos os pixels que compõem a imagem corresponde a NImg.
- Use a equação de contraste para determinar N Img - N o e execute-a selecionando Processo > Matemática > Subtrair. Divida o resultado por N0 - B. Finalmente, encontre o contraste para a imagem focalizada (C 0) e desfocada (C1).
- Obtenção do perfil de altura
- Para a obtenção do perfil estatural, utiliza-se o método já descrito13. Em resumo, utilizar a transformada de Hartley (FHT) para obter a espessura das hemácias. No ImageJ, clique em Process > FFT > FFT Options e escolha FHT.
- Subtrair as imagens C0 e C1 no ImageJ usando Process > Math > Subtrair para obter a imagem para o procedimento a seguir, C= C0 - C1.
- Para a imagem C, clique em Process > FFT > FFT Options e, em seguida, escolha FHT para executar a transformação Hartley da imagem. Em seguida, divida pela frequência espacial q2 usando o plugin personalizado DivideQ2. Clique em Plugin > DivideQ2.
Observação : O arquivo DivideQ2.class deve ser copiado no diretório plugins onde o ImageJ está instalado. O plugin é fornecido como um arquivo .class (Arquivo Suplementar 1) a ser incluído na pasta do plugin do ImageJ. - Em seguida, execute a transformação inversa FHT usando Process > FFT > FFT para obter uma imagem com um nível de cinza proporcional à altura da célula.
- Finalmente, clique em Process > Math > Multiply para multiplicar a imagem resultante usando a seguinte constante:
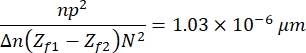
que é definida com base nas características das imagens, da amostra e do arranjo experimental13. Aqui, n = 1,51 é o índice de refração do óleo, p = 0,0721 μm é a relação entre micrômetros e pixels das imagens, Δn = 0,058 é a diferença entre o CH e os índices de refração do meio aquoso, a distância entre as imagens focalizadas e desfocadas (Z f1 - Z f2) = 2μm e o tamanho das imagens, N 2 = 256 pxl2. - Use a imagem resultante para obter o perfil de altura (Figura 7). A imagem resultante é usada no ImageJ para observar diferentes perfis de altura. Depende de onde na célula a linha vertical amarela é colocada, por exemplo, na Figura 7 obtém-se um perfil de altura limitado pela linha vertical, observe isso pressionando Ctrl + K.
- Fator de forma
- Depois de encontrar a imagem que contém o perfil de altura RBC, use o contraste desfocado de 2 μm para criar um conjunto de duas imagens no ImageJ. Clique em Image > Stacks e, em seguida, selecione a opção Images to Stack. Para localizar o fator forma, use uma macro personalizada do ImageJ para analisar a pilha. A macro personalizada do ImageJ está disponível para download como Arquivo Suplementar 2.
Observação : O programa usa a imagem de desfocagem para determinar as bordas da célula. Em seguida, determina o perímetro, usando a imagem que contém o perfil de altura, para cada posição horizontal. Além das bordas, determina o perímetro, bem como o inverso do perímetro. A soma dos valores inversos do perímetro, multiplicada pela espessura do pixel, corresponde ao inverso do fator forma. - Insira a relação pixel/micrômetro no programa. Obter este valor a partir do experimento de calibração objetiva do microscópio. No exemplo usado, o valor é 13,87 pixel/μm.
- Escolha a posição inicial horizontal para colocar a linha amarela na primeira imagem da pilha. Inicie a linha antes do início da célula e desenhe-a além dos limites verticais da célula. No exemplo, a linha amarela tem 153 pixels de comprimento e a posição inicial está entre i = 70, y1 = 80 e i = 70, e y2 = 195. Em seguida, mova a linha amarela horizontalmente até que a posição final seja f = 245, y1 = 80 e f = 245, e y2 = 195.
- Finalmente, para encontrar as bordas da célula, o perímetro e o inverso do perímetro, selecione a guia Macro e clique em Executar Macro. A macro entregará uma tabela com a posição das bordas, o perímetro e o inverso do perímetro, e uma imagem da célula analisada. Verifique se as bordas desta imagem são semelhantes às bordas da Figura 7, caso contrário, repita o procedimento.
- Use a soma do inverso do perímetro para encontrar o fator de forma8.
- Depois de encontrar a imagem que contém o perfil de altura RBC, use o contraste desfocado de 2 μm para criar um conjunto de duas imagens no ImageJ. Clique em Image > Stacks e, em seguida, selecione a opção Images to Stack. Para localizar o fator forma, use uma macro personalizada do ImageJ para analisar a pilha. A macro personalizada do ImageJ está disponível para download como Arquivo Suplementar 2.
7. Modelo de reologia vítrea macia e análise experimental
- Organizando os dados experimentais em uma tabela
- Crie uma nova tabela no software de análise clicando na guia Arquivo . Determinar 10 colunas diferentes (de c0 a c9) para os seguintes parâmetros: frequências angulares utilizadas (rad/s): c0; Valores de K' (pN/μm) obtidos para cada frequência angular: c1; ErrK': c2; Valores de K" (pN/μm) obtidos para cada frequência angular: c3; ErrK": c4; Valores de G'(Pa) obtidos para cada frequência angular: c5; ErrG': c6; G" (Pa) valores obtidos para cada frequência angular: c7; ErrG": c8 e Ff com seu respectivo erro: c9 (Figura 8). Use a seguinte fórmula para preencher as seguintes colunas:
c5 = (3 x c1) / (8 x 1,28 x 0,087)
c6 = (3 / (8 x 0,087 x 1,28)) x sqrt (c2^2 + (c1 x 0,01 / 1,28)^2 + (c1 x 0,008 / 0,087) ^ 2)
c7 = (3 x c3) / (8 x 1,28 x 0,087)
c8 = (3 / (8 x 0,087 x c4)) x sqrt (c4^2 + (c3 x 0,01 / 1,28)^2 + (c3 x 0,008 / 0,087)^2)
- Crie uma nova tabela no software de análise clicando na guia Arquivo . Determinar 10 colunas diferentes (de c0 a c9) para os seguintes parâmetros: frequências angulares utilizadas (rad/s): c0; Valores de K' (pN/μm) obtidos para cada frequência angular: c1; ErrK': c2; Valores de K" (pN/μm) obtidos para cada frequência angular: c3; ErrK": c4; Valores de G'(Pa) obtidos para cada frequência angular: c5; ErrG': c6; G" (Pa) valores obtidos para cada frequência angular: c7; ErrG": c8 e Ff com seu respectivo erro: c9 (Figura 8). Use a seguinte fórmula para preencher as seguintes colunas:
- Traçando a curva G' (ω) versus G" (ω)
- Para gerar a curva G' (ω) versus G" (ω) no software de análise, utilize os dados da tabela anterior. Clique em Galeria > Linear e escolha o formato Gráfico de Dispersão.
- Uma nova janela será aberta. Selecione a coluna G " como eixo x e a coluna G' como eixo y. Por fim, clique no botão Plotar para obter o gráfico.
- Para adicionar as barras de erro no gráfico, clique na janela Plotar e, em seguida, clique em Plotar > Barras de Erro. Uma nova janela será aberta. Primeiro, marque a opção Y Err. Outra janela, chamada Configurações da Barra de Erros, será aberta.
- Clique em % do Valor, selecione Coluna de Dados e, em seguida, clique na coluna ErrG'. Finalmente, clique em OK > Plotar, as barras de erro para os valores y aparecerão. Repita o mesmo procedimento para valores do eixo x selecionando agora o quadrado X Err e a coluna correta para o ErrG". O gráfico final será semelhante ao mostrado na Figura 9.
- Adequação dos parâmetros ao modelo de reologia vítrea macia
OBS: A análise dos dados é dividida em duas partes: 1) curva que ajusta o gráfico G' (ω) versus G" (ω) para obtenção dos parâmetros Γ e Gm; 2) ajuste da curva G' (ω) e G" (ω) em função da frequência ω, para obter o expoente da lei de potência α.- Curva ajustando-se o gráfico G' (ω) versus G" (ω) para obter os parâmetros Γ e Gm.
- Clique na guia Ajuste de curva , selecione Ajuste1. Uma nova janela será aberta. Selecione o quadrado e clique no botão Definir . Uma janela chamada definição geral de ajuste de curva será exibida. Digite a seguinte equação:
m1 + m0/m2
onde m1 = 61,576; m2 = 1, com erro admissível de 1 x 10-5. Aqui m0 representa G ", m1 representa Gm e m2 representa Γ.
OBS: Para m1 e m2 é necessário estimar seus respectivos valores durante a definição do ajuste da curva. As estimativas acima foram baseadas no experimento de exemplo mostrado. Nos experimentos, estimar os números de acordo com os valores observados na parcela. - Clique no botão OK em ambas as janelas e o encaixe aparecerá, como mostrado na Figura 10 - uma curva preta. Verifique os valores corretos para m1 e m2, listados na tabela de ajuste de curva que apareceu com o gráfico.
- Clique na guia Ajuste de curva , selecione Ajuste1. Uma nova janela será aberta. Selecione o quadrado e clique no botão Definir . Uma janela chamada definição geral de ajuste de curva será exibida. Digite a seguinte equação:
- Ajuste da curva G ' e G" em função da frequência angular ω
- Em seguida, crie dois outros gráficos, a saber, G' como uma função de ω e G' como uma função de ω. Coloque as barras de erro somente no eixo y, conforme demonstrado anteriormente.
- Repita o procedimento de ajuste de curva, mas agora na opção Seleções de ajuste de curva , selecione G" e, em Ajuste geral > definição de curva, escreva a seguinte equação:
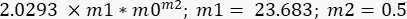
Nesse caso, estimou-se m1 como sendo o valor de G" (ω) = 23,683 Pa, quando ω = 6,3 rad/s; O m2 foi estimado em 0,5 (lembre-se que o m2 é o expoente α e varia entre 0 e 1). Insira o valor para Γ = 2,0293, de acordo com o resultado de m2 na Figura 10. - Marque a opção Dados de peso. Após todos esses procedimentos, clique em OK, e um ajuste de curva semelhante ao da Figura 11 - uma curva azul aparecerá. Os valores de α, α = 0,63 ± 0,02 e G 0, G 0 = (3,7 ± 0,3)Pa aparecerão. Use esses valores para ajustar a próxima curva, G' (ω).
- Clique na próxima curva, repita o procedimento de ajuste de curva, mas agora na Definição Geral da Curva de Ajuste, escreva a seguinte equação:
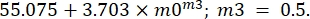
Neste caso, m3 é apenas um valor estimado de α para confirmar o valor obtido anteriormente. Use os valores para G 0 = 3,703 e G' (ω) = 23,683 Pa quando ω = 6,3 rad/s. - Novamente, adicione 1 x 10-5 como erro permitido e marque a opção Dados de peso. Após todos esses procedimentos, clique em OK, e um ajuste de curva semelhante ao da Figura 11 - uma curva verde aparecerá.
- Curva ajustando-se o gráfico G' (ω) versus G" (ω) para obter os parâmetros Γ e Gm.
Access restricted. Please log in or start a trial to view this content.
Resultados
A Figura 1 representa os esquemas do sistema OT utilizado para as medidas reológicas. A Figura 2 mostra os esquemas do experimento de microrreologia com ambas as esferas e uma hemácia representativa também é mostrada. A Figura 3 mostra uma curva típica para as amplitudes de ambas as esferas em função do tempo em que os movimentos senoidais são produzidos pelo estágio piezelétrico. Enquanto a esfera de referência (Figura 3...
Access restricted. Please log in or start a trial to view this content.
Discussão
Neste protocolo, um método integrado baseado em pinças ópticas e microscopia de desfocagem é apresentado para mapear quantitativamente as propriedades viscoelásticas das hemácias. Os resultados para os módulos de cisalhamento de armazenamento e perda, juntamente com o expoente de escala que caracteriza a reologia vítrea mole das hemácias são determinados. A aplicação deste protocolo para diferentes condições experimentais, como na situação fisiológica8 ou ao longo de cada estágio...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm interesses financeiros nos produtos descritos neste manuscrito e não têm mais nada a divulgar.
Agradecimentos
Os autores gostariam de agradecer a todos os membros da instalação de microscopia avançada do CENABIO pela ajuda importante. Este trabalho contou com o apoio das agências brasileiras Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES) - Código Financeiro 001, Fundação de Amparo à Pesquisa do Estado do Rio de Janeiro (FAPERJ) e Instituto Nacional de Ciência e Tecnologia de Fluidos Complexos (INCT-FCx) em conjunto com a Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP). B.P. foi apoiado por uma bolsa JCNE da FAPERJ.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 35mm culture dishes | Corning | 430165 | |
| Bovine serum albumin | Sigma-Aldrich | A9418 | |
| Coverslips | Knittel Glass | VD12460Y1A.01 and VD12432Y1A.01 | |
| Glass-bottom dishes | MatTek Life Sciences | P35G-0-10-C | |
| Glucose | Sigma-Aldrich | G7021 | |
| ImageJ | NIH | https://imagej.nih.gov/ij/ | |
| Immersion oil | Nikon | MXA22165 | |
| Inverted microscope | Nikon | Eclipse TE300 | |
| KaleidaGraph | Synergy Software | https://www.synergy.com/ | |
| KCl | Sigma-Aldrich | P5405 | |
| KH2PO4 | Sigma-Aldrich | P5655 | |
| Microscope camera | Hamamatsu | C11440-10C | |
| Na2HPO4 | Sigma-Aldrich | S5136 | |
| NaCl | Sigma-Aldrich | S5886 | |
| Neubauer chamber | Sigma-Aldrich | BR717805-1EA | |
| Objective lens | Nikon | PLAN APO 100X 1.4 NA DIC H; PLAN APO 60x 1.4 NA DIC H and Plan APO 10x XXNA PH2 | |
| Optical table | Thorlabs | T1020CK | |
| OT laser | IPG Photonics | YLR-5-1064-LP | |
| Polystyrene microspheres | Polysciences | 17134-15 | |
| rubber ring | Forever Seals | NBR O-Ring | |
| Silicone grease | Dow Corning | Z273554 | |
| Stage positioning | PI | P-545.3R8S | |
| Pipette | Gilson | P1000 |
Referências
- Fowler, V. M. The human erythrocyte plasma membrane: a Rosetta Stone for decoding membrane-cytoskeleton structure. Current Topics in Membranes. 72, 39-88 (2013).
- Tomaiuolo, G. Biomechanical properties of red blood cells in health and disease towards microfluidics. Biomicrofluidics. 8 (5), 051501(2014).
- Depond, M., Henry, B., Buffet, P., Ndour, P. A. Methods to investigate the deformability of RBC during malaria. Frontiers in Physiology. 10, 1613(2019).
- Boal, D. Mechanics of the Cell. 2 edn. , Cambridge University Press. (2012).
- Balland, M., et al. Power laws in microrheology experiments on living cells: Comparative analysis and modeling. Physical Review E. 74 (2), 021911(2006).
- Amin, M. S., et al. Microrheology of red blood cell membranes using dynamic scattering microscopy. Optics Express. 15 (25), 17001-17009 (2007).
- Puig-de-Morales-Marinkovic, M., Turner, K. T., Butler, J. P., Fredberg, J. J., Suresh, S. Viscoelasticity of the human red blood cell. American Journal of Physiology Cell Physiology. 293 (2), 597-605 (2007).
- Gomez, F., et al. Effect of cell geometry in the evaluation of erythrocyte viscoelastic properties. Physical Review E. 101 (6-1), 062403(2020).
- Gomez, F., et al. Plasmodium falciparum maturation across the intra-erythrocytic cycle shifts the soft glassy viscoelastic properties of red blood cells from a liquid-like towards a solid-like behavior. Experimental Cell Research. 397 (2), 112370(2020).
- Pompeu, P., et al. Protocol to measure the membrane tension and bending modulus of cells using optical tweezers and scanning electron microscopy. STAR Protocols. 2 (1), 100283(2021).
- Agero, U., Mesquita, L. G., Neves, B. R., Gazzinelli, R. T., Mesquita, O. N. Defocusing microscopy. Microscopy Research and Technique. 65 (3), 159-165 (2004).
- Agero, U., Monken, C. H., Ropert, C., Gazzinelli, R. T., Mesquita, O. N. Cell surface fluctuations studied with defocusing microscopy. Physical Review E. 67 (5), 051904(2003).
- Roma, P. M. S., Siman, L., Amaral, F. T., Agero, U., Mesquita, O. N. Total three-dimensional imaging of phase objects using defocusing microscopy: Application to red blood cells. Applied Physics Letters. 104 (25), 251107(2014).
- Köhler, A. New method of illumination for photomicrographical purposes. Journal of the Royal Microscopical Society. 14, 261-262 (1894).
- Nans, A., Mohandas, N., Stokes, D. L. Native ultrastructure of the red cell cytoskeleton by cryo-electron tomography. Biophysical Journal. 101 (10), 2341-2350 (2011).
- Ayala, Y. A., et al. Rheological properties of cells measured by optical tweezers. BMC Biophysics. 9, 5(2016).
- Ayala, Y. A., et al. Effects of cytoskeletal drugs on actin cortex elasticity. Experimental Cell Research. 351 (2), 173-181 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados