Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse spatiale à haute résolution des niveaux d’expression génique dans les tendons
Dans cet article
Résumé
Cet article décrit comment réaliser un protocole in situ optimisé pour les tendons. Cette méthode traite de la préparation des tissus, de la perméabilisation des sections, de la conception de la sonde et des méthodes d’amplification du signal.
Résumé
Au cours des dernières années, de nombreux protocoles ont été développés pour la transcriptomique à haute résolution dans de nombreux domaines médicaux et biologiques différents. Cependant, les tissus riches en matrice, et plus particulièrement les tendons, ont été laissés pour compte en raison de leur faible nombre de cellules, de leur faible quantité d’ARN par cellule et de leur teneur élevée en matrice, ce qui les rendait difficiles à analyser. L’un des outils unicellulaires les plus récents et les plus importants est l’analyse spatiale des niveaux d’expression génique dans les tendons. Ces outils spatiaux d’ARN ont une importance particulièrement élevée dans les tendons pour localiser des cellules spécifiques de populations nouvelles et inconnues, valider les résultats du RNA-seq sur cellule unique et ajouter un contexte histologique aux données de RNA-seq sur cellule unique. Ces nouvelles méthodes permettront d’analyser l’ARN dans les cellules avec une sensibilité exceptionnelle et de détecter des cibles d’ARN à molécule unique au niveau de la cellule unique, ce qui contribuera à caractériser moléculairement les tendons et à promouvoir la recherche sur les tendons.
Dans cet article méthodologique, nous nous concentrerons sur les méthodes disponibles pour analyser les niveaux d’expression génique spatiale sur les coupes histologiques en utilisant de nouveaux tests d’hybridation in situ pour détecter l’ARN cible dans des cellules intactes à des niveaux unicellulaires. Tout d’abord, nous nous concentrerons sur la façon de préparer le tissu tendineux pour les différents tests disponibles et sur la façon d’amplifier des signaux spécifiques à la cible sans bruit de fond mais avec une sensibilité et une spécificité élevées. Ensuite, l’article décrira des méthodes de perméabilisation spécifiques, les différents modèles de sondes et les stratégies d’amplification du signal actuellement disponibles. Ces méthodes uniques d’analyse des niveaux de transcription de différents gènes en résolution unicellulaire permettront l’identification et la caractérisation des cellules du tissu tendineux dans des populations jeunes et âgées de divers modèles animaux et tissus tendineux humains. Cette méthode aidera également à analyser les niveaux d’expression génique dans d’autres tissus riches en matrice tels que les os, le cartilage et les ligaments.
Introduction
Les tendons sont des tissus conjonctifs qui permettent la transmission de la force entre le muscle et l’os1. Au cours du développement, les ténocytes axiaux sont dérivés de cellules mésenchymateuses dans le sclérotome des somites2 ; les tendons des membres dérivent du mésoderme de la plaque latérale ; et les tendons crâniens proviennent de la lignée 3,4 de la crête neurale crânienne. Le tendon peut être caractérisé par l’expression du facteur de transcription5 de la sclérexie, bien que plusieurs marqueurs jouent également un rôle clé dans le développement du tendon, notamment la ténomoduline, la mohawk et la réponse de croissance précoce 1/2 6,7,8,9.
Malgré le peu de marqueurs connus du tendon, en général, une caractérisation plus approfondie reste difficile car le tendon contient des cellules qui s’étendent sur un gradient de propriétés biomécaniques. À partir de la jonction myotendineuse, du tendon au milieu du corps et de l’enthèse plus calcifiée, les cellules tendineuses résident dans des matrices extracellulaires dont les propriétés de traction varient. Étant donné que le tendon doit résister à la tension imposée par la différence de résistance mécanique entre les tissus mous et durs, l’organisation spatiale des cellules du tendon est particulièrement importante pour son fonctionnement. Cependant, on sait peu de choses sur ces sous-populations de tendons.
De nombreux outils de transcriptomique spatiale à haute résolution peuvent être utilisés pour commencer à élucider les sous-populations cellulaires, y compris, mais sans s’y limiter, le séquençage de l’ARN unicellulaire ou l’hybridation in situ . Cependant, bien que ces tests de profilage spatial aident à découvrir l’expression de l’ARN dans les tissus après la microdissection ou la section, ces méthodes peuvent être difficiles lorsqu’elles sont effectuées sur des tissus tendineux. Les tendons sont des tissus riches en matrice composés de près de 86 % de collagène en masse sèche10, ce qui rend difficile l’extraction des cellules pour le séquençage. En raison des complications liées à l’isolement des cellules de la matrice, de la nature hypocellulaire du tendon11 et du nombre relativement faible d’ARN, le tendon est un tissu difficile à analyser.
Dans cet article, nous présentons une méthode pour optimiser de nouveaux tests d’hybridation in situ afin de les exploiter pour les tendons en fournissant des méthodes de préparation des tissus, de perméabilisation et de conception de sondes. Associé aux technologies de séquençage existantes, cela pourrait aider les chercheurs à caractériser spatialement les sous-populations de tendons en développement, adultes ou blessés avec une sensibilité et une spécificité accrues du dosage.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les expériences sur les animaux ont été effectuées conformément aux directives de l’Institutional Animal Care and Use Committee (IACUC) et de l’AAALAC. Les expériences ont été réalisées selon un protocole approuvé #2013N000062 au Massachusetts General Hospital. Dans cette étude, des souris C57BL/J6 (âgées de 5 semaines et P0) ont été utilisées. Voir le tableau des matériaux pour plus de détails sur tous les matériaux, réactifs et instruments utilisés dans ce protocole.
1. Préparation et fixation de l’échantillon
- Euthanasier des souris dans une chambre de CO2 suivie d’une luxation cervicale comme méthode secondaire d’euthanasie. Ensuite, utilisez des ciseaux pour couper les membres postérieurs de la souris le long de l’articulation de la hanche12. Dans un flacon à scintillation, immerger le membre postérieur de la souris dans une solution de paraformaldéhyde (PFA) à 4 % suffisante pour couvrir complètement l’échantillon et fixer le tissu pendant 24 h.
REMARQUE : Au lieu de 4 % de PFA, 10 % de formol peut également être utilisé pour la fixation. Après fixation, l’échantillon peut être stocké dans une solution saline tamponnée au phosphate (PBS) jusqu’à un traitement ultérieur. En fixant l’ensemble de la patte postérieure, nous sommes en mesure de préserver la tension sur le tendon et avons la possibilité d’examiner l’enthèse, le milieu du corps tendineux ou la jonction myotendineuse. - Après avoir retiré le PFA du flacon à scintillation, laver l’échantillon pendant 3 x 10 min avec 1 x PBS en ajoutant suffisamment de PBS pour couvrir l’échantillon. Ensuite, immergez les membres postérieurs dans 0,5 M d’EDTA à 4 °C avec agitation pendant 1 à 2 semaines, en remplaçant par de l’EDTA frais tous les 2-3 jours.
- Après avoir retiré l’EDTA, lavez l’échantillon pendant 3 x 10 min avec 1x PBS, déshydratez les échantillons dans 70 % d’EtOH et incorporez-les dans de la paraffine dans l’orientation souhaitée. Utilisez les techniques conventionnelles de sectionnement FFPE pour sectionner le tissu d’une épaisseur de 3 μm et transférer les coupes sur une lame de microscope traitée et chargée (voir le tableau des matériaux)13.
REMARQUE : Les sections peuvent être stockées à 4 °C avant de passer aux étapes suivantes. - Déparaffinisation
- Faites cuire les diapositives sur une plaque chauffante à 60 °C pendant 1 h, puis laissez les diapositives revenir à température ambiante (RT). Pour retirer la cire de paraffine des lames, placez les lames sur un support à lames et abaissez-les dans un plat de coloration rempli de xylène. Répéter dans le xylène frais pendant 3 x 5 min.
ATTENTION : Le xylène est dangereux ; manipuler avec précaution. - Pour réhydrater les sections, immergez les lames dans les éléments suivants pendant 5 min par lavage, le tout à RT : laver 2x dans 100 % EtOH, 1x dans 75 % EtOH dilué dans dH2O, 1x dans 50 % EtOH dilué dans PBS, 1x dans 25 % EtOH dilué dans PBS, et 2x dans PBS.
REMARQUE : Toutes les étapes de réhydratation se font avec les lames sur un support de lames et avec les différentes solutions EtOH dans les plats de coloration.
- Faites cuire les diapositives sur une plaque chauffante à 60 °C pendant 1 h, puis laissez les diapositives revenir à température ambiante (RT). Pour retirer la cire de paraffine des lames, placez les lames sur un support à lames et abaissez-les dans un plat de coloration rempli de xylène. Répéter dans le xylène frais pendant 3 x 5 min.
2. Adaptation du protocole 14 du RNAscope (ISH commercialisé)
- Préparation des solutions et des sondes
- Préparez une solution TEG pour le prétraitement des échantillons sectionnés en combinant 25 mM de Tris-HCl à pH 8, 10 mM d’EDTA et 50 mM de glucose. Faire un volume suffisant pour tous les échantillons à traiter ou préparer l’excédent ; filtrer-stériliser et stocker à RT ; Mélangez correctement la solution avant de l’utiliser.
- Préparez suffisamment de 1x tampon de lavage (voir le tableau des matériaux) pour tous les lavages et rangez-le chez RT.
- Dans un bain-marie à 40 °C ou un incubateur, réchauffez les sondes d’intérêt pendant 10 min. Ensuite, préparez les sondes selon le protocole ISH14.
- Prétraitement des échantillons
- Plutôt que d’utiliser la solution de prétraitement recommandée, plongez les lames dans une boîte de coloration contenant un tampon TEG pendant 4 h à 60 °C.
REMARQUE : L’incubation du tampon TEG peut être prolongée jusqu’à 6 h si le tissu est particulièrement dense ou si la récupération de l’antigène du motif de liaison à l’ARN d’intérêt est difficile. Dans ce cas, un point d’arrêt peut être ajouté ici, les lames séchées et conservées dans un réfrigérateur à 4 °C pendant la nuit. - Retirez les échantillons du tampon et laissez-les sécher. Ensuite, prélevez les échantillons à l’aide d’un stylo barrière hydrophobe, plongez-les dans un autre plat de coloration contenant de la protéase IV et incubez-les dans un four pendant 45 min à 40 °C.
REMARQUE : Nous recommandons la protéase IV pour les souris adultes. Si vous utilisez des souris plus jeunes, comme la P0, nous vous recommandons d’utiliser une enzyme plus douce, comme la protéase III, pendant 30 min. Lors du dépannage pour différents âges, observez l’intégrité du tendon et vérifiez qu’il n’y a pas de dégradation. Si le tissu se dissocie ou se dégrade, diminuez le temps d’incubation ou changez la protéase utilisée. - Laver à nouveau les échantillons dans le tampon TEG pendant 30 min puis hybrider les sondes selon le protocole ISH14.
- Ajouter 50 à 100 μL de réactif de montage sur l’échantillon. Placez une lamelle sur les échantillons pour la microscopie. Lors de l’imagerie, utilisez un grossissement élevé (objectif 40x ou plus) pour visualiser le signal.
- Plutôt que d’utiliser la solution de prétraitement recommandée, plongez les lames dans une boîte de coloration contenant un tampon TEG pendant 4 h à 60 °C.
3. Adaptation du protocole 15 du HCR ISH
- Post-fixation
- Préparer une solution de PFA à 4 % (dans du PBS), une solution de PBT (0,1 % de Tween-20 dans du PBS) et de la protéinase K dans du PBS (concentration mère de 10 mg/mL, concentration finale de 5 μg/mL).
- Pour commencer la première étape de postfix, plongez les lames dans un plat de coloration contenant 4 % de PFA pendant 5 min. ensuite, retirez la solution de PFA à 4 % et stockez-la pour l’utiliser dans les étapes ultérieures. Rincez les lames pendant 2 x 5 minutes dans une solution PBT et retirez la solution PBT. Placez les lames dans un plat de coloration contenant une solution de protéinase K (5 μg/mL) pendant 5 min et rincez-les dans la solution PBT 2 x 5 min.
- Commencez la deuxième étape postfix et plongez les lames dans le PFA à 4 % pendant 5 min.
REMARQUE : Il s’agit du même PFA de 4 % économisé à l’étape 3.1.2. - Rincer les lames pendant 3 x 5 min dans la solution PBT.
REMARQUE : Toutes les étapes de post-fixation sont effectuées avec les lames sur un support à lames et dans des plats de coloration contenant les solutions respectives.
- Acétylation
- Préparez la solution d’acétylation contenant 625 μL d’anhydride acétique, 3,3 mL de tampon 1 M de triéthanolamine (TEA) et 246 mL de ddH2O. Assurez-vous de bien mélanger et d’utiliser immédiatement après la préparation.
- Placez les lames dans la solution d’acétylation pendant 10 minutes, puis rincez 3 x 5 min dans une solution PBT. Rincez les lames au jdH2O et laissez-les sécher à l’air libre pendant 30 min. Ensuite, dessinez autour des échantillons avec un stylo barrière hydrophobe.
REMARQUE : Toutes les étapes d’acétylation sont effectuées avec les lames sur un support de lames et dans des boîtes de coloration contenant les solutions respectives
- Hybridation
- Préchauffez le tampon d’hybridation à 37 °C et préchauffez une chambre humidifiée à 37 °C.
ATTENTION : Le tampon contient du formamide, une matière dangereuse. Si les lames ne sont pas suffisamment sèches, épongez les bords avec une lingette de laboratoire pour éliminer l’excès de dH2O. - Préparez les solutions de sonde en ajoutant 0,4 pmol de chaque mélange de sonde à 100 μL de tampon d’hybridation.
REMARQUE : Si vous utilisez l’imagerie dHCR, utilisez une concentration plus élevée de sonde pour améliorer l’efficacité de l’hybridation de la sonde. - Préparez 4 L de tampon de citrate de sodium (20x SSC) en combinant 3 M de NaCl (701,1 g de NaCl dans un volume final de 4 L) et 0,3 M de Na3CH6H5O7•2H2O (352,8 g), puis ajustez le pH final à 7 en ajoutant du HCl ou 10 N NaOH. Ajouter suffisamment de H2O pour atteindre 4 L. Préparer 5 fois SSCT en diluant 20 fois SSC à 5 fois et en ajoutant suffisamment de 10 % de Tween 20 pour obtenir 0,1 % du volume total.
- Ajoutez 200 μL de tampon d’hybridation à l’échantillon, placez les lames dans une chambre humidifiée et laissez les lames incuber pendant 10 min. Retirez la solution d’hybridation et égouttez l’excédent de tampon sur la lame en épongeant les bords avec une lingette de laboratoire.
- Ajouter 100 μL de la solution de sonde préparée sur l’échantillon et placer une lamelle sur l’échantillon. Incuber au four pendant la nuit ou pendant environ 12 à 16 h dans une chambre humidifiée avec une solution contenant 5x SSC et 50 % de formamide. Réglez la température à 37 °C.
REMARQUE : Comme l’incubation dure toute la nuit, l’ajout de lamelles vise à minimiser l’évaporation. - Laisser une aliquote de 5x solution SSCT dans un bain-marie à 37 °C et une deuxième aliquote à RT. Anticiper les volumes de ces aliquotes nécessaires en fonction du nombre d’échantillons et du nombre de lavages à l’étape suivante.
- Préchauffez le tampon d’hybridation à 37 °C et préchauffez une chambre humidifiée à 37 °C.
- Lavage
- À l’aide de la solution 5x SSCT préalablement préchauffée, préparez des solutions à 75 % de tampon de lavage/25 % de 5x SSCT, 50 % de tampon de lavage/50 % de 5x SSCT et 25 % de tampon de lavage/75 % de 5x SSCT.
- Pour éliminer l’excès de sondes, immergez les lames en série pendant 15 min par lavage à 37 °C dans 75 % de tampon de lavage/25 % 5x SSCT, 50 % de tampon de lavage/50 % 5x SSCT et 25 % de tampon de lavage/75 % 5x SSCT. Laissez les lamelles se détacher des échantillons.
- Incuber les lames 2 x 15 min avec 100 % 5x SSCT puis les immerger dans 5x SSCT pendant 5 min à RT.
REMARQUE : Les étapes de lavage peuvent être effectuées avec les lames sur un support à lames et dans des plats tachés contenant les solutions respectives ou avec les solutions ajoutées sous forme de gouttelettes.
- Amplification
- Préparez 6 pmol d’épingle à cheveux h1 et 6 pmol d’épingle à cheveux h2 en refroidissant par pression 2 μL d’un stock de 3 μM (chauffer à 95 °C pendant 90 s et refroidir à RT). Protégez les épingles à cheveux de la lumière pendant ce processus.
REMARQUE : Les épingles à cheveux HCR h1 et h2 sont fournies dans un tampon de stockage en épingle à cheveux et sont prêtes à être refroidies par enclenchement. Enclenchez les épingles à cheveux h1 et h2 dans des tubes séparés. - Séchez les lames en épongeant leurs bords avec une lingette de laboratoire. Ensuite, ajoutez 200 μL de tampon d’amplification sur l’échantillon et placez-le dans une chambre humidifiée pendant 30 min à RT. Préparez le mélange d’épingles à cheveux en ajoutant des épingles à cheveux h1 refroidies et des épingles à cheveux h2 refroidies à 100 μL de tampon d’amplification à RT.
- Retirez le tampon d’amplification et égouttez l’excès de liquide sur la lame en épongeant les bords avec une lingette de laboratoire. Ajouter 100 μL du mélange d’épingles à cheveux préalablement préparé et placer du parafilm sur l’échantillon. Incuber les lames pendant au moins 4 h ou toute la nuit dans une chambre sombre et humidifiée à RT.
- Pour éliminer l’excès d’épingles à cheveux, plongez les lames dans 5x SSCT dans une boîte de coloration et incubez à RT pendant 30 min. Répétez le lavage dans 5 fois SSCT frais pendant 30 min, et une troisième fois pendant 5 min.
- Séchez les lames en épongeant les bords avec une lingette de laboratoire et ajoutez 50 à 100 μL de réactif de montage sur l’échantillon. Placez une lamelle sur les échantillons pour la microscopie. Lors de l’imagerie, utilisez un grossissement élevé (objectif 40x ou plus) pour visualiser le signal.
- Préparez 6 pmol d’épingle à cheveux h1 et 6 pmol d’épingle à cheveux h2 en refroidissant par pression 2 μL d’un stock de 3 μM (chauffer à 95 °C pendant 90 s et refroidir à RT). Protégez les épingles à cheveux de la lumière pendant ce processus.
Access restricted. Please log in or start a trial to view this content.
Résultats
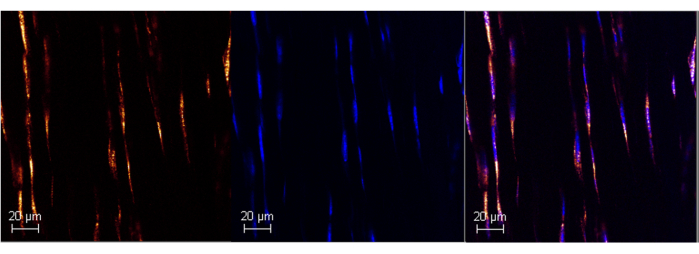
Figure 1 : Expression de l’ARN Poly A dans le tendon d’Achille de souris adulte à l’aide de RNAScope. Image représentative d’un marquage réussi de Poly A dans le tendon d’Achille de souris (panneau de gauche) à l’aide du test ISH commercialisé. La colocalisation avec DAPI confirme la spécificité ...
Access restricted. Please log in or start a trial to view this content.
Discussion
Dans cet article, nous décrivons les modifications apportées pour tirer parti des outils ISH existants afin qu’ils puissent être utilisés dans les tissus tendineux avec un haut degré de spécificité et de sensibilité. Étant donné que le tendon est un tissu très dense en matrice, des ajustements de protocole doivent souvent être effectués pour obtenir des degrés similaires de pénétration et de spécificité de la sonde. Ces méthodes spécifiques de perméabilisation et d...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs remercient Jenna Galloway et les membres du Galloway Lab pour leur soutien et leurs encouragements dans le développement et le dépannage de ces protocoles.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 1 M triethanolamine buffer | |||
| 10% Formalin solution | |||
| 10% Tween-20 | |||
| 20x Saline Sodium Citrate buffer | |||
| 4% PFA | |||
| ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 | ACD | 323100 | |
| Acetic Anhydride | |||
| Axio Imager Microscope | ZEISS | ||
| C57BL/J6 mice | JAX ID: 000664 | ||
| Coverslips | Fisher | 12-541-042 | |
| ddH2O | |||
| ETDA | Thermofisher | AM9262 | |
| EtOH | |||
| Glucose | VWR Chemicals BDH | BDH9230-500G | |
| HCR RNA-FISH Bundle | Molecular Instruments Inc. | ||
| HybEZ II Hybridization System | ACD | ||
| Immedge Barrier Pen | Vector Laboratories | H4000 | |
| Leica SPE Confocal Microscope | Leica | ||
| Parafilm | Fisher | ||
| Phosphate-buffered saline (PBS, 1x) | Invitrogen | AM9625 | Dilute 10x PBS in milli-Q water to get 1x solution |
| Protease IV | |||
| Proteinase K | Roche | 3115836001 | |
| RNAscope H2O2 and Protease Reagents | ACD | PN 322381 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V3 |
| RNAscope Multiplex Fluorescent Detection Kit | ACD | PN 323110 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 |
| RNAscope Target Retrieval reagents | ACD | 322000 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V4 |
| RNAscope Wash Buffer | ACD | PN 310091 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V5 |
| RNAscope Probe Diluent | ACD | 300041 | |
| Slide holder | StatLab | 4465A | |
| Staining Dish with Lid | StatLab | LWS20WH | |
| Superfrost Plus Microscope slides | Fisher | 1255015 | treated, charged slides |
| Tris-HCl | |||
| Xylene | Sigma-Aldrich | 534056-4L |
Références
- Sharma, P., Maffulli, N. Tendon injury and tendinopathy: healing and repair. Journal of Bone and Joint Surgery. 87 (1), 187-202 (2005).
- Brent, A. E., Schweitzer, R., Tabin, C. J. A somitic compartment of tendon progenitors. Cell. 113 (2), 235-248 (2003).
- Noden, D. M. The role of the neural crest in patterning of avian cranial skeletal, connective, and muscle tissues. Developmental Biology. 96 (1), 144-165 (1983).
- Chen, J. W., Galloway, J. L. The development of zebrafish tendon and ligament progenitors. Development. 141 (10), 2035-2045 (2014).
- Schweitzer, R., et al. Analysis of the tendon cell fate using Scleraxis, a specific marker for tendons and ligaments. Development. 128 (19), 3855-3866 (2001).
- Tsai, S., Nödl, M., Galloway, J. Bringing tendon biology to heel: Leveraging mechanisms of tendon development, healing, and regeneration to advance therapeutic strategies. Developmental Dynamics. 250 (3), 393-413 (2021).
- Kannus, P., et al. Location and distribution of non-collagenous matrix proteins in musculoskeletal tissues of rat. The Histochemical Journal. 30 (11), 799-810 (1998).
- Kannus, P. Structure of the tendon connective tissue. Scandinavian Journal of Medicine & Science in Sports. 10 (6), 312-320 (2000).
- Thorpe, C. T., Birch, H. L., Clegg, P. D., Screen, H. R. C. The role of the non-collagenous matrix in tendon function. International Journal of Experimental Pathology. 94 (4), 248-259 (2013).
- Lin, T. W., Cardenas, L., Soslowsky, L. J. Biomechanics of tendon injury and repair. Journal of Biomechanics. 37 (6), 865-877 (2004).
- Grinstein, M., et al. A distinct transition from cell growth to physiological homeostasis in the tendon. eLife. 8, e48689(2019).
- Villaseñor, S., Grinstein, M. Two-photon microscopy for the study of tendons. Journal of Visualized Experiments. , e65853(2023).
- Qin, C., et al. The cutting and floating method for paraffin-embedded tissue for sectioning. Journal of Visualized Experiments. (139), e58288(2018).
- Wang, F., et al. RNAscope®: A Novel In Situ RNA Analysis Platform for Formalin-Fixed Paraffin-Embedded Tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Choi, H. M. T., et al. Third-generation situ hybridization chain reaction: multiplexed, quantitative, sensitive, versatile, robust. Development. 145 (12), 165753(2018).
- Jackson, D. J., Herlitze, I., Hohagen, J. A whole mount in situ hybridization method for the gastropod mollusc Lymnaea stagnalis. Journal of Visualized Experiments. (109), e53968(2016).
- Young, A. P., Jackson, D. J., Wyeth, R. C. A technical review and guide to RNA fluorescence in situ hybridization. PeerJ. 8, e8806(2020).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon