È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi spaziale ad alta risoluzione dei livelli di espressione genica nei tendini
In questo articolo
Riepilogo
Questo articolo descrive come eseguire un protocollo in situ ottimizzato per i tendini. Questo metodo illustra la preparazione dei tessuti, la permeabilizzazione delle sezioni, la progettazione della sonda e i metodi di amplificazione del segnale.
Abstract
Negli ultimi anni, sono stati sviluppati molti protocolli per la trascrittomica ad alta risoluzione in diversi campi della medicina e della biologia. Tuttavia, i tessuti ricchi di matrice e, in particolare, i tendini sono stati lasciati indietro a causa del loro basso numero di cellule, della bassa quantità di RNA per cellula e dell'alto contenuto di matrice, che li ha resi complicati da analizzare. Uno degli strumenti più recenti e più importanti per le singole cellule è l'analisi spaziale dei livelli di espressione genica nei tendini. Questi strumenti spaziali dell'RNA hanno un'importanza particolarmente elevata nei tendini per localizzare cellule specifiche di popolazioni nuove e sconosciute, convalidare i risultati dell'RNA-seq a singola cellula e aggiungere un contesto istologico ai dati dell'RNA-seq a singola cellula. Questi nuovi metodi consentiranno l'analisi dell'RNA in cellule con eccezionale sensibilità e il rilevamento di bersagli di RNA a singola molecola a livello di singola cellula, il che aiuterà a caratterizzare molecolarmente i tendini e a promuovere la ricerca sui tendini.
In questo articolo di metodo, ci concentreremo sui metodi disponibili per analizzare i livelli di espressione genica spaziale su sezioni istologiche utilizzando nuovi saggi di ibridazione in situ per rilevare l'RNA bersaglio all'interno di cellule intatte a livello di singola cellula. In primo luogo, ci concentreremo su come preparare il tessuto tendineo per i diversi saggi disponibili e su come amplificare i segnali specifici del bersaglio senza rumore di fondo ma con alta sensibilità e alta specificità. Quindi, l'articolo descriverà specifici metodi di permeabilizzazione, i diversi design delle sonde e le strategie di amplificazione del segnale attualmente disponibili. Questi metodi unici di analisi dei livelli di trascrizione di diversi geni nella risoluzione di singole cellule consentiranno l'identificazione e la caratterizzazione delle cellule del tessuto tendineo in popolazioni giovani e anziane di vari modelli animali e tessuti tendinei umani. Questo metodo aiuterà anche ad analizzare i livelli di espressione genica in altri tessuti ricchi di matrice come ossa, cartilagine e legamenti.
Introduzione
I tendini sono tessuti connettivi che consentono la trasmissione della forza tra muscolo e osso1. Dal punto di vista dello sviluppo, i tenociti assiali derivano da cellule mesenchimali all'interno dello sclerotomo dei somiti2; i tendini degli arti derivano dal mesoderma della placca laterale; e i tendini cranici derivano dalla linea della cresta neurale cranica 3,4. Il tendine può essere caratterizzato dall'espressione del fattore di trascrizione5 della sclerassi, sebbene anche diversi marcatori svolgano un ruolo chiave nello sviluppo del tendine, tra cui la tenomodulina, la mohawk e la risposta di crescita precoce 1/2 6,7,8,9.
Nonostante i pochi marcatori noti del tendine, in generale, una caratterizzazione più approfondita rimane impegnativa perché il tendine contiene cellule che si estendono attraverso un gradiente di proprietà biomeccaniche. Dalla giunzione miotendinea, al tendine a metà del corpo e all'entesi più calcificata, le cellule tendinee risiedono in matrici extracellulari che variano nelle proprietà di trazione. Poiché il tendine deve resistere alle sollecitazioni di trazione imposte dalla differenza di resistenza meccanica tra tessuti molli e duri, l'organizzazione spaziale delle cellule del tendine è particolarmente importante per la sua funzione. Tuttavia, si sa poco su queste sottopopolazioni tendinee.
Molti strumenti di trascrittomica spaziale ad alta risoluzione possono essere utilizzati per iniziare a chiarire le sottopopolazioni cellulari, tra cui, a titolo esemplificativo ma non esaustivo, il Seq di RNA a singola cellula o l'ibridazione in situ . Tuttavia, mentre questi saggi di profilazione spaziale aiutano a scoprire l'espressione dell'RNA attraverso il tessuto dopo la microdissezione o il sezionamento, questi metodi possono essere impegnativi se eseguiti sul tessuto tendineo. I tendini sono tessuti ricchi di matrice composti da quasi l'86% di collagene per massa secca10, il che rende difficile l'estrazione delle cellule per il sequenziamento. A causa sia delle complicazioni nell'isolamento delle cellule dalla matrice, della natura ipocellulare del tendine11 e del numero relativamente basso di RNA, il tendine è un tessuto difficile da analizzare.
In questo articolo, presentiamo un metodo per ottimizzare nuovi saggi di ibridazione in situ per sfruttarli per i tendini, fornendo metodi di preparazione dei tessuti, permeabilizzazione e progettazione della sonda. Accoppiato con le tecnologie di sequenziamento esistenti, questo può aiutare i ricercatori a caratterizzare spazialmente le sottopopolazioni tendinee in tendini in via di sviluppo, adulti o feriti con una maggiore sensibilità e specificità del test.
Protocollo
Tutti gli esperimenti sugli animali sono stati eseguiti in conformità con le linee guida dell'Institutional Animal Care and Use Committee (IACUC) e dell'AAALAC. Gli esperimenti sono stati eseguiti secondo il protocollo approvato #2013N000062 presso il Massachusetts General Hospital. In questo studio sono stati utilizzati topi C57BL/J6 (5 settimane di età e P0). Consultare la Tabella dei materiali per i dettagli relativi a tutti i materiali, i reagenti e gli strumenti utilizzati in questo protocollo.
1. Preparazione e fissazione del campione
- Eutanasia dei topi in una camera di CO2 seguita da lussazione cervicale come metodo secondario di eutanasia. Quindi, usa le forbici per tagliare gli arti posteriori del topo lungo l'articolazione dell'anca12. In una fiala di scintillazione, immergere l'arto posteriore del topo in una soluzione di paraformaldeide (PFA) al 4% sufficiente a coprire completamente il campione e fissare il tessuto per 24 ore.
NOTA: Invece del 4% di PFA, per la fissazione può essere utilizzato anche il 10% di formalina. Dopo la fissazione, il campione può essere conservato in soluzione salina tamponata con fosfato (PBS) fino a un'ulteriore elaborazione. Fissando l'intero arto posteriore, siamo in grado di preservare la tensione sul tendine e avere l'opportunità di esaminare l'entesi, il tendine medio del corpo o la giunzione miotendinea. - Dopo aver rimosso il PFA dalla fiala di scintillazione, lavare il campione per 3 x 10 minuti con 1x PBS aggiungendo abbastanza PBS per coprire il campione. Quindi, immergere gli arti posteriori in EDTA 0,5 M a 4 °C agitando per 1-2 settimane, sostituendo con EDTA fresco ogni 2-3 giorni.
- Dopo aver rimosso l'EDTA, lavare il campione per 3 x 10 minuti con 1x PBS, disidratare i campioni in EtOH al 70% e incorporarli in paraffina con l'orientamento desiderato. Utilizzare le tecniche convenzionali di sezionamento FFPE per sezionare il tessuto con uno spessore di 3 μm e trasferire le sezioni su un vetrino da microscopio trattato e caricato (vedere la Tabella dei materiali)13.
NOTA: Le sezioni possono essere conservate a 4 °C prima di procedere con i passaggi successivi. - Deparaffinazione
- Cuocere i vetrini su una piastra calda a 60 °C per 1 ora e poi lasciare che i vetrini raggiungano la temperatura ambiente (RT). Per rimuovere la cera di paraffina dai vetrini, posizionare i vetrini su una griglia per vetrini e abbassarli in una vaschetta per la colorazione piena di xilene. Ripetere in xilene fresco per 3 x 5 min.
ATTENZIONE: Lo xilene è pericoloso; maneggiare con cura. - Per reidratare le sezioni, immergere i vetrini per 5 minuti per lavaggio, tutti a RT: lavare 2 volte in 100% EtOH, 1 volta in 75% EtOH diluito in dH2O, 1 volta in 50% EtOH diluito in PBS, 1 volta in 25% EtOH diluito in PBS e 2 volte in PBS.
NOTA: Tutte le fasi di reidratazione vengono eseguite con i vetrini su un rack per vetrini e con le varie soluzioni di EtOH in piastre di colorazione.
- Cuocere i vetrini su una piastra calda a 60 °C per 1 ora e poi lasciare che i vetrini raggiungano la temperatura ambiente (RT). Per rimuovere la cera di paraffina dai vetrini, posizionare i vetrini su una griglia per vetrini e abbassarli in una vaschetta per la colorazione piena di xilene. Ripetere in xilene fresco per 3 x 5 min.
2. Adattamento del protocollo RNAscope (ISH commercializzato)14
- Preparazione di soluzioni e sonde
- Preparare la soluzione TEG per il pretrattamento dei campioni sezionati combinando 25 mM di Tris-HCl a pH 8, 10 mM di EDTA e 50 mM di glucosio. Realizzare un volume sufficiente per tutti i campioni da trattare o preparare l'eccesso; filtrare-sterilizzare e conservare presso RT; Mescolare correttamente la soluzione prima di utilizzarla.
- Preparare una quantità sufficiente di 1x Wash Buffer (vedere la Tabella dei materiali) per tutti i lavaggi e conservarla presso RT.
- In un bagnomaria a 40 °C o in un'incubatrice, riscaldare le sonde di interesse per 10 minuti. Quindi, preparare le sonde secondo il protocollo ISH14.
- Pretrattamento dei campioni
- Invece di utilizzare la soluzione di pretrattamento consigliata, immergere i vetrini in una capsula di colorazione contenente tampone TEG per 4 ore a 60 °C.
NOTA: L'incubazione del tampone TEG può essere prolungata fino a 6 ore se il tessuto è particolarmente denso o se il recupero dell'antigene del motivo di legame dell'RNA di interesse è difficile. In tal caso, è possibile aggiungere un punto di arresto, asciugare i vetrini e conservarli in frigorifero a 4 °C per una notte. - Rimuovere i campioni dal tampone e lasciarli asciugare. Quindi, disegnare i campioni con una penna barriera idrofobica, immergerli in un'altra piastra di colorazione contenente proteasi IV e incubarli in forno per 45 minuti a 40 °C.
NOTA: Raccomandiamo la proteasi IV per i topi adulti. Se si utilizzano topi più giovani, come P0, si consiglia di utilizzare un enzima più delicato, come la proteasi III, per 30 minuti. Quando si risolvono i problemi per età diverse, osservare l'integrità del tendine e verificare la degradazione. Se il tessuto si dissocia o si degrada, diminuire il tempo di incubazione o cambiare la proteasi utilizzata. - Lavare nuovamente i campioni in tampone TEG per 30 minuti e quindi ibridare le sonde secondo il protocollo ISH14.
- Aggiungere 50-100 μl di reagente di montaggio sulla parte superiore del campione. Posizionare un vetrino coprioggetti sopra i campioni per microscopia. Durante l'imaging, utilizzare un ingrandimento elevato (obiettivo 40x o superiore) per visualizzare il segnale.
- Invece di utilizzare la soluzione di pretrattamento consigliata, immergere i vetrini in una capsula di colorazione contenente tampone TEG per 4 ore a 60 °C.
3. Adattamento del protocollo HCR ISH15
- Postfissazione
- Preparare PFA al 4% (in PBS), soluzione di PBT (0,1% Tween-20 in PBS) e proteinasi K in PBS (concentrazione madre di 10 mg/mL, concentrazione finale di 5 μg/mL).
- Per iniziare la prima fase del postfisso, immergere i vetrini in una capsula colorante contenente il 4% di PFA per 5 minuti; quindi, rimuovere la soluzione di PFA al 4% e conservarla per utilizzarla nei passaggi successivi. Sciacquare i vetrini per 2 x 5 minuti in una soluzione di PBT e rimuovere la soluzione di PBT. Porre i vetrini in una capsula colorante contenente una soluzione di proteinasi K (5 μg/mL) per 5 minuti e sciacquarli nella soluzione di PBT 2 x 5 minuti.
- Iniziare il secondo passaggio di suffisso e immergere i vetrini nel PFA al 4% per 5 minuti.
NOTA: Questo è lo stesso 4% di PFA risparmiato dal passaggio 3.1.2. - Sciacquare i vetrini per 3 x 5 minuti nella soluzione PBT.
NOTA: Tutte le fasi di postfissazione vengono eseguite con i vetrini su un rack per vetrini e in piastre di colorazione contenenti le rispettive soluzioni.
- Acetilazione
- Preparare la soluzione di acetilazione contenente 625 μL di anidride acetica, 3,3 mL di tampone 1 M trietanolammina (TEA) e 246 mL di ddH2O. Assicurarsi di mescolare bene e utilizzare immediatamente dopo la preparazione.
- Mettere i vetrini nella soluzione di acetilazione per 10 minuti e poi risciacquare 3 x 5 minuti in soluzione di PBT. Sciacquare i vetrini in ddH2O e lasciarli asciugare all'aria per 30 min. Quindi, disegna intorno ai campioni con una penna barriera idrofobica.
NOTA: Tutte le fasi di acetilazione vengono eseguite con i vetrini su un rack per vetrini e in piastre di colorazione contenenti le rispettive soluzioni
- Ibridazione
- Preriscaldare il tampone di ibridazione a 37 °C e preriscaldare una camera umidificata a 37 °C.
ATTENZIONE: Il tampone contiene formammide, un materiale pericoloso. Se i vetrini non sono sufficientemente asciutti, tamponare i bordi con un panno da laboratorio per rimuovere l'eccesso di dH2O. - Preparare le soluzioni della sonda aggiungendo 0,4 pmol di ciascuna miscela di sonde a 100 μL di tampone di ibridazione.
NOTA: Se si utilizza l'imaging dHCR, utilizzare una concentrazione maggiore di sonda per migliorare l'efficienza dell'ibridazione della sonda. - Preparare 4 L di tampone citrato di sodio cloruro di sodio (20x SSC) combinando 3 M di NaCl (701,1 g di NaCl in un volume finale di 4 L) e 0,3 M di Na3CH6H5O7•2H2O (352,8 g) e quindi regolando il pH finale a 7 aggiungendo HCl o 10 N NaOH. Aggiungere una quantità sufficiente di H2O per raggiungere i 4 L. Preparare 5 SSCT diluendo 20 SSC a 5 volte e aggiungendo abbastanza 10% di Tween 20 in modo che sia lo 0,1% del volume totale.
- Aggiungere 200 μl di tampone di ibridazione al campione, posizionare i vetrini in una camera umidificata e lasciarli incubare per 10 minuti. Rimuovere la soluzione di ibridazione e drenare il tampone in eccesso sul vetrino tamponando i bordi con un panno da laboratorio.
- Aggiungere 100 μl della soluzione della sonda preparata sopra il campione e posizionare un vetrino coprioggetti sul campione. Incubare in forno per una notte o per circa 12-16 ore in una camera umidificata con una soluzione contenente 5x SSC e 50% di formammide. Impostare la temperatura a 37 °C.
NOTA: Poiché l'incubazione avviene durante la notte, l'aggiunta del vetrino coprioggetti serve a ridurre al minimo l'evaporazione. - Lasciare un'aliquota di 5 volte la soluzione di SSCT in un bagno d'acqua a 37 °C e una seconda aliquota a RT. Anticipare i volumi di queste aliquote necessari in base al numero di campioni e al numero di lavaggi nella fase successiva.
- Preriscaldare il tampone di ibridazione a 37 °C e preriscaldare una camera umidificata a 37 °C.
- Lavaggio
- Utilizzando la soluzione 5x SSCT precedentemente preriscaldata, preparare soluzioni di tampone di lavaggio al 75%, 25% di SSCT, 50% di tampone di lavaggio/50% 5x SSCT e 25% di tampone di lavaggio/75% 5x SSCT.
- Per rimuovere le sonde in eccesso, immergere i vetrini in serie per 15 minuti per lavaggio a 37 °C in tampone di lavaggio al 75%, 25% 5x SSCT, 50% tampone di lavaggio/50% 5x SSCT e 25% tampone di lavaggio/75% 5x SSCT. Lasciare che i vetrini coprioggetti fluttuino via dai campioni.
- Incubare i vetrini 2 x 15 min con 100% 5x SSCT e poi immergerli in 5x SSCT per 5 min a RT.
NOTA: Le fasi di lavaggio possono essere eseguite con i vetrini su un carrello per vetrini e in vaschette di colorazione contenenti le rispettive soluzioni o con le soluzioni aggiunte sotto forma di goccioline.
- Amplificazione
- Preparare 6 pmol di hairpin h1 e 6 pmol di hairpin h2 raffreddando a scatto 2 μL di un brodo da 3 μM (riscaldare a 95 °C per 90 s e raffreddare a RT). Proteggi le forcine dalla luce durante questo processo.
NOTA: Le forcine HCR h1 e h2 sono fornite in un buffer di stoccaggio a forcina e sono pronte per il raffreddamento a scatto. Agganciare le forcine h1 e h2 in tubi separati. - Asciugare i vetrini tamponandone i bordi con una salvietta da laboratorio. Quindi, aggiungere 200 μL di tampone di amplificazione sopra il campione e metterlo in una camera umidificata per 30 minuti a RT. Preparare la miscela a forcina aggiungendo forcine h1 raffreddate a scatto e forcine h2 raffreddate a scatto a 100 μl di tampone di amplificazione a RT.
- Rimuovere il tampone di amplificazione e scolare il liquido in eccesso sul vetrino tamponando i bordi con una salvietta da laboratorio. Aggiungere 100 μl della miscela a forcina precedentemente preparata e posizionare il parafilm sopra il campione. Incubare i vetrini per un minimo di 4 ore o per una notte in una camera buia e umidificata a RT.
- Per rimuovere le forcine in eccesso, immergere i vetrini in 5x SSCT in una piastra colorante e incubare a RT per 30 minuti. Ripetere il lavaggio in SSCT fresco 5 volte per 30 minuti e una terza volta per 5 minuti.
- Asciugare i vetrini tamponando i bordi con un panno da laboratorio e aggiungere 50-100 μl di reagente di montaggio sulla parte superiore del campione. Posizionare un vetrino coprioggetti sopra i campioni per microscopia. Durante l'imaging, utilizzare un ingrandimento elevato (obiettivo 40x o superiore) per visualizzare il segnale.
- Preparare 6 pmol di hairpin h1 e 6 pmol di hairpin h2 raffreddando a scatto 2 μL di un brodo da 3 μM (riscaldare a 95 °C per 90 s e raffreddare a RT). Proteggi le forcine dalla luce durante questo processo.
Risultati
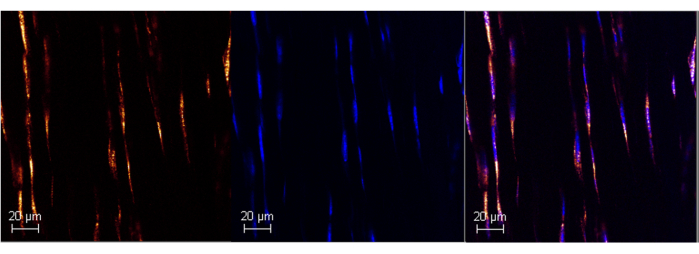
Figura 1: Espressione dell'RNA poli A nel tendine d'Achille di topo adulto utilizzando RNAScope. Immagine rappresentativa del successo della marcatura Poly A nel tendine d'Achille di topo (pannello sinistro) utilizzando il test ISH commercializzato. La colocalizzazione con DAPI conferma la specificità della sonda ...
Discussione
In questo articolo, descriviamo le modifiche apportate per sfruttare gli strumenti ISH esistenti in modo che possano essere utilizzati nel tessuto tendineo con un alto grado di specificità e sensibilità. Poiché il tendine è un tessuto altamente denso di matrice, spesso è necessario apportare modifiche al protocollo per ottenere gradi simili di penetrazione e specificità della sonda. Questi specifici metodi di permeabilizzazione e strategie di amplificazione del segnale del tessuto ...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Gli autori ringraziano Jenna Galloway e i membri del Galloway Lab per il loro supporto e incoraggiamento nello sviluppo e nella risoluzione dei problemi di questi protocolli.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 M triethanolamine buffer | |||
| 10% Formalin solution | |||
| 10% Tween-20 | |||
| 20x Saline Sodium Citrate buffer | |||
| 4% PFA | |||
| ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 | ACD | 323100 | |
| Acetic Anhydride | |||
| Axio Imager Microscope | ZEISS | ||
| C57BL/J6 mice | JAX ID: 000664 | ||
| Coverslips | Fisher | 12-541-042 | |
| ddH2O | |||
| ETDA | Thermofisher | AM9262 | |
| EtOH | |||
| Glucose | VWR Chemicals BDH | BDH9230-500G | |
| HCR RNA-FISH Bundle | Molecular Instruments Inc. | ||
| HybEZ II Hybridization System | ACD | ||
| Immedge Barrier Pen | Vector Laboratories | H4000 | |
| Leica SPE Confocal Microscope | Leica | ||
| Parafilm | Fisher | ||
| Phosphate-buffered saline (PBS, 1x) | Invitrogen | AM9625 | Dilute 10x PBS in milli-Q water to get 1x solution |
| Protease IV | |||
| Proteinase K | Roche | 3115836001 | |
| RNAscope H2O2 and Protease Reagents | ACD | PN 322381 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V3 |
| RNAscope Multiplex Fluorescent Detection Kit | ACD | PN 323110 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 |
| RNAscope Target Retrieval reagents | ACD | 322000 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V4 |
| RNAscope Wash Buffer | ACD | PN 310091 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V5 |
| RNAscope Probe Diluent | ACD | 300041 | |
| Slide holder | StatLab | 4465A | |
| Staining Dish with Lid | StatLab | LWS20WH | |
| Superfrost Plus Microscope slides | Fisher | 1255015 | treated, charged slides |
| Tris-HCl | |||
| Xylene | Sigma-Aldrich | 534056-4L |
Riferimenti
- Sharma, P., Maffulli, N. Tendon injury and tendinopathy: healing and repair. Journal of Bone and Joint Surgery. 87 (1), 187-202 (2005).
- Brent, A. E., Schweitzer, R., Tabin, C. J. A somitic compartment of tendon progenitors. Cell. 113 (2), 235-248 (2003).
- Noden, D. M. The role of the neural crest in patterning of avian cranial skeletal, connective, and muscle tissues. Developmental Biology. 96 (1), 144-165 (1983).
- Chen, J. W., Galloway, J. L. The development of zebrafish tendon and ligament progenitors. Development. 141 (10), 2035-2045 (2014).
- Schweitzer, R., et al. Analysis of the tendon cell fate using Scleraxis, a specific marker for tendons and ligaments. Development. 128 (19), 3855-3866 (2001).
- Tsai, S., Nödl, M., Galloway, J. Bringing tendon biology to heel: Leveraging mechanisms of tendon development, healing, and regeneration to advance therapeutic strategies. Developmental Dynamics. 250 (3), 393-413 (2021).
- Kannus, P., et al. Location and distribution of non-collagenous matrix proteins in musculoskeletal tissues of rat. The Histochemical Journal. 30 (11), 799-810 (1998).
- Kannus, P. Structure of the tendon connective tissue. Scandinavian Journal of Medicine & Science in Sports. 10 (6), 312-320 (2000).
- Thorpe, C. T., Birch, H. L., Clegg, P. D., Screen, H. R. C. The role of the non-collagenous matrix in tendon function. International Journal of Experimental Pathology. 94 (4), 248-259 (2013).
- Lin, T. W., Cardenas, L., Soslowsky, L. J. Biomechanics of tendon injury and repair. Journal of Biomechanics. 37 (6), 865-877 (2004).
- Grinstein, M., et al. A distinct transition from cell growth to physiological homeostasis in the tendon. eLife. 8, e48689 (2019).
- Villaseñor, S., Grinstein, M. Two-photon microscopy for the study of tendons. Journal of Visualized Experiments. , e65853 (2023).
- Qin, C., et al. The cutting and floating method for paraffin-embedded tissue for sectioning. Journal of Visualized Experiments. (139), e58288 (2018).
- Wang, F., et al. RNAscope®: A Novel In Situ RNA Analysis Platform for Formalin-Fixed Paraffin-Embedded Tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Choi, H. M. T., et al. Third-generation situ hybridization chain reaction: multiplexed, quantitative, sensitive, versatile, robust. Development. 145 (12), 165753 (2018).
- Jackson, D. J., Herlitze, I., Hohagen, J. A whole mount in situ hybridization method for the gastropod mollusc Lymnaea stagnalis. Journal of Visualized Experiments. (109), e53968 (2016).
- Young, A. P., Jackson, D. J., Wyeth, R. C. A technical review and guide to RNA fluorescence in situ hybridization. PeerJ. 8, e8806 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon