A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
קביעת מתילציה DNA של גנים מוטבעים ארבידופסיס האנדוספרם
In This Article
Summary
החתמה היא תופעה במפעל ורבייה יונק. מתילציה DNA ממלא תפקיד חשוב במנגנוני של הטבעה. האנדוספרם בידוד וקביעת מעמד מתילציה של גנים מוטבעים ארבידופסיס יכול להיות קשה. בפרוטוקול זה, אנו מתארים כיצד לבודד את האנדוספרם ולקבוע מתילציה של רצפי bisulfite.
Abstract
Thaliana ארבידופסיס הוא אורגניזם מודל מצוין לחקר מנגנוני epigenetic. אחת הסיבות היא מוטציה הפסד-of-null פונקציה של methyltransferases DNA היא בת קיימא, ובכך לספק מערכת ללמוד איך הפסד של מתילציה DNA בגנום משפיע על הגדילה וההתפתחות. החתמה מתייחס הביטוי ההפרש של אללים אימהי ואבהי ובעל תפקיד חשוב בהתפתחות רבייה הן יונקים וצמחים. מתילציה DNA הוא קריטי לקביעת אם אללים אימהי או אבהי של הגן המוטבע מבוטא או מושתק. בשנת צמחים פורחים, יש אירוע הפריה כפולה רפרודוקציה: תא הזרע מפרה את הביצית ליצירת העובר ואת הנתיכים הזרע השני עם תא מרכזי להצמיח האנדוספרם. האנדוספרם היא הרקמה בה החתמה מתרחשת בצמחים. MEDEA, תחום SET גן Polycomb הקבוצה, FWA, גורם שעתוק ויסות פריחה, הם הראשונים שני גנים הראו להיות טבועה האנדוספרם והביטוי שלהם נשלטת על ידי מתילציה של דנ"א demethylation צמחים. על מנת לקבוע את מעמדם החתמה של גן דפוס המתילציה של האנדוספרם, אנחנו צריכים להיות מסוגלים לבודד את האנדוספרם הראשון. מאז הוא זרע זעיר ארבידופסיס, הוא נותר מאתגר לבודד את האנדוספרם ארבידופסיס ולבחון מתילציה שלה. בפרוטוקול זה וידאו, אנו מדווחים איך לנהל את צלב גנטית, לבודד רקמת האנדוספרם מזרעים, כדי לקבוע את מצב המתילציה על ידי רצף bisulfite.
Protocol
I. גנטיות מעבר
1. מסרס ההורה הנקבי
כדי להבחין בין אללים אימהי ואבהי באמצעות רצף ה-DNA פולימורפיזם, שני ecotypes שונים, כגון קולומביה-0 (Col-0) ו הימלר, ייבחרו כהורים הנשית והגברית. הצמחים צריכים להיות צעירים ובריאים. אפשר לסרס ההורה הנקבי באמצעות מיקרוסקופ לנתח, מגדלת מגן, או בעין בלתי מזוינת. אתר הבמה 12 פרחים (סמית. Et al 1990) והסר פרחים או siliques מעל ומתחת להם על ידי חיתוך הבסיס של pedicel עם המספריים. לעקר מלקחיים על ידי טבילה הבסיס של קצה בעדינות לתוך מבחנה של אתנול 95%, אשר תסיר את כל גרגרי אבקה על מלקחיים ולהרוג את האבקה גם כן. לחטט בעדינות לגזרים את ניצני פרחים באמצעות מלקחיים, להסיר בעדינות 4 עלי גביע, 4 עלי כותרת, ו -6 אבקנים, עוזב פיסטיל החשוף ללא פגע. נסו להימנע נזק carpels במהלך תהליך זה.
2. בחירת התורם אבקה וביצוע ההאבקה
התורמים הטובים ביותר הם אבקה anthesed פרחים לפתוח בשלב 14 עם עלי הכותרת הארכת בזווית של 90 מעלות על פיסטיל (סמית. Et al 1990), שבו הרבה אבקה היא לשפוך. תפוס את פרח בבסיס בדיוק מעל pedicel, אשר תגרום לפרח להפיץ פתוח. אבק את הסטיגמה של פיסטיל הכינו עם מאבק. לאחר ההאבקה, הסטיגמה יהיה מכוסה אבקה צהובה, אשר ניתן לצפות בקלות תחת מיקרוסקופ.
3. סימון הצלב
לאחר מאביקים כל עליים מסורס על צמח, ומתייגים את הצלב עם תג תכשיטים מידע להורים הנשית והגברית ותאריך. המקום יתד באדמה קרוב הצמח, להשתמש במחרוזת לקשור את גזע של תפרחת אל המוקד, ולכסות את עליים pollinated עם שקית פלסטיק.
השנייה. בידוד של רקמות האנדוספרם של ארבידופסיס
1. הכנת חומרים
בדרך כלל אנחנו מקבלים חומרים הדרושים מוכן לפני קצירת האנדוספרם ורקמות העובר: מיכל חנקן נוזלי, חנקן נוזלי, מיקרוסקופ לנתח, שני זוגות חדשים של עדין קצה מלקחיים (5 Inox FST ידי דומון ביולוגיה, שוויץ.) זכוכית שקופיות מיקרוסקופ ( 3 "X 1" X 1.0 מ"מ), 5.7 pH פתרון של M 0.3 ו - 5 מ"מ סורביטול MES.
2. איסוף siliques
ב - 7 או 8-יום לאחר ההאבקה-(DAP), זרעים מוכנים להיות שנקטפו על אמצע לטרפד לשלב מאוחר של העובר ואת גזור עבור האנדוספרם ואת העובר. לפעמים, זה עלול לקחת 9-10 DAP אם הפרחים מסורס צעירים מדי.
3. האנדוספרם בידוד העובר
מאז זרעי ארבידופסיס זעירים, אנו משתמשים במיקרוסקופ לנתח לבודד האנדוספרם ואת העובר. שים silique 8-DAP תחת מיקרוסקופ, להשתמש זוג מלקחיים להחזיק את pedicel silique ולהשתמש קצה זוג נוסף של מלקחיים להחליק silique לפתוח את בשוליים שבו השניים מתמזגים carpels. השתמש זוג מלקחיים לאסוף זרע אחד על 5.7 פתרונות ה-pH של 0.3 M ו - 5 מ"מ סורביטול MES, לעשות חתך קטן בסוף micropyle להחליק את העובר, לסחוט את הסוף חתוכים לדחוף את האנדוספרם ו האנדוספרם נפרד מן המעיל זרע (Kinoshita et al. 2004). שים את העובר לתוך האנדוספרם microtubes נפרד חנקן נוזלי. המשך עד שאנחנו צוברים העובר או האנדוספרם 10-15 siliques ולאחר מכן לאחסן את הצינורית למקפיא -80 מעלות צלזיוס. במקרים מסוימים, האנדוספרם לא צריך להפריד את המעיל זרע, כלומר, אפשר לבודד RNA הכולל האנדוספרם ואת מעיל תערובת זרעי לניתוח החתמה גן.
ג. Bisulfite סידור
1. חובה ריאגנטים
אמוניום ברומיד Cetyltrimethyl (CTAB) להכנת דנ"א גנומי, אנזימי הגבלה, 3 M NaOH (טריים), 6.42 מ 'אוריאה / 4 bisulfite M נתרן (2 metabisulfite M נתרן, סיגמא אולדריץ, S9000, Na2S2O5, מולקולרית משקל: 190), הידרוקינון 10 מ"מ, ערכת טיהור דנ"א (DNA Promega אשף לנקות מערכת, חתול. # A7280), TE חיץ, 6.3 M NaOH (טריים), 10 מ NH 4 OAc, 20 מיקרוגרם / tRNA μL, אתנול ו - 100% .
2. פרטי פרוטוקול הטיפול bisulfite
- האנדוספרם או לבודד DNA גנומי העובר באמצעות הליך CTAB (רוג'רס Bendich, 1988).
- תקציר 100 ננוגרם - 2 מיקרוגרם של ה-DNA הגנומי של μL 20-100 בנפח כולל μL עם אנזימי הגבלה, כי לחתוך מחוץ לאזור כדי להיות מנותח. עבור האמרגן MEA, אנו משתמשים XhoI, NdeI ו PstI או HindIII.
- לפגל אנזימי הגבלה על ידי ה-DNA רותחים במשך חמש דקות ואז להרוות על הקרח.
- מוסיפים 1 / 9 נפח (2.2 μL עבור DNA 20 מתעכל μL) של 3 M NaOH ו לדגור על 37 מעלות צלזיוס במשך 15 דקות.
- העברת הפתרון צינור 250 PCR μL.
- להמיס 7.5 גרם של אוריאה ב 10 מ"ל של מים מזוקקים סטריליים; לאט להוסיף 7.6 גרם נתרן metabisulfite מעל 1-2 שעות חימום בדרך כלל עוזר המסת; התאם את ה-pH ל 5 עם טריים 10 M NaOH, הוסף מים מזוקקים סטריליים על סופי נפח מ"ל 20. זוהי 6.24 אוריאה ז / 4 bisulfite M פתרון נתרן.
- הוסף 6.24 אוריאה ז / 4 bisulfite M נתרן פתרון ריכוז סופי של 5.36 מ 'ו 3.44 מ', בהתאמה (Paulin et al., 1998). לדוגמה, להוסיף 208 μL של אוריאה 6.42 M / M 4 bisulfite נתרן פתרון μL 22.2 לעיל של הדנ"א הגנומי מפוגל (שיאו et al., 2003).
- הוסף הידרוקינון 10 mM ל-DNA בריכוז סופי של 0.5 מ"מ (12 μL לעיכול 20 μL).
- התנהגות טיפול bisulfite במכונה PCR: 30 מחזורים של 55 מעלות צלזיוס במשך 15 דקות ו 95 מעלות צלזיוס למשך 30 שניות.
- Desalt את ה-DNA bisulfite שטופלו באמצעות מערכת לנקות אשף דנ"א Promega ומעקב פרוטוקול (ג'ייקובסן et al., 2000).
- מדוד את נפח המדויק של TE התאושש הטור לאחר desalting ולהוסיף 6.3 M NaOH ריכוז סופי של 0.3 מ 'לדגור על 37 מעלות צלזיוס במשך 15 דקות.
- הוסף 10 M NH 4 OAc (pH 7.0) לריכוז סופי של M 3, 2 μL של 20 מיקרוגרם / tRNA μL, 3 כרכים של 100% אתנול ואז לערבב. צנטריפוגה במשך 15 דקות ב 14,000 סל"ד.
- שטפו את הכדור פעם אחת עם 70% אתנול, לעשות צנטריפוגה קצר, ולהסיר אתנול נוספת.
- יבש גלולה ב speedvac למשך 5-10 דקות ו resuspend ב 25-100 μL חיץ TE בהתאם החל כמות ה-DNA. נתרן bisulfite שטופלו DNA עכשיו הוא מוכן לניתוח PCR.
3. PCR הגברה
- מאז cytosines unmethylated מומרים אורציל, קשה כדי להגביר ושבר גדול באמצעות DNA bisulfite שטופלו כתבנית. לפיכך, אנו בדרך כלל עיצוב primers להגביר מוצר לא יותר מ 500 נ"ב. רצף האמרגן 4-kb MEA, עיצבנו קבוצות רבות של primers ואת מוגבר 14 שברים חופפים כדי לכסות את האזור כולו (שיאו et al., 2003).
- כדי רצף העליונה גדיל, בעיצוב פריימר קדימה, אני) לבחור (G גואנין) עשיר באזור על מנת להיות חישול בטמפרטורה גבוהה ללא נוקלאוטידים ארוך במיוחד של primers; II) לשנות C (ציטוזין) ל-Y ( פירימידין) בשעה CG והקשרים CNG ולשנות את C הנותרים T (תימין). בעיצוב פריימר הפוכה, אני) בוחר אזור C עשיר; II) לשנות את G R (purine) בשעה CG והקשרים CNG ולשנות את G הנותרים A (אדנין).
- רצף התחתונה גדיל, בעיצוב פריימר קדימה, אני) בוחר אזור C עשיר; II) לשנות את G R ב CG והקשרים CNG ולשנות את G הנותרים א בעיצוב פריימר הפוכה, אני) לבחור אזור ה-G-עשיר; II) לשנות את C ל-Y (פירימידין) בשעה CG והקשרים CNG ולשנות את C הנותרים ט
- אנחנו בדרך כלל להשתמש 1-2 μL של ה-DNA נתרן bisulfite שטופלו כתבנית הגברה כל PCR (שיאו et al., 2003). מוצר ה-PCR צריך להיות מנותח על ידי אלקטרופורזה בג'ל על מנת לאשר את הגודל הנכון של שבר, ולאחר מכן להיות מטוהרים ג'ל משובטים לתוך ת"א Topo שיבוט וקטור pCR2.1 (Invitrogen) כמו להוסיף. מושבת יחיד הרים ותרבותיים; DNA פלסמיד חילוץ ושלח עבור סידור.
4. רצף ניתוח
העיקרון של רצף bisulfite היא ציטוסין unmethylated יומרו אורציל בשל deamination hydrolytic על ידי ריכוז גבוה של bisulfite נתרן ב pH5.0 אשר יהיה מוגבר כמו תימין במוצר ה-PCR, בעוד 5-מתיל ציטוזין לא יהיה שונה על ידי bisulfite נתרן ולהישאר להיות ציטוזין לאחר הגברה PCR (Clark et al, 1994;. פרומר et al, 1992).. לאחר השגת התוצאה ריצוף, נשווה את זה עם תבנית גדיל ספציפי המשמש הגברה PCR. אם שאריות ציטוסין בתבנית קורא כמו תימין בתוצאה רצף, מעידה על כך ציטוסין אינו מפוגל. אם שאריות ציטוסין בתבנית נשאר ציטוסין ב רצף, זה אומר כי ציטוסין הוא מפוגל.
IV. נציג תוצאות
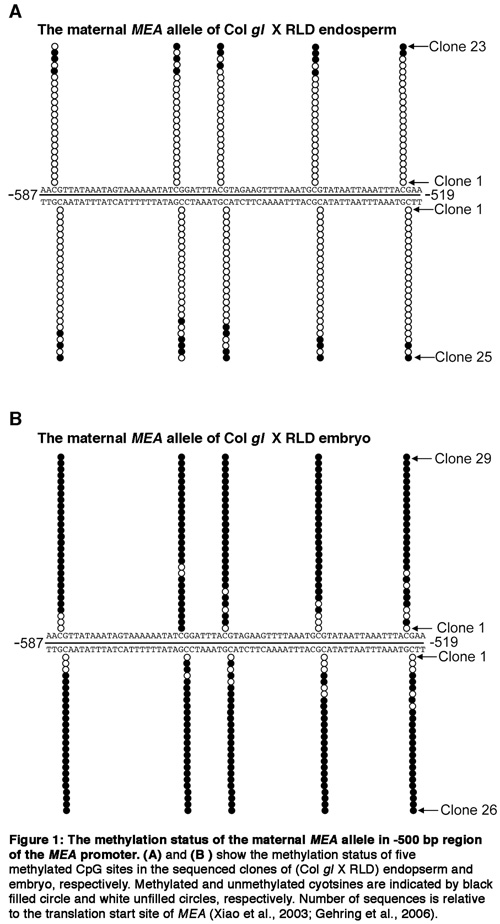 באיור 1.
באיור 1.
Discussion
קל יחסית להפריד העובר מן האנדוספרם ואת המעיל זרע, אבל זה משעמם להפריד האנדוספרם מן המעיל זרע, במיוחד עבור זרע בשלב מוקדם או אמצע לטרפד של העובר. מאז מעיל הזרע רק תורם כמות קטנה מאוד של רקמה, עבור גנים מסוימים, למשל, MEA ו FWA, אין לנו להפריד את האנדוספרם מן המעיל ז?...
Disclosures
Acknowledgements
המחברים מודים לגב 'ג'ניפר מ Lommel וטרה נ Rognan לצורך תחזוקה של צמחי ארבידופסיס. עבודה זו נתמכה על ידי סטארט כספים אוניברסיטת סנט לואיס ו - National Institutes of Health מענקים 1R15GM086846-01 ו-01S1 3R15GM086846 אל שיאו וו.
Materials
ספקי
- הביתור מיקרוסקופ
- מספרים
- עצה מלקחיים פיין
- תכשיטים תגית
- הצמח תחרות
- מחרוזת או טוויסט-Ties
- 4 "X 2" X 8 "שקיות פוליאתילן
- 3 "X 1" X מיקרוסקופ שקופיות 1.0 מ"מ
- חנקן נוזלי
- מיכלי חנקן נוזלי
- מחממים חסום
- מנורות ה-PCR
- Thermocycler
- Microcentrifuge צינורות
- Microcentrifuge
- ג'ל אלקטרופורזה מתקן
- ארבידופסיס thaliana קולומביה-0 צמחים
- ארבידופסיס thaliana לנדסברג erecta צמחים
ריאגנטים
- 70% אתנול
- 95% אתנול
- 100% אתנול
- 0.3 מ סורביטול ו 5 מ"מ MES-pH 5.7
- אמוניום ברומיד Cetyltrimethyl (CTAB)
- 100% אתנול
- Cholorform
- הגבלת אנזימים
- 3 M NaOH
- 6.3 M NaOH
- 6.24 מ 'דשן / 4 bisulfite M נתרן
- סטרילי מזוקקים H 2 O
- 10 mM הידרוקינון
- אשף ה-DNA לנקות מערכת (Promega)
- 10 מ NH 4 OAc
- 20 מיקרוגרם / tRNA μL
- TE חיץ
- מדד ת"א Topo שיבוט Kit (Invitrogen)
References
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Cokus, S. J., Feng, S., Zhang, X., Chen, Z., Merriman, B., Haudenschild, C. D., Sriharsa Pradhan, S., Nelson, S. F., Pellegrini, M., Jacobsen, S. E. Shotgun bisulphite sequencing of the Arabidopsis genome reveals DNA methylation patterning. Nature. 452, 215-219 (2008).
- Frommer, M., McDonald, L. E., Millar, D. S., Collis, C. M., Watt, F., Grigg, G. W. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc Natl Acad Sci USA. 89, 1827-1831 (1992).
- Gehring, M., Huh, J. H., Hsieh, T. F., Penterman, J., Choi, Y., Harada, J. J., Goldberg, R. B., Fischer, R. L. D. E. M. E. T. E. R. DNA glycosylase establishes MEDEA polycomb gene self-imprinting by allele-specific demethylation. Cell. 124, 495-506 (2006).
- Henderson, I. R., Chan, S. R., Cao, X., Johnson, L., Jacobsen, S. E. Accurate sodium bisulfite sequencing in plants. Epigenetics. 5, 47-49 (2010).
- Hsieh, T. F., Ibarra, C. A., Silva, P., Zemach, A., Eshed-Williams, L., Fischer, R. L., Zilberman, D. Genome-wide demethylation of Arabidopsis endosperm. Science. 324, 1451-1454 (2009).
- Jacobsen, S. E., Sakai, H., Finnegan, E. J., Cao, X., Meyerowitz, E. M. Ectopic hypermethylation of flower-specific genes in Arabidopsis. Curr. Biol. 10, 179-186 (2000).
- Kinoshita, T., Miura, A., Choi, Y., Kinoshita, Y., Cao, X., Jacobsen, S. E., Fischer, R. L., Kakutani, T. One-way control of FWA imprinting in Arabidopsis endosperm by DNA methylation. Science. 303, 521-523 (2004).
- Lister, R., O'Malley, R. C., Tonti-Filippini, J., Gregory, B. D., Berry, C. C., Millar, A. H., Ecker, J. R. Highly integrated single-base resolution maps of the epigenome in Arabidopsis. Cell. 133, 523-536 (2008).
- Lister, R., Pelizzola, M., Dowen, R. H., Hawkins, R. D., Hon, G., Tonti-Filippini, J., Nery, J. R., Lee, L., Ye, Z., Ngo, Q. -. M. Human DNA methylomes at base resolution show widespread epigenomic differences. Nature. 462, 315-322 (2009).
- Paulin, R., Grigg, G. W., Davey, M. W., Piper, A. A. Urea improves efficiency of bisulfite-mediated sequencing of 5'- methylcytosine in genomic DNA. Nucl. Acids Res. 26, 5009-5010 (1998).
- Rogers, S. O., &, B. e. n. d. i. c. h., J, A. Extraction of DNA from plant tissues. Plant Molecular Biology Manual. A6, 1-10 (1988).
- Smyth, D. R., Bowman, J. L., Elliot, M., Meyerowitz, E. M. Early Flower Development in Arabídopsis. Plant Cell. 2, 755-767 (1990).
- Xiao, W., Gehring, M., Choi, Y., Margossian, L., Pu, H., Harada, J. J., Goldberg, R. B., Pennell, R. I., Fischer, R. L. Imprinting of the MEA Polycomb gene is controlled by antagonism between MET1 methyltransferase and DME glycosylase. Dev. Cell. 5, 891-901 (2003).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved