È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Determinazione della metilazione del DNA di geni impressa Arabidopsis Dell'endosperma
In questo articolo
Riepilogo
L'imprinting è un fenomeno in impianti e riproduzione dei mammiferi. Metilazione del DNA gioca un ruolo importante nei meccanismi di imprinting. Isolando endosperma e determinare lo stato di metilazione dei geni impressa Arabidopsis Può essere difficile. In questo protocollo, si descrive come isolare endosperma e determinare metilazione mediante sequenziamento bisolfito.
Abstract
Arabidopsis thaliana è un organismo modello eccellente per lo studio dei meccanismi epigenetici. Uno dei motivi è la perdita di funzione mutante nullo di DNA metiltransferasi è vitale, fornendo così un sistema per studiare come la perdita di metilazione del DNA in un genoma influisce sulla crescita e lo sviluppo. Imprinting si riferisce alla espressione differenziale di alleli paterni e materni e svolge un ruolo importante nello sviluppo riproduzione in entrambi i mammiferi e piante. Metilazione del DNA è essenziale per determinare se gli alleli materni o paterni di un gene impresso è espressa o tacere. In piante da fiore, c'è un evento doppia fecondazione in riproduzione: una cellula spermatozoo feconda l'ovulo per formare embrione e un fusibili sperma secondo con la cella centrale per dar luogo a endosperma. Endosperma è il tessuto in cui imprinting si verifica nelle piante. MEDEA, un dominio gene SET Polycomb gruppo e FWA, un fattore di trascrizione che regolano la fioritura, sono i primi due geni dimostrato di essere impressa nella endosperma e la loro espressione è controllata da metilazione del DNA e demetilazione in piante. Al fine di determinare lo stato imprinting di un gene e pattern di metilazione in endosperma, dobbiamo essere in grado di isolare endosperma prima. Dal seme è piccolo in Arabidopsis, rimane difficile da isolare endosperma Arabidopsis ed esaminare la sua metilazione. In questo protocollo video, si segnala come condurre un incrocio, per isolare il tessuto dell'endosperma da semi, e per determinare lo stato di metilazione mediante sequenziamento bisolfito.
Protocollo
I. genetica Crossing
1. Castrante il genitore femmina
Al fine di distinguere gli alleli materni e paterni, utilizzando sequenza polimorfismo del DNA, due ecotipi diversi, ad esempio, la Columbia-0 (Col-0) e Ler, saranno scelti come genitori femminile e maschile. Le piante devono essere giovani e sani. Si può evirare il genitore femminile utilizzando un microscopio da dissezione, visiera d'ingrandimento, o occhio nudo. Individuare fase-12 fiori (Smyth et al., 1990) e rimuovere eventuali fiori o silique sopra e sotto di loro da taglio alla base del pedicello con le forbici. Sterilizzare pinze immergendo la base della punta delicatamente in un bicchiere di etanolo al 95%, che eliminerà qualsiasi grani di polline sulle pinze e uccidere il polline pure. Sollevare delicatamente a parte i boccioli dei fiori con pinze, e rimuovere delicatamente 4 sepali, 4 petali e 6 stami, lasciando il pistillo nudo e intatto. Cercate di evitare di danneggiare la carpelli durante questo processo.
2. La raccolta del donatore polline e lo svolgimento impollinazione
I donatori migliori polline sono anthesed fiori aperti nella fase 14 con i petali si estende ad un angolo di 90 ° al pistillo (Smyth et al., 1990), in cui un sacco di polline è spargimento. Prendete il fiore alla base e appena sopra il peduncolo, che farà sì che il fiore di diffondere aperto. Polvere lo stigma del pistillo preparato con le antere. Dopo l'impollinazione, lo stigma sarà coperto con il polline giallo, che può essere facilmente osservati al microscopio.
3. Etichettatura la croce
Dopo impollinatori tutti i pistilli castrato su una pianta, si etichetta la croce con un tag gioielli e informazione dei genitori femminili e maschili e la data. Mettere un palo nel terreno vicino alla pianta, utilizzare una stringa per legare il fusto della infiorescenza al rogo, e coprire i pistilli impollinati con un sacchetto di plastica.
II. Isolamento dei tessuti dell'endosperma in Arabidopsis
1. Preparazione dei materiali
Noi di solito ottengono materiali necessari pronto prima della raccolta della mandorla farinosa e tessuti dell'embrione: un serbatoio di azoto liquido, azoto liquido, un microscopio da dissezione, due nuove coppie di fine-punta pinza (5 INOX FST da Dumont biologia, in Svizzera.), Vetrini microscopio ( 3 "X 1" X 1.0 mm), una soluzione di 5,7 pH di 0,3 M sorbitolo e 5 mM MES.
2. Silique raccolta
A 7 - 8-o giorno dopo l'impollinazione (DAP), i semi sono pronte per essere raccolte a metà e la fine della fase siluro embriogenesi e sezionato per endosperma ed embrione. A volte, potrebbe prendere 9-10 DAP se i fiori evirati sono troppo giovani.
3. Endosperma isolare ed embrione
Dal momento che i semi di Arabidopsis sono piccoli, usiamo un microscopio da dissezione per isolare endosperma ed embrione. Mettere un 8-DAP Silique al microscopio, usare un paio di pinze per tenere il pedicello Silique e utilizzare la punta di un altro paio di pinze a scivolare aprire il Silique a margine dove fondono due carpelli. Usare un paio di pinze per prendere un seme sul pH 5,7 soluzioni di 0,3 M sorbitolo e 5 mM MES, fare un piccolo taglio alla fine micropilo a scivolare fuori embrione, spremere alla fine non tagliato per spingere fuori endosperma e endosperma separato da tegumento (Kinoshita et al., 2004). Mettere l'embrione e endosperma in microtubi separati in azoto liquido. Continuare fino a quando questo si accumulano embrione o endosperma 10-15 silique e quindi memorizzare il tubo in un congelatore C ° -80. In alcuni casi, dalla mandorla farinosa non deve essere separato da tegumento, cioè, si può isolare l'RNA totale da mandorla farinosa e del cappotto miscela di semi per l'analisi del gene imprinting.
III. Bisolfito Sequencing
1. Reagenti necessari
Cetyltrimethyl ammonio bromuro (CTAB) per la preparazione di DNA genomico, enzimi di restrizione, 3 M di NaOH (appena fatto), 6,42 M urea / 4 bisolfito di sodio M (2 metabisolfito di sodio M, Sigma-Aldrich, S9000, Na2S2O5, peso molecolare: 190), 10 mM idrochinone, un kit di purificazione del DNA (Promega Wizard DNA Clean-up di sistema, cat. # A7280), TE buffer, 6,3 M di NaOH (appena fatto), 10 M NH 4 OAc, 20 mg / tRNA microlitri, e il 100% di etanolo .
2. I dettagli del protocollo di trattamento bisolfito
- Isolare endosperma del DNA genomico o embrione utilizzando una procedura CTAB (Rogers e Bendich, 1988).
- Digest 100 ng - 2 mg di DNA genomico in 20 l per 100 l di volume totale con enzimi di restrizione che tagliano fuori della regione da analizzare. Per il promotore MEA, usiamo XhoI, NdeI e PstI o HindIII.
- Denaturare gli enzimi di restrizione del DNA facendo bollire per cinque minuti e poi spegnete il ghiaccio.
- Aggiunta del volume 09/01 (2,2 microlitri per 20 microlitri di DNA digerito) di 3 milioni di NaOH e incubare a 37 ° C per 15 minuti.
- Trasferire la soluzione in una provetta da 250 microlitri PCR.
- Sciogliere 7,5 g di urea in 10 ml di acqua distillata sterile; lentamente aggiungere 7,6 g di metabisolfito di sodio nell'arco di 1-2 ore e il riscaldamento di solito aiuta la dissoluzione; Regolare il pH a 5 con appena fatto 10 M NaOH; Aggiungere acqua distillata sterile ad una finale volume a 20 mL. Si tratta di 6,24 M urea / 4 M bisolfito di sodio.
- Aggiungi 6,24 M urea / 4 M bisolfito di sodio ad una concentrazione finale di 5,36 M e 3,44 M, rispettivamente (Paulin et al., 1998). Ad esempio, aggiungere 208 ml di 6,42 M urea / 4 M bisolfito di sodio al precedente 22,2 ml di DNA genomico denaturato (Xiao et al., 2003).
- Aggiungere 10 mM idrochinone al DNA ad una concentrazione finale di 0,5 mm (12 microlitri per 20 digestione mL).
- Condotta trattamento bisolfito in una macchina di PCR: 30 cicli di 55 ° C per 15 minuti e 95 ° C per 30 secondi.
- Desalificare il DNA bisolfito trattato con il DNA guidata Clean-Up del sistema da Promega e seguire il protocollo (Jacobsen et al., 2000).
- Misurare il volume esatto di TE recuperato dalla colonna dopo desalinizzazione e aggiungere 6,3 M di NaOH ad una concentrazione finale di 0,3 M. Incubare a 37 ° C per 15 minuti.
- Aggiungere 10 M NH 4 OAc (pH 7,0) ad una concentrazione finale di 3 milioni, 2 ml di 20 mg / tRNA microlitri, e 3 volumi di etanolo 100% e poi mescolare. Centrifugare per 15 minuti a 14.000 giri al minuto.
- Lavare il pellet una volta con etanolo al 70%, fare una centrifuga breve, ed eliminare l'etanolo extra.
- Secco pellet in un speedvac per 5-10 minuti e risospendere in 25-100 microlitri di buffer TE seconda partenza quantità di DNA. Il bisolfito di sodio-trattati DNA è ora pronto per l'analisi PCR.
3. Amplificazione PCR
- Dal momento che non metilato cytosines vengono convertiti in uracile, è difficile per amplificare un frammento di grandi dimensioni utilizzando il bisolfito trattati con il DNA come un modello. Così, di solito disegno primer per amplificare un prodotto non più di 500 bp. Per la sequenza di 4 KB promotore MEA, abbiamo progettato molti set di primer e amplificati 14 frammenti sovrapposti per coprire l'intera regione (Xiao et al., 2003).
- Per la sequenza in alto a filo, nella progettazione di un primer forward, I) scegliere un (G guanina)-regione ricca in modo da avere una temperatura superiore di ricottura senza più nucleotidi nella primer; II) cambiare C (citosina) a Y ( pirimidina) in CG e contesti metano e cambiare il rimanente C a T (timina). Nella progettazione di un primer reverse, I) scegliere una regione ricca di C; II) cambiare G R (purine) in CG e contesti metano e cambiare il G rimanenti A (adenina).
- Per la sequenza in basso a filo, nella progettazione di un primer forward, I), scegliere una regione ricca di C; II) cambiare G a R a CG e contesti metano e cambiare il G rimanenti ad A. Nella progettazione di un primer reverse, I) scegliere un G-regione ricca; II) C di variazione di Y (pirimidina) in CG e contesti metano e cambiare il rimanente C a T.
- Usiamo solitamente 1-2 ml di DNA bisolfito di sodio, trattato come un modello per ogni amplificazione PCR (Xiao et al., 2003). Il prodotto di PCR deve essere analizzato mediante elettroforesi su gel per confermare la corretta dimensione del frammento, poi da gel purificato e clonato nel vettore di clonaggio TOPO TA pCR2.1 (Invitrogen) come inserto. Una singola colonia viene prelevato e colto; DNA plasmidico estratto e inviato per il sequenziamento.
4. Analisi di sequenze
Il principio di sequenziamento bisolfito è che citosina non metilato verrà convertito in uracile a causa di deaminazione idrolitici da elevata concentrazione di bisolfito di sodio a pH5.0 che sarà amplificato come timina nel prodotto di PCR, mentre 5-metil citosina non verrà modificato da bisolfito di sodio e restano da citosina dopo amplificazione PCR (Clark et al, 1994;. Frommer et al, 1992).. Dopo aver ottenuto il risultato di sequenziamento, la confrontiamo con il filone specifico modello utilizzato per l'amplificazione PCR. Se un residuo citosina nel modello si legge come una timina nel risultato di sequenziamento, indica che la citosina non è metilato. Se un residuo citosina nel modello rimane una citosina in sequenza, significa che la citosina è metilato.
IV. Rappresentante Risultati
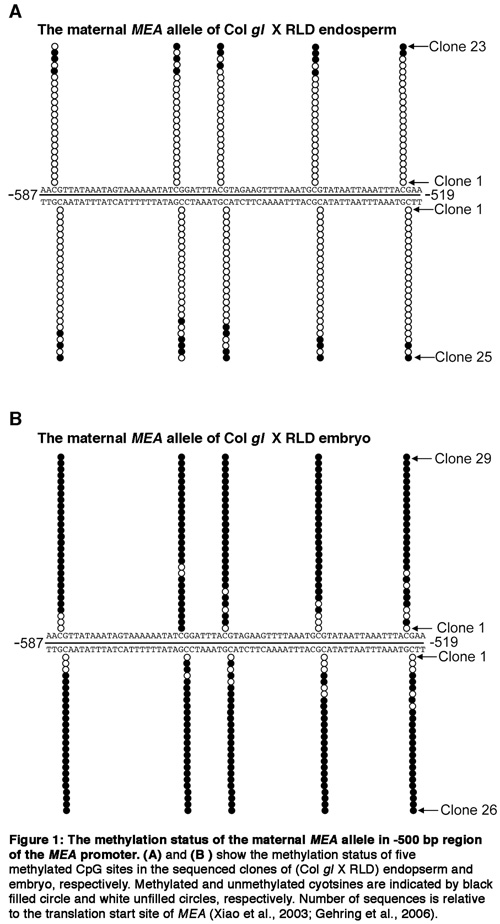 Figura 1.
Figura 1.
Discussione
E 'relativamente facile separare embrione da endosperma e tegumento, ma è noioso per separare endosperma da tegumento, soprattutto per le sementi in fase iniziale o medio-siluro di embriogenesi. Dal cappotto di seme contribuisce solo una piccola quantità di tessuto, per alcuni geni, ad esempio, MEA e FWA, non abbiamo a separare endosperma da tegumento. Questo significa che siamo in grado di isolare RNA da una miscela di endosperma e tessuti tegumento, controllare l'espressione degli alleli ma...
Divulgazioni
Riconoscimenti
Gli autori ringraziano la signora Jennifer M. Lommel e Tara N. Rognan per la manutenzione degli impianti di Arabidopsis. Questo lavoro è stato sostenuto da fondi di start-up da Saint Louis University e il National Institutes di borse di Salute 1R15GM086846-01 e 3R15GM086846-01S1 a W. Xiao.
Materiali
Forniture
- Microscopio da dissezione
- Forbici
- Pinza punta fine
- Gioielli Tag
- Impianto Stakes
- Stringa o Twist-Ties
- 4 "X 2" X "in polietilene 8 Borse
- 3 "X 1" X preparati microscopici 1,0 millimetri
- Azoto liquido
- I contenitori di azoto liquido
- Blocco di calore
- Tubi di PCR
- Termociclatore
- Microcentrifuga Tubi
- Microcentrifuga
- Gel elettroforesi impianto
- Arabidopsis thaliana Columbia-0 Piante
- Arabidopsis thaliana Landsberg erecta Piante
Reagenti
- 70% di etanolo
- 95% di etanolo
- 100% di etanolo
- 0,3 M Sorbitolo e 5 mM MES-pH 5,7
- Cetyltrimethyl Bromuro di ammonio (CTAB)
- 100% di etanolo
- Cholorform
- Gli enzimi di restrizione
- 3 M NaOH
- 6,3 M di NaOH
- 6,24 M urea / 4 bisolfito di sodio M
- Distillata sterile H 2 O
- 10 mM Idrochinone
- Wizard DNA Clean-Up System (Promega)
- 10 M NH 4 OAc
- 20 mg / tRNA microlitri
- TE buffer di
- Il TOPO TA Cloning Kit (Invitrogen)
Riferimenti
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Cokus, S. J., Feng, S., Zhang, X., Chen, Z., Merriman, B., Haudenschild, C. D., Sriharsa Pradhan, S., Nelson, S. F., Pellegrini, M., Jacobsen, S. E. Shotgun bisulphite sequencing of the Arabidopsis genome reveals DNA methylation patterning. Nature. 452, 215-219 (2008).
- Frommer, M., McDonald, L. E., Millar, D. S., Collis, C. M., Watt, F., Grigg, G. W. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc Natl Acad Sci USA. 89, 1827-1831 (1992).
- Gehring, M., Huh, J. H., Hsieh, T. F., Penterman, J., Choi, Y., Harada, J. J., Goldberg, R. B., Fischer, R. L. D. E. M. E. T. E. R. DNA glycosylase establishes MEDEA polycomb gene self-imprinting by allele-specific demethylation. Cell. 124, 495-506 (2006).
- Henderson, I. R., Chan, S. R., Cao, X., Johnson, L., Jacobsen, S. E. Accurate sodium bisulfite sequencing in plants. Epigenetics. 5, 47-49 (2010).
- Hsieh, T. F., Ibarra, C. A., Silva, P., Zemach, A., Eshed-Williams, L., Fischer, R. L., Zilberman, D. Genome-wide demethylation of Arabidopsis endosperm. Science. 324, 1451-1454 (2009).
- Jacobsen, S. E., Sakai, H., Finnegan, E. J., Cao, X., Meyerowitz, E. M. Ectopic hypermethylation of flower-specific genes in Arabidopsis. Curr. Biol. 10, 179-186 (2000).
- Kinoshita, T., Miura, A., Choi, Y., Kinoshita, Y., Cao, X., Jacobsen, S. E., Fischer, R. L., Kakutani, T. One-way control of FWA imprinting in Arabidopsis endosperm by DNA methylation. Science. 303, 521-523 (2004).
- Lister, R., O'Malley, R. C., Tonti-Filippini, J., Gregory, B. D., Berry, C. C., Millar, A. H., Ecker, J. R. Highly integrated single-base resolution maps of the epigenome in Arabidopsis. Cell. 133, 523-536 (2008).
- Lister, R., Pelizzola, M., Dowen, R. H., Hawkins, R. D., Hon, G., Tonti-Filippini, J., Nery, J. R., Lee, L., Ye, Z., Ngo, Q. -. M. Human DNA methylomes at base resolution show widespread epigenomic differences. Nature. 462, 315-322 (2009).
- Paulin, R., Grigg, G. W., Davey, M. W., Piper, A. A. Urea improves efficiency of bisulfite-mediated sequencing of 5'- methylcytosine in genomic DNA. Nucl. Acids Res. 26, 5009-5010 (1998).
- Rogers, S. O., &, B. e. n. d. i. c. h., J, A. Extraction of DNA from plant tissues. Plant Molecular Biology Manual. A6, 1-10 (1988).
- Smyth, D. R., Bowman, J. L., Elliot, M., Meyerowitz, E. M. Early Flower Development in Arabídopsis. Plant Cell. 2, 755-767 (1990).
- Xiao, W., Gehring, M., Choi, Y., Margossian, L., Pu, H., Harada, J. J., Goldberg, R. B., Pennell, R. I., Fischer, R. L. Imprinting of the MEA Polycomb gene is controlled by antagonism between MET1 methyltransferase and DME glycosylase. Dev. Cell. 5, 891-901 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon