Method Article
שיטה לבידוד וזיהוי mRNAs, רנ"א ורכיבי חלבון של מכלולי Ribonucleoprotein מתמציות סלולריות באמצעות שבב RIP
In This Article
Summary
צעד אחר צעד לפרוטוקול בידוד וזיהוי RNA מתחמים הקשורים באמצעות RIP שבב.
Abstract
כתוצאה מההתפתחות של רצף תפוקה גבוהה וניתוח microarray יעיל, ניתוח ביטוי גנים העולמי הפך צורה קלה וזמינה של איסוף נתונים. במחקר רב ומודלים למחלות עם זאת, רמות המצב יציבות של mRNA גן היעד לא תמיד לתאם ישירות עם רמות חלבון המצב יציבות. רגולציה של גנים לאחר השעתוק היא הסבר סביר של הסטייה בין שתיים. מונע על ידי הקשירה של חלבונים מחייבים RNA (RBP), רגולציה שלאחר השעתוק משפיעה לוקליזציה-mRNA, יציבות ותרגום על ידי יצירת Ribonucleoprotein (RNP) מורכבת עם mRNAs היעד. זיהוי מטרות de mRNA נובו הידועות אלה מתמציות סלולריות במתחם RNP הוא מרכזי להבנת מנגנונים ופונקציות של RBP וההשפעה שלהם על תוצאת פלט חלבון. פרוטוקול זה מתאר שיטה כינתה RNP immunoprecipitation-microarray (RIP-Chip), המאפשרת לזיהוי שלmRNAs pecific קשור במתחם ribonucleoprotein, תחת תנאי ניסוי משתנה, יחד עם אפשרויות לייעול נוסף לניסוי לחוקר הבודד. בכלי ניסיוני חשוב זה, חוקרים יכולים לחקור את המנגנונים המורכבים הקשורים לרגולציה של גנים שלאחר שעתוק, כמו גם אינטראקציות ribonucleoprotein אחרות.
Protocol
הכנת ניסוי
לפני תחילת ניסוי, זה קריטי שיש את כל מגיבים, מכולות וכלי RNase חינם. פנק את הזכוכית עם RNase מעכב (RNaseZAP, Ambion) ואחרי השטיפה במים DEPC טופל. ודא שכל המגיבים הם אשרו כRNase חינם.
1. הכן mRNP lysate
- לגדול ולקצור תאי רקמת גידול אקספוננציאלי לייצר בין 2-5 מ"ג של חלבון הכולל לכל RIP.
- שתי מנות התרבות P150 מספיקות בדרך כלל.
- עבור כל RBP נחקר, כמויות חלבון ומספר סלולריים כלל חייבות להיות מותאמות למקסם mRNA היעד המתאים ואינטראקציות RBP.
- RIP לניתוח qRT-PCR עשוי לדרוש פחות lysate הסלולרי (כ -400 מיקרוגרם חלבון כולל) בשל ההגברה שלו ושיטת זיהוי במיוחד עבור RBPs שפע גבוה כגון חור, AUF1, TIAR.
- תאים גלולים באמצעות צנטריפוגה XG ב 200 במשך 8-10 דקות ב 4 ° C, אז לשטוף שלוש פעמים עם הקרח הקר PBS.
- לאחר שטיפה סופית, בעדינות להתיז את התחתית של תחתית ותא הגלול resuspend בנפחים שווים של חיץ מוכן polysome תמוגה (PLB) עם מעכבי פרוטאז RNase ו.
- מומלץ למדוד את ההיקף המדויק של תא גלול.
- בעדינות תערובת pipet (לא מערבולת) להתפרק גושים של תאים ודגירת lysate על קרח למשך 5 דקות.
- צנטריפוגה ב XG 13000 עבור 20 דקות ב 4 ° C כדי לנקות את lysate של פסולת.
- מייד להעביר פינה supernatant לצינור microfuge מראש מצונן.
- שמור על קרח ולאחסן ב -80 ° C.
- הקפאה מיידית משלימה תמוגה התקינה של התאים ומונעת מחייבים לא רצוי. המשך עם הפשרת מדגם מייד לאחר RIP ולשמור דגימות על קרח, כדי למנוע הידרדרות רנ"א.
- lysate זה ניתן לאחסן עד שישה חודשים ב-80 ° C. הימנע ממחזורי הפשרת הקפאה חוזרת ונשנים כמו זה יכול להוביל לחלבון ו / או שפלת mRNA.
- לכמת ריכוז חלבון בlysate באמצעות בדיקת חלבון ברדפורד הסטנדרטי.
2. חלבון מעייל חרוזי Sepharose עם נוגדן ושטפים
- חלבון טרום להתנפח בSepharose (PAS) חרוזי לילה בNT2 חיץ (כרכים 3-4) עם BSA 5%. לאחסן ב 4 ° C.
- אחסון לטווח ארוך של עד כמה חודשים ב 4 ° C אפשרי כאשר בתוספת 0.1% יזידו נתרן.
- חרוזי Sepharose חלבון צריכים להיבחר על בסיס אלוטיפ של נוגדן RBP היעד. חלבונים, G ו / G כל יש מטרות אלוטיפ ספציפיות ומשתנים בזיקת היעד. חרוזי sepharose חלבון שמשו מבוססים על סגולי והזיקה אלוטיפ לנוגדן חלבון חור.
- לפני השימוש, הסר חיץ NT2 עודף, ולכןשהחרוזים אחרונים ליחס חיץ הוא 1:1.
- באמצעות צינורות 1.5 microcentrifuge RNAse ללא מ"ל, להסיר 100 μl של PAS slurry ולהוסיף 30 מיקרוגרם של נוגדנים עבור כל תגובת IP בודדה (כלומר, RBP הספציפי ושליטת אלוטיפ).
- הוסף 100-200 μl של NT2 חיץ לתמהיל חרוז נוגדן.
- תערובת עשויה להיות מאוחסנת במשך מספר שבועות ב 4 ° C, כאשר בתוספת 0.1% יזידו נתרן.
- נוגדן אלוטיפ התאמה או סרום נורמלי לגמרי מאותם מינים יש להשתמש במקביל כשליטת נוגדנים נגד RNA רקע.
- הוסף נוגדן מתאים לתמהיל חרוז ודגירת הלילה, מתגלגל מקצה אל קצה ב 4 ° C.
- לייעל כייל נוגדנים לחלבון מסוים נחקר (1, 5, 10 או 30 מיקרוגרם של נוגדנים הוא בדרך כלל מספיק).
- הכן את חרוזים המצופים בנוגדנים מייד לפני שימוש על ידי שטיפה עם 1 מ"ל שלקר כקרח NT2 חיץ 5 פעמים.
- שטוף את תמהיל חרוז ידי צנטריפוגה ב13000 XG במשך 1-2 דקות ב 4 ° C.
- מוציאים בזהירות את הסכום המקסימאלי של supernantant עם pipettor יד או aspirator אבל להיות זהיר, כדי למנוע שיבוש גלולה.
- כביסה מסייעת להסיר נוגדן מאוגד, כמו גם מזהמי RNase מתערובת נוגדנים.
- לאחר השטיפה הסופית הושלמה, resuspend את החרוזים ב700 μl של חיץ קר כקרח NT2 אחרי טיפול במעכבי RNase שונים כדי להגן על mRNAs היעד, לרבות 10 μl של RNase אאוט ב 40 U / μl, 10 μl של 100 מ"מ DTT ו15 EDTA μl (15 מ"מ). הבא נפח עד 1,000 μl עם NT2 חיץ.
- מתחמי ribonucleoside Vanadyl אינם בשימוש בשל השפעה מעכבת של EDTA.
3. Immunoprecipitation ורנ"א רטיבות
- שלב פרקליר אופציונלי:
- כדי להפחית את הרקע, חרוזים ונוגדני שליטה עשויים לשמש לlysate מראש ברור. זה עשוי להפחית את האות בפלט.
- צעד זה עשוי להיות נחוץ כדי לצמצם רקע כאשר עושה IP אחרי microarray. זה בדרך כלל לא הכרחי לIP אחרי qRT-PCR.
- פרקליר עם 15 מיקרוגרם של שליטת אלוטיפ למשך 30 דקות / 4 ° C הסוף נופל על הסוף.
- הוסף 50 μl של חרוזי preswollen PAS הלא מצופים עם נוגדן מצעד 2.1.
- דגירת 30 דקות / 4 ° C עם תום סיבוב על קצה
- צנטריפוגה ב -10,000 XG ב 4 ° C. שמור supernatant עבור ה-IP.
- הוסף 100 μl של lysate פינה המבודד (כ 2-5 מ"ג) לתמהיל נוגדנים מוכנים.
- דילול lysate יעזור להפחית רקע ומחייב לא ספציפי.
- כמות קלט lysate עשויה להשתנות בהתאם לשיטת זיהוי ושפע של RBP oיעילות של נוגדן r RBP כאמורה בצעד 1.1.c.
- (אופציונלי) מייד לערבב צינור מצליף בעדינות על ידי מספר פעמים ואחריו צנטריפוגה הקצרה ב -10,000 XG ב 4 ° C כדי חרוזי גלולה ולהסיר 100 μl של supernatant כייצוג mRNA קלט הכולל לניתוח qPCR באמצעות טכניקות בידוד RNA רגילות באופן מיידי.
- צעד זה נועד לאשר שקלט lysate RNA הוא אופטימלי עבור ה-IP וצריך להתבצע רק כצעד המחאה או כצעד הבא לפתרון בעיות RIP עם תוצאות עניות RNA.
- צינור גלישה בparafilm כדי להבטיח אטימה הדוקה ולדגור על 4 מעלות צלזיוס למשך 2-4 שעות, מתגלגל מקצה אל קצה.
- עיתוי בדגירה צריך להיות מותאם המבוסס על שפע והיעד צריך להיות ממוזער כדי למנוע התארגנות מחודשת או שפלה מורכבת. לRBPs כמה incubations קצר יותר עשויה להיות יותר אופטימלי.
- חרוזים גלולים ב5000 xgfאו 5 דקות ב 4 ° C ולשמור supernatant לניתוח פוטנציאל על ידי כתם מערבי. aliquots בחנויות ב-80 ° C.
- Aliquots של supernatant עם כמויות גבוהות של חלבון המטרה השיורי עשוי להצביע על כישלון של החלבון שזרז ידי חרוזי sepharose.
- כפי שתואר לעיל, לשטוף חרוזים 5 פעמים עם 1 מ"ל של חיץ וצנטריפוגה NT2 קר כקרח (5000 XG למשך 5 דקות ב 4 ° C) ולאחר מכן להסיר supernatant עם pipettor יד או aspirator.
- שיטות שטיפה יותר מחמירות ניתן להשתמש כדי להפחית את הרקע על ידי השלמת NT2 חיץ עם deoxycholate נתרן, אוריאה או SDS.
- שמור דגימות על קרח ככל האפשר ולעבוד במהירות כדי למזער את ההשפלה של mRNA היעד.
- אופציונלי: שמור aliquot קטן (10%) של תמהיל חרוז או להפעיל מדגם נוסף במקביל לבדיקת יעילות ה-IP של חלבון המטרה באמצעות ניתוח כתם מערבי.
4. טיפולי proteinase K DNase ו
- לאחר שטיפה, חרוזי resuspend עם 100 μl של NT2 חיץ בתוספת 5 μl של RNase-1 חינם DNase (2 U / μl).
- שמור על 37 מעלות צלזיוס במשך דקות 5-10. הוסף 1 מ"ל של חיץ וNT2 סרכזת ב5000 XG ל1 דקות בטמפרטורת חדר.
- קטן aliquot (~ 10%) של חרוזים ניתן להשתמש כדי לבדוק את יעילות ה-IP על ידי ניתוח SDS-PAGE.
- Resuspend PAS הגלולה בNT2 חיץ 100 μl, 5 μl proteinase K (10 מ"ג / מ"ל), וμl 10% SDS 1.
- דגירה את תערובת חרוז resuspended למשך 30 דקות ב 55 מעלות צלזיוס באמבט מים, בעדינות מצליפה בכל 10 דקות.
- Proteinase K יסייע בשחרורו של מרכיבי RNP.
- חרוזים גלולים ב5000 XG למשך 5 דקות בטמפרטורת חדר (RT) ולאסוף את supernatant (~ 100 μl).
- הוסף 200 NT2 חיץ μl לחרוזים, centri5000 XG fuge עבור 2 דקות בRT, לאסוף supernatant (~ 200 μl), וחרוזים שנזרקו.
- שלב supernatants (100 ו 200 μl μl) ולהוסיף שכבה 300 μl תחתונה של 3 חומצות פנול CHCl.
- מערבולת, 1 דקות RT (או 37 מעלות צלזיוס בייקר) וצנטריפוגה XG ב 16000 בRT לדקות 1.
- לאסוף 250 μl של שכבה העליונה (אל ממשק לשבש), להוסיף נתרן יצטט 25 μl ב-pH 5.2, 625 μl 100% ETOH וglycoblue 5 μl ומערבב היטב.
- אחסן לילה ב -20 ° C.
- כדי להבטיח התאוששות נאותה של רנ"א, התוספת של glycoblue תגדיל את הנראות של רנ"א הגלולה על ידי מתנהג כמו מולקולה מובילה.
- למחרת, צינורות לערבב על ידי היפוך ספין, 3-5 פעמים ב12000 XG ב 4 מעלות צלזיוס למשך 30 דקות וזורקים supernatant.
- הוסף 1 מ"ל של 70% לETOH הכחול הגלולה והתמהיל ידי היפוך או vortexing.
- צנטריפוגה XG 12000 ב 4 מעלות צלזיוס למשך 2 דקות.
- Discard supernatant וסרכזת 12000 XG ל1 דקות ב 4 ° C.
- הסר את כל ETOH 70% שיירים עם פיפטה ואוויר יבש בגלולת RT למשך 5 דקות.
- Resuspend בμl 20-40 מי RNase / DNase חינם או אחר נפח המתאים לפי צורך.
- לדוגמה עכשיו הוא מוכן ליישומים במורד זרם נוספים כגון qRT-PCR או microarray.
- ספקטרופוטומטריה Nanodrop ניתן להשתמש בהערכת ריכוז מדגם, אולם עם דגימות יקרות, זה עשוי להיות מועיל כדי להימנע מדגימות nanodropping כפי שהוא יכול להיות בזבזני ויחסית לא מדויק. אנו מציעים דגימות נרמול לתעתיקי גני המשק להעריך טוהר ויעילות ה-IP לדגימות תשואה יקרות או נמוכות.
5. נציג תוצאות
אם ההליך הוא מותאם ומבוצע כהלכה, immunoprecipitation אמור להניב העשרה משמעותית של מטרות mRNA. בדרך כלל, בהתאם ליעד וRBP mRNA שלו (הים), אנו רואים את ההעשרה של כ 10 - ל 50-קיפול כאשר העריכו ידי qRT-PCR. מטרות רבות של RBPs ניתן לגלות בהמוניהם באמצעות ניתוח microarray. עם זאת, שיטה זו היא רגישה יותר להידרדרות בהשוואה לqRT-PCR. בהתאם RBP, מספר היעדים והיעילים של התגובה, microarray יכול לחשוף מאה יעדים חדשים, או שהוא עשוי לחשוף רק מעט, אם בכלל. לדוגמה, אחד מהמאופיין טוב יותר RNA מחייב חלבוני החור, לאחר התעתיק מסדיר את הביטוי והתרגום של גנים רבים חשובים פיסיולוגיים 1, 2. בידוד של-מורכב החור ribonucleo דרך RIP שבב בשורות תאי סרטן השד, למשל, חשף את ההעשרה של מספר מטרות חשובות ידועה חור, כוללים β-תקטין באמצעות qRT-PCR כפי שמוצג באיור 1. בשתי שורות תאי סרטן β-תקטין מועשר 12 - ל15-קיפול. בדרך כלל, כאשר מבוצעים כראוי אנו רואים Enr משמעותיichment של β-תקטין במגוון רחב של שורות תאים. עם זאת, אם RIP אינו חושף שום העשרה משמעותית עבור β-תקטין זה מצביע על בעיה בRIP וההליך עשוי צורך לחזור. יתר על כן, ניתוח microarray של דגימות immunoprecipitated משורות תאים אלה גילו תת ביטוי ברור של מטרות חור בקולטן אסטרוגן השונה (ER) תאים MCF-7 סרטניים חיוביים לעומת MB-231 קווי ER שליליים סרטן שד תאים כפי שמודגמים באיור 2 1. מטרות אלה נופלות למספר קטגוריות: יעדי חור ידועים ובלתי ידועים, שהיו קשור או שאינה קשור לסרטן. לדוגמה, CALM2 וCD9 שניהם גנים סרטניים שלא אותרו בעבר כיעדי חור. שימוש microarray ומאשר עם qRT-PCR, CALM2 וCD9 נמצאו 5 - ל180-לקפל מועשר בגלולת החור המציין אינטראקציה בולטת בין חלבון החור וגני המטרה הבאה.
איור 1. Immunoprecipitation וRIP בMB-231 (ER-) וMCF-7 (ER +) תאי סרטן השד. Immunoprecipitations בוצעו מMB-231-7 או MCF תא lysates באמצעות נוגדן אנטי חור חד שבטי (3A2) ושליטת אלוטיפ IgG1. א IP המערבי של חור חשף להקת גודל צפוי זוהה על ידי 3A2. לוח בצד ימין מגלה כמויות של חור בקלט lysates שימוש משתי שורות תאים, עם β-טובולין כבקרת טעינה. אימות ב 'על ידי כמותית RT-PCR והראתה 15-11-enrichments קיפול של β-תקטין, יעד חור ידוע, בשב"ס 3A2 מMB-231 וMCF-7, בהתאמה. כל ערכי ΔΔCT היו מנורמלים GAPDH. ניסויים נעשו בשני עותקים (n = 2).
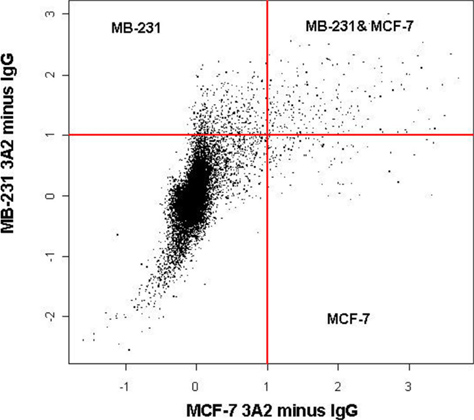
איור 2. החור RIP השבב מזהה פרופילים גנטיים שונים בחדר מיון + ותאי סרטן שד ER-.immunoprecipitations החור בוצע מMB-231-7 או MCF תא lysates באמצעות נוגדן חור ושליטת אלוטיפ IgG1 הכלאת מערכי Sentrix Illumina (47,000 גנים). אותות בקרה נגרעו. תוצאות מייצגות נתונים מצטברים מ12 מערכים שונים. ניסויים נעשו בשלושה עותקים (n = 3) עבור כל שורת תאים עם בקרות מתאימות. קשקשים הם log2.
Discussion
בשל האופי של ניסוי, אופטימיזציה וניסיון זה יהיו הדרכים מובטחות רק כדי לרכוש את התוצאות המצופות בהצלחה. בשלבים רבים של הליך זה, טמפרטורה וטיפול יעיל של חומרים הכימיים ומוצרים הם חשובים וקריטיים. תכנון וביצוע של טכניקה נכון יעזרו להבטיח כי הניסוי בוצע במסגרת זמן מתאימה בטמפרטורה האופטימלית המומלצת. נושא מרכזי בניסויי בידוד RNA הוא הרגישות של RNAs לשפלה ידי RNases. כל ריאגנטים צריכים להיות RNase חופשי ומאוחסן או משמש במכולות RNase חינם. זה הוא שלב קריטי בהבטחת השלמות של מדגם mRNA שלך. גם כאשר הניסוי מבוצע כראוי, עם זאת, התוצאה הרצויה לא יכולה להיות מושגת בשל האופי של האינטראקציה בין RBP וmRNAs היעד שלה.
בעיה פוטנציאלית אחת היא שיש נמוכה או אפילו אין אות מרנ"א מבודד על ידי RIP שבב.למרות שעשוי להיות אות מ-RNA המוחלט, זה עשוי להיות תוצאה של חלבון מחייב לקוי שנגרר על ידי החרוזים. הצעד הראשון הוא פתרון הבעיות כדי לאשר שlysate הסלולרי בשימוש יש ביטוי נאות RBP הספציפי. עם האישור, חלבון יכול להיות מבודד לאחר NT2 השטיפה הסופית וresuspended במאגר Laemmli או אחר חיץ denaturing מתאים ומחומם על 95 מעלות צלזיוס למשך 5 דקות. ניתוח הכתם מערבי ניתן להשתמש בדגימות אלה בתיאום עם lysate קלט כמו גם בקרות שליליות כדי להבטיח מספיק למשוך למטה של חלבון קשור.
יתר על כן, בגלל lysing התא נדרש כדי לגשת לרכיבים אלה, הפוטנציאל לאינטראקציות בלתי תקינות ובלתי רצויות בין חלבונים וmRNA בדרך מופרדים עשוי להיות מוצג. אינטראקציות אלה עלולות להיקשר ו" לספוג "mRNAs היעד שלך או החלבונים מחייבים דרך אינטראקציות לא ספציפיות. בנוסף, חלבונים אלו בשיתוף שונהnditions יכול לקפל בוריאציות שונות, והמוטיבים המחייבים שלהם עשויים להיות נגישים לmRNAs היעד שלהם, ומונעים האינטראקציות ביניהם. שני אלה מחזקים את החשיבות של עבודה ביעילות, כמו גם ניצול האופטימלי הטמפרטורות מפורטים להגביל אינטראקציות בלתי רצויות אלה. בנוסף, אופטימיזציה של שטיפת תנאים לכל חלבון המטרה ספציפי תהיה קריטית למקסם את הטוהר של האינטראקציה. תנאים מחמירים יותר להדחה עשויים להיות נחוצים. לדוגמה, החיץ לשטוף ניתן להשלים עם SDS או כמות מתאימה של אוריאה להפחית אינטראקציות ורקע לא ספציפיות באות הפלט שלך. זה יהיה תלוי לחלוטין בRBP היעד של הנסיין, כמו גם את ה-mRNA היעד בתנאים הפיסיולוגיים הייחודיים שלהם. תנאים מסוימים לא יהיו מתאימים לכלים מסוימים mRNA ניתוח, שיצוינו בהכנת דגימות.
לבסוף, למרות RIP הוא מוצלח בהעשרה של רנ"אאינטראקציות RBP, בעיה מוכרת היטב עם שיטת RIP (שבב) היא חוסר היכולת לזהות את התחומים המחייבים הספציפיים של RBP על יעדי mRNA החולפים. כמה טכניקות צלב מקשר-יכולים לשמש אחרי RIP לבודד יעדי רצף ייחודיים, עם זאת, השימוש בקרינת UV בגלים קצרים נוטה להוביל לניזק חומצות גרעין. שיטה חדשה המכונית PAR-CLIP, או crosslinking ribonucleoside photoactivatable וimmuoprecipitation, מעסיקה ארוך UV-גל לשלב thiouridine לרנ"א המתהווה המאפשר זיהוי של אתרי קישור ייחודיים מאינטראקציות RNA הוא יציבות וחולפות.
בסך הכל, שבב RIP הוקם ככלי מצוין המשמש לבודד ולבחון את יחסי גומלין בין חלבונים המחייבים RNA ויעדי mRNA שלהם על ידי הקבוצה שלנו, כמו גם רבות קבוצות מחקר אחרות. למרות רגיש בטבע ובפועל, ביצוע נכון של הליך זה יניב את הבידוד של קומפלקסי RNP אלה, שעד לא מזמן, היה inaccessible לגילוי וניתוח.
Disclosures
החוקרים מצהירים כי אין להם אינטרסים מתחרים.
Acknowledgements
משרד ההגנה (פרס הרעיון W81XWH-07-0406) - כדי Ulus אטסוי
NIH RO1 A1080870 - כדי Ulus אטסוי
NIH R21 A1079341 - כדי Ulus אטסוי
אוניברסיטת מיזורי קרנות מוסדיות - כדי Ulus אטסוי
Materials
| Name | Company | Catalog Number | Comments |
| שם מגיב | חברה | מספר קטלוגים | תגובות |
| 1 ז dithiothreitol | דיג | BP172-5 | DTT |
| 1 DNase | Ambion | 2235 | RNase חופשי |
| Ethylendiamine חומצת Tetraccetic | דיג | BP118-500 | EDTA |
| גליקוגן | Ambion | 9516 | |
| 1 HEPES | סיגמא | H3375-100G | pH 7.0 |
| Igepal Nonidet P-40 | USB | 78641 | NP40 |
| 1 ז KCl | דיג | BP366-500 | |
| 1 ז MgCl 2 | דיג | BP214-500 | |
| NT2 מאגר | * ראה להלן | ||
| מאגר תמוגה Polysome | * ראה להלן | ||
| טבליות קוקטייל מעכב פרוטאז | רוש | 11873580001 | |
| חלבון חרוזי Sepharose | סיגמא | P3391 | |
| Proteinase K | דיג | BP1700-100 | |
| RNase אאוט RNase המעכב | Invitrogen | 10777-019 | 40 U / μl |
| 1 ז NaCl | דיג | BP358-212 | |
| סולפט נתרן dodecyl | דיג | BP166-500 | SDS |
| 1 ז טריס-HCl | דיג | BP153-500 | pH 7.4 |
| Trizol | Invitrogen | 15596-026 | |
| מכלולי Ribonucleoside Vanadyl | מעבדות ניו אינגלנד | S1402S | VRC |
| בירור מגיב |
| הכן ריאגנטים בRNase / DNase חינם, כלי הזכוכית DEPC טופלה |
| מאגר תמוגה Polysome |
| 100 המ"מימ KCl |
| 5 המ"מ MgCl 2 |
| 10 HEPES מ"מ (pH 7.0) |
| 0.5% NP40 |
| 1 המ"מ DTT |
| 100 יחידות / מיליליטר RNase אאוט |
| 400 מיקרומטר VRC |
| לוח קוקטייל מעכב פרוטאז |
| 5 מ"ל של חיץ תמוגה Polysome |
| הוסף 50 μl של 1 HEPES M (pH 7.0) |
| 500 μl של ז KCL 1 |
| 25 μl של ז MgCl 1 2 |
| 25 μl של NP40 |
| 4.7 המ"ל RNase-DNase ללא H 2 O |
| 50 μl של M-DTT 1 |
| 12.5 μl של 100 U / המ"ל RNase אאוט |
| קוקטייל 200 μl פרוטאז מעכב (מומס על פי יצרן) |
| 10 μl 200 המ"מ VRC (בזמן שימוש) |
| NT2 מאגר |
| 50 mM טריס-HCl (pH 7.4) |
| 150 המ"מ NaCl |
| 1 המ"מ MgCl 2 |
| 0.05% NP40 |
| 1 ליטר של NT2 מאגר |
| 50 המ"ל טריס (pH 7.4) |
| 30 מ"ל 5 M NaCl |
| 1 מ"ל 1 ז MgCl 2 |
| 500 NP40 μl |
| 820 המ"ל RNase-DNase ללא H 2 O |
References
- Calaluce, R., Gubin, M. M., Davis, J. W., Magee, J. D., Chen, J., Kuwano, Y., Gorospe, M., Atasoy, U. The RNA binding protein HuR differentially regulates unique subsets of mRNAs in estrogen receptor negative and estrogen receptor positive breast cancer. BMC Cancer. 10, 126-140 (2010).
- Gubin, M. M., Calaluce, R., Davis, J. W., Magee, J. D., Strouse, C. S., Shaw, D. P., Ma, L., Brown, A., Hoffman, T., Rold, T. L., Ulus Atasoy, U. Overexpression of the RNA binding protein HuR impairs tumor growth in triple negative breast cancer associated with deficient angiogenesis. Cell Cycle. 9, 3337-3346 (2010).
- Keene, J. D., Komisarow, J. M., Friedersdorf, M. B. RIP-Chip: the isolation and identification of mRNAs, microRNAs and protein components of ribonucleoprotein complexes from cell extracts. Nature Protocols. 1, 302-307 (2006).
- Tenenbaum, S. A., Lager, P. J., Carson, C. C., Keene, J. D. Ribonomics: identifying mRNA subsets in mRNP complexes using antibodies to RNA-binding proteins and genomic arrays. Methods. 26, 191-198 (2002).
- Tenenbaum, S. A., Carson, C. C., Atasoy, U., Keene, J. D. Genome-wide regulatory analysis using en masse nuclear run-ons and ribonomic profiling with autoimmune sera. Gene. 317, 79-87 (2003).
- Tenenbaum, S. A., Carson, C. C., Lager, P. J., Keene, J. D. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. Proc. Natl. Acad. Sci. U.S.A. 97, 14085-14090 (2000).
- Baroni, T. E., Chittur, S. V., George, A. D., Tennebaum, S. A. Advances in RIP-Chip Analysis. Methods in Molecular Biology. 419, 93-108 (2008).
- de Silanes Lopez, I., Zhan, M., Lal, A., Yang, X., Gorospe, M. Identification of a target RNA motif for RNA-binding protein HuR. Proc. Natl. Acad. Sci. U.S.A. 101, 2987-2992 (2004).
- de Silanes Lopez, I., Lal, A., Gorospe, M. HuR: post-transcriptional paths to malignancy. RNA Biol. 2, 11-13 (2005).
- Grigull, J., Mnaimneh, S., Pootoolal, J., Robinson, M. D., Hughes, T. R. Genome-wide analysis of mRNA stability using transcription inhibitors and microarrays reveals posttranscriptional control of ribosome biogenesis factors. Mol. Cell Biol. 24, 5534-5547 (2004).
- Hieronymus, H., Silver, P. A. Genome-wide analysis of RNA-protein interactions illustrates specificity of the mRNA export machinery. Nat. Genet. 33, 155-161 (2003).
- Hieronymus, H., Yu, M. C., Silver, P. A. Genome-wide mRNA surveillance is coupled to mRNA export. Genes Dev. 18, 2652-2662 (2004).
- Mukherjee, N., Corcoran, D. L., Nusbaum, J. D. Integrative Regulatory Mapping Indicates that the RNA-Binding Protein HuR Couples Pre-mRNA Processing and mRNA Stability. Molecular Cell. 43, 1-13 (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved