Method Article
Verfahren zur Isolierung und Identifizierung von mRNAs, microRNAs und Protein-Komponenten des Ribonucleoprotein Komplexe aus Zellextrakten mit RIP-Chip
In diesem Artikel
Zusammenfassung
Eine Schritt für Schritt-Protokoll zur Isolierung und Identifizierung von RNA assoziiert Komplexe durch RIP-Chip.
Zusammenfassung
Als Folge der Entwicklung von High-Throughput-Sequenzierung und effiziente Mikroarray-Analyse hat die globale Genexpressionsanalyse sich eine einfache und leicht zugängliche Form der Datenerfassung. In vielen Forschungs-und Krankheitsmodelle jedoch nicht steady state Ebenen der Zielgen-mRNA nicht immer direkt mit steady state Protein-Spiegel korrelieren. Post-transkriptionelle Genregulation ist eine wahrscheinliche Erklärung für die Diskrepanz zwischen den beiden. Angetrieben durch die Bindung von RNA-bindende Proteine (RBP), wirkt posttranskriptionale Regulierung mRNA Lokalisierung, Stabilität und Translation durch Bilden einer Ribonucleoprotein (RNP)-Komplex mit Ziel-mRNAs. Identifizierung dieser unbekannten de novo mRNA Targets aus Zellextrakten in der RNP-Komplex schwenkbar ist, um das Verständnis Mechanismen und Funktionen des RBP und deren resultierende Wirkung auf die Protein-Ausgang. Dieses Protokoll beschreibt ein Verfahren genannt RNP Immunpräzipitations-Mikroarray (RIP-Chip), die für die Identifizierung von s ermöglichtpezifische mRNAs in der Ribonukleoproteinkomplex verbunden sind, unter wechselnden experimentellen Bedingungen, zusammen mit Optionen weiter zu optimieren, um ein Experiment für den einzelnen Forscher. Mit diesem wichtigen experimentelles Werkzeug, können die Forscher erkunden die komplizierten Mechanismen mit post-transkriptionellen Genregulation sowie andere ribonucleoprotein Wechselwirkungen verbunden.
Protokoll
Versuchsvorbereitung
Vor Beginn Experiments ist es kritisch, alle Reagenzien, Behältern und Geräten RNase frei zu haben. Saures Glaswaren mit RNase-Inhibitor (RNaseZAP, Ambion) durch Spülen mit DEPC-behandeltem Wasser. Stellen Sie sicher, dass alle Reagenzien wie RNase free bestätigt werden.
Ein. Bereiten mRNP Lysate
- Züchten und zu ernten exponentiell wachsenden Gewebezellen zwischen 2-5 mg Gesamtprotein für jede RIP erzeugen.
- Zwei P150 Kulturschalen sind in der Regel ausreichend.
- Für jede RBP untersucht, müssen zelluläre Gesamt-Anzahl und Proteinmengen optimiert, um entsprechende Ziel-mRNA und RBP Wechselwirkungen zu maximieren.
- RIP für qRT-PCR-Analyse erfordert möglicherweise weniger Zelllysat (ca. 400 pg Gesamtprotein) aufgrund seiner Amplifikation und Detektion Verfahren besonders für hohe Fülle RBPs wie HuR, AUF1, TIAR.
- Pellet-Zellen über Zentrifugation bei 200 xg für 8-10 min bei 4 ° C, dann dreimal mit eiskaltem PBS.
- Nach dem letzten Waschen sanft Fingern an die Unterseite des Rohres und resuspendieren Zellpellet in gleichen Volumina von vorbereiteten Polysomen Lysepuffer (PLB) mit RNase und Proteaseinhibitoren.
- Es wird empfohlen, zu messen, die genaue Volumen des Zellpellets.
- Gently Pipette Mischung (nicht vortexen) auseinander zu brechen Klumpen von Zellen und inkubieren Lysat auf Eis für 5 min.
- Zentrifuge bei 13.000 × g für 20 min bei 4 ° C, um das Lysat von Schutt zu löschen.
- Sofortüberweisung Überstand gelöscht, um vorgekühlten Mikrozentrifugenröhrchen.
- Keep on ice und bei -80 ° C.
- Sofortige Einfrieren rundet das richtige Lyse der Zellen und verhindert unerwünschte Bindung. Fahren Sie mit dem RIP sofort folgende Beispiel Tauwetter und halten Proben auf Eis RNA-Abbau zu vermeiden.
- Das Lysat kann gelagert werden bis zu sechs Monaten bei -80 ° C. Vermeiden wiederholten Gefrier-Auftau-Zyklen, wie dies an Protein und / oder mRNA-Abbau führen kann.
- Quantifizieren Proteinkonzentration im Lysat unter Verwendung von Standard Bradford Protein Assay.
2. Coat Protein A Sepharose Beads mit Antikörper und Wash
- Pre-Protein A-Sepharose Dünung (PAS)-Perlen über Nacht in NT2 Puffer (3-4 Volumina) mit 5% BSA. Lagerung bei 4 ° C.
- Langzeitlagerung von bis zu mehreren Monaten bei 4 ° C ist möglich, wenn mit 0,1% Natriumazid ergänzt.
- Protein Sepharoseperlen sollte basierend auf Isotyp des Antikörpers RBP Target ausgewählt werden. Proteine A, G und A / G haben alle spezifischen Isotyp Ziele und unterscheiden sich in Ziel-Affinität. Protein A-Sepharose Kügelchen wurden basierend auf Isotyp Spezifität und Affinität für Protein HuR Antikörper verwendet.
- Vor Gebrauch entfernen überschüssiges NT2-Puffer, sodass die endgültige Perlen Puffer beträgt 1:1.
- Verwendung von 1,5 ml RNAse-frei Mikrozentrifugenröhrchen, entfernen 100 ul PAS Aufschlämmung und stell 30 ug Antikörper für jede einzelne IP-Reaktion (dh spezifische RBP und Isotyp-Kontrolle).
- Fügen 100-200 ul NT2 Puffer Antikörper Beadgemisch.
- Gemisch kann für mehrere Wochen bei 4 ° C gelagert werden, wenn sie mit 0,1% Natriumazid ergänzt.
- Ein Isotyp-passenden Antikörper oder ganzen normalen Seren von derselben Spezies sollten parallel als ein Antikörper gegen Hintergrund Steuerung RNA verwendet werden.
- Fügen entsprechenden Antikörper zu Wulst-Mix und über Nacht inkubieren, Tumbling Ende über Ende bei 4 ° C.
- Optimieren Antikörpertiter für spezifische Protein untersucht (1, 5, 10 oder 30 ug Antikörper ist normalerweise ausreichend).
- Planen Antikörper-beschichteten Kügelchen unmittelbar vor der Verwendung durch Waschen mit 1 mleiskalter NT2-Puffer 5 mal.
- Waschen Beadgemisch durch Zentrifugation bei 13.000 × g für 1-2 Minuten bei 4 ° C.
- Entfernen Sie vorsichtig die maximale Menge an Überstandes mit Hand Pipette oder Aspirator aber vorsichtig sein, zu verhindern, dass Pellets.
- Waschen hilft bei der Entfernung ungebundenen Antikörper sowie RNase Verunreinigungen von Antikörper-Gemisch.
- Nach der letzten Wäsche abgeschlossen wurde, resuspendieren die Perlen in 700 ul eiskaltem NT2 Puffer durch Behandlung mit verschiedenen RNase-Inhibitoren gefolgt, um die Ziel-mRNAs, einschließlich 10 ul RNase Out bei 40 U / ul, 10 ul 100 mM zu schützen DTT und 15 ul EDTA (15 mM). Bringt Volumen 1.000 ul mit NT2 Puffer.
- Vanadyl-Ribonucleosid-Komplexe werden nicht verwendet aufgrund Hemmwirkung von EDTA.
3. Immunpräzipitation und RNA Niederschlag
- Optional Preclear Schritt:
- In den Hintergrund zu reduzieren, können Perlen und Kontroll-Antikörper vor, klare Lysat verwendet werden. Dies kann die Signal in der Ausgabe.
- Dieser Schritt kann es notwendig sein Hintergrund zu reduzieren, wenn dabei IP mittels Microarray gefolgt. Es ist im Allgemeinen nicht notwendig, IP mittels qRT-PCR gefolgt.
- Preclear mit 15 ug Isotypenkontrolle für 30 min / 4 ° C Taumeln Ende über Ende.
- Fügen Sie 50 ul vorgequollenen PAS Perlen nicht mit Antikörper beschichtet ab Schritt 2,1.
- Inkubieren 30 min / 4 ° C unter Rotation Ende über Ende
- Zentrifugieren bei 10000 × g bei 4 ° C. Speichern Überstand für IP.
- Fügen Sie 100 ul der isolierten geklärte Lysat (ca. 2-5 mg) auf vorbereitete Antikörper-Mix.
- Verdünnen Lysat wird dazu beitragen, Hintergrund und unspezifische Bindung zu reduzieren.
- Anzahl der Lysat-Eingang kann je nach Nachweismethode und Fülle von RBP or Effizienz der RBP Antikörper, wie in Schritt 1.1.c. beachten
- (Optional) sofort mischen Rohr durch leichtes flicking mehrfach durch kurze Zentrifugation bei 10.000 × g bei 4 ° C bis Pellet Perlen und umgehend entfernen 100 ul des Überstandes als Gesamteingangsleistung mRNA Darstellung für qPCR Analyse unter Verwendung von Standard-Techniken RNA-Isolierung.
- Dieser Schritt ist zu bestätigen, dass Eingangs-Lysat RNA ist optimal für IP und sollte nur als Prüfschritt oder als Schritt zur Problembehandlung nach RIP mit schlechter RNA Ergebnisse durchgeführt werden.
- Wrap Rohr in Parafilm eindeutige Abdichtung und Inkubieren bei 4 ° C für 2 bis 4 Stunden zu gewährleisten, Taumeln Ende über Ende.
- Timing der Inkubation sollte basierend auf dem Soll Fülle optimiert werden und sollte minimiert werden, um komplexe Umlagerung oder Zersetzung zu vermeiden. Für einige RBPs kürzere Inkubationen kann mehr optimal.
- Pellet Perlen bei 5.000 xgfoder 5 min bei 4 ° C und bis auf Potenzialanalyse Überstand durch Western blot. Shop Aliquots bei -80 ° C.
- Aliquote des Überstandes mit hohen Mengen an restlichem Zielprotein kann auf ein Versagen des Proteins durch Sepharosebeads ausgefällt werden.
- Wie zuvor beschrieben, zu waschen Perlen 5 mal mit 1 ml eiskaltem NT2 Puffer und Zentrifugation (5.000 × g für 5 min bei 4 ° C) und dann mit der Hand zu entfernen Pipette oder einem Aspirator Überstand.
- Strengere Waschverfahren kann verwendet werden, um Hintergrund durch Ergänzung NT2 Puffer mit Natriumdeoxycholat, Harnstoff oder SDS verringern.
- Die Proben auf Eis so viel wie möglich und arbeiten schnell, um den Abbau der Ziel-mRNA zu minimieren.
- Optional: Speichern eines kleines Aliquot (10%) der Beadgemisch oder führen eine zusätzliche Probe parallel zur Kontrolle der Effizienz von IP Zielprotein mittels Western Blot-Analyse.
4. DNase und Proteinase K Behandlungen
- Nach dem Waschen resuspendieren Perlen mit 100 ul NT2-Puffer mit 5 ul RNase-freie DNase 1 (2 U / ul) ergänzt.
- Halten bei 37 ° C für 5-10 min. 1 ml NT2 Puffer und Zentrifuge bei 5.000 xg für 1 min bei Raumtemperatur.
- Eine kleine (~ 10%) Aliquot von Perlen verwendet werden, um die IP-Effizienz durch SDS-PAGE-Analyse zu überprüfen.
- Resuspendieren PAS Pellet in 100 ul NT2-Puffer, 5 ul Proteinase K (10 mg / ml) und 1 ul 10% SDS.
- Inkubieren der resuspendierten Kügelchen Mischung für 30 min bei 55 ° C in einem Wasserbad leicht auf jeder flicking 10 min.
- Proteinase K wird in der Veröffentlichung der RNP-Komponenten unterstützen.
- Pellet Perlen bei 5.000 xg für 5 min bei Raumtemperatur (RT) und den Überstand (~ 100 ul).
- Fügen Sie 200 ul NT2 Puffer Perlen, Zentrifugenfuge 5.000 xg für 2 min bei RT, sammeln Überstand (~ 200 ul) und discard Perlen.
- Kombinieren Überstände (100 ul und 200 ul) und 300 ul untere Schicht der Säure Phenol-CHCl 3.
- Vortex, 1 min RT (oder 37 ° C im Shaker) und Zentrifuge bei 16.000 xg bei RT für 1 min.
- Sammeln Sie 250 ul der oberen Schicht (nicht stören Schnittstelle), fügen Sie 25 ul Natriumacetat bei pH 5,2, 625 ul 100% EtOH und 5 ul glycoblue und gut mischen.
- Bewahren Sie über Nacht bei -20 ° C.
- Um eine einwandfreie Rückgewinnung der RNA zu gewährleisten, wird die Zugabe von glycoblue die Sichtbarkeit des RNA-Pellet, indem sie als Träger-Molekül zu erhöhen.
- Am folgenden Tag Spin Mischung Rohre durch Inversion 3-5 mal, bei 12.000 × g bei 4 ° C für 30 min und der Überstand verworfen.
- 1 ml 70% EtOH auf die blaue Pellet und mischen durch Umdrehen oder Vortexen.
- Zentrifuge 12.000 × g bei 4 ° C für 2 min.
- Discard Überstand abzentrifugieren und 12.000 × g für 1 Minute bei 4 ° C.
- Entfernen Sie alle verbleibenden 70% EtOH mit einer Pipette und an der Luft trocknen Pellet bei RT für 5 min.
- Resuspendieren in 20-40 ul RNase / DNase-freiem Wasser oder einem anderen geeigneten Volumen benötigt.
- Probe ist nun bereit für weitere Downstream-Anwendungen wie qRT-PCR oder Microarray.
- Nanodrop Spektrophotometrie kann bei der Beurteilung Probenkonzentration verwendet werden, jedoch mit wertvollen Proben kann es von Vorteil sein, nanodropping Proben zu vermeiden, da es sein kann, verschwenderisch und relativ ungenau. Wir schlagen vor, die Normalisierung Proben Housekeeping-Gen-Transkripte auf Reinheit und IP Effizienz für Edelsteine oder geringe Ausbeute Proben zu beurteilen.
5. Repräsentative Ergebnisse
Wenn das Verfahren optimiert und richtig ausgeführt, sollte der Immunpräzipitation ergeben signifikante Anreicherung von mRNA Ziele. Typisch, Abhängig von der RBP und seine mRNA Target (s), so sehen wir die Anreicherung von etwa 10 - bis 50-fache, wenn sie durch qRT-PCR untersucht. Viele Ziele RBPs kann en masse entdeckt werden mittels Microarray-Analyse. Jedoch ist dieses Verfahren anfällig für Degradation wie bei qRT-PCR verglichen. Abhängig von der RBP, die Anzahl von Zielen und den Wirkungsgrad der Reaktion kann Mikroarray offenbaren hunderte neuer Targets, oder es kann nur ein paar zu entdecken, wenn überhaupt. Beispielsweise einer der besseren dadurch RNA-Bindeproteinen HuR, post-transkriptionell reguliert die Expression und Translation von Genen viele wichtige physiologische 1, 2. Isolierung des HuR-ribonucleo-Komplexes mittels RIP-Chip in Brustkrebs-Zelllinien, beispielsweise offenbart Anreicherung von mehreren wichtigen bekannten HuR Ziele, einschließlich β-Actin-PCR unter Verwendung qRT wie in Abbildung 1 gezeigt. In beiden Krebszelllinien β-Aktin 12 angereichert - bis 15-fache. Normalerweise, wenn richtig durchgeführt sehen wir eine deutliche enrichment von β-Aktin in einer Vielzahl von Zelllinien. Allerdings, wenn die RIP nicht verraten keinen wesentlichen Bereicherung für β-Aktin deutet auf ein Problem mit dem RIP und das Verfahren kann wiederholt werden muss. Außerdem ergab Mikroarray-Analyse von immunpräzipitierten Proben aus diesen Zellinien deutliche Expression Teilmengen HuR Targets in verschiedenen Östrogenrezeptor (ER) positiven MCF-7 Krebszellen gegenüber ER negativen MB-231 Brustkrebs-Zelllinien, wie in 2 gezeigt, ein. Diese Ziele lassen sich in mehrere Kategorien: bekannte und unbekannte HuR Ziele, die entweder verbunden waren oder nicht im Zusammenhang mit Krebs. Zum Beispiel sind sowohl CALM2 und CD9 Krebsgene die nicht zuvor als HuR Targets identifiziert. Verwendung des Mikroarrays und Bestätigung mit qRT-PCR, CALM2 und CD9 erwiesen sich 5 - bis 180-fache in HuR Pellet angereichert, die eine Wechselwirkung zwischen dem prominenten HuR Protein und dieser Zielgene.

Abbildung 1. Immunpräzipitation und RIP in MB-231 (ER-) und MCF-7 (ER +) Brustkrebszellen. Immunpräzipitationen wurden von MB-231 oder MCF-7 Zelllysaten unter Verwendung von Anti-HuR monoklonalen Antikörper (3A2) und IgG1 Isotyp-Kontrolle. A. IP Western of HuR zeigte erwartete Größe band von 3A2 erkannt. Panel auf der rechten Seite zeigt Mengen von HuR im Eingang Lysate aus beiden Zelllinien verwendet, mit β-Tubulin als Ladekontrolle. B. Überprüfung durch quantitative RT-PCR zeigte fünfzehn und elf-fache Anreicherung von β-Aktin, ein bekannter HuR Ziel, in den 3A2 IPs von MB-231 und MCF-7, bzw.. Alle ΔΔCT Werte wurden auf GAPDH normalisiert. Experimente wurden in doppelter Ausführung (n = 2) erfolgt.
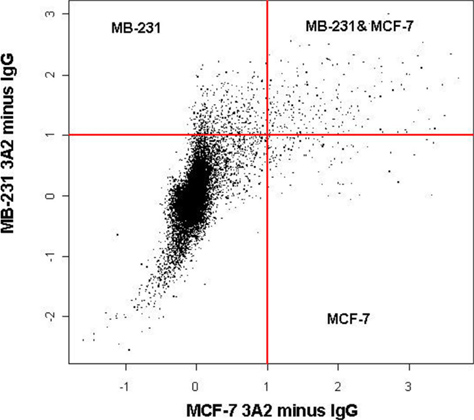
Abbildung 2. HuR RIP-CHIP identifiziert verschiedene genetische Profile in ER + und ER-Brustkrebszellen.HuR Immunpräzipitationen wurden von MB-231 MCF-7 oder Zelllysaten unter Verwendung HuR Antikörper und IgG1 Isotyp-Kontrolle hybridisiert Illumina Sentrix Arrays (47.000 Gene) durchgeführt. Steuersignale wurden abgezogen. Die Ergebnisse stellen die kumulativen Daten aus 12 verschiedenen Arrays. Experimente wurden in dreifacher Ausführung (n = 3) für jede Zelllinie mit passenden Kontrollen durchgeführt. Waagen sind log2.
Diskussion
Aufgrund der Natur dieses Experiments, Optimierung und Erfahrung wird die einzigen Möglichkeiten, um erfolgreich gewährleistet beschaffen die beabsichtigten Ergebnisse sein. In vielen Schritte dieses Verfahrens sind die Temperatur und effiziente Handhabung der Reagenzien und Produkte von entscheidender Bedeutung. Die richtige Planung und Ausführung der Technik wird dazu beitragen, sicherzustellen, dass das Experiment innerhalb eines angemessenen Zeitrahmens an den optimalen Temperaturen empfohlen wurde durchgeführt. Ein Hauptproblem mit RNA Isolation Experimenten ist die Empfindlichkeit von RNAs den Abbau durch RNasen. Alle Reagenzien müssen RNase frei und gelagert oder verwendet in RNase-freiem Container sein. Dies ist ein entscheidender Schritt bei der Sicherstellung der Integrität Ihrer mRNA Probe. Selbst wenn das Experiment korrekt durchgeführt wird, kann jedoch das gewünschte Ergebnis nicht durch die Art der Wechselwirkung zwischen dem RBP und seiner Ziel-mRNAs erreicht werden.
Ein potentielles Problem wird mit einer geringen oder gar kein Signal von der RNA durch die RIP-Chip isoliert.Obwohl es Signals von Gesamt-RNA sein kann, kann dies das Ergebnis einer unzureichenden Bindungsprotein ist heruntergezogen werden, die Perlen sein. Der erste Schritt bei der Fehlerbehebung ist zu bestätigen, dass die zelluläre Lysat verwendet adäquaten Ausdruck der spezifischen RBP hat. Nach Bestätigung kann Protein nach der endgültigen NT2 Waschen isoliert werden gewaschen und in Laemmli-Puffer oder einer anderen geeigneten denaturierenden Puffer und erhitzt bei 95 ° C für 5 min. Western-Blot-Analyse kann an diesen Proben in Koordination mit Eingangs Lysat sowie negative Kontrollen verwendet werden, um eine ausreichende Pulldown von assoziierten Protein.
Darüber hinaus, weil Lysieren der Zelle ist erforderlich, um diese Komponenten zugreifen zu können, kann das Potential für abnorme und unerwünschte Wechselwirkungen zwischen normalerweise getrennten Proteine und mRNA eingeführt werden. Diese Interaktionen könnten binden und "tanken" Ihre Ziel-mRNAs oder Proteine durch unspezifische Wechselwirkungen. Zusätzlich Proteine in diesen unterschiedlichen conditions kann in mehreren Varianten falten und ihre Bindungsmotive kann nicht zugegriffen werden, um ihre Ziel-mRNAs, die Verhinderung ihrer Wechselwirkungen. Beide stärken die Bedeutung der Zusammenarbeit effizient als auch die Nutzung der optimalen Temperaturen aufgeführt, um diese unerwünschte Wechselwirkungen zu begrenzen. Darüber hinaus wird die Optimierung der Waschbedingungen für jeden spezifischen Zielproteins kritisch sein, um die Reinheit der Wechselwirkung maximieren. Mehr stringenten Waschbedingungen erforderlich sein. Zum Beispiel kann der Waschpuffer mit SDS oder einer entsprechenden Menge an Harnstoff um unspezifische Wechselwirkungen und Hintergrund im Signalausgang reduzieren ergänzt werden. Dies wird völlig abhängig von der angestrebten Experimentator RBP sowie die Ziel-mRNA in ihrer einzigartigen physiologischen Bedingungen. Einige Bedingungen nicht geeignet sein für bestimmte mRNA-Analyse-Tools, die zur Vorbereitung von Proben von Bedeutung.
Schließlich, obwohl RIP ist bei der Anreicherung von RNA erfolgreiche-RBP Interaktionen, ein gut bekanntes Problem mit RIP (CHIP)-Methode ist die Unfähigkeit, die spezifischen bindenden Domänen des RBP auf die vorübergehende mRNA Targets zu identifizieren. Mehrere Vernetzung Techniken können durch RIP einzigartige Sequenz Ziele zu isolieren verwendet befolgt werden, allerdings neigt die Verwendung von kurzwelligem UV-auf Nukleinsäure-Schäden führen. Ein neues Verfahren PAR-CLIP oder photoaktivierbaren Ribonukleosid Vernetzen und immuoprecipitation bekannt beschäftigt langwelligen UV bis in Thiouridin naszierende RNA, die eine Identifizierung der eindeutigen Bindungsstellen von sowohl stabil als auch transient RNA-Wechselwirkungen zu integrieren.
Insgesamt hat RIP-Chip als hervorragendes Werkzeug zur Isolierung und Untersuchung der Wechselwirkungen zwischen RNA-bindende Proteine und ihre mRNA Zielsetzungen unserer Gruppe sowie viele andere Forschergruppen etabliert. Obwohl sensible Natur und Praxis wird ordnungsgemäße Durchführung dieses Verfahrens ergeben die Isolierung dieser RNP-Komplexe, die bis vor kurzem INACC habenessible für die Entdeckung und Analyse.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden Interessen haben.
Danksagungen
Department of Defense (Idea Award W81XWH-07-0406) - Zum Ulus Atasoy
NIH RO1 A1080870 - Um Ulus Atasoy
NIH R21 A1079341 - Um Ulus Atasoy
University of Missouri Institutional Funds - Um Ulus Atasoy
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenz | Firma | Catalog Number | Kommentare |
| 1 M Dithiothreit | Fischer | BP172-5 | DTT |
| DNase 1 | Ambion | 2235 | RNase-frei |
| Ethylendiamin Tetraccetic Säure | Fischer | BP118-500 | EDTA |
| Glykogen | Ambion | 9516 | |
| 1 HEPES | Sigma | H3375-100G | pH 7,0 |
| Igepal Nonidet P-40 | USB | 78641 | NP40 |
| 1 M KCl | Fischer | BP366-500 | |
| 1 M MgCl 2 | Fischer | BP214-500 | |
| NT2 Buffer | * Unten finden | ||
| Polysom Lysis Buffer | * Unten finden | ||
| Proteaseinhibitor-Cocktail-Tabletten | Roche | 11873580001 | |
| Protein A-Sepharose Kügelchen | Sigma | P3391 | |
| Proteinase K | Fischer | BP1700-100 | |
| RNase Out RNase Inhibitor | Invitrogen | 10777-019 | 40 U / ul |
| 1 M NaCl | Fischer | BP358-212 | |
| Natriumdodecylsulfat | Fischer | BP166-500 | SDS |
| 1 M Tris-HCl | Fischer | BP153-500 | pH 7,4 |
| Trizol | Invitrogen | 15596-026 | |
| Vanadyl-Ribonucleosid-Komplexe | New England Labs | S1402S | VRC |
| Reagent Aufarbeitung |
| Planen Reagenzien in RNase / DNase-frei, DEPC behandeltem Glaswaren |
| Polysomen Lysepuffer |
| 100 mM KCl |
| 5 mM MgCl 2 |
| 10 mM HEPES (pH 7,0) |
| 0,5% NP40 |
| 1 mM DTT |
| 100 Einheiten / ml RNase Out |
| 400 uM VRC |
| Protease-Inhibitor-Cocktail Tablette |
| 5 ml Lysepuffer Polysomen |
| Fügen Sie 50 ul 1 M HEPES (pH 7,0) |
| 500 ul 1 M KCL |
| 25 ul 1 M MgCl 2 |
| 25 ul NP40 |
| 4,7 ml RNase-DNase-freie H 2 O |
| 50 ul 1 M DTT |
| 12,5 ul 100 U / ml RNase Out |
| 200 ul Protease-Inhibitor-Cocktail (gelöst laut Hersteller) |
| 10 ul 200 mM VRC (zum Zeitpunkt der Verwendung) |
| NT2 Buffer |
| 50 mM Tris-HCl (pH 7,4) |
| 150 mM NaCl |
| 1 mM MgCl 2 |
| 0,05% NP40 |
| 1 l Puffer NT2 |
| 50 ml Tris (pH 7,4) |
| 30 ml 5 M NaCl |
| 1 ml 1 M MgCl 2 |
| 500 ul NP40 |
| 820 ml RNase-DNase-freie H 2 O |
Referenzen
- Calaluce, R., Gubin, M. M., Davis, J. W., Magee, J. D., Chen, J., Kuwano, Y., Gorospe, M., Atasoy, U. The RNA binding protein HuR differentially regulates unique subsets of mRNAs in estrogen receptor negative and estrogen receptor positive breast cancer. BMC Cancer. 10, 126-140 (2010).
- Gubin, M. M., Calaluce, R., Davis, J. W., Magee, J. D., Strouse, C. S., Shaw, D. P., Ma, L., Brown, A., Hoffman, T., Rold, T. L., Ulus Atasoy, U. Overexpression of the RNA binding protein HuR impairs tumor growth in triple negative breast cancer associated with deficient angiogenesis. Cell Cycle. 9, 3337-3346 (2010).
- Keene, J. D., Komisarow, J. M., Friedersdorf, M. B. RIP-Chip: the isolation and identification of mRNAs, microRNAs and protein components of ribonucleoprotein complexes from cell extracts. Nature Protocols. 1, 302-307 (2006).
- Tenenbaum, S. A., Lager, P. J., Carson, C. C., Keene, J. D. Ribonomics: identifying mRNA subsets in mRNP complexes using antibodies to RNA-binding proteins and genomic arrays. Methods. 26, 191-198 (2002).
- Tenenbaum, S. A., Carson, C. C., Atasoy, U., Keene, J. D. Genome-wide regulatory analysis using en masse nuclear run-ons and ribonomic profiling with autoimmune sera. Gene. 317, 79-87 (2003).
- Tenenbaum, S. A., Carson, C. C., Lager, P. J., Keene, J. D. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. Proc. Natl. Acad. Sci. U.S.A. 97, 14085-14090 (2000).
- Baroni, T. E., Chittur, S. V., George, A. D., Tennebaum, S. A. Advances in RIP-Chip Analysis. Methods in Molecular Biology. 419, 93-108 (2008).
- de Silanes Lopez, I., Zhan, M., Lal, A., Yang, X., Gorospe, M. Identification of a target RNA motif for RNA-binding protein HuR. Proc. Natl. Acad. Sci. U.S.A. 101, 2987-2992 (2004).
- de Silanes Lopez, I., Lal, A., Gorospe, M. HuR: post-transcriptional paths to malignancy. RNA Biol. 2, 11-13 (2005).
- Grigull, J., Mnaimneh, S., Pootoolal, J., Robinson, M. D., Hughes, T. R. Genome-wide analysis of mRNA stability using transcription inhibitors and microarrays reveals posttranscriptional control of ribosome biogenesis factors. Mol. Cell Biol. 24, 5534-5547 (2004).
- Hieronymus, H., Silver, P. A. Genome-wide analysis of RNA-protein interactions illustrates specificity of the mRNA export machinery. Nat. Genet. 33, 155-161 (2003).
- Hieronymus, H., Yu, M. C., Silver, P. A. Genome-wide mRNA surveillance is coupled to mRNA export. Genes Dev. 18, 2652-2662 (2004).
- Mukherjee, N., Corcoran, D. L., Nusbaum, J. D. Integrative Regulatory Mapping Indicates that the RNA-Binding Protein HuR Couples Pre-mRNA Processing and mRNA Stability. Molecular Cell. 43, 1-13 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten