A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מדידת מחזור התא קינטיקס התקדמות עם סימון מטבוליות cytometry זרימה
In This Article
Summary
שינויים עדינים מעקב בהתקדמות ו קינטיקה של שלבי מחזור התא ניתן להשיג זאת על ידי שימוש בשילוב של תיוג חילוף החומרים של חומצות גרעין עם BrdU ו מכתים סך הדנ"א הגנומי באמצעות יודיד Propidium. שיטה זו מונעת את הצורך של סינכרון כימי של תאים רכיבה על אופניים, ובכך למנוע את כניסתה של נזק שאינו ספציפי בדנ"א, אשר בתורו משפיע על התקדמות מחזור התא.
Abstract
Precise control of the initiation and subsequent progression through the various phases of the cell cycle are of paramount importance in proliferating cells. Cell cycle division is an integral part of growth and reproduction and deregulation of key cell cycle components have been implicated in the precipitating events of carcinogenesis 1,2. Molecular agents in anti-cancer therapies frequently target biological pathways responsible for the regulation and coordination of cell cycle division 3. Although cell cycle kinetics tend to vary according to cell type, the distribution of cells amongst the four stages of the cell cycle is rather consistent within a particular cell line due to the consistent pattern of mitogen and growth factor expression. Genotoxic events and other cellular stressors can result in a temporary block of cell cycle progression, resulting in arrest or a temporary pause in a particular cell cycle phase to allow for instigation of the appropriate response mechanism.
The ability to experimentally observe the behavior of a cell population with reference to their cell cycle progression stage is an important advance in cell biology. Common procedures such as mitotic shake off, differential centrifugation or flow cytometry-based sorting are used to isolate cells at specific stages of the cell cycle 4-6. These fractionated, cell cycle phase-enriched populations are then subjected to experimental treatments. Yield, purity and viability of the separated fractions can often be compromised using these physical separation methods. As well, the time lapse between separation of the cell populations and the start of experimental treatment, whereby the fractionated cells can progress from the selected cell cycle stage, can pose significant challenges in the successful implementation and interpretation of these experiments.
Other approaches to study cell cycle stages include the use of chemicals to synchronize cells. Treatment of cells with chemical inhibitors of key metabolic processes for each cell cycle stage are useful in blocking the progression of the cell cycle to the next stage. For example, the ribonucleotide reductase inhibitor hydroxyurea halts cells at the G1/S juncture by limiting the supply of deoxynucleotides, the building blocks of DNA. Other notable chemicals include treatment with aphidicolin, a polymerase alpha inhibitor for G1 arrest, treatment with colchicine and nocodazole, both of which interfere with mitotic spindle formation to halt cells in M phase and finally, treatment with the DNA chain terminator 5-fluorodeoxyridine to initiate S phase arrest 7-9. Treatment with these chemicals is an effective means of synchronizing an entire population of cells at a particular phase. With removal of the chemical, cells rejoin the cell cycle in unison. Treatment of the test agent following release from the cell cycle blocking chemical ensures that the drug response elicited is from a uniform, cell cycle stage-specific population. However, since many of the chemical synchronizers are known genotoxic compounds, teasing apart the participation of various response pathways (to the synchronizers vs. the test agents) is challenging.
Here we describe a metabolic labeling method for following a subpopulation of actively cycling cells through their progression from the DNA replication phase, through to the division and separation of their daughter cells. Coupled with flow cytometry quantification, this protocol enables for measurement of kinetic progression of the cell cycle in the absence of either mechanically- or chemically- induced cellular stresses commonly associated with other cell cycle synchronization methodologies 10. In the following sections we will discuss the methodology, as well as some of its applications in biomedical research.
Protocol
1. תא הכנה
- תאים הצלחת להשיג צפיפות של כ 60% confluency. התאים חייבים להיות בשלב יומן בעת האיסוף. עבור mcf7 תאים, עושים זאת על ידי זריעה על 5 x 10 תאים 5/10 ס"מ צלחת באמצעי התקשורת המתאימים. HT29 ו LS180 תאים זרע לאחר 6 x 10 תאים 5/6 צלחת ס"מ. השתמשנו DMEM התקשורת השלימו עם 10% FBS ו 1x פניצילין / סטרפטומיצין. הקפד צלחות זרע שולטת חיוביות ושליליות הנכונותבנוסף דגימות הבדיקה שלך. הם כוללים:
| אני | בקרה חיובית | BrdU בלבד |
| ב | בקרה חיובית | לצרכן בלבד |
| ג | בקרה שלילית | BrdU שלילית, PI שלילית |
(יש לציין כי ניסוי ראשוני עם timepoints מרובים כמו זה שמתואר בפרוטוקול זה ניתן להשתמש כדי לצמצם את מסגרת הזמן שבו לבצע את האוספים הבאים.)
- התאים מצופה מודגרת על 37 מעלות צלזיוס, 5% CO 2 של 24-48 שעות, ובכך לאפשר לתאים להתאושש לצרף.
- לדופק תאים תווית עם bromodeoxyuridine (BrdU), DMEM מוחלף Wiהתקשורת טריים המכילים 10 מיקרומטר ה BrdU. תאים מודגרת עבור 1 H ב 37 מעלות צלזיוס, 5% CO 2, כדי לאפשר שילוב BrdU לתוך ה-DNA. הקפד להשאיר צלחת 1 מטופל לשמש בקרת שלילית.
- הדופק תיוג התקשורת מוסר לבין תאים שטף בקצרה עם 1X PBS.
- התקשורת הצמיחה טריים נוסף (מינוס BrdU) ותאי מותר להמשיך לדגור על 37 מעלות צלזיוס, 5% CO 2 עד timepoint מתאים קצירת הוא הגיע.
2. קציר קיבוע
- בתאים שלא טופלו משלב 1.3 נחשבים timepoint אפס. מדגם זה ניתן לקצור יחד עם 1 H timepoint אשר נאסף מיד לאחר הטיפול BrdU.
- כדי לקצור את התאים, מדיה מוסר והצלחות הם שטף פעם אחת עם 1X PBS.
- תאים trypsinized, אסף בתקשורת culturing ו pelleted לאחר מכן על ידי צנטריפוגה בסל"ד 1500 עבור 5 דקות סופernatant נמחק.
- יש לשטוף 1-10 x 10 6 תאים 5 מ"ל קרח קר 1X PBS. סרכזת בסל"ד 1500 עבור Supernatant 5 דקות נמחק.
- תא גלולה הוא resuspended אז μl 100 של קר כקרח PBS / 1% FBS. תוספת של 1% FBS PBS מסייע במניעת clumping התא.
- הוסף ירידה תאים אלה על ידי ירידה של 5 מ"ל ל -20 ° C, 70% אתנול לתקן את התאים.
- דגירה על קרח למשך 30 דקות או בחנות ב 4 ° C למשך הלילה. זהו צעד אידיאלי שבו להפסיק איסוף דגימות מן timepoints שונים. דוגמאות ניתן להשאיר באתנול במשך כמה ימים, ולאפשר להם להיות מעובד בו זמנית באמצעות יתרת לפרוטוקול.
3. BrdU ו PI מכתים
- גלולה תאים על ידי צנטריפוגה 5 דקות בסל"ד 1500.
- הסר מקבע, אבל להשאיר ~ 50 μl בו כדי לשחרר את הכדור על ידי vortexing.
- כדי לפגל את ה-DNA, לאט להוסיף 1 מ"ל של HCL 2N / טריטון X-100dropwise תוך vortexing. דגירה דגימות בטמפרטורת החדר למשך 30 דקות.
- גלולה תאים על ידי צנטריפוגה דגימות 5 דקות בסל"ד 1500. Aspirate וזורקים supernatant.
- Resuspend תא גלולה ב 1 מ"ל של 0.1M נתרן tetraborate, pH 8.5, לנטרל את הצעד denaturation.
- סרכזת תאים 5 דקות בסל"ד 1500. Aspirate וזורקים supernatant.
- Resuspend תא גלולה ב μl 75 תמהיל מכתים BrdU (50 0.5% μl tween 20/1% BSA / PBS + 20 μl FITC מצומדות נגד BrdU + 5 μl 10 מ"ג / מ"ל RNase).
- דגירה בטמפרטורת החדר למשך 45 דקות מפני האור.
- לתאים גלולה, צנטריפוגות מדגמים דקות 5 בסל"ד 1500. Aspirate וזורקים supernatant.
- Resuspend תא גלולה ב 1 מ"ל של PBS המכיל 5 מיקרוגרם / מ"ל propidium יודיד.
4. תזרים Cytometry
- כדי לנתח את התאים, cytometer זרימת מצויד לייזר 488 ננומטרמסננים מתאימים הוא הכרחי. תוכנת ניתוח כגון CellQuest יש ליצור החלקות המפורטות להלן.
- כאשר פועל באמצעות מדגם cytometer את זרימת, ליצור פיזור קדימה (SSC-H) לעומת הצד פיזור (FSC-H) העלילה כדי להבטיח את התפלגות גודל תקין של התאים (איור 1 א). שני פרמטרים אלה נרשמות על בקנה מידה ליניארי.
- במקביל להצגת חלקת FL3-H (PI כתם) לעומת fl2-W (1B איור). עלילה זו משמשת ליצירת השער (R3) כדי לבודד את החלק היחסי של תאים הנמצאים בתוך דפוס ההתפלגות הנורמלית של מחזור התא. באופן כללי, דפוס זה מאופיין על ידי חלוקת שני אשכולות של תאים מתוכן את ה-DNA של 2N ו 4n מכתים לצרכן מייצג את ה-G1 ו G2 של שלבי מחזור התא, בהתאמה. שורה של תאים הממוקמים בין 2 קבוצות הוא נציג של שכפול ה-DNA מתמשכת המתרחשת בשלב S של מחזור התא.
- צור displayi העלילהng התאים סגורות (R3) של 4.3 צעד, עם FL1-H (FITC, העלילה יומן) על ציר y ו - PI (העלילה ליניארי) על ציר x (איור 1 ג). העלילה זה ישמש כדי לקבוע את הפרמטרים הנותרים באמצעות הפקדים השונים המתוארים 1.1, וכן כדי לאסוף נתונים לצורך ניתוח.
- מניחים את PI רק דגימה מוכתמת על cytometer. התאם רווח להציב G1 ב ~ 200 על ציר ה-x. זה יהיה קל יותר לדמיין בעלילה היסטוגרמה של PI הכתם.
- פקד 2 כרוך פועל מדגם BrdU בלבד על cytometer את הזרימה. התאם רווח כך 2 אוכלוסיות של תאים (BrdU חיובית לעומת BrdU שלילי) נראה על המגרש. באופן אידיאלי, התאים BrdU שליליות ממוקמים להופיע מתחת 10 -1.
- השליטה הסופית היא מדגם BrdU / PI שלילית. הפעל בקרה שלילית על מנת להבטיח כי התאים אינם מופיעים בכל אחד הרביעים העליונה או ימינה.
- לאחר הפרמטרים של החלקות השונות נקבעו,cytometer הזרימה הוא מכויל ומוכן לעיבוד של תאים BrdU ו PI מוכתמים. מינימום של 10,000 תאים הנמצאים מגודרת בשבריר לצרכן הנכון (מתייחס לשלב 4.3) יש לקרוא לכל אוסף.
- כדי לנתח את הנתונים שנאספו, תוכנה כגון FlowJo או FacsDiva מנוצלים. כל החלקות הנ"ל ניתן ליצור מחדש בתוכניות אלה וניתוח כמותי סטטיסטי ביצע.
- נקודת הסיום של ניתוח כולל כמה שלבים רצופים. יצירת חלקת FITC לעומת PI עם תאים סגורות כמו PI חיובית העלילה לצרכן לעומת fl2-W מאפשר להבחין בין האוכלוסייה רכיבה על אופניים. שער 2 נוצרת העלילה זה על ידי בידוד האוכלוסייה BrdU חיובית. להביע את האוכלוסייה הייחודי על מגרש היסטוגרמה עם PI על ציר ה-x מאפשר לעקוב אחר מהלך הזמן של התקדמות מחזור התא. זה יכול להיות דמיינו עוד יותר על ידי מתכנן מספר BrdU חיובי תאים עם G1 או תוכן G2 כפונקציה של הזמן.
5. נציג תוצאות
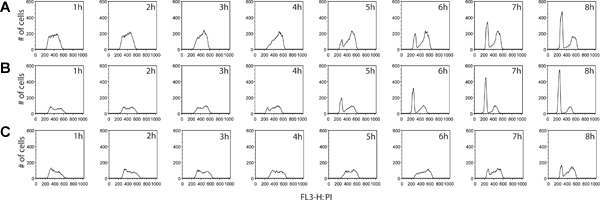
בדרך כלל תאים אופניים מוכתמים יודיד propidium יש שיאים שונים ב G1 ו G2, המתאימים תאים המכילים תוכן DNA 2N ו 4n, בהתאמה (איור 2). הדופק סימון BrdU מאפשר תיוג סלקטיבית של אוכלוסיית משנה של תאים באופן פעיל סינתזה DNA (שלב S IE). זמן קצר לאחר הסרת מגיב BrdU, כל התאים המסומנים נמצאים בשלב S (איור 3). על ידי הגבלת תיוג לדופק קצר אחד הוא מסוגל לעקוב אחרי זה ברור עכשיו תת אוכלוסיה של תאים בכל נקודות זמן רב כשהם עוברים דרך השלבים הבאים של מחזור התא. זה יכול להיות דמיינו בנקודה H 1 זמן באיור 3 כחוסר הברור של ה-G1 ו G2 פסגות.
מידע נוסף ניתן להסיק בשלב תיוג BrdU. לא רק יכול חלקם של התאים המתחלקים באופן פעיל להימדד, אבל ESTimate ההפצה מחזור התא בשלב בין שתי דגימות ניתן לקבוע גם כן. על ידי איסוף תאים במרווחים גדולים לא פחות לאחר הסרת מגיב BrdU, ניתן לאתר תאים כאשר הם ממשיכים בהתקדמות מחזור, כדי G2, ובסופו של דבר לעבור כדי חלוקת התא של התאים המקוריים, סוף סוף מתעוררים כמו BrdU חיובי לתאי הבת עם G1 ה-DNA תוכן (איור 4). דוגמה כימות מחזור התא בשלב וניתוח הקינטית ניתן באיור 5.
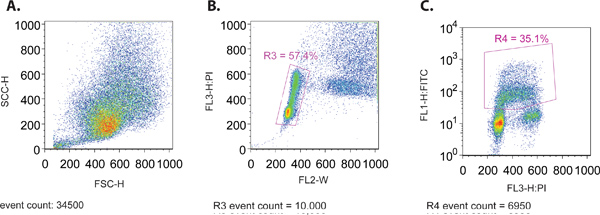
איור 1 (א) פיזור קדימה לעומת פיזור מגרש הצד של האוכלוסייה mcf7 נציג התא. (ב) לצרכן לעומת רוחב (FL-W) מגרש של האוכלוסייה mcf7 נציג התא. Gating מוצג להוציא כפילויות התא בסופו של דבר (R3). כפילויות התא יהיה רוחב הדופק עולה על תא בודד, כפי שהם דורשים זמן רב יותר לעבור דרך קרן לייזר ו therefעפרות ניתן לשלול מן הניתוח. (ג) PI לעומת FITC (BrdU) מגרש של האוכלוסייה mcf7 נציג התא. Gating מוצג לכלול רק את (BrdU) תאים FITC חיוביות (R4).
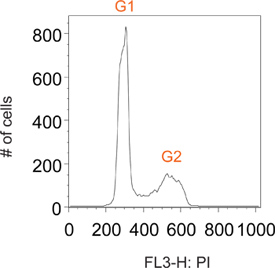
איור 2. היסטוגרמה נציג חלקת אוכלוסייה של תאים בדרך כלל אופניים.

איור 3. העלילה היסטוגרמה של התאים שנאספו 1h לאחר הסרת הדופק BrdU, לאחר gating עבור FITC (BrdU) חיובי תאים. BrdU חיובי תאים ב timepoint מוקדם לאחר הסרת התווית הצגת פרופילים לצרכן המתאימים דגימות DNA המציגים תוכן בקנה אחד עם תאים הנמצאים בשלב-S של מחזור התא, המאשר תיוג מוצלח של תאים רק במהלך סינתזת ה-DNA.
 LT = "איור 4" />
LT = "איור 4" />
איור 4. השוואה של מחזור התא קינטיקה התקדמות בין שורות תאים סרטניים, סרטן המעי הגס HT29 (א) ו LS180 (ב '), כמו גם סרטן השד mcf7 (C). תאים נאספו כל שעה במשך 8 שעות, בעקבות הסרת הדופק BrdU. בניסוי זה, צפינו על פרופיל ברור של התפתחות תאים מואצת לעבור שלב G2 של מחזור התא LS180 תא המעי הגס קו. השוואת קינטיקה בין שני פרופילים של תאים סרטן המעי הגס, הופעת שיא G1 ניכר ב ט = 4H שלאחר BrdU ב LS180 תאים, לעומת נקודת הזמן המתאימה לקו HT-29 בתא שבו אין זה השיא. בהשוואה בין אחת משתי שורות תאים המעי הגס, mcf7 תאים רכיבה על אופניים בקצב משמעותי. לחץ כאן כדי להציג דמות גדולה .
מחדש 5 "/>
איור 5 (א) תא כמותי מחזור שלב ניתוח BrdU, שכותרתו תאים סרטניים. האלגוריתם דין / ג'ט / פוקס היה מוחל על HT29, LS180 ו mcf7 (מאויר בירוק). מחזור וכתוצאה מכך על תאים בשלב הפצות מכל מדגם מבוטאות% מסך BrdU חיובי תאים עבור כל שלב. רק לבחור נקודות זמנים לקו כל תא מוצגים, כמו ניתוחים של כמה נקודות זמן קודמות הניבו תוצאות לא חוקיים. אלה נקודות הזמן קודמות, כל BrdU חיובי תאים הם בשלב-S ולכן אין G1 ברורים פסגות G2, אשר נחוצים יישום אלגוריתם. (ב) Histograms של תאים סרטניים מראה את הקינטיקה של התקדמות דרך G2 / M שלבי מחזור התא. התקדמות דרך G2 / M שלבים הוא המהיר ביותר LS180 התאים, ואחריו HT29 ו mcf7. לחץ כאן כדי להציג דמות גדולה .
"alt =" jpg איור S1 "/>
איור S1. Cytometry בקרות זרימה. mcf7 תאים מוצגים הביקורת (A) שלילי שבו התאים הם לא לצרכן ולא מוכתם BrdU. (ב) לצרכן מוכתם בלבד. (ג) BrdU-FITC שכותרתו דגימות.

איור איור S2. סכמטי המציג את הקשר בין ספקטרום פליטה הניאון של PI לעומת FITC (BrdU). החלונות איסוף ספקטרליים עבור ערוץ ניאון 1 (FL1, עבור FITC) וערוץ ניאון 3 (FL3, עבור PI) מוצגים בתיבות המתאימות. אין חפיפה בין ספקטרום fluorophore FL1 ו FL3 זיהוי. ככל הנראה, פיצוי הקרינה אינו הכרחי כאשר נתוני הניסוי של PI ו FITC-BrdU שיתוף שכותרתו התאים נאספים על FL1/FL3 ערוצי בהתאמה. ספקטרום הקרינה התקבלו באתר BioSciences BD:target = "_blank"> http://www.bdbiosciences.com/research/multicolor/spectrum_viewer. לחץ כאן כדי להציג דמות גדולה .
Access restricted. Please log in or start a trial to view this content.
Discussion
על ידי שילוב של cytometry זרימה עם שילוב BrdU, יש לנו את הכלים הדרושים כדי ללמוד קינטיקה מחזור התא. המאפיין הייחודי של BrdU לתפקד אנלוגי תימידין מה מאפשרת כימות DNA תוכן התא רכיבה על אופניים. שילוב של BrdU לתוך גדיל DNA צומח הבת בשלב הסינתזה של מחזור התא היא מה מאפשר לעקוב subpopulation של ...
Access restricted. Please log in or start a trial to view this content.
Disclosures
אין לנו מה למסור.
Acknowledgements
אנו מודים אנדי ג'ונסון של מרכז מחקר ביו רפואית UBC לקבלת סיוע בניתוח FACS. לחקר הסרטן הגיוס וונג מעבדה ניתן על ידי האגודה למלחמה בסרטן הקנדית Research Institute (ההפעלה מענק # 019250) ומ מהשקעה קרנות מחקר של הפקולטה למדעי התרופות, UBC. JMYW נתמך על ידי הכיסאות מחקר בקנדה סמית מיכאל קרן לקריירה בריאות תוכניות מחקר ופיתוח.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| מגיב | חברה | מספר קטלוגי | תגובות |
| bromodeoxyuridine | בקטון דיקינסון | 55089 | |
| propidium יודיד | סיגמא | 287075 | 1mg/ml המניות |
| FITC נגד BrdU | בקטון דיקינסון | 347583 | |
| נתרן tetraborate | דיג | S80172 | 0.1M, pH 8.5 |
| FACS קליבר | בקטון דיקינסון |
References
- Musgrove, E. A., Caldon, C. E., Barraclough, J., Stone, A., Sutherland, R. L. Cyclin D as a therapeutic target in cancer. Nat. Rev. Cancer. 11, 558-572 (2011).
- Molchadsky, A., Rivlin, N., Brosh, R., Rotter, V., Sarig, R. p53 is balancing development, differentiation and de-differentiation to assure cancer prevention. Carcinogenesis. 31, 1501-1508 (2010).
- Dickson, M. A., Schwartz, G. K. Development of cell-cycle inhibitors for cancer therapy. Curr. Oncol. 16, 36-43 (2009).
- Banfalvi, G. Cell cycle synchronization of animal cells and nuclei by centrifugal elutriation. Nat. Protoc. 3, 663-673 (2008).
- van Opstal, A., Boonstra, J. Inhibitors of phosphatidylinositol 3-kinase activity prevent cell cycle progression and induce apoptosis at the M/G1 transition in CHO cells. Cell Mol. Life Sci. 63, 220-228 (2006).
- Gasnereau, I. Flow cytometry to sort mammalian cells in cytokinesis. Cytometry. A. 71, 1-7 (2007).
- Harper, J. V. Synchronization of cell populations in G1/S and G2/M phases of the cell cycle. Methods Mol. Biol. 296, 157-166 (2005).
- Pedrali-Noy, G. Synchronization of HeLa cell cultures by inhibition of DNA polymerase alpha with aphidicolin. Nucleic Acids Res. 8, 377-387 (1980).
- Merrill, G. F. Cell synchronization. Methods Cell Biol. 57, 229-249 (1998).
- Fleisig, H. B., Wong, J. M. Telomerase promotes efficient cell cycle kinetics and confers growth advantage to telomerase-negative transformed human cells. Oncogene. , (2011).
- Cai, D., Byth, K. F., Shapiro, G. I. AZ703, an imidazo[1,2-a]pyridine inhibitor of cyclin-dependent kinases 1 and 2, induces E2F-1-dependent apoptosis enhanced by depletion of cyclin-dependent kinase 9. Cancer Res. 66, 435-444 (2006).
- Pozarowski, P., Darzynkiewicz, Z. Analysis of cell cycle by flow cytometry. Methods Mol. Biol. 281, 301-311 (2004).
- Sherwood, S. W., Rush, D. F., Kung, A. L., Schimke, R. T. Cyclin B1 expression in HeLa S3 cells studied by flow cytometry. Exp. Cell Res. 211, 275-281 (1994).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved