Method Article
גדיל DNA עקירת עיר רשתות תגובת השרשת כימית המופק מפלסמיד
In This Article
Summary
This protocol describes a method for deriving DNA strand displacement gates from plasmids and testing them using fluorescence kinetics measurements. Gates can be modularly composed into multi-component systems to approximate the behavior of formal chemical reaction networks (CRN), demonstrating a new use for CRNs as a molecular programming language.
Abstract
DNA nanotechnology requires large amounts of highly pure DNA as an engineering material. Plasmid DNA could meet this need since it is replicated with high fidelity, is readily amplified through bacterial culture and can be stored indefinitely in the form of bacterial glycerol stocks. However, the double-stranded nature of plasmid DNA has so far hindered its efficient use for construction of DNA nanostructures or devices that typically contain single-stranded or branched domains. In recent work, it was found that nicked double stranded DNA (ndsDNA) strand displacement gates could be sourced from plasmid DNA. The following is a protocol that details how these ndsDNA gates can be efficiently encoded in plasmids and can be derived from the plasmids through a small number of enzymatic processing steps. Also given is a protocol for testing ndsDNA gates using fluorescence kinetics measurements. NdsDNA gates can be used to implement arbitrary chemical reaction networks (CRNs) and thus provide a pathway towards the use of the CRN formalism as a prescriptive molecular programming language. To demonstrate this technology, a multi-step reaction cascade with catalytic kinetics is constructed. Further it is shown that plasmid-derived components perform better than identical components assembled from synthetic DNA.
Introduction
יכולת החיזוי של זיווג הבסיס ווטסון-קריק אפשרה DNA ננוטכנולוגיה הדינמי לצוץ כדרך הניתנת לתכנות לעיצוב התקנים מולקולריים עם תכונות דינמיות 1,2. בפרט, עקירת גדיל DNA - תגובת הכלאת תכנות, תחרותית - הוכיחה את עצמו מנגנון רב עוצמה להנדסת מערכות ה- DNA דינמיות. בתגובת עקירת גדיל DNA, oligonucleotide הנכנס דוחק גדיל קשור בעבר "פלט" משותף מחייב משלים. ניתן לקשר תגובות כגון מרובות יחד לתוך מפלי תגובה רב-שלבים עם רמה גבוהה של שליטה על הסדר והעיתוי של תגובה בודדת שלבים 3. מפלי עקירת גדיל DNA נעשו שימוש כדי ליצור מעגלים דיגיטליים ואנלוגיים מולקולריים 4-7, ננו החלפת 8-10, מנועים אוטונומיים מולקולריים 11-15, ומגברי קטליטי שאינם קוולנטיים 13,16-21. יתר על כן, Dניתן לדמות מכשירי NA באמצעות תגובות עקירת גדיל ומיועדים ליישומים מגוונים באמצעות תוכנת עיצוב בעזרת מחשב 22-24.
נכון לעכשיו, ה- DNA מסונתז כימי משמש כחומר העיקרי לננוטכנולוגיה DNA. עם זאת, טעויות בתהליך הסינתזה של DNA, וכתוצאה מכך oligonucleotides מושלם, הם האמינו להגביל את הביצועים של מכשירי ה- DNA דינמיים על ידי גרימת תופעות תגובות שגויות. לדוגמא, תגובות "דליפה" יכולות לגרום לשחרורו של oligonucleotide תפוקה גם בהיעדרו של הדק תגובה. תופעות כאלה הן ברורות ביותר במפלי תגובה עצמן שבו אפילו כמות מזערית של דליפה הראשונית סופו של דבר לגרום להפעלה המלאה של 19,20 המפל. לעומת זאת, תגובות לעתים קרובות אינן מצליחות להגיע לרמה הצפויה של הפעלה כי חלק מרכיבים לא לעורר אפילו בנוכחות של הקלט המיועד 7,25. מבוסס DNA כדי להפוך את הביצועים שלננו-התקנים דומים לעמיתיהם המבוססים על החלבונים הביולוגיים שלהם, מצבי שגיאה כזו צריך להיות מופחת באופן דרמטי.
פלסמידים חיידקים או DNA הביולוגי אחר יכולים לשמש כמקור זול יחסית של ה- DNA הטהור ביותר עבור יישומי ננוטכנולוגיה. כמויות גדולות של DNA יכולות להיות שנוצרו על ידי שכפול בחיידקים ויכולות ההגהה הפנימיות של מערכות חיות להבטיח את הטוהר של ה- DNA וכתוצאה מכך. למעשה, כמה מאמרים האחרונים הכירו בשירות הפוטנציאלי של ה- DNA הביולוגי ליישומי ננוטכנולוגיה 21,26-28. עם זאת, הטבע-גדילים כפולים המלאה של פלסמיד דנ"א עד כה אסור שימוש בו כחומר לייצור מכשירי ה- DNA דינמיים, אשר בדרך כלל מורכב מהמספר הרב של oligonucleotides ומכילים שני תחומים פעמים התקועות וחד-גדילים. במאמר האחרון 29 סוגיה זו הייתה ממוענת ואדריכלות שער DNA חדשה שמורכבת בעיקר מגדילי דנ"א כפול חתוכות (ndsDNA) היה להציגד.
חשוב לציין, יכולות להיות מתוכננות מערכות שערי ndsDNA שמבינות את הדינמיקה שצוינה על ידי כל רשת פורמאלית תגובה הכימית (CRN) 29. יכולים אפוא לשמש שערי ndsDNA, בעיקרון, כדי ליצור מערכות דינמיות שמפגינות תנודות ותוהו ובוהו, bistability וזיכרון, לוגיקה בוליאנית או התנהגויות אלגוריתמיות 30-38. לדוגמא, אסמכתא. 29 הפגינו CRN שלוש-תגובה שסיפק יישום מולקולרי של פרוטוקול "קונסנסוס", סוג של אלגוריתם מחשוב מבוזר 29,39,40. עבודה זו הוצגה לראשונה שימוש חדשני לפורמליזם CRN כ" שפת תכנות "למהירות סינתזת מערכות מולקולריות פונקציונליות (איור 1 א).
כאן, פרוטוקול מפורט לנובעים שערי ndsDNA מפלסמיד דנ"א מסופק. הראשון הוא סקירה של תהליך עיצוב הרצף. לאחר מכן, בעקבות הסבר של oligonucleotides איך סינטטי המכילרצפי השער הם משובטים לתוך פלסמידים והרצף אומת ומוגבר באמצעות תרבות חיידקים. לאחר מכן, הוא הראה כיצד ניתן לגזור ndsDNA שערים מפלסמידים על ידי עיבוד האנזימטית (ראה איור 2). לבסוף, שיטה לבדיקת שער התנהגות באמצעות מבחני קינטיקה הקרינה מתוארת.
מנגנון תגובה
כדוגמא, הפרוטוקול מתמקד בתגובה כימית + B-> B + C קטליטי. מיני A, B, ו- C ("אותות", איור 1) מתאים לכל מולקולת DNA חד-גדילים שונה. הרצפים של מולקולות אלה הם עצמאיים לחלוטין והגדילים לא מגיבים אחד עם השני באופן ישיר. יש הרצפים של כל האותות שני תחומים שונים פונקציונליים, כלומר, subsequences שפועל יחד בתגובות עקירת גדיל: 1) תחום דריסת רגל קצר (תוויות ת"א, שחפת, TC) המשמש לייזום של r עקירת הגדילeaction, ו -2) תחום ארוך (תוויות, B, C) הקובע את זהות האות.
אינטראקציות בין גדילי אות מתווכות על ידי מתחמי חתוכות פעמיים גדילי דנ"א (ndsDNA) שער (נקראים הצטרפו AB ומזלג לפני הספירה) ומינים חד-גדילי עזר (, , <ג TB> ו- ). התגובה הרשמית + B-> B + C מתבצע באמצעות סדרה של צעדי תגובת עקירת גדיל, שבו כל צעד תגובה חושף דריסת רגל לתגובה הבאה (איור 1). בדוגמא זו, אותות ו- B הם בתחילה בחינם בפתרון תוך C אות קשור לשער המזלג. בסוף התגובה B ו- C הם בפתרון. באופן כללי יותר, אותות שחייבים שער אינם פעילים ואילו אותות שהם חופשיים בפתרון הם פעילים, כלומר, הם יכולים להשתתף בתגובת עקירת גדיל כקלט. במהלך זמן התגובה ואחריו באמצעות אסטרטגית כתב ניאון (איור 1 ג). בעבודה קודמת 29, זה היה הראה כי מנגנון תגובה זו מבין stoichiometry הנכון אלא גם קינטיקה של תגובת היעד לא רק.
Protocol
1. רצף עיצוב
הערה: סקירה כללית עיצוב רצף: בסעיף זה, האסטרטגיה לעיצוב שערי DNA הנגזר פלסמיד מתואר. אתרי אנזים ממוקמים משני סוף השערים כדי לאפשר את שחרורו של שערים כפולים במלואו תקוע לאחר עיכול. אז אתרים שסחבו ממוקמים כך שאנזימים ליצור ניקס על הגדיל העליון כדי ליצור את שערי ndsDNA הסופיים. לבסוף, הרצפים שנותרו נבחרים כך שהתחומים עצמאיים הם מאונך זה לזה ואינו מציגים מבנה משני.
- מניחים את אתר nicking Nt.BstNBI ארבעה נוקלאוטידים מהסוף '3 של כל תחום ארוך (A, B, C, R, ואני). מניחים את אתר nicking Nb.BsrDI בסוף '5 של כל תחום ארוך (A, B, C, ו- R. שים לב שהתחום לא שיש לי כל אתר שסחב Nb.BsrDI). איור 2C מציג את השקפת הרצף מפורטת של הצטרף לשערי AB והמזלג לפני הספירה.
- מניחים את אתר הגבלת PvuII בשני הקצוות של שערי ndsDNA כך שעיכול PvuII יכול לשחרר את השערים מפלסמידים (ראה איור 2 ג).
- עיצוב רצפים מאולץ אחרים על ידי שני עקרונות הבאים: (א) גדילים לא צריכים להפגין מבנים משניים (ניתן לחזות מבני DNA באמצעות Nupack 41), ו- (ב) בכל התחומים צריכים להיות מאונך כדי למזער crosstalk.
- מניחים רצפי ndsDNA במרכז תבנית שער. מניחים 30-40 רצפי spacer נ"ב אקראיים בשני הקצוות של תבנית השער, כל spacer משמש כאתר קישור ייחודי לתגובה הבאה שרשרת פולימראז (PCR).
2. שיבוט של NdsDNA גייטס לפלסמידים
הערה: סעיף זה מתאר את שיטת שיבוט גיבסון להוספת 4 עותקים של השער לשדרת פלסמיד.
- תבניות שער להזמין ndsDNA כלוקים הגנומי פעמיים תקועים של יצרן ה- DNA (רצפי תבנית שער מוצגותבטבלה 1; גדילים להתרחש בשערי ndsDNA מוצגים בטבלה 2; רצפי רמת תחום מוצגים בטבלה 3).
- לאחר שקבל את ה- DNA הורה, לסובב את הצינורות המכילים בלוקים הגנומי ב10,000-14,000 XG ל1 דק 'על מנת להבטיח שכל ה- DNA המיובש הוא בחלק התחתון של הצינור.
- Resuspend הלוקים הגנומי המיובשים במים ללא DNase כדי להשיג ריכוז סופי של 10 ng / μl.
הערה: לחלופין, ה- DNA יכול להיות resuspended באמצעות חיץ 1x טריס חומצת ethylenediaminetetraacetic (EDTA) (TE חיץ: 10 מ"מ טריס ו1 mM EDTA, pH 8.0). עם זאת, EDTA הוא סוכן chelating לקטיונים דו ערכיים ויכול לעכב PCR. - צור 4 ברי שער עם אזורים שונים חפיפה באמצעות PCR סטנדרטי עם DNA פולימראז גבוהים פידליטי (ראה איור 3 א). רצפי פריימר מפורטים בטבלה 4 (טמפרטורת התכה של פריימרים אלה היא 62 מעלות צלזיוס).
- הפעל ג'ל agarose 2% ליום 1 ב40 V למשך 30 דקות ב RT (לפרוטוקול ג'ל אלקטרופורזה agarose מפורט לראות 42) ולחתוך את הלהקות המתאימות לכל PCR מוגברים בר מתוך ג'ל. אז לטהר את פרוסות ג'ל באמצעות ערכת חילוץ ג'ל (עיין בחומרים) הבא הוראות היצרן.
- לעכל עמוד השדרה פלסמיד המספר גבוה עותק (ראה חומרים) עם PvuII-HF וPstI-HF על 37 מעלות צלזיוס במשך שעה 1 (ראה לוח 5) על פי הפרוטוקול של היצרן. PvuII-HF וPstI-HF הם אנזימי הגבלת איכות גבוהה, אשר להפחית באופן דרמטי קיצוצים נוקבים.
- הפעל ג'ל agarose 1.5% ולחתוך את עמוד השדרה לינארית (בדרך כלל להפעיל את הג'ל על 140 V במשך 30-40 דקות ב RT). אז לחלץ את ה- DNA מפרוסת ג'ל באמצעות ערכת חילוץ ג'ל הבאה הוראות היצרן.
- לבצע הרכבה גיבסון 43 עם וקטור ינארית ושברי PCR מטוהרים (ראה טבלה 6 ואיור 3 ) במעלות צלזיוס 50 לשעה 1.
- להפוך את מוצר ההרכבה גיבסון מצעד 2.8 לEscherichia coli (E. coli) וצלחת על צלחת אגר Lysogeny מרק (LB) המכילה אנטיביוטיקה אמפיצילין (בריכוז של 100 מיקרוגרם / מיליליטר). לבצע שינוי באמצעות electroporation או שיטת הלם חום 44,45, ולהשתמש בE. המתאים זן coli. לדוגמא, השתמש E. JM109 coli זן לשינוי הלם חום, ולהשתמש DH5α electrocompetent E. קולי תאים לelectroporation.
הערה: עמוד השדרה פלסמיד המשמש מכילה קלטת התנגדות אמפיצילין. אם באמצעות סמן בחירה שונה, להשתמש באנטיביוטיקה המתאימה במקום אמפיצילין.
3. בקטריאלי תרבות הגברה ובקרת איכות
הערה: סעיף זה מתאר את הייצור ובידוד ההמוניים של פלסמידים המכילים את שערי DNA לאחר בקרת איכות.
- פיק מושבה אחתמהצלחת סלקטיבית אמפיצילין מהצעד 2.9 דגירה תרבות של 3 מיליליטר בינוני מועשרים המכיל אנטיביוטיקה אמפיצילין (בריכוז של 100 מיקרוגרם / מיליליטר). סמן את המושבה כזאת שזה יכול להיות מנוצל שוב בצעדים ניסיוניים שלאחר מכן. לגדול התרבות ב 37 מעלות CO / N עם (סל"ד 200-300) הטלטול הנמרץ. בדרך כלל, דגירה של 16-24 שעות.
- לחלץ את ה- DNA פלסמיד מתרבות החיידקים באמצעות ערכת מיני-הכנה הבאה הוראות היצרן.
- מדוד את ה- DNA פלסמיד מטוהר באמצעות ספקטרופוטומטר הבא הוראות היצרן. טווחי תשואה אופייניים מ50-1,000 ng / μl.
- קבל את ה- DNA פלסמיד חילוץ רצף על ידי שליחת הדגימה לחברת רצפי DNA. צריכים להיות ממוקמים פריימרים רצף על 100 נוקלאוטידים במורד זרם של האזור ללהיות רצף; פריימר הרצף לפלסמיד (ראה חומרים לפלסמיד) יש הרצף הבא: ATTACCGCCTTTGAGTGAGC.
לֹאטה: אם יש שגיאת רצף או רקומבינציה בשערי ndsDNA הוכנסו, בחר מושבה שונה מהצלחת מהשלב 2.9. בצע את השלבים 3.1-3.4 כדי לוודא שהרצפים של השערים הוכנסו נכונים. - אחרי שאימת כי הרצפים נכונים, לבחור את המושבה המקבילה מהצלחת סלקטיבית אמפיצילין (משלב 2.9), ודגירת תרבות של 800 מיליליטר נהדר מרק (TB) המכיל אנטיביוטיקה אמפיצילין (בריכוז של 100 מיקרוגרם / מיליליטר). לגדול התרבות ב 37 מעלות צלזיוס במשך שעה 16-24 עם רעד נמרץ (200-300 סל"ד). TB במיוחד הוא גם מתאים לייצור פלסמיד תשואה גבוהה.
הערה: לחלופין, LB יכול לשמש גם כדי לגדול חיידקים למרות תשואת פלסמיד יכולה להיות בעיה. - לטהר את ה- DNA באמצעות ערכת מקסי-ההכנה הבאה הוראות היצרן.
- בצע את השלב 3.3-3.4 כדי לבדוק אם הרצפים נכונים. אם כל רקומבינציה אירעה, ראה את ההערה הבאה. אחרת, עבור לשלב 4.
הערה: נושא אפשרי אחד הוא שעותקים רבים של שערים הוכנסו בפלסמיד עשויים להשתלב מחדש בשל תיקון DNA. כדי לטפל בבעיה זו, השתמש בE. זן coli חסר חלבון recA (חלבון הקשורים לתיקון DNA) כגון JM109 או DH5α להפוך פלסמיד בעבר-מאומת רצף (כלומר, ללא כל שגיאות רצף ורקומבינציה). לאחר מכן לבחור מושבה אחת מצלחת זו ולאמת את רצף פלסמיד על ידי שליחת הדגימה לחברת רצפי DNA.
4. אנזימתי עיבוד
הערה: סעיף זה מתאר את התהליך לעיכול פלסמידים כך שהם נחתכים ופצעו במקומות הנכונים ומוכן לשימוש עבור ניסויי קינטיקה.
- לעכל את ה- DNA פלסמיד המטוהר מצעד 3.7 עם אנזים הגבלת PvuII-HF עבור שעה 1 ב 37 ° C (ראה לוח 7). בדרך כלל לעכל את הפלסמיד עם 4 יחידות של PvuII-HF לכל 1 מ"ג של פלסמיד. fid הגבוהאנזימי הגבלת elity מומלצים לשימוש כי הם להפחית באופן דרמטי קיצוצים נוקבים.
- בצע משקעים אתנול במדגם.
- הוסף 2 כרכים שווים של אתנול מוחלט קר כקרח לדוגמא.
- דגירה את התערובת ב -80 ° C למשך שעה לפחות 1 (תערובת זו יכולה גם לשבת ב -80 ° C לO / N).
- צנטריפוגה ב 10,000-14,000 XG ב 0 מעלות צלזיוס למשך 30 דקות.
- הסר את supernatant.
- להוסיף 1,000 μl של RT אתנול 95% למדגם, ולהפוך 10-15 פעמים.
- צנטריפוגה ב 10,000-14,000 XG ב 4 מעלות צלזיוס למשך 10 דקות.
- הסר את supernatant יבש ואוויר על ספסל במשך 10-20 דקות.
- Resuspend את כדורי ה- DNA בנפח מתאים של H 2 O Nuclease בחינם (בדרך כלל 100-200 μl). הוספה יותר מ -200 μl בדרך כלל לעשות המדגם מדי לדלל לשימוש בניסויי קינטיקה.
- מדוד את ה- DNA resuspended באמצעות ספקטרופוטומטר הבא מ 'ההוראות של anufacturer.
- תקציר להצטרף שערים עם סחב אנזים Nb.BsrDI על 65 מעלות צלזיוס במשך שעה 1 באמצעות 4 יחידות של אנזים לכל 1 מיקרוגרם של פלסמיד (ראה לוח 8); לעכל מזלג שערים עם סחב אנזים Nt.BstNBI על 55 מעלות צלזיוס במשך שעה 1 באמצעות 8 יחידות של אנזים לכל 1 מיקרוגרם של פלסמיד (ראה לוח 9).
הערה: שלב 4.2 מסיר חיץ עיכול אנזים ועוזר להתרכז השערים לניסויי קינטיקה. שלב 4.2 ניתן לדלג לשערים להצטרף משום ששניהם אנזים הגבלת PvuII-HF ואנזים שסחב Nb.BsrDI חולקים את אותה חיץ עיכול. בשלב 4.2.8, H 2 O nuclease ללא משמש במקום TE כי EDTA הוא סוכן chelating לקטיונים דו ערכיים ויכול לעכב אנזימי הגבלה שצריך יונים אלה לתפקד.
הערה: התוספת של כמויות עודפות של אנזימים עלולה להוביל לכמויות גבוהות של דליפה ראשונית מעגל (איור 4), שככל הנראה נגרמת על ידי על-עיכול 46. נושא זה can לטפל על ידי אופטימיזציה של כמויות האנזים (ראה איור 4). מגוון טיפוסי של אנזימים הוא בין 1-10 יחידות / 1 מיקרוגרם פלסמיד.
5. הכנה של oligonucleotides חד-גדילים
הערה: סעיף זה מתאר את הפרוטוקול לresuspending וquantitating DNA synthetized כימי חד-גדילים (ssDNA) שישמש לגדילי אות וגדילי עזר. לרצפי גדיל ראה טבלה 10. שים לב שהפרוטוקול הבא הוא דוגמא להכנת 10 ssDNA מיקרומטר. ריכוזים אחרים של ssDNA יכולים להיות מוכנים באופן דומה.
- לאחר קבלת oligos מיצרן DNA, לסובב את הצינורות המכילים DNA ב10,000-14,000 XG ל1 דק 'על מנת להבטיח שכל ה- DNA המיובש הוא בחלק התחתון של הצינור.
- Resuspend את ה- DNA באמצעות חיץ 1x טריס חומצת ethylenediaminetetraacetic (EDTA) (TE חיץ: 10 מ"מ טריס ו1 המ"מ EDTA, pH 8.0) כדי להשיג ריכוז סופי של 100 מיקרומטר. בשביל שדוגמא, resuspend 8 nmol של ה- DNA בμl 80 של חיץ TE.
- מערבבים 10 μl של ה- DNA בעם 90 μl מים מולקולריים בצינור microcentrifuge, שאמור להגיע לריכוז סופי של 10 מיקרומטר 100 מיקרומטר.
- למדוד את הריכוז של דגימת DNA באמצעות ספקטרופוטומטר הבא הוראות היצרן המדויק. הפרוטוקול הבא נותן דוגמא לאופן שריכוז ה- DNA ניתן למדוד.
- ריק ספקטרופוטומטר עם 2 μl של מים מולקולריים.
- מדוד את הספיגה ב 260 ננומטר (260) של דגימת DNA. השתמש במשוואה הבאה כדי לחשב את ריכוז המניות.
הערה: ריכוז המדגם הוא M = מקדם 260 / הכחדה. ניתן למצוא את מקדם ההכחדה על גליון נתוני המפרט ידי יצרן ה- DNA.
6. הכנת כתבי ניאון
הערה: סעיף זה מתאר אתפרוטוקול להכנת C כתב, ניתן להרכיב כתבי ניאון אחרים באופן דומה.
- להזמין כרומטוגרפיה ביצועים גבוהים נוזלי (HPLC) oligonucleotides מטוהר ROX- <* ג * TC> (גדיל ראש C כתב) ו< ג> -RQ (הגדיל התחתון של C כתב) מיצרן DNA (ראה לוח 10 לרצפים ).
- לאחר שקבל את oligonucleotides, הגלול המסונתזת ולכמת דגימות כפי שמוסברות בשלב 5.
- מערבבים את ראש הכתב וגדילים תחתון (כלומר, ROX- <ג * TC *> ו- <ג> -RQ) ב1x טריס-אצטט-EDTA (טה) עם 12.5 מ"מ Mg 2 + (ראה לוח 11 למתכון מפורט ). שים לב שכאן 30% -RQ עודף גדיל כותרת מרווה <ג> מתווסף להרכיב את הכתב, אשר מבטיח כי כל החוטים שכותרת fluorophore הרוו אפילו עם stoichiometry מושלם.
- לחשל מורכב C הכתב באמצעות Cycler תרמי, קירור מ -95 מעלות צלזיוס עד 20 מעלות צלזיוס בשיעור של 1 ° C / דקה. ניתן לאחסן הדגימות ב 4 מעלות צלזיוס.
7. מדידות הקרינה
הערה: הסעיף זה מתאר פרוטוקול כללי למדידות הקרינה קינטיקה (ראה איור 5 להליך ניסיוני), ופרוטוקול זה ישמש בשלבי 8, 9, ו -10 כמו כן שימו לב שפרוטוקול זה הוא לשימוש בspectrofluorimeter. לחלופין, ניסויים אלה יכולים גם להתבצע בקורא צלחת למרות וריאציות וחוסר השליטה בטמפרטורה בניסויים ארוך טווח רגישות, גם ל- היטב יכולות להיות בעיה.
- הגדר את בקר הטמפרטורה ל -25 מעלות צלזיוס, ולחכות לטמפרטורה לייצב. שימוש בבקר טמפרטורה יכול להפחית השתנות באות שיכול להיגרם כתוצאה משינויי הטמפרטורה.
- פרמטרים נכונים להגדיר FOמדידות קינטיקה r בתוכנת רכישת נתונים של spectrofluorimeter. הגדרות דוגמא מפורטות הן כדלקמן:
- הגדר את רוחב החריץ ל2.73 ננומטר עבור שני monochromators העירור והפליטה.
- הגדר את זמן האינטגרציה עד 10 שניות לכל נקודת זמן 60 שניות. הגדר את משך מדידת 24 שעות.
- הגדר את אורכי גל עירור / פליטה כדי להתאים את fluorophores השתמשה בניסוי. אורכי גל דוגמא הם כדלקמן: רוקס (588 ננומטר / 608 ננומטר), וטמרה (559 ננומטר / 583 ננומטר).
- להוסיף H 2 O Nuclease החופשי ו10x חיץ טריס-אצטט-EDTA המכיל 125 מ"מ Mg 2 + (10x טה / Mg 2 +) לתא קוורץ סינטטי. ראה לוחות 12, 13, ו -14 לכרכי דוגמא לשימוש.
- להוסיף גדילי polyT כדי להשיג ריכוז סופי של מיקרומטר ~ 1 (ראה לוח 12, 13, ו -14 לכרכים), ולאחר מכן מערבולת סינתטיתתאי קוורץ במשך 10-15 שניות. באופן כללי, טיפים פיפטה יהיו בלתי במיוחד להיקשר DNA. הוספת ריכוזים גבוהים של גדילי polyT יכולה להפחית השגיאה מחייבת שאינו הספציפית הזה.
- להוסיף כתבים וגדילי עזר. ראה טבלה 12, 13, ו -14 לכרכי דוגמא לשימוש. שים לב כי עבור כיול כתב, אין צורך בחוטי עזר.
- הוסף 10% סולפט dodecyl נתרן (SDS) כדי להשיג ריכוז סופי של 0.15% SDS. הערה: SDS משמש ללנתק אנזימים מהשערים הנגזר פלסמיד כי אנזימים עלולים להפריע לתגובת עקירת גדיל (ראה איור 6). SDS מומלץ כאן במקום denaturation חום של אנזימים כדי למנוע את הניתוק ורקומבינציה לא נכונה של קווצות שער, שעלול להשפיע לרעה על תפקוד המעגל.
- [דלג על שלב זה לכיול כתב.]
- להוסיף להצטרף ושערים מזלג (ראו לוח 13, ו -14 לכרכים)לתא קוורץ הסינתטי ומערבבים את הפתרון על ידי pipetting למעלה ולמטה במשך לפחות 20 פעמים (לא מערבולת קובט כי פתרונות vortexing עם SDS יכולים לגרום לבועות שישפיעו מדידות הקרינה קינטיקה).
- כמו כן, תעבור לשלבים הבאים המדידה בהקדם האפשרי משום שתגובת הדליפה יוזמת מייד לאחר התוספת של שערים להצטרף ומזלג לתא קוורץ סינטטי.
- מניחים את תאי קוורץ סינטטיים לתוך התא של spectrofluorimeter.
- התחל מדידת קינטיקה.
- לאחר 5 דקות של מדידה, להוסיף גדילי קלט (ראה לוח 12, 13, ו -14 לכרכים) לתא קוורץ הסינתטי ומערבבים התגובה על ידי pipetting למעלה ולמטה במשך לפחות 20 פעמים. שים לב שהמדגם צריך להיות מעורב בעדינות כדי למנוע בועות. לבצע שלב זה בזמן שתכנית רכישת נתונים מושהה להימנע מדידת אותות מופעל על ידי חיצוניאוֹר.
- רשום את קינטיקה התגובה עד שהוא מגיע למצב יציב. קינטיקה התגובה מוצגות במחשב.
8. כתבי ניאון כיול
הערה: סעיף זה מתאר את הפרוטוקול להכנת עקומות כיול של כתבי ניאון. עקומות כיול ישמשו להמיר יחידות הקרינה שרירותיות לריכוז אות טוחנת.
- לכייל כתבי ניאון הבאים הפרוטוקול המתואר בשלב 7. השתמש בכרכים של מגיבים ומאגרים כפי שסוכם בלוח 12 הריכוז הסטנדרטי לדוגמא זו הוא 50 ננומטר (1x).; כתבים הם ב3x; קלט הוא 1x. למקרים שבהם הקלט נמצאים ב0.25x, 0.5x, 0.75x, להתאים את עוצמת הקול של H 2 O nuclease ללא בהתאם כדי לשמור על הנפח הסופי של כל תגובה יהיה 600 μl. נתוני דוגמא מוצגים באיור 7 א.
- הפוך עקומת כיול שלC כתב ידי ליניארי בכושר של ערכי הקרינה הגמר נגד הריכוז הראשוני של C אות (עקומת כיול דוגמא מוצגת באיור 7). עקומת כיול זה יכול לשמש כדי להמיר יחידות הקרינה שרירותיות לריכוז האות המקביל שלה.
9. לכמת את הריכוז של גייטס ndsDNA נגזר פלסמיד
הערה: כל אצווה מעובד באופן עצמאי של תוצאות שערי ndsDNA נגזר פלסמיד בתשואה של שערים פונקציונליים שונה, וסעיף זה מתאר פרוטוקול לכימות הריכוז של שערי ndsDNA נגזר פלסמיד.
- לכמת את הריכוז של שערי ndsDNA נגזר פלסמיד הבא הפרוטוקול המתואר בשלב 7. השתמש בכמויות של חומרים כימיים כפי שסוכם בטבלה 13 הערה:. לוח 13 מתאר מתכון דוגמא לכימות המזלג לפנה"ס הצטרף. AB ושערים אחרים יכולים להתבצע באופן דומה, אך באמצעות גדילי קלט שונים, גדילים וכתבי עזר.
- להמיר את ערך הקרינה הסופי נמדד בניסוי זה לריכוז של C אות באמצעות עקומת הכיול מהצעד 8.2. לאחר מכן חזרה-לחשב את ריכוז שער ndsDNA. לדוגמא, ערך הקרינה סופי לניסוי כימות השער של מקבילה 25 ננומטר C האות (0.5x) המבוסס על עקומת הכיול באיור 7. מאז המניה של מזלג לפנה"ס מדוללת 40 פעמים בתגובה זו, מניית הריכוז של שער המזלג לפני הספירה הוא 1 מיקרומטר.
10. מדידות קינטיקס לתגובת C + B-> B +
הערה: סעיף זה מתאר פרוטוקול לבדיקת מימוש DNA של תגובה כימית פורמאלית באמצעות מדידות הקרינה קינטיקה.
- לְכָלמדידת קינטיקה טופס הבא הפרוטוקול מתואר בשלב 7. כרכי שימוש בחומרים כימיים ומאגרים כפי שסוכם בלוח 14.
תוצאות
למבחן פונקציונלי, יישום ה- DNA של התגובה קטליטי bimolecular (כלומר, + B-> B + C) נוצר. הביצועים של שערים הנגזר פלסמיד הושוו לשערים מורכבים מדנ"א הסינתטי. תגובות קטליטי הן מבחן טוב לטוהר שער כי שער פגום יכול באופן בלתי הפיך מלכודת זרז, גורם השפעה בלתי מידתית בכמות המוצר מיוצר 18,19. במקביל, תגובת דליפה קטנה וכתוצאה מכך שחרור untriggered של אות קטליטי תהיה מוגברת באופן ליניארי, שהובילה לאות שגיאה בלתי מידתית. נתוני ניסוי לשערים ומסונתזים הנגזר פלסמיד מוצגים באיור 8B ו8C, בהתאמה. בניסויים, הריכוז של גדיל אות קבוע ואילו הסכום של אות B קטליטי מגוון. אות C משמש לקריאה מתוך ההתקדמות של התגובה בלי להפריע קטליטימַחזוֹר. קטליזה ניתן לצפות בנתונים מאז תגובות מתקרבות השלמה גם עם כמויות של הזרז B קטנים בהרבה מהסכום. מאז SDS לא הוסיף לניסויים שנעשו במערכת המסונתזת, מהירות תגובה (שיכול להיות מושפע על ידי התוספת של SDS) אינה השוואה ומוקד האנליטיות הוא במקום במחזור קטליטי (שיפורט להלן).
ניתוח נוסף של המחזור הקטליטית של תגובה זו נערך. מחזור מוגדר כאות C כמות המיוצרת עבור כל זרז B בזמן נתון. באופן ספציפי, מחזור חושבו מנתוני הניסוי שלנו על ידי חלוקת האות C דליפת מופחתים על ידי הסכום הראשוני של זרז B הוסיף. למערכת קטליטי אידיאלית, מספר המחזור הזה צריך להגדיל באופן ליניארי עם זמן ולהיות עצמאי מהסכום של זרז כל עוד המצע לא מגביל. במערכת אמיתית, שערים פגומים יכולים להשבית חתול alysts, והמחזור יגיע ערך מרבי, גם אם לא את כל המצע זמין מומר למוצר. ערך המחזור המרבי מציין כיצד מצעים רבים (אות) זרז (אות ב ') יכולים להמיר לפני שהפכו מובטל. כאן, הוא ציין כי המערכת המסונתזת חורגת מהגידול האידיאלי ליניארי המחזור הרבה יותר מוקדם מהמערכת הנגזר פלסמיד עושה, המציין תפיסה של הזרז דרך צד תגובה לא רצויה (איור 8D). השוואת המחזור מוצגת רק לריכוזים נמוכים כי בריכוזים גבוהים של זרזים, כל השערים יופעלו ולשחרר C אות. דליפת המעגל גם בהשוואה, והוא ציין כי יחס אות דליפה באמצעות שערים נגזר פלסמיד הוא כ -8% פחות מזה שערים מסונתזים באמצעות לאחר 10 שעות של תגובה (איור 8E).
Iles / ftp_upload / 53,087 / 53087fig1.jpg "/>
1. CRNs איור () משמש כשפת תכנות התיישנות. יכולות להיות מהונדסות רשתות תגובת DNA משוער הדינמיקה של CRN רשמי יישום ה- DNA (ב) להוראה כימית דוגמא:. + B-> B + C. גדילי דנ"א נמשכים כקווים עם חצים בסוף '3 ו* מציין השלמה. כל אות גדילים (<ת"א>, ירוק), B (, כתום), ו- C (, אדום) כללו תחום דריסת רגל אחת (שכותרתו ת"א, שחפת, וTC) ו תחום אחד זהות (שכותרתו, B, C). תגובת bimolecular + B-> B + C דורש שני מתחמים תקועים רב הצטרפו לAB ומזלג לפני הספירה, וארבעה גדילי עזר , , <ג TB>, ו- . התגובה ממשיכה דרך שבעה שלבים של עקירת גדיל, שבו כל צעד התחלהשל עם. אסטרטגית דריסת רגל מחייבת (ג) כתב. התגובה ואחריו באמצעות כתב שבגדיל התחתון הוא שכותרתו עם fluorophore (נקודה אדומה) והגדיל העליון מחובר למרווה (נקודה שחורה). בגלל שיתוף הלוקליזציה של fluorophore והמרווה, הקרינה כתב הוא הרווה בכתב שלם. C האות יכול להחליף את הגדיל העליון של הכתב, שהוביל לגידול של הקרינה. (נתון זה שונה מאסמכתא 29.) אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
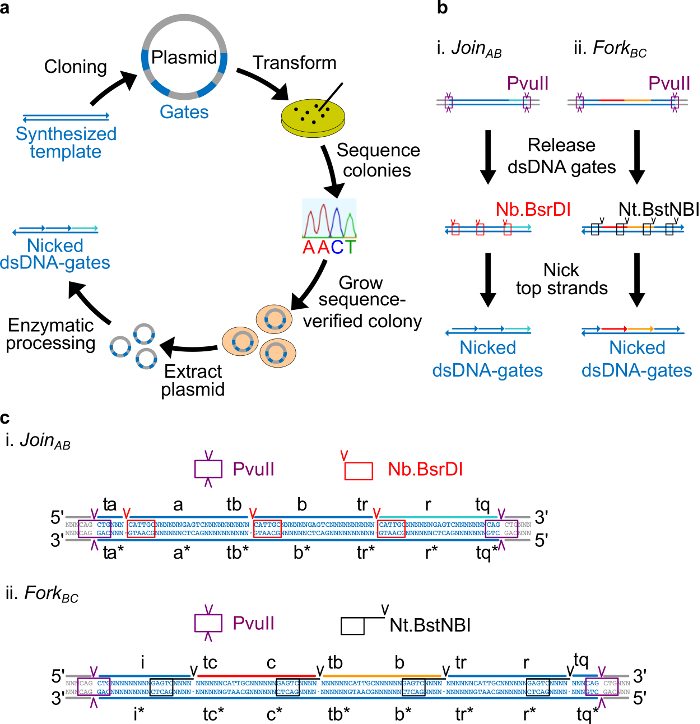
שערי 2. NdsDNA () איור עשויים מפלסמיד דנ"א חיידקים. כמה עותקים של תבנית שער גדילים הכפולה ndsDNA הם משובטים לתוך פלסמיד. פלסמידים המשובטים הםn הפך E. תאי coli ומושבות על הצלחת הם רצף מאומת. ברגע שהרצף הוא אישר, פלסמיד דנ"א מוגבר וחילוץ. לבסוף, פלסמיד גדילים הכפול מעובד לשערי ndsDNA הרצויים באמצעות עיבוד האנזימטית. עיבוד אנזימתי (ב) לשערי ndsDNA. אנזים ההגבלה PvuII משמש כדי לשחרר את השער מפלסמיד. השערים שוחררו מעובדים נוסף באמצעות אנזימים שסחבו: Nb.BsrDI משמש להפקת ניקס להצטרפות AB (לוח i); Nt.BstNBI משמש להפקת ניקס למזלג לפנה"ס (לוח ב '). אתרי הגבלה וסחב מצוינים כמו תיבות בצבעים. צפה ברצף (C) של תבנית השער של הצטרפות AB (לוח i) ומזלג לפנה"ס (לוח ב). אתר PvuII ההגבלה (מסומן בתיבה סגולה) הוא בשני קצותיו השערים ndsDNA. Nb.BsrDI ו- NTאתרים שסחבו .BstNBI מודגשים בקופסות אדומות ושחורות, בהתאמה. המקומות של חתך מסומנים בראשי חץ. רצף N הוא כל נוקלאוטיד. (נתון זה השתנה באישור אסמכתא 29.) אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
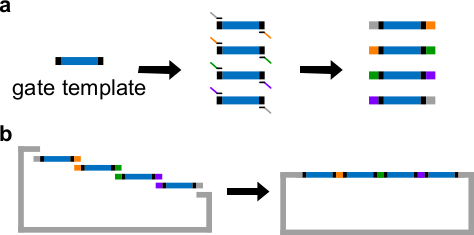
3. PCR איור (א) לתבנית שער DNA. תבנית שער DNA מכילה רצפי ndsDNA שער במרכז (אזור כחול), ורצפי spacer בשני הקצוות (אזורים שחורים; שני רצפי הסוף אלה הם מאונך). Primers יכול להיקשר לרצפי spacer של תבנית השער, וליצור ארבעה שברי DNA חופפים באמצעות PCR (רצפים חופפים נמצאים בדמות בצבעים). הרכבה (ב) גיבסון. ארבע fragmen DNA המוגבראז TS הם התאספו לתוך עמוד השדרה פלסמיד לינארית באמצעות שיטת ההרכבה גיבסון 43. (נתון זה השתנה באישור אסמכתא 29.) אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
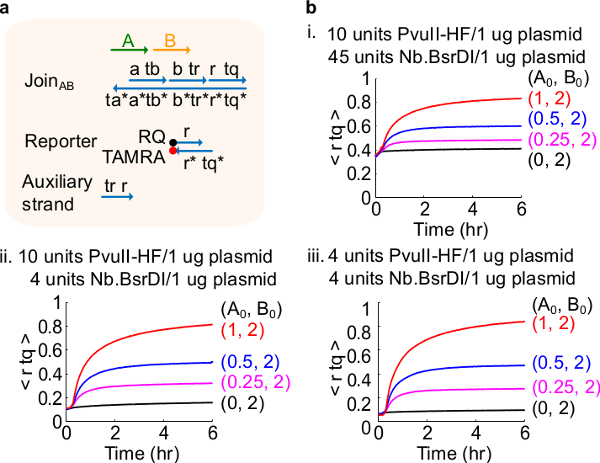
ביצועי איור 4. מעגל עם כמויות אנזים שונות. (א) ייצוג מופשט של השער, גדילי כתב, גדילי עזר, ואות המשמשים לניסויים המקביל. (ב) ניסויי קינטיקס עם נגזרות פלסמיד הצטרף מעובד AB עם כמויות אנזים שונות . אני. 10 יחידות של PvuII-HF ו -45 יחידות של Nb.BsrDI לכל 1 מיקרוגרם של פלסמיד; ii. 10 יחידות של PvuII-HF ו -4 יחידות של Nb.BsrDI לכל 1 מיקרוגרם של פלסמיד; iii. 4 יחידות של PvuII-HF ו -4 יחידות של Nb.BsrDI לכל 1 מיקרוגרם של פלסמיד. כל גדילי העזר היו ב2x (1x = 10nm). מתחם השער היה 1.5x, והניסויים בוצעו על 35 מעלות צלזיוס ב1x טה / Mg 2 +. (נתון זה השתנה באישור אסמכתא 29.) אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
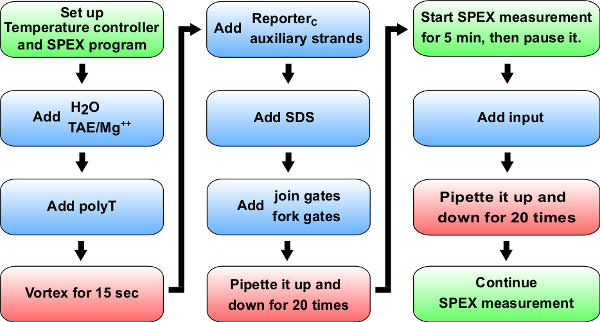
. איור 5 תרשים הזרימה של ניסויי קינטיקה כחולה:. חומרים להוסיף לקובט (תא קוורץ 0.875 מיליליטר סינטטי). טבלת עזר 14 לכרכים ספציפיים להוסיף לניסוי הקינטית של A + B -> B + C. ירוקות: הוראות של spectrofluorimeter (שכותרתו SPEX). אדום: ערבוב הוראות."Target =" _ 53087fig5large.jpg blank "> לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
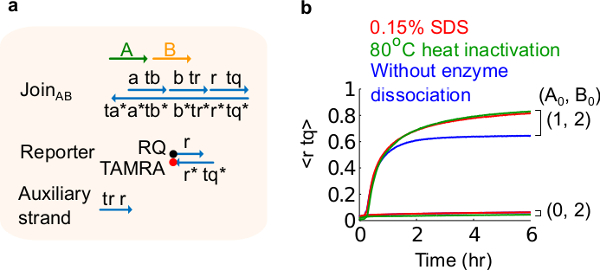
איור 6. אנזימים ניתוק ומעגל התנהגות. (א) ייצוג פשוטים של השער, גדילי כתב, גדילי עזר, ואות המשמשים לניסויים המקביל. (ב) ניסויי קינטיקה של חום 80 מעלות צלזיוס הצטרף AB באמצעות נגזרות פלסמיד איון (עקבות ירוקות), 0.15% סולפט dodecyl נתרן (SDS) (אדום), ושליטה ללא איון חום או תוספת של SDS (כחול). הריכוז הסטנדרטי היה 1x = 10 ננומטר, וכל גדילי העזר והקלט B היו ב2x. מתחם השער היה 1.5x, והניסויים בוצעו על 35 מעלות צלזיוס במאגר 1x טריס-אצטט-EDTA המכיל 12.5 מ"מ Mg 2 + (1x טה / Mg 2 + ). (נתון זה שונה מאסמכתא 29.) אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
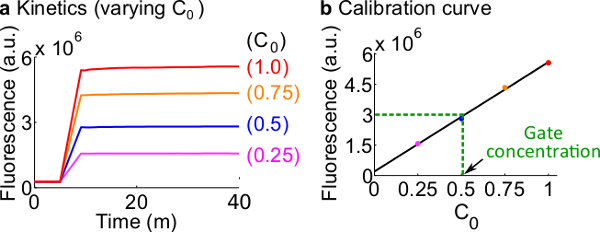
כיול איור 7. כתב. () כתב ג קינטיקה. ריכוז הכתב היה ב3x (1x = 50 ננומטר), והריכוז הראשוני של C אות מצויינים בדמות. (ב) רמות הקרינה של C אות בנקודה (40 דקות) סוף המדידה מראה קשר לינארי עם ריכוז ראשוני של C אות. בדוגמא כימות של מזלג לפנה"ס שער (קו מקווקו ירוק), ערך הקרינה של מזלג לפני הספירה היה מידהד כמו 3 x 10 6 (AU), אשר תואם את 25 ננומטר (0.5x) המבוסס על עקומת הכיול. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
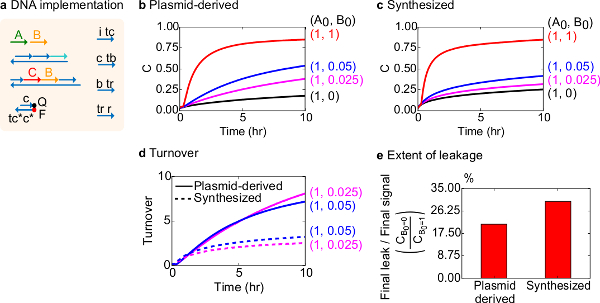
איור 8. קינטיקה תגובה קטליטי bimolecular (+ B-> B + C). () פשוטים ייצוג של השער, הכתב, גדילי עזר, וגדילי אות משמש לניסויים המקביל. ניסויים היו לרוץ במאגר 1x טריס-אצטט-EDTA המכיל 12.5 מ"מ Mg 2 + (1x טה / Mg 2 +). כל מתחמי השער היו בריכוז 75 ננומטר (1.5x), וגדילי עזר היו בריכוז 100 ננומטר (2x). קינטיקס נתונים לשערים הנגזר פלסמיד ונתונים לשערים מסונתזים מוצג ב( ב) ו- (ג) , בהתאמה. האות הייתה בגיל 50 ננומטר (1x). היקף כמויות שונות של אות (זרז) הוכנסו במערכת, והתגובה הייתה נבדקה ב- C ° 35. שערים נגזר פלסמיד (ד ') הציגו מחזור גבוה משערי DNA מסונתזים כאשר כמויות נמוכות של קלט נוספו. (E) של דְלִיפָה. התרשים בר מראה את היחס של הדליפה הסופית לאות הסופית (C B0 = 0 / C B0 = 1) בנקודות הקצה (10 שעות). (נתון זה שונה מאסמכתא 29.) אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
| תבניות שער | רצפים | אורך (NT) |
| JoinAB | TCTAGTTCGATCAGAGCGTTATTACCAGTAGTCGATTGCTCAG CTGCTACATTGCTTCTACGAGTCATCCTTCCACCATTGCACCTTAGAGTCCGAATCCTACCATTGCTTAACCGAGTCTCACAACCAGCTGTCATTATGGACTTGACACACAGATTACACGGGAAAGTTGC | 173 |
| FORKBC | TCTAGTTCGATCAGAGCGTTATTACCAGTAGTCGATTGCTCAGCTGCCATCATAAGAGTCACCATACCCACATTGCCACATCGAGTCCCTTTTCCACCATTGCACCTTAGAGTCCGAATCCTACCATTGCTTAACCGAGTCTCACAACCAGCTGTCATTATGGACTTGACACACAGATTACACGGGAAAGTTGC | 194 |
vious.within עמודים = "תמיד"> טבלת 1. רצפים של תבניות שער ndsDNA.
| שַׁעַר | גָדִיל | אורכו של הגדיל תחתון (NT) |
| JoinAB | JoinAB-תחתון, tb> , | 87 |
| ForkBC | ForkBC-תחתון, , , , | |
| 108 |
טבלה 2. קווצות הכוללות הצטרף AB והמזלג לפני הספירה. (טבלה זו שונות מאסמכתא 29.)
| תְחוּם | סדר פעולות | אורך (NT) |
| ת"א | CTGCTA | 6 |
| שַׁחֶפֶת | TTCCAC | 6 |
| TC | TACCCA | 6 |
| TR | TCCTAC | 6 |
| TQ | AACCAG | 6 |
| א | CATTGCTTCTACGAGTCATCC | 21 |
| ב | CATTGCACCTTAGAGTCCGAA | 21 |
| ג | CATTGCCACATCGAGTCCCTT | 21 |
| r | CATTGCTTAACCGAGTCTCAC | 21 |
| אני | CTGCCATCATAAGAGTCACCA | 21 |
| גדיל פריימר | רצפים | אורך (NT) |
| פריימר-1 קדימה | AAGAGAGACCACATGGTCCTTCTTGAGTTTGTAACAG CGTTATTACCAGTAGTCGATTGC | 60 |
| פריימר-1 הפוך | ACTACTATTTACTAATCCCATTGCGTGTTCTTATT TAATCTGTGTGTCAAGTCCATAATG | 60 |
| פריימר-2 קדימה | AATAAGAACACGCAATGGGATTAGTAAATAGTAGT CGTTATTACCAGTAGTCGATTGC | 58 |
| פריימר-2 ההפוך | GCGAAACTAGCTTGTGGTGATATTGTCTCGTGTGT TAATCTGTGTGTCAAGTCCATAATG | 60 |
| פריימר-3 קדימה | ACACACGAGACAATATCACCACAAGCTAGTTTCGC CGTTATTACCAGTAGTCGATTGC | 58 |
| פריימר-3 ההפוך | ACATTGTACGCCTAAATCATCAAGAATAATTGTTG TAATCTGTGTGTCAAGTCCATAATG | 60 |
| פריימר-4 קדימה | CAACAATTATTCTTGATGATTTAGGCGTACAATGT CGTTATTACCAGTAGTCGATTGC | 58 |
| פריימר-4 ההפוך | GAGCGCAGCGAGTCAGTGAGCGAGGAAGCCTGCAG TAATCTGTGTGTCAAGTCCATAATG | 60 |
לוח 4. רצפי פריימר לתבניות שער PCR של ndsDNA.
| מֵגִיב | נפח לתגובת 1x (μl) |
| עמוד השדרה פלסמיד גבוהה עותק (~ 300 ng / μl) | 10 |
| PvuII-HF (20,000 יחידות / מיליליטר) | 2 |
| PstI-HF (20,000 יחידות / מיליליטר) | 2 |
| מאגר חכם 10x Cut | 2 |
| H 2 O | 4 |
| נפח כולל | 20 ד> |
לוח 5. פרוטוקול עמוד השדרה פלסמיד לעכל.
| מֵגִיב | נפח לתגובת 1x (μl) |
| וקטור DNA (~ 50 ng / μl) | 1 |
| PCR מוגבר בר-1 (~ 50 ng / μl) | 1 |
| PCR מוגבר בר-2 (~ 50 ng / μl) | 1 |
| PCR מוגבר בר-3 (~ 50 ng / μl) | 1 |
| PCR מוגבר בר-4 (~ 50 ng / μl) | 1 |
| מיקס מאסטר 2x עצרת גיבסון | 5 |
| נפח כולל | 10 |
| מֵגִיב | נפח לתגובת 1x (μl) |
| פלסמיד דנ"א (~ 1 מיקרוגרם / μl ריכוז) | 1,000 |
| PvuII-HF (20,000 יחידות / מיליליטר) | 200 |
| מאגר חכם 10x Cut | 133.3 |
| נפח כולל | 1,333.3 |
לוח 7. פרוטוקול לשערים ndsDNA הוכנסו פלסמיד לעכל עם אנזים הגבלת PvuII-HF.
| מֵגִיב | נפח (μl) |
| שערים הצטרפו (~ 5 מיקרוגרם / ריכוז μl) | 150 |
| Nb.BsrDI (10,000 יחידות / מיליליטר) | 300 |
| מאגר חכם 10x Cut | 50 |
| נפח כולל | 500 |
לוח 8. פרוטוקול להצטרף שערים לעכל עם סחב אנזים Nb.BsrDI.
| מֵגִיב | נפח (μl) |
| מזלג שערים (~ 5 מיקרוגרם / ריכוז μl) | 150 |
| Nt.BstNBI (10,000 יחידות / מיליליטר) | 600 |
| חיץ 10x חוד 3.1 | 83.3 |
| נפח כולל | 833.3 |
together.within עמודים = "תמיד" FO:. לשמור-עם-previous.within עמודים = "תמיד"> לוח 9 פרוטוקול למזלג שערים לעכל עם סחב אנזים Nt.BstNBI.
ג. לוח 10 רצפי סטרנד ליישום התגובה הכימית + B -.> B + C (. טבלה זו שונה מאסמכתא 29)
| מֵגִיב | נפח (μl) | ריכוז סופי |
| ROX- <ג * TC *> ב 100 מיקרומטר | 10 | 10 מיקרומטר (1x) |
| <ג> -RQ ב 100 μ;M | 13 | 13 מיקרומטר (1.3x) |
| 10x טה עם 125 מ"מ Mg 2 + | 10 | 1x טה עם 12.5 מ"מ Mg 2 + |
| H 2 O | 67 | - |
| נפח כולל | 100 | 10 מיקרומטר (1x) |
שולחן פרוטוקול 11. להרכבת C כתב.
| מֵגִיב | נפח (μl) | ריכוז סופי |
| H 2 O | 514 | - |
| 10x טה עם 125 מ"מ Mg 2 + | 60 | 1x טה עם 12.5 מ"מ Mg 2 + |
| PolyT ב 300 μ;M | 2 | 1 מיקרומטר |
| C הכתב ב 10 מיקרומטר | 9 | 150 ננומטר (3x) |
| 10% SDS | 9 | 0.15% |
| 5 מיקרומטר | 6 | 50 ננומטר (1x) |
| נפח כולל | 600 | - |
לוח 12. פרוטוקול לכיול של C כתב. הכרכים הניתנים כאן הוא בסך הכל תגובת נפח של 600 μl (מקביל לשימוש בתא קוורץ הסינתטי מיליליטר 0.875), אך יכול להיות מותאם לעבודה עם תאים בגדלים שונים.
| מֵגִיב | נפח (μl) | קון הסופיcentration |
| H 2 O | 493 | - |
| 10x טה עם 125 מ"מ Mg 2 + | 60 | 1x טה עם 12.5 מ"מ Mg 2 + |
| polyT ב 300 מיקרומטר | 2 | 1 מיקרומטר |
| C הכתב ב 10 מיקרומטר | 9 | 150 ננומטר (3x) |
| ב 100 מיקרומטר | 3 | 10x |
| <ג TB> ב 100 מיקרומטר | 3 | 10x |
| 3 | 10x | |
| 10% SDS | 9 | 0.15% |
| מזלג לפנה"ס ב ~ 1 מיקרומטר (ריכוז לא ידוע) | 15 | ~ 0.5x |
| ב 100 מיקרומטר | 3 | 10x |
| נפח כולל | 600 | - |
לוח 13. פרוטוקול לכיול של מזלג לפני הספירה. הכרכים הניתנים כאן הוא בסך הכל תגובת נפח של 600 μl, אבל יכול להיות מותאם לעבודה עם תאים בגדלים שונים.
| מֵגִיב | נפח (μl) | ריכוז סופי | |
| H 2 O | 407.2 | - | |
| 10x טה עם 125 מ"מ Mg 2 + | 52.8 | 12.5 מ"מ Mg 2 + | |
| polyT ב 300 מיקרומטר | 2 | 1 מיקרומטר | |
| C הכתב ב 10 מיקרומטר | 9 | 150 ננומטר (3x) | |
| 10 מיקרומטר ב | 6 | 100 ננומטר (2x) | |
| <ג TB> 10 מיקרומטר ב | 6 | 100 ננומטר (2x) | |
| 6 | 100 ננומטר (2x) | ||
| 6 | 100 ננומטר (2x) | ||
| 10% SDS | 9 | 0.15% | |
| הצטרפות AB במיקרומטר 1 | 45 | 75 ננומטר (1.5x) | |
| מזלג לפני הספירה במיקרומטר 1 | 45 | 75 ננומטר (1.5x) | |
| <ת"א> 10 מיקרומטר ב | 3 | 50 ננומטר (1x) | |
| 10 מיקרומטר ב | 3 | 50 ננומטר (1x) | |
| נפח כולל | 600 | - | |
שולחן פרוטוקול 14. לתגובה כימית + B-> B + C. הכרכים הניתנים כאן הוא בסך הכל תגובת נפח של 600 μl, אבל יכול להיות מותאם לעבודה עם תאים בגדלים שונים.
| שערים מסונתזים | שערים הנגזר פלסמיד | ||||
| תֵאוּר | עֲלוּת | הצטרף לשערים | שערי מזלג | ||
| גדיל עמוד מטוהר ארוך (100 NT; שימש כגדילים התחתון של שער) | ~ 75 $ | תיאור < / Strong> | עֲלוּת | תֵאוּר | עֲלוּת |
| גדיל עמוד מטוהר קצר (~ 30 NT, שימשו כגדילים העליונים של שער) | ~ 185 $ | תבנית שער | ~ 100 $ | תבנית שער | ~ 100 $ |
| סה"כ | ~ 260 $ | ערכת חילוץ פלסמיד | ~ 26 $ | ערכת חילוץ פלסמיד | ~ 26 $ |
| אנזים הגבלה (PvuII-HF) | ~ 11 $ | אנזים הגבלה (PvuII-HF) | ~ 11 $ | ||
| סחבו אנזים (Nt.BsrDI, הצטרף לשערים) | ~ 29 $ | אנזים שסחב (Nt.BstNBI, שערי מזלג) | ~ 62 $ | ||
| סה"כ | ~ 166 $ | סה"כ | ~ 199 $ |
בעמודים = "תמיד" FO:.. לשמור-עם-previous.within עמודים = "תמיד"> לוח 15 השוואה בין עלות שערים נגזר פלסמיד ושערים מסונתזים (. טבלה זו שונה מאסמכתא 29)
| שערים מסונתזים | שערים הנגזר פלסמיד | ||
| עיבוד | זמן עיבוד | עיבוד | זמן עיבוד |
| רִכּוּך | שעה 1 | שיבוט | 5 שעות |
| טיהור עמוד | 2 שעות | חילוץ פלסמיד | 2 שעות |
| סה"כ | 3 שעות | שני שלבים של עיכול אנזים | 0.5 שעות |
| משקעים אתנול | 1 שעה | ||
| סה"כ | 8.5 שעות | ||
לוח 16. השוואת עיבוד זמן בין שערים הנגזר פלסמיד ושערים סינטטיים. (טבלה זו שונה מאסמכתא 29.)
Discussion
מאמר זה מתאר שיטה להפקת שערי ndsDNA מפלסמיד דנ"א הטהור ביותר. יתר על כן, פרוטוקול מוצג לאפיון ביצועי שער באמצעות assay קינטיקה הקרינה. נתוני ניסוי מראה כי מערכת הנגזר פלסמיד בביצועיו עמיתו הסינתטי שלה, גם אם המערכת סינתטית מורכבת מחוטים מטוהרים באמצעות ג'ל אלקטרופורזה polyacrylamide (עמוד). סביר להניח, הביצועים המשופרים של שערים הנגזר פלסמיד הוא בעיקר בשל גבוה מאוד טוהר של ה- DNA הביולוגי. DNA סינטטי מכיל מגוון של טעויות, במחיקות מסוימות שתגרומנה oligonucleotides של n-1 אורך, וצד מוצרים אלה בדרך כלל לא הוסרו לחלוטין בעמוד או הליכי טיהור כרומטוגרפיה נוזלית ביצועים גבוהים (HPLC). שיפורים דומים לאלה שדווחו כאן גם נצפו במחקר קודם של מגבר סיכת ראש זרז שהשתמש בדנ"א המופק ממקורות ביולוגיים 21.
התחת = "jove_content"> עם זאת, גם השימוש בשערים הנגזר פלסמיד לא יכולים לחסל לחלוטין טעויות בביצועי השער, שיש לפחות שתי סיבות: ראשונה על מערכת עיכול או חוסר הדיוק לחתוך יכול להוביל לשערים עם יותר מדי ניקס או חתכים בעמדות הלא נכונות. בכל מקרה, שערים נוטים יותר להשתתף בתגובות לא רצויות. בעיות מסוג זה עשויים להיות הקלה על ידי אופטימיזציה של כמות האנזים המשמשת (ראה איור 4). שנית, בניסויים אלה, רוב התשומות וגדילי דנ"א סינטטי עזר היו וכך הכילו מחיקות ומוטציות. באופן עקרוני, יכולים להיות גם השיגו את כל הקלט חד-גדילים וגדילי עזר מ- DNA phagemid דרך עיכול אנזים nicking של הגנום הנגיפי M13 קידוד מראש 26. אולי ביצועי המעגל ניתן לשפר עוד יותר באמצעות ssDNA נגזר מהגנום של החיידקים.
בעוד שהשימוש בשערים הנגזר פלסמיד נמצא כדי לשפר את ביצועי מעגל, ניתוחשל הפעמים העלות ועיבוד גילה כי בעוד שהייצור של שערים הנגזר פלסמיד הוא מעט זול יותר (לוח 15), זה לוקח 2-3 פעמים יותר זמן עיבוד בהשוואה להרכבה וטיהור של שערים מoligos המסונתזת מסחרי (לוח 16). העלויות העיקריות של שערים הנגזר פלסמיד הן סינתזת גן והשימוש באנזימי הגבלה. עבור 300 pmole של שערים (15 תגובות מספיק ליום 30 בננומטר), העלות המשוערת להצטרפות שערים היא כ 170 $ ו 200 $ עבור מזלג שערים, להיות הבדל העלות בשל השימוש באנזימים שסחבו שונים. לעומת זאת, הסינתזה הכימית של הגדילים לאותו שער העלויות כ 260 $ כוללים דמי טיהור עמוד. עלות הזמן העיקרית לשערים הנגזר פלסמיד היא בתהליך השכפול, ש, בדיוק כמו סינתזת DNA, יכול להיות במיקור חוץ לחברת סינתזת גן. עם זאת, יש לי שערים לאחר הרכבה, הנגזר פלסמיד היתרון שיכולים בקלות להיות משוכפלים פלסמידים המארחניתן לאחסן nd בצורה של מניות גליצרול בקטריאלי. זה מאפשר לעשות שימוש חוזר בשערים פעמים רבות.
במבט קדימה, ביצועים המשופרים של שערים הנגזר פלסמיד יכולים לאפשר מגוון הרבה יותר גדול של התנהגויות דינמיקה מאשר הודגם בניסוי עד כה עם ה- DNA CRNs. לדוגמא, 47,48 עבודה תיאורטית האחרון הציע שדפוסים מרחביים באמצעות התארגנות עצמית במקרו בקנה המידה יכולים להתממש עם CRNs DNA באמצעות מנגנון דיפוזיה תגובה. השיטה המוצגת כאן מספקת נתיב קיימא לבניית הרכיבים המולקולריים שבבסיס לחומרי DNA דפוסים עצמיים כזה. למרות מאתגר, פיתוח מורפולוגיות מאקרו בקנה מידה בדרך לתכנות יהיו השלכות משמעותיות בתחומים החל מחקר בביו-חומרים לרפואת רגנרטיבית.
Disclosures
The authors declare no competing financial interests.
Acknowledgements
איורים 1, 2, 3, 4, 6, 8 ולוחות 2, 3, 10, 15, 16 הם שונה מאסמכתא 29. עבודה זו נתמכה על ידי הקרן הלאומית למדע (NSF-להעניק CCF 1,117,143 וNSF-CCF 1,162,141 לGS). י '-JC נתמכה על ידי מלגות ממשלה הטיוואנית. SDR נתמכה על ידי התכנית הלאומית למדע קרן בוגר מחקר המלגה (GRFP).
Materials
| Name | Company | Catalog Number | Comments |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531S | |
| PvuII-HF | NEB | R3151L | |
| PstI-HF | NEB | R3140S | |
| Gibson Assembly Master Mix | NEB | E2611S | |
| Terrific Broth, Modified | SIGMA-ALDRICH | T0918-250G | |
| QIAprep Spin Miniprep Kit (250) | QIAGEN | 27106 | |
| QIAGEN Hispeed Maxi-prep Kit | QIAGEN | 12662 | |
| Nb.BsrDI | NEB | R0648L | |
| Nt.BstNBI | NEB | R0607L | |
| NanoDrop 2000c | Thermo Scientific | ||
| Double-stranded Genomic Blocks | IDT | ||
| Horiba Jobin-Yvon Spex Fluorolog-3 Fluorimeter | Horiba/Jobin Yvon | ||
| Synthetic Quartz Cells | Starna | 23-5.45-S0G-5 | |
| QIAGEN Gel Extraction Kit | QIAGEN | 28706 | |
| Plasmid Backbones | BioBrick | E0240-pSB1A2 | High copy number plasmid with Ampicillin resistance. Sequence can be found from http://parts.igem.org |
References
- Zhang, D. Y., Seelig, G. Dynamic DNA nanotechnology using strand-displacement reactions. Nat. Chem. 3, 103-113 (2011).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angew. Chem. Int. Ed. Engl. 50, 3124-3156 (2011).
- Zhang, D. Y., Winfree, E. Control of DNA strand displacement kinetics using toehold exchange. J. Am. Chem. Soc. 131, 17303-17314 (2009).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475, 368-372 (2011).
- Qian, L., Winfree, E. Scaling up digital circuit computation with DNA strand displacement cascades. Science. 332, 1196-1201 (2011).
- Zadegan, R. M., Jepsen, M. D., Hildebrandt, L. L., Birkedal, V., Kjems, J. Construction of a fuzzy and boolean logic gates based on DNA. Small. 11, 1811-1817 (2015).
- Seelig, G., Soloveichik, D., Zhang, D. Y., Winfree, E. Enzyme-free nucleic acid logic circuits. Science. 314, 1585-1588 (2006).
- Zadegan, R. M., et al. Construction of a 4 zeptoliters switchable 3D DNA box origami. ACS Nano. 6, 10050-10053 (2012).
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459, 73-76 (2009).
- Zhang, D. Y., Hariadi, R. F., Choi, H. M., Winfree, E. Integrating DNA strand-displacement circuitry with DNA tile self-assembly. Nat. Commun. 4, (1965).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406, 605-608 (2000).
- Green, S. J., Lubrich, D., Turberfield, A. J. DNA hairpins: fuel for autonomous DNA devices. Biophys. J. 91, 2966-2975 (2006).
- Venkataraman, S., Dirks, R. M., Rothemund, P. W., Winfree, E., Pierce, N. A. An autonomous polymerization motor powered by DNA hybridization. Nat. Nanotechnol. 2, 490-494 (2007).
- Green, S. J., Bath, J., Turberfield, A. J. Coordinated chemomechanical cycles: a mechanism for autonomous molecular motion. Phys. Rev. Lett. 101, 238101 (2008).
- Omabegho, T., Sha, R., Seeman, N. C. A bipedal DNA Brownian motor with coordinated legs. Science. 324, 67-71 (2009).
- Turberfield, A. J., et al. DNA fuel for free-running nanomachines. Phys. Rev. Lett. 90, 118102 (2003).
- Dirks, R. M., Pierce, N. A. Triggered amplification by hybridization chain reaction. Proc. Natl. Acad. Sci. U. S. A. 101, 15275-15278 (2004).
- Seelig, G., Yurke, B., Winfree, E. Catalyzed relaxation of a metastable DNA fuel. J. Am. Chem. Soc. 128, 12211-12220 (2006).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318, 1121-1125 (2007).
- Yin, P., Choi, H. M., Calvert, C. R., Pierce, N. A. Programming biomolecular self-assembly pathways. Nature. 451, 318-322 (2008).
- Chen, X., Briggs, N., McLain, J. R., Ellington, A. D. Stacking nonenzymatic circuits for high signal gain. Proc. Natl. Acad. Sci. U. S. A. 110, 5386-5391 (2013).
- Phillips, A., Cardelli, L. A programming language for composable DNA circuits. J. R. Soc. Interface. 6, S419-S436 (2009).
- Lakin, M. R., Youssef, S., Polo, F., Emmott, S., Phillips, A. Visual DSD: a design and analysis tool for DNA strand displacement systems. Bioinformatics. 27, 3211-3213 (2011).
- Lakin, M. R., Youssef, S., Cardelli, L., Phillips, A. Abstractions for DNA circuit design. J. R. Soc. Interface. 9, 470-486 (2012).
- Zhang, D. Y., Winfree, E. Robustness and modularity properties of a non-covalent DNA catalytic reaction. Nucleic Acids Res. 38, 4182-4197 (2010).
- Ducani, C., Kaul, C., Moche, M., Shih, W. M., Hogberg, B. Enzymatic production of 'monoclonal stoichiometric' single-stranded DNA oligonucleotides. Nat. Methods. 10, 647-652 (2013).
- Lin, C., et al. In vivo cloning of artificial DNA nanostructures. Proc. Natl. Acad. Sci. U. S. A. 105, 17626-17631 (2008).
- Bhatia, D., et al. Icosahedral DNA nanocapsules by modular assembly. Angew. Chem. Int. Ed. Engl. 48, 4134-4137 (2009).
- Chen, Y. J., et al. Programmable chemical controllers made from DNA. Nat. Nanotechnol. 8, 755-762 (2013).
- Arkin, A., Ross, J. Computational functions in biochemical reaction networks. Biophys. J. 67, 560-578 (1994).
- Érdi, P., Tóth, J. . Mathematical models of chemical reactions: theory and applications of deterministic and stochastic models. , (1989).
- Magnasco, M. O. Chemical kinetics is Turing universal. Phys. Rev. Lett. 78, 1190 (1997).
- Oishi, K., Klavins, E. Biomolecular implementation of linear I/O systems. IET Syst. Biol. 5, 252-260 (2011).
- Senum, P., Riedel, M. Rate-independent constructs for chemical computation. PLoS One. 6, (2011).
- Soloveichik, D., Cook, M., Winfree, E., Bruck, J. Computation with finite stochastic chemical reaction networks. Natural Computing. 7, 615-633 (2008).
- Soloveichik, D., Seelig, G., Winfree, E. DNA as a universal substrate for chemical kinetics. Proc. Natl. Acad. Sci. U. S. A. 107, 5393-5398 (2010).
- Tyson, J. J., Chen, K. C., Novak, B. Sniffers, buzzers, toggles and blinkers: dynamics of regulatory and signaling pathways in the cell. Curr. Opin. Cell. Biol. 15, 221-231 (2003).
- Cardelli, L. Two-domain DNA strand displacement. Math. Struct. Comput. Sci. 23, 247-271 (2013).
- Angluin, D., Aspnes, J., Eisenstat, D. A simple population protocol for fast robust approximate majority. Distrib. Comput. 21, 87-102 (2008).
- Cardelli, L., Csikasz-Nagy, A. The cell cycle switch computes approximate majority. Sci. Rep. 2, 656 (2012).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. J. Comput. Chem. 32, 170-173 (2011).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. , (2012).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat. Methods. 6, 343-345 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J. Vis. Exp. , e253 (2007).
- Lessard, J. C. Transformation of E. coli via electroporation. Methods Enzymol. 529, 321-327 (2013).
- Nasri, M., Thomas, D. Alteration of the specificity of PvuII restriction endonuclease. Nucleic Acids Res. 15, 7677-7687 (1987).
- Dalchau, N., Seelig, G., Phillips, A. Computational design of reaction-diffusion patterns using DNA-based chemical reaction networks. DNA Computing and Molecular Programming. , 84-99 (2014).
- Scalise, D., Schulman, R. Designing modular reaction-diffusion programs for complex pattern formation. Technology. 2, 55-66 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved