Method Article
3' סיום רצף ספריית הכנה עם א-seq2
In This Article
Summary
פרוטוקול זה מתאר שיטת ה-pre-mRNA מיפוי 3' עיבוד אתרי קצה.
Abstract
מחקרים בעשור האחרון נחשפו מגוון מורכב ודינמי, של ה-pre-mRNA המחשוף ותגובות פוליאדנילציה. mRNAs עם זמן 3' ללא תרגום אזורים (UTRs) נוצרות בתאים הבדיל ואילו תאים מתרבים מעדיפים לבטא תעתיקים עם קצר 3' UTRs. אנו מתארים פרוטוקול A-seq, (י) בהגרסה השנייה שלה, אשר פותחה כדי למפות פוליאדנילציה אתרים ברמת הגנום וללמוד ברגולציה של ה-pre-mRNA 3' סיום העיבוד. גם בפרוטוקול הנוכחי זה מנצל polyadenylate (זנב poly(A)) הנוספים במהלך להן של mRNAs יונקים ביותר להעשיר עבור mRNAs מעובד באופן מלא. מתאם ה-DNA עם deoxyuracil על מעמדה הרביעי מאפשר עיבוד מדויק של קצה 3' mRNA קטעים על רצף. לא כולל התרבות תאים, את ligations בין לילה, הפרוטוקול דורש כ 8 h הפעם תרגולים. יחד עם זאת, חבילת תוכנה קלה לשימוש עבור הניתוח של הנתונים רצף נגזר מסופק. A-seq2 ותוכנת ניתוח המשויך לספק פתרון יעיל ואמין המיפוי של 3' ה-pre-mRNA מסתיים במגוון רחב של מחלות, מ-106 או פחות תאים.
Introduction
לכידת ורצף של mRNA 3' הקצוות מאפשר חקר עיבוד mRNA כימות של ביטוי גנים. עקב הזנבות שלהם poly(A), mRNAs האיקריוטים יכול ביעילות להיטהר מן התא סך הכל lysates עם חרוז. מרותק למיטה oligo-deoxythymidine (oligo(dT)) מולקולות, אשר יכול גם פריים cDNA סינתזה. עם זאת, גישה זו יש שני חסרונות. ראשית, המופרת של א' כי הם פנימי תעתיקים יכול גם פריים cDNA סינתזה, וכתוצאה מכך אתרי poly(A) כדין. Poly(A) השני, הומוגניות מותח מהוות אתגרים ספציפיים עבור רצף, מלבד היותו לא אינפורמטיבי לזיהוי תעתיק. גישות שונות הוצעו כדי לעקוף את מגבלות אלו, כגון שעתוק במהופך דרך poly(A) זנבות ואחריו RNase H עיכול (seq P 3- 1), שימוש פריימר רצף מותאם אישית המסתיימת ב- 20 Ts (2, P-seq 2), פולסים עם כיוון מוקדם של שברי RNA עם poly(A) זנבות של נוקלאוטידים מעל 50 עם פריימר45 CU5T ולאחריו RNase H עיכול (3' קריאות 3), ושימוש של פריימר oligo-dT המכיל 3' המתאם סיכת ראש (A-seq 4).
שיטה א'-seq2 פיתחה לאחרונה 5 שואפת לעקוף את רצף דרך poly(A), באותו זמן כדי למזער את הפרופורציה של הדימרים הנוצרים על-ידי עצמית מצדו של מתאמי, במיוחד המתרחשים כאשר ריכוז מולרי של מתאמי עולה ריכוז הוספה. בעיה זו ניתן לסלק כאשר שני מתאמי מאתרים לאותו סוג של polynucleotide מסתיים כמו א-seq2, שבו 3' המתאמים מאתרים לסוף 5' קטעים RNA, המתאמים 5' 5' בסוף cDNAs לאחר שעתוק במהופך. השיטה היא נוחה יותר מאשר שלנו בעבר המוצע A-seq - שבו היה רצף של 5'-כדי-3 ' כיוון ובכך הדורשות דיוק נשלט RNA פיצול-, תוך שמירה על רמת דיוק גבוהה של זיהוי אתר poly(A). סביב 80% הקריאות ברצף בדגימות טיפוסי למפות באופן ייחודי הגנום, להוביל זיהוי של יותר מ-20,000 poly(A) אתר אשכולות, יותר מ-70% אשר חופפים עם המבואר 3' UTRs.
בקצרה, פרוטוקול א-seq2 מתחיל עם ה-mRNA ולהפיק מצדו של מתאמי הפוכה המשלים 3' 5' בקצוות של קטעים RNA. פוליפוני (א)-RNAs המכיל נארזים הפוכה עיבד עם פריימר oligo(dT) ארוך נוקלאוטיד 25 (nt) המכיל של נוקלאוטיד עיגון בקצה 3', על dU במיקום 4 ו של ביוטין 5' בסוף, המאפשר קשירה של cDNA כדי streptavidin מגנטי חרוזים. רוב הראשיים, כולל של ביוטין, יוסר cDNA על-ידי פצילות ב- dU על ידי שילוב אנזים המשתמש, המכילה DNA אורציל glycosylase (UDG), את ה-DNA glycosylase-lyase Endonuclease השמיני. התגובה הזו יוצא מסתיימת ללא פגע מצדו של מתאם 5', ואת השמאלית Ts שלושה לאחר המחשוף להישאר כדי לסמן את המיקום של הזנב poly(A). כי 5' והן 3' מתאמים המחוברים על ידי מצדו הנמען 5' הקצוות, הדימרים מתאם לא נוצרים. ארבעה נוקלאוטיד אקראי-שהרובוט הציג בראשית קורא מאפשר רזולוציה אשכול בכלים רצף המדינה-of-the-art, יכול לשמש גם מזהה ייחודי מולקולרית (UMI) באיתור ובהסרה של PCR הגברה חפצים. הגודל של UMI ניתן להגדיל עוד יותר כפי שנעשה מחקרים אחרים 6. הפרוטוקול יוצר קריאות שמבטלות משלימים mRNA 3' הקצוות, הכל מתחיל עם tetramer אקראי ואחריו 3 Ts. עיבוד של קורא את יש את 3 Ts האבחון שלהם 5' סוף מתחיל התיקון של ה-PCR הגברה חפצים על ידי ניצול של UMIs, הסרה של 3' מתאם רצפים, ולהפוך קומפלמנטציה. קריאות כי ייתכן שיש מקורם oligo(dT) לקרקע באתרים A עשיר פנימי גם מזוהה שהמפתחות, יימחקו. האתרים כדין בדרך כלל חסר אחד 18 מאופיין היטב וממוקם אותות poly(A) שנשמרת שאמור להיות ~ 21 נוקלאוטידים במעלה הזרם של האתר המחשוף לכאורה 7.
הפרוטוקול דורש כ 8 הפעם תרגולים h, לא כולל תרבית תאים, את ligations לילה. המשויך לקרוא ניתוח תוכנה מאפשרת זיהוי אתר של poly(A) ומדויקים. מתוך אתר poly(A) אשכולות שנוצרו בהתבסס על דגימות 4 מודגשת עוד יותר את כתב היד (שני ביולוגי ושכפול של שליטה siRNA ותאים סי HNRNPC-שטופלו) 84% חפיפה עם ג'ין המבואר, ועל אלה, 75% חפיפה עם 3' UTR, 86% עם כל אחד 3' UTR או של אקסון מסוף. מקדם המתאם פירסון של ביטוי של 3' קצוות הדגימות שכפל 0.92, הערכים של מעל 0.9 מתקבלים בדרך כלל עם השיטה. לפיכך, A-seq2 היא שיטה נוחה זה נותן תוצאות מאוד לשחזור.
Protocol
1. צמיחת תאים, ה-mRNA בידוד
- לגדול תאים על פי ניסיוני בעיצוב טוב 6 צלחות ~ 1 x 10 6 תאים לכל במעגן למפגש 80%.
- להסיר את מדיום הגידול ולשטוף את התאים פעם עם פוספט buffered מלוחים. ישירות lyse התאים בצלחת על-ידי הוספת 1 מ"ל של פירוק מאגר הערכה mRNA-בידוד. להעביר את צמיגה lysate לתוך צינור פלסטיק 15 מ"ל עם טיפ פיפטה 1 מ"ל. להשתמש במרית גומי לנתק לחלוטין את החומר תאים מפני השטח צלחת-
- להטות הדי צמיגה המכילה lysate עם מזרק 1 מ"ל המצורפת מחט תת-עורית 23 גרם על ידי כמה נמרצת לאורך תנועות הבוכנה עד lysate זה כבר לא צמיגה. הצבע המחט מזרק במרכז החלק התחתון כדי למנוע הוצאת lysate יצאה מהשפופרת.
- להעביר את lysate לתוך צינור 1.5 mL באמצעות המזרק. ספין 5 דקות ב 20,000 x g ו- C ° 4 לפנות את הפסולת. להשתמש DNA bind נמוך 1.5 mL הבקבוקונים בכל הפרוטוקול.
- בזמן לצנטריפוגה פועל, לשטוף µL 300 של resuspended oligo (dT) 25 beads מגנטי על מתלה מגנטי עם 500 µL פירוק המאגר. מערבבים את צינורות 2 - 3 פעמים על המדף. הסרת המאגר לאחר הפתרון ברור. לאסוף את תגובת שיקוע ברור מהשלב 1.4 ולהוסיף החרוזים. Resuspend ומניחים צינורות על הגלגל מסתובב במשך 10 דקות
- במקום הצינורות על מתלה מגנטי. הסר את נוזל שקוף לאחר 2 דק הוסף 0.8 מ ל מאגר של ערכת ה-mRNA-בידוד. הפעל את הצינור ב- 180° מעלות על המדף, 2 - 3 פעמים. חזור על שלב זה כביסה פעם נוספת עם מאגר א
- לרחוץ את החרוזים 2 פעמים עם 0.8 מ של מאגר B כפי שמתואר בשלב 1.6.
- כדי elute את ה-mRNA מאוגד של החרוזים, הוסף 33 µL H 2 O ' resuspend את החרוזים. מחממים עד 75 מעלות צלזיוס למשך 5 דקות על גוש מחוממת. מיד לסובב את הצינורות עבור 1 s ומקום אותם על מתלה מגנטי. להעביר את תגובת שיקוע צינור חדש. דוגמאות שניתן לאחסן ב- 80 ° C עד שימוש נוסף. µL
- מאגר הידרוליזה אלקליין µL 66 להוסיף את ה-33 mRNA (שלב 1.8), לערבב וחום בדיוק 5 דקות ב 95 מעלות צלזיוס על בלוק חימום. תירגע מיד צינורות על קרח
- לבודד RNA עם ערכת ניקוי RNA.
הערה: לאשר את אמצעי האחסון; זה צריך להיות 100 µL.- להוסיף 350 µL RLT מאגר הערכה, 250 µL אתנול. לטעון עמודה ועל ספין בשביל 30 s ב 8000 g x בטמפרטורת החדר (RT). לשטוף עם 500 µL RPE מאגר הערכה. לשטוף עם 500 µL 80% אתנול. ספין למשך 5 דקות ב g x 20,000 לייבש את העמודה. להוסיף 36 µL H 2 O עמודה, ספין 1 דקות ב- 20,000 x g... למחוק את העמודה, להציל את eluate.
2-5 ' לסיים את זרחון היתר וטיפול DNase
- להוסיף 5 µL polynucleotide קינאז מאגר, 5 µL 10 מ מ ATP, 1 µL ריבונוקלאז, 1 µL DNase ו- µL 2 polynucleotide קינאז כדי דוגמאות של דגירה ב 37 מעלות צלזיוס במשך 30 דקות להכין באופן אופציונלי תגובת האב mixes לאורך כל הפרוטוקול על ידי ערבוב כרכים 1.1 x n (n = מספר דוגמאות) של כל מרכיב.
- לשנות מאגר ולהסיר ATP על עמודה-ספין כדי למנוע poly(A) תוספת בשלב הבא.
- Prespin ספין-עמודות ב- g 735 x עבור מינימלית 1 להעביר את העמודות בקבוקונים 1.5 mL החדש וטען את התגובות קינאז על גבי העמודים. ספין העמודות 2 דקות ב ג x 735 למחוק העמודות ו במקום הצינורות עם תגובות שנאספו על הקרח או לאחסן ב-80 מעלות צלזיוס
3. חסימת 3 ' מסתיים Cordycepin טריפוספט
הערה: זה חיוני כדי לחסום את 3 ' הקצוות של קטעים RNA להימנע שלהם concatemerization תגובות עוקבות מצדו. 3 ' מסתיים כי לא נחסמים כבר על ידי ( פוספט מחזורית) לאחר הידרוליזה מטופלים על ידי תוספת של 3 ' dATP (cordycepin טריפוספט) שרשרת המחסל נוקלאוטיד בסיועם של poly(A) פולימראז. כאן, שמרים poly(A) פולימראז (yPAP), כי היה בא לידי ביטוי טהור כפי שמתואר 8 שימשה-ריכוז של 0.5 מ"ג/מ"ל. שמרים או e. coli PAP שני יש כמעט את אותה פעילות עבור תוספת של 3 ' dATP ניתן לרכוש מסחרית (ראו טבלה של החומרים).
- µL 13.5 להוסיף 5 x poly(A) מרוכז מאגר התגובה פולימראז, 2 µL 10 מ מ 3 ' dATP, מעכב RNase 1 µL ו- 1 µL poly(A) פולימראז כדי מהתגובה צעד 2.2.1. מערבבים ו ספין 1 ס' Incubate ב 37 מעלות צלזיוס במשך 30 דק להוסיף 32.5 µL H 2 O על כל תגובה. לטהר את הרנ א כמו שלב 1.10.1. Elute את הרנ א עם 14 µL H 2 O.
4. מצדו של 3 הפוכה ' מתאמי 5 ' קצה של RNA מקטעי
- מקום לתגובות מיכשור 10 דקות כדי להקטין את עוצמת הקול כדי 6 µL. להוסיף 3 µL 10 x T4 RNA מצדו מאגר, 3 µL 10 מ מ ATP , 15 µL PEG-8000, מעכב RNase 1 µL µL 1 של 0.1 מ"מ הפוכה המשלים 3 ' מתאם " revRA3 " (ראה טבלה של החומרים) ומערבבים µL 1 ריכוז גבוה רנ א ליגאז 1,.
- תדגור התגובות ב 24 ° C עבור 16 h במיקסר מחוממת עם ערבוב לסירוגין-1000 סל ד להוסיף 70 µL H 2 O כל התגובה ומערבבים. לטהר את הרנ א כמו שלב 1.10.1. Elute את הרנ א עם 14 µL H 2 O. דוגמאות שניתן לאחסן ב-80 מעלות צלזיוס בנקודה זו.
5. הפוך שעתוק (RT)
- המקום eluates ב רכז ואקום במשך 3 דקות כדי לצמצם את אמצעי האחסון 11 µL. העברת התגובות 200 µL PCR צינורות. להוסיף 1 µL 0.05 מ"מ RT פריימר " ביו-dU-dT25 ". חום במשך 5 דקות ב- 70 מעלות צלזיוס הצנטרפוגה PCR ולהשאיר RT עבור 5 דק
- הוסף 1 dNTPs 10 מ מ µL, 4 µL 5 x רוורס טרנסקריפטאז מאגר, 1 µL 0.1 M DTT, מעכב RNase µL 1 1 µL רוורס טרנסקריפטאז. לערבב וחום התגובות למשך 10 דקות עד 55 ° C ו- 10 דקות עד 80 ° C ב הצנטרפוגה PCR. לשמור על הקרח או ב-80 מעלות צלזיוס לאחסון ארוך יותר.
6. עיכול במיקס האנזים DNA Glycosylase אורציל
- µL Pipet 100 Streptavidin-חרוזים לתוך בקבוקון 1.5 mL, resuspend במאגר איגוד ביוטין µL 800 ומניחים על מתלה מגנטי. היפוך צינורות 2 - 3 פעמים. הסר את המאגר כאשר ברור. חזור על השלב כביסה. Resuspend את החרוזים במאגר איגוד ביוטין µL 200-
- הוסף תגובה שעתוק במהופך הפתרון חרוזים, דגירה 20 דקות ב 4 ° C על גלגל מסתובב. תשטוף החרוזים 2 x עם איגוד ביוטין מאגר כמו צעד x 6.1 ו- 2 עם מאגר עשר על מתלה מגנטי. Resuspend את החרוזים במאגר עשר µL 50, להוסיף 2 µL DNA אורציל glycosylase האנזים לערבב אותם דגירה h 1 ב 37 מעלות צלזיוס במיקסר עם ערבוב לסירוגין. מאגר
- להוסיף 50 µL H 2 O, µL 11 של RNase H, µL 1 RNase H לתגובות. דגירה ב 37 מעלות צלזיוס במשך 20 דקות צינורות המקום על מתלה מגנטי ולהעביר את הנוזל המכיל את cDNA cleaved לרכבת התחתית החדשה
- לטהר את cDNA cleaved.
- µL 550 להוסיף מאגר PB מתוך ערכת טיהור PCR לתגובות המחשוף. להוסיף 10 µL של 3 מ' סודיום אצטט, pH 5.2 כדי להוריד את רמת ה-pH. לטעון את התגובות • תנאי מינימלי ספין עמודות ו ספין-17,000 g x עבור מינימלית 1
- µL 750 להוסיף מאגר PE עמודות ו ספין-17,000 g x עבור מינימלית 1 למחוק את הזרימה דרך. לסובב את העמודות ב- 17,000 g x עבור 1 דקות לייבוש. להעביר את העמודות בקבוקון 1.5 mL, להוסיף 16 µL H 2 O ו ספין-17,000 g x עבור מינימלית 1 מקום לתגובות concentrator ואקום במשך 8 דקות להתרכז לאמצעי אחסון של 7 µL.
7. מצדו של 5 ' מתאמים ל- 5 ' קצוות cDNA
- כדי cDNA מבודד, להוסיף 3 µL 10 x T4 RNA ליגאז 1 מאגר, 3 µL 10 מ מ ATP, 15 µL PEG-8000, 1 מיקרומטר µL 50 " revDA5 " oligo , ו- 1 µL ריכוז גבוה T4 RNA ליגאז 1. דגירה ב 24 ° C עבור ה 20 להוסיף 70 µL H 2 O על כל תגובה. דוגמאות שניתן לאחסן ב-20 ° C בשלב זה.
8. טייס ה-PCR, הגברה של ספריות ובחירת גודל
- בתגובה פיילוט, לקבוע מספר אופטימלי של ה-PCR מחזורי כדי להגיע אל הספריה הגברה בטווח לשלב מעריכית.
- Pipet 25 µL DNA פולימראז מיקס, 20 תגובה מצדו µL, 2 µL H 2 O, 1.5 µL 10 מיקרומטר קדימה PCR פריימר (RP1) ו 1.5 µL 10 מיקרומטר הפוכה PCR מדד פריימר לתוך צינור PCR µL 200-
- לרוץ הצנטרפוגה עם התוכנית הבאה: 3 דקות 95 ° C, ואחריו 20 מחזורים של 20 s 98 ° C, 20 s 67 ° C ו- s 72 30 ° C. לאסוף 7 aliquots µL לאחר 6, 8, 10, 12, 14, 16 ו 18 מחזורים ישירות מתוך הצנטרפוגה. להוסיף 1 µL 10 x טעינת מאגר (50% גליצרול, 0.05% קסילן cyanol). הערה: נא פעל בהתאם להמלצות של הספק, אם באמצעות ריבוב כאשר שילוב של ברקודים.
- מוצרים נפרדים בחריצים קטן על ג'ל agarose 2% 1 x TBE מאגר המכיל 1:10, 00 דילול של הפלורסנט ירוק.
- טען aliquots על agarose 2% ג'ל ולהפעיל את הג'ל ב100 וולט במשך 15 דקות Visualize העברה של מוצרי ה-PCR על מערכת תיעוד ג'ל.
- השתמש במספר מחזורים בתחילת הגברה מעריכי התגובה פיילוט לתגובה PCR בקנה מידה גדול עם פעמיים את הכרכים משמש עבור התגובה פיילוט ( איור 2).
- עבור תגובות PCR בקנה מידה גדול, להתרכז, desalt את התגובות קודם עם ערכת טיהור PCR, להפריד את המוצרים על משבצות רחב על 2% agarose ג'לים במאגר x TBE 1-
- לחתוך פרוסות ג'ל המכיל 200-350 nt DNA מוצרים. ממיסים את הג'ל במאגר של chaotropic ב RT למשך עד 30 דקות. לחלץ את ה-DNA מ הפרוסות ג'ל עם ערכת חילוץ ג'ל. שלא לחמם עד 50 ° C כדי למנוע הטיה באיגוד של דנ א-עשיר 9.
- הגשה על רצף.
הערה: בדרך כלל, 50 מחזורים יחיד-קריאה (SR50) מספיקים (ראו, למשל, https://www.illumina.com/technology/next-generation-sequencing.html).
9. עיבוד נתונים
הערה: רצף הנתונים שיתקבלו (בפורמט fastq) מעובדים עם תוכנה הזמינים במאגר gitlab (https://git.scicore.unibas.ch/zavolan_public/A-seq2-processing). הניתוח כולל ארבעה שלבים עיקריים: (1) הורדת המאגר לגית, (2) התקנה של סביבה וירטואלית, (3) הגדרת פרמטרים ספציפיים בקובץ התצורה ואת (4) משיקה את הניתוח דרך ‘ snakemake ’ 10. הניתוח כולו נעשה בשלב 4 דורש פקודה אחת בלבד. ניתן למצוא תיאור מפורט שלב אחר שלב של הניתוח בקובץ ה-README gitlab במאגר וזמינה תיאור קצר מתחת. כל שלבי עיבוד בודדים מושגת על ידי הביצוע של כלי זמין לציבור, גם ממקורות חיצוניים או מוכן בתוך הבית. הצינור חישובית תלוי מבוססת-אנקונדה 11 פיתון 3 וירטואלית סביבה בעלת זמינות חבילת 10 snakemake. היא פועלת במחשבים עם מערכת הפעלה דמויית-יוניקס, נבדקה סביבת לינוקס עם התקנת מערכת ההפעלה CentOS 6.5 ו 40 GB RAM זמין. תוכנה תלות נשלטים באופן אוטומטי בתוך הסביבה הוירטואלית. כלי תוכנה זמין לציבור הבאים הם נדרש ו ובכך מותקן יחד עם הסביבה: snakemake (v3.9.1) 10, ערכת הכלים (v0.0.14) fastx 12, כוכב (v2.5.2a) 13 , cutadapt (v 1.12) 14, 14 , samtools (v1.3.1) 15, 16 , bedtools (v2.26.0) 17.
- עיבוד נתונים מראש של קריאות ל cDNAs
הערה: העומק רצף עשויים להשתנות בין פועל, בהתאם לכלי-ניתן לפצל נתונים מ דוגמא אחת על פני רצף קבצים מרובים. אם זה המקרה, לשרשר את הקבצים התואמים דוגמא אחת לתוך קובץ קלט יחיד המשמש בשלבים הבאים.- להמיר את הקובץ מ fastq לתבנית fasta.
- תמצית קורא עם מבנה הנכון (3 thymidines ומעמדות 5, 6 ו-7 של הקריאה).
הערה: קריאה המוכנה כראוי על פי פרוטוקול הניסוי המתואר לעיל צריך המבנה (בין 5 ' סוף): 4-נוקלאוטיד ברקוד - 3 thymidines - הפוך המשלים של התעתיק 3 ' סוף. - לאחסן מידע אודות tetramer המוצא הקו תיאור של הרצף.
הערה: tetramer משמש כמזהה ייחודי מולקולרית (UMI) המקל על התיקון של הגברה חפצים בהמשך הניתוח. - להסיר את שבעת נוקלאוטידים קרא ' s 5 ' סוף.
- תקן עבור הגברה חפצים על-ידי שמירת עותק אחד בלבד של הקריאות עם אותו הוספת רצף ו- UMI-
- הסרת החלק של 3 ' סיום התואם את רצף מתאם ואת הפוך ואז משלים הרצף. רק להמשיך עם קריאות בעלות אורך מינימלי (ברירת מחדל: 15 nt).
הערה: בהתאם לאורך השבר mRNA המקורי ואת המספר של רצף מחזורי, 3 ' סוף קרא עשוי להכיל חלק 3 ' מתאם, אשר יוסר בשלב זה.
- לחלץ קריאות כל העומדים בקריטריונים הבאים: מקסימום 2 נוקלאוטידים לא ידוע (' N '), מקסימום 80% כמו ולאחר נוקלאוטיד האחרון של הקריאה לא א קריאות אלה נחשבים באיכות מספיק כדי לשמש בניתוח.
- למפות את הקריאות הגנום עם כלי מטפל קריאות משולבים, מייצרת קובץ הפלט בפורמט בום.
- כוכבים אם משמש, צור קובץ עם האינדקס של הגנום שאליו ניתן למפות את הקריאות. על הגנום האנושי, שלב זה דורש 35 ג'יגה-בתים של זיכרון (RAM).
- את הקריאות למפות הגנום.
הערה: (הערות ספציפיות כוכב) קלים-מסיכה ללא זמין כדי לאלץ את המיפוי של 3 ' הסיום של כל אחד לקרוא כמו זה נוקלאוטיד מיד במעלה הזרם של אתר מחשוף.
- להמיר את בום לקובץ-מיטה. אם לקרוא מפות במספר מיקומים, לשמור רק אלה עם הנמוך ביותר לערוך מרחק.
הערה: המספר עותק של קרא ממופה במיקום ספציפי משמש הציון. קריאות הממפות מיקומים מרובים נספרות fractionally בכל מקום עם משקל שווה ל- 1 או המספר של מיקומים שאליהם מפות קריאה.
קריאות - התמוטטות להשתנות על-ידי שגיאת רצף סביר. אם שתי קריאות שונות למפות באותו המיקום (מיקום ההתחלה והסיום המיפויים זהים) והם חולקים את UMI באותו, מחשיב אותם PCR כפילויות, רק אחד.
- יסיק pre-mRNA בודדות כל 3 ' סוף אתרי עיבוד.
הערה: קריאה בודדים מספק ראיות 3 ' מסתיימת כאשר נוקלאוטידים ארבע שלו ממופים הגנום ללא שגיאה. המיקום שאליו 3 ' סוף המפות קריאה מאוחסן האתר מחשוף. - זיהוי 3 ' סוף אתרים יכול להיות שמקורם מן לקרקע פנימי. מגדירים את האתר החפץ הפנימי לקרקע כאשר 10 nt במורד הזרם של אתר המחשוף הגנום לספק אחד הקריטריונים הבאים: מכיל יותר משישה כמו, מכיל 6 ברציפות כמו או מתחילה באחד tetramers הבאים: AAAA, AGAA, AAGA, AAAG .
- ליצור טבלה של 3 בודדים ' סוף אתרי עיבוד בתבנית למיטה.
- זיהוי מוסדר באופן עצמאי אשכולות באתר poly(A).
הערה: השלבים המתוארים כאן בצע את ההליך שהוצגה ב פרסום מראש 5.- להתחיל על-ידי איסוף 3 בודדים ' סוף אתרי עיבוד התקבלו אצל כל דוגמאות של המחקר-
- ביאור אותות ידועים poly(A) 7 באזור של-60 כדי +10 נוקלאוטידים סביב כל 3 בודדים ' סוף עיבוד אתר.
- Poly(A) זיהוי אתרים הביעו מעל הרקע בכל מדגם כדלקמן.
- למיין את האתרים שלהם ביטוי raw בתוך המדגם הנוכחי. חוצים את רשימת האתרים מלמעלה למטה, שיוך אתרים מדורגת נמוך אתר מדורגת גבוה יותר אם הם ממוקמים במרחק מוגדר מראש הגנום (ברירת מחדל: למעלה nt 25 - או במורד הזרם) מאתר בכיר.
הערה: כל האתרים בדרג נמוך המשויך אתר בכיר להגדיר אשכול אשר הביטוי הוא מספר הקריאות המתעדים את כל האתרים האלה. - למיין אשכולות אלה לפי הביטוי וחוצות את הרשימה של אשכולות מהגבוהה לביטוי הנמוך, קביעת הסף ביטוי c שבו האחוז של אשכולות עם poly(A) מוערת אותות ירידה מתחת (הסף המוגדרות מראש ברירת מחדל: 90%).
- התעלם מאתרים כל אשכול מתחת החיתוך.
- למיין את האתרים שלהם ביטוי raw בתוך המדגם הנוכחי. חוצים את רשימת האתרים מלמעלה למטה, שיוך אתרים מדורגת נמוך אתר מדורגת גבוה יותר אם הם ממוקמים במרחק מוגדר מראש הגנום (ברירת מחדל: למעלה nt 25 - או במורד הזרם) מאתר בכיר.
- אשכול היטב במרווחים 3 ' סוף אתרי שהושג בין מדגמים.
הערה: מיון 3 ' ספירת קריאה של סוף עיבוד אתרי תחילה לפי מספר התומכים דגימות ולאחר מכן לפי סכום מנורמל (קורא לכל מיליון (סל ד)) מדגמים. חוצים את הרשימה מלמעלה למטה, שיוך המדורגים התחתונה אתרי אתרי המדורגים גבוה יותר כאשר המרחק שלהם לאתר בדרגה גבוהה אינו גדול יותר מאשר גבול מוגדר מראש (ברירת מחדל: 12 nt). בכל פעם כל אחד 3 קונסטיטוטיבי ' סוף האתר חופף עם אות המבואר poly(A) או יש אות poly(A) ישירות במורד הזרם, באשכול המקביל מסומן לבדיקה נוספת לזהות לקרקע פנימי. - מיזוג poly(A) באתר אשכולות.
הערה: כאשר אשכול מסומן בתור מועמד בשם לקרקע פנימי, זה התמזגו לתוך הזרם אשכול אם שני האשכולות לחלוק את אותות poly(A) שלהם או אם האתר. ביותר במורד הזרם באשכול יש אות poly(A) ממוקם לכל הפחות מרחק במעלה הזרם (ברירת מחדל: 15 nt). לבסוף, אשכולות צפופים ימוזגו אם: (i) הם חולקים את אותו signal(s) poly(A), או (ii) אורך של האשכול וכתוצאה מכך אינו עולה על מקסימום (ברירת מחדל: 25 nt). - לאחסן אשכולות בתוך תבנית קובץ מיטה עם הסכום הכולל מנורמל ספירת קריאה 3 כל ' לסיים אתרים כל אשכול כמו הציון.
תוצאות
פוליפוני (א)-המכיל RNA היה מבודד מן התאים בתרבית, מפוצל על ידי הידרוליזה אלקליין, cDNAs נעשו על ידי הפוך תמלול עם תחל oligo(dT). CDNA וכתוצאה מכך היה מתאושש על streptavidin חרוזים, dU היה ביקע ב התגובה כריתה מסוים אורציל, מתאמי היו מאתרים כדי 5' ו 3' קצוות השבר cleaved ושל השרוול היו וסודרו. איור 1 מציג קו מיתאר גרפי של הניסוי.
עבור תאים הלה ו- HEK293, 106 תאים היו מספיקות לזהות אתרי poly(A) עבור הרוב המכריע של חלבונים גנים בסופו של ההליך. עם זאת, עבור סוגי תאים או רקמות שייתכן צורך לבדוק את הרוויה במספר אתרים poly(A) מזוהה כמספר של תאים המשמשים את הניסוי אחרים מגדילה. תוצאות נציג ה-PCR פיילוט צעד, של המקטע דנ א ניתוח המדגם לפני רצף מוצגים באיור2.
איור 3 מראה את השלבים מראש עיבוד של ניתוח חישובית, החל מקובץ fastq המתקבל הרצפים (sequencer) ומסתיימת בדקנו איכות, חיתוך מתאם קריאות לפיהן ניתן למפות את הגנום. איור 4 מציג את השלבים ניתוח המתחילות המיפוי הקריאות הגנום ולסוף המתאים עם הקטלוג של mRNA 3' end עיבוד אתרים המזוהים ב מדגם מסוים. כאשר מספר דוגמאות מנותחות, פעולות נוספות מתבצעות כדי להתאים את הקצה 3' עיבוד אתרים שנמצאו דוגמאות בודדות ולדווח שלהם שפע מדגמים. השלבים הבאים מוצגים באיור5.
לכן, ברגע שיש כבר וסודרו דגימות, הניתוח של הרצף וכתוצאה מכך. לקרוא את הקבצים (בפורמט fastq) דרך צינור עיבוד זמין היא פשוטה. לאחר הוספת מידע אודות הדגימות לקובץ התצורה, ההוצאה להורג של הצינור תגרום שני סוגים עיקריים של קבצי פלט: 1) מיטה-קבצים עם כל 3' סוף אתרי עיבוד מזוהה בדגימות בודדים (למשל " sample1.3pSites.noIP.bed.gz"), ו- 2) מיטה-קובץ עם כל אשכולות באתר poly(A) (clusters.merged.bed) בכל הדוגמאות של המחקר. הפלט כולל גם את נקודות הציון של הגנום עבור כל פעולות הקריאה של כל דגימה בודדת (למשל "sample1. STAR_out/Aligned.sortedByCoord.out.bam") אשר מאוחר יותר ניתן להציג בדפדפן הגנום igv ב16. בדיקה ויזואלית של פרופילי קרא בדרך כלל מספק הצצה ראשונה של ההתפלגות poly(A) אתרים הגנום, השינויים המתרחשים בעת לפליטת ספציפיות שבוצעו במסגרת המחקר. לדוגמה, באיור 6 מוצג התגובה של גנים ספציפיים הדפיקה למטה של החלבון HNRNPC.
סיכומים של הפצות הגנום כולו אלה הינם גם מסופקים (טבלה 1). באופן ספציפי, קבצי פלט בספריה "ספירות/annotation_overlap" מכילים שברים של אתרים חפיפה עם תכונות המבואר ספציפיות (מהקובץ gtf סיפק כקלט; מבואר מסודרים: 3' UTR, אקסון מסוף, אקסון, אינטרון, intergenic). לבסוף, עבור כל דגימה, התוצאות של שלבי עיבוד בודדים גם נשמרים (למשל "sample1.summary.tsv"). זה כולל את המספרים של: קריאות raw כל דגימה, קריאות בעלות המבנה הצפוי בקצה 5', קריאות שנותרו לאחר כיווץ כפילויות PCR מלא, באיכות גבוהה קורא על-פי הקריטריונים שהוגדרו בשלב 9.2, קורא את המפה באופן ייחודי כדי הגנום (לאחר כיווץ אלו שנבעו רצף שגיאות, ראה שלב 9.5), מיפוי מרובה קורא (לאחר כיווץ אלו שנבעו רצף שגיאות, ראה שלב 9.5), raw (לא מקובצים באשכולות) 3' סוף עיבוד אתרים בשני צדדיו דוגמה, raw 3' עיבוד אתרים בלי מועמדים פוטנציאליים לקרקע פנימי, ייחודי 3' סוף אתרי עיבוד של כל הדגימות בלי מועמדים פנימיים לקרקע, קבוצה סופית של poly(A) אתר אשכולות.
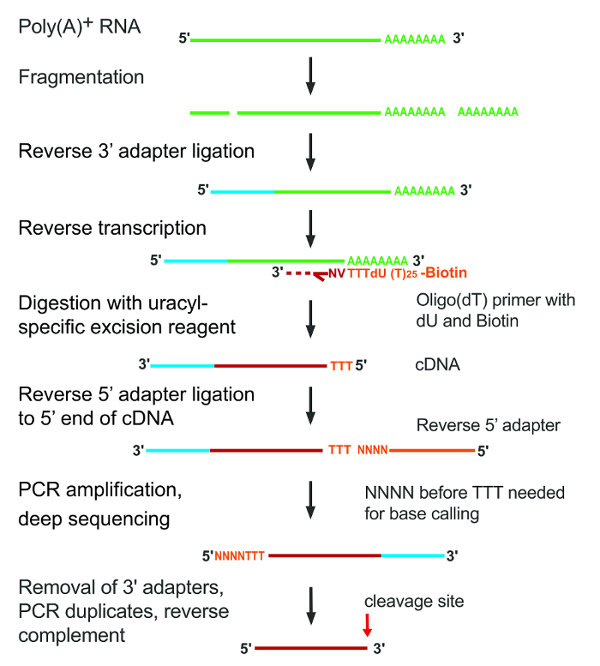
איור 1: שלבים עיקריים של פרוטוקול א-seq2. צעדים בודדים נקובים על הצד השמאלי של האיור. הוספה RNA מקטעי מתוארים בקווים זה הופך לאדום עבור cDNA לאחר שעתוק במהופך; מתאמי הם בצבע כחול בהיר או תפוז. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
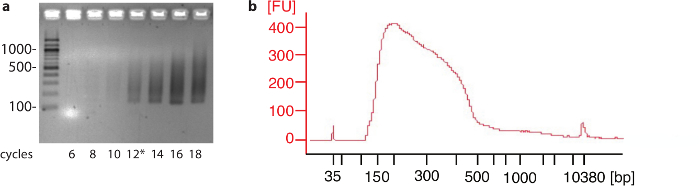
איור 2: טייס ה-PCR ופרופיל המוצר הסופי- () Aliquots מן התגובה PCR היו שנאספו מחזורים שונים ומופרדות על 2% agarose ג'לים. המספרים שמאלה מציינים גודל נוקלאוטידים הלהקות בהתאמה בסולם הדנ א. בניסוי זה נבחרו 12 מחזורים (*) עבור התגובה PCR בקנה מידה גדול. (b) דוגמה של מדגם לאחר בחירת גודל לרוץ על מנתח גודל המקטע חושפני עם גודל ממוצע של נוקלאוטידים בסביבות 280. המספרים שמאלה [פו] מציינים עוצמת האות יחסית. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
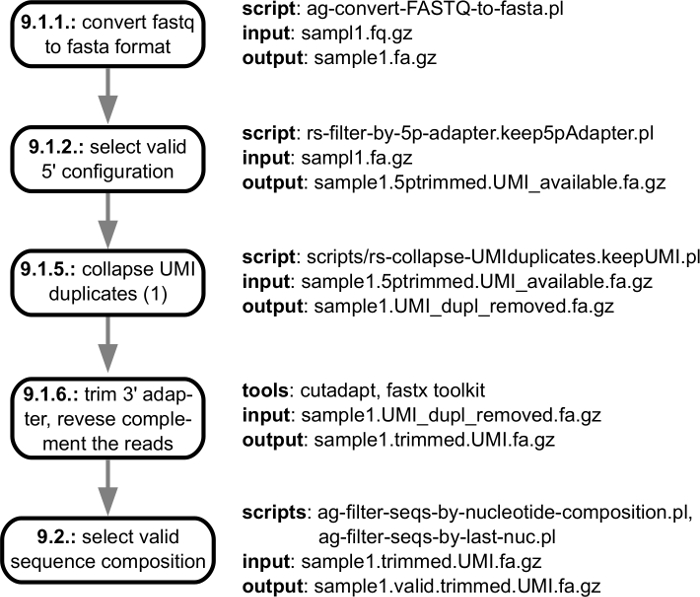
איור 3: חלוקה של עיבוד קדם רצף קריאות. הקבצים fastq עם קריאות הנוצרים על-ידי התוכנה כלי-הקשורים רצף מעובדים לזהות קריאות באיכות גבוהה זה ניתן למפות את הגנום המתאימים. האיור מציג את המפרט קלט/פלט של צעדים בודדים בצנרת, עם קישורים אל המדרגות בודדים של הפרוטוקול המתואר בסעיף "עיבוד נתונים". אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
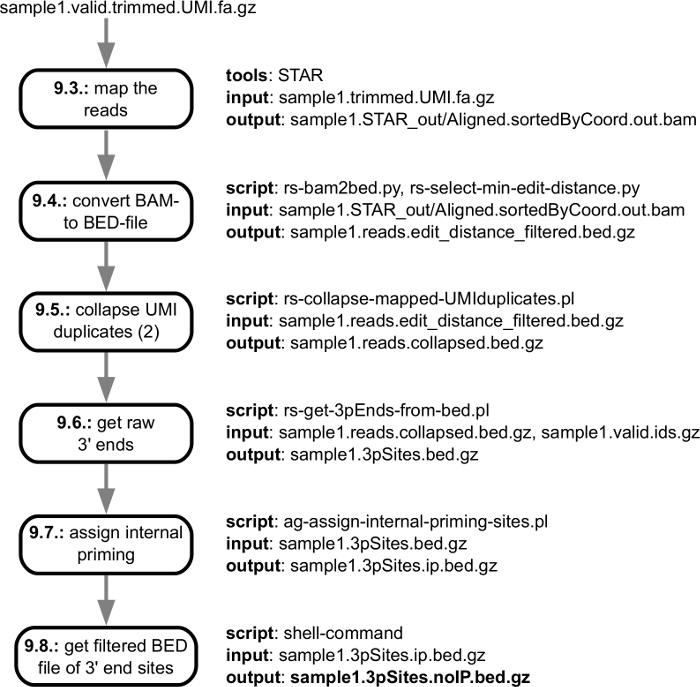
איור 4: חלוקה של רצף לקרוא עיבוד, מן השלב של מיפוי הגנום לדור של הפרט 3' סוף אתרי עיבוד. האיור מציג את המפרט קלט/פלט של צעדים בודדים בצנרת, עם קישורים iצעדים והקבוצתית של הפרוטוקול המתואר בסעיף "עיבוד נתונים". קובץ הפלט הראשי הנשלחת למשתמש מסומן באותיות מודגשות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
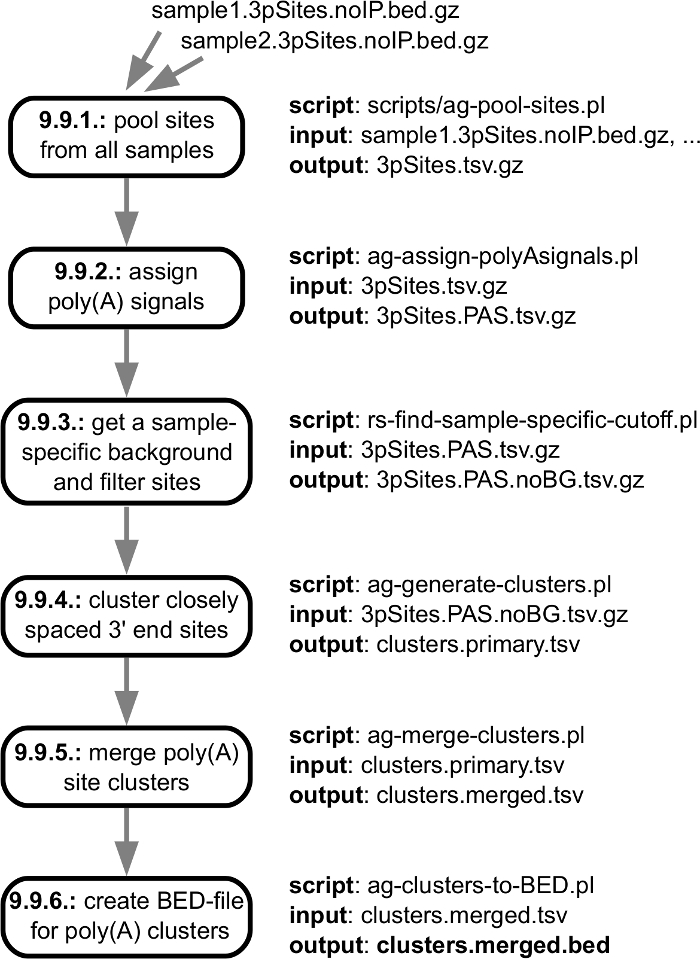
איור 5: קווי המתאר של הפעולות הננקטות כדי להפיק אשכולות של שיתוף מוסדר 3' סיום רצף אתרים- האיור מציג את המפרט קלט/פלט של צעדים בודדים בצנרת, עם קישורים אל המדרגות בודדים של הפרוטוקול המתואר בסעיף "עיבוד נתונים". קובץ הפלט הראשי מסומן באותיות מודגשות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
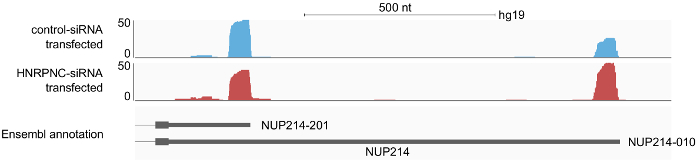
איור 6: תוצאות דוגמה לפרופיל של 3' לסיים את עיבוד קריאות לאורך אקסון מסוף של הגן NUP214, שמוצג בדפדפן הגנום 16 igv ב. A-seq2 קריאות הוכנו מדגימות שני תאים HEK 293, מטופלים עם שליטה-siRNA או עם siRNA HNRNPC. קריאות שתועדו אתרי poly(A) היו מוערת על-ידי הצבר ניתוח נשמרו בתבנית באם ששימש כקלט לדפדפן הגנום igv. הקצוות 3' של הפסגות לקרוא מפה 3' ה-mRNA מסתיים כי הם מבואר ב- Ensembl. הפרופילים מצביעים על שימוש מוגבר ארוך 3' UTR isoform על HNRNPC דפיקה למטה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| סי-בקרת שכפול 1 | סי-בקרת שכפול 2 | |
| קוד: 29765 | קוד: 32682 | |
| מספר הקריאות raw | 44210258 | 68570640 |
| מספר הקריאות בתוקף לאחר קיצוץ וסינון | 14024538 | 21211793 |
| מספר ייחודי מיפוי קריאות | 6953674 | 13946436 |
| מספר הקריאות מיפוי מנחלת מרובים | 2040646 | 2925839 |
| מספר בודדים 3' סוף אתרי עיבוד | 1107493 | 1710353 |
טבלה 1: דוגמה פלט של הצינור ניתוח. סיכומים של קורא את התקבלו ב צעדים בודדים.
Discussion
ריבוי של עזר הגורמים המעורבים בעיבוד pre-mRNA 3' הקצה והליבה משתקף נוף פוליאדנילציה מורכב בהתאמה. בנוסף, פוליאדנילציה הוא גם מגיב לשינויים בתהליכים אחרים כגון שעתוק ו שחבור. 3' סוף המחשוף אתרים ב- pre-mRNAs מזוהים בדרך כלל בהתבסס על זנבות poly(A) האופיינית הנוספים למוצרים המחשוף 5'. רוב שיטות להשתמש oligo(dT) תחל באורכים משתנים המאפשרות המרה ספציפי של פולי (א)-המכיל mRNAs ל cDNAs בתגובה שעתוק במהופך. בעיה נפוצה של גישה זו הוא פנימי לקרקע כדי רצפים א-עשיר וכתוצאה מכך המחשוף מלאכותית אתרים. הוצעו שתי שיטות שמטרתן לעקוף את החפץ הזה בשלב של הכנת הדוגמא. P 3-seq בשיטה 1, מתאמי במיוחד מאתרים בקצוות של זנבות poly(A) עם עזרה של oligo סד ואחריו לעיכול RNase T1 חלקי, שעתוק במהופך עם טרומבוציטופניה, בעקבות התגובות כמו deoxynucleotide היחידה. Heteroduplexes poly(A)-poly(dT) וכתוצאה מכך מתעכלים ואז עם RNase H, והשריד RNA הם מבודדים מאתרים למתאמים, וסודרו. פשוט ואלגנטי שיטה, P 2-seq, המשתמשת פריימר רצף מותאם אישית דילוג רצועת oligo(dT) שנותרו בעקבות רצף תגובות דווח על ידי מחברים אותו 2. בשיטת קשורים, 3' קורא 3, פריימר רב מהמשוער מתוך 5 לנו 45 Ts, מכיל גם של ביוטין הוא annealed ל RNA מפוצלים, ואחריו שוטף מחמירים כדי לבחור עבור מולקולות RNA עם poly(A) זנבות של נוקלאוטידים מעל 50. למרות 3' קריאות באופן דרסטי מפחית את התדירות של לקרקע פנימי, זה לא מונע לחלוטין את זה 3. גם יש כבר הציע פרוטוקולים עבור רצפי RNA ישיר, אבל קריאות וכתוצאה מכך הם קצרים ואם יש שיעור גבוה של שגיאה, גישה זו לא הייתה יותר מפותחת 18,19,20. תיקים עשויים-Seq, הפרוטוקולים חשבים Seq ממוסחר לשלב oligo(dT) מבוסס לקרקע עם צעד לקרקע אקראי עבור ה cDNA השני סטרנד סינתזה 20. השימוש של התגובה שעתוק במהופך בורר תבנית עם רוורס טרנסקריפטאז Moloney מאתר לוקמיה וירוס (MMLV) מוביל אל הדור של cDNAs עם linkers בשלב אחד, ובכך אין הדימרים מתאם יכול להופיע PAS-Seq ופעולות SAPAS 21 , 22.
בשיטת א-seq2 המובאת כאן בולט שלה ניצול נוקלאוטיד cleavable (דו) בתוך פריימר oligo(dT) biotinylated. שינוי זה משלב את התועלת של העשרת oligo(dT) hybridized, polyadenylated מטרות עם ההסרה של רוב הרצף25 oligo (dT) מן השברים מבודד לפני ספריות מוכנות ושמירה על שלושה Ts, אשר מצביעים על נוכחות מוקדמת של הזנב poly(A). לעומת זאת, שיטות לנצל RNase H כדי להסיר poly(A) של מולקולות RNA באקראי להשאיר מספר כמו. מאז ב- A-seq2, רצף נעשית מקצה 3' גדילים תחושה אנטי, המחשוף אתרים הם חזו את להיות ממוקם לאחר מוטיב NNNNTTT בראשית קורא רצף raw. Tetramers אקראי לשמש לא רק כדי לאפשר בסיס התקשרות אלא גם חיסול של PCR הגברה חפצים. גם ניתן לאכלס יותר UMIs. האפשרות לראשוניות פנימי נשאר ב- A-seq2, מופנית שהמפתחות, תחילה על-ידי מחיקת 3' מסתיים עם רצף במורד הזרם מקודד genomically, A-עשיר ולאחר מכן על-ידי מחיקת 3' סוף אשכולות שלא ניתן להסביר על ידי לקרקע פנימי- האות A עשיר poly(A) עצמה. ניתוח האחרונות של אתרי poly(A) להסיק באופן ייחודי על ידי מספר רב של פרוטוקולים מציין באתרים ייחודיים A-seq2 יש את התפלגות נוקלאוטיד הצפוי ואת המיקום בתוך גנים, הדומה אחרים 3' לסיים את רצפי פרוטוקולים.
שלב קריטי ב- A-seq2 הוא הבחירה של polyadenylated RNA הסרת ribosomal RNAs RNAs קטנים שונים. הדבר מתבצע בקלות הרבה ביותר על-ידי ערכת ה-mRNA-בידוד עם oligo (dT)25 beads מגנטי. בעקרון, הכולל RNA מבודדת עם פנול המכיל פתרונות גם נותן באיכות גבוהה RNA יכולה להיות עוד יותר נתונה לבחירה על ידי ערכת ה-mRNA-בידוד או oligo (dT) agarose. צעד זה יכולים להיות מגוונים ב- A-seq2 הוא הטיפול עם אלקליין הידרוליזה שניתן מקוצר או מורחב כדי להשיג RNA מקטעי בגדלים שונים. קריטי הוא גם תוספת של 3' dATP 3' הקצוות של קטעים RNA באמצעות פולימראז poly(A) זה יעיל. בפרוטוקול המתוארים כאן, טיפול זה מוחל על כל חלקי רנ א, כדי להימנע concatemerization במהלך התגובה מצדו. ולבסוף, נציין כי למרות רנ א ליגאז 1 משמש בדרך כלל ליגאז RNA, זה גם ligates ה-DNA נטושים יחיד ביעילות, כפי שעשינו כאן. מאתרים ומפסיקים להשתמש במתאם כדי 5' סוף cDNA מולקולות.
לכן, הוא א-seq2 יעיל וקל ליישם פרוטוקול לצורך זיהוי ה-pre-mRNA 3' סוף עיבוד אתרים. התפתחויות עתידיות יכולות לכלול שמצמצם עוד יותר את המורכבות את הפרוטוקול ואת כמות החומר הנדרש. קבוצת כלי ניתוח נתונים חישובית נוספים הקשורים מאפשרים עיבוד הומוגנית של 3' סיום רצף קריאות שהושג עם מגוון רחב של פרוטוקולים.
Disclosures
המחברים אין לחשוף.
Acknowledgements
המחברים תודה גברת Béatrice Dimitriades על העזרה עם התרבות תאים. עבודה זו נתמכה על ידי קרן המדע הלאומית השוויצרית מענקים #31003A_170216 ו 51NF40_141735 (NCCR RNA & המחלה).
Materials
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Agarose, ultra pure | Invitrogen | 16500-500 | |
| 2100 Bioanalyzer | Agilent | G2940CA | |
| Cordycepin triphosphate (3’ dATP) | SIGMA | C9137 | |
| DNA low bind vials, 1.5 ml | Eppendorf | 22431021 | |
| Dulbecco’s Phosphate Buffered Saline | SIGMA | D8637 | |
| Dynabeads mRNA-DIRECT Kit | Ambion | AM61012 | |
| GR-Green dye | Excellgen | EG-1071 | use 1:10,000 dillution |
| HiSeq 2500 or NextSeq 500 next generation sequencers | Illumina | inquire with supplier | |
| KAPA HiFi Hotstart DNA polymerase mix | KAPA/Roche | KK2602 | |
| Nuclease free water | Ambion | AM9937 | |
| Poly(A) polymerase, yeast | Thermo Fisher Scientific | 74225Z25KU | |
| Poly(A) polymerase, E.coli | New England Biolabs | M0276L | |
| Polynucleotide kinase | Thermo Fisher Scientific | EK0032 | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| RNA ligase 1, high concentration | New England Biolabs | M0437M | includes PEG-8000 |
| RNeasy MinElute RNA Cleanup kit | Qiagen | 74204 | |
| RNase H | New England Biolabs | M0279 | |
| RNasin Plus, ribonuclease inhibitor | Promega | N2618 | |
| Superscript IV reverse transcriptase | Thermo Fisher Scientiific | 18090050 | |
| Turbo DNase | Ambion | AM2238 | |
| USER enzyme mix | New England Biolabs | M5505 | |
| Dyna-Mag-2 magnetic rack | Thermo Fisher Scientific | 12321D | |
| Thermomixer C | Eppendorf | 5382000015 | Heated mixer with heated lid |
| MicroSpin columns | GE-Healthcare | 27-5325-01 | |
| Name | Company | Catalog Number | Comments |
| Buffers | |||
| Alkaline hydrolysis buffer, 1.5 x | Mix 1 part 0.1 M Na2CO3 and 9 parts 0.1 M NaHCO3. Add EDTA to 1 mM. Adjust pH to 9.2. Store aliquots at -20 °C. | ||
| 5x poly(A) polymerase buffer | Thermo Fisher Scientiific | 100 mM Tris-HCl, pH 7.0, 3 mM MnCl2, 0.1 mM EDTA, 1 mM DTT, 0.5 mg/ml acetylated BSA, 50% glycerol | |
| Biotin binding buffer | 20 mM TrisCl pH 7.5, 2 M NaCl, 0.1% NP40 | ||
| TEN buffer | 10 mM TrisCl, pH 7.5, 1 mM EDTA, 0.02% NP40 | ||
| Name | Company | Catalog Number | Sequence |
| Oligonucleotides according to Illumina TruSeq Small RNA Sample Prep Kits, for GA-IIx and Hiseq2000/2500 sequencers | Microsynth | ||
| revRA3 (RNA) | Microsynth | 5’ amino CCUUGGCACCCGAGAAUUCCA 3’ | |
| revDA5 | Microsynth | 5’ amino GTTCAGAGTTCTACAGTCCGAC GATCNNNN-3’ | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' (V = G, A or C) | |
| PCR primer forward, RP1 | Microsynth | 5' AATGATACGGCGACCACCGAGA TCTACACGTTCAGAGTTCTACAG TCCGA 3' | |
| PCR primer reverse, RPI1, barcode in bold | Microsynth | 5' CAAGCAGAAGACGGCATACGAG ATCGTGATGTGACTGGAGTTCCT TGGCACCCGAGAATTCCA 3' | |
| Name | Company | Catalog Number | Comments |
| Oligonucleotides according to Illumina TruSeq HT-Small RNA Sample Prep Kits, for HiSeq2000/2500 and NextSeq500 sequencers | |||
| HT-rev3A (DNA/RNA) | Microsynth | 5'-amino-GTGACTGGAGTTCAGACGTGTG CTCTTCCrGrAUrC-3' | |
| HT-rev5A | Microsynth | 5' amino-ACACTCTTTCCCTACACGACGCT CTTCCGATCTNNNN 3' | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' | |
| PCR primers forward (D501-506) | Microsynth or Illumina | 5'-AATGATACGGCGACCACCGAGAT CTACAC[i5]ACACTCTTTCCCTACA CGACGCTCTTCCGATCT -3' | |
| PCR primers reverse (D701-D712) | Microsynth or Illumina | 5'-CAAGCAGAAGACGGCATACGAG A[i7]GTGACTGGAGTTCAGACGTG TGCTCTTCCGATC-3' | |
| Documentation for Illumina multiplexing: | Illumina | https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/illumina-adapter-sequences_1000000002694-01.pdf |
References
- Jan, C. H., Friedman, R. C., Ruby, J. G., Bartel, D. P. Formation, regulation and evolution of Caenorhabditis elegans 3'UTRs. Nature. 469 (7328), 97-101 (2011).
- Spies, N., Burge, C. B., Bartel, D. P. 3' UTR-isoform choice has limited influence on the stability and translational efficiency of most mRNAs in mouse fibroblasts. Genome Res. 23 (12), 2078-2090 (2013).
- Hoque, M., Ji, Z., et al. Analysis of alternative cleavage and polyadenylation by 3' region extraction and deep sequencing. Nat. methods. 10 (2), 133-139 (2013).
- Martin, G., Gruber, A. R., Keller, W., Zavolan, M. Genome-wide analysis of pre-mRNA 3’ end processing reveals a decisive role of human cleavage factor I in the regulation of 3' UTR length. Cell Rep. 1 (6), 753-763 (2012).
- Gruber, A. R., Martin, G., et al. Global 3' UTR shortening has a limited effect on protein abundance in proliferating T cells. Nat. Commun. 5, 5465 (2014).
- Kivioja, T., Vähärautio, A., et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat. methods. 9 (1), 72-74 (2011).
- Gruber, A. J., Schmidt, R., et al. A comprehensive analysis of 3' end sequencing data sets reveals novel polyadenylation signals and the repressive role of heterogeneous ribonucleoprotein C on cleavage and polyadenylation. Genome Res. 26 (8), 1145-1159 (2016).
- Lingner, J., Keller, W. 3'-end labeling of RNA with recombinant yeast poly(A) polymerase. Nucleic Acids Res. 21 (12), 2917-2920 (1993).
- Quail, M. A., Kozarewa, I., et al. A large genome center's improvements to the Illumina sequencing system. Nat. methods. 5 (12), 1005-1010 (2008).
- Rahmann, S. Snakemake--a scalable bioinformatics workflow engine. Bioinformatics. 28 (19), 2520-2522 (2012).
- . Anaconda Software Distribution Available from: https://continuum.io (2016)
- Dobin, A., Davis, C. A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Li, H., Handsaker, B., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Robinson, J. T., Thorvaldsdóttir, H., et al. Integrative genomics viewer. Nat. Biotechnol. 29 (1), 24-26 (2011).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ozsolak, F., Platt, A. R., et al. Direct RNA sequencing. Nature. 461 (7265), 814-818 (2009).
- Yao, C., Biesinger, J., et al. Transcriptome-wide analyses of CstF64-RNA interactions in global regulation of mRNA alternative polyadenylation. Proc. Natl. Acad. Sci. U. S. A. 109 (46), 18773-18778 (2012).
- Lin, Y., Li, Z., et al. An in-depth map of polyadenylation sites in cancer. Nucleic Acids Res. 40 (17), 8460-8471 (2012).
- Shepard, P. J., Choi, E. -. A., Lu, J., Flanagan, L. A., Hertel, K. J., Shi, Y. Complex and dynamic landscape of RNA polyadenylation revealed by PAS-Seq. RNA. 17 (4), 761-772 (2011).
- Fu, Y., Sun, Y., et al. Differential genome-wide profiling of tandem 3' UTRs among human breast cancer and normal cells by high-throughput sequencing. Genome Res. 21 (5), 741-747 (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved