Method Article
3' 끝 시퀀싱 A seq2 라이브러리 준비
요약
이 프로토콜 매핑 사전 mRNA 3' 최종 사이트를 처리 하는 방법을 설명 합니다.
초록
지난 10 년간에서 연구 사전 mRNA 절단, polyadenylation 반응의 복잡 하 고 동적인 다양 한을 밝혀졌다. 반면 확산 셀 우선적으로 표현 하는 성적 증명서와 함께 짧은 3' Utr mRNAs 긴 3' 번역 되지 않은 영역 (Utr) 차별화 된 세포에서 생성 됩니다. A-seq 프로토콜 polyadenylation 사이트 게놈 넓은 고 사전 mRNA 3' 끝 처리의 규칙을 공부 하기 위해 개발 되었다 그것의 두 번째 버전에서 설명 합니다. 또한이 현재 프로토콜 활용 polyadenylate (poly(A)) 꼬리 완전히 처리 된 mRNAs에 대 한 풍부 하 대부분 포유류 mRNAs의 속은 동안 추가 됩니다. 4 위치에 deoxyuracil와 DNA 어댑터의 mRNA 3' 끝 조각 시퀀싱에 대 한 정확한 처리 수 있습니다. 세포 배양과 하룻밤 ligations, 포함 하 여 아닙니다 프로토콜에는 약 8 h 실습 시간이 필요합니다. 그것은, 함께 파생된 시퀀싱 데이터의 분석을 위한 사용 하기 쉬운 소프트웨어 패키지를 제공 됩니다. A seq2 및 관련된 분석 소프트웨어 제공 사전-mRNA 3'의 매핑 하는 효율적이 고 신뢰할 수 있는 솔루션 10에서 조건의 넓은 범위에서 끝나는6 또는 적은 셀.
서문
캡처 및 mRNA 3' 끝의 연속 mRNA 처리의 연구 및 유전자 발현의 정량화를 허용 한다. 때문에 그들의 많은 꼬리, 진 핵 mRNAs 수 효율적으로 정화 구슬 움직일 올리고-deoxythymidine (oligo(dT)) 분자, cDNA 합성 프라임도 수는로 총 세포 lysates에서. 그러나,이 방법은 두 가지 단점이 있다. 첫째, a의 성적을 내부 뻗어 cDNA 합성, 가짜 많은 사이트 결과 프라임 또한 수 있습니다. 두 번째, 균질 많은 뻗어 시퀀싱, 제외 되지 않는 사본 식별에 대 한 정보에 대 한 구체적인 과제를 포즈. 다양 한 방법을 통해 많은 반전 녹음 방송 등 이러한 제한을 우회 하 제안 되었습니다 꼬리 뒤에 RNase H 소화 (3 P-seq 1), 20에서 끝나는 사용자 지정 시퀀싱 뇌관의 사용 Ts (2 P-seq 2)의 미리 RNase H 소화 (3' 읽기 3), 그리고 머리 핀 (A-seq 4)에서 3' 어댑터 포함 된 oligo dT 뇌관의 사용 다음 CU5T45 뇌관 50 개 이상의 뉴클레오티드의 많은 꼬리와 RNA 파편.
최근에 개발 된 A seq2 방법 5 시퀀싱 및 어댑터, 특히 발생의 각자 결 찰에 의해 생성 되는 이합체의 비율을 최소화 하기 위해 동시에 많은 통해 우회를 목표로 때 어댑터의 어 금 니 농도 삽입 농도를 보다 큽니다. 두 어댑터는 동일한 유형의 polynucleotide 끝 A-seq2, 후 반전 녹음 방송 RNA 조각과 cDNAs의 5' 끝에 5' 어댑터의 5' 끝에 3' 어댑터는 출혈 하는 어디에서 출혈 하는 경우에이 문제를 제거할 수 있습니다. 방법은 우리의 이전 제안된 A seq-는 5'-3 시퀀스는 보다 더 편리 하 게 ' 그로 인하여 정확 하 게 요구 하는 방향 제어 RNA 조각-, 많은 사이트 확인의 높은 정확도 유지 하면서. 전형적인 샘플에서 시퀀스 읽기의 약 80% 고유는 게놈 지도 고 20000 이상의 많은 사이트 클러스터와 주석 3' Utr는 중복의 70% 이상의 식별.
간단 하 게, A seq2 프로토콜 mRNA 조각화 및 역 보수 3' 5' RNA 파편의 끝에 어댑터의 결 찰으로 시작합니다. 폴 리 (A)-포함 된 RNAs는 다음 역 3' 끝, 위치 4에서 뒤 끝에 5', biotin에 앵커 뉴클레오티드를 포함 하는 25 뉴클레오티드 (nt) 긴 oligo(dT) 뇌관 복사할 자기 streptavidin 구슬에 cDNA의 바인딩 허용. 뇌관, biotin, 등의 대부분 사용자 효소 혼합, Uracil DNA glycosylase (UDG)와 DNA glycosylase lyase Endonuclease VIII를 포함 하 여 분열 뒤에 여는 cDNA에서 제거 됩니다. 이 반응 후 분열 남아 많은 꼬리의 위치를 표시 하는 5' 어댑터, 그리고 3 Ts 왼쪽의 결 찰에 대 한 그대로 끝 나뭇잎. 5'과 3' 어댑터 받는 사람 5' 끝에 결 찰에 의해 연결 된 때문에 아무 어댑터 이합체 생성 됩니다. 4 개의 뉴클레오티드 무작위-메 읽기의 시작 부분에 도입-의 상태--예술 시퀀싱 악기 클러스터 해상도 허용 하 고 또한 검색 및 제거의 PCR 증폭 유물에 대 한 독특한 분자 식별자 (우미) 될 수 있습니다. 다른 연구 6일 우미의 크기 더 늘릴 수 있습니다. 프로토콜 생성 mRNA 3' 끝, 모든 무작위 tetramer 3 Ts. 처리는 그들의 5' 끝 시작의 PCR 증폭 유물에 의해 교정에 3 진단 Ts 읽기의 뒤와 시작에 무료 리버스는 읽기 악용 하는 Umi, 3' 어댑터 시퀀스의 제거 및 보완성을 역방향. 내부 A-풍부한 사이트에서 oligo(dT) 못쓰게에서 발생 할 수 읽기 또한 컴퓨터로 확인 하 고 삭제. 가짜 사이트는 일반적으로 하나 잘 특징 18의 부족 고 되어야 보존된 많은 신호 명백한 분열 사이트 7~ 21 뉴클레오티드 상류.
프로토콜 세포 배양과 하룻밤 ligations 안 세 약 8 h 실습 시간에 필요 합니다. 관련 된 읽기 분석 소프트웨어를 사용 하면 매우 정확 하 게 많은 사이트 확인. 많은 사이트에서 클러스터 추가이 원고 (제어 siRNA와 si HNRNPC 대우 셀의 두 생물 학적 복제) 84% 중복, 주석이 달린된 유전자와 이들의 75%와 3' UTR, 중복 및 중 86%에서 강조 4 샘플을 기반으로 생성 한 3' UTR 또는 터미널 exon입니다. 피어슨 상관 계수는 복제 예제에 3' 끝의 표현의 0.92, 이며 0.9의 값 일반적으로 방법으로 얻을 수 있습니다. 따라서, A seq2 재현성 결과 제공 하는 편리한 방법입니다.
프로토콜
1. 세포 성장 및 mRNA 격리
- 1 ~ 80% 합류에 잘 당 10 6 셀 x 6 잘 플레이트에 실험적인 디자인에 따라 세포 성장.
- 성장 매체를 제거 하 고 씻어 셀 한번 버퍼링 인산 염. 직접 mRNA 격리 키트에서 세포의 용 해 버퍼의 1 mL을 추가 하 여 접시에 세포를 lyse. 점성 전송 1 mL 피 펫 팁 15 mL 플라스틱 튜브로 lysate. 고무 주걱을 사용 하 여 완전히 분리 판 표면에서 셀 자료.
- 전단 1 mL와 lysate 포함 점성 DNA 주사기는 lysate는 점도 더 이상 때까지 플런저의 움직임을 위아래로 힘으로 23 G 피하 주사 바늘에 연결 된. 꺼내기는 튜브 lysate 피하기 위해 바닥의 중앙에 주사기 바늘을 가리킨.
- 는 주사기를 사용 하 여 1.5 mL 튜브에 lysate를 전송할. 20000 x g에서 5 분와 파편을 제거 하 4 ° C 회전 합니다. 프로토콜에 걸쳐 DNA 낮은 바인딩 1.5 mL 튜브 사용.
- 는 원심 분리기를 실행 하는 동안 세포의 용 해 버퍼의 500 µ L와 자석 선반에 resuspended 올리고 (dT) 25 자석 구슬의 300 µ L를 씻어. 믹스 2-3 번 선반에 튜브. 솔루션은 분명 후 버퍼를 제거 합니다. 1.4 단계에서 명확한 상쾌한을 수집 하 고 구슬에 추가. Resuspend 및 10 분에 대 한 회전 휠에 튜브를 배치
- 자석 선반에 튜브를 놓습니다. 2 분 추가 0.8 mL 버퍼 A 후 mRNA 격리 키트에서 투명 한 액체를 제거 합니다. 2-3 시간 180 °도 선반에 의해 관을 설정 합니다. A. 버퍼와 한 번 더 세척이 반복
- 1.6 단계에서 설명한 대로 2 번 버퍼 B의 0.8 mL와 구슬 세척.
- Elute 구슬에서 바인딩된 mRNA를 33 µ L H 2 O를 추가 하 고 resuspend 구슬. 5 분가 열된 블록에 75 ° C에가 열. 즉시 1 s와 장소에 대 한 튜브를 회전 자석 선반에 그들. 새로운 튜브에는 상쾌한을 전송. 샘플 추가 사용까지-80 ° C에 저장 될 수 있다.
- 추가 66 µ L 알칼리 가수분해 버퍼는 33 µ L mRNA (단계 1.8), 혼합 하 고 정확 하 게 5 분가 열 블록에 95 ° C에서 열. 얼음에 튜브를 즉시 진정
- RNA 정리 키트 RNA 분리.
참고: 확인 볼륨; 100 µ L 이어야 한다입니다.- 추가 350 µ L 실내 버퍼 키트 및 250 µ L의 에탄올 열 및 30에 대 한 회전에 부하에서 실 온 (RT)에서 8000 x g s. 키트에서 500 µ L RPE 버퍼 씻어. 500 µ L 80% 에탄올으로 세척. 열 건조 20000 x g에 5 분에 대 한 스핀. 36 µ L H 2 O 열을 추가 하 고 20000 x g.에 1 분 열을 삭제 하 고 저장은 eluate 스핀.
2. 5 ' 인 산화 및 DNase 치료 끝
- 추가 5 µ L polynucleotide 키 버퍼, 5 µ L 10 mM ATP, 1 µ L ribonuclease 억제제, 1 µ L DNase와 2 µ L polynucleotide 키 니 아 제를 샘플링 하 고 30 분 1.1 볼륨을 혼합 하 여 프로토콜을 통해 마스터 반응 혼합 준비에 대 한 37 ° C에서 품 어 x n (n = 샘플의 수)의 각 구성 요소.
- 버퍼를 변경 하 고 다음 단계에서 많은 추가 방지 하기 위해 스핀 열에 ATP 제거.
- Prespin
- 스핀-735 x g 1 분 대에 열 새로운 1.5 mL 튜브에 열을 전송 하 고 열에 키 반응을 로드 합니다. 735 x g. 2 분 열 열 삭제 얼음에 수집 된 반응 튜브를 장소 또는-80에서 저장 스핀 ° c.
3. 3 차단 ' Cordycepin 3 인산 염으로 끝나는
참고: 3 차단 하는 것이 필수적입니다 '에 후속 결 찰 반응. 3 그들의 concatemerization을 피하기 위해 RNA 파편의 끝 '는 (에 의해 이미 차단 되지 않은 끝 순환) 인산 가수분해는 3의 추가 의해 처리 됩니다 후 ' dATP (cordycepin 3 인산 염) 체인 터미네이터 뉴클레오티드 많은 중 합 효소의 도움으로. 여기, 효 모 많은 중 합 효소 (yPAP) 하 여 표현 8에 설명 된 대로 정화 0.5 mg/mL의 농도에서 사용 되었다. 효 모 또는 대장균 두 아빠는 거의 3의 추가 대 한 동일한 동작 ' dATP 상업적으로 구입하실 수 있습니다 (재료의 표 참조).
- 중 합 효소 반응 버퍼, 10 m m 3의 2 µ L ' dATP, 1 µ L RNase 억제 물 및에서 반응에 중 하는 1 합 한 µ L 많은 효소 2.2.1 단계. 믹스 및 스핀 1 30 분 추가 32.5 µ L H 2 O 각 반응에 대 한 37 ° C에서 품 s.. 1.10.1 단계에서 RNA를 정화. 14 µ L H 2 o. RNA elute
4. 역 3의 결 찰 ' 5 어댑터 ' 끝의 RNA 조각
- 6 µ L. 추가 3 µ L 10 T4 RNA 결 찰 버퍼, 3 µ L 10 mM ATP x을 볼륨을 줄이기 위해 10 분 동안 진공 집중 장치는 반응 에서도 15 µ L PEG-8000, 1 µ L RNase 억제제, 0.1 m m 역 보완 3의 1 µ L ' 어댑터 " revRA3 " (재료의 표 참조)와 1 µ L 높은 농도 RNA 리가 1, 섞어.
- 1000 rpm에서 간헐적으로 혼합과 온수 믹서에 16 h 24 ° C에서 반응 품 어. 각 반응에 70 µ L H 2 O를 추가 하 고 혼합. 1.10.1 단계에서 RNA를 정화. 14 µ H L 2 O. 샘플-80 ° C에이 시점에서 저장 될 수 있다 RNA elute.
5. 전사 (RT) 리버스
- 장소 11 µ L. 전송에 대 한 반응 200 µ L PCR 볼륨을 줄이기 위해 3 분 동안 진공 집중에 있다 관. 추가 1 µ L 0.05 mM RT 뇌관 " 바이오-뒤-dT25 ". PCR cycler에 70 ° C에서 5 분 동안가 열 하 고 5 분 RT에서 떠나
- 1 µ L 10 mM dNTPs, 4 µ L 5 배 역전사 버퍼, 1 µ L 0.1 M DTT, 1 µ L RNase 억제제, 및 1 µ L 역전사를 추가합니다. 혼합 하 고 PCR cycler에 80 ° c ~ 55 ° C 및 10 분 10 분에 대 한 반응을 열. 얼음에 또는 저장을 위해-80 ° C에서 유지.
6. Uracil DNA Glycosylase 효소 혼합 소화
- 피펫으로 100 µ L 1.5 mL 유리병으로 Streptavidin 구슬 800 µ L biotin 바인딩 버퍼에 resuspend 및 자석 선반에 두십시오. 튜브 2-3 번 반전. 분명 때 버퍼를 제거 합니다. 세척 단계를 반복 합니다. 200 µ L biotin 바인딩 버퍼에 구슬 resuspend.
- 반전 녹음 방송 반응 비즈 솔루션에 추가 하 고 회전 바퀴에 4 ° C에서 20 분을 품 어. Biotin 바인딩으로 2 x 버퍼링으로 구슬 자석 선반에 6.1 / 2 x 10 버퍼를 단계 세척. 50 µ L 10 버퍼에 구슬 resuspend, 2 µ L Uracil DNA glycosylase 효소 혼합을 추가 하 고 간헐적으로 혼합과 믹서에서 37 ° C에서 1 시간을 품 어.
- 추가 50 µ L H 2 O, RNase H의 11 µ L 버퍼 및 1 µ L RNase H 반응을 한다. 마그네틱 선반에 20 분 장소 튜브에 대 한 37 ° C에서 품 어와 새로운 튜브에 쪼개진된 cDNA를 포함 하는 액체를 전송
- 쪼개진된 cDNA 정화.
- 분열 반응에 PCR 정화 키트에서 PB 버퍼의 추가 550 µ L
- . 3 M 아세트산 나트륨, pH 5.2 낮은 pH의 10 µ L를 추가 합니다. 로드는 반응 최소한의 차입 스핀 열 및 17000 x g 1 분에서 스핀
- 추가 750 µ L 버퍼 PE 열과 1 분에 17000 x g에서 스핀을 통해 흐름 삭제합니다. 17000 x g 1 분 건조에 열을 회전 합니다. 1.5 mL 유리병에 열을 전송, 16 µ L H 2 O와 스핀 17000 x g 1 분에서 8 분 7 µ L의 볼륨 집중 진공 집중 장치에 반응 장소 추가.
7. 5 결 찰 ' 5 어댑터 ' cDNA의 끝
- 격리 된 cDNA 추가 T4 RNA 리가 1 버퍼, 3 µ L 10 mM ATP, 15 µ L x 3 µ L 10 PEG-8000, 1 µ L 50 µ M " revDA5 " 올리고 고 1 µ L 높은 농도 T4 RNA 리가 1. H 2 O 각 반응에 20 h. 추가 70 µ L 24 ° C에서 품 어. 샘플이 시점에서-20 ° C에서 저장 될 수 있다.
8. 안내 하는 PCR, 증폭의 라이브러리와 크기 선택
- 파일럿 반응에서 PCR의 최적의 수 지 수 단계 내에서 라이브러리 증폭 도달 주기 결정.
- 피펫으로 25 µ L DNA 중 합 효소 혼합, 20 µ L 결 찰 반응, 2 µ L H 2 O, 1.5 µ L가 10 µ M에 앞으로 PCR 뇌관 (RP1) 및 1.5 µ L 10 µ M 역 PCR 인덱스 뇌관 200 µ L PCR 튜브에.
- Cycler는 다음 프로그램 실행: 3 분 뒤에 20의 20 주기 95 ° C, s 98 ° C, 20 s 67 ° C와 30 s 72 ° C. 수집 7 µ L aliquots는 cycler에서 직접 6, 8, 10, 12, 14, 16, 18 주기 후. (50% 글리세롤, 0.05% 자일 렌 cyanol) 버퍼 로드 x 1 µ L 10을 추가 합니다. 참고: 따르십시오 공급 업체의 권장 사항을 멀티플렉싱 바코드를 결합할 때 사용 하는 경우.
- 1:10, 00 희석 형광 녹색 염료를 포함 하는 1 x TBE 버퍼에 2 %agarose 젤에 작은 슬롯에 별도 제품.
- 부하 aliquots에 2 %agarose 젤과 15 분 PCR 제품의 시각화 마이그레이션에 대 한 100 볼트에 젤 문서 시스템에서 실행 하는 젤.
- 파일럿 반응에서 지 수 증폭의 시작 부분에 사이클 수를 사용 하 여 두 번 볼륨 파일럿 반응 ( 그림 2)에 사용 된 대규모 PCR 반응.
- 대규모 PCR 반응에 대 한 집중 및 PCR 정화 키트와 함께 먼저 반응을 desalt 및 1 x TBE 버퍼에 2 %agarose 젤에 넓은 슬롯에 제품을 분리.
- 200-350 nt DNA를 포함 하는 젤 조각 잘라 제품. 최대 30 분 RT에서 chaotropic 버퍼에 젤을 녹여. 젤 추출 키트 젤 조각에서 DNA를 추출 합니다. A-부자 DNA 9의 바인딩에서 바이어스를 방지 하기 위해 50 ° C에가 열 하지 마십시오.
- 제출 시퀀싱.
참고: 일반적으로, 50 사이클 단일 읽기 (SR50)는 충분 한 (참조, 예를 들어, https://www.illumina.com/technology/next-generation-sequencing.html).
9. 데이터 처리
참고: 결과 시퀀싱 데이터 (fastq 형식) gitlab 저장소 (https://git.scicore.unibas.ch/zavolan_public/A-seq2-processing)에서 사용할 수 있는 소프트웨어와 함께 처리 됩니다. 분석 포함 4 가지 주요 단계: (1) 다운로드 git 저장소, 가상 환경, (2) 설치 (3) 설정 특정 매개 변수는 구성 파일에 (4)을 통해 분석을 시작 ‘ snakemake ’ 10. 단계 4에서 전체 분석 필요 하나의 명령. 분석의 상세한 단계별 설명 gitlab 저장소에 추가 정보 파일에서 찾을 수 있으며 간단한 설명 아래 수 있습니다. 모든 개별 처리 단계는 외부 소스에서 공개적으로 사용 가능한 도구를 실행 하 여 수행할 또는 사내 준비. 계산 파이프라인 snakemake 패키지 사용할 수 있는 10 아나콘다 기반 11 파이썬 3 가상 환경에 따라 달라 집니다. 유닉스 계열 운영 체제와 컴퓨터에서 실행 하 고 설치 된 CentOS 6.5 운영 체제와 리눅스 환경에 40 기가바이트 RAM 사용할 수 있는 테스트 되었습니다. 소프트웨어 종속성 가상 환경 내에서 자동으로 제어 됩니다. 다음 공개 소프트웨어 도구 필요 및 환경과 함께 설치 함으로써: snakemake (v3.9.1) 10, fastx 툴킷 (v0.0.14) 12, 스타 (v2.5.2a) 13 , cutadapt (v1.12) 14, samtools (v1.3.1) 14 , 15, bedtools (v2.26.0) 16 , 17.
- CDNAs 읽기에서 데이터 사전 처리
참고: 시퀀싱 깊이 실행 간에 다를 수 있습니다 그리고, 악기에 따라 하나의 샘플에서 데이터는 여러 시퀀스 파일 분할 될 수 있습니다. 이 경우 하나의 샘플에 해당 하는 다음 단계에 사용 되는 단일 입력된 파일에는 파일을 연결 합니다.- Fasta 형식 fastq에서 파일을 변환.
- 올바른 구조 (3 thymidines는 읽기의 위치 5, 6 및 7에서) 추출 읽습니다.
참고: 위에서 설명한 실험 프로토콜에 따라 준비 제대로 읽기 구조 있어야 (5에서 ' 끝): 4-뉴클레오티드 바코드-3 thymidines-반대로 사본 3의 보수 ' 끝. - 시퀀스의 설명 줄에 시작 tetramer에 대 한 정보를 저장.
참고:는 tetramer 역할 증폭 유물 분석 뒷부분에서의 보정을 용이 하 게 하는 독특한 분자 식별자 (우미). - 읽기에서 처음 7 개의 뉴클레오티드를 제거 ' 5 ' 끝.
- 같은 읽기의 복사본을 하나만 유지 하 여 증폭 아티팩트 삽입 시퀀스와 우미에 대 한 수정.
- 3의 부분을 제거 ' 어댑터 시퀀스 및 다음 역방향 보완 시퀀스 일치 하는 끝. 최소 길이 읽기로만 진행 (기본: 15 nt).
참고: 원래 mRNA 조각 및 시퀀싱, 3의 숫자의 길이 따라 '는 읽기의 끝 3의 일부를 포함할 수 있습니다 ' 어댑터,이 단계에서 제거 됩니다.
- 추출 다음 조건을 충족 하는 모든 읽기: 최대 2 알 수 없는 뉴클레오티드 (' N '), 최대 80%, 그리고 읽기 하지 대답의 마지막 뉴클레오티드 이러한 읽기는 분석에 사용 될 충분 한 품질의 것으로 간주 됩니다.
- 접합된 읽기를 처리 하 고 BAM 형식에서 출력 파일을 생성 하는 도구와 게놈 읽기 매핑됩니다.
- 경우 스타
- 사용, 읽기 매핑해야 한다 게놈의 인덱스 파일을 만듭니다. 인간 게놈이이 단계 35 g B의 메모리 (RAM) 필요 합니다.
- 게놈 읽기 매핑됩니다.
참고: (스타 전용 노트) 소프트 클리핑 3의 매핑을 강제 하기 위하여 사용할 수 없습니다 ' 각 끝이 읽기 뉴클레오티드는 분열 위치의 바로 업스트림.
- 게놈 읽기 매핑됩니다.
- BAM 침대 파일 변환. 경우에 읽기는 여러 위치에 지도, 계속 그 최저 편집 거리만.
참고: 특정 위치에 매핑된 읽기 복사 수 점수로 사용 됩니다. 여러 위치에 매핑되는 읽기 fractionally 위치 읽기 지도의 무게 1/번호와 같은 각 위치에서 계산 됩니다. - 붕괴 가능성이 시퀀싱 오류로 다 읽습니다. 두 개의 별개 읽기 동일한 위치에 지도 하는 경우 (매핑 시작 및 끝 위치는 동일)와 같은 우미 공유, PCR 중복으로 그들을 고려 하 고 하나만 유지.
- 유추 모든 개별 사전-mRNA 3 ' 끝 처리 사이트.
참고: 3에 대 한 증거를 제공 하는 개별 읽기 ' 끝낼 때 그것의 마지막 4 개의 뉴클레오티드 오류 없이 게놈에 매핑됩니다. 위치는 3 ' 끝 읽기 지도의 분열 사이트도 저장 됩니다. - 검색 3 ' 내부 못쓰게에서 유래 할 수 있는 사이트를 종료. 10는 게놈에서 분열 위치의 하류 nt 만족 내부 못쓰게 유물으로 사이트 정의 다음 기준 중 하나: 이상으로 포함, 6으로, 연속 포함 또는 다음 tetramers 중 하나에서 시작: AAAA, AGAA, AAGA, AAAG .
- 개별 3의 테이블을 생성 ' 침대 형태로 처리 사이트 끝.
- 식별 많은 사이트 클러스터를 독립적으로 통제.
참고: 여기에 설명 된 단계는 사전 게시 5에 도입 된 절차를 따릅니다.- 개별 3을 수집 하 여 시작 ' 연구의 모든 샘플에서 가져온 처리 사이트 끝.
- 알려진된 많은 신호 7-60 + 10의 지역에서 주석을 주위에 각 각 3 뉴클레오티드 ' 끝 처리 사이트.
- 식별 많은 사이트는 다음과 같이 각 샘플에서 배경 위에 표현.
- 현재 샘플 내의 그들의 원시 식으로 사이트를 정렬. 위에서 아래로, 그들은 게놈에 있는 미리 정의 된 거리 내에 있는 경우 낮은 순위 사이트 더 높은 순위 사이트 연결 사이트의 목록을 통과 (기본값: 25 nt 업 또는 다운스트림) 높은 순위 사이트에서.
참고: 모든 낮은 순위 사이트 고위 사이트와 관련 된 정의 인 식 읽기가이 사이트의 모든 문서화 수 클러스터. - 식으로 이러한 클러스터를 정렬 하 고 결정 하는 클러스터의 주석된 많은 비율 신호는 미리 정의 된 임계값 (아래 드롭 식 임계값 c 최저 식 높은에서 클러스터의 목록을 통과합니다 기본값: 90%).
- 컷오프 아래 어떤 클러스터에서 사이트를 삭제.
- 현재 샘플 내의 그들의 원시 식으로 사이트를 정렬. 위에서 아래로, 그들은 게놈에 있는 미리 정의 된 거리 내에 있는 경우 낮은 순위 사이트 더 높은 순위 사이트 연결 사이트의 목록을 통과 (기본값: 25 nt 업 또는 다운스트림) 높은 순위 사이트에서.
- 클러스터 밀접 하 게 간격 3 ' 사이트 샘플에서 얻은 끝.
참고: 정렬 3 ' 끝 샘플 지원의 수에 의해 그리고는 정규화의 합으로 사이트를 먼저 처리 읽을 수 (당 읽고 백만 (RPM)) 샘플에서. 위에서 아래로, 그들의 거리는 더 높은 순위 사이트에 미리 정의 된 제한 보다 큰 경우 낮은 순위 사이트 더 높은 순위 사이트 연결 목록을 탐색 (기본값: 12 nt). 때마다 창설 3의 ' 끝 사이트 주석된 많은 신호 중복 되었거나 많은 신호 감지 내부 못쓰게 하 추가 검사에 대 한 해당 클러스터를 표시 하는 하류, 직접. - 병합 많은 클러스터 사이트.
참고: 클러스터 상 상속 내부 못쓰게 후보자로 표시 되어, 그들의 많은 신호를 공유 하는 두 클러스터 경우 다운스트림 클러스터에 병합 하거나 클러스터에서 가장 다운스트림 사이트에 최소한에 있는 많은 신호 유지 상류 거리 (기본: 15 nt). 마지막으로, 간격이 클러스터 경우 병합 됩니다: (i) 그들은 공유 하는 같은 많은 signal(s), 또는 (ii) 결과 클러스터의 범위는 최대를 초과 하지 않습니다 (기본: 25 nt). - 클러스터 정규화 총 침대-파일 형식에서 모든 3에서 카운트를 읽는 저장 ' 점수 각 클러스터에 사이트 끝.
결과
폴 리 (A)-RNA를 포함 된 격리 배양된 세포, 알칼리 가수분해에 의해 조각에서 되었고 cDNAs 여 전사 oligo(dT) 뇌관으로 반전 되었다. 결과 cDNA streptavidin 구슬에 움직일 수 있었다, 뒤 uracil 특정 절단 반응에 죽 습 했다, 어댑터에 5' 출혈 했다 고 쪼개진된 조각 및 삽입의 3' 끝 시퀀싱 했다. 그림 1 실험의 그래픽 개요를 보여 줍니다.
헬러 HEK293 세포 106 세포 절차의 끝에서 단백질 코딩 유전자의 대부분에 대 한 많은 사이트를 식별 하기에 충분 했다. 그러나, 다른 세포 유형 또는 조직 실험에 사용 하는 셀의 수로 확인 된 많은 사이트의 수에는 채도 테스트 해야 할 수 있습니다 증가 합니다. 파일럿 PCR의 대표적인 결과 단계와 DNA 파편의 시퀀싱 하기 전에 샘플의 분석은 그림 2에 표시 됩니다.
그림 3 시퀀서에서 얻은 고 게놈에 매핑할 준비가 된 어댑터 손질, 품질 검사 읽기로 끝나는 fastq 파일에서 시작 계산 분석의 전처리 단계를 보여 줍니다. 그림 4 는 mRNA 3' 끝 처리는 특정 샘플에서 식별 되는 사이트의 카탈로그와 해당 게놈 및 끝에 읽기의 매핑으로 시작 하는 분석 단계를 보여 줍니다. 여러 샘플을 분석할 때 개별 샘플에서 발견 된 사이트 처리 3' 끝과 일치 하는 샘플에서 그들의 풍부를 보고 추가 단계가 수행 됩니다. 다음이 단계는 그림 5에 나와 있습니다.
따라서, 일단 샘플을 시퀀스 사용 가능한 처리 파이프라인을 통해 (fastq 형식) 파일을 읽고 결과 시퀀싱의 분석은 간단 합니다. 샘플에 대 한 정보를 구성 파일에 추가한 후 파이프라인 실행 될 출력 파일의 두 가지 주요 유형: 1)와 모든 3' 침대-파일 끝 처리 사이트 (예: "개별 샘플에서 확인 된 sample1.3pSites.noIP.bed.gz"), 그리고 2) 연구의 모든 샘플에서 모든 많은 사이트 클러스터 (clusters.merged.bed)와 침대 파일. 출력 또한 모든 읽기 (예: "sample1 각각의 개별 샘플에서에 대 한 게놈 좌표를 포함. STAR_out/Aligned.sortedByCoord.out.bam")는 나중 IGV16같은 게놈 브라우저에서 볼 수 있습니다. 읽기 프로 파일의 검사는 일반적으로 게놈 연구에서 수행 된 특정 섭 시 발생 하는 변화에 많은 사이트의 배포의 첫 번째 엿볼을 제공 합니다. 예를 들어 그림 6에서 HNRNPC 단백질의 노크 다운에 대 한 특정 유전자의 응답은 표시 됩니다.
이러한 게놈 넓은 배포판의 요약도 (표 1)를 제공 됩니다. "수/annotation_overlap" 디렉터리에 출력 파일 특정 주석된 기능와 중복 사이트의 분수를 포함 하는 특히, (gtf 파일에서 입력으로 제공;는 주석: 3' UTR, 터미널 엑손 exon, intergenic intron). 마지막으로, 각 샘플에 대 한 개별 처리 단계의 결과 (예: "sample1.summary.tsv") 저장 됩니다. 이것의 숫자를 포함 한다: 게놈을 고유 하 게 그 지도 읽고 각 샘플, 5' 끝의 예상된 구조를 읽기, 읽기 전체 PCR 중복 축소 후에 남아 있는 원시 읽기, 높은-품질 단계 9.2에서 정의 된 기준에 따라 읽고 (연속 오류에서 발생 하는 그 붕괴 이후 단계 9.5 참조), 다중 매핑 읽습니다 (시퀀싱 오류에서 발생 하는 그 붕괴 이후 단계 9.5 참조), 각 샘플, 원시 3' 끝 처리 사이트에에서 사이트를 처리 원시 (하지 클러스터) 3' 끝 잠재적인 내부 못쓰게 후보자 없이 독특한 3' 끝 처리 사이트 내부 못쓰게 후보자, 및 많은 사이트 클러스터의 최종 세트 없이 모든 견본에서.
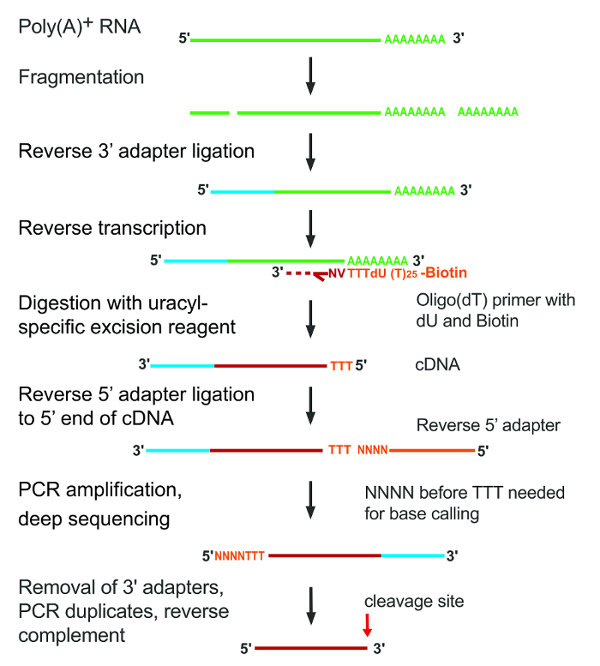
그림 1: A seq2 프로토콜의 주요 단계. 각 단계는 그림의 왼쪽에 표시 됩니다. 삽입 RNA 파편 반전 녹음 방송; 후 cDNA에 빨갛게 녹색 선으로 표시 됩니다. 어댑터는 밝은 파랑 또는 오렌지 컬러는. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
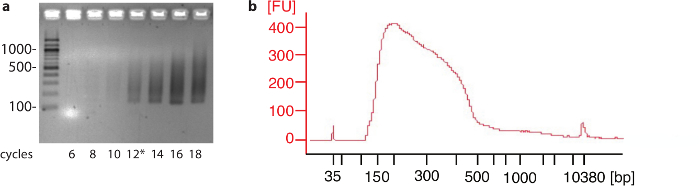
그림 2: 파일럿 PCR 및 최종 제품 프로필. PCR 반응에서 (는) Aliquots 다른 사이클에서 수집 된 고 2 %agarose 젤에서 분리. 왼쪽 숫자는 뉴클레오티드 DNA 사다리에 각각 밴드의 크기를 나타냅니다. 이 실험에서 12 사이클 (*)는 대규모 PCR 반응에 대 한 선정 됐다. (b) 약 280 뉴클레오티드의 평균 크기를 공개 하는 조각 크기 분석기에서 실행 하는 크기 선택 후 샘플의 예. [푸] 왼쪽 숫자 상대적 신호 강도를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
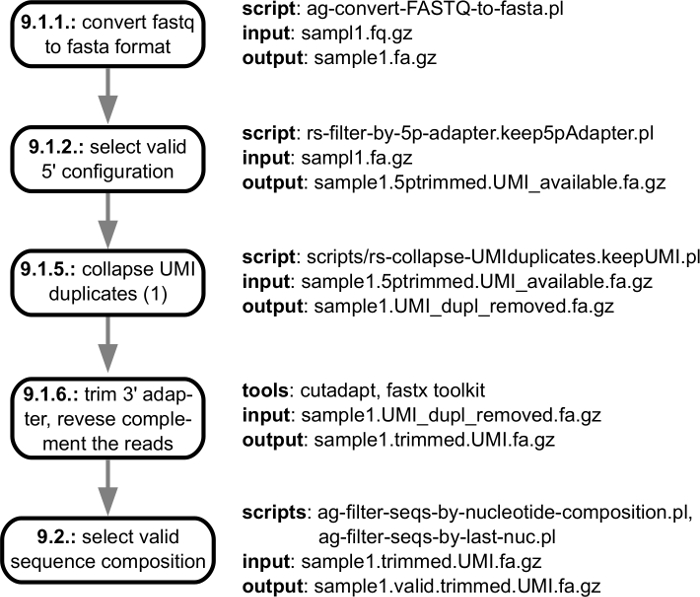
그림 3: 읽기 시퀀싱의 사전 처리의 개요. 시퀀싱 악기 관련 소프트웨어에 의해 생성 되는 읽기로 fastq 파일 해당 게놈에 매핑되는 높은-품질 읽기를 식별 하기 위해 처리 됩니다. 그림의 개별 단계는 입력/출력 사양 "데이터 처리" 섹션에 설명 된 프로토콜의 각 단계에 대 한 링크와 함께 진행중에서입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
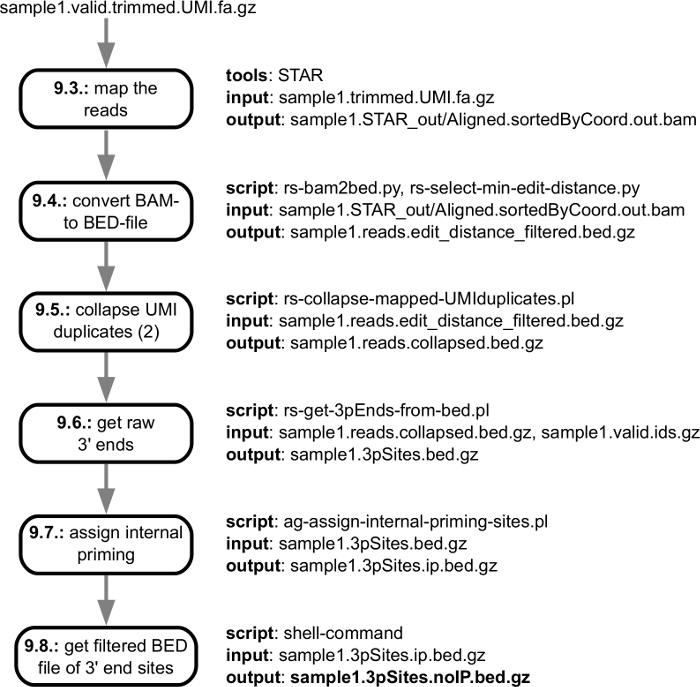
그림 4: 시퀀스의 개요 읽기 처리, 개별 3' 끝의 세대에 게놈의 매핑 단계에서 처리 사이트. 그림 i에 대 한 링크와 파이프라인의 개별 단계는 입력/출력 사양"데이터 처리" 섹션에 설명 된 프로토콜의 ndividual 단계. 주 출력 파일을 사용자에 게 전달 되는 굵게 표시 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
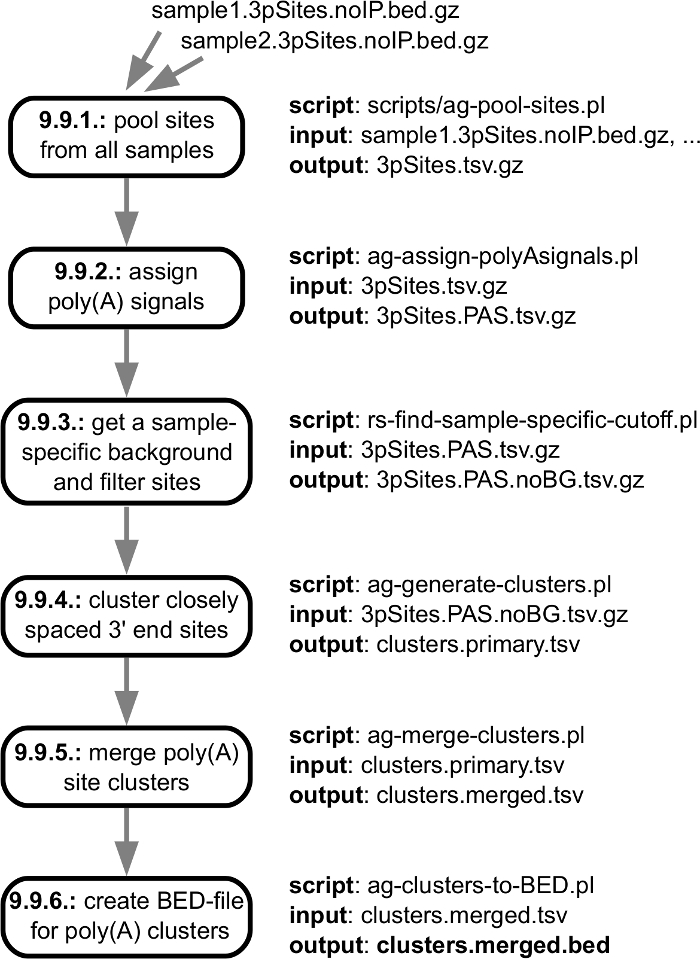
그림 5: 이동 공동 규제 3' 끝 사이트 시퀀싱의 클러스터를 생성 하는 단계의 개요. 그림의 개별 단계는 입력/출력 사양 "데이터 처리" 섹션에 설명 된 프로토콜의 각 단계에 대 한 링크와 함께 진행중에서입니다. 주 출력 파일은 굵게 표시 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
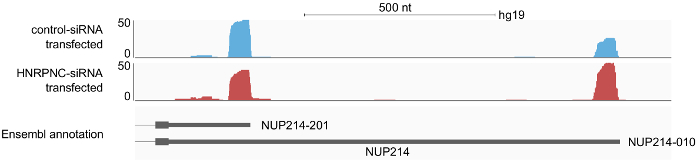
그림 6: 3'의 프로필의 예제 결과 IGV 16 게놈 브라우저에 표시 하는 NUP214 유전자의 터미널 exon 따라 처리 읽기 끝. A seq2 읽기 제어 siRNA 또는 HNRNPC siRNA 치료 HEK 293 세포의 두 샘플에서 준비 되었다. 분석 파이프라인 주석 된 많은 사이트를 문서화 읽기 IGV 게놈 브라우저에 대 한 입력으로 사용 된 BAM 형태로 저장 되었다. 읽기 봉우리의 3' 끝은 mRNA 3' 합에 주석 끝 매핑됩니다. 프로필은 HNRNPC 눕 시 긴 3' UTR isoform의 증가 사용을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| 시-제어 복제 1 | 복제 시 제어 2 | |
| id: 29765 | id: 32682 | |
| 원시 읽기 수 | 44210258 | 68570640 |
| 트리밍 및 필터링 후 유효한 읽기 수 | 14024538 | 21211793 |
| 수 읽기를 고유 하 게 매핑 | 6953674 | 13946436 |
| 여러 개의 loci를 읽기 수 | 2040646 | 2925839 |
| 개별 3' 끝 처리 사이트의 수 | 1107493 | 1710353 |
표 1: 분석 파이프라인의 출력 예. 각 단계에서 가져온 읽기 요약
토론
핵심 및 보조 요인 사전 mRNA 3' 끝 처리에 관련 된 수많은 대응 하 게 복잡 한 polyadenylation 풍경에 반영 됩니다. 또한, polyadenylation 녹음 및 접합 등 다른 프로세스에 있는 변화에 반응 이기도합니다. 사전-mRNAs에 3' 끝 분열 사이트는 일반적으로 5' 분열 제품에 추가 된 특성 많은 꼬리에 따라 식별 됩니다. 대부분의 방법은 사용 폴 리 (A)의 특정 변환을 허용 하는 가변 길이의 oligo(dT) 뇌관-반전 녹음 방송 반응에서 cDNAs mRNAs를 포함. 이 방법의 일반적인 문제는 A 풍부한 시퀀스 artifactual 분열 사이트 결과에 내부 못쓰게. 샘플 준비의 단계에서이 유물을 우회 하는 것을 목표로 하는 두 가지 방법이 제안 되었습니다. 3 P-seq 방법 1, 어댑터는 특히 부분 RNase T1 소화 및 유일한 deoxynucleotide로 반응에서 TTP와 반전 녹음 방송 부 목 oligo의 도움으로 많은 꼬리의 끝에 출혈 됩니다. 결과 poly(A)-poly(dT) heteroduplexes 다음 RNase H로 소화 하 고 나머지 RNA 파편 절연, 어댑터, 출혈 및 시퀀스. 간단 하 고 우아한 방법, 2 P-seq, 시퀀싱 반응에서 나머지 oligo(dT) 스트레치 2동일한 저자 의해 보고 되었다 건너뛰는 사용자 지정 시퀀싱 뇌관을 사용 하. 관련 방법에서 3' 읽고 3, 5의 비정상적으로 긴 뇌관 우리와 45도는 비오 틴을 포함 하는 Ts 조각난된 RNA, 50 개 이상의 뉴클레오티드의 많은 꼬리와 RNA 분자에 대 한 선택에 엄격한 세척 뒤에 단련. 3' 읽기 대폭 줄여 내부 못쓰게의 주파수, 그것은 완전히 제거 하지 그것 3. 또한 직접 RNA 시퀀싱에 대 한 프로토콜 제안 되었습니다, 하지만 결과 읽기 짧은 오류의 높은 비율을가지고 고이 접근 하지 않은 추가 개발된 18,,1920. PolyA Seq와 상용화 양의 Seq 프로토콜 기반 oligo(dT) 못쓰게 cDNA 두 번째 가닥 종합 20임의의 못쓰게 단계를 결합 한다. 서식 파일 스위치 반전 녹음 방송 반응 역전사 Moloney Murine 백혈병 바이러스 (MMLV)와 함께 사용 단일 단계에서 링커 cDNAs의 세대를 리드 하 고 그로 인하여 아무 어댑터 이합체는 파-Seq 및 SAPAS 메서드에 나타날 수 있습니다. 21 , 22.
A seq2 메서드 여기 biotinylated oligo(dT) 뇌관 내 쪼갤 뉴클레오티드 (뒤)의 그것의 사용에서 밖으로 서 제시. 이 수정 결합 풍부 때 교배 하는 oligo(dT), polyadenylated 라이브러리를 준비 하기 전에 격리 된 조각에서 올리고 (dT)25 시퀀스의 대부분의 제거와 대상과 3 ts, 보존의 유틸리티는 많은 꼬리의 이전 존재를 나타냅니다. 반면, RNase H 임의로 많은 RNA 분자에서 제거를 사용 하는 방법을 여러 가지로 둡니다. A-seq2, 시퀀싱 방지 감각 가닥의 3' 끝에서 이루어집니다, 이후 분열 사이트를 원시 시퀀스 읽기의 시작 부분에 NNNNTTT 모티브 후 예상 됩니다. 무작위 tetramers PCR 증폭 유물의 제거에서 뿐만 아니라 호출 기본 허용 뿐만 아니라 제공 합니다. 더 이상 Umi 또한 가질 수 있습니다. 내부 못쓰게 가능성 A seq2에 남아 및 계산 해결, 먼저 3'를 삭제 하 여 genomically 인코딩, A 부유한 다운스트림 순서 그리고 3'에서 내부 못쓰게에 의해 설명 될 수 있는 최종 클러스터를 삭제 하 여 종료 합니다 자체 A 풍부한 많은 신호. 프로토콜의 많은 수에 의해 고유 하 게 유추 하는 많은 사이트의 최근 분석 A seq2에 고유한 사이트 예상된 뉴클레오티드 유통 및 유전자, 다른 3'와 비슷한 내 위치 시퀀싱 프로토콜을 종료 했음을 나타냅니다.
A seq2에서 중요 한 단계는 polyadenylated RNA의 선택과 리보솜 RNAs 및 다양 한 작은 RNAs의 제거. 이것은 가장 쉽게 올리고 (dT)25 자석 구슬 가진 mRNA 격리 키트에 의해 이루어집니다. 원칙적으로, 총 RNA 격리 솔루션을 또한 포함 하는 페 놀과 고품질 대상이 될 수 있는 추가 선택에 mRNA-절연 키트 또는 올리고 (dT) agarose RNA를 제공 합니다. A seq2에 변화 될 수 있는 단계 단축 하거나 다양 한 크기의 RNA 파편을 얻을 수 확장 될 수 있는 알칼리 가수분해와 치료입니다. 중요 한 많은 중 합 효소에 의해 RNA 파편의 3' 끝에 3' dATP의 추가 효율적 이기도 합니다. 여기에 설명 된 프로토콜에이 치료는 동안을 피하기 위해 concatemerization 결 찰 반응 모든 RNA 조각에 적용 됩니다. 마지막으로, 우리는 주의 RNA 리가 1은 일반적으로 RNA 리가로 사용 됩니다, 그것은 또한 ligates 효율적으로 단 하나 좌초 된 DNA, 우리 선 cDNA 분자의 5' 끝에 어댑터를 여기 다.
따라서, A seq2 효율적이 고 사전 mRNA 3' 끝 처리 사이트의 식별을 위한 프로토콜을 구현 하기 쉬운 이다. 향후 개발 더 필요한 자료의 양과 프로토콜의 복잡성을 포함할 수 있습니다. 전산 데이터 분석 도구를 더 관련 된 세트의 3' 끝 읽기 다양 한 프로토콜 얻은 시퀀싱 균질 처리 가능
공개
저자는 공개 없다.
감사의 말
저자는 세포 배양에 대 한 부인 베아트리스 Dimitriades 감사합니다. 이 작품은 스위스 국립 과학 재단 교부 금 #31003A_170216 및 51NF40_141735에 의해 지원 되었다 (NCCR RNA & 질병).
자료
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Agarose, ultra pure | Invitrogen | 16500-500 | |
| 2100 Bioanalyzer | Agilent | G2940CA | |
| Cordycepin triphosphate (3’ dATP) | SIGMA | C9137 | |
| DNA low bind vials, 1.5 ml | Eppendorf | 22431021 | |
| Dulbecco’s Phosphate Buffered Saline | SIGMA | D8637 | |
| Dynabeads mRNA-DIRECT Kit | Ambion | AM61012 | |
| GR-Green dye | Excellgen | EG-1071 | use 1:10,000 dillution |
| HiSeq 2500 or NextSeq 500 next generation sequencers | Illumina | inquire with supplier | |
| KAPA HiFi Hotstart DNA polymerase mix | KAPA/Roche | KK2602 | |
| Nuclease free water | Ambion | AM9937 | |
| Poly(A) polymerase, yeast | Thermo Fisher Scientific | 74225Z25KU | |
| Poly(A) polymerase, E.coli | New England Biolabs | M0276L | |
| Polynucleotide kinase | Thermo Fisher Scientific | EK0032 | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| RNA ligase 1, high concentration | New England Biolabs | M0437M | includes PEG-8000 |
| RNeasy MinElute RNA Cleanup kit | Qiagen | 74204 | |
| RNase H | New England Biolabs | M0279 | |
| RNasin Plus, ribonuclease inhibitor | Promega | N2618 | |
| Superscript IV reverse transcriptase | Thermo Fisher Scientiific | 18090050 | |
| Turbo DNase | Ambion | AM2238 | |
| USER enzyme mix | New England Biolabs | M5505 | |
| Dyna-Mag-2 magnetic rack | Thermo Fisher Scientific | 12321D | |
| Thermomixer C | Eppendorf | 5382000015 | Heated mixer with heated lid |
| MicroSpin columns | GE-Healthcare | 27-5325-01 | |
| Name | Company | Catalog Number | Comments |
| Buffers | |||
| Alkaline hydrolysis buffer, 1.5 x | Mix 1 part 0.1 M Na2CO3 and 9 parts 0.1 M NaHCO3. Add EDTA to 1 mM. Adjust pH to 9.2. Store aliquots at -20 °C. | ||
| 5x poly(A) polymerase buffer | Thermo Fisher Scientiific | 100 mM Tris-HCl, pH 7.0, 3 mM MnCl2, 0.1 mM EDTA, 1 mM DTT, 0.5 mg/ml acetylated BSA, 50% glycerol | |
| Biotin binding buffer | 20 mM TrisCl pH 7.5, 2 M NaCl, 0.1% NP40 | ||
| TEN buffer | 10 mM TrisCl, pH 7.5, 1 mM EDTA, 0.02% NP40 | ||
| Name | Company | Catalog Number | Sequence |
| Oligonucleotides according to Illumina TruSeq Small RNA Sample Prep Kits, for GA-IIx and Hiseq2000/2500 sequencers | Microsynth | ||
| revRA3 (RNA) | Microsynth | 5’ amino CCUUGGCACCCGAGAAUUCCA 3’ | |
| revDA5 | Microsynth | 5’ amino GTTCAGAGTTCTACAGTCCGAC GATCNNNN-3’ | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' (V = G, A or C) | |
| PCR primer forward, RP1 | Microsynth | 5' AATGATACGGCGACCACCGAGA TCTACACGTTCAGAGTTCTACAG TCCGA 3' | |
| PCR primer reverse, RPI1, barcode in bold | Microsynth | 5' CAAGCAGAAGACGGCATACGAG ATCGTGATGTGACTGGAGTTCCT TGGCACCCGAGAATTCCA 3' | |
| Name | Company | Catalog Number | Comments |
| Oligonucleotides according to Illumina TruSeq HT-Small RNA Sample Prep Kits, for HiSeq2000/2500 and NextSeq500 sequencers | |||
| HT-rev3A (DNA/RNA) | Microsynth | 5'-amino-GTGACTGGAGTTCAGACGTGTG CTCTTCCrGrAUrC-3' | |
| HT-rev5A | Microsynth | 5' amino-ACACTCTTTCCCTACACGACGCT CTTCCGATCTNNNN 3' | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' | |
| PCR primers forward (D501-506) | Microsynth or Illumina | 5'-AATGATACGGCGACCACCGAGAT CTACAC[i5]ACACTCTTTCCCTACA CGACGCTCTTCCGATCT -3' | |
| PCR primers reverse (D701-D712) | Microsynth or Illumina | 5'-CAAGCAGAAGACGGCATACGAG A[i7]GTGACTGGAGTTCAGACGTG TGCTCTTCCGATC-3' | |
| Documentation for Illumina multiplexing: | Illumina | https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/illumina-adapter-sequences_1000000002694-01.pdf |
참고문헌
- Jan, C. H., Friedman, R. C., Ruby, J. G., Bartel, D. P. Formation, regulation and evolution of Caenorhabditis elegans 3'UTRs. Nature. 469 (7328), 97-101 (2011).
- Spies, N., Burge, C. B., Bartel, D. P. 3' UTR-isoform choice has limited influence on the stability and translational efficiency of most mRNAs in mouse fibroblasts. Genome Res. 23 (12), 2078-2090 (2013).
- Hoque, M., Ji, Z., et al. Analysis of alternative cleavage and polyadenylation by 3' region extraction and deep sequencing. Nat. methods. 10 (2), 133-139 (2013).
- Martin, G., Gruber, A. R., Keller, W., Zavolan, M. Genome-wide analysis of pre-mRNA 3’ end processing reveals a decisive role of human cleavage factor I in the regulation of 3' UTR length. Cell Rep. 1 (6), 753-763 (2012).
- Gruber, A. R., Martin, G., et al. Global 3' UTR shortening has a limited effect on protein abundance in proliferating T cells. Nat. Commun. 5, 5465 (2014).
- Kivioja, T., Vähärautio, A., et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat. methods. 9 (1), 72-74 (2011).
- Gruber, A. J., Schmidt, R., et al. A comprehensive analysis of 3' end sequencing data sets reveals novel polyadenylation signals and the repressive role of heterogeneous ribonucleoprotein C on cleavage and polyadenylation. Genome Res. 26 (8), 1145-1159 (2016).
- Lingner, J., Keller, W. 3'-end labeling of RNA with recombinant yeast poly(A) polymerase. Nucleic Acids Res. 21 (12), 2917-2920 (1993).
- Quail, M. A., Kozarewa, I., et al. A large genome center's improvements to the Illumina sequencing system. Nat. methods. 5 (12), 1005-1010 (2008).
- Rahmann, S. Snakemake--a scalable bioinformatics workflow engine. Bioinformatics. 28 (19), 2520-2522 (2012).
- . Anaconda Software Distribution Available from: https://continuum.io (2016)
- Dobin, A., Davis, C. A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Li, H., Handsaker, B., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Robinson, J. T., Thorvaldsdóttir, H., et al. Integrative genomics viewer. Nat. Biotechnol. 29 (1), 24-26 (2011).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ozsolak, F., Platt, A. R., et al. Direct RNA sequencing. Nature. 461 (7265), 814-818 (2009).
- Yao, C., Biesinger, J., et al. Transcriptome-wide analyses of CstF64-RNA interactions in global regulation of mRNA alternative polyadenylation. Proc. Natl. Acad. Sci. U. S. A. 109 (46), 18773-18778 (2012).
- Lin, Y., Li, Z., et al. An in-depth map of polyadenylation sites in cancer. Nucleic Acids Res. 40 (17), 8460-8471 (2012).
- Shepard, P. J., Choi, E. -. A., Lu, J., Flanagan, L. A., Hertel, K. J., Shi, Y. Complex and dynamic landscape of RNA polyadenylation revealed by PAS-Seq. RNA. 17 (4), 761-772 (2011).
- Fu, Y., Sun, Y., et al. Differential genome-wide profiling of tandem 3' UTRs among human breast cancer and normal cells by high-throughput sequencing. Genome Res. 21 (5), 741-747 (2011).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유