Method Article
3' fine sequenziamento preparazione libreria con A-seq2
In questo articolo
Riepilogo
Questo protocollo descrive un metodo per la mappatura pre-mRNA 3' fine elaborazione siti.
Abstract
Gli studi nell'ultimo decennio hanno rivelato una varietà complessa e dinamica di pre-mRNA clivaggio e poliadenilazione reazioni. mRNA con lungo regioni 3' non tradotta (UTR) vengono generati in cellule differenziate, mentre le cellule proliferanti esprimono preferenzialmente trascrizioni con breve 3' UTR. Descriviamo il protocollo A-seq, ora alla sua seconda versione, che è stato sviluppato per mappare siti di poliadenilazione genoma e studiare la regolazione del pre-mRNA processamento dell'estremità 3'. Anche questo protocollo attuale sfrutta la polyadenylate nel (poly(A)) code che vengono aggiunti durante la biogenesi dei mRNAs più mammiferi per arricchire per mRNA completamente elaborato. Un adattatore di DNA con deoxyuracil alla sua quarta posizione permette la lavorazione precisa di mRNA 3' fine frammenti per la sequenza. Non compresa la coltura delle cellule e le legature durante la notte, il protocollo richiede circa 8 h tempo di hands-on. Insieme ad essa, viene fornito un pacchetto facile da usare software per l'analisi dei dati di sequenziamento derivata. A-seq2 e il software di analisi associati forniscono una soluzione efficiente e affidabile per il mapping del pre-mRNA 3' finisce in una vasta gamma di condizioni, da 106 o meno cellule.
Introduzione
La cattura e la sequenza di mRNA 3' estremità permette lo studio dell'elaborazione di mRNA e la quantificazione dell'espressione genica. A causa di loro code di poli (a), mRNA eucariotico può essere efficacemente purificato da lisati cellulari totali con perlina-immobilizzata oligo-deossitimidina (molecole di oligo(dT)), che può anche innescare la sintesi di cDNA. Tuttavia, questo approccio presenta due svantaggi. In primo luogo, tratti della A interne a trascrizioni possono anche innescare la sintesi di cDNA, conseguente spurie poli (a) siti. Poli (a) in secondo luogo, omogeneo tratti pongono sfide specifiche per il sequenziamento, oltre a non essere ben informato per l'identificazione di trascrizione. Vari approcci sono stati proposti per aggirare queste limitazioni, come trascrizione d'inversione attraverso poli (a) Code seguite da digestione RNasi H (3P-seq 1), utilizzo di un primer di sequenziamento personalizzato che termina in 20 Ts (2P-seq 2), preselezione di Frammenti di RNA con code di poli (a) di oltre 50 nucleotidi con un primer di45 CU5T seguita da digestione RNasi H (3' letture 3) e l'utilizzo di un primer oligo-dT contenente l'adattatore 3' in un tornante (A-seq 4).
L'A-seq2 metodo sviluppato di recente 5 mira a bypassare il sequenziamento attraverso poli (a) e allo stesso tempo per ridurre al minimo la percentuale di dimeri generati da auto-legatura degli adattatori, particolare che si verificano quando la concentrazione molare di adattatori supera la concentrazione di inserto. Questo problema può essere eliminato quando entrambe le schede sono legate allo stesso tipo di polinucleotide estremità come in A-seq2, dove le schede 3' sono legate all'estremità 5' di frammenti di RNA e le schede di 5' a 5' estremità dei cDNA dopo trascrizione inversa. Il metodo è più conveniente della nostra precedentemente proposta A-seq - in cui sequenziamento è stato in 5'-a-3' direzione quindi che richiedono precisamente controllata RNA frammentazione-, pur mantenendo un'elevata precisione di identificazione del sito di poli (a). Circa l'80% delle letture sequenziate in campioni tipici associati in modo univoco al genoma e condurre all'identificazione di oltre 20.000 cluster di sito di poli (a), più del 70% delle quali sovrapposte con annotata 3' UTR.
In breve, il protocollo A-seq2 inizia con frammentazione di mRNA e la legatura del inverso-complemento 3' adattatori alle estremità 5' dei frammenti di RNA. Poli (A)-contenente RNA è quindi inverso trascritto con un primer oligo lungo 25 nucleotidi (nt) che contiene un nucleotide di ancoraggio all'estremità 3', un dU in posizione 4 e una biotina all'estremità 5', consentendo l'associazione del cDNA di branelli magnetici streptavidina. La maggior parte del primer, tra cui la biotina, viene rimosso da cDNA tramite fenditura al dU tramite il mix di enzima di utente, contenente Uracil DNA glicosilasi (UDG) e il DNA glicosilasi-liasi endonucleasi VIII. Questa reazione lascia intatti finisce per la legatura di un adattatore per 5' e tre Ts sinistra dopo fenditura rimangono per contrassegnare la posizione della coda di poli (a). Perché schede di 5' e 3' sono collegate tramite la legatura al destinatario 5' estremità, non vengono generati dimeri di adattatore. Quattro nucleotidi casuale-mers introdotto all'inizio di letture permette la risoluzione di cluster su strumenti di sequenziamento di state-of-the-art e può anche servire come identificatore univoco molecolare (UMI) per il rilevamento e la rimozione di artefatti di amplificazione di PCR. La dimensione della Umi può essere ulteriormente aumentata come fatto in altri studi 6. Il protocollo genera letture che sono invertire complementare al mRNA 3' estremità, tutti che iniziano con un tetramero randomizzato seguito da 3 Ts. elaborazione di letture che hanno le 3 Ts diagnostica a loro 5' fine inizia con la correzione di artefatti di amplificazione di PCR di sfruttando l'UNMIS, rimozione delle sequenze 3' adattatore e invertire la complementazione. Si legge che potrebbero trarre origine da oligo adescamento a siti A ricchi interni sono anche identificati informaticamente e scartati. I siti spuri generalmente privi di uno dei 18 ben caratterizzati e segnali di poli (a) conservati che dovrebbero essere situato ~ 21 nucleotidi a Monte del clivaggio apparente sito 7.
Il protocollo richiede circa 8 h tempo di hands-on, senza contare la coltura delle cellule e le legature durante la notte. L'associato leggere analisi software consente un'identificazione del sito di poli (a) altamente accurate. Dal sito di poli (a) i cluster creato in basati a 4 campioni ulteriormente evidenziati in questa sovrapposizione di 84% manoscritto (due replicati biologici di controllo siRNA e cellule si-HNRNPC-trattate) con un gene con annotazioni e di questi, sovrapposizione di 75% con una 3' UTR e l'86% sia con un 3' UTR o un esone terminale. Il coefficiente di correlazione di Pearson di espressione di 3' estremità nei campioni replicati è 0.92, e valori di superiore allo 0,9 sono in genere ottenuti con il metodo. Così, A-seq2 è un metodo conveniente che dà risultati molto riproducibili.
Protocollo
1. crescita cellulare e mRNA isolamento
- crescere cellule secondo il vostro disegno sperimentale in piastre da 6 pozzetti a ~ 1 x 10 6 cellule per pozzetto di confluenza 80%.
- Rimuovere il mezzo di crescita e lavare le cellule una volta con tamponato fosfato salino. Direttamente lisare le cellule sul piatto aggiungendo 1 mL di tampone di lisi dal kit di mRNA-isolamento. Trasferire il viscoso lisato in un tubo di plastica da 15 mL con una pipetta da 1 mL. Utilizzare una spatola di gomma per staccare completamente la cella di materiale dalla superficie della placca.
- Cesoia il DNA viscoso contenente lisato con una siringa da 1 mL, inserita un ago ipodermico 23 G da diversi vigorosa su e giù per i movimenti dello stantuffo fino il lisato non è più viscoso. Puntare l'ago della siringa al centro della parte inferiore per evitare l'espulsione di lisato fuori dal tubo.
- Trasferire il lisato in una provetta da 1,5 mL con la siringa. Rotazione di 5 min a 20.000 x g e 4 ° C per rimuovere i detriti. Utilizzare DNA binding basso 1,5 mL flaconi in tutto il protocollo.
- Mentre la centrifuga è in esecuzione, lavare 300 µ l di sedimento oligo (dT) 25 biglie magnetiche su un rack magnetico con 500 µ l di tampone di lisi. Mescolare 2 - 3 volte a rack i tubi. Rimuovere il buffer dopo la soluzione è limpida. Raccogliere il surnatante chiaro dal passaggio 1.4 e aggiungere le perline. Risospendere e inserire le provette su una ruota rotante per 10 min.
- Posizionare i tubi su un rack magnetico. Rimuovere il liquido chiaro dopo 2 min. aggiungere 0,8 mL tampone A dal kit di mRNA-isolamento. Ruotare il tubo di 180° gradi sul rack, 2 - 3 volte. Ripetere questo passaggio di lavaggio ancora una volta con buffer di r.
- Lavare le perle 2 volte con 0,8 mL di tampone B come descritto al punto 1.6.
- Per eluire il mRNA associato dai branelli, aggiungere 33 µ l H 2 O e risospendere le perline. Riscaldare fino a 75 ° C per 5 minuti su un blocco riscaldante. Girare immediatamente le provette per 1 s e posto loro sul rack magnetico. Trasferire il surnatante in una nuova provetta. Campioni possono essere conservati a-80 ° C fino a ulteriore uso.
- Aggiungere 66 µ l di tampone idrolisi alcalina per la 33 µ l mRNA (passo 1,8), mescolare e scaldare per esattamente 5 minuti a 95 ° C su un blocco di riscaldamento. Raffreddare immediatamente tubi su Ice.
- RNA isolare con un kit di pulitura RNA.
Nota: Confermare il volume; dovrebbe essere 100 µ l. Etanolo:- aggiungere 350 µ l di tampone RLT da kit e 250 µ l. Carico sulla colonna e spin per 30 s a 8.000 x g a temperatura ambiente (TA). Lavare con 500 µ l di tampone RPE dal kit. Lavare con 500 µ l 80% di etanolo. Centrifugare per 5 min a 20.000 x g per asciugare la colonna. Aggiungere 36 µ l H 2 O a colonna e girare per 1 min a 20.000 x g. scartare la colonna e salvare l'eluato.
2. 5 ' fosforilazione e dnasi trattamento fine
- inibitore della ribonucleasi di aggiungere 5 µ l polinucleotide chinasi buffer, 5 µ l 10 mM ATP, 1 µ l, 1 µ l 2 µ l polinucleotide chinasi a e dnasi campioni e incubare a 37 ° C per 30 min. facoltativamente preparare miscele di reazione principale in tutto il protocollo mescolando 1,1 volumi x n (n = numero di campioni) di ogni componente.
- Modificare il buffer e rimuovere ATP su una colonna di spin per impedire l'aggiunta di poli (a) nel passaggio successivo.
- Prespin filare-colonne a 735 x g per 1 min. trasferire le colonne in nuove fiale da 1,5 mL e caricare le reazioni di chinasi sulle colonne. Selezione delle colonne 2 min a 735 x g. scartare le colonne e posizionare i tubi con le reazioni raccolte sul ghiaccio o conservare a -80 ° C.
3. Blocco 3 ' termina con la cordicepina trifosfato
Nota: è essenziale per bloccare il 3 ' le estremità dei frammenti di RNA per evitare loro concatemerization le reazioni di legatura successiva. 3 ' estremità che non sono già bloccate da un ( ciclico) fosfato dopo idrolisi sono trattati tramite l'aggiunta di un 3 ' nucleotide di terminatore di catena dATP (cordicepina trifosfato) con l'ausilio di poli (a) polimerasi. Qui, la polimerasi di poli (a) di lievito (yPAP), che è stato espresso e purificata come descritto in 8 è stata utilizzata ad una concentrazione di 0,5 mg/mL. Lievito o e. coli PAP entrambi hanno quasi la stessa attività per l'aggiunta di 3 ' dATP e può essere acquistato in commercio (vedere la tabella dei materiali).
Tampone di reazione della polimerasi di- 5 µ l di aggiungere 13,5 x concentrato poli (a), 2 µ l di 10 mM 3 ' dATP, inibitore di RNAsi 1 µ l e 1 µ l poli polimerasi alla reazione da punto 2.2.1. Mix e spin per 1 s. Incubare a 37 ° C per 30 min., aggiungere 32.5 µ l H 2 O ad ogni reazione. Purificare il RNA come descritto al punto 1.10.1. Eluire il RNA con 14 µ l H 2 O.
4. Legatura di Reverse 3 ' adattatori al 5 ' fine di frammenti di RNA
- posto le reazioni in un concentratore a vuoto per 10 min per ridurre il volume a 6 µ l. Add 3 µ l 10 x buffer legatura di RNA di T4, 3 µ l 10 mM ATP , 15 µ l PEG-8000, inibitore di RNAsi 1 µ l, 1 µ l di complemento inverso 0,1 mM 3 ' adattatore " revRA3 " (Vedi la tabella materiali) e ligasi di 1 µ l alta concentrazione RNA 1, mescolare.
- Incubare le reazioni a 24 ° C per 16 h su un mixer riscaldato con miscelazione intermittente a 1.000 giri/min. Aggiungere 70 µ l H 2 O ad ogni reazione e mescolare. Purificare il RNA come descritto al punto 1.10.1. Eluire il RNA con 14 µ l H 2 o. i campioni possono essere conservati a-80 ° C a questo punto.
5. Invertire la trascrizione (RT)
- posto eluati in un concentratore a vuoto per 3 min per ridurre il volume a 11 µ l. reazioni di trasferimento di 200 µ l PCR tubi. Aggiungere 1 primer di 0,05 mM RT µ l " Bio-dU-dT25 ". Riscaldare per 5 minuti a 70 ° C in un cycler PCR e RT di lasciare per 5 min.
- Aggiungere 1 µ l 10 mM dNTPs, 4 buffer di trascrittasi inversa x 5 µ l, 1 µ l 0,1 M DTT, inibitore di RNAsi 1 µ l e 1 µ l trascrittasi inversa. Mescolare e riscaldare le reazioni per 10 minuti a 55 ° C e a 10 minuti a 80 ° C in un cycler PCR. Tenere in ghiaccio o a-80 ° C per periodi più lunghi.
6. Digestione con uracile DNA glicosilasi enzima Mix
- dispensare 100 µ l streptavidina-perline in una fiala di 1,5 mL, risospendere in 800 µ l tampone di legame biotina e metterle su un ripiano magnetico. Capovolgere tubi 2 - 3 volte. Rimuovere il buffer quando chiaro. Ripetere la fase di lavaggio. Risospendere le sfere in 200 µ l tampone di legame biotina.
- Aggiungere la reazione di trascrizione inversa per la soluzione di perline e incubare per 20 minuti a 4 ° C su una ruota in movimento. Lavare le perle 2x con associazione biotina del buffer come in passo 6.1 e 2x con buffer di dieci su un rack magnetico. Risospendere le sfere in 50 µ l di tampone dieci, aggiungere 2 µ l uracile DNA glicosilasi enzima mix e Incubare 1h a 37 ° C in un mixer con miscelazione intermittente. Buffer di
- aggiungere 50 µ l di H 2 O, 11 µ l di RNasi H e 1 µ l della RNasi H alle reazioni. Incubare a 37 ° C per 20 min. tubi di posto su una griglia magnetica e trasferire il liquido contenente il cDNA fenduto ad un nuovo tubo
- purificare il cDNA fenduto.
- Aggiungere 550 µ l di tampone PB dal kit di purificazione di PCR per le reazioni di scissione. Aggiungere 10 µ l di acetato di sodio 3 M, pH 5.2 per abbassare il pH. Caricare le reazioni su colonne di spin di eluizione minimal e spin a 17.000 x g per 1 min.
- Aggiungere 750 µ l di tampone PE a colonne e spin a 17.000 x g per 1 min. scartare il flusso continuo. Girare le colonne a 17.000 x g per 1 min ad asciugare. Trasferire le colonne in una fiala di 1,5 mL, aggiungere 16 µ l di H 2 O e spin a 17.000 x g per 1 min. Posizionare le reazioni in un concentratore a vuoto per 8 min di concentrare in un volume di 7 µ l.
7. Legatura di 5 ' adattatori a 5 ' le estremità del cDNA
- il cDNA isolato, aggiungere 3 µ l 10 x buffer di T4 RNA ligasi 1, 3 µ l 10 mM ATP, 15 µ l PEG-8000, 1 µ l 50 µM " revDA5 " oligo e ligasi di 1 µ l alta concentrazione T4 RNA 1. Incubare a 24 ° C per 20 h. aggiungere 70 µ l di H 2 O ad ogni reazione. I campioni possono essere conservati a-20 ° C a questo punto.
8. Pilota PCR, amplificazione delle biblioteche e selezione dimensione
- In una reazione di pilota, determinare il numero ottimale di PCR cicli per raggiungere amplificazione biblioteca entro la fase esponenziale.
- Dispensare 25 µ l DNA polimerasi mix, reazione di legatura 20 µ l, 2 µ l di H 2 O, 1,5 µ l 10 µM avanti PCR primer (RP1) e primer di 1,5 µ l 10 µM inversa PCR indice in provetta PCR 200 µ l.
- Eseguire il ciclatore con il seguente programma: 3 min 95 ° C, seguita da 20 cicli di 20 s 98 ° C, 20 s 67 ° C e 30 aliquote di µ l ° C. raccogliere 7 s 72 dopo 6, 8, 10, 12, 14, 16 e 18 cicli direttamente dal ciclatore. Aggiungere 1 µ l 10 x caricamento buffer (glicerolo al 50%, 0,05% xilene cianolo). Nota: Si prega di seguire le raccomandazioni del fornitore se utilizza multiplexing quando si combinano i codici a barre.
- Prodotti separati in piccole fessure su un gel di agarosio 2% in 1 tampone di x TBE contenente un 01:10, 00 diluizione della tintura di verde fluorescente.
- Aliquote di carico su un 2% di agarosio gel ed esegua il gel a 100 volt per 15 min Visualize migrazione dei prodotti di PCR su un sistema di documentazione gel.
- Utilizzare il numero di cicli all'inizio di amplificazione esponenziale nella reazione pilota per una reazione di PCR su larga scala con due volte i volumi come usato per reazione del pilota ( Figura 2).
- Per le reazioni di PCR su larga scala, concentrarsi e dissalare le reazioni prima con un kit di purificazione di PCR e separare i prodotti vasta slot machine su gel di agarosio al 2% in 1 tampone di x TBE.
- Tagliato a fette del gel contenente 200-350 nt DNA prodotti. Sciogliere il gel nel buffer caotropici RT per fino a 30 min. Estrarre il DNA dal gel fette con un kit di estrazione del gel. Non scaldare a 50 ° C per evitare bias nel grippaggio del DNA A ricchi 9.
- Invia per il sequenziamento.
Nota: In genere, 50 cicli singolo-lettura (SR50) sono sufficienti (Vedi, per es., https://www.illumina.com/technology/next-generation-sequencing.html).
9. Elaborazione dati
Nota: I dati di sequenziamento risultante (in formato fastq) vengono elaborati con software disponibile nel repository gitlab (https://git.scicore.unibas.ch/zavolan_public/A-seq2-processing). L'analisi include quattro passaggi principali: (1) scaricare il repository git, (2) installazione di un ambiente virtuale, (3) impostazione di parametri specifici nel file di configurazione e (4) lanciare l'analisi attraverso ‘ snakemake ’ 10. l'intera analisi fatta nel passaggio 4 richiede un solo comando. Una dettagliata descrizione dettagliata dell'analisi può essere trovata nel file README nel repository gitlab e una breve descrizione è disponibile qui sotto. Tutte le fasi di lavorazione individuali sono compiuti tramite l'esecuzione di strumenti disponibili pubblicamente, sia da fonti esterne o preparate in casa. La pipeline di calcolo dipende da un ambiente virtuale di python 3 Basato su anaconda 11 con il snakemake pacchetto disponibile 10. Funziona su macchine con sistema operativo Unix-like ed è stato testato in un ambiente Linux con installato il sistema operativo CentOS 6.5 e 40 GB di RAM disponibile. Delle dipendenze software sono controllate automaticamente all'interno dell'ambiente virtuale. I seguenti strumenti software pubblicamente disponibili sono necessari e quindi installati insieme con l'ambiente: snakemake (v3.9.1) 10, fastx toolkit (v0.0.14) 12, STAR (v2.5.2a) 13 , cutadapt (v 1.12) 14, samtools (v. 1.3.1) 14 , 15, bedtools (v2.26.0) 16 , 17.
- Pre-elaborazione dei dati da letture a cDNA
Nota: la profondità di sequenziamento può variare tra le esecuzioni e, a seconda dello strumento, i dati da un campione possono essere suddivise in più file di sequenza. Se questo è il caso, concatenare i file che corrispondono a un campione in un singolo file di input che viene utilizzato nei passaggi seguenti.- Convertire il file da fastq in formato fasta.
- Estratto di legge con una struttura corretta (3 thymidines alle posizioni 5, 6 e 7 della lettura).
Nota: Una lettura che è stata preparata correttamente secondo il protocollo sperimentale descritto in precedenza deve avere la struttura (dal 5 ' fine): codice a barre 4-nucleotide - 3 thymidines - invertire complemento della trascrizione 3 ' fine. - Memorizzare le informazioni sul tetramero iniziale nella riga di descrizione della sequenza.
Nota: Il tetramero funge da identificatore univoco molecolare (UMI) che facilita la correzione di artefatti di amplificazione più avanti nell'analisi. - Rimuovere i primi sette nucleotidi dal leggere ' s 5 ' fine.
- Correggere per inserire gli artefatti di amplificazione conservando solo una copia della legge con la stessa sequenza e UMI.
- Rimuovere la parte dei 3 ' fine che corrisponde alla sequenza di adattatore e il complemento inverso quindi la sequenza. Procedere solo con letture che hanno una lunghezza minima (impostazione predefinita: 15 nt).
Nota: a seconda della lunghezza del frammento originale di mRNA e il numero di cicli di sequenziamento, 3 ' fine di lettura può contenere parte dei 3 ' adattatore, che è stata rimossa in questo passaggio.
- Estrarre tutte le letture che soddisfano i criteri seguenti: massimi 2 nucleotidi sconosciuti (' N '), massimo 80% come e ultimo nucleotide di lettura non A. Queste letture sono considerate di qualità sufficiente per essere utilizzato nell'analisi.
- Mappa le letture al genoma con uno strumento che gestisce impiombate letture e genera un file di output in formato BAM.
- STAR se viene utilizzato, creare un file con l'indice del genoma a cui la legge devono essere mappata. Per il genoma umano, questo passaggio richiede 35 GB di memoria (RAM).
- Mappa le letture al genoma.
Nota: (note STAR-specifiche) il Soft-clipping è disabilitato per forzare il mapping dei 3 ' fine di ogni letto come questo è il nucleotide immediatamente a Monte del sito di clivaggio.
- Convertire il BAM in un letto-file. Se una lettura esegue il mapping a più posizioni, mantenere solo quelle con il più basso modifica distanza.
Nota: Il numero di copia di lettura mappato a una posizione specifica viene utilizzato come punteggio. Letture che eseguono il mapping a più posizioni sono contati frazionata in ogni posizione con un peso pari a 1/numero di posizioni a cui una lettura mappe.
Letture di - collasso che variano da un errore probabile sequenziamento. Se due letture distinte mappano nella stessa posizione (posizione di inizio e fine dei mapping sono identiche) e condividono la stessa UMI, li considerano come PCR duplicati e tenere solo uno.
- Dedurre tutti i singoli pre-mRNA 3 ' fine elaborazione siti.
Nota: Una lettura individuale fornisce la prova per un 3 ' termina quando i suoi ultimi quattro nucleotidi vengono mappati al genoma senza errori. La posizione a cui il 3 ' fine delle mappe lettura viene memorizzato come sito di clivaggio. - Rileva 3 ' fine siti che potrebbero avere avuto origine da innesco interno. Definire il sito come artefatto di innesco interno quando il 10 nt a valle del sito di clivaggio nel genoma soddisfare uno dei seguenti criteri: contiene più di sei come, contiene sei consecutivi come o inizia con uno dei tetrameri seguenti: AAAA, AGAA, ALÒS, alaimo .
- Generare una tabella di 3 singoli ' fine elaborazione siti in letto formato.
- Identificare regolato indipendentemente i cluster del sito di poli (a).
Nota: La procedura descritta qui seguire la procedura che è stata introdotta in una pubblicazione preliminare 5.- Iniziare raccogliendo 3 singoli ' fine elaborazione siti che sono stati ottenuti in tutti i campioni dello studio.
- Annotare poli (a) noto segnali 7 nella regione di 9 nucleotidi intorno ogni 3 singoli ' sito di elaborazione end.
- Poli (a) identificare siti espressi sopra lo sfondo in ogni campione come segue.
- Ordinare i siti da loro espressione crudo all'interno del campione corrente. Attraversare l'elenco di siti da cima a fondo, associando i siti classificato inferiore con un sito Classificato al superiore se si trovano all'interno di una distanza predefinita nel genoma (impostazione predefinita: 25 nt - up o a valle) dal sito di alto rango.
Nota: Tutti i siti di basso rango, associati a un sito di alto rango definiscono un cluster in cui l'espressione è il numero di letture che documentano tutti questi siti. - Ordinare questi ammassi di espressione e attraversare l'elenco di cluster dalla più alta alla più bassa espressione, determinare la soglia di espressione c in cui la percentuale dei cluster con un poli (a) con annotazioni di segnale goccia sotto una soglia predefinita ( predefinito: 90%).
- Scartare siti da qualsiasi cluster sotto il cutoff.
- Ordinare i siti da loro espressione crudo all'interno del campione corrente. Attraversare l'elenco di siti da cima a fondo, associando i siti classificato inferiore con un sito Classificato al superiore se si trovano all'interno di una distanza predefinita nel genoma (impostazione predefinita: 25 nt - up o a valle) dal sito di alto rango.
- Cluster ravvicinati 3 ' fine siti ottenuti attraverso campioni.
Nota: Tipo 3 ' fine elaborazione siti prima per il numero di campioni di supporto e quindi la somma della normalizzazione leggere totali (letture al milione (RPM)) attraverso campioni. Attraversare l'intero elenco dall'alto verso il basso, associando i siti più bassi in classifica con più alto in classifica siti quando loro distanza per il sito di rango più elevato non è più un limite predefinito (impostazione predefinita: 12 nt). Ogni volta che uno qualsiasi dei 3 che costituiscono ' sito end si sovrappone a un segnale di poli (a) con annotazioni o ha un segnale di poli (a) direttamente a valle, il cluster corrispondente è contrassegnato per l'ulteriore ispezione rilevare innesco interno. - Poli (a) merge sito cluster.
Nota: Quando un cluster viene contrassegnato come candidato di innesco interno putativo, è fusa in un cluster a valle se i due cluster condividono i loro segnali di poli (a) o mantenuto se il sito più a valle del cluster ha un segnale di poli (a) si trova a un minimo distanza a Monte (impostazione predefinita: 15 nt). Infine, i cluster ravvicinati sono Uniti se: (i) che condividono gli stessi segnali di poli (a), o (ii) la durata del cluster risultante non superi un massimo (impostazione predefinita: 25 nt). - Negozio di cluster nel formato di file di letto con il totale normalizzato leggere il conteggio da tutti e 3 ' fine siti in ogni cluster come punteggio.
Risultati
Poli (A)-contenente RNA è stato isolato dalle cellule coltivate, frammentate di idrolisi alcalina e cDNA sono state fatte di retrotrascrizione con primer oligo (distacco). Il cDNA risultante è stato immobilizzato su perle di streptavidina, dU è stato fenduto nella reazione di asportazione specifico di uracile, adattatori erano legati al 5' e 3' estremità del frammento spaccato e gli inserti sono state sequenziate. Figura 1 raffigura un contorno grafico dell'esperimento.
Per cellule HEK293 e HeLa, 106 celle erano sufficienti per identificare i siti di poli (a) per la stragrande maggioranza dei geni di proteina-codificazione alla fine della procedura. Tuttavia, per altri tipi di cellule o tessuti che può essere necessario testare la saturazione del numero di poli (a) identificati siti come il numero di celle utilizzato nell'esperimento aumenta. Risultati rappresentativi della PCR pilota passo e del frammento del DNA analisi del campione prima di sequenziamento sono mostrati nella Figura 2.
La figura 3 Mostra i passaggi di pre-elaborazione di analisi computazionale, a partire dal file fastq ottenuti dal sequencer e terminando con le letture di qualità controllata, bordato di adattatore che sono pronte ad essere mappato il genoma. La figura 4 Mostra la procedura di analisi che iniziano con la mappatura delle letture al genoma corrispondente e fine con catalogo di siti che sono identificati in un campione particolare di elaborazione estremità 3' mRNA. Quando si analizzano campioni multipli, vengono eseguite ulteriori operazioni per abbinare all'estremità 3' elaborazione di siti che sono stati trovati nei campioni individuali e segnalare loro abbondanza attraverso campioni. Questi passaggi sono illustrati nella Figura 5.
Così, una volta che i campioni sono stati sequenziati, l'analisi della sequenziazione risultante leggere i file (in formato fastq) attraverso la pipeline di elaborazione disponibili è semplice. Dopo aver aggiunto le informazioni sugli esempi nel file di configurazione, l'esecuzione della pipeline si tradurrà in due tipi principali di file di output: 1) letto-file con tutte le 3' finiscono elaborazione siti individuati nei singoli campioni (ad es. " Sample1.3pSites.noIP.Bed.gz") e 2) un letto-file con tutti i poli (a)-cluster sito (clusters.merged.bed) attraverso tutti i campioni dello studio. La produzione comprende anche le coordinate del genoma per tutte le letture da ogni singolo campione (ad esempio "sample1. STAR_out/aligned.sortedByCoord.out.Bam") che più tardi possono essere visualizzati in un browser del genoma come IGV16. Ispezione visiva dei profili letti fornisce generalmente un primo assaggio della distribuzione dei siti di poli (a) nel genoma e i cambiamenti che si verificano al momento delle perturbazioni specifiche che sono state effettuate nello studio. Ad esempio, nella Figura 6 è illustrata la risposta di un gene specifico per il knock-down della proteina HNRNPC.
Sintesi di queste distribuzioni di genoma sono inoltre forniti (tabella 1). In particolare, i file di output nella directory "conteggi/annotation_overlap" contengono frazioni di siti che si sovrappongono con specifiche caratteristiche con annotazioni (dal gtf file fornito come input; annotati sono: 3' UTR, essone terminale, esone, introne, intergenica). Infine, per ogni campione, risultati delle fasi di lavorazione individuali vengono salvati (ad esempio "sample1.summary.tsv"). Questo include i numeri di: crude letture in ogni campione, si legge che hanno la struttura prevista dell'estremità 5', si legge che rimangono dopo essere collassato completo duplicati PCR, alta qualità legge secondo i criteri definiti al punto 9.2, legge che la mappa in modo univoco al genoma (dopo essere collassato a quelli derivanti da errori di sequenziamento, vedi punto 9.5), multi-mappatura legge (dopo essere collassato a quelli derivanti da errori di sequenziamento, vedi punto 9.5), raw (non cluster) 3' fine elaborazione siti in ogni campione, crudo 3' fine elaborazione siti senza candidati potenziali di innesco interno, unico 3' fine elaborazione siti da tutti i campioni senza innesco interno candidati e set finale di poli (a) sito cluster.
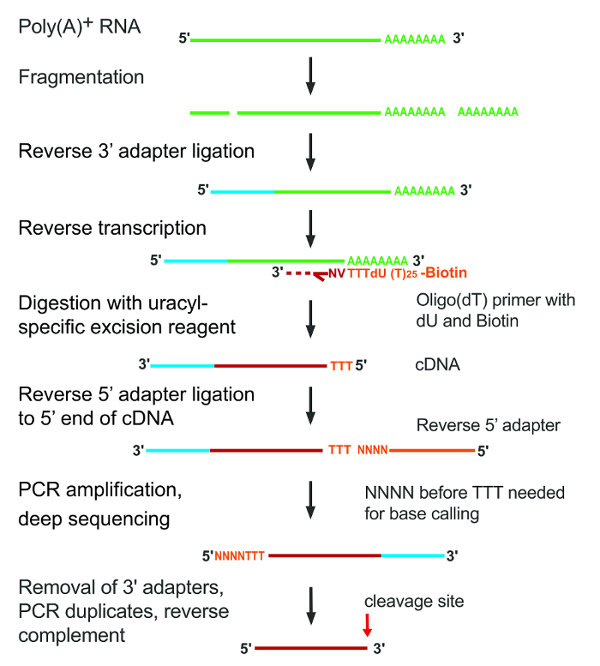
Figura 1: fasi principali del protocollo A-seq2. Singoli passi sono indicati sul lato sinistro della figura. Inserisci frammenti di RNA sono rappresentati come linee verdi che diventano rossi per cDNA dopo trascrizione inversa; gli adattatori sono colorati in azzurro o in arancione. Clicca qui per visualizzare una versione più grande di questa figura.
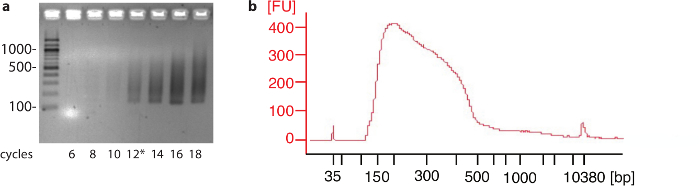
Figura 2: pilota PCR e prodotto finale profilo. (un) aliquote dalla reazione di PCR sono stati raccolte a cicli diversi e separati su gel di agarosio 2%. I numeri a sinistra indicano dimensione in nucleotidi delle rispettive bande nella scala del DNA. In questo esperimento 12 cicli (*) sono stati scelti per la reazione di PCR su larga scala. (b) esempio di un campione dopo il formato selezione correre su un analizzatore di dimensione del frammento rivelando una dimensione media di circa 280 nucleotidi. I numeri a sinistra [FU] indicano l'intensità relativa del segnale. Clicca qui per visualizzare una versione più grande di questa figura.
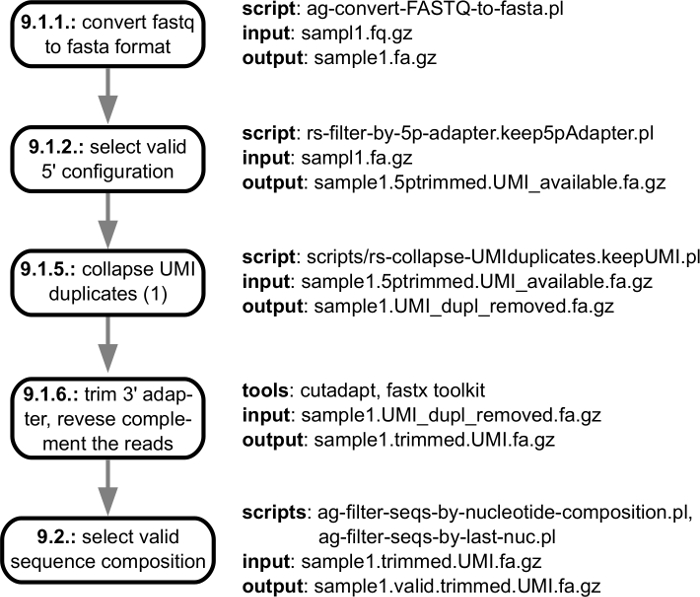
Figura 3: struttura del pre-trattamento di sequenziamento letture. Per identificare le letture di alta qualità che verranno mappate al genoma corrispondente vengono elaborati i file fastq con letture che vengono generati dal software strumento-collegata di sequenziamento. La figura mostra la specifica di ingresso/uscita delle singole fasi della pipeline, con collegamenti per i singoli passaggi del protocollo descritto nella sezione "Elaborazione dei dati". Clicca qui per visualizzare una versione più grande di questa figura.
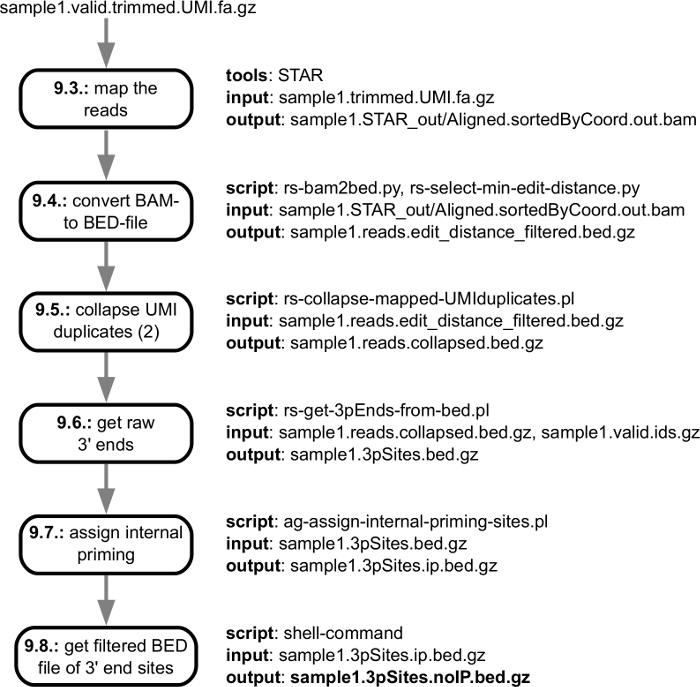
Figura 4: schema di sequenza leggere elaborazione, del passaggio di mapping per il genoma per la generazione di singoli 3' fine elaborazione siti. Nella figura viene illustrata la specifica di ingresso/uscita delle singole fasi della pipeline, con collegamenti per la individual passaggi del protocollo descritto nella sezione "Elaborazione dei dati". Il file di output principale che viene consegnato all'utente è segnato in neretto. Clicca qui per visualizzare una versione più grande di questa figura.
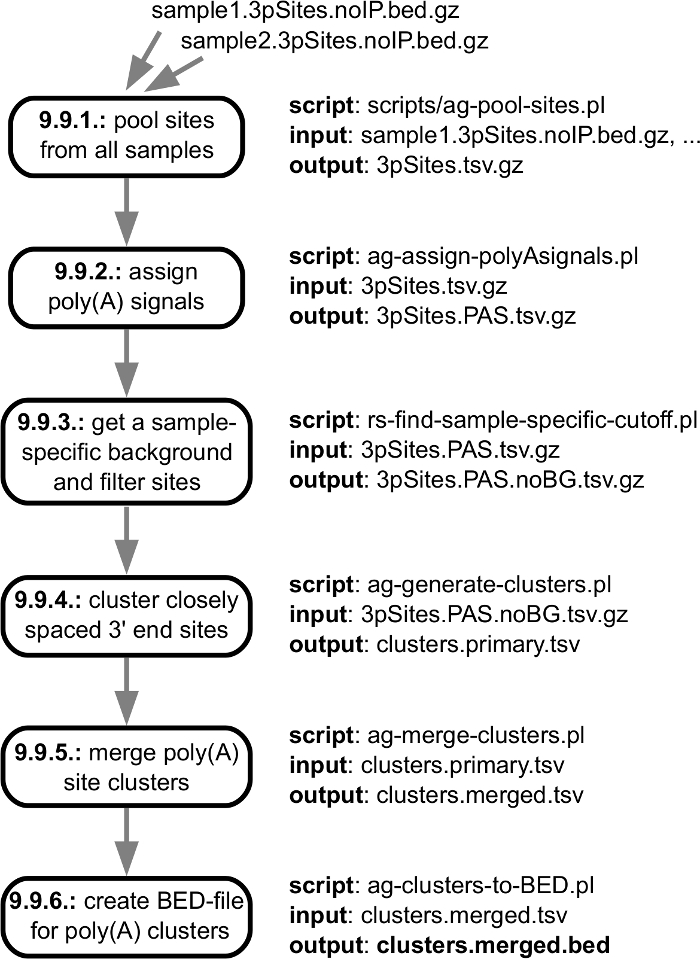
Figura 5: struttura dei passaggi che sono presi per generare cluster di co-regolati da 3' estremità sequenziamento siti. La figura mostra la specifica di ingresso/uscita delle singole fasi della pipeline, con collegamenti per i singoli passaggi del protocollo descritto nella sezione "Elaborazione dei dati". Il file di output principale è segnato in neretto. Clicca qui per visualizzare una versione più grande di questa figura.
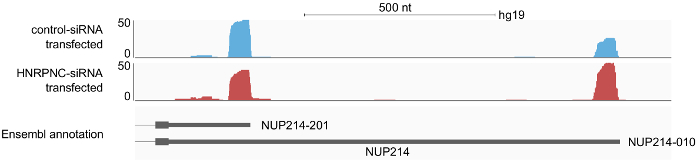
Figura 6: risultati di esempio del profilo del 3' finiscono elaborazione letture lungo il terminale esone del gene NUP214, mostrato nel browser del genoma IGV 16 . A-seq2 letture sono state preparate da due campioni di cellule HEK 293, trattate con un controllo-siRNA o con un HNRNPC siRNA. Le letture che documentato siti di poli (a) che sono stati annotati dalla pipeline di analisi erano salvate nel formato BAM che è stato utilizzato come input per il browser del genoma IGV. Le estremità 3' dei picchi letti mappa mRNA 3' estremità che vengono annotate nello Ensembl. I profili indicano un uso aumentato dell'isoforma lunga 3' UTR su HNRNPC knock-down. Clicca qui per visualizzare una versione più grande di questa figura.
| si-controllo replicare 1 | si-controllo replicare 2 | |
| ID: 29765 | ID: 32682 | |
| numero di letture crude | 44210258 | 68570640 |
| numero di letture valide dopo la rifilatura e filtraggio | 14024538 | 21211793 |
| numero di mapping in modo univoco letture | 6953674 | 13946436 |
| numero di letture mapping ai luoghi multipli | 2040646 | 2925839 |
| numero di singoli 3' fine elaborazione siti | 1107493 | 1710353 |
Tabella 1: esempio di output della pipeline analisi. Sintesi della legge che sono stati ottenuti alle singole fasi.
Discussione
La moltitudine di core e fattori ausiliari che sono coinvolti nel processamento dell'estremità 3' del pre-mRNA si riflette in un paesaggio di poliadenilazione corrispondentemente complesse. Inoltre, è sensible a reagire ai cambiamenti in altri processi come la trascrizione e splicing poliadenilazione. 3' siti di fine taglio in pre-mRNA vengono in genere identificati in base le code di poli (a) caratteristica che vengono aggiunti ai prodotti di clivaggio 5'. Maggior parte dei metodi utilizzare primer oligo (distacco) di lunghezza variabile che consentono la conversione specifica di poli (A)-contenente mRNA da cDNA in una reazione di trascrizione inversa. Un problema comune di questo approccio è innesco interno alle sequenze A ricchi conseguente siti di fenditura artifactual. Sono stati proposti due metodi che mirano ad eludere questo artefatto nella fase di preparazione del campione. Nel metodo 3P-seq 1, adattatori sono specificamente legati alle estremità delle code di poli (a) con l'aiuto di una stecca oligo seguita da digestione parziale di RNAsi T1 e trascrizione d'inversione con TTP nella reazione come l'unico deossinucleotidi. Il risultante eteroduplici poly(A)-poly(dT) quindi sono digeriti con RNAsi H e i rimanenti frammenti di RNA sono isolati, legati agli adattatori e sequenziati. Un metodo più semplice ed elegante, 2P-seq, che utilizza un primer di sequenziamento personalizzato saltando il rimanente tratto di oligo (distacco) nella reazione di sequenziamento è stato segnalato dalla stessa autori 2. In un metodo correlato, 3' legge 3, un primer insolitamente lungo di 5 noi e 45 Ts, contenente anche una biotina è temprato a RNA frammentato, seguito da lavaggi stringenti per selezionare per molecole di RNA con code di poli (a) di oltre 50 nucleotidi. Anche se 3' letture drasticamente riduce la frequenza di innesco interno, questa non elimina completamente e 3. Protocolli per l'ordinamento diretto del RNA sono stati proposti, ma le letture risultante sono brevi e hanno un alto tasso di errore e questo approccio non è stato ulteriormente sviluppato 18,19,20. PolyA-Seq e i protocolli di Quant Seq commercializzati combinano oligo (distacco) basata di adescamento con un passo di adescamento casuale per il cDNA secondo strand sintesi 20. L'uso della reazione di trascrizione inversa della interruttore modello con della trascrittasi inversa del Virus di leucemia murina Moloney (MMLV) conduce alla generazione dei cDNAs con linker in un unico passaggio e quindi nessun dimeri adattatore possono apparire nel PAS-Seq e metodi S.A.P.A.S. 21 , 22.
Il metodo A-seq2 presentato qui spicca nel suo utilizzo di un nucleotide spaccabili (dU) all'interno di un primer oligo biotinilati. Questa modifica unisce l'utilità di arricchire oligo ibridato, poliadenilazione obiettivi con la rimozione della maggior parte della sequenza di25 oligo (dT) tra i frammenti isolati prima di librerie sono preparate e la conservazione delle tre "t", che indicare la presenza precedente della coda di poli (a). Al contrario, i metodi che utilizzano la RNasi H per rimuovere poli (a) dalle molecole di RNA in modo casuale lasciano diversi come. Poiché in A-seq2, sequenziamento è fatto dall'estremità 3' dei filamenti anti-senso, siti di taglio sono preveduti per essere collocato dopo il motivo NNNNTTT all'inizio della sequenza crudo letture. I tetrameri randomizzati servono non solo per consentire base chiamata ma anche l'eliminazione degli artefatti di amplificazione di PCR. UNMIS più lunghi possono essere sistemati. La possibilità di innesco interno rimane in A-seq2 e si rivolge informaticamente, prima scartando 3' termina con una sequenza a valle genomicamente codificati, A ricchi e quindi scartando 3' fine di cluster che potrebbe essere spiegato da innesco interno presso la A-ricco di poli (a) segnale stesso. Una recente analisi dei siti di poli (a) dedotto in modo univoco da un gran numero di protocolli indica che i siti che sono unici per A-seq2 hanno la distribuzione prevista del nucleotide e la posizione all'interno dei geni, simile a altri 3' fine protocolli di sequenziamento.
Un passaggio fondamentale nella A-seq2 è la selezione di poliadenilazione RNA e la rimozione di RNA ribosomiale (rRNA) e vari piccoli RNA. Ciò avviene più facilmente da un kit di isolamento di mRNA con oligo (dT)25 biglie magnetiche. In linea di principio, RNA totale isolato con fenolo contenenti soluzioni anche dà alta qualità RNA che può essere ulteriormente sottoposto a selezione dal kit di mRNA-isolamento o dell'agarosi oligo (dT). Un passaggio che può essere variato in A-seq2 è il trattamento con idrolisi alcalina che possono essere ridotto o esteso per ottenere frammenti di RNA di diverse dimensioni. Fondamentale è anche che l'aggiunta di dATP 3' a 3' estremità dei frammenti di RNA da parte della polimerasi di poli (a) è efficiente. Nel protocollo descritto qui, questo trattamento viene applicato a tutti i frammenti di RNA, per evitare concatemerization durante la reazione di legatura. Si segnala infine che, sebbene ligasi RNA 1 è usata normalmente come una ligasi di RNA, lega anche in modo efficiente singolo DNA incagliato, come abbiamo fatto qui per legare un adattatore all'estremità 5' delle molecole di cDNA.
Così, A-seq2 è un efficiente e facile da implementare il protocollo per l'identificazione di siti di elaborazione estremità 3' del pre-mRNA. Gli sviluppi futuri potrebbero includere riducendo ulteriormente la complessità del protocollo e la quantità di materiale necessario. Set di strumenti di analisi computazionale dati ulteriormente associato abilitare l'elaborazione omogenea dell'estremità 3' letture ottenute con una vasta gamma di protocolli di sequenziamento.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori ringraziano la signora Béatrice Dimitriades per aiuto con la coltura delle cellule. Questo lavoro è stato supportato dalla Swiss National Science Foundation sovvenzioni n. 31003A_170216 e 51NF40_141735 (PRN RNA & malattia).
Materiali
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Agarose, ultra pure | Invitrogen | 16500-500 | |
| 2100 Bioanalyzer | Agilent | G2940CA | |
| Cordycepin triphosphate (3’ dATP) | SIGMA | C9137 | |
| DNA low bind vials, 1.5 ml | Eppendorf | 22431021 | |
| Dulbecco’s Phosphate Buffered Saline | SIGMA | D8637 | |
| Dynabeads mRNA-DIRECT Kit | Ambion | AM61012 | |
| GR-Green dye | Excellgen | EG-1071 | use 1:10,000 dillution |
| HiSeq 2500 or NextSeq 500 next generation sequencers | Illumina | inquire with supplier | |
| KAPA HiFi Hotstart DNA polymerase mix | KAPA/Roche | KK2602 | |
| Nuclease free water | Ambion | AM9937 | |
| Poly(A) polymerase, yeast | Thermo Fisher Scientific | 74225Z25KU | |
| Poly(A) polymerase, E.coli | New England Biolabs | M0276L | |
| Polynucleotide kinase | Thermo Fisher Scientific | EK0032 | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| RNA ligase 1, high concentration | New England Biolabs | M0437M | includes PEG-8000 |
| RNeasy MinElute RNA Cleanup kit | Qiagen | 74204 | |
| RNase H | New England Biolabs | M0279 | |
| RNasin Plus, ribonuclease inhibitor | Promega | N2618 | |
| Superscript IV reverse transcriptase | Thermo Fisher Scientiific | 18090050 | |
| Turbo DNase | Ambion | AM2238 | |
| USER enzyme mix | New England Biolabs | M5505 | |
| Dyna-Mag-2 magnetic rack | Thermo Fisher Scientific | 12321D | |
| Thermomixer C | Eppendorf | 5382000015 | Heated mixer with heated lid |
| MicroSpin columns | GE-Healthcare | 27-5325-01 | |
| Name | Company | Catalog Number | Comments |
| Buffers | |||
| Alkaline hydrolysis buffer, 1.5 x | Mix 1 part 0.1 M Na2CO3 and 9 parts 0.1 M NaHCO3. Add EDTA to 1 mM. Adjust pH to 9.2. Store aliquots at -20 °C. | ||
| 5x poly(A) polymerase buffer | Thermo Fisher Scientiific | 100 mM Tris-HCl, pH 7.0, 3 mM MnCl2, 0.1 mM EDTA, 1 mM DTT, 0.5 mg/ml acetylated BSA, 50% glycerol | |
| Biotin binding buffer | 20 mM TrisCl pH 7.5, 2 M NaCl, 0.1% NP40 | ||
| TEN buffer | 10 mM TrisCl, pH 7.5, 1 mM EDTA, 0.02% NP40 | ||
| Name | Company | Catalog Number | Sequence |
| Oligonucleotides according to Illumina TruSeq Small RNA Sample Prep Kits, for GA-IIx and Hiseq2000/2500 sequencers | Microsynth | ||
| revRA3 (RNA) | Microsynth | 5’ amino CCUUGGCACCCGAGAAUUCCA 3’ | |
| revDA5 | Microsynth | 5’ amino GTTCAGAGTTCTACAGTCCGAC GATCNNNN-3’ | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' (V = G, A or C) | |
| PCR primer forward, RP1 | Microsynth | 5' AATGATACGGCGACCACCGAGA TCTACACGTTCAGAGTTCTACAG TCCGA 3' | |
| PCR primer reverse, RPI1, barcode in bold | Microsynth | 5' CAAGCAGAAGACGGCATACGAG ATCGTGATGTGACTGGAGTTCCT TGGCACCCGAGAATTCCA 3' | |
| Name | Company | Catalog Number | Comments |
| Oligonucleotides according to Illumina TruSeq HT-Small RNA Sample Prep Kits, for HiSeq2000/2500 and NextSeq500 sequencers | |||
| HT-rev3A (DNA/RNA) | Microsynth | 5'-amino-GTGACTGGAGTTCAGACGTGTG CTCTTCCrGrAUrC-3' | |
| HT-rev5A | Microsynth | 5' amino-ACACTCTTTCCCTACACGACGCT CTTCCGATCTNNNN 3' | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' | |
| PCR primers forward (D501-506) | Microsynth or Illumina | 5'-AATGATACGGCGACCACCGAGAT CTACAC[i5]ACACTCTTTCCCTACA CGACGCTCTTCCGATCT -3' | |
| PCR primers reverse (D701-D712) | Microsynth or Illumina | 5'-CAAGCAGAAGACGGCATACGAG A[i7]GTGACTGGAGTTCAGACGTG TGCTCTTCCGATC-3' | |
| Documentation for Illumina multiplexing: | Illumina | https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/illumina-adapter-sequences_1000000002694-01.pdf |
Riferimenti
- Jan, C. H., Friedman, R. C., Ruby, J. G., Bartel, D. P. Formation, regulation and evolution of Caenorhabditis elegans 3'UTRs. Nature. 469 (7328), 97-101 (2011).
- Spies, N., Burge, C. B., Bartel, D. P. 3' UTR-isoform choice has limited influence on the stability and translational efficiency of most mRNAs in mouse fibroblasts. Genome Res. 23 (12), 2078-2090 (2013).
- Hoque, M., Ji, Z., et al. Analysis of alternative cleavage and polyadenylation by 3' region extraction and deep sequencing. Nat. methods. 10 (2), 133-139 (2013).
- Martin, G., Gruber, A. R., Keller, W., Zavolan, M. Genome-wide analysis of pre-mRNA 3’ end processing reveals a decisive role of human cleavage factor I in the regulation of 3' UTR length. Cell Rep. 1 (6), 753-763 (2012).
- Gruber, A. R., Martin, G., et al. Global 3' UTR shortening has a limited effect on protein abundance in proliferating T cells. Nat. Commun. 5, 5465 (2014).
- Kivioja, T., Vähärautio, A., et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat. methods. 9 (1), 72-74 (2011).
- Gruber, A. J., Schmidt, R., et al. A comprehensive analysis of 3' end sequencing data sets reveals novel polyadenylation signals and the repressive role of heterogeneous ribonucleoprotein C on cleavage and polyadenylation. Genome Res. 26 (8), 1145-1159 (2016).
- Lingner, J., Keller, W. 3'-end labeling of RNA with recombinant yeast poly(A) polymerase. Nucleic Acids Res. 21 (12), 2917-2920 (1993).
- Quail, M. A., Kozarewa, I., et al. A large genome center's improvements to the Illumina sequencing system. Nat. methods. 5 (12), 1005-1010 (2008).
- Rahmann, S. Snakemake--a scalable bioinformatics workflow engine. Bioinformatics. 28 (19), 2520-2522 (2012).
- . Anaconda Software Distribution Available from: https://continuum.io (2016)
- Dobin, A., Davis, C. A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Li, H., Handsaker, B., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Robinson, J. T., Thorvaldsdóttir, H., et al. Integrative genomics viewer. Nat. Biotechnol. 29 (1), 24-26 (2011).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ozsolak, F., Platt, A. R., et al. Direct RNA sequencing. Nature. 461 (7265), 814-818 (2009).
- Yao, C., Biesinger, J., et al. Transcriptome-wide analyses of CstF64-RNA interactions in global regulation of mRNA alternative polyadenylation. Proc. Natl. Acad. Sci. U. S. A. 109 (46), 18773-18778 (2012).
- Lin, Y., Li, Z., et al. An in-depth map of polyadenylation sites in cancer. Nucleic Acids Res. 40 (17), 8460-8471 (2012).
- Shepard, P. J., Choi, E. -. A., Lu, J., Flanagan, L. A., Hertel, K. J., Shi, Y. Complex and dynamic landscape of RNA polyadenylation revealed by PAS-Seq. RNA. 17 (4), 761-772 (2011).
- Fu, Y., Sun, Y., et al. Differential genome-wide profiling of tandem 3' UTRs among human breast cancer and normal cells by high-throughput sequencing. Genome Res. 21 (5), 741-747 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon