Method Article
3' 末端配列 A seq2 とライブラリの準備
要約
このプロトコルでは、マッピング mRNA 3' 末端のサイトを処理する方法について説明します。
要約
過去 10 年間の研究は、複雑で動的なさまざまな mRNA の開裂と起こる反応を明らかにしました。長い 3' 非翻訳領域 (UTRs) の Mrna は、細胞の増殖は優先的に短い 3' UTRs と成績証明書を表現に対し分化した細胞で生成されます。今起こるサイト ゲノムのマップ、mRNA の 3' 終わり処理の規制を検討し開発された、2 番目のバージョンでは seq プロトコルについて述べる。また現在このプロトコルを活用 polyadenylate (豊かに完全に処理された mRNAs のためにほとんどの哺乳類の mRNAs の器官の中に追加される poly(A)) 尾。4 番目の位置にある deoxyuracil の DNA アダプター mRNA 3' 端フラグメントの配列のための精密加工が可能します。細胞培養、一晩を含まないプロトコルは約 8 h ハンズオン時間を必要があります。それに伴い派生シーケンス データの分析のための簡単に使用できるソフトウェア パッケージを提供しています。A seq2 と関連付けられた解析ソフトウェア 10 から、条件の広い範囲で最終的に、mRNA 3' のマッピングに効率的かつ信頼性の高いソリューションを提供する6または少ないセル。
概要
キャプチャと mRNA の 3' 端のシーケンスは、mRNA プロセシングの研究と遺伝子の発現定量をことができます。真核生物 Mrna がポリ a の尾のためビードを固定化したオリゴ チミジン (cDNA 合成の主なことができます oligo(dT)) 分子と合計セル lysates から効率的に精製することができます。ただし、このアプローチには、2 つ欠点があります。最初、トラン スクリプト内部にある A の伸張できる cDNA 合成、スプリアス多サイトの結果の主要なも。第二に、均質多ストレッチは、トラン スクリプト識別のための情報されていない別のシーケンスの特定課題を提起します。ポリ a を逆転写など、これらの制限を回避するために様々 なアプローチが提案されているツインテール RNase H 消化 (3 P seq 1)、続いて 20 で終わるカスタム配列のプライマーの使用 Ts (2 P seq 2) の選択基準RNase H 消化 (3' 読み取り3)、そしてヘアピン (A seq 4) で 3' アダプターを含むオリゴ dT プライマーの使用によって続いて CU5T45プライマーで 50 以上のヌクレオチドのポリ a の尾を持つ RNA 断片。
最近開発された A seq2 方法5は多と特に発生するアダプターの自己 ligation によって生成されるダイマーの割合を最小限に抑えるために同時にシーケンスをバイパスすることを目的とするときのアダプターのモル濃度挿入濃度を上回る。この問題は、両方のアダプターが、seq2、3' アダプターが逆転写後の RNA のフラグメントと Cdna の 5' 末端に 5' アダプターの 5' 端に結紮がようにポリヌクレオチド終了の同じ型に組み合わされてときに排除できます。方法は当社従来 - seq - シーケンスが、5'-に - 3 年より便利な ' により、正確に方向制御 RNA 断片化-、ポリ a サイトの識別の高精度を維持しながら。典型的なサンプルのシーケンス処理された読み取りの約 80% はゲノムに一意にマップし、以上 20,000 多サイト クラスター、注釈付き 3' UTRs と重複の 70% 以上の識別に 。
簡単に言えば、A seq2 プロトコルは mRNA の断片化と逆の補数 3' 5' RNA のフラグメントの端にアダプターの ligation から始まります。ポリ (A)-含む Rna は、リバース 3' 末端、位置 4 dU および 5' 端、ビオチンでアンカー ヌクレオチドを含む 25 のヌクレオチド (nt) 長いランダムプライマー プライマーと転写磁気ストレプトアビジン ビーズに cDNA のバインディングが可能です。、ビオチンを含むプライマーのほとんどはウラシル DNA グリコシラーゼ (UDG) と DNA グリコシラーゼ リアーゼ エンドヌクレアーゼ VIII を含むユーザー酵素ミックスによるデュで胸の谷間で cDNA から削除されます。胸の谷間に残るポリ a テールの位置をマークするこの反応 5' アダプター、および 3 つの Ts 左の結紮のためそのまま終了を残します。受信者の 5' 端に結紮 5' と 3' の両方のアダプターが添付されているので、アダプターの二量体は生成されません。4 ヌクレオチド ランダム-mers 読み取りの初めに導入された最新のシーケンシングの楽器のクラスター分解能を可能し、検出と PCR 増幅アーチファクト除去のため分子意識 (UMI) としても利用できます。UMI のサイズは、他の研究6ではさらに増加することができます。プロトコルすべて無作為化の四量体で、5' 端始まるによって PCR 増幅デジタルアーティファクトの補正で 3 診断 Ts を持っている読み取りの 3 ts 処理に続いて始まる mRNA 3' 端に相補的な逆が読み取りを生成します。行政、3' アダプター配列除去を悪用し、逆に補完。A 豊富なサイトは内部でランダムプライマー プライミングから由来している可能性があります読み取りまた計算識別、破棄されます。偽のサイトは一般的に 18 の特徴の一つを欠いているとする必要があります保存された多信号 〜 21 ヌクレオチド上流明らか胸の谷間サイト7の。
プロトコル細胞培養と一晩をカウントされません約 8 h の実践的な時間が必要です。解析ソフトウェアで高精度な多サイト id を関連付けられている読みます。ポリ a サイトからクラスターが作成この注釈付きの遺伝子とこれらの原稿 (コントロール siRNA と si HNRNPC 処理細胞の 2 つの生物学的複製) 84% 重複と 3' UTR、75% の重複、86% どちらかとさらに強調表示 4 のサンプルに基づいて、3' UTR またはターミナル エクソン。3' 端レプリケートのサンプルでの発現のピアソンの相関係数は 0.92 と、0.9 以上の値は、通常、メソッドで取得します。したがって、A seq2、非常に再現性のある結果を与える便利な方法です。
プロトコル
1 ですセル成長および mRNA 分離
- ~ 80% 合流でよくあたり 10 の 6 セル × 1 6 ウェル プレートの実験的設計に応じて細胞を成長。 。
- は、成長培地を削除し、セルで 1 回洗浄リン酸緩衝生理食塩水。直接 mRNA 分離キットから 1 mL の溶解バッファーを追加することによって、プレート上のセルを溶解させます。転送粘性 1 mL ピペット チップを 15 mL プラスチック チューブに溶解。ゴム製へらを使用してプレート表面から細胞材料を完全にデタッチします 。
- せんライセート含む粘性 DNA ライセート、もはや粘性までいくつか上下にピストンの動き活発で 23 G 注射針に接続されている 1 mL 注射器。注射針をチューブから溶解液を取り出すように底の中心にポイントします 。
- は、注射器を使用して 1.5 mL チューブにライセート転送します。20,000 × g で 5 分間、残骸を削除する 4 ° C をスピンします。プロトコルを通して DNA 低いバインド 1.5 mL バイアルを使用します 。
- 遠心分離機の実行中は、換散バッファーの 500 μ L と磁気ラックに再懸濁オリゴ (dT) 25 磁気ビーズの 300 μ L を洗ってください。ラックに 2-3 回チューブをミックスします。ソリューションが明確だった後、バッファーを削除します。手順 1.4 から明確な上澄みを収集し、ビーズに追加します。再懸濁し、ホイールの回転で 10 分間のチューブを配置
- は、磁気ラックにチューブを置きます。2 分追加 0.8 mL バッファー A 後 mRNA 分離キットから透明な液体を削除します。2-3 回、ラックに 180 ° 度の管の電源を入れます。この洗濯、手順 A. のバッファーをもう一度
- 手順 1.6 0.8 mL のバッファー B にビードを 2 回洗浄しなさい 。 ビーズ、バインドされた mRNA を溶出する
- は、33 μ H 2 O を追加し、ビードを再停止しなさい。5 分加熱ブロック 75 ° C まで加熱します。チューブ、1 の s および場所をすぐにスピン磁気ラックにそれら。上清を新しいチューブに転送します。さらに使用するまで-80 ° c のサンプルを格納できます 。 アルカリ加水分解、33 バッファー追加 66 μ
- μ L mRNA (ステップ 1.8) 混合し、加熱ブロック 95 ° C で 5 分間まったく熱します。すぐに氷のチューブをチル
- 分離 RNA、RNA のクリーンアップ キットレンズ
。 メモ: ボリュームを確認します。それは、100 μ L をする必要があります。- 追加 350 μ L RLT バッファー キット 250 から μ L エタノール。列と 30 のスピンに負荷室温 (RT) で 8,000 の x g で s。キットから 500 μ L RPE バッファーで洗浄します。500 μ L 80% エタノールで洗浄します。列を乾燥する 20,000 × g で 5 分間スピンします。36 μ L H 2 O 列を追加、列を破棄し、溶出液を保存する 20,000 x g に 1 分のためにスピンします 。
2. 5 ' リン酸化と DNase 処理終了
- 追加 5 μ L ポリヌクレオチド バッファー、5 μ L 10 mM ATP 1 μ L リボヌクレアーゼ阻害剤 1 μ L DNase と 2 μ L ポリヌクレオチド キナーゼサンプルし、, 37 ° C で 30 分必要に応じて 1.1 ボリュームを混合することによりプロトコルを通してマスター反応混合物の準備のため n × (n = サンプル数) の各コンポーネント 。
- バッファーを変更し、次の手順でポリ a 付加しないようにスピン列に ATP を削除します。
- Prespin スピン - 735 x g で 1 分間に列は列を新しい 1.5 mL バイアルに転送し、列にキナーゼ反応を読み込みます。スピン 735 x g に 2 分の列列を破棄収集反応チューブを氷の場所や-80 デパート ° C
3。ブロック 3 ' 三リン酸コルジセピンで終わる
注: ブロック、3 が不可欠です ' 以降ライゲーション反応 3 で自分の concatemerization を避けるために RNA のフラグメントの端 '、(既にブロックされていない終了。3 の付加によって扱われる加水分解後の繰返し) リン酸 ' dATP (三リン酸コルジセピン) チェーン ターミネーター塩基ポリ a ポリメラーゼの助けを借りて。ここでは、0.5 mg/mL の濃度で表現され、 8 で説明されているように精製酵母ポリ a ポリメラーゼ (yPAP) が使用されました。酵母や 大腸菌 PAP 両方 3 を追加するためほぼ同じ活動がある ' dATP をし、商業的に購入することができます (資料の表を参照してください).
- 追加 13.5 μ L 5 倍濃縮多ポリメラーゼ反応バッファー、10 mM 3 の 2 μ L ' dATP、1 μ L RNase 阻害剤との反応に 1 μ L のポリ a ポリメラーゼ ステップ 2.2.1。ミックスと各反応を 30 分追加 32.5 μ L H 2 O の 37 ° C で 1 s. 加温のためのスピン。1.10.1 のステップのように RNA を浄化します。溶出が 14 μ L H 2 o ・ RNA
4。逆に 3 の結紮 ' 5 アダプター ' RNA のフラグメントの末尾
- が 6 μ L 追加 3 μ l 10 x T4 RNA 結紮バッファー、3 μ l の 10 mM ATP にボリュームを減らすために 10 分間真空濃縮、反応。、15 μ L 0.1 mM 逆の補数 3 1 μ、1 μ L RNase 阻害剤 PEG 8000 ' アダプター " revRA3 " (材料表参照) 1 μ L 高濃度 RNA リガーゼ 1、ミックスと。
- では、1,000 rpm で断続的な混合加熱ミキサー上で 16 時間 24 の ° C で反応を孵化させなさい。各反作用に 70 μ L H 2 O を加え、混ぜます。1.10.1 のステップのように RNA を浄化します。14 μ L H 2 o. サンプルを-80 ° C でこの時点で格納できる RNA を溶出します 。
5。逆に転写 (RT)
- 場所 11 μ。 転送に 200 μ l の PCR 反応に、量を削減する 3 分の真空濃縮で溶出チューブ。1 μ L 0.05 mM RT プライマーを追加 " バイオ デュ dT25 "。PCR サーマルサイクラーの 70 ° C で 5 分間加熱し、5 分の RT のまま
- は、1 μ L の 10 mM dNTPs、4 μ L 5 x 逆転写酵素バッファー、1 μ L 0.1 M DTT、1 μ L RNase 阻害剤と 1 μ L 逆転写酵素を追加します。ミックスし、PCR サーマルサイクラーの 80 ° c 10 分 55 ° C まで、10 分の反応の熱します。氷の上や長期保管-80 ° C で維持します 。
6。ウラシル DNA グリコシラーゼ酵素ミックス消化
- ピペット 100 μ L 1.5 mL バイアルにストレプトアビジン ビーズ 800 μ L ビオチン結合バッファーで再懸濁し、磁気ラックに置き。2-3 回チューブを反転します。クリア時のバッファーを削除します。洗浄手順を繰り返します。200 μ L ビオチン結合バッファーのビードを再停止しなさい 。
- は、ビーズ ソリューションへの逆転写反応を追加し、ホイールの回転に 4 ° C で 20 分を孵化させなさい。2 倍のビオチン結合バッファーでビーズを踏む磁気ラック 6.1 および 2 x 10 のバッファーで洗浄。50 μ L の 10 のバッファーのビードを再懸濁します 2 μ L ウラシル DNA グリコシラーゼ酵素ミックスを追加して、断続的な混合ミキサーで 37 ° C で 1 時間インキュベートします 。
- 追加 50 μ L H 2 O、リボヌクレアーゼ H の 11 μ L バッファー、1 μ L RNase H、反応します。磁気ラックに 20 分位チューブの 37 ° C で孵化させなさい、新しいチューブに劈開の cDNA を含有する液体を転送
- 劈開の cDNA を浄化します。
- 胸の谷間反応する PCR 精製キットの PB のバッファーの追加 550 μ L。10 μ L の 3 M 酢酸ナトリウム、pH が 5.2 pH を下げるために追加します。反応をロードします。最小限の溶出スピン列と 1 分 17,000 x g でスピン
- 列および 17,000 x g で 1 分間にスピンを追加 750 μ L バッファー PE を流れを破棄します。乾燥する 1 分 17,000 x g で列をスピンします。1.5 mL のバイアルに列を転送、16 μ L H 2 O および 17,000 x g で 1 分間でスピン 7 μ L のボリュームに集中する 8 分の真空濃縮で反応の場所を追加します 。
7。5 の結紮 ' 5 アダプター ' cDNA の端
- 分離 cDNA T4 RNA リガーゼ 1 バッファー、3 μ L 10 mM ATP 15 μ L x 3 μ 10 を追加 PEG-8000、1 μ L 50 μ M " revDA5 " オリゴ、、1 μ L 高濃度 T4 RNA リガーゼ 1。H 2 O 各反応に 24 ° C、20 h. 追加 70 μ L インキュベートします。サンプルは-20 ° C でこの時点で保存できます 。
8。PCR 増幅のライブラリ、サイズ選択をパイロット
- パイロットの反応指数段階の増幅をライブラリに到達するサイクルの PCR の最適な数を決定します。
- ピペット 25 μ L の DNA ポリメラーゼのミックス、20 μ L の ligation の反作用、2 μ L H 2 O、1.5 μ 10 μ M 前方 PCR プライマー (RP1)、1.5 μ 10 μ M 逆 PCR インデックス プライマー 200 μ L の PCR チューブにします 。
- は、次のプログラムで、サーマルサイクラーを実行: 95 ° C、20 の 20 のサイクルが続く 3 分 s 98 ° C、20 s 67 ° C と 30 s 72 ° c. を集める 7 μ 因数 cycler から直接 6、8、10、12、14、16、18 のサイクル後。バッファー (50% のグリセロール、0.05% キシレンシアノール) x 1 μ 10 を追加します。注: 場合はバーコードを結合するときに多重化を使用してサプライヤーの推奨事項に従ってください 。
- 、1:10、緑の蛍光染料の 00 希釈を含む 1 x TBE バッファーに 2% の agarose のゲルの小さなスロット別々 の製品。
- ロード因数 2% の agarose のゲル、ゲルのドキュメンテーション システムの 100 ボルト 15 分 PCR の製品の可視化移行のためのゲルを実行します 。
- パイロットの反応 ( 図 2) のボリューム 2 倍に大規模な PCR の反作用のためのパイロットの反応指数関数的増幅の先頭サイクル数を使用して。
- 大規模な PCR の反作用の集中、PCR 精製キットの最初の反応を脱塩し、1 x TBE バッファー内の 2% アガロースゲル上ワイド スロット上の製品を分離します 。
- 200-350 nt DNA を含むゲルのスライスをカット製品。最大 30 分間常温カオトロ ピック バッファーのゲルを溶かします。ゲル抽出キットとゲルのスライスから DNA を抽出します。A 豊富な DNA 9 のバインディングでバイアスを防ぐために 50 ° C に加熱しないでください 。 シーケンスの
- 送信します
。 注: 通常、50 サイクル シングル読み取り (SR50) は十分です (例えば、については https://www.illumina.com/technology/next-generation-sequencing.html を参照).
9。データ処理
注: gitlab リポジトリ (https://git.scicore.unibas.ch/zavolan_public/A-seq2-processing) で使用可能なソフトウェア (fastq 形式) での結果のシーケンス データが処理されます。分析には 4 つの主な手順が含まれています: (1) ダウンロード git リポジトリ、仮想環境のインストール (2) (3) 特定のパラメーターの設定構成ファイルと (4) で起動による分析 ‘ snakemake ’ 10. ステップ 4 で実行全体の分析 1 つだけコマンドが必要です。分析の詳細なステップバイ ステップの説明は、gitlab リポジトリ内の README ファイルで見つけることができます、簡単な説明は以下。すべての個別の処理ステップが公開されているツールは、いずれかの外部ソースからの実行あるいは社内準備します。計算のパイプラインは、snakemake パッケージの利用できる 10 anaconda ベース 11 python 3 仮想環境に依存します。それは Unix ライクなオペレーティング システムを持つマシン上で実行し、インストールされている CentOS 6.5 オペレーティング システムと Linux 環境で 40 GB 使用可能な RAM 試験しました。ソフトウェアの依存関係は、仮想環境内で自動的に制御されます。次の公に利用可能なソフトウェア ツールが必要でありそれにより環境と共にインストール: snakemake (v3.9.1) 10, fastx ツールキット (v0.0.14) 12 星 (v2.5.2a) 13、cutadapt (v1.12) 14, samtools (v1.3.1) 14 , 15, bedtools (v2.26.0) 16 , 17.
- Cdna を読み取りからデータ前処理
注: シーケンスの深さは実行の間に異なる場合があり、楽器によって 1 つのサンプルからのデータを複数のシーケンスのファイルに分割できます。この場合は、次の手順で使用されている 1 つの入力ファイルに 1 つのサンプルに対応するファイルを連結します。- Fastq から fasta 形式にファイルを変換します 。
- (読み取りの位置 5、6、7 で 3 thymidines) の正しい構造を持つエキスを読み取ります
。 メモ: 上記実験的プロトコルに従って準備が正しく読み取りが必要構造 (5 から ' 終わり): 4 ヌクレオチド バーコード - 3 thymidines - 逆に成績証明書 3 の補数 ' 終わり 。
- は、シーケンスの説明の行で開始四量体に関する情報を格納します
。 注: 後における増幅デジタルアーティファクトの補正を容易にするユニークな分子識別子 (UMI) として、テトラマー 。
- 読んでから最初の 7 つのヌクレオチドを削除 ' s 5 ' 終了します 。
- と同じ読み取りの 1 つだけのコピーを保つことによって増幅アイテム挿入のシーケンスと海を修正します 。
- 3 の部分を削除する ' エンド アダプター シーケンスとその逆の補数シーケンスに一致します。のみ、最小長さの読み取りを続行 (既定: 15 nt).
注: 元の mRNA フラグメントと、3 サイクル シーケンス数の長さに応じて '、読み取りの終了は 3 の部分を含めることができます ' アダプターは、この手順で削除されます 。
- 次の条件を満たすすべての読み取りを抽出: 最大 2 不明なヌクレオチド (' N ')、として、最大 80% と読むない A. 最後のヌクレオチドこれらの読み取り、解析で使用される十分な品質であることと見なされます 。
- 読み取りをスプライシングの読み取りを処理し、BAM 形式で出力ファイルを生成するツールをゲノムにマップします。
- スターの場合は、読み取りする必要がありますマップするゲノムのインデックス ファイルを作成します。人間のゲノムのこのステップは 35 GB のメモリ (RAM) を必要があります 。
- は、読み取りをゲノムにマップします
。 注: (星固有の注意事項) ソフト クリップは 3 のマッピングを強制するために無効になって ' それぞれの終わりを読んでこれはヌクレオチド胸の谷間サイトのすぐ上流 。
- BAM をベッド ファイルに変換します。読み取りは、複数の場所にマップする場合これら最低の編集距離だけを維持します
。 注: 特定の場所でマップを読むのコピー数はスコアとして使用されます。複数の場所にマップされる読み取り、読み取りのマップ先の場所の 1/数と等しい重量の各場所で分数カウントされます 。
可能性が高い順序の誤りによって異なる - 崩壊を読み取ります。同じ場所に 2 つの異なる読み取りマップする場合 (マッピングの開始と終了位置が同じ) と同じ海を共有、PCR 重複としてそれらを考慮して 1 つしか保存します 。
- 推論すべての個々 の mRNA 3 ' 処理サイトを終了します
。 注: 個々 の読みは 3 で証拠を提供する ' その最後の 4 つのヌクレオチドがエラーなしゲノムにマップされるときに終了します。先の位置、3 ' 読み取りのマップの端、胸の谷間のサイトとして格納されます 。
- 検出 3 ' 内部プライミングから由来するかもしれないサイトを終了します。10 ゲノムで胸の谷間サイトの下流に nt を満たすとき内部のプライミング加工品としてサイトを定義する次の条件の 1 つ: として六つ以上が含まれています、6 年連続を含むまたは次テトラマーのいずれかから始まります: AAAA, AGAA, 佐賀, AAAG.
- 3 の個々 のテーブルを生成 ' ベッド形式の処理サイトを終了します 。
- 識別がポリ a サイト クラスターを個別調整
。 注: ここで説明する手順に従って前の発行 5 で導入された手順に従います。- 個々 の 3 を集めることによって開始 ' 研究のすべてのサンプルで得られた処理サイトを終了します 。
- 9 の地域で知られている多信号 7 に注釈を付ける各個別 3 周りヌクレオチド ' エンド処理サイト 。
- 識別多サイト各サンプルの背景の上次のように表されます。
- は、現在のサンプル内の元の式でサイトを並べ替えます。上から下、ゲノムのあらかじめ定義された距離内にある場合より高いランク付けされたサイトとランクの低いサイトを関連付けるサイトのリストを走査 (既定: 25 nt アップまたはダウン ストリーム) 上位サイトから
。 注: 上位サイトに関連付けられたすべての低ランキングのサイト定義式はこれらのサイトのすべてを文書化する読み取りの数クラスター 。
- 式でこれらのクラスターを並べ替えるし、最高から最低式に式しきい値 c で注釈付きポリ a を持つクラスターの割合信号定義済みしきい値 (下のドロップを決定するクラスターのリストを走査既定値: 90%).
- サイトのカットオフ以下の任意のクラスターからを破棄します 。
- は、現在のサンプル内の元の式でサイトを並べ替えます。上から下、ゲノムのあらかじめ定義された距離内にある場合より高いランク付けされたサイトとランクの低いサイトを関連付けるサイトのリストを走査 (既定: 25 nt アップまたはダウン ストリーム) 上位サイトから
- クラスター近接 3 ' のサンプルの間で得られるサイトを終了します
。 注: 並べ替え 3 ' 終了のサンプルをサポートの数し、正規化の合計によってサイトをまず処理カウントを読む (あたりの読み取り数百万 (RPM)) のサンプルの間で。上から順に、上位サイトへの間隔で定義済みの上限よりも大きくない場合、低ランクのサイトをより高いランク サイトに関連付けるリスト内の移動 (デフォルト: 12 nt)。いずれかの構成する 3 ' エンド サイト注釈付き多信号と重複または多信号の直接内部のプライミングを検出する詳細な検査に対応するクラスターをマークする下流 。
- マージ多サイト クラスター
。 注: クラスターが推定内部プライミング候補としてマークされると、それが 2 つのクラスターを共有、多信号に応じて下流クラスターにマージまたはクラスターで最もダウン ストリーム サイト最低に位置する多信号がある場合は保持上流の距離 (既定: 15 nt)。場合に密接に間隔をあけられたクラスターを結合する最後に、: 同じ多込むを共有 (i) または (ii) 結果のクラスターのスパンが最大値を超えない (既定: 25 nt). - すべての 3 からカウントを読む正規化総ベッド ファイル形式でクラスターを保存 ' スコアと各クラスター内のサイトを終了します 。
結果
ポリ (A)-アルカリ加水分解による断片化の培養細胞から分離された RNA を含むと Cdna のランダムプライマー プライマーが付いているトランスクリプションを逆で作られました。結果として得られる cDNA はストレプトアビジン ビーズに固定化した、ウラシル特定切除反応でデュ開裂、5 ' アダプターを結紮、3' 端に裂かれたフラグメントの挿入されました。図 1は、実験のグラフィカルな概要を示しています。
Hela 細胞 HEK293 細胞の 10 の6セルされたプロシージャの終わりに蛋白質のコーディングの遺伝子の大半のためのポリ a サイトを特定するのに足りる。ただし、他の種類の細胞や組織の識別された多サイト数実験で使用されているセルの数と彩度をテストする必要があります増加します。パイロットの PCR の代表の結果のステップし、DNA のフラグメントのシーケンスの前にサンプルの分析は、図 2に示します。
シーケンサーから得られるゲノムにマップする準備ができている品質チェック、アダプターによりトリミングされた読み取りで終わる fastq ファイルから始まって計算の分析の前処理のステップを図 3に示します。図 4は、mRNA 3' 末端処理特定のサンプルで特定されたサイトのカタログに対応するゲノムと末尾に読み取りのマッピングで始まる解析手順を示します。複数のサンプルを分析するときの追加手順が個々 のサンプルで発見されたサイトを処理して、3' 末端に一致し、サンプルの間で彼らの余剰を報告する実行されます。これらの手順は、図 5のとおりです。
したがって、サンプル化されている、利用可能な処理パイプラインを通じて (fastq 形式) のファイルを読み取り結果のシーケンスの分析は簡単です。サンプルについての情報を構成ファイルに追加すると、パイプラインの実行は出力ファイルの 2 つの主なタイプになります: 1) ベッド-ファイルとすべて 3' 終了 (は例えば「個々 のサンプルの識別処理サイトsample1.3pSites.noIP.bed.gz「)、2) 研究のすべてのサンプルの間ですべてのポリ a サイト クラスター (clusters.merged.bed) とベッド ファイル。出力では、すべての読み取り (例: "sample1 それぞれの個々 のサンプルからのゲノム座標も含まれています。STAR_out/Aligned.sortedByCoord.out.bam") その後 IGV16のようなゲノムのブラウザーで表示できます。読み取りのプロファイルの外観検査は一般的にポリ a ゲノムの研究で実施された特定の摂動によって発生する変更サイトで配信の最初の一見を提供します。たとえば、図 6の HNRNPC 蛋白質のノックダウンする特定の遺伝子の応答が表示されます。
これらのゲノム広い分布の概要は (表 1) を設けています。具体的には、"カウント/annotation_overlap"ディレクトリ内の出力ファイルを含む特定の注釈機能と重複サイトの分数 (gtf ファイルから入力として提供; 注釈: 3' UTR、ターミナルのエクソン、エクソン、イントロン、遺伝子間)。最後に、各サンプルは、個別の処理ステップの結果は、("sample1.summary.tsv"など) をも保存されます。これの数が含まれています: ゲノムに一意に対応する各サンプル、5' 末端の予想される構造の読み取り、読み取りにおける PCR 重複を崩壊した後で、読み取り、高品質 9.2 のステップで定義されている条件に従って読む読み取り(シーケンス エラーに起因するそれらを崩壊した後手順 9.5 を参照)、複数のマッピングを読み取り (シーケンス エラーに起因するそれらを崩壊した後手順 9.5 を参照)、生 (クラスター化されていない) 3' 端の各サンプルでは、raw 3' 末端処理サイトのサイトを処理候補内部プライミングすることがなくユニークな 3' 終了処理サイト内部プライミング候補と最終的なポリ a サイトのクラスターのセットなしすべてのサンプルから。
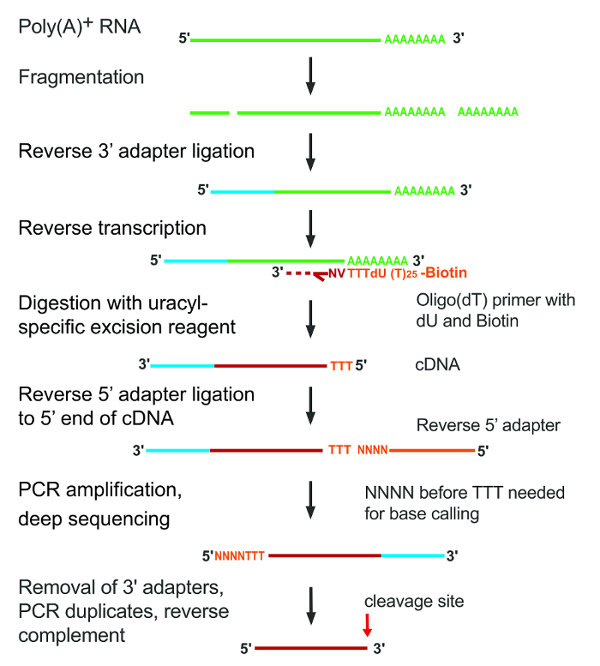
図 1: A seq2 プロトコルの主な手順です。個々 のステップは、図の左側に示されます。挿入の RNA のフラグメントは、逆のトランスクリプション; 後の cDNA の赤く緑の線として描かれています。アダプターは、明るい青またはオレンジに着色されています。この図の拡大版を表示するのにはここをクリックしてください。
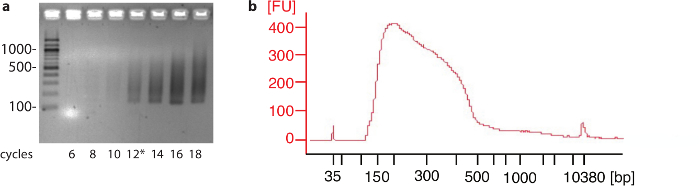
図 2: パイロット PCR および最終製品プロファイル。PCR の反作用から (、) の因数は異なるサイクルで収集された、2% アガロースゲル上区切られます。左に数字は、ヌクレオチド DNA の梯子のそれぞれのバンドのサイズを示します。この実験で 12 サイクル (*) は、大規模な PCR の反作用のため選ばれました。サイズの後サンプルの例 (b) 選択は、明らかに平均サイズは約 280 のヌクレオチドのフラグメント サイズ アナライザーで実行します。[FU] 左に数字は相対的な信号強度を示します。この図の拡大版を表示するのにはここをクリックしてください。
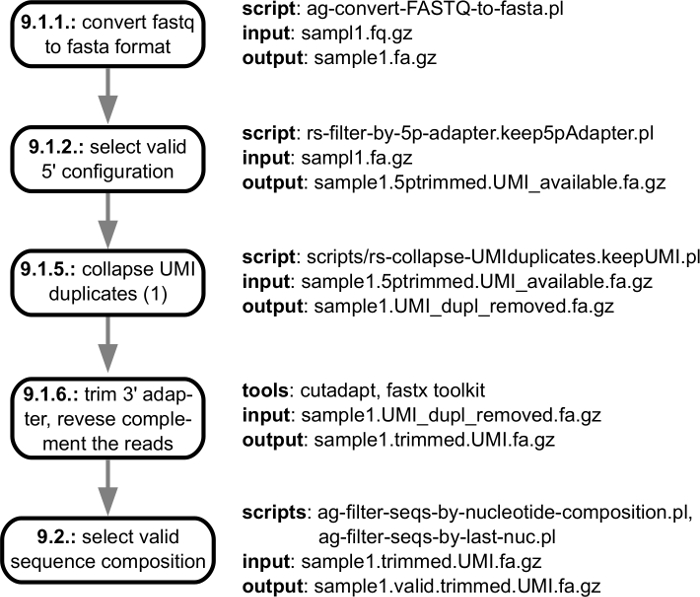
図 3: 読み取りをシーケンシングの前処理のアウトライン。シーケンサー機器関連ソフトウェアによって生成される読み取り fastq ファイルは、対応するゲノムにマップする高品質の読み取りを識別するために処理されます。「データ処理」に記載されているプロトコル個々 手順へのリンクが、パイプライン個々 ステップの入力/出力仕様を示します。この図の拡大版を表示するのにはここをクリックしてください。
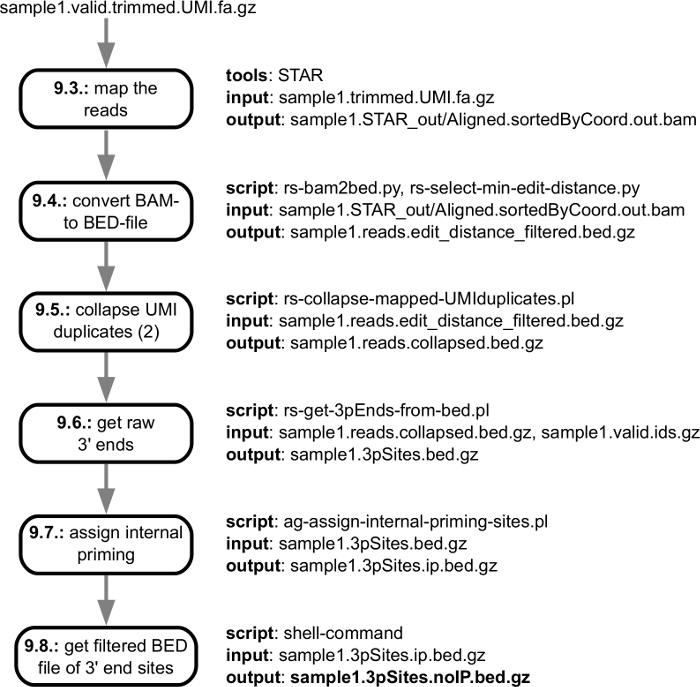
図 4: シーケンスの概要読んで処理、個別 3' 末端の世代にゲノムにマッピングの手順から処理サイト。図は i へのリンクと、パイプラインの個々 のステップの入力/出力仕様「データ処理」に記載されているプロトコル個々 手順を実行。ユーザーに配信される主な出力ファイルは、太字で示されます。この図の拡大版を表示するのにはここをクリックしてください。
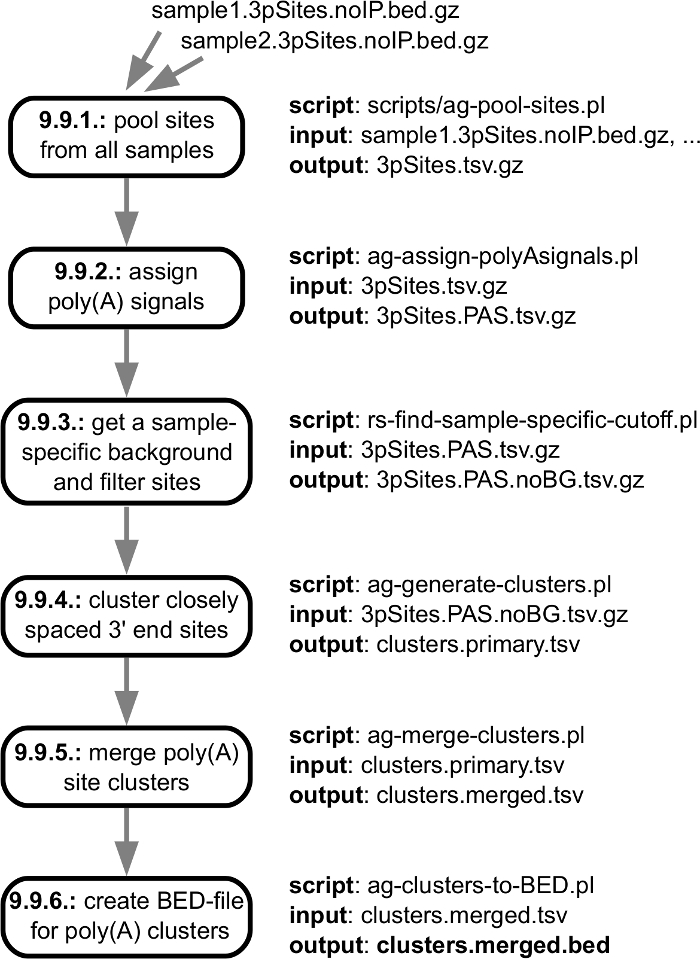
図 5: 共同規制 3' 末端配列サイトのクラスターを生成する撮影されている手順の概要です。「データ処理」に記載されているプロトコル個々 手順へのリンクが、パイプライン個々 ステップの入力/出力仕様を示します。メイン出力ファイルは、太字で示されます。この図の拡大版を表示するのにはここをクリックしてください。
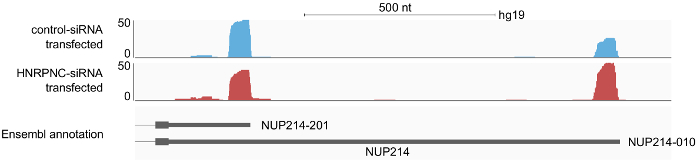
図 6: 3' のプロファイルの例の結果終了 IGV 16ゲノム ブラウザーに表示される NUP214 遺伝子のターミナルのエクソンに沿って処理読み取ります。A seq2 読み取り HEK 293 細胞、コントロール siRNA または HNRNPC siRNA との 2 つのサンプルの調製。解析パイプラインによって注釈が付けられる多サイト記載されている読み取り IGV のゲノムのブラウザーへの入力として使用された BAM 形式で保存されました。MRNA 3' にマップ読み取りピークの 3' 端 Ensembl で注釈が終了します。プロファイルは、HNRNPC ノック ダウン時に長い 3' UTR アイソの使用の増加を示します。この図の拡大版を表示するのにはここをクリックしてください。
| si コントロール複製 1 | si コントロール複製 2 | |
| id: 29765 | id: 32682 | |
| raw 読み取りの数 | 44210258 | 68570640 |
| トリミングとフィルタ リングは有効な読み取りの数 | 14024538 | 21211793 |
| 一意にマッピングの読み取り数 | 6953674 | 13946436 |
| 多遺伝子座にマップの読み込み回数 | 2040646 | 2925839 |
| 個別 3' 末端処理サイトの数 | 1107493 | 1710353 |
表 1: 解析パイプラインの出力例。個々 のステップで得られた読み取りの概要。
ディスカッション
コアや mRNA の 3' 終わり処理に関与している補助要因の多くは、それに応じて複雑な起こる風景に反映されます。また、起こるも転写、スプライシングなど他のプロセスの変化に敏感です。Mrna の 3' 終わり胸の谷間サイトは通常 5' 開裂製品に追加される特性多尾に基づいて識別されます。ほとんどのメソッドを使用して、ポリ (A) の特定の変換を許可する変数の長さのランダムプライマー プライマー-逆転写反応の Cdna に Mrna を含みます。このアプローチの一般的な問題は、人工胸の谷間サイトの結果の豊富なシーケンスに内部起爆剤です。サンプル準備の段階でこのアーティファクトを回避することを目指す 2 つの方法が提案されています。3 P seq の方法1のアダプターが具体的にポリ a の尾の部分の RNase T1 消化し、唯一の deoxynucleotide として反作用で TTP と逆のトランスクリプションが続くスプリント oligo の助けを借りての両端に組み合わされて。結果 poly(A)-poly(dT) heteroduplexes は、RNase H で消化され、残りの RNA のフラグメントの分離、アダプターに結紮およびシーケンスします。シンプルでエレガントな方法、2 P seq、シーケンシング反応で残りのランダムプライマー ストレッチは同じ作者2によって報告されたスキップ カスタム シーケンス プライマーを使用します。関連メソッドで 3' 35 の異常に長いプライマー私たちと読み取り 45 Ts、また、ビオチンを含む 50 以上のヌクレオチドのポリ a の尾を持つ RNA 分子を選択する厳しい洗浄に続いて、断片化された RNA に焼鈍します。3' 読み取り内部プライミングの周波数を大幅に短縮、なくなるわけでなく完全にそれ3。直接 RNA 配列のためのプロトコルも提案されているが、結果の読み取りエラー率が高いをして、短いとこのアプローチされていないさらに先進18,19,20。ポリア Seq と製品化のクオンツ Seq プロトコル cDNA の第 2 鎖合成20のランダムなプライミング手順に基づくランダムプライマー プライミングを兼ね備えています。モロニー マウス白血病ウイルス (MMLV) 逆転写酵素とテンプレート スイッチの逆転写反応の使用単一ステップのリンカーと Cdna の生成につながるし、それにより PAS Seq および SAPAS メソッドでないアダプター ダイマーが表示21,22。
A seq2 メソッドは、ビオチン化ランダムプライマー プライマー内劈開ヌクレオチド (dU) の活用で際立ってを紹介しました。この変更を組み合わせたランダムプライマーを交配、ライブラリを準備する前に孤立した断片からオリゴ (dT)25シーケンスのほとんどの除去と polyadenylated ターゲットおよび 3 Ts の保全の充実のユーティリティをポリ a の尾の前の存在を示します。対照的に、ランダムに RNA 分子からポリ a を削除するリボヌクレアーゼ H を利用する方法をいくつかとして残します。A seq2、シーケンスはアンチセンス鎖の 3' 末端から行われる、ので、胸の谷間のサイトは塩基配列読み取りの先頭 NNNNTTT モチーフの後に位置する予測されます。無作為化のテトラマー PCR 増幅アーティファクトの除去も呼び出す基本を許可するだけではなくサービスを提供します。長い行政にも対応できます。内部プライミングの可能性 A seq2 内に残りますと負荷に対処は、最初 3' を破棄して終了で内部のプライミングによって説明することができる 3' 側クラスターを破棄して、genomically でエンコードされた、A 豊富な下流のシーケンスと、A 豊富な多信号自体。多数のプロトコルによって一意に推論多サイトの最近の分析は、A seq2 に固有のサイトが予想されるヌクレオチドの分布とシーケンス プロトコルを終了し遺伝子のような他の 3' 内の場所をいるを示します。
A seq2 の重要なステップは、polyadenylated RNA の選択とリボソーム Rna と様々 な低分子 Rna の除去です。オリゴ (dT)25磁気ビーズの mRNA 分離キットでこれは最も簡単です。原則として、ソリューションも含むフェノールで分離された総 RNA は、高品質の mRNA 分離キットまたはオリゴ (dT) agarose によって選択をさらに受けることが RNA を与えます。A seq2 に変化するステップは、短縮または拡張サイズの異なる RNA のフラグメントを取得することができるアルカリ加水分解による治療です。重要なは、3' 3' ポリ a ポリメラーゼによって RNA のフラグメントの端に dATP 添加効率がよいこともです。ここで説明したプロトコルで、この治療法は ligation の反作用の間に concatemerization を避けるために、すべての RNA のフラグメントに適用されます。最後に、我々 はここで cDNA 分子の 5' 端にアダプターを縛ること事がある、RNA リガーゼ 1 は RNA リガーゼとして通常使用されますが、また効率的に単一の鎖の DNA を郭を注意します。
したがって、A seq2 は、効率的で mRNA 3' 末端処理サイトの識別のためのプロトコルを実装する簡単です。今後の展開は、プロトコルと必要な材料の量の量をさらに削減を含めることができます。さらの計算データ解析ツールの関連するセットは、3' 末端のさまざまなプロトコルを用いての読み取りをシーケンスの均一処理を有効にします。
開示事項
著者が明らかに何もありません。
謝辞
著者らは、細胞培養のヘルプについて夫人 Béatrice Dimitriades をありがとうございます。この作品は、スイスの全米科学財団の補助金 #31003A_170216 と 51NF40_141735 によって支持された (国立がん RNA & 病)。
資料
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Agarose, ultra pure | Invitrogen | 16500-500 | |
| 2100 Bioanalyzer | Agilent | G2940CA | |
| Cordycepin triphosphate (3’ dATP) | SIGMA | C9137 | |
| DNA low bind vials, 1.5 ml | Eppendorf | 22431021 | |
| Dulbecco’s Phosphate Buffered Saline | SIGMA | D8637 | |
| Dynabeads mRNA-DIRECT Kit | Ambion | AM61012 | |
| GR-Green dye | Excellgen | EG-1071 | use 1:10,000 dillution |
| HiSeq 2500 or NextSeq 500 next generation sequencers | Illumina | inquire with supplier | |
| KAPA HiFi Hotstart DNA polymerase mix | KAPA/Roche | KK2602 | |
| Nuclease free water | Ambion | AM9937 | |
| Poly(A) polymerase, yeast | Thermo Fisher Scientific | 74225Z25KU | |
| Poly(A) polymerase, E.coli | New England Biolabs | M0276L | |
| Polynucleotide kinase | Thermo Fisher Scientific | EK0032 | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| RNA ligase 1, high concentration | New England Biolabs | M0437M | includes PEG-8000 |
| RNeasy MinElute RNA Cleanup kit | Qiagen | 74204 | |
| RNase H | New England Biolabs | M0279 | |
| RNasin Plus, ribonuclease inhibitor | Promega | N2618 | |
| Superscript IV reverse transcriptase | Thermo Fisher Scientiific | 18090050 | |
| Turbo DNase | Ambion | AM2238 | |
| USER enzyme mix | New England Biolabs | M5505 | |
| Dyna-Mag-2 magnetic rack | Thermo Fisher Scientific | 12321D | |
| Thermomixer C | Eppendorf | 5382000015 | Heated mixer with heated lid |
| MicroSpin columns | GE-Healthcare | 27-5325-01 | |
| Name | Company | Catalog Number | Comments |
| Buffers | |||
| Alkaline hydrolysis buffer, 1.5 x | Mix 1 part 0.1 M Na2CO3 and 9 parts 0.1 M NaHCO3. Add EDTA to 1 mM. Adjust pH to 9.2. Store aliquots at -20 °C. | ||
| 5x poly(A) polymerase buffer | Thermo Fisher Scientiific | 100 mM Tris-HCl, pH 7.0, 3 mM MnCl2, 0.1 mM EDTA, 1 mM DTT, 0.5 mg/ml acetylated BSA, 50% glycerol | |
| Biotin binding buffer | 20 mM TrisCl pH 7.5, 2 M NaCl, 0.1% NP40 | ||
| TEN buffer | 10 mM TrisCl, pH 7.5, 1 mM EDTA, 0.02% NP40 | ||
| Name | Company | Catalog Number | Sequence |
| Oligonucleotides according to Illumina TruSeq Small RNA Sample Prep Kits, for GA-IIx and Hiseq2000/2500 sequencers | Microsynth | ||
| revRA3 (RNA) | Microsynth | 5’ amino CCUUGGCACCCGAGAAUUCCA 3’ | |
| revDA5 | Microsynth | 5’ amino GTTCAGAGTTCTACAGTCCGAC GATCNNNN-3’ | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' (V = G, A or C) | |
| PCR primer forward, RP1 | Microsynth | 5' AATGATACGGCGACCACCGAGA TCTACACGTTCAGAGTTCTACAG TCCGA 3' | |
| PCR primer reverse, RPI1, barcode in bold | Microsynth | 5' CAAGCAGAAGACGGCATACGAG ATCGTGATGTGACTGGAGTTCCT TGGCACCCGAGAATTCCA 3' | |
| Name | Company | Catalog Number | Comments |
| Oligonucleotides according to Illumina TruSeq HT-Small RNA Sample Prep Kits, for HiSeq2000/2500 and NextSeq500 sequencers | |||
| HT-rev3A (DNA/RNA) | Microsynth | 5'-amino-GTGACTGGAGTTCAGACGTGTG CTCTTCCrGrAUrC-3' | |
| HT-rev5A | Microsynth | 5' amino-ACACTCTTTCCCTACACGACGCT CTTCCGATCTNNNN 3' | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' | |
| PCR primers forward (D501-506) | Microsynth or Illumina | 5'-AATGATACGGCGACCACCGAGAT CTACAC[i5]ACACTCTTTCCCTACA CGACGCTCTTCCGATCT -3' | |
| PCR primers reverse (D701-D712) | Microsynth or Illumina | 5'-CAAGCAGAAGACGGCATACGAG A[i7]GTGACTGGAGTTCAGACGTG TGCTCTTCCGATC-3' | |
| Documentation for Illumina multiplexing: | Illumina | https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/illumina-adapter-sequences_1000000002694-01.pdf |
参考文献
- Jan, C. H., Friedman, R. C., Ruby, J. G., Bartel, D. P. Formation, regulation and evolution of Caenorhabditis elegans 3'UTRs. Nature. 469 (7328), 97-101 (2011).
- Spies, N., Burge, C. B., Bartel, D. P. 3' UTR-isoform choice has limited influence on the stability and translational efficiency of most mRNAs in mouse fibroblasts. Genome Res. 23 (12), 2078-2090 (2013).
- Hoque, M., Ji, Z., et al. Analysis of alternative cleavage and polyadenylation by 3' region extraction and deep sequencing. Nat. methods. 10 (2), 133-139 (2013).
- Martin, G., Gruber, A. R., Keller, W., Zavolan, M. Genome-wide analysis of pre-mRNA 3’ end processing reveals a decisive role of human cleavage factor I in the regulation of 3' UTR length. Cell Rep. 1 (6), 753-763 (2012).
- Gruber, A. R., Martin, G., et al. Global 3' UTR shortening has a limited effect on protein abundance in proliferating T cells. Nat. Commun. 5, 5465 (2014).
- Kivioja, T., Vähärautio, A., et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat. methods. 9 (1), 72-74 (2011).
- Gruber, A. J., Schmidt, R., et al. A comprehensive analysis of 3' end sequencing data sets reveals novel polyadenylation signals and the repressive role of heterogeneous ribonucleoprotein C on cleavage and polyadenylation. Genome Res. 26 (8), 1145-1159 (2016).
- Lingner, J., Keller, W. 3'-end labeling of RNA with recombinant yeast poly(A) polymerase. Nucleic Acids Res. 21 (12), 2917-2920 (1993).
- Quail, M. A., Kozarewa, I., et al. A large genome center's improvements to the Illumina sequencing system. Nat. methods. 5 (12), 1005-1010 (2008).
- Rahmann, S. Snakemake--a scalable bioinformatics workflow engine. Bioinformatics. 28 (19), 2520-2522 (2012).
- . Anaconda Software Distribution Available from: https://continuum.io (2016)
- Dobin, A., Davis, C. A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Li, H., Handsaker, B., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Robinson, J. T., Thorvaldsdóttir, H., et al. Integrative genomics viewer. Nat. Biotechnol. 29 (1), 24-26 (2011).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ozsolak, F., Platt, A. R., et al. Direct RNA sequencing. Nature. 461 (7265), 814-818 (2009).
- Yao, C., Biesinger, J., et al. Transcriptome-wide analyses of CstF64-RNA interactions in global regulation of mRNA alternative polyadenylation. Proc. Natl. Acad. Sci. U. S. A. 109 (46), 18773-18778 (2012).
- Lin, Y., Li, Z., et al. An in-depth map of polyadenylation sites in cancer. Nucleic Acids Res. 40 (17), 8460-8471 (2012).
- Shepard, P. J., Choi, E. -. A., Lu, J., Flanagan, L. A., Hertel, K. J., Shi, Y. Complex and dynamic landscape of RNA polyadenylation revealed by PAS-Seq. RNA. 17 (4), 761-772 (2011).
- Fu, Y., Sun, Y., et al. Differential genome-wide profiling of tandem 3' UTRs among human breast cancer and normal cells by high-throughput sequencing. Genome Res. 21 (5), 741-747 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved