A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
קו 1 ניתוח מתילציה בתאי גזע מסנמצ'אל מטופלים עם אוסטאוסרקומה-מסחטות שלפוחיות
In This Article
Summary
המתואר כאן הוא השימוש בשיטה ספציפית הגברה בדיקה כדי לנתח את רמות המטלציה של האלמנטים קו 1 בתאי גזע mesenchymal שטופלו אוסטאוסרקומה נגזר שלפוחיות. בנוסף, הליך פופולרי עבור הפרדת שלפוחיות ושלפוחית של הפרה העובר סרום, הוא הפגינו גם.
Abstract
מתילציה-הגברה בדיקה ספציפית (MSPA) היא טכניקה פשוטה ואיתנה שניתן להשתמש בה כדי לזהות הבדלים יחסיים ברמות מתילציה של דגימות דנ א. זה רב תושייה, דורש כמויות קטנות של DNA, ולוקח סביב 4 – 5 h של הידיים על העבודה. בטכניקה המוצגת, דגימות ה-DNA הם הראשון מאוד לאחר מכן הוכלא בדיקות כי היעד DNA על מתילאו אתרים התייחסות כפקד. ה-DNA הוכלא מופרד לתגובות מקבילות, אחד עובר רק הארכה והשני עובר לאחר ואחריו העיכול HhaI-תיווך ברצפי GCGC unמתילated. שברי הדי. אנ. איי התוצאה הם מוגבר על ידי ה-PCR ומופרדים על ידי אלקטרופורזה קפילר. אתרים GCGC מתילated אינם מתעכלים על ידי HhaI ולייצר אותות שיא, בעוד האתרים GCGC מתעכל מתעכלים ולא אותות שיא נוצרות. השוואת הפסגות מנורמל השליטה של גירסאות מתעכל ובלתי מתעכלות של כל מדגם מספק את יחס המינון מתילציה של דגימת דנ א. כאן, MSPA משמש כדי לזהות את ההשפעות של אוסטאוסרקומה (EVs) על מעמד מתילציה של אלמנט גרעיני ארוך וביניהם-1 (קו -1) בתאי גזע mesenchymal. LINE-1s הם רכיבי DNA חוזרים שבדרך כלל עוברים hypomethylation בסרטן, בקיבולת זו, עשוי לשמש ביואריקר. משמש גם כשיטה חסכונית כדי להפריד ושלפוחיות מתוך נוזלים ביולוגיים (כלומר, כאשר הכנת סרום העוברי EV-מרוקנת [FBS] ובידוד EVs מתוך מדיה אוסטאוסרקומה ממוזג [צנטריפוגה]). עבור ניתוח מתילציה, בדיקות קו 1 מותאמות אישית מיועדות למקד שלושה אתרי מתילציה ברצף המקדם של קו 1 ובשבעה אתרי בקרה. פרוטוקול זה מדגים את השימוש ב-MSPA עבור ניתוח מתילציה של קו 1 ומתאר את הכנת הגושים המרוקנים ב-EV על ידי הסתבכות.
Introduction
מתילציה DNA היא שינוי. משמעותי בתאי האדם DNA מתילציה מתייחס לחיבור של קבוצות מתיל לשקעים ציטוסין ב-CpG dinucleotides. בדרך כלל מצויים באשכולות (איי ה-CpG) באזור 5 ' של גנים1. בתאים נורמליים, רוב הדיקלאוסים האלה קיימים במצב לא מתילנטי, המאפשר תמלול של דנ א. אגב, סרטן רבים קשורים באיים ה-CpG היפרמתיל והשתקה של2, בעיקר בגנים מדכא הגידול, אשר בתורו לתרום הסימנים השונים של סרטן3.
מצד שני, הרבה מרכיבים גרעיניים שונים-1 (קו-1 או L1s) הם חוזרים ונשנים, רכיבי DNA שבדרך כלל יש רמות גבוהות של מתילציה ב איי ה-CpG. מתילציה של קו -1 מונעת טרנסלוקציה ומסייעת לשמור על שלמות הגנום. במספר סוגים של סרטן, קו -1 הוא hypomethylated, וכתוצאה מכך הפעלה ושלאחר מכן ההפעלה העוקבת-מתווך אי-יציבות כרומוזומלית4. קו 1 חשבונות עבור כמעט 17% של הגנום האנושי5, ומצב מתילציה שלה יכול לשמש כאינדיקטור לרמה העולמית של מתיונין גנולציה6. לפני המעבר שלתאים. לפנוטיפלגידול לכן, הוא מחזיק הבטחה כסמן פוטנציאלי לתחילת הסרטן המוקדמים.
כיום, ישנן מספר שיטות לניתוח מתילציה, כולל פירולציה, PCR ספציפי, מיקרו-מערכים וכרומטין immunoprecipitation1. השימוש ברצף הדור הבא הפך גם את האפשרות לשלב גישות הגנום רחב לגילוי של מתילציה DNA. רבות מהשיטות הללו מסתמכות על דנ א המטופל בביסולפיט, שבו מומרות הציטוסינוסים בלתי מגנטיים לאורצילין ומתילטוסינוסים נותרים ללא שינוי. עם זאת, העבודה עם ה-DNA bisulfite תייחס שטופלו יש מלכודות מספר, כגון המרות שלא הושלמה של ציטוסינוסים unמתיליום כדי uracil, הגברה מוטה של רצפים, ו רצף שגיאות8.
ב מתילציה-הגברה בדיקה ספציפית (MSPA), בדיקות המורכב משני ליגונואותים רצפי DNA היעד המכיל אתר הגבלה (GCGC) עבור אנזים מתילציה רגיש הגבלה HhaI9. לאחר הבדיקות hybridize ל-DNA, כל מדגם מחולק לשתי קבוצות. רגשים בסדרה הראשונה עוברים הארכה, בעוד הבדיקות בסדרה השנייה עוברים הארכה ואחריו העיכול HhaI-תיווך באתרי CGCG unמתילated. שני סטים של דגימות מוגבר אז על ידי ה-PCR, והמוצרים מופרדים על ידי אלקטרופורזה קפיצית. הזונדים באתרים לא מתיללים מתעכלים על ידי hhaI ולא מוגבר במהלך ה-PCR, וכתוצאה מכך אין אותות שיא. לעומת זאת, הגששים באתרי מתיליום מוגנים מפני העיכול, ולכן מוגברת במהלך ה-PCR, ולאחר מכן מייצרים אותות שיא10.
MSPA יש מספר יתרונות על פני שיטות חלופיות. ראשית, זה דורש כמות נמוכה של DNA (50-100 ng) והוא מתאים היטב לניתוח של ה-DNA מ פורמאלין-קבוע פרפין מוטבע דגימות10. זה לא דורש DNA התייחס bisulfite פיט; למעשה, זה לא מתאים DNA כי הוא שונה בדרך זו. דגימות רבות ניתן לנתח באותו זמן, ו MSPA בדיקות יכול להיות מעוצב כך שהם מכוונים גנים מרובים או רצפים בו. בנוסף, הבדיקות הן ספציפיות ורגישות עבור ה-DNA מתילנטי כאתר ההגבלה hhaI מתאים לרצף האופייני לאיי ה-CpG10.
מחקר זה חקר את ההשפעות של אוסטאוסרקומה (מערכת ההפעלה)-שלפוחיות נגזרות (EVs) על קו-1 מתילציה ברקמת השומן הנגזר של הרקמה המטבית (AT-MSCs; איור 1). EVs הם ננו-סקאלה, ממברנה מאוגד שלפוחיות מופרש על ידי סוגי התאים ביותר. הם נושאים חלבונים, שומנים, mrna, מיקרווריה, ומולקולות נוספותמתאיההורה 11,12. EVs תווך תקשורת בין-תאית והפעל תפקידים חשובים במספר מצבים פתופסלוגיים13,14. מחקר שנערך לאחרונה הראה כי סרטן נגזר EVs עשוי להעביר הפעיל קו -1 לתאים הנמען15. זה דווח מוקדם יותר כי EVs מ-HOS-143B קו התא יכול לשנות את מצב מתילציה של קו 1 ב MSCs, בנוסף להשפעות גנטיות אחרות16.
כאשר התאים הגדלים עבור ev בידוד, חשוב להשתמש ev-מרוקן סרום שור העובר [fbs] במדיום הצמיחה, מאז fbs-נגזר EVs יכול להפריע EVs ממקורות אחרים ולפגוע בתוצאות17,18. ההפוגות היא אחת השיטות הנפוצות ביותר עבור מכלה EVs מ FBS. זהו הליך פשוט וחסכוני יחסית בהשוואה לחלופות כגון אולטראפילטרציהו-FBSמסחרי. כאן, הפרוטוקול גם מדגים כיצד להכין את FBS מרוקן-EV על-ידי הסתבכות.
מאמר זה מציג פרוטוקול מפורט עבור הטכניקות הנ ל, מבידוד של EVs מקו מערכת ההפעלה לניתוח מתילציה של קו 1 ב-OS-EV טיפל MSCs (איור 1).
Protocol
מחקר זה אושר על ידי ועדת האתיקה של הלסינקי ומחוז החולים אוסיסיאא (אישור מוסרי D. No. 217/13/03/02/2015).
1. הכנת הפבס מרוקנת על ידי הפוגות
- לקחת FBS (אולטרה) הצינורות צנטריפוגה ולמקם אותם דליים ultracentrifuge. כדי להבטיח שהפוגות מסוימות יפעלו בצורה חלקה ובטוחה, תאזן את הדליים בתוך 10 מ"ג אחד מהשני.
- טען את הדליים ברוטור מתנדנד (סוג SW28, k-factor 246). מניחים את הרוטור ultracentrifuge ולרוץ ב 100,000 x g עבור 19 h ב 4 ° c.
- אספו בזהירות את השכבה העליונה בצבע האור של סופרנאטנט (בערך 9 עשיריות) והעבירו לצינור 50 mL. אין להפריע או פיפטה את הגלולה חום כהה, כפי שהוא מכיל EVs מ FBS.
- להעביר את supernatant באמצעות מסנן 0.22 יקרומטר לתוך צינור חדש 50 mL.
- הוסף את ה-FBS המועקר של המסנן למדיה של תרבות התא בעת גידול תאים עבור בידוד EV.
2. בידוד של אוסטאוסרקומה-נגזר EVs
- לוח מערכת ההפעלה תאים (HOS-143B קו התא) בבקבוקון T-175 עם RPMI 1640 בינונית, שיושלם עם 10% FBS רגילה ו 1% אנטיביוטיקה (100 U/mL פניצילין, 0.1 mg/mL סטרפטומיצין). מניחים את הבקבוקון בחממה ב 37 ° c ו 5% CO2.
- כאשר הבקבוקון הוא 70%-80% שוטפת, לשטוף את התאים עם מלוחים באגירה פוספט (PBS) ולאחר מכן לגדל אותם במדיה המכילה 10% EV-מרוקן FBS (להלן המכונה התקשורת EV-לרוקן).
הערה: 1 x 106 HOS-143b תאים מצופה T175 בקבוקון מגיע 70% שליטה לאחר כ 60 h. - במהלך 48 h הבא, לאסוף מדיה ממוזג מתאי אוסטאוסרקומה אחרי כל 24 h ולהוסיף טרי EV מדיה דלה.
- צנטריפוגה את המדיה הממוזגת ב 2500 x g עבור 20 דקות ב 4 ° צ' כדי להסיר תאים ופסולת תא. העבר את הסופרנטאנט לצינור חדש, והשאיר כ-2 מ ל של מדיה בתחתית.
הערה: אם לא ישירות להמשיך עם בידוד EV בשלב זה, הסופרנטאנט ניתן לאחסן ב-80 oC. - יוצקים את supernatant לתוך ultracentrifuge צינורות ולאזן אותם כמו מוקדם יותר. צנטריפוגה את הצינורות ב 100,000 x g עבור 2 h ב 4 ° c.
- , בזהירות, ביטול הסופרנטאנט. משאיר כ-100 מ ל בתחתית הוסף סביב 20 מ ל של PBS (0.1 יקרומטר מסוננים) לצינור ופיפטה בעדינות כדי לשטוף ולהשעות מחדש את הגלולה EV.
- מאזן את הצינורות ולבצע סיבוב נוסף של הקשר עם הגדרות זהות.
- הסר בזהירות את הסופרנטנט והשהה מחדש את הגלולה EV ב 200 μL של PBS על ידי ליטוף עדין. אחסן את EVs בצינורות קשירה נמוכה.
הערה: ניתן להשתמש EVs מייד או אחר מאוחסן ב-80 ° c עד שהם נחוצים.
3. אפיון מערכת ההפעלה-EVs
הערה: EVs מטוהרים יכול להיות מאופיין על ידי בלוק מערבי (WB), ניתוח מעקב ננו-חלקיק (נ. ת. ע), ומיקרוסקופ אלקטרון שידור (TEM)16.
- לבצע WB לפי פרוטוקול סטנדרטי16 עם EV סמנים CD63, TSG101, ו Hsp70, ועם calnexin כפקד שלילי כדי לציין את הטוהר של לדוגמה EV20.
- עבור נ. ת. ע, הראשון לדלל את דגימת EV ב 0.1 יקרומטר מסוננים הPBS של dulbecco להשיג (באופן אידיאלי) 30-100 חלקיקים לכל מסגרת.
- לקחת סביב 500 μL של המדגם לתוך מזרק 1 mL ולטעון אותו לתוך הנמל מפרץ של המכשיר נ. ת. ע. בדקו שיש מספיק חלקיקים לכל מסגרת, למדידות מדויקות.
- פתח את התוכנה נ. ת. ע ולהקליט חמישה קטעי וידאו של 60 s משך, באמצעות מצלמה ברמה 13 בטמפרטורת הסביבה. במהלך הניתוח, השתמש בהסף לזיהוי = 5 והשג = 10.
- לנתח דגימות EV על ידי TEM כמתואר בעבר21.
4. בתרבות החברה
הערה: רקמת השומן של האדם לבידוד תא גזע mesenchymal סופק כמו שאיבת שומן (המחלקה לכירורגיה פלסטית, לייזר Tilkka בע מ, פינלנד). הסכמה מושכלת בכתב נלקחה מן התורמים ליפוף, אשר עברו בחירה שאיבת שומן.
- בידוד AT-MSCs מתוך שאיבת שומן באמצעות שיטות הבידוד מכני ואנזימטי סטנדרטיים22.
הערה: ניתן גם להשתמש בMSCs ממקורות אחרים (כולל קווי תא מסחרי). - תאי תרבות במדיה של DMEM דיה/F-12 בתוספת של 10% FBS ו 1% אנטיביוטיקה.
5. טיפול בMSCs עם מערכת ההפעלה-EVs
- צלחת 15,000 AT-MSCs לבאר 24 היטב צלחת.
- לאחר 24 שעות, להסיר את התקשורת הישנה, לשטוף תאים עם PBS, ולשנות את התקשורת EV-לרוקן.
- לטפל בתאים עם OS-EVs (בריכוז חלקיקים של 1 x 106 EVs לכל תא) ביום 1 (24 שעות לאחר הדבקה תא), יום 3 (48 h אחרי יום 1), ויום 5 (96 h אחרי יום 1).
- להפסיק את הטיפול OS-EV עבור נקודת זמן (TP) 0 דגימות ביום 1, TP 3 דגימות ביום 3 ו-TP 7 דגימות ביום 7 (48 h אחרי יום 5).
הערה: ניתן לעקוב אחר נקודות זמן נוספות לפי התוכניות הנסיוניות.
- להפסיק את הטיפול OS-EV עבור נקודת זמן (TP) 0 דגימות ביום 1, TP 3 דגימות ביום 3 ו-TP 7 דגימות ביום 7 (48 h אחרי יום 5).
- חלץ דנ א מ MSCs באמצעות שיטה המתאימה. כלול דגימות שליטה חיוביות ושליליות עבור ניתוח הנתונים.
6. שורה-1 מתילציה
- עיצוב שורה 1 מותאם אישית בדיקה התחל, כפי שנעשה בעבר על ידי Pavicic et al.23. לבדיקות מתילציה, בחר שלושה רצפים המכילים את אתר ההגבלה של HhaI בתוך אזור המקדם של LINE-1. עבור הבדיקות שליטה, בחר שבעה רצפים חסר ההגבלה HhaI מתוך שאר רצף קו 1.
הערה: רצף קו -1 זמין במסד הנתונים של GenBank24 (l 1.2, הצטרפות לא. AH 005269.2). השתמש בהוראות יצרן MSPA לעיצוב הגששים25. - לדלל 70 ng של דגימת DNA ב TE מאגר לנפח 5 μL.
- בצע את צעדי התרמותרמיים הבאים והPCR כפי שהוזכר בטבלה 1. מחממים את הדגימות ל -10 דקות ב-98 מעלות צלזיוס, ואז מצננים עד 25 ° c.
- הוסף 3 μL של תערובת הכלאה היברידיזציה לכל מדגם ולהפעיל את הציקלטרמר כדי לאפשר את הבדיקות כדי hybridize DNA.
- בטמפרטורת החדר (RT), להוסיף 13 μL של ערבוב שלאחר הכלאה לכל מדגם. העבר 10 μL לשפופרת שנייה.
- מניחים את שני סטים של צינורות בתוך הציקלייט ו הדגירה ב 48 ° c עבור לפחות 1 דקות.
- בעוד דגימות הן ב 48 ° c, להוסיף 10 μL של תערובת ליטל את הקבוצה הראשונה של צינורות (סדרה לא מתעכל) ו 10 μL של ערבוב שילוב העיכול לקבוצה השנייה של צינורות (סדרה מתעכל). תריץ את התוכנית. התרמוטרטרנר הבאה
- מסובבים את הצינורות ומשטים בו את הציקלטרטרזה עד 72 ° c.
- הוסף 5 μL של מערבבים פולימראז לכל צינור ומניחים את הצינורות של הציקלהטרטרנר. הפעל את תוכנית ה-PCR.
- בעוד תוכנית ה-PCR פועלת, הכן פתרון של מערכת מסוג 1 mL המכילה 2.5 μL של תקן גודל. פיפטה 10 μL של פתרון זה כל טוב של אופטי 96 צלחת הבאר (עם ברקוד).
- לאחר ה-PCR, לדלל את הדגימות מתעכל ומתעכל כדי 1:100 ו 1:200 בהתאמה במים באולטרטהורים. הוסף 2 μL של מוצר PCR מדולל ל 96 צלחת היטב. צנטריפוגה את הצלחת ב 200 x g עבור 15 – 20 s כדי להסיר בועות אוויר.
- לבצע ניתוח רסיס של דגימות על ידי אלקטרופורזה קפילר.
הערה: יש לאחסן את הלוח ב -4 ° c בחשכה עד לניתוח.
7. ניתוח נתוני קטע
- פתח את האלקטרופורזה נימים תוצאות בתוכנת ניתוח electropherogram.
- תחת עמודת החלונית , עבור אחת הדגימות, בחר mlpa מהתפריט. לחץ על כותרת החלונית והקש Ctrl + D כדי להחיל mlpa על כל הדגימות.
- באותו אופן, הגדר את שיטת הניתוח לברירת מחדל למיקרו-לווין עבור כל הדגימות.
- בחר את כל הדגימות ולחץ על לחצן הפעל ירוק כדי לנתח את הדגימות לפי ההגדרות שנבחרו.
- בחר את כל הדגימות ולחץ על כפתור הגרף כדי להמחיש את פסגות בדיקה.
- התקרבות באזור השיא לרזולוציה גבוהה יותר של פסגות הגישוש הבודדות. ודא כי כל 10 הפסגות המתאימות הגששים מן הבדיקה LINE-1 לערבב מתויגים. השמט פסגות נוספות (< 95 bp ו > 160 bp).
- בכרטיסיה גנוסוגים , יצא את התוצאות בתבנית ערכים מופרדים באמצעות פסיקים (CSV).
- פתח את קובץ ה-CSV בתוכנת ניתוח נתונים ומיין את הנתונים לעמודות.
- סמן את שלושת הפסגות באתר מתילציה בהתבסס על הגדלים המשוער שלהם (L1-1m ב 153 bp, L1-2m ב 119 bp, L1-3m ב 133 bp). שבעת הפסגות הנותרות מתאימות לבדיקות הבקרה.
הערה: כאן, הערכים של פסגות הבדיקה L1-2m משמשים, אשר בגודל של 117 bp, מאז האזור הזה נעשה שימוש ברוב שורה 1 מתירחון בחני23. - לכל מדגם (לא מתעכל ומתעכל), חשב את אזור השיא של כל שבעת פסגות הבקרה. חלק את אזור השיא של כל אחד מבין שורה-1 בסכום זה.
- עבור כל דגימת DNA, לחלק את הערך של המדגם מתעכל על ידי זה של המדגם לא מתעכל כדי להשיג את המינון מתילציה יחס (DM) באמצעות המשוואה הבאה:
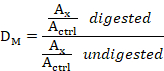
היכן: DM הוא יחס המינון של מתילציה,x הוא האזור שמתחת לשיא x (לדוגמה, L1-2m שיא), ו-ctrl הוא אזור השיא של כל שבעת הבדיקות בקרה.
- סמן את שלושת הפסגות באתר מתילציה בהתבסס על הגדלים המשוער שלהם (L1-1m ב 153 bp, L1-2m ב 119 bp, L1-3m ב 133 bp). שבעת הפסגות הנותרות מתאימות לבדיקות הבקרה.
תוצאות
המטרה העיקרית של מחקר זה היה להעריך את ההשפעות epigenetic של OS-EVs ב MSCs. OS-EVs היו מבודדים מתאי-143B באמצעות השיטה הדיפרנציאליות סטנדרטית. הביטוי של האופייני EV סמנים CD63, Hsp70, ו TSG101 על ידי בלוק המערבי אישר את הנוכחות של OS-EVs. (איור 2א). העדר אות calnexin הצביע על טוהר של מערכת ההפעלה-EV ב...
Discussion
מחקר זה ממחיש כיצד MSPA ניתן להשתמש כדי לזהות ולכמת את סטטוס מתילציה של אלמנט גנטי ספציפי. קו -1 היה המוקד כאן, אבל הגששים יכולים להיות מיועדים למקד את מגוון של גנים ורצפים. יתר על כן, יש רשימה גוברת של מתערבב בדיקה זמין עבור יישומים שונים. Mspa היא טכניקה פשוטה ואיתנה עבור ניתוח מתיונין DNA שאינו ד...
Disclosures
. למחברים אין מה לגלות
Acknowledgements
עבודה זו ממומנת על ידי אוניברסיטת הלסינקי מימון הפרויקט (WBS490302, WBS73714112) הלסינקי אוניברסיטת החולים המדינה מימון לחקר הבריאות ברמת האוניברסיטה (Y1014SUL05, TYH2016130), הקרן לרפואה פינית-נורווגית, ואת סלמה ו קרן מינרווה-ליסה סלאנדר (קרן מינרבה). אנו מודים לוולטר Pavicic על מתן פרוטוקול MSPA שהשתנה ולתמיכה טכנית קשורה. אנחנו אסירי תודה לטאמו Masalin (אוניברסיטת הלסינקי) על שסייע לנו עם הפקת וידאו.
Materials
| Name | Company | Catalog Number | Comments |
| 1 mL syringe | Terumo | SS+01T1 | for NTA |
| 24-well plate | Corning | 3524 | MSC cell culture |
| 3730xl DNA Analyzer | Applied Biosystems, ThermoFisher Scientific | 3730XL | |
| 50 mL centrifuge tube | Corning | 430829 | |
| Beckman Optima LE-80K Ultracentrifuge | Beckman | ||
| BlueStar Prestained Protein Marker | Nippon Genetics | MWP03 | WB: protein marker |
| Calnexin (clone C5C9) | Cell Signaling Technology | 2679 | WB, dilution 1:800 |
| CD63 (clone H5C6) | BD Biosciences | 556019 | WB, dilution 1:1000 |
| Centrifuge 5702 R | Eppendorf | 5703000010 | For conditioned media and cells |
| Centrifuge 5810 | Eppendorf | 5810000010 | For spinning down 96-well plate |
| Centrifuge tube (polyallomer, 14x95 mm) | Beckman | 331374 | Ultracentrifugation |
| DMEM/F-12 + GlutaMAX medium | Gibco, Life Technologies | 31331-028 | For AT-MSC culture |
| Fetal bovine serum | Gibco, Life Technologies | 10270-106 | |
| GeneScan 500 LIZ size standard | Applied Biosystems, Life Technologies | 4322682 | for capillary electrophoresis |
| GenomePlex Complete Whole Genome Amplification (WGA) Kit | Sigma | WGA2-10RXN | for MSPA negative control |
| Hi-Di formamide | Applied Biosystems, Life Technologies | 4311320 | for capillary electrophoresis |
| HOS-143B cell line | ATCC | CRL-8303 | |
| Hsp70 (clone 5G10) | BD Biosciences | 554243 | WB, dilution 1:1000 |
| IRDye 800CW Goat anti-mouse | Li-Cor | 926-32210 | WB: secondary |
| IRDye 800CW Goat anti-rabbit | Li-Cor | 926-32211 | WB: secondary |
| LINE-1 probe-mix primers | IDT | Sequences in Table 1 | |
| MicroAmp Optical 96-well reaction plate with barcode | Applied Biosystems, Life Technologies | 4306737 | also requires sealing film |
| Micro BCA Protein Assay kit | ThermoFisher Scientific | 23235 | measure protein concentration |
| MiniProtean TGX 10% gels | Bio-Rad | 456-1034 | WB: gel electrophoresis |
| NanoSight LM14C | Malvern Instruments | for NTA | |
| Nitrocellulose membrane 0.2 µm | Bio-Rad | 1620112 | WB: protein transfer |
| NucleoSpin Tissue XS | Macherey-Nagel | 740901.50 | for DNA extraction |
| Odyssey Blocking Buffer | Li-Cor | 927-40000 | WB: blocking, antibodies |
| PBS, 1X | Corning | 21-040-CVR | |
| Penicillin-streptomycin | Gibco, Life Technologies | DE17-602E | Antibiotics for culture media |
| Protein LoBind tube, 0.5 mL | Eppendorf | 22431064 | For storing Evs |
| REVERT Total Protein Stain and Wash Solution Kit | Li-Cor | 926-11015 | WB: total protein staining |
| RKO cell line | ATCC | CRL-2577 | for MSPA positive control |
| RPMI medium 1640 + GlutaMAX | Gibco, Life Technologies | 61870-010 | For HOS-143B cell culture |
| SALSA MLPA HhaI enzyme | MRC-Holland | SMR50 | |
| SALSA MLPA reagent kit | MRC-Holland | EK1-FAM | |
| SALSA MLPA P300 probe-mix | MRC-Holland | P300-100R | |
| Swinging rotor SW-28 | Beckman Coulter | 342207 | Ultracentrifugation |
| Syringe filter, 0.22 µm | Jet Biofil | FPE-204-030 | sterile filtering FBS |
| Tecnai 12 | FEI Company | equipped with Gatan Orius SC 1000B CCD-camera (Gatan Inc., USA); for TEM | |
| TBS, 1X tablets | Medicago | 09-7500-100 | WB: buffer |
| Trans-Blot Turbo | Bio-Rad | WB: transfer | |
| Thermal cycler | ThermoFisher Scientific | TCA0096 | |
| TrypLE Express | Gibco | 12604-021 | for trypsinization of cells |
| TSG101 (clone 4A10) | Sigma | SAB2702167 | WB, dilution 1:500 |
References
- Esteller, M. Cancer epigenomics: DNA methylomes and histone-modification maps. Nature Reviews Genetics. 8, 286-298 (2007).
- Herman, J. G., Baylin, S. B. Gene silencing in cancer in association with promoter hypermethylation. New England Journal of Medicine. 349, 2042-2054 (2003).
- Hanahan, D., Weinberg, R. A. Hallmarks of Cancer: The Next Generation. Cell. 144 (5), 646-674 (2011).
- Howard, G., Eiges, R., Gaudet, F., Jaenisch, R., Eden, A. Activation and transposition of endogenous retroviral elements in hypomethylation induced tumors in mice. Oncogene. 27, 404-408 (2008).
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409, 860-921 (2001).
- Rodriguez, J., et al. Chromosomal instability correlates with genome-wide DNA demethylation in human primary colorectal cancers. Cancer Research. 66, 8462 (2006).
- Feinberg, A. P., Ohlsson, R., Henikoff, S. The epigenetic progenitor origin of human cancer. Nature Reviews Genetics. 7, 21-33 (2006).
- Bock, C., et al. BiQ Analyzer: visualization and quality control for DNA methylation data from bisulfite sequencing. Bioinformatics. 21 (11), 4067-4068 (2005).
- Schouten, J. P., et al. Relative quantification of 40 nucleic acid sequences by multiplex ligation-dependent probe amplification. Nucleic Acids Research. 30, 57 (2013).
- Nygren, A. O. H., et al. Methylation-Specific MLPA (MS-MLPA): simultaneous detection of CpG methylation and copy number changes of up to 40 sequences. Nucleic Acids Research. 33 (14), 128 (2005).
- El Andaloussi, S., Mager, I., Breakefield, X. O., Wood, M. J. Extracellular vesicles: biology and emerging therapeutic opportunities. Nature Reviews Drug Discovery. 12, 347-357 (2013).
- Vallabhaneni, K. C., et al. Extracellular vesicles from bone marrow mesenchymal stem/stromal cells transport tumor regulatory microRNA, proteins, and metabolites. Oncotarget. 6 (7), 4953-4967 (2015).
- Ratajczak, J., Wysoczynski, M., Hayek, F., Janowska-Wieczorek, A., Ratajczak, M. Z. Membrane- derived microvesicles: important and underappreciated mediators of cell-to-cell communication. Leukemia. 20, 1487-1495 (2006).
- Lee, T. H., et al. Microvesicles as mediators of intercellular communication in cancer- the emerging science of cellular "debris". Seminars in Immunopathology. 33, 455-467 (2011).
- Kawamura, Y., Calle, A. S., Yamamoto, Y., Sato, T., Ochiya, T. Extracellular vesicles mediate the horizontal transfer of an active LINE-1 retrotransposon. Journal of Extracellular Vesicles. 8, 1643214 (2019).
- Mannerström, B., et al. Epigenetic alterations in mesenchymal stem cells by osteosarcoma-derived extracellular vesicles. Epigenetics. 14 (4), 352-364 (2019).
- Shelke, G. V., Lässer, C., Gho, Y. S., Lötvall, J. Importance of exosome depletion protocols to eliminate functional and RNA-containing extracellular vesicles from fetal bovine serum. Journal of Extracellular Vesicles. 3, 24783 (2014).
- Wei, Z., Batagov, A. O., Carter, D. R., Krichevsky, A. M. Fetal bovine serum RNA interferes with the cell culture derived extracellular RNA. Scientific Reports. 6, 31175 (2016).
- Kornilov, R., et al. Efficient ultrafiltration-based protocol to deplete extracellular vesicles from fetal bovine serum. Journal of Extracellular Vesicles. 7, 1422674 (2018).
- Théry, C., Amigorena, S., Raposo, G., Clayton, A. Isolation and characterization of exosomes from cell culture supernatants and biological fluids. Current Protocols in Cell Biology. 30 (1), 1-29 (2006).
- Puhka, M., et al. Metabolomic profiling of extracellular vesicles and alternative normalization methods reveal enriched metabolites and strategies to study prostate cancer-related changes. Theranostics. 7 (16), 3824 (2017).
- Peltoniemi, H. H., et al. Stem cell enrichment does not warrant a higher graft survival in lipofilling of the breast: A prospective comparative study. Journal of Plastic, Reconstructive & Aesthetic Surgery. 66 (11), 1494-1503 (2013).
- Pavicic, W., Joensuu, E. I., Nieminen, T., Peltomäki, P. LINE-1 hypomethylation in familial and sporadic cancer. Journal of Molecular Medicine. 90, 827-835 (2012).
- . L1.2 sequence on GenBank (accession number: AH005269.2) Available from: https://www.ncbi.nlm.nih.gov/nuccore/AH005269 (2000)
- Designing synthetic MLPA probes (v04). MRC-Holland Available from: https://support.mlpa.com/downloads/files/designing-synthetic-mlpa-probes (2018)
- . Takara Bio Inc Available from: https://www.takarabio.com/us/products/cell_biology_and_epigenetics/epigenetics/dna_preparation/msre_overview (2018)
- Pisanic, R., et al. Long interspersed nuclear element 1 retrotransposons become deregulated during the development of ovarian cancer precursor lesions. The American Journal of Pathology. 189 (3), 513-520 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved