Method Article
ניטור דינמיקת אינטראקציה חלבון-רנ"א ב-Vivo ברזולוציה טמפורלית גבוהה באמצעות χCRAC
In This Article
Erratum Notice
Summary
הצלבה קינטית וניתוח של cDNA היא שיטה המאפשרת לחקור את הדינמיקה של אינטראקציות חלבון-RNA בתאים חיים ברזולוציה טמפורלית גבוהה. כאן מתואר בפירוט הפרוטוקול, כולל גידול תאי שמרים, הצלבת UV, קציר, טיהור חלבונים ושלבי הכנת ספריית הריצוף של הדור הבא.
Abstract
האינטראקציה בין חלבונים קושרי RNA (RBPs) לבין מצעי הרנ"א שלהם מפגינה נזילות ומורכבות. במהלך חייו, רנ"א יחיד יכול להיות קשור על ידי RBPs רבים ושונים אשר יווסתו את ייצורו, יציבותו, פעילותו והתפרקותו. לכן, נעשה הרבה כדי להבין את הדינמיקה הקיימת בין שני סוגי מולקולות אלה. פריצת דרך חשובה במיוחד הגיעה עם הופעתו של 'c ross-linking and immunoprecipitation' (CLIP). טכניקה זו אפשרה חקירה קפדנית של אילו רנ"א קשורים ל-RBP מסוים. בקיצור, החלבון המעניין מקושר UV למצעי הרנ"א שלו in vivo, מטוהרים בתנאים מחמירים ביותר, ואז הרנ"א המוצלבים באופן קוולנטי לחלבון מומרים לספריות cDNA ומרוצפים. מאז תפיסתו, טכניקות נגזרות רבות פותחו על מנת להפוך את CLIP מקובל לתחומי לימוד מסוימים. עם זאת, קישור צולב באמצעות אור אולטרה סגול ידוע לשמצה לא יעיל. התוצאה היא זמני חשיפה ארוכים יותר שהופכים את המחקר הזמני של אינטראקציות RBP-RNA לבלתי אפשרי. כדי להתגבר על בעיה זו, תכננו ובנינו לאחרונה מכשירי הקרנת UV וקצירת תאים משופרים בהרבה. באמצעות כלים חדשים אלה, פיתחנו פרוטוקול לניתוח בזמן של אינטראקציות RBP-RNA בתאים חיים ברזולוציה טמפורלית גבוהה: Kinetic CRoss-linking ו-Analysis of cDNAs (χCRAC). לאחרונה השתמשנו בטכניקה זו כדי לחקור את התפקיד של שמרים RBPs בהסתגלות לעקה תזונתית. כתב יד זה מספק סקירה מפורטת של שיטת χCRAC ומציג תוצאות אחרונות שהתקבלו עם Nrd1 RBP.
Introduction
רנ"א מסתמכים לעתים קרובות על RBPs כדי להפעיל את תפקידם, מה שהוביל להתעניינות רבה בהבנת הדינמיקה בין מולקולות אלה. RBPs רבים זוהו במגוון רחב של אורגניזמים. עם זאת, זה תמיד היה ידוע לשמצה קשה לחקור אינטראקציות RBP-RNA in vivo. פריצת דרך משמעותית בחקר אינטראקציות כאלה הגיעה עם הופעתו של CLIP1. שיטה זו משתמשת בקרינה אולטרה סגולה (UV, 254 ננומטר) כדי לגרום לקשרים קוולנטיים בין RBPs לבין RNA הקשורים ישירות שלהם (כלומר, קישור צולב למרחק אפס). לאחר מכן, RBP של עניין הוא immunopurified בתנאים מחמירים כדי להבטיח כי רק RNAs crosslinked קוולנטית חלבונים מזוהים. רנ"א קשור מתעכל חלקית עם RNases ולאחר מכן מומר לספריות cDNA לצורך ריצוף. חומרת הטיהור הגבוהה חשובה מכיוון שהיא מגדילה מאוד את הספציפיות של התאוששות חלבון ו- RNA, אשר משופרת עוד יותר באמצעות טיהור SDS-PAGE של קומפלקס ריבונוקלאו פרוטאין צולב (RNP). CLIP ושיטות קשורות מספקות גם תובנה ברזולוציית הנוקלאוטידים לאתר קשירת החלבון, מכיוון שבמהלך הכנת ספריית הריצוף, חומצות אמינו המקושרות לרנ"א לעיתים קרובות מסיימות את השעתוק ההפוך או גורמות לאנזים להציג מוטציות באתר זה 1,2,3.
מאז הצגתו, פרוטוקול CLIP המקורי הפיק מגוון מדהים של מתודולוגיות נגזרות. פריצת דרך חשובה במיוחד הגיעה עם פיתוח HITS-CLIP (או CLIP-seq), הממזג רצף בתפוקה גבוהה עם גישת CLIP3. זה אומץ מאז על ידי כל המתודולוגיות מבוססות CLIP. iCLIP הציגה שיפורים בטכניקות חיתוך וקשירת מתאמים בתיווך RNase המאפשרים מיפוי מדויק יותר של אתרי הקישור של RBP4. PAR-CLIP שילב תיוג 4thio-uridine/uracil עם cross-linking ב-365 ננומטר, מה שמאפשר למפות אתרי קישור צולב על ידי ניתוח תחליפי T-C5. CRAC, urea-iCLIP, dCLIP ו-uvCLAP הציגו תנאי דנטורינג ושלבי טיהור זיקה כפולה המפחיתים עוד יותר את קשירת הרקע לשרף הזיקה ומגדילים עוד יותר את הספציפיות של לכידת חלבונים 2,6,7,8,9. בנוסף, CRAC, uvCLAP ו-dCLIP הציגו תיוג RBP של עניין עם תג זיקה, ובכך התגברו על הצורך לייצר נוגדנים ספציפיים.
מספר אופטימיזציות נעשו גם כדי לזרז את מתודולוגיית CLIP. פרוטוקול CLIP המקורי השתמש בתיוג רדיו של הרנ"א שנלכד על מנת להמחיש את קומפלקסי RBP-RNA לאחר SDS-PAGE. עם זאת, השימוש ברדיואקטיביות יכול להיות בעייתי עבור מעבדות שאינן ערוכות לעבודה כזו. irCLIP משלב מתאם מצומד פלואורופור המאפשר הדמיה באמצעות דימות אינפרא אדום10 ו-sCLIP משתמש בביוטינילציה של רנ"א שנלכד על מנת להמחיש אותם באמצעות HRP11 מצומד סטרפטווידין. יתר על כן, eCLIP מוותר לחלוטין על תיוג RNA; במקום זאת, החלבון נכרת אך ורק על סמך גודלו הידוע12. טיהור מבוסס סטרפטווידין שימש גם להאצת תהליך הכנת הספרייה ב- FAST-iCLIP, שם מתאם 3' ביוטינילציה קשור לרנ"א ומשמש כדי לאפשר טיהור לאחר שעתוק לאחור ומעגליזציה13. שיפורים נוספים בפרוטוקול iCLIP גם הגדילו מאוד את המורכבות של הספריות4.
לבסוף, CLIP שונה כדי לאפשר לכידה של RBPs מתת-תאים שונים 14,15, כדי להמחיש RNA משועתק חדש באמצעות השראה פועמת של ריבונוקלאוזידים פוטואקטיביים 5,16,17, כדי ללכוד RNA מתילציה18,19,20, כדי לבחון אינטראקציות RNA-RNA 21,22, ולמפות 3' קצוות 23,24.
למרות התרומות הגדולות של טכניקות מבוססות קליפ בסיוע להבנתנו את האינטראקציות בין RBPs ורנ"א, היא הוגבלה על ידי חוסר היעילות של קישור צולב UV. למרות שתאי תרבית הגדלים בשכבה חד-שכבתית הם בדרך כלל קלים יחסית להצלבה, זה מאתגר יותר באופן משמעותי ברקמות או בתאים בתמיסה. רקמות יכולות לדרוש סבבים מרובים של חשיפה לקרינת UV על מנת לחדור לשכבות התאים הנדרשות, בעוד שתאים מיקרוביאליים גדלים לעתים קרובות במדיומים עשירים המכילים תרכובות ארומטיות סופגות UV25. ואכן, זמני הקרנת UV של עד 30 דקות שימשו ליצירת קישור צולב מספיק בין RBPs לבין RNA קשור שלהם עבור דגימות כאלה26,27,28. חשיפה ממושכת זו לקרינת UV גורמת לתגובות לחץ בתוך התא, כגון נזק לדנ"א המושרה על ידי UV, אשר יכול לזהם את הנתונים הסופיים ביישומים מסוימים.
רוב מחקרי CLIP התמקדו ביצירת "תצלומי בזק" בודדים של אינטראקציות חלבון-רנ"א ספציפיות בתא. עם זאת, אינטראקציות חלבון-רנ"א הן דינמיות מטבען, במיוחד כאשר תאים נתונים לשינויים בסביבתם. זה יכול לכלול ירידה פתאומית בזמינות של חומרים מזינים חיוניים או שינויים מהירים בטמפרטורה. לכן, כדי להבין באמת את תפקידו של RBP במהלך מתח, עדיף לבצע ניתוחים שנפתרו בזמן מכיוון שהם יכולים ללכוד את הספקטרום המלא של מטרות RBP במהלך הלחץ ולקבוע באיזה שלב של תגובת הלחץ RBP שנבחר פעיל. בפרט, מחקרים בשמרים הראו כי הדקות הראשונות של הסתגלות הן קריטיות לחלוטין להישרדות וזמן מחצית החיים של RNA בחיידקים יכול לנוע בין דקות לשניות 29,30,31,32,33. לכן, ניתוחים כאלה שנפתרו בזמן צריכים להתבצע באופן אידיאלי ברזולוציה זמנית גבוהה. עם זאת, זמני ההצלבה הארוכים הופכים את המחקר של תגובות אדפטיביות בשלב מוקדם למאתגר במיוחד.
כדי להתגבר על בעיות אלה, פיתחנו לאחרונה שיטה משופרת המסוגלת להצליב ולקצור תאים בטווחי זמן של דקה. שיטת χCRAC שלנו מאפשרת מדידה כמותית של שינויים דינמיים באינטראקציות RBP-RNA ברזולוציה שלא נראתה בעבר. קריטי לשיטה זו היה פיתוח של מכשיר קרינת UVחדשני 32 אשר מפחית את זמן הקישור הצולב, הנדרש בשמרים ובחיידקים בתמיסה בערך פי 10, ולמעשה מקפיא אינטראקציות RBP-RNA באופן מיידי. בנוסף, על מנת לקצור במהירות את התאים לאחר קרינת UV, פיתחנו מכשיר סינון ואקום שיכול לקצור שמרים הגדלים באופן אקספוננציאלי בתרבית של 0.5 ליטר בסביבות 30 s32. חידושים טכנולוגיים אלה מאפשרים ללמוד את הדינמיקה של RBP-RNA ברזולוציה דקה. בנוסף, הצגנו מספר אופטימיזציות לפרוטוקול CRACהמקורי 2 על מנת להגביר את המעשיות שלו.
באמצעות χCRAC, חקרנו לאחרונה את המטרה של RBP גרעיני שמרים, Nab3, בתגובה למחסור בגלוקוז. ב Saccharomyces cerevisiae, Nab3 יכול ליצור קומפלקס עם Nrd1, RBP, ואת RNA helicase Sen1 כדי ליצור קומפלקס NNS. NNS נקשר ל-RNA פולימראז ולתעתיק המתהווה יכול לגרום לסיום שעתוק34. קומפלקס זה מעורב בעיקר בהסרת תעתיקי RNA מוצפנים שאינם מקודדים, אך הוכח גם כמווסת ביטוי של גנים מקודדי חלבונים. המחקר הראה מיקוד דיפרנציאלי של Nab3 לתמלילים ללא קידוד וקידוד לאחר דקה בלבד של לחץ32. הראינו כי סיום שעתוק משותף על ידי Nab3 גורם לביטוי חולף מאוד, דמוי דופק, של גנים רטרוטרנספוזון, אשר היה קשה לזהות באמצעות גישות מסורתיות מבוססות CLIP. בנוסף, זמני הקרנת UV הקצרים בקרוס-לינקר UV שלנו גם הגדילו באופן משמעותי את ההתאוששות של RNA קצר מועד שאינו מקודד32. χCRAC יהיה ככל הנראה כלי מכריע בהבהרת לא רק האופן שבו RBPs מעצבים את התגובה ללחץ בטווחי זמן מיידיים, אלא גם את תפקידיהם המשתנים במהלך כל מחזור החיים של תגובה. כתב יד זה מספק סקירה מפורטת של כל השלבים בפרוטוקול χCRAC. לשם המחשה, השיטה שימשה לחקר חלבון השמרים Nrd1, המעורב בסיומת שעתוק ודעיכת RNA35,36, ואת מטרת ה-RNA שלו בתגובה למחסור בגלוקוז על פני מספר רב של נקודות זמן. לבסוף, אנו גם מראים כי יחידת הקרנת UV שלנו יכולה להצליב במהירות RBPs ל- RNA בתאי HeLa, מה שמאפשר גם לבצע ניתוחים ברזולוציה גבוהה בזמן בתאים דבקים.
Protocol
| TN150 |
| 50 מ"מ טריס pH 7.8 |
| 150 mM NaCl |
| 0.1% NP-40 |
| 1X מעכב פרוטאז |
| TN1000 |
| 50 מ"מ טריס pH 7.8 |
| 1M NaCl |
| 0.1% NP-40 |
| NP-PNK |
| 50 מ"מ Tris-HCl pH 7.8 |
| 10 מ"מ מ"ג Cl2 |
| 0.1% NP-40 |
| 5 מ"מ בטא-מרקפטואתנול |
| 5 x PNK |
| 250 מ"מ Tris-HCl pH 7.8 |
| 50 מ"מ MgCl2 |
| 50 מ"מ בטא-מרקפטואתנול |
| WB I |
| 50 מ"מ Tris-HCl pH 7.8 |
| 300 מ"מ NaCl |
| 10 מ"מ אימידזול |
| 6M guanidine-HCl |
| 0.1% NP-40 |
| 5 מ"מ בטא-מרקפטואתנול |
| WB II |
| 50 מ"מ Tris-HCl pH 7.8 |
| 50 מ"מ NaCl |
| 10 מ"מ אימידזול |
| 0.1% NP-40 |
| 5 מ"מ בטא-מרקפטואתנול |
| חיץ אלוציה |
| 50 מ"מ טריס pH 7.8 |
| 50 מ"מ NaCl |
| 250 מ"מ אימידזול |
| 0.1% NP-40 |
| 5 מ"מ בטא-מרקפטואתנול |
| חיץ פרוטאז K |
| 50 מ"מ טריס |
| 0.1% NP-40 |
| 5 מ"מ β-מרקפטואתנול |
| 1% SDS |
| 5 מ"מ EDTA |
| 50 מ"מ NaCl2 |
| חיץ ליזה של יונקים |
| 50 מ"מ Tris-HCl pH 8 |
| 100 mM NaCl |
| 0.5% לעומת טריטון X-100 |
| 0.25% עם Na-deoxycholate |
| 0.1% עם SDS |
| 5 מ"מ EDTA |
| 1 mM DTT (נוסף טרי) |
| 1X מעכב פרוטאז |
טבלה 1: המאגרים הדרושים עבור χCRAC והרכביהם.
1. קרינת UV צולבת וייצור ליזט
- מיקרואורגניזמים בתמיסה
- יש לחסן 3.5 ליטר של המדיום הרצוי בשמרים מתרבית לילה ל-OD600 התחלתי של 0.05. גדל ב-30°C עם רעידות מתמשכות ב-180 סל"ד.
- במהלך הצמיחה, להכין חומרים הדרושים אחרים.
- הכינו מיכל של חנקן נוזלי.
- הכינו 3 ליטר של מדיום מעורר מתח וחם עד 30 מעלות צלזיוס באמבט מים.
- הגדירו את מנגנון המסנן, הפעילו את הקרוס-לינקר (איור 2A) ותייגו צינורות חרוטיים בנפח 50 מ"ל, אחד לכל נקודת זמן.
- ברגע שהתאים מגיעים ל- OD 600 הרצוי, שפכו500 מ"ל של תאים היישר לתוך הקרוס-לינקר והקרינו UV עם 250 mJ של 254 ננומטר UV. ראו איור 2A ואיור 3A לפרטים על השימוש בקרוסלינקר.
הערה: אנרגיית קרינת UV חייבת להיות מותאמת בקפידה עבור כל חלבון מעניין. ראה דיון לפרטים נוספים. - לאחר קישור צולב, סננו את התאים באמצעות אחד ממכשירי סינון הוואקום (איור 2B,C). לגלגל את הממברנה עם התאים המסוננים, מניחים בצינור החרוטי t = 0 (זמן אפס) 50 מ"ל, ולהקפיא הבזק בחנקן נוזלי.
- סנן את התאים הנותרים בשישה מסננים שונים. להשהות מחדש את התאים שנאספו ב 3 L של מדיום גורם מתח שחומם בעבר על ידי הטלת הממברנות בתווך וערבוב נמרץ עם סטריפט במשך 50 שניות. לאחר 50 שניות, להתכונן לקחת את t = 1 דגימה.
- לאחר דקה אחת, הצליבו 500 מ"ל תאים וקצרו באמצעות סינון כמו בשלבים 1.1.3–1.1.4. חזור על הפעולה לאחר 2, 4, 8, 14 ו- 20 דקות, או נקודות זמן שונות לפי הצורך.
- אחסן את הצינורות החרוטיים המכילים את התאים ב -80 ° C. הציבו מי מלח חוצצים פוספט (PBS) על 4°C (75°F) למשך הלילה.
- למחרת, קח כל צינור חרוטי המכיל דגימה מוצלבת והשהה מחדש את התאים ב 25 מ"ל של PBS קר על ידי רעד נמרץ.
- מעבירים את מתלי התא לצינורות חרוטיים חדשים ומסתובבים ב-4,600 x גרם, 5 דקות ב-4°C.
- יוצקים את PBS, מסתובבים במהירות שוב כדי לאסוף PBS שאריות ולאחר מכן לרוקן את הנוזל שנותר עם פיפטה.
- חשב את משקל הגלולה בצינור על ידי השוואתו לצינור ריק.
- הוסף שני נפחי גלולה של TN150 קר כקרח, 60 μL של DNase 1, ו 10 μL של מעכב RNase. דוגרים על קרח במשך 30 דקות.
- לדוגמה, עבור 400 מ"ג של תאים, להוסיף 800 μL של TN150 קר כקרח.
- הוספת DNase אינה חיונית עבור רוב החלבונים המסיסים, אך היא חשובה מאוד כאשר חוקרים חלבונים הקשורים לכרומטין, כגון RNA פולימראז. בנוסף, הוא מפחית את צמיגות הליזאטים החיידקיים. חשוב מאוד להשתמש בדיוק בשני נפחי גלולה של חיץ הליזיס, אחרת יעילות הליזה יכולה לרדת.
- הוסף שלושה נפחי גלולה (במ"ל) של חרוזי זירקוניה לתרחיף התא. עבור שמרים, להשתמש חרוזים בקוטר 0.5 מ"מ עבור חיידקים להשתמש 0.1 מ"מ.
- לדוגמה, עבור 400 מ"ג של תאים, למדוד 1.2 מ"ל של חרוזי זירקוניה בצינור 1.5 מ"ל ולהוסיף אותם לתאים resuspended ב lysis buffer.
- מערבבים את מתלי התא למשך דקה אחת, ואז מניחים על קרח למשך דקה. חזור על הפעולה במשך 5 פעמים בסך הכל.
- הוסף שני נפחי גלולה של חיץ TN150 ומערבולות במרץ כדי לערבב.
- צנטריפוגה את המתלה בצינור החרוטי ב 4,600 גרם למשך 20 דקות ב 4 ° C בצנטריפוגה ספסל.
- לאחר הצנטריפוגה, קחו דגימה של 50 מיקרוליטר של הסופרנאטנט לניתוח כתם מערבי עתידי כדי לבחון את ביטוי החלבון בתא כולו.
- מעבירים את הסופרנאטנטים לצינורות של 1.5 מ"ל ומסובבים את הליזט במשך 20 דקות ב-20,000 x גרם ב-4°C, במיקרופוגה.
- לחלופין, אם אתה משתמש בצינורות 5 מ"ל, צנטריפוגה ב 13,000 x גרם למשך 20 דקות.
- לאחר הצנטריפוגה, יש לקחת דגימה של 50 μL של הסופרנאטנט לניתוח כתם מערבי עתידי כדי לבחון את הביטוי המסיס של החלבון.
- המשך ללכידת RBP (סעיף 2).
- תאי דבק בתרבית
- זרעו מספיק תאים דבקים בצלחת פטרי 24 שעות לפני קישור צולב UV כך שהם יכולים להגיע 80% מפגש למחרת. לגדול לילה בתווך הרצוי באינקובטור תרבית תאים ב 37 ° C, 5% CO2.
הערה: אם משתמשים בצלחות קוורץ פטרי, כדאי לקדם הידבקות תאים באמצעות טיפול בכלי התרבית עם פולי-D-ליזין (70,000-140,000 wt) וסרום עגל עובר (FCS) 2.5 שעות לפני הזריעה. הוסיפו מספיק פולי-D-ליזין כדי לכסות את כל משטח הגידול ודגרו בטמפרטורת החדר (RT) במשך 5 דקות. לאחר מכן, צלחת פטרי קוורץ צריך להיות שטוף היטב עם מים מיובש אינקובטור תרבית התא במשך 2 שעות או עד יבש לחלוטין. לאחר מכן, להוסיף מספיק FCS כדי לכסות לחלוטין את משטח הגידול ומניחים באינקובטור לפחות 30 דקות. יש להסיר לחלוטין את ה- FCS לפני זריעת תאים. - ברגע שהתאים הגיעו למפגש של 80%, הסר את המדיה ושטוף עם 15 מ"ל של PBS קר כקרח. לאחר מכן, להסיר לחלוטין את כל הנוזלים שנותרו ולהמשיך מיד לשלב הבא.
- העבירו את צלחת הפטרי למגש עבור תאים דבקים (איור 3B) והקרינו UV עם 300 mJ של 254 ננומטר UV. ראו איור 2A ואיור 3B לפרטים על השימוש בקרוסלינקר.
הערה: אנרגיית קרינת UV חייבת להיות מותאמת בקפידה עבור כל חלבון מעניין. ראה דיון לפרטים נוספים. - מיד לאחר קישור צולב, מניחים את צלחת הפטרי על קרח ומוסיפים 10 מ"ל של PBS קר כקרח. לאסוף תאים על ידי גירוד ולהעביר צינור חרוטי 15 מ"ל. גלולה דרך צנטריפוגה ב 300 x גרם במשך 5 דקות ב 4 ° C.
- הסר את PBS והשהה מחדש את גלולת התא ב -1 מ"ל של PBS קר כקרח, והעבר לצינור מיקרוצנטריפוגה של 1.5 מ"ל. תאי גלולה שוב על ידי צנטריפוגה במשך 5 דקות ב 300 x גרם ב 4 ° C.
- הסר את PBS והקפיא את כדורי התא על קרח יבש. יש לאחסן את כדורי התא בטמפרטורה של -80°C עד לצורך.
- חזור על שלבים 1.2.3–1.2.6 עבור כל נקודת זמן.
- להשעות מחדש את כדורי התא ב 1 מ"ל של חיץ ליזה ולהעביר צינור חרוטי 15 מ"ל. לאחר מכן, הוסף 1 מ"ל של חיץ ליזיס עבור סך של 2 מ"ל.
- הוסף 5 μL של מעכב RNase יונקים.
- סוניק 5x במשך 10 שניות על קרח ב 10 אמפר. המתן 30 שניות בין סבבי סוניקציה.
- חישוב ריכוז החלבון של כל דגימה ולנרמל לריכוז הנמוך ביותר.
- העברת 1.98 מ"ל של ליזט לצינור 2 מ"ל.
- הוסף 10 μL של DNase I ודגר ב 37 ° C במשך 5 דקות עם רעד ב 1,200 סל"ד.
- צנטריפוגה את הליזט ב 16,000 x גרם במשך 20 דקות ב 4 ° C.
- לאחר הצנטריפוגה, קחו דגימה של 50 מיקרוליטר של הסופרנאטנט לניתוח כתמים מערביים עתידיים כדי לבחון ביטוי מסיס של החלבון.
- המשך ללכידת RBP (סעיף 2).
- זרעו מספיק תאים דבקים בצלחת פטרי 24 שעות לפני קישור צולב UV כך שהם יכולים להגיע 80% מפגש למחרת. לגדול לילה בתווך הרצוי באינקובטור תרבית תאים ב 37 ° C, 5% CO2.
2. לכידת RBP
- שטפו את האנטי-FLAG המגנטי (75 מיקרוליטר של slurry לדגימה) או IgG agarose (500 μL של slurry לדגימה) חרוזים 3x עם 5 מ"ל של TN150. השהה מחדש בנפח סופי של 700 μL של TN150 והוסף 100 μL של חרוזים שטופים לשבעה צינורות חרוטי 15 מ"ל.
- יש לאחסן על קרח עד לצורך.
- לאחר הבהרת הליזטים, הוסיפו את הסופרנאטנט לשפופרת המכילה את חרוזי האנטי-FLAG/IgG.
- אגוז ב 4 ° C במשך 2 שעות.
הערה: פרוטוקולים מסוימים מתארים דגירות לילה עם החרוזים, אך זה לא מומלץ, מכיוון שזמני דגירה ארוכים יכולים להפחית באופן דרמטי את ההתאוששות של RNA צולב.
3. שטיפת החרוזים ומחשוף TEV של התגים
- קוצרים את החרוזים ומסירים את הליזט.
- קח דגימה של 50 μL של supernatant לניתוח כתם מערבי עתידי כדי לבחון את החלבון שלא נלכד.
- להשעות מחדש את החרוזים ב TN1000 קר כקרח ולהעביר צינור 1.5 מ"ל. יש לשטוף במשך 10 דקות, 4°C, עם אגוז. חזרו על הפעולה במשך שלוש שטיפות בסך הכל.
- אם אתה משתמש חרוזי אגרוז IgG, לשטוף עם 5 מ"ל של TN1000. אם אתם משתמשים בחרוזים נגד FLAG, השתמשו ב-2 מ"ל.
- לאחר מכן, לשטוף את החרוזים 3x עם TN150, עם אותו נפח כמו לעיל.
- לאחר הכביסה השלישית, להשעות מחדש את החרוזים ב 600 μL של TN150.
- הוסף 30 U של פרוטאז GST-TEV תוצרת בית למתלה החרוזים וסובב במשך שעתיים ב- RT.
הערה: פרוטאז רקומביננטי GST-TEV זמין כעת גם באופן מסחרי, אך הוא לא נבדק עם פרוטוקול זה.- במהלך העיכול, להתכונן לשלבים הבאים על ידי הגדרת עמודות של שלוש צינורות 1.5 מ"ל עבור כל דגימה (כלומר, עבור שבע דגימות, יש שלוש שורות של שבע עמודות).
- לשורה האחרונה של צינורות, להוסיף 0.4 גרם של guanidium hydrochloride, 27 μL של 5M נתרן כלורי ו 3 μL של 2.5 M imidazole (pH = 8). שים לב כי ה- pH של imidazole חייב להיות 8. זה קריטי כדי לשמור על שלמות RNA.
- בנוסף, לשטוף את הנפח הנדרש של חרוזי ניקל WB I 3x. השתמש 100 μL של slurry לכל דגימה. לאחר השטיפה הסופית, להשעות מחדש את החרוזים באותו נפח מקורי של WB I ולאחסן על קרח.
- לאחר השלמת עיכול ה-EV, אספו את הסופרנאטנט באמצעות מתלה מגנטי לחרוזי אנטי-FLAG או צנטריפוגה לחרוזי IgG, והעבירו לשורה הראשונה של הצינורות שהותקנו קודם לכן.
- קח 50 μL של דגימה של TEV eluate עבור ניתוח כתמים מערביים.
- הגדר אינקובטור thermoblock ל 37 °C. לשורה השנייה של צינורות, להוסיף 1 μL של קוקטייל RNase (דילול 1:50).
- קח 550 μL של TEV לפלוט משורת הצינורות הראשונה ולהוסיף לשורה השנייה (המכילה את קוקטייל RNase). פיפטה במרץ כדי להבטיח ערבוב.
- לאחר השלמת זה עבור המדגם הראשון, מיד לשים את הצינור לתוך thermoblock ולהפעיל טיימר. עבור לדגימות הבאות, כך שכל אחת מהן מדורגת.
- דוגרים במשך 5 דקות בדיוק. לאחר השלמתה, להסיר את הדגימה הראשונה מן thermoblock ולהעביר את הפתרון לשורה השלישית של צינורות (המכיל אבקת hydrochloride guanidium).
הערה: דגירה של 5 דקות עם דילול של 1:50 של קוקטייל RNase מתאימה בדרך כלל לרוב החלבונים, אך שלב זה יצטרך להיות מותאם בזהירות עם זמני דגירה או ריכוזים שונים עבור כל חלבון כדי לוודא שהרנ"א הצולבים הם בגודל הנכון (30-100 nt). - מערבלים מיד במשך כמה שניות במלוא המהירות כדי להמיס את אבקת הגואנידיום ואז עוברים לדגימה הבאה.
- לאחר שכל הדגימות הועברו לאבקת הגואנידיום, מערבלים שוב כדי להבטיח שכל האבקה מומסת במלואה.
- הוסף 100 μL של חרוזי ניקל שטופים וסובב לילה ב 4 ° C. ניתן לקצר דגירה זו לשעתיים.
4. טיפול פוספטאז אלקליין על חרוז
- הגדר thermoblock ל 37 ° C.
- מניחים עמוד ספין טיהור בצינור של 2 מ"ל, אחד לכל דגימה. מעבירים את חרוזי הניקל לעמודים ומאפשרים לסופרנאטנט להתנקז דרכם. לאחר מכן, ודא שכל חרוזי הניקל הוסרו מצינור 1.5 מ"ל על ידי שטיפה עם WB I ומריחה על העמודה.
- הגדר 2 צינורות מ"ל, שישה לכל דגימה (אחד לאסוף כל שטיפה). שמרו על החלק החיצוני של העמודים יבש כדי לשמור על זרימה. לשטוף את החרוזים 3x עם 500 μL של WB I ולאחר מכן 3x עם 500 μL של NP-PNK.
- סגור את המכסה של עמוד הסחיטה וסובב חרוזים לזמן קצר כדי להסיר מאגר עודף.
- שים את הפקק על העמודה, הכנס עמודות לתוך צינורות של 1.5 מ"ל והוסף 60 μL של תערובת התגובה הנראית בטבלה 2.
| רכיב | 1x | פי 7.5 |
| 5 x מאגר PNK | 12 | 90 |
| אלקליין פוספטאז | 4 | 30 |
| מעכב RNase | 2 | 15 |
| ח2O | 42 | 315 |
| נפח סופי | 60 מיקרוליטר | 450 מיקרוליטר |
טבלה 2: תערובת תגובת פוספטאז אלקליין.
- לדגור את החרוזים במשך 1 שעה ב 37 ° C.
- שטפו את החרוזים 1x עם 500 μL של WB I כדי להשבית את phosphatase אלקליין ולאחר מכן 3x עם 500 μL של מאגר NP-PNK. הקפד לשטוף היטב את החלק הפנימי של העמוד עם מאגר NP-PNK כדי להסיר עקבות של גואנידיום.
5. קשירת חרוזים של מקשר App-PE לקצה 3' של ה-RNA
- סובב החוצה את המאגר הנותר והוסף 60 μL של התערובת שצוינה בטבלה 3 (ראה טבלה 4 עבור רצף App-PE) לעמודות. לדגור את התגובה במשך 6 שעות ב 25 ° C.
| רכיב | 1x | פי 7.5 |
| 5 x מאגר PNK | 12 | 90 |
| מתאם App-PE (100 מיקרומטר) | 0.6 | 4.5 |
| T4 RNA ליגאז 2 K227Q קטוע | 3 | 22.5 |
| מעכב RNase | 1.5 | 11.25 |
| 50% PEG 8000 | 12 | 90 |
| ח2O | 30.9 | 231.75 |
| נפח סופי | 60 מיקרוליטר | 450 מיקרוליטר |
טבלה 3: תערובת תגובת קשירת קישור App-PE.
| שם אוליגונוקלאוטיד | רצף (5'-3') | |||
| L5Aa | invddT-ACACrGrArCrUrUrCrUrNrNrNrUrArArGrCrN-OH | |||
| L5Ab | invddT-ACACrGrArCrUrUrCrUrCrUrUrNrNrArUrUrUrArGrCrN-OH | |||
| L5Ac | invddT-ACACrGrArCrUrUrCrCrGrUrUrCrUrCrUrUrNrNrNrGrCrCrCrCrN-OH | |||
| L5Ad | invddT-ACACrGrArCrUrUrCrUrCrUrUrNrNrNrCrCrCrUrUrUrArGrCrN-OH | |||
| L5Ba | invddT-ACACrGrArCrUrUrUrCrUrCrUrUrCrUrNrNrArArGrArGrCrN-OH | |||
| L5Bb | invddT-ACACrGrArCrUrUrUrCrCrArUrUrCrUrUrNrNrNrUrUrGrGrN-OH | |||
| L5Bc | invddT-ACACrGrArCrUrUrCrUrCrUrUrCrUrNrNrCrArCrCrUrUrGrCrN-OH | |||
| L5Bd | invddT-ACACrGrArCrUrUrUrCrCrGrUrCrUrUrNrNrUrCrUrCrUrUrCrUrGrCrN-OH | |||
| L5Ca | invddT-ACACrGrArCrUrUrCrCrGrArUrUrCrUrUrCrUrNrNrCrUrUrArGrCrN-OH | |||
| L5Cb | invddT-ACACrGrArCrUrUrUrCrCrGrUrUrCrUrUrCrUrNrNrUrGrGrN-OH | |||
| L5Cc | invddT-ACACrGrArCrUrUrUrCrCrArUrUrCrUrUrNrNrArCrCrCrCrArGrCrN-OH | |||
| L5Cd | invddT-ACACrGrArCrUrUrUrCrUrCrUrUrNrNrGrArCrUrArGrCrN-OH | |||
| L5Da | invddT-ACACrGrArCrUrUrUrCrCrCrUrCrUrUrNrNrNrCrGrUrGrUrUrN-OH | |||
| L5Db | invddT-ACACrGrArCrUrUrUrCrCrGrUrCrUrUrNrNrGrCrArCrUrArN-OH | |||
| L5Dc | invddT-ACACrGrArCrUrUrCrUrCrUrUrNrNrUrUrUrGrGrGrGrCrN-OH | |||
| L5Dd | invddT-ACACrGrArCrUrUrUrCrUrNrNrArArCrArCrCrGrN-OH | |||
| L5Ea | invddT-ACACrGrArCrUrUrUrCrCrCrUrCrUrNrNrCrArCrCrUrUrN-OH | |||
| L5Eb | invddT-ACACrGrArCrUrUrUrCrCrArUrCrUrUrNrNrNrUrGrGrGrArCrArN-OH | |||
| L5Ec | invddT-ACACrGrArCrUrUrUrCrUrCrUrUrNrNrUrUrUrUrCrCrCrN-OH | |||
| L5Ed | invddT-ACACrGrArCrUrUrCrUrCrUrUrNrNrNrArCrArGrUrGrN-OH | |||
| App_PE | App-NAGATCGGAAGAGCACACGTCTG-ddC | |||
טבלה 4: רצפי מתאמי הדנ"א והרנ"א הדרושים לקשירה בקצוות 5' ו-3' של רנ"א שנלכד. אלה טוהרו באמצעות HPLC ללא RNase.
- לשטוף חרוזים 1x עם 500 μL של WB I ו 3x עם 500 μL של מאגר NP-PNK. שים את העמודה בצינור חדש וסובב החוצה את המאגר שנותר.
6. זרחן חרוזים של 5' קצוות הרנ"א
- הוסף 80 μL מהתערובת שצוינה בטבלה 5 לעמודות. לדגור על התגובה במשך 40 דקות ב 37 ° C.
הערה: הדגימות יהיו כעת רדיואקטיביות מאוד. לכן, כל העבודה הבאה צריכה להתבצע מאחורי מסך מגן ויש להשליך פסולת על פי כללי הבריאות והבטיחות המקומיים.
| רכיב | 1x | פי 7.5 |
| 5 x מאגר PNK | 16 | 120 |
| 32P-ɣATP (10 μCi/μL) | 3 | 22.5 |
| T4 PNK | 3 | 22.5 |
| ח2O | 58 | 435 |
| נפח סופי | 80 מיקרוליטר | 600 מיקרוליטר |
טבלה 5: תערובת תגובת זרחון.
- הוסף 1 μL של 100 mM ATP ולתת לתגובה להמשיך עוד 20 דקות. זה יבטיח שכמעט בכל קצוות ה-5' יהיו פוספטים כדי להקל על קשירת המקשר 5'.
- הגדר 2 צינורות מ"ל, חמישה לכל דגימה.
- לשטוף חרוזים 1x עם 500 μL של WB I ו 3x עם 500 μL של מאגר NP-PNK. שים לב כי elutions אלה יהיו רדיואקטיביים מאוד ולכן יש להיפטר כראוי.
- העבר את העמודה לצינור הסופי וסובב החוצה את המאגר שנותר.
7. קשירת חרוזים של מקשר 5'
הערה: מקשרי 5' מכילים ברקוד RNA המשמש לזיהוי כל דגימה לאחר ריצוף. לכן, זה בהחלט חיוני לציין איזה מקשר משמש עבור איזה מדגם.
- הוסף 78 μL של התערובת המתוארת בטבלה 6 לעמודות. יש להוסיף 2 μL של מתאם 5' (100 מיקרומטר; ראה טבלה 4) לכל צינור ולדגור למשך הלילה בטמפרטורה של 18°C.
| רכיב | 1x | פי 7.5 |
| 5 x מאגר PNK | 16 | 120 |
| ATP (10 מ"מ) | 8 | 60 |
| מעכב RNase | 2 | 15 |
| T4 RNA ליגאז | 4 | 30 |
| ח2O | 48 | 360 |
| נפח סופי | 78 מיקרוליטר | 585 מיקרוליטר |
טבלה 6: תערובת תגובת קשירת מקשר 5 אינץ'.
- למחרת, לשטוף חרוזים 1x עם 500 μL של WB I ו 3x עם 500 μL של WB II ולהעביר את העמודות לצינור חדש 2 מ"ל.
8. Elution, SDS-PAGE ומיצוי RNA
- הגדר את הצנטריפוגה ל 4 °C. הכינו שתי שורות של צינורות 1.5 מ"ל לכל דגימה לצורך הדבקה.
- סובב החוצה את נפח הריק של העמודות עם חרוזי ניקל עם סיבוב מהיר. מקם את העמודות בשורה הראשונה של צינורות אלוציה והוסף 200 μL של חיץ אלוציה. המתינו 2 דקות ולאחר מכן אלצו את המאגר לעבור דרך הטור בסיבוב מהיר.
- העבר את העמודות לשורת הצינורות השניה וחזור על שלב 8.2. כל דגימה תכיל כעת 400 μL של פליטה בסך הכל, המפוצלת על פני שני צינורות של 1.5 מ"ל.
- קח את כל eluates ולהעביר אותם יחד צינור 5 מ"ל. הוסף 2 μL של 20 מ"ג / מ"ל של גליקוגן. לכן, אם באמצעות שבע דגימות, יהיה עכשיו 2.8 מ"ל של eluate מאגר בצינור 5 מ"ל.
- הוסף 100 μL של חומצה טריכלורואצטית (TCA) לכל דגימה [למשל 700 μL של TCA עבור 7 דגימות (2.8 מ"ל של eluate מאוגם)] לצינור 5 מ"ל, ומערבול היטב במשך 30 שניות.
- דוגרים על קרח במשך 20 דקות.
- צנטריפוגה למשך 30 דקות במהירות של 17,000 x גרם, 4 מעלות צלזיוס, בצנטריפוגה על ספסל.
- בזהירות להסיר את supernatant מן הצינור החרוט, לבדוק את פיפטה עם מונה גייגר כדי לוודא את הגלולה לא הוסר בטעות. אם כן, החזירו את הסופרנאטנט לצינור ולצנטריפוגה למשך 10 דקות נוספות.
הערה: הסופרנאטנט עדיין עשוי להיות רדיואקטיבי מאוד. הקפידו להשתמש במיגון מתאים. - לחלוטין להשעות את הגלולה ב 2 מ"ל של אצטון קר כקרח.
- צנטריפוגה למשך 15 דקות ב 17,000 x גרם, 4 ° C.
- הסר כמה שיותר מהאצטון עם פיפטה P1000. לאחר מכן, סובב בקצרה את הצינור כדי לאסוף טיפות קטנות של אצטון, ולאחר מכן להסיר עם פיפטה P10. יבש במשך 2 דקות במכסה אדים.
הערה: סופרנאטנט אצטון עדיין יכול להיות רדיואקטיבי. הקפידו להשתמש במיגון מתאים. - יש להשעות מחדש את הדגימה ב-30 מיקרוליטר של מאגר העמסת חלבון 1x. כדי להבטיח שהגלולה תלויה מחדש כראוי, בדוק שהרוב המכריע של הרדיואקטיביות נמצא כעת במאגר ההעמסה ולא נשאר בצינור של 1.5 מ"ל על ידי הסרת התמיסה בפיפטת P200 ומדידת הפעילות שנותרה בצינור 1.5 מ"ל באמצעות מונה גייגר.
- חממו את הדגימה במשך 10 דקות ב-65°C. יש להעמיס על ג'ל Bis-Tris פריקאסט בקוטר 1 מ"מ, 4-12% ולרוץ במשך שעה וחצי ב-125 וולט במאגר MOPS.
- לאחר שהג'ל סיים לפעול, פתח את קלטת הג'ל. הג'ל צריך להישמר על הצלחת התחתונה. יש להשליך את החלק העליון.
- עטפו את הג'ל בסרט נצמד ולאחר מכן הדקו אותו באמצעות נייר דבק לחלק הפנימי של קלטת אטומה לאור. ודא שלקלטת יש מסך הגברה לשיפור האות.
- חשוף סרט אוטורדיוגרפי לג'ל ואחסן את הקלטת ב -80 מעלות צלזיוס במהלך החשיפה. זמן החשיפה ישתנה בין חלבונים בעלי יעילות הצלבה שונה.
- בעת הצבת הסרט, חייבת להיות דרך ליישר אותו מחדש לקלטת על מנת לחתוך את רצועת העניין בשלב הבא. כדי להבטיח זאת, השתמש בסרגל ניאון וגם לוודא כי הג'ל נמצא בפינה של הקלטת, אשר מכוסה על ידי הסרט ממוקם גם בפינה העליונה.
הערה: ככלל אצבע, חוצץ העמסה שנותן קריאה של לפחות ~250 cps כאשר הוא מוצג למונה גייגר נותן אות מספיק לחשיפה של 3 שעות. אחרת, מתבצעת חשיפה למשך הלילה.
- בעת הצבת הסרט, חייבת להיות דרך ליישר אותו מחדש לקלטת על מנת לחתוך את רצועת העניין בשלב הבא. כדי להבטיח זאת, השתמש בסרגל ניאון וגם לוודא כי הג'ל נמצא בפינה של הקלטת, אשר מכוסה על ידי הסרט ממוקם גם בפינה העליונה.
- לפתח את הסרט. חותכים את סרט ההיצמדות המכסה את הג'ל אך לא מזיזים את הג'ל. אחרת, התמונה תוסט מהג'ל.
הערה: סביר להניח שהג'ל יהיה רדיואקטיבי מאוד. הקפידו להשתמש במיגון מתאים בעת חיתוך פרוסת הג'ל. - מניחים את הסרט מעל הג'ל ומוציאים את רצועת העניין. שים את פרוסת הג'ל לתוך צינור 2 מ"ל.
- מרסקים את פרוסת הג'ל באמצעות קצה פיפטה P1000 ומוסיפים 600 מיקרוליטר של חיץ פרוטאינאז K בתוספת 200 מיקרוגרם של פרוטאינאז K (פרוטוקול זה משתמש ב-10 מיקרוליטר של תמיסת פרוטאינאז K במינון 20 מ"ג/מ"ל). לדגור במשך 2 שעות ב 55 ° C עם רעד נמרץ.
- לאחר מכן, חתכו את קצה קצה P1000 בעזרת אזמל נקי והעבירו את חתיכות הסופרנאטנט והג'ל לעמוד סיבוב הממוקם בצינור בנפח 2 מ"ל.
- סובב את העמודה במשך דקה אחת במהירות של 17,000 x גרם ב- RT. אסוף את הזרימה, המכילה את הרנ"א הרדיואקטיבי והמבודד.
- בצע מיצוי פנול:כלורופורם.
- הוסף 50 μL של 3 M נתרן אצטט, pH = 5.2, ו 500 μL של פנול:chloroform ומערבולת היטב. סבבו במשך 5 דקות במהירות של 17,000 x גרם. הסר את השכבה העליונה המימית ומניחים בצינור חדש של 1.5 מ"ל.
- מוסיפים 500 מיקרוליטר כלורופורם ומערבולות במרץ למשך 10-15 שניות. סבבו במשך 5 דקות במהירות של 17,000 x גרם ב-RT. הסירו את השכבה המימית והניחו אותה בצינור חדש של 1.5 מ"ל.
- הוסף 1 μL של 20 מ"ג / מ"ל גליקוגן ו 1 מ"ל של קר כקרח, 96% אתנול. יש לזרז במשך 30 דקות ב-80°C (-80°C) או למשך הלילה ב-20°C (-20°C).
- צנטריפוגה למשך 30 דקות ב-4°C, 17,000 x גרם. הסר את supernatant, להוסיף 500 μL של 70% אתנול וצנטריפוגה במשך 5 דקות, 4 ° C ב 17,000 x גרם. מוציאים את כל האתנול, מבצעים סיבוב מהיר לאיסוף שאריות ומסירים עודפים עם פיפטה P10.
- יבש את הגלולה במשך ~ 3 דקות במכסה אדים. יש להשהות מחדש ב-20 מיקרוליטר של מים שטופלו ב-DEPC.
- אחסנו את הרנ"א בטמפרטורה של -80°C למשך הלילה או המשיכו מיד לשלב השעתוק ההפוך.
9. תמלול הפוך
- הוסף 2 μL של 10 μM RT oligo (PE_reverse; ראה טבלה 7) ו- 4 μL של 5 mM dNTPs ל- 20 μL של RNA.
| שם אוליגונוקלאוטיד | רצף (5'-3') | |||
| P5 קדימה | AATGATACGGCGACCACCGAGATCTACACTTTCCCTACACGACGCTCTTCCGATCT | |||
| BC1 | CAAGCAGAAGACGGCATACGAGATCGTGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| BC3 | CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGTTCAGACGTGTGCTCTTCCGATCTCT | |||
| BC4 | CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| BC5 | CAAGCAGAAGACGGCATACGAGATCACTGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTCT | |||
| BC7 | CAAGCAGAAGACGGCATACGAGATCAGATCGTGACTGGTTCAGACGTGTGCTCTTCCGATCTCT | |||
| BC8 | CAAGCAGAAGACGGCATACGAGATTAGCTTGACTGGAGTTCAGACGTGCTCTTCCGATCTCT | |||
| BC9 | CAAGCAGAAGACGGCATACGAGATGATCAGGTGACTGGTTCAGACGTGTGCTCTTCCGATCTCT | |||
| BC10 | CAAGCAGAAGACGGCATACGAGATATCACGGTGACTGGAGTTCAGACGTGCTCTTCCGATCTCT | |||
| PE_reverse | CAGACGTGTGCTCTTCCGATCT | |||
טבלה 7: פריימרים PCR (כולל רצפי ברקוד) ופריימר תמלול הפוך.
- מעבירים לתרמובלוק שחומם מראש ב-85°C למשך 3 דקות, ואז מצננים על קרח למשך 5 דקות. לאסוף את התוכן של הצינור על ידי צנטריפוגה קצרה ולאחר מכן להוסיף 8 μL של 5x חיץ שעתוק הפוך, 2 μL של 100 mM DTT, ו 2 μL של מעכב RNase.
- לדגור על התערובת ב 50 ° C במשך 3 דקות ולאחר מכן להוסיף 2 μL של transcriptase הפוך לדגור במשך 1 שעה ב 50 ° C.
- השבת את התעתיק ההפוך על ידי דגירה ב 65 ° C למשך 15 דקות.
- מעבירים את הצינורות לתרמובלוק שחומם מראש ב-37°C ומשאירים למשך 3 דקות להתאקלמות.
- הוסף 2 μL של RNase H ודגור במשך 30 דקות ב 37 ° C.
- בודדו את ה-cDNA באמצעות חרוזי SPRI.
- הוסף שני כרכים של 84 μL של חרוזים. דוגרים במשך 15 דקות. הניחו את החרוזים על מדף מגנטי והשאירו למשך דקה אחת כדי לקצור את החרוזים.
- הסר ולהיפטר supernatant ולהוסיף 200 μL של 70% אתנול. אין להסיר את החרוזים מהמדף המגנטי. לדגור את החרוזים עם אתנול במשך 30 שניות.
- מוציאים את האתנול וחוזרים על שלב השטיפה. הסר את כל שאריות האתנול באמצעות קצה P10.
- שים את החרוזים במכסה אדים למשך 2 דקות כדי לייבש אותם. הסר את החרוזים מהמדף, השהה אותם מחדש ב -12 מיקרוליטר מים ולאחר מכן החזיר את החרוזים למתלה. יש להסיר 11 μL של supernatant.
- להקפיא את cDNA ב -20 ° C או להמשיך מיד לשלב PCR.
10. תגובת qPCR
- לפני ה-PCR הסופי להגברה של cDNAs, מבוצעת תגובת שרשרת כמותית של פולימראז (qPCR) כדי לזהות את מספר המחזורים האופטימלי להגברת cDNA כדי למנוע הגברה יתר של הספרייה.
- הגדר תגובת qPCR על קרח לפי טבלה 8. ראו טבלה 7 לכל הפריימרים.
| רכיב | 1x |
| 2x תערובת מופת של תגובת qPCR | 5 |
| פריימר P5 0.1 מיקרומטר (קדימה) | 0.8 |
| פריימר 0.1 מיקרומטר BC (הפוך) | 0.8 |
| cDNA (או מים כבקרה שלילית) | 1 |
| ח2O | 2.4 |
| נפח סופי | 10 מיקרוליטר |
טבלה 8: תערובת תגובת qPCR.
- לכימות נכון של המחזורים הדרושים להגברה, השתמש בשלושה עותקים טכניים עבור cDNA ושלושה פקדים שליליים (כלומר מים).
- אטמו את הצלחות בסרט אופטי שקוף והפעילו את ה-qPCR בהתאם להוראות יצרן הערכה.
- נתחו את הדגימות באמצעות שיטת כימות מוחלטת כדי לזהות את מספר המחזורים (n) שבהם מגיעים לברך של גדילה מעריכית (ראו איור 4C לדוגמה). מספר זה של מחזורים משמש לאחר מכן להגברה הסופית של שאר cDNA.
11. תגובת PCR ומיצוי ג'ל
- הגדר את תגובת ה-PCR על הקרח לפי טבלה 9. ראו טבלה 7 לכל הפריימרים.
הערה: רק 5 μL של ספריית cDNA משמש.
| רכיב | 1x |
| 10 x חיץ פולימראז להגהה | 5 |
| פריימר P5 בגודל 10 מיקרומטר (קדימה) | 1 |
| פריימר 10 מיקרומטר BC (הפוך) | 1 |
| 5 mM dNTPs | 2.5 |
| הוכחת קריאת אנזים פולימראז | 1 |
| cDNA | 5 |
| ח2O | 34.5 |
| נפח סופי | 50 מיקרוליטר |
טבלה 9: תערובת תגובת PCR.
- הפעל PCR באופן הבא: 95 °C למשך 2 דקות; n מחזורים של 98 ° C במשך 20 שניות, 52 ° C עבור 30 שניות, ו 72 ° C במשך 1 דקות; ו 72 ° C במשך 5 דקות. מספר (n) המחזורים להגברת ספריית χCRAC נקבע על ידי qPCR המתואר בסעיף 10.
- הוסף 1 μL של exonuclease I ודגר ב 37 ° C במשך 60 דקות.
- נקה את ה- cDNA המוגבר באמצעות חרוזי SPRI כמתואר לעיל באמצעות שני נפחים של חרוזים (כלומר 100 μL). Elute ב 11 μL.
- הוסף 3 μL של צבע העמסה 6x והפעל על ג'ל TBE פריקאסט 6% ב 100 V למשך שעה אחת במאגר TBE 1x. השתמש בסולם המתאים לכימות של מקטעי DNA קצרים.
- לאחר שתסיים, הסר את הג'ל מהקלטת ושים בכלי מתאים, אטום לנוזל עם מספיק 1x TBE כדי לכסות את הג'ל (למשל ~ 50 מ"ל). הוסף כמות מתאימה של צבע בטוח לשימוש SYBR (לדוגמה, עבור 50 מ"ל, השתמש ב-5 μL של צבע 10,000x)
- הניחו לג'ל להכתים באמצעות ערבוב עדין למשך 15 דקות ב-RT. סננו את ה-1x TBE המכיל SYBR והוחלפו ב-1x TBE נקי. שטפו את הג'ל במשך 10 דקות בניעור עדין ב-RT.
- מסננים את ה-1x TBE ומניחים את הג'ל בתיקייה שקופה. גזור את התיקיה לגודל מתאים.
- דמיינו את הג'ל באמצעים מתאימים כגון זרחן. בלו מקטעי DNA בין ~ 175 bp ו ~ 400 bp. שים את פרוסת הג'ל בצינור 1.5 מ"ל.
- יש לרסק היטב את פרוסת הג'ל בעזרת קצה P1000 ולהוסיף 400 μL של H2O. לדגור בטמפרטורה של 37°C עם ניעור במשך שעה בתרמובלוק.
- הקפיאו את הדגימה על קרח יבש למשך 10 דקות, ולאחר מכן הניחו בחזרה בתרמובלוק בטמפרטורה של 37°C עם ניעור במשך שעה אחת.
- צור יחידת סינון על-ידי לקיחת עמודת מסנן והכנסת שני מסנני מיקרופייבר זכוכית פנימה. מניחים את היחידה בצינור של 1.5 מ"ל.
- חתכו את קצה קצה P1000 בעזרת אזמל נקי וספגו את מתלה הג'ל TBE המרוסק, ולאחר מכן העבירו ליחידת המסנן שנוצרה בשלב 11.12. סובב ב 17,000 x גרם במשך 30 שניות.
- הוסף 1 μL של גליקוגן supernatant, יחד עם 40 μL של נתרן אצטט, pH = 5.2, ו 1 מ"ל של 96% אתנול. יש לדגור בטמפרטורה של -80°C למשך 30 דקות.
- צנטריפוגה למשך 30 דקות ב-17,000 x גרם, 4°C. יש להשליך את הסופרנאטנט ולשטוף עם 500 μL של 70% אתנול.
- יש לסחור במשך 5 דקות, להסיר את האתנול לחלוטין ולאחר מכן לייבש את הגלולה במכסה אדים למשך 3 דקות.
- השהה מחדש ב 10 μL של H2O ולמדוד את ריכוז ה- DNA.
תוצאות
כדי להדגים את יעילותה של שיטת χCRAC, בוצע ניסוי בזמן עם זני שמרים המבטאים חלבון Nrd1 המתויג על ידי HTP. ייצוג סכמטי מפורט המתאר כיצד השיטה פועלת מוצג באיור 1. כמו Nab3, Nrd1 מעורב בדעיכת RNA גרעיני של מגוון תעתיקי RNA37. עבודה קודמת ממעבדת קורדן הצביעה על כך שקישור Nrd1 למטרות הרנ"א שלו משתנה באופן משמעותי כאשר תאים נתונים לרעב גלוקוז28,38. לפיכך, תאים שגדלו באופן אקספוננציאלי בתווך המכיל גלוקוז (SD-TRP) הועברו לאותו תווך ללא גלוקוז (S-TRP) לאורך מסלול זמן כדי לעקוב אחר שינויים דינמיים באינטראקציות Nrd1-RNA. הדגימות נלקחו והוצלבו בתא Vari-X-linker (איור 3A) לפני המשמרת ולאחר מכן לאחר 1, 2, 4, 8, 14 ו-20 דקות. המדיום המשמש לצמיחת תאים היה חסר במכוון בטריפטופן כדי להפחית את ספיגת UV על ידי חומצת אמינו ארומטית זו. שימו לב שעדיף להשתמש במדיום סינתטי שעובר סינון מעוקר מכיוון שאוטוקלאבינג של המדיום יכול להוביל לקירמול של הסוכרים. פעולה זו מפחיתה את יעילות הקישור הצולב.
איור 4A מראה אוטורדיוגרף מייצג מניסוי χCRAC. שים לב שבדוגמה זו, הדגימות לא אוגדו יחד. במקום זאת, כל אחד מהם הופעל בנפרד על הג'ל. זה מומלץ לבדיקות ניסיוניות ראשוניות כדי להראות כי החלבון מצטלב ביעילות עם RNA בכל נקודות הזמן שנבדקו. אות חזק במיוחד נצפה במשקל המולקולרי הצפוי של ה-RBP, המייצג את החלבון הקשור לרנ"א קצר מאוד, רדיואקטיבי שאינו ניתן לריצוף. לכן, בודד אות המריחה מעל רצועה זו, שהוא החלבון המוצלב למקטעי רנ"א ארוכים יותר. השבר נחתך ממש מעל רצועת החלבונים בתוספת כ-30 kDa. איור 4B מראה אוטורדיוגרמה לאחר כריתה, כאשר החלבון מקושר לרנ"א קצר שנותר בג'ל והאות שנמרח בעבר נכרת כעת.
לאחר שעתוק לאחור, יש להגביר את ספריית cDNA באמצעות PCR. עם זאת, יש להימנע מהגברה מוגזמת של הספרייה מכיוון שזה יכול להציג הטיה לכיוון רצפים המוגברים באופן מועדף על ידי הפולימראז וליצור חפצי PCR. ספריות מוגברות יתר מכילות גם מספר רב של רצפים כפולים שמבזבזים קריאות על הרצף. על מנת לחשב את המספר האידיאלי של מחזורי PCR להגברה של הספרייה הסופית, Aliquot של cDNA הוגבר באמצעות qPCR באמצעות אוליגונוקלאוטידים P5 ו- BC. המחזור הראשון שבו הגיעה הספרייה לשיא הפלואורסצנטיות נבחר כספירת מחזורי PCR. איור 4C נותן דוגמה של qPCR מספריית cDNA טיפוסית, שהניבה ספירת מחזור שיא של 16. ערך זה שימש לאחר מכן עבור χCRAC PCR הסופי. על מנת לעבד את הנתונים הרצפים, השתמשנו בתוכנה שפותחה בעבר במעבדה שלנו (pyCRAC) ובצנרת המתאימה לניתוח נתוני CRAC קינטיים (Nues et al., 2017; https://git.ecdf.ed.ac.uk/sgrannem/pycrac, https://bitbucket.org/sgrann/kinetic_crac_pipeline/src/default/). כלי תוכנת קוד פתוח אלה מאפשרים דה-ריבוב וחיתוך של הנתונים, הסרת כפילויות PCR, זיהוי שיאים מובהקים סטטיסטית, קריאות אשכולות לרצפים רציפים וזיהוי מוטיבים מחייבים39. פרטים נוספים על אופן הפעולה של כלים אלה נמצאים בדפי האינטרנט שלהם.
התחלנו גם לפתח פרוטוקול χCRAC עבור תאי יונקים. רוב קווי התאים של יונקים גדלים כחד-שכבתיים, והמגש בקרוסלינקר שלנו עם השקית החדירה UV אינו מתאים לניסויים בתאים דבקים. כדי להתגבר על הבעיה הזו, פיתחנו שלב שבו משתמשים יכולים להקרין קרינת על-סגול של 1-2 צלחות פטרי (קוטר של 150 מ"מ ועומק של 25 מ"מ) עם תאים דבקים (איור 3B). כבדיקה ראשונה, נמדדה יעילות הקרוס-לינקר עבור תאי יונקים באמצעות הצלבה ולכידה של GFP-RBM7 המתויג ביציבות באמצעות נוגדנים נגד GFP וטיהור מסורתי מבוסס CLIP. כפי שניתן לראות באיור 5A, הקרוס-לינקר הצליח לשחזר קומפלקסים של חלבון-רנ"א מתאי יונקים שגדלו כחד-שכבה באמצעות קרינת UV של 254 ננומטר ביעילות דומה למכשיר קרינת UV הנמצא בשימוש נרחב. עם זאת, כלי פלסטיק סטנדרטיים של תרביות תאים המשמשים בדרך כלל לניסויי קישור צולב UV הם בלתי חדירים ל- UV של 254 ננומטר. לכן, בקרוס-לינקר שלנו התאים יקבלו רק קרינה מהגדה העליונה של מנורות UV. כדי להתגבר על כך, פיתחנו צלחת קוורץ פטרי חדירה UV לצמיחת תאים וקישור צולב. השימוש בכלי תרבית הקוורץ הראה התאוששות חזקה של קומפלקסים חלבוניים-רנ"א עם 2 שניות בלבד של קרינת UV (איור 5B). בשילוב עם שיטות לכידת RBP עבור תאי יונקים כגון טכנולוגיות CLIP, זמני קישור צולבים קצרים אלה ניתנים להתאמה עם מסלולי זמן לשחזור פרופילים קושרי RNA מרחביים-זמניים של RBPs בתגובה לעקות גנוטוקסיות או דלדול מהיר של גורמי חלבון, או במקביל לסנכרון שעתוק או מחזור התא.
איור 6 מציג מספר דוגמאות של נתוני Nrd1 המעובדים על-ידי צינור χCRAC. איור זה הוכן באמצעות קבצי הבדגרף שנוצרו על ידי הצינור וחבילת Python GenomeBrowser (https://pypi.org/project/GenomeBrowser/1.6.3/), שעיצבנו כדי לפשט את יצירת תמונות דפדפן גנום באיכות פרסום של הנתונים. המלבנים האפורים מייצגים אזורים גנומיים שביטאו רנ"א לא מקודד, כגון תעתיק לא יציב מוצפן (CUTs), תעתיק יציב לא מאופיין (SUTs)40 ותעתיקים לא יציבים רגישים ל-Xrn1 (XUTs)41. הנתונים באיור 6 מראים ש-Nrd1 נקשר לרבים מתעתיקי הרנ"א הלא-מקודדים האלה, מה שעולה בקנה אחד עם הרעיון שחלבון זה מעורב בפירוק של סוג זה של תעתיקים42. איור 6A מראה אזור של ~15 קילו-בתים בכרומוזום IV. כאן חלה עלייה משמעותית בקשירה של Nrd1 לתעתיקים המקודדים את מובילי הגלוקוז בעלי הזיקה הגבוהה HXT6 ו- HXT7, שניהם מווסתים במהלך רעב גלוקוז. סביר להניח כי סיום שעתוק על ידי קומפלקס NNS עשוי להשפיע על קינטיקה אינדוקציה של גנים אלה במהלך רעב גלוקוז. איור 6B מראה דוגמה של קישור צולב Nrd1 לתמלול Imd3, אשר ידוע כי הוא מוסדר על-ידי Nab343. במקרה זה הנתונים הראו ירידה משמעותית בקשירה לרעב גלוקוז. עבודות קודמות הראו ירידה בקשירה של Nab3 לתמלול Tye7 במהלך הרעבת גלוקוז44. בהתאם לתצפית זו, נתוני χCRAC מצביעים על כך שהקישור של Nrd1 פחת במהלך הרעבת גלוקוז והקישור הצולבות של Nrd1 ל-Tye7 היה הנמוך ביותר לאחר 8 דקות של לחץ (איור 4C). עם זאת, נראה כי השפעה זו הייתה חולפת בלבד, מכיוון שלאחר 14 דקות של רעב גלוקוז, קשירת Nrd1 חזרה לרמות ההתחלה.
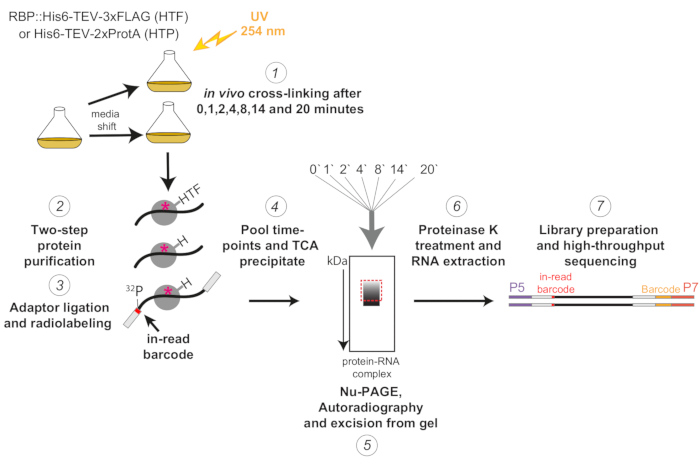
איור 1: ייצוג סכמטי של פרוטוקול χCRAC. זנים מתויגים גודלו עד לצפיפות הרצויה. RBP מציין חלבון קושר RNA. לאחר מכן, נלקחה דגימת ייחוס והוצלבה עם אור UV 254 ננומטר. התאים הנותרים נקצרו על ידי סינון ולאחר מכן הועברו במהירות למדיום מעורר הלחץ. עבור ניסוי χCRAC המתואר כאן, דגימות נלקחו והצולבו 1, 2, 4, 8, 14 ו -20 דקות לאחר המשמרת (1). לאחר מכן ה-RBP של העניין טוהר באמצעות טיהור זיקה דו-שלבי מחמיר ביותר (2). לאחר מכן, הרנ"א הצולבות שנלכדו עוכל חלקית באמצעות RNases, סומנו ברדיו בקצה ה-5' ומתאמים נקשרו אליהם (3). מתאמי 5' הכילו רצפי ברקוד ייחודיים "בתוך קריאה", כך שניתן היה להפריד את הדגימות הבודדות באופן ביואינפורמטי לאחר ריצוף. קומפלקסי RBP-RNA הוצאו, אוגמו והואצו יחד (4), נפתרו על ידי SDS-PAGE והודמיינו באמצעות אוטורדיוגרפיה (5). לאחר מכן, פרוסת ג'ל בודדת המכילה את האות הרדיואקטיבי ממש מעל הרצועה הראשית, מאוירת עם קופסה אדומה מקווקו בתמונת האוטורדיוגרפיה, נחתכה מהג'ל (5). פרוסות הג'ל טופלו בפרוטאז K והרנ"א חולץ לאחר מכן (6), הומר ל- cDNA והוגבר באמצעות PCR (7). צעד ה-PCR הכניס ברקודים נוספים (בלוק צהוב שהוצג על ידי אוליגו P7) כך שניתן היה להכפיל ספריות רבות לנתיב אחד. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.

איור 2: קישור צולב וסינון ואקום. (A) הקרוס-לינקר. מתלה התא מוזג לתוך משפך הממוקם בפינה הימנית העליונה של המכונה (ראו גם איור 3A לתקריב) ומוחזק בשקית שקופה UV הממוקמת במגש האמצעי. שקית זו מוקפת בשני תריסים שנשארים סגורים עד שהמשתמש מורה למכונה להתחיל את שלב ההקרנה. התאים מוקרנים באור UV מהמגשים שמעל ומתחת. המכונה מגיעה עם מנורות UV 254 ו 365 ננומטר, כאשר האחרונה ישימה לניסויי PAR-CLIP. המכשיר מופעל באמצעות לוח מסך מגע הממוקם בפינה השמאלית העליונה המאפשר לשלוט במינון UV או בזמן החשיפה. (B) לאחר קישור צולב, התאים מנוקזים מהצד השמאלי של המכונה. מתלי התאים מוחזרים באמצעות ואקום ומנוקזים לבקבוק זכוכית, שם ניתן לשפוך אותם לאחר מכן למכשיר סינון ואקום לקציר. (C) מכשירי סינון ואקום. אלה נפתחים ונסגרים באמצעות קליפ ומוכנס מסנן ביניהם. ארבעה מכשירי סינון שימשו במקביל לסדרות זמן קצרות מאוד כדי לא לאבד זמן כתוצאה משינוי פילטרים. (D) לאחר הסינון, נוקז חומר התקשורת לצלוחיות לסילוק לאחר מכן. מתחת להתקני סינון הוואקום הותקנו שסתומים כדי לשמור על הוואקום במערכת בעת הסרת המסנן. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.

איור 3: קישור צולב בין תאים מרחפים לעומת תאים דבקים. (A) הקרוס-לינקר עם תא Vari-X-linker לתאי תרחיף. תרבית התאים נשפכת לתוך כניסת הדגימה (משפך) הממוקם בפינה השמאלית העליונה של המגש. (B) מגש שיכול להכיל צלחות פטרי מפלסטיק או קוורץ לצורך קישור צולב בין תאים דבקים או נפחים קטנים של תאי תרחיף. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.

איור 4: הכנת הספרייה. (A) דוגמה לאוטורדיוגרמה מניסוי Nrd1-HTP χCRAC. האות החזק והמרוכז מייצג את החלבון המוצלב לרנ"א קצר מאוד, ואילו הכתם לעיל מייצג את החלבון המקושר לרנ"א באורך מספיק לריצוף. (B) הכתם נכרת כפי שמוצג באוטורדיוגרמה שנלקחה לאחר כריתת ג'ל. (C) qPCR מייצג מספריית cDNA χCRAC. בדוגמה זו, הגברה מקסימלית של cDNA הושגה ב -16 מחזורים. לפיכך, 16 מחזורים שימשו להגברה הסופית. סרגל השגיאה מייצג את סטיית התקן של שלושה עותקים משוכפלים טכניים של qPCR. (D) דוגמה של זרחן מספריית cDNA על ג'ל TBE של 6%. (E) ניתוח אורך ואיכות cDNA מאלקטרופורזה נימית מבוססת שבב. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
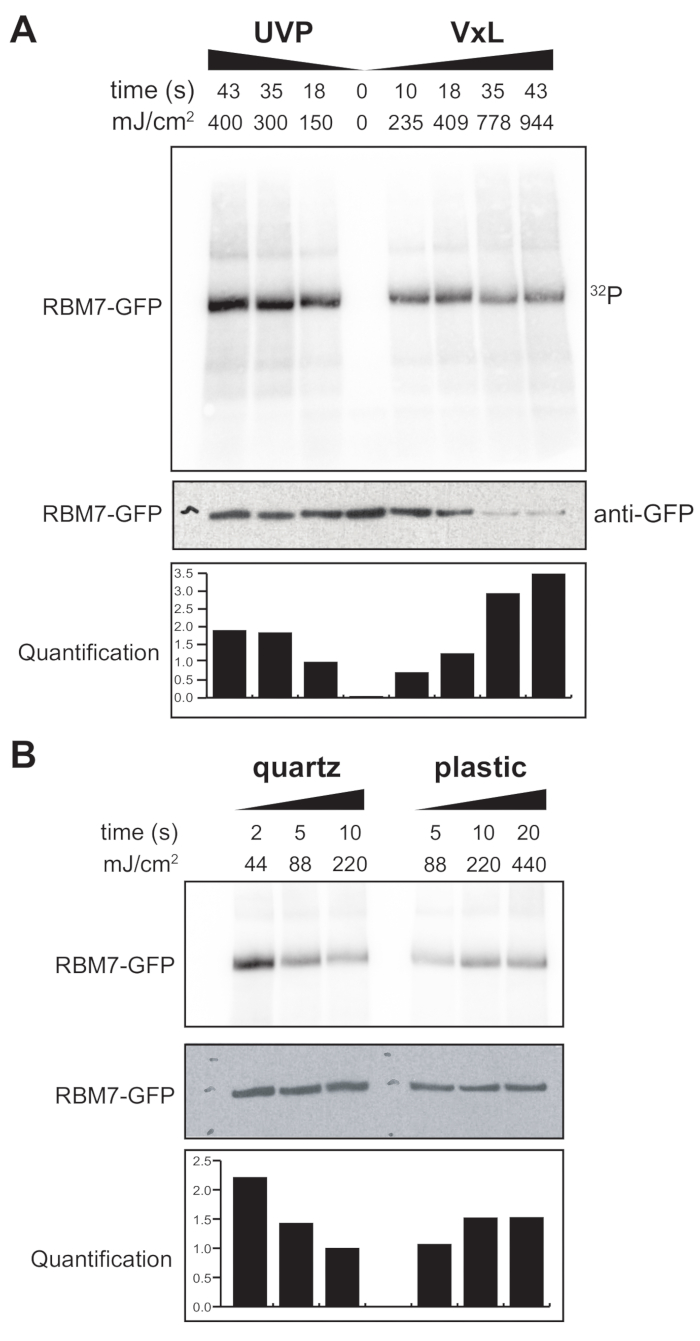
איור 5: ניסוי iCLIP לבדיקת High RNase לבדיקת קרוסלינקינג בתאי יונקים. מוצגים אוטורדיוגרמות מניסויי GFP-RBM7 iCLIP אשר בדקו את יעילות התאוששות RNP על פני אנרגיות צולבות שונות. משקעים חיסוניים בוצעו באמצעות נוגדנים נגד GFP המוצמדים לחרוזים מגנטיים על תאים צולבים אשר ביטאו ביציבות GFP-RBM7. Immunoprecipitates הודגרו עם ריכוזים גבוהים של RNase I על מנת לחתוך RNA הקשורים לאורכים קצרים ואחידים. RNPs הודגמו על ידי תיוג 32P ו- SDS-PAGE ונדדו כפס מוגדר, קרוב לנדידה של חלבון שאינו מקושר. כימות מציין את התוצאות של ניתוחים דנסיטומטריים של אות RBM7-RNA רדיואקטיבי מנורמל לאות הכתם המערבי נגד GFP. (A) מסלול זמן קרוס-לינקינג של קרוס-לינקר UVP הנפוץ לעומת קרוס-לינקר שלנו (Vari-X-linker; VxL). (B) מסלול זמן של קרוס-לינקר שלנו על כלי תרבות קוורץ (משמאל) ופלסטיק (מימין). אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
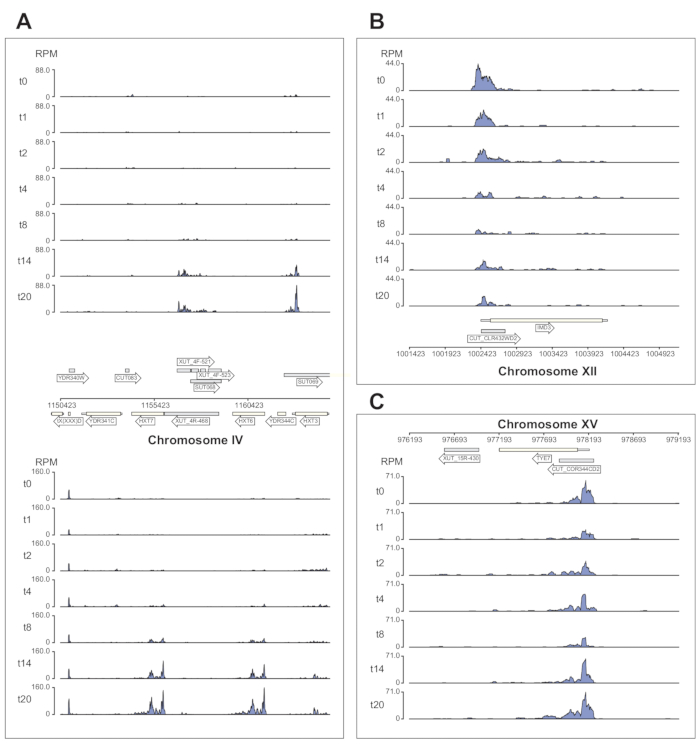
איור 6: תרשימי דפדפן גנום לדוגמה המראים את כוחו של χCRAC להראות קשירה זמנית דיפרנציאלית של Nrd1 למטרותיו. כל תיבה מציגה תרשימים עבור אזורים גנומיים בודדים. החצים מציינים על איזה גדיל מקודדים הגנים (חץ מצביע שמאלה = מינוס גדיל; חץ מצביע ימינה = גדיל פלוס). נקודות הזמן (דקות) מסומנות על ידי t0, t1, t2 וכו 'על צירי y של כל חלקת משנה. ספרות רומיות המציינות את הכרומוזומים ואת הקואורדינטות מוצגות. (A) עם חסך גלוקוז, Nrd1 קושר שני מובילי גלוקוז בעלי זיקה גבוהה, HXT6 ו-HXT7, אשר שניהם מווסתים במצב זה. (B) Nrd1 נצפה נקשר ל-Imd3, יעד שכבר אומת של Nab344, עם עוצמה מופחתת בעקבות רעב גלוקוז. (C) קשירת Nrd1 של Tye7 מציגה אופי דינמי וחולף, ופוחתת לאחר רעב גלוקוז למינימום לאחר 8 דקות של מתח. עם זאת, הקשירה חוזרת לאחר מכן לרמות הבסיס לאחר 14 דקות. הקריאות נורמלו ל"קריאות למיליון" (סל"ד; ציר y). תיבות אפורות מציינות אזורים המקודדים רנ"א שאינו מקודד. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Discussion
לשיטת χCRAC, בשילוב עם ההתקנים החדשים לקישור צולב וקצירת תאים, יש פוטנציאל גדול מכיוון שהיא ישימה למגוון רחב של אורגניזמים לדוגמה ולכן צריכה להיות בעלת עניין כללי בתחום הרנ"א. ישנם תחומים רבים בהם ניתן להשתמש ב-χCRAC. לדוגמה, השיטה יכולה לשמש למדידת ההרכבה ההיררכית של חלבונים לקומפלקסים מקרומולקולריים גדולים, כגון spliceosome ו- ribosome, אשר לעתים קרובות כרוך באינטראקציות דינמיות בין חלבונים ומולקולות RNA. כיום אנו גם משתמשים בו באופן שגרתי כדי לנטר אינטראקציות בין גורמי דעיכת רנ"א לבין הסובסטרטים שלהם כאשר תאים נתונים לסוגים שונים של עקה. זה מאפשר לנו לקבוע באיזה שלב של התגובה האדפטיבית הגורמים האלה הם הפעילים ביותר, לאילו מצעים הם נקשרים, ועד כמה דינמיים אינטראקציות אלה. נתונים כאלה אמורים לאפשר לחוקרים לקבוע את תרומתו היחסית של כל גורם להסתגלות לשינויים סביבתיים.
χCRAC משתמש בתגי טיהור זיקה כפולה (HTF או HTP) כדי לטהר את החלבון בתנאים מחמירים ביותר ודנטורציה. זה מבטיח שהרנ"א המקודש מועשר מאוד עבור רנ"א שהיו מקושרים באופן קוולנטי לחלבון המעניין. עם זאת, להסתמכות על תגי זיקה יש חסרונות. לדוגמה, התג עלול להפריע לתפקוד החלבון, מה שעלול לתת קריאה מעוותת של האינטראקטום קושר הרנ"א שלו. בנוסף, עבור חלק מאורגניזמי המודל לא תמיד ניתן להשתמש בתגיות מכיוון שהכלים הגנטיים לשילוב מקטעי דנ"א בגנום או להתמרת פלסמידים של ביטוי עדיין אינם זמינים. עם זאת, קל לשנות חלקים מסוימים של פרוטוקול χCRAC כדי להתאים אותו לפרוטוקולים מבוססי קליפ המסתמכים על נוגדנים לטיהור RBP. ואכן, מחקר זה הראה כי ניתן לשלב טיהורים מבוססי iCLIP עם crosslinker שלנו. אנו נמצאים כעת בתהליך של פיתוח פרוטוקולי CLIP לחקר הקשר הזמני של חלבונים קושרי RNA אנושיים עם תעתיקי RNA מתהווים.
בעת ביצוע χCRAC על חלבון חדש, החשיפה לקרינת UV חייבת להיות אופטימלית על מנת לגרום לקישור צולב מקסימלי. זה חשוב מכיוון שחשיפות UV גבוהות יכולות להפחית את התאוששות ה- RNA במהלך שלב הטיהור. תאים המבטאים RBP רקומביננטי נחשפו למנות UV שונות, 100 mJ/cm 2, 250 mJ/cm 2, 500 mJ/cm 2 ו-1 J/cm 2. לאחר מכן נלכדו ה-RNPs והרנ"א פוצל ותויג ברדיו. לאחר מכן, ה-RNPs נפתרו על ידי SDS-PAGE ונלקחה אוטורדיוגרמה על מנת להסיק איזו חשיפה נתנה את האות החזק ביותר (כלומר ההצלבה המקסימלית).
לאחר אופטימיזציה של תנאי הניסוי, מומלץ לבצע מספר ניסויי בקרה בעת ביצוע χCRAC. ראשית, ניתן להשתמש בדגימה מוקרנת UV ולא מתויגת כדי לפקח על קשירת הרקע לחרוזי הטיהור. שנית, כאשר מיישמים χCRAC במהלך ניסוי משמרת, סדרת זמן שנייה שבה התאים מוסטים חזרה לתווך המקורי מאפשרת לחקור אם סינון התאים עצמו גורם לשינויים ברמות הרנ"א או באינטראקציות חלבון-רנ"א.
כפי שהוזכר במבוא, מאמרים רבים שפורסמו לאחרונה מציעים מספר אופטימיזציות לפרוטוקול CLIP. זה כולל את השימוש במתאמים המסומנים באופן פלואורסצנטי לזיהוי קומפלקס חלבון-רנ"א באמצעות סריקת אינפרא אדום10, כמו גם אופטימיזציות לטיהור חומצות גרעין שונות ושלבי בחירת גודל המוצגים כמגדילים את המורכבות של הספריות המתקבלות 12,45. אנו מיישמים כעת חלק מהשיפורים הללו כדי לחדד עוד יותר את פרוטוקול χCRAC. הפרוטוקול המוצג כאן כבר מכיל מספר שיפורים לפרוטוקולי CRAC ו-χCRAC המקוריים המגדילים את מורכבות הנתונים. לדוגמה, בעבר, לאחר פתרון קומפלקסים צולבים של חלבון-רנ"א רדיואקטיבי על ג'לים SDS-PAGE, הם הועברו לקרום ניטרוצלולוז והרנ"א הצולב, בודד מהכתם. עם זאת, העברת ה-RNP ומיצוי ה-RNA שלאחר מכן יכולה להיות מאוד לא יעילה, במיוחד כאשר מדובר ב-RBP גדולים כגון תת-יחידות RNA פולימראז. זה יכול לגרום לירידה משמעותית בהתאוששות של RNA צולב. בפרוטוקול הנוכחי, הרנ"א הצולב-מקושר מופק ישירות מפרוסות ג'ל SDS-PAGE כפי שמודגם באיור 1. זה הגביר את ההתאוששות של RNA צולב. בנוסף, לאחר הגברה PCR של cDNAs המוצר נפתר במקור על 3%, טמפרטורת התכה נמוכה ג'ל agarose, ולאחר מכן 175-300 bp מוצרי PCR הופקו מן הג'ל. עם זאת, ג'לים אלה יכולים להיות עמוסים בקלות, וכתוצאה מכך הפרדה גרועה מאוד של ה- DNA. החלפת ג'ל אגרוז בג'ל TBE טרומי הביאה להפרדת גודל עקבית יותר ולהתאוששות טובה יותר של מוצרי PCR.
Disclosures
A. Langford ו- W. Worboys קשורים ל- UVO3, חברה מסחרית. לא היה להם כל תפקיד בעיצוב המחקר, באיסוף הנתונים ובפרשנותם, או בהחלטה להגיש את העבודה לפרסום.
Acknowledgements
עבודה זו נתמכה על ידי מענקים מקרן Wellcome (091549 ל- S.G ו- 109093/Z/15/A ל- S.M.), מענק הליבה של Wellcome Trust Centre for Cell Biology (092076) ומלגת מחקר בכירה לא קלינית של המועצה למחקר רפואי (MR/R008205/1 ל- S.G.), הארגון האירופי לביולוגיה מולקולרית תחת מלגת פוסט-דוקטורט ארוכת טווח (ALTF 1070-2017 ל- R.A.C), וקרן המחקר העצמאית דנמרק (T.H.J).
Materials
| Name | Company | Catalog Number | Comments |
| 1,4-dithioreitol | Merck | 10708984001 | Buffer component in mammalian cell lysis |
| 1.5 mL tubes | Eppendorf | 0030 120.086 | General reaction tube |
| 2 mL tubes | Eppendorf | 0030 123.344 | For holding columns and collection of waste |
| 32P-yATP | Perkin Elmer | NEG502Z-250 | For radiolabelling the 5' end of the RNA |
| 4-12% Bis-Tris gel | Invitrogen | NP0321BOX | SDS-PAGE gel |
| 4X loading buffer | Novex | NP0008 | Protein loading dye concentrate |
| 50 bp ladder | New England Biolabs | N3236 | Reference ladder for excising region of interest from the amplified cDNA library |
| 50% PEG | NEB | B100045 | For the L5 linker ligation |
| 6% TBE gel | Invitrogen | EC6265BOX | For separation and purification of the cDNA library |
| Acetone | ACROS Organics | 423245000 | Washing of TCA-precipitated proteins |
| anti-FLAG beads | Sigma Aldrich | M8823-1ML | For purifcation of FLAG-tagged RBPs |
| ATP (100 mM) | Thermo Fisher Scientific | R0441 | For ligation of the L5 linker onto the 5' end of captured RNAs |
| Beta-mercaptoethanol | Sigma Aldrich | M3148-100ML | Buffer component |
| Biomax MS intensifying screen | Sigma Aldrich | Z363162-1EA | For intensifying the autoradiogram signal |
| Chloroform | Thermo Fisher Scientific | 1010219 | For phenol-chloroform extraction following RNA purification |
| cOmplete EDTA-free protease inhibitor cocktail | Roche | 11873580001 | For inhibition of cellular proteases after lysis |
| Complete supplement mixture -TRP | Formedium | DCS0149 | For preparation of synthetic defined medium |
| Costar Spin-X 0.22 µm filters | Sigma Aldrich | CLS8160 | For isolating the excised cDNAs following gel extraction |
| DNase RQ1 | Promega | M6101 | For DNA digest following cell lysis |
| dNTPs (10 mM) | Sigma Aldrich | 4638956001 | For reverse transcription and PCR |
| Ethanol | Thermo Fisher Scientific | 10041814 | For phenol-chloroform extraction following RNA purification and DNA precipitation |
| Ethylenediaminetetraacetic acid | Invitrogen | AM9261 | For protease K buffer |
| Exonuclease I | New England Biolabs | M0293 | For degradation of primers following PCR |
| Glass microfiber filters | Whatman | 1823-010 | For isolating the excised cDNAs following gel extraction |
| Glucose | Formedium | GLU03 | For preparation of glucose-containing, synthetic defined medium |
| Glycogen (20 mg/mL) | Roche | 10901393001 | Precipitation of proteins, RNA and DNA |
| GST-TEV | Homemade | Construct and purification protocol is available upon request | |
| Guanidium hydrochloroide | Thermo Fisher Scientific | 10071503 | Required for pulldown denaturing conditions and washing buffer |
| IgG beads | GE Healthcare | 17-0969-01 | For purification of protein A-tagged RBPs |
| Imidazole | Sigma Aldrich | I2399-100G | For elution of captured proteins from Nickel beads |
| Isoamyl alcohol | Thermo Fisher Scientific | A393-500 | For phenol-chloroform extraction following RNA purification |
| Luna Universal One-Step RT-qPCR | NEB | E3005S | For qPCR of the cDNA in order to calculate required number of PCR cycles |
| Magnesium chloride | Fluka Analytical | 63020-1L | For PNK buffer |
| Membrane filters | Millipore | AAWP09000 for yeast or HAWP09000 for bacteria | For vacuum filtration of cells |
| Micro bio-spin columns | Biorad | 732-6204 | For collecting eluate after gel extraction |
| Ni-NTA beads | Qiagen | 30210 | For secondary protein capture |
| NP-40 | Sigma Aldrich | I8896-100ML | Buffer component |
| Pfu polymerase | Promega | M7741 | For amplification of the cDNA library |
| Phenol | Sigma Aldrich | P4682-400ML | For phenol-chloroform extraction following RNA purification |
| Pierce spin columns | Thermo Fisher Scientific | 69725 | For on-column enzymatic reactions |
| Protease K | Roche | 3115887001 | For degradation of the RBP following gel extraction |
| Quartz Petri dish | UVO3 | N/A | For cross-linking of adherent cells. Available from https://www.vari-x-link.com for 400 GBP |
| Radiography films | Amersham | 28906843 | For autoradiography visualisation |
| RNAClean XP beads | Beckmann | A63987 | SPRI beads for clean up of RNAs and cDNAs |
| RNase H | New England Biolabs | M0297 | For degradation of RNAs following reverse transcription |
| RNase-It | Agilent | 400720 | For RNA digestion |
| rRNasin | Promega | N2511 | For inhibition of any contaminating RNases during enzymatic reaction |
| Sodium acetate | Sigma Aldrich | S2889-1KG | For phenol-chloroform extraction following RNA purification and DNA precipitation |
| Sodium chloride | Thermo Fisher Scientific | 7647-14-5 | Buffer component |
| Sodium deoxycholate | Sigma Aldrich | D6750-100G | Buffer component in mammalian cell lysis |
| Sodium dodecylsulfate | Sigma Aldrich | L3771-1KG | For protease K buffer |
| SUPERase-In | Invitrogen | AM2694 | For inhibition of cellular RNases after lysis |
| SuperScript IV | Thermo Fisher Scientific | 18090010 | For reverse transcription |
| T4 PNK | New England Biolabs | M0201 | For radiolabelling the 5' end of the RNA |
| T4 RNA ligase 1 | New England Biolabs | M0204 | For ligation of the L5 adaptor onto the RNA 5' end |
| T4 RNase ligase 2, truncated K222Q | NEB | M0351S | For ligation of the App_PE linker onto the 3' end of captured RNAs |
| TBE buffer (10X) | Invitrogen | 15581-028 | For running TBE gels |
| TEV protease | Homemade | For eluting captured proteins following FLAG capture | |
| Thermosensitive alkaline phosphatase | Promega | M9910 | For 5' and 3' dephosphorylation of RNAs |
| Trichloroacetic acid (100%) | Sigma Aldrich | T0699-100ML | For precipitation of RBP-RNA complexes |
| Tris hydrochloride | Invitrogen | 15504-020 | Buffer component |
| Triton X-100 | Sigma Aldrich | T8787-100ML | Buffer component in mammalian cell lysis |
| Vari Filter | UVO3 | N/A | Device for vacuum harvesting cells. Available from https://www.vari-x-link.com for 100 GBP |
| Vari-X-Linker | UVO3 | N/A | Cross-linker for cross-linking cells. Available from https://www.vari-x-link.com for 16,000 GBP |
| Yeast nitrogen base | Formedium | CYN0410 | For preparation of synthetic defined medium |
| Zirconia beads | Thistle | 11079105Z for yeast or 11079101Z for bacteria | For cell lysis via bead beating |
References
- Ule, J., et al. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Granneman, S., Kudla, G., Petfalski, E., Tollervey, D. Identification of protein binding sites on U3 snoRNA and pre-rRNA by UV cross-linking and high-throughput analysis of cDNAs. Proceedings of the National Academy of Sciences. 106 (24), 9613-9618 (2009).
- Licatalosi, D. D., et al. HITS-CLIP yields genome-wide insights into brain alternative RNA processing. Nature. 456 (7221), 464-469 (2008).
- König, J., et al. iCLIP reveals the function of hnRNP particles in splicing at individual nucleotide resolution. Nature Structural & Molecular Biology. 17 (7), 909-915 (2010).
- Hafner, M., et al. Transcriptome-wide Identification of RNA-Binding Protein and MicroRNA Target Sites by PAR-CLIP. Cell. 141 (1), 129-141 (2010).
- Aktaş, T., et al. DHX9 suppresses RNA processing defects originating from the Alu invasion of the human genome. Nature. 544 (7648), 115-119 (2017).
- Huppertz, I., et al. iCLIP: Protein–RNA interactions at nucleotide resolution. Methods. 65 (3), 274-287 (2014).
- Li, X., et al. Comprehensive in vivo RNA-binding site analyses reveal a role of Prp8 in spliceosomal assembly. Nucleic Acids Research. 41 (6), 3805-3818 (2013).
- Rosenberg, M., et al. Denaturing CLIP, dCLIP, Pipeline Identifies Discrete RNA Footprints on Chromatin-Associated Proteins and Reveals that CBX7 Targets 3′ UTRs to Regulate mRNA Expression. Cell Systems. 5 (4), 368-385 (2017).
- Zarnegar, B. J., et al. irCLIP platform for efficient characterization of protein–RNA interactions. Nature Methods. 13 (6), 489-492 (2016).
- Kargapolova, Y., Levin, M., Lackner, K., Danckwardt, S. sCLIP—an integrated platform to study RNA–protein interactomes in biomedical research: identification of CSTF2tau in alternative processing of small nuclear RNAs. Nucleic Acids Research. 45 (10), 6074-6086 (2017).
- Van Nostrand, E. L., et al. Robust transcriptome-wide discovery of RNA-binding protein binding sites with enhanced CLIP (eCLIP). Nature Methods. 13 (6), 508-514 (2016).
- Flynn, R. A., et al. Dissecting noncoding and pathogen RNA–protein interactomes. RNA. 21 (1), 135-143 (2015).
- Brugiolo, M., Botti, V., Liu, N., Müller-McNicoll, M., Neugebauer, K. M. Fractionation iCLIP detects persistent SR protein binding to conserved, retained introns in chromatin, nucleoplasm and cytoplasm. Nucleic Acids Research. 45 (18), 10452-10465 (2017).
- Sanford, J. R., et al. Identification of Nuclear and Cytoplasmic mRNA Targets for the Shuttling Protein SF2/ASF. PLOS ONE. 3 (10), e3369 (2008).
- Garzia, A., Meyer, C., Morozov, P., Sajek, M., Tuschl, T. Optimization of PAR-CLIP for transcriptome-wide identification of binding sites of RNA-binding proteins. Methods. 118-119, 24-40 (2017).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Research. 22 (10), 2031-2042 (2012).
- Chen, K., et al. High-Resolution N6-Methyladenosine (m6A) Map Using Photo-Crosslinking-Assisted m6A Sequencing. Angewandte Chemie International Edition. 54 (5), 1587-1590 (2015).
- Ke, S., et al. A majority of m6A residues are in the last exons, allowing the potential for 3′ UTR regulation. Genes & Development. 29 (19), 2037-2053 (2015).
- Linder, B., et al. Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome. Nature Methods. 12 (8), 767-772 (2015).
- Kudla, G., Granneman, S., Hahn, D., Beggs, J. D., Tollervey, D. Cross-linking, ligation, and sequencing of hybrids reveals RNA–RNA interactions in yeast. Proceedings of the National Academy of Sciences. 108 (24), 10010-10015 (2011).
- Sugimoto, Y., et al. hiCLIP reveals the in vivo atlas of mRNA secondary structures recognized by Staufen 1. Nature. 519 (7544), 491-494 (2015).
- Hwang, H. W., et al. cTag-PAPERCLIP Reveals Alternative Polyadenylation Promotes Cell-Type Specific Protein Diversity and Shifts Araf Isoforms with Microglia Activation. Neuron. 95 (6), 1334-1349 (2017).
- Hwang, H. W., et al. PAPERCLIP Identifies MicroRNA Targets and a Role of CstF64/64tau in Promoting Non-canonical poly(A) Site Usage. Cell Reports. 15 (2), 423-435 (2016).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Beckmann, B. M. RNA interactome capture in yeast. Methods. 118-119, 82-92 (2017).
- Granneman, S., Petfalski, E., Tollervey, D. A cluster of ribosome synthesis factors regulate pre-rRNA folding and 5.8S rRNA maturation by the Rat1 exonuclease. The EMBO Journal. 30 (19), 4006-4019 (2011).
- Schaughency, P., Merran, J., Corden, J. L. Genome-Wide Mapping of Yeast RNA Polymerase II Termination. PLOS Genetics. 10 (10), e1004632 (2014).
- Bernstein, J. A., Khodursky, A. B., Lin, P. H., Lin-Chao, S., Cohen, S. N. Global analysis of mRNA decay and abundance in Escherichia coli at single-gene resolution using two-color fluorescent DNA microarrays. Proceedings of the National Academy of Sciences. 99 (15), 9697-9702 (2002).
- Kresnowati, M. T. A. P., et al. When transcriptome meets metabolome: fast cellular responses of yeast to sudden relief of glucose limitation. Molecular Systems Biology. 2, 49 (2006).
- Marguerat, S., Lawler, K., Brazma, A., Bähler, J. Contributions of transcription and mRNA decay to gene expression dynamics of fission yeast in response to oxidative stress. RNA Biology. 11 (6), 702-714 (2014).
- van Nues, R., et al. Kinetic CRAC uncovers a role for Nab3 in determining gene expression profiles during stress. Nature Communications. 8 (1), 12 (2017).
- Selinger, D. W., Saxena, R. M., Cheung, K. J., Church, G. M., Rosenow, C. Global RNA Half-Life Analysis in Escherichia coli Reveals Positional Patterns of Transcript Degradation. Genome Research. 13 (2), 216-223 (2003).
- Tudek, A., Candelli, T., Libri, D. Non-coding transcription by RNA polymerase II in yeast: Hasard or nécessité?. Biochimie. 117, 28-36 (2015).
- Lingaraju, M., et al. The MTR4 helicase recruits nuclear adaptors of the human RNA exosome using distinct arch-interacting motifs. Nature Communications. 10 (1), 1-11 (2019).
- Lubas, M., et al. Interaction Profiling Identifies the Human Nuclear Exosome Targeting Complex. Molecular Cell. 43 (4), 624-637 (2011).
- Conrad, N. K., et al. A yeast heterogeneous nuclear ribonucleoprotein complex associated with RNA polymerase II. Genetics. 154 (2), 557-571 (2000).
- Darby, M. M., Serebreni, L., Pan, X., Boeke, J. D., Corden, J. L. The Saccharomyces cerevisiae Nrd1-Nab3 Transcription Termination Pathway Acts in Opposition to Ras Signaling and Mediates Response to Nutrient Depletion. Molecular and Cellular Biology. 32 (10), 1762-1775 (2012).
- Webb, S., Hector, R. D., Kudla, G., Granneman, S. PAR-CLIP data indicate that Nrd1-Nab3-dependent transcription termination regulates expression of hundreds of protein coding genes in yeast. Genome Biology. 15 (1), R8 (2014).
- Jensen, T. H., Jacquier, A., Libri, D. Dealing with Pervasive Transcription. Molecular Cell. 52 (4), 473-484 (2013).
- van Dijk, E. L., et al. XUTs are a class of Xrn1-sensitive antisense regulatory non-coding RNA in yeast. Nature. 475 (7354), 114-117 (2011).
- Thiebaut, M., et al. Futile Cycle of Transcription Initiation and Termination Modulates the Response to Nucleotide Shortage in S. cerevisiae. Molecular Cell. 31 (5), 671-682 (2008).
- Merran, J., Corden, J. L. Yeast RNA-Binding Protein Nab3 Regulates Genes Involved in Nitrogen Metabolism. Molecular and Cellular Biology. 37 (18), e00154-e00117 (2017).
- Bresson, S., Tuck, A., Staneva, D., Tollervey, D. Nuclear RNA Decay Pathways Aid Rapid Remodeling of Gene Expression in Yeast. Molecular Cell. 65 (5), 787-800 (2017).
- Buchbender, A., et al. Improved library preparation with the new iCLIP2 protocol. Methods. , (2019).
Erratum
Formal Correction: Erratum: Monitoring Protein-RNA Interaction Dynamics in vivo at High Temporal Resolution using χCRAC
Posted by JoVE Editors on 2/17/2023. Citeable Link.
An erratum was issued for: Monitoring Protein-RNA Interaction Dynamics in vivo at High Temporal Resolution using χCRAC. The Authors section was updated from:
Stuart W. McKellar1
Ivayla Ivanova1
Robert W. van Nues2
Ross A. Cordiner3
Will Worboys4
Andrew Langford4
Torben Heick Jensen3
Sander Granneman1
1Centre for Synthetic and Systems Biology, University of Edinburgh
2Institute of Cell Biology, University of Edinburgh
3Department of Molecular Biology and Genetics, Aarhus University, 4UVO3 Ltd.
to:
Stuart W. McKellar1
Ivayla Ivanova1
Robert W. van Nues2
Ross A. Cordiner3
Mehak Chauhan1
Niki Christopoulou1
Will Worboys4
Andrew Langford4
Torben Heick Jensen3
Sander Granneman1
1Centre for Engineering Biology, University of Edinburgh
2Institute of Cell Biology, University of Edinburgh
3Department of Molecular Biology and Genetics, Aarhus University, 4UVO3 Ltd.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved