Method Article
Мониторинг динамики взаимодействия белок-РНК in vivo при высоком временном разрешении с использованием χCRAC
В этой статье
Erratum Notice
Резюме
Кинетическое сшивание и анализ кДНК – метод, позволяющий исследовать динамику белок-РНК-взаимодействий в живых клетках с высоким временным разрешением. Здесь подробно описан протокол, включая рост дрожжевых клеток, УФ-сшивание, сбор урожая, очистку белка и этапы подготовки библиотеки секвенирования следующего поколения.
Аннотация
Взаимодействие между РНК-связывающими белками (RBP) и их РНК-субстратами проявляет текучесть и сложность. В течение своей жизни одна РНК может быть связана множеством различных RBP, которые будут регулировать ее производство, стабильность, активность и деградацию. Таким образом, многое было сделано для понимания динамики, существующей между этими двумя типами молекул. Особенно важным прорывом стало появление «cross-l inking and immunoprecipitation» (CLIP). Этот метод позволил тщательно исследовать, какие РНК связаны с конкретным RBP. Короче говоря, интересующий белок представляет собой УФ-сшитый с его субстратами РНК in vivo, очищенный в очень строгих условиях, а затем ковалентно сшитые с белком РНК превращаются в библиотеки кДНК и секвенируются. С момента его создания было разработано множество производных методов, чтобы сделать CLIP пригодным для определенных областей обучения. Однако сшивание с использованием ультрафиолетового света, как известно, неэффективно. Это приводит к увеличению времени воздействия, что делает невозможным временное исследование взаимодействий RBP-РНК. Чтобы решить эту проблему, мы недавно разработали и построили значительно улучшенные устройства для ультрафиолетового облучения и сбора клеток. Используя эти новые инструменты, мы разработали протокол для анализа взаимодействий RBP-РНК с временным разрешением в живых клетках с высоким временным разрешением: кинетическое связывание CRoss и анализ A c-ДНК(χCRAC). Недавно мы использовали этот метод для изучения роли дрожжей RBP в адаптации к стрессу питательных веществ. В этой рукописи представлен подробный обзор метода χCRAC и последние результаты, полученные с помощью Nrd1 RBP.
Введение
РНК часто полагаются на RBP для выполнения своей функции, что привело к большому интересу к пониманию динамики между этими молекулами. Многие RBP были идентифицированы в самых разных организмах. Тем не менее, всегда было трудно изучать взаимодействия RBP-РНК in vivo. Крупный прорыв в изучении таких взаимодействий произошел с появлением CLIP1. Этот метод использует ультрафиолетовое (УФ, 254 нм) облучение для индуцирования ковалентных связей между RBP и их непосредственно связанными РНК (т.е. сшивание на нулевом расстоянии). Впоследствии, интересующий RBP иммуноочищают в строгих условиях, чтобы гарантировать, что идентифицируются только РНК, ковалентно сшитые с белками. Затем связанные РНК частично расщепляются с помощью РНКаз и впоследствии превращаются в библиотеки кДНК для секвенирования. Высокая строгость очистки важна, поскольку она значительно увеличивает специфичность восстановления белка и РНК, которая также дополнительно усиливается за счет очистки SDS-PAGE сшитого комплекса рибонуклеопротеина (RNP). CLIP и связанные с ним методы также обеспечивают понимание нуклеотидного разрешения сайта связывания белка, поскольку во время подготовки библиотеки секвенирования аминокислоты, которые сшиты с РНК, часто прекращают обратную транскриптазу или заставляют фермент вводить мутации в этом сайте 1,2,3.
С момента своего появления оригинальный протокол CLIP породил замечательное разнообразие производных методологий. Особенно важным прорывом стала разработка HITS-CLIP (или CLIP-seq), который объединяет высокопроизводительное секвенирование с подходомCLIP 3. С тех пор это было принято во всех методологиях, основанных на CLIP. В iCLIP были усовершенствованы методы тримминга, опосредованного РНКазой, и лигирования адапторов, которые облегчают более точное картирование сайтов связывания RBP4. PAR-CLIP объединил маркировку 4тио-уридина/урацила с кросслинкингом на длине волны 365 нм, что позволило картировать сайты кросс-линкинга путем анализа T-Cзамен 5. CRAC, мочевина-iCLIP, dCLIP и uvCLAP ввели условия денатурации и этапы очистки с двойным сродством, которые еще больше снижают фоновое связывание с аффинной смолой и еще больше повышают специфичность захвата белка 2,6,7,8,9. Кроме того, CRAC, uvCLAP и dCLIP ввели маркировку интересующего RBP аффинной меткой, тем самым преодолев необходимость генерации специфических антител.
Кроме того, был внесен ряд оптимизаций для ускорения внедрения методологии CLIP. Оригинальный протокол CLIP использовал радиоактивное мечение захваченных РНК для визуализации комплексов RBP-РНК после SDS-PAGE. Однако использование радиоактивности может быть проблематичным для лабораторий, не созданных для такой работы. irCLIP включает в себя адаптер с флуорофорной связью, который облегчает визуализацию с помощью инфракрасной визуализации10 , а sCLIP использует биотинилирование захваченных РНК для их визуализации с помощью стрептавидин-конъюгированного HRP11. Кроме того, eCLIP полностью отказывается от маркировки РНК; вместо этого белок иссекается исключительно на основе его известного размера12. Очистка на основе стрептавидина также использовалась для ускорения процесса подготовки библиотеки в FAST-iCLIP, где биотинилированный 3'-адаптер лигируется с РНК и используется для обеспечения очистки после обратной транскрипции и циркуляции13. Дополнительные усовершенствования протокола iCLIP также значительно увеличили сложность библиотек4.
Наконец, CLIP был модифицирован для обеспечения захвата RBP из различных клеточных субкомпартментов 14,15, для визуализации вновь транскрибируемых РНК с использованием импульсной индукции фотоактивируемых рибонуклеозидов 5,16,17, для захвата метилированных РНК18,19,20, для изучения РНК-РНК-взаимодействий 21,22 и для картирования 3' концов 23,24.
Несмотря на большой вклад методов, основанных на CLIP, в помощь нашему пониманию взаимодействий между RBP и РНК, он был ограничен неэффективностью УФ-сшивания. Хотя культуральные клетки, выращенные в монослое, как правило, относительно легко сшиваются, это значительно сложнее в тканях или клетках в растворе. Тканям может потребоваться несколько раундов воздействия ультрафиолета для проникновения в требуемые клеточные слои, в то время как микробные клетки часто выращиваются в богатых средах, содержащих ароматические, поглощающие ультрафиолетовое излучение соединения25. Действительно, время ультрафиолетового облучения до 30 минут использовалось для создания достаточного сшивания между RBP и связанными с ними РНК для таких образцов26,27,28. Это расширенное воздействие ультрафиолета вызывает стрессовые реакции внутри клетки, такие как УФ-индуцированное повреждение ДНК, которое может загрязнить конечные данные в некоторых приложениях.
Большинство исследований CLIP были сосредоточены на создании единичных «снимков» специфических взаимодействий белка и РНК в клетке. Однако белок-РНК-взаимодействия по своей природе динамичны, особенно когда клетки подвержены изменениям в окружающей среде. Это может включать внезапное снижение доступности необходимых питательных веществ или быстрые изменения температуры. Таким образом, чтобы по-настоящему понять роль RBP во время стресса, лучше всего провести анализ с временным разрешением, потому что он может охватить весь спектр целей RBP во время стресса и определить, на какой стадии реакции на стресс активен выбранный RBP. В частности, исследования на дрожжах показали, что первые несколько минут адаптации абсолютно необходимы для выживания, а период полураспада РНК у бактерий может варьироваться от минут до секунд 29,30,31,32,33. Поэтому такие анализы с временным разрешением в идеале должны выполняться с высоким временным разрешением. Однако длительное время сшивания делает изучение адаптивных реакций на ранних стадиях особенно сложным.
Чтобы преодолеть эти проблемы, мы недавно разработали улучшенный метод, который способен сшивать и собирать клетки в течение нескольких минут. Наш метод χCRAC позволяет количественно измерять динамические изменения во взаимодействиях RBP-РНК при ранее ненаблюдаемом разрешении. Решающее значение для этого метода имела разработка нового устройства32 УФ-облучения, которое сокращает требуемое время сшивания у дрожжей и бактерий в растворе примерно в 10 раз, эффективно замораживая взаимодействия RBP-РНК мгновенно. Кроме того, чтобы быстро собирать клетки после УФ-облучения, мы разработали устройство вакуумной фильтрации, которое может собирать экспоненциально растущие дрожжи в культуре объемом 0,5 л примерно за 30 с32. Эти технологические инновации позволяют изучать динамику RBP-РНК с поминутным разрешением. Кроме того, мы также внесли несколько оптимизаций в исходный протокол CRAC2 , чтобы повысить его практичность.
Используя χCRAC, мы недавно изучили мишень дрожжевого ядерного RBP, Nab3, в ответ на депривацию глюкозы. В Saccharomyces cerevisiae Nab3 может образовывать комплекс с Nrd1, RBP и геликазой РНК Sen1 с образованием комплекса NNS. Связывание NNS с РНК-полимеразой и зарождающимся транскриптом может вызвать терминацию транскрипции34. Этот комплекс в основном участвует в удалении загадочных некодирующих транскриптов РНК, но также было показано, что он регулирует экспрессию генов, кодирующих белок. Исследование показало дифференциальное нацеливание Nab3 на некодирующие и кодирующие транскрипты после всего лишь минуты стресса32. Мы продемонстрировали, что котранскрипционное прекращение Nab3 приводит к очень преходящей, импульсной экспрессии генов ретротранспозона, которую было бы трудно обнаружить с помощью традиционных подходов, основанных на CLIP. Кроме того, короткое время УФ-облучения в нашем УФ-сшивающем агенте также значительно увеличило восстановление короткоживущих некодирующих РНК32. χCRAC, вероятно, станет важнейшим инструментом для выяснения не только того, как ОДП формируют реакцию на стресс в непосредственных временных рамках, но и их меняющихся ролей в течение всего жизненного цикла ответных мер. В этой рукописи представлен подробный обзор всех этапов протокола χCRAC. В иллюстративных целях метод был использован для изучения белка дрожжей Nrd1, который участвует в терминации транскрипции и распаде РНК35,36, и его РНК-мишени в ответ на депривацию глюкозы во множестве временных точек. Наконец, мы также демонстрируем, что наш блок УФ-облучения может быстро сшивать RBP с РНК в клетках HeLa, что позволяет также выполнять анализы с высоким разрешением по времени в адгезивных клетках.
протокол
| ТН150 |
| 50 мМ Tris pH 7,8 |
| 150 мМ NaCl |
| 0,1% НП-40 |
| 1X ингибитор протеазы |
| ТН1000 |
| 50 мМ Tris pH 7,8 |
| 1М NaCl |
| 0,1% НП-40 |
| НП-ПНК |
| 50 мМ Tris-HCl pH 7,8 |
| 10 мМ MgCl2 |
| 0,1% НП-40 |
| 5 мМ бета-меркаптоэтанол |
| 5 х PNK |
| 250 мМ Tris-HCl pH 7,8 |
| 50 мМ MgCl2 |
| 50 мМ бета-меркаптоэтанол |
| ВБ I |
| 50 мМ Tris-HCl pH 7,8 |
| 300 мМ NaCl |
| 10 мМ имидазол |
| 6М гуанидин-HCl |
| 0,1% НП-40 |
| 5 мМ бета-меркаптоэтанол |
| ВБ II |
| 50 мМ Tris-HCl pH 7,8 |
| 50 мМ NaCl |
| 10 мМ имидазол |
| 0,1% НП-40 |
| 5 мМ бета-меркаптоэтанол |
| Буфер для элюирования |
| 50 мМ Tris pH 7,8 |
| 50 мМ NaCl |
| 250 мМ имидазол |
| 0,1% НП-40 |
| 5 мМ бета-меркаптоэтанол |
| Буфер протеазы К |
| 50 мМ Трис |
| 0,1% НП-40 |
| 5 мМ β-меркаптоэтанол |
| 1% SDS |
| 5 мМ ЭДТА |
| 50 мМ NaCl2 |
| Буфер лизиса млекопитающих |
| 50 мМ Tris-HCl pH 8 |
| 100 мМ NaCl |
| 0,5% об./об. Triton X-100 |
| 0,25% мас./об. Na-дезоксихолат |
| 0,1% мас./об. SDS |
| 5 мМ ЭДТА |
| 1 мМ DTT (добавлен свежий) |
| 1X ингибитор протеазы |
Таблица 1: Буферы, необходимые для χCRAC, и их составы.
1. УФ-сшивание и производство лизата
- Микроорганизмы в растворе
- Прививают 3,5 л желаемой среды дрожжами из ночной культуры до исходного OD600 0,05. Выращивание при 30 °C при непрерывном встряхивании при 180 об/мин.
- Во время роста подготовьте другие необходимые материалы.
- Подготовьте емкость с жидким азотом.
- Приготовьте 3 л среды, вызывающей стресс, и нагрейте до 30 °C на водяной бане.
- Настройте фильтрующий аппарат, включите сшивающий агент (рис. 2A) и наклейте этикетки на конические трубки объемом 50 мл, по одной для каждого момента времени.
- Как только клетки достигнут желаемого OD 600, вылейте500 мл клеток прямо в сшивающий агент и облучайте УФ-излучением 250 мДж 254 нм УФ. Подробнее об использовании сшивающего агента см. на рисунках 2A и 3A .
ПРИМЕЧАНИЕ: Энергия ультрафиолетового излучения должна быть тщательно оптимизирована для каждого интересующего белка. Более подробную информацию см. в обсуждении. - После сшивания отфильтруйте ячейки с помощью одного из вакуумных фильтрующих устройств (рис. 2B, C). Сверните мембрану с отфильтрованными клетками, поместите в коническую пробирку t = 0 (нулевое время) объемом 50 мл и заморозьте в жидком азоте.
- Отфильтруйте оставшиеся ячейки по шести различным фильтрам. Ресуспендируют собранные клетки в 3 л предварительно нагретой среды, вызывающей стресс, опуская мембраны в среду и энергично перемешивая с полоской в течение 50 с. По прошествии 50 с подготовьтесь к взятию пробы t = 1.
- Через 1 минуту сшивают 500 мл клеток и собирают через фильтрацию, как показано на этапах 1.1.3–1.1.4. Повторите через 2, 4, 8, 14 и 20 минут или в другие моменты времени по мере необходимости.
- Храните конические трубки, содержащие ячейки, при температуре -80 °C. Установите фосфатный буферный физиологический раствор (PBS) на 4 °C на ночь.
- На следующий день возьмите каждую коническую пробирку, содержащую сшитый образец, и ресуспендируйте клетки в 25 мл холодного PBS, энергично встряхивая.
- Перенесите клеточные суспензии в новые конические трубки и отжим при 4 600 x g, 5 мин при 4 ° C.
- Слейте PBS, быстро открутите еще раз, чтобы собрать остатки PBS, а затем сцедите оставшуюся жидкость пипеткой.
- Рассчитайте вес гранулы в пробирке, сравнив ее с пустой пробиркой.
- Добавьте два объема гранул ледяного TN150, 60 мкл ДНКазы 1 и 10 мкл ингибитора РНКазы. Инкубировать на льду 30 мин.
- Например, на 400 мг клеток добавьте 800 мкл ледяного TN150.
- Добавление ДНКазы не является существенным для большинства растворимых белков, но очень важно при изучении связанных с хроматином белков, таких как РНК-полимераза. Кроме того, он снижает вязкость бактериальных лизатов. Очень важно использовать ровно два объема гранул лизисного буфера, иначе эффективность лизиса может снизиться.
- Добавьте три объема гранул (в мл) гранул диоксида циркония в клеточную суспензию. Для дрожжей используйте шарики диаметром 0,5 мм, а для бактерий - 0,1 мм.
- Например, для 400 мг клеток отмерьте 1,2 мл шариков диоксида циркония в пробирке объемом 1,5 мл и добавьте их к клеткам, ресуспендированным в буфере лизиса.
- Перемешивайте клеточные суспензии в течение 1 мин, затем поместите на лед на 1 мин. Повторите в общей сложности 5 раз.
- Добавьте два объема гранул буфера TN150 и энергично перемешайте.
- Центрифугу суспензию в конической трубке по 4 600 г в течение 20 мин при 4 °С в настольной центрифуге.
- После центрифугирования возьмите образец надосадочной жидкости объемом 50 мкл для будущего анализа вестерн-блоттинга, чтобы изучить экспрессию белка всей клетки.
- Перенесите надосадочные жидкости в пробирки объемом 1,5 мл и вращайте лизат в течение 20 мин при 20 000 x g при 4 ° C в микрофуге.
- В качестве альтернативы, при использовании пробирок объемом 5 мл, центрифугу при 13 000 x g в течение 20 мин.
- После центрифугирования возьмите образец надосадочной жидкости объемом 50 мкл для будущего анализа вестерн-блоттинга, чтобы изучить растворимую экспрессию белка.
- Перейдите к захвату RBP (раздел 2).
- Культивируемые адгезивные клетки
- Засейте достаточное количество адгезивных клеток в чашку Петри за 24 часа до УФ-сшивания, чтобы они могли достичь 80% слияния на следующий день. Выращивают в течение ночи в желаемой среде в инкубаторе клеточных культур при 37 °C, 5% CO2.
ПРИМЕЧАНИЕ: При использовании кварцевых чашек Петри полезно стимулировать клеточную адгезию путем обработки культуральной посуды поли-D-лизином (70 000–140 000 мас.) и фетальной телячьей сывороткой (FCS) за 2,5 часа до посева. Добавьте достаточное количество поли-D-лизина, чтобы покрыть всю поверхность роста, и инкубируйте при комнатной температуре (RT) в течение 5 минут. Далее кварцевую чашку Петри следует тщательно промыть водой и высушить в инкубаторе клеточных культур в течение 2 ч или до полного высыхания. После этого добавьте достаточное количество FCS, чтобы полностью покрыть поверхность роста, и поместите в инкубатор не менее чем на 30 минут. FCS должна быть полностью удалена перед посевом клеток. - Как только клетки достигнут 80% слияния, удалите носитель и промойте 15 мл ледяного PBS. Далее полностью удалите всю оставшуюся жидкость и приступайте сразу к следующему шагу.
- Перенесите чашку Петри в лоток для адгезивных клеток (рис. 3B) и облучайте УФ-излучением 300 мДж 254 нм УФ. Подробнее об использовании сшивающего агента см. на рисунках 2A и 3B .
ПРИМЕЧАНИЕ: Энергия ультрафиолетового излучения должна быть тщательно оптимизирована для каждого интересующего белка. Более подробную информацию см. в обсуждении. - Сразу после сшивания поместите чашку Петри на лед и добавьте 10 мл ледяного PBS. Соберите клетки путем соскабливания и перенесите в коническую пробирку объемом 15 мл. Гранулы подвергают центрифугированию при 300 x g в течение 5 мин при 4 °C.
- Удалите PBS и ресуспендируйте гранулу клетки в 1 мл ледяного PBS и перенесите в микроцентрифужную пробирку объемом 1,5 мл. Грануляторы снова центрифугируют в течение 5 мин при 300 x g при 4 °C.
- Снимите PBS и заморозьте гранулы ячейки на сухом льду. Храните гранулы ячейки при температуре -80 °C до тех пор, пока они не потребуются.
- Повторите шаги 1.2.3–1.2.6 для каждого момента времени.
- Ресуспендируют клеточные гранулы в 1 мл лизисного буфера и переносят в коническую пробирку объемом 15 мл. После этого добавьте 1 мл буфера для лизиса, в общей сложности 2 мл.
- Добавьте 5 мкл ингибитора РНКазы млекопитающих.
- Ультразвук 5x в течение 10 с на льду при 10 амперах. Подождите 30 с между раундами обработки ультразвуком.
- Рассчитайте концентрацию белка в каждом образце и нормализуйте до самой низкой концентрации.
- Перенесите 1,98 мл лизата в пробирку объемом 2 мл.
- Добавьте 10 мкл ДНКазы I и инкубируйте при 37 ° C в течение 5 минут при встряхивании при 1 200 об/мин.
- Центрифугируют лизат при 16 000 x g в течение 20 мин при 4 °C.
- После центрифугирования возьмите образец надосадочной жидкости объемом 50 мкл для будущего анализа вестерн-блоттинга, чтобы изучить растворимую экспрессию белка.
- Перейдите к захвату RBP (раздел 2).
- Засейте достаточное количество адгезивных клеток в чашку Петри за 24 часа до УФ-сшивания, чтобы они могли достичь 80% слияния на следующий день. Выращивают в течение ночи в желаемой среде в инкубаторе клеточных культур при 37 °C, 5% CO2.
2. Захват RBP
- Промойте магнитные шарики анти-FLAG (75 мкл суспензии на образец) или IgG agarose (500 мкл суспензии на образец) 3 раза 5 мл TN150. Ресуспендировать в конечном объеме 700 мкл TN150 и добавить 100 мкл промытых шариков в семь конических пробирок по 15 мл.
- Хранить на льду до тех пор, пока они не потребуются.
- После того, как лизаты будут осветлены, добавьте надосадочную жидкость в пробирку, содержащую шарики анти-FLAG/IgG.
- Нутат при 4 °C в течение 2 ч.
ПРИМЕЧАНИЕ: Некоторые протоколы описывают ночную инкубацию с шариками, но это не рекомендуется, потому что длительное время инкубации может значительно снизить восстановление сшитых РНК.
3. Промывка бусин и расщепление ТЭВ бирок
- Соберите бусины и удалите лизат.
- Возьмите 50 мкл образца надосадочной жидкости для будущего анализа вестерн-блоттинга, чтобы исследовать незахваченный белок.
- Ресуспендируйте шарики в ледяной TN1000 и переложите в пробирку объемом 1,5 мл. Мыть 10 мин, 4 °C, с нутацией. Повторите в общей сложности три стирки.
- Если вы используете шарики агарозы IgG, промойте их 5 мл TN1000. Если вы используете шарики против флага, используйте 2 мл.
- Далее простираем бусины 3х с помощью TN150, с тем же объемом, что и выше.
- После третьей стирки ресуспендируйте шарики в 600 мкл TN150.
- Добавьте 30 U самодельной протеазы GST-TEV в суспензию шарика и вращайте в течение 2 ч при RT.
ПРИМЕЧАНИЕ: Рекомбинантная протеаза GST-TEV теперь также коммерчески доступна, но она не тестировалась с этим протоколом.- Во время разложения подготовьтесь к следующим шагам, установив колонки по три пробирки по 1,5 мл для каждого образца (т.е. для семи образцов сделайте три ряда по семь столбцов).
- В последний ряд пробирок добавляют 0,4 г гидрохлорида гуанидия, 27 мкл 5М хлорида натрия и 3 мкл 2,5 М имидазола (рН = 8). Учтите, что рН имидазола должен быть 8. Это имеет решающее значение для поддержания целостности РНК.
- Дополнительно промойте необходимый объем никелевых шариков в WB I 3x. Используйте 100 мкл суспензии на образец. После окончательной стирки ресуспендируйте шарики в том же первоначальном объеме WB I и храните на льду.
- После завершения сбраживания TEV соберите надосадочную жидкость с помощью магнитной стойки для шариков против FLAG или центрифугирования для шариков IgG и перенесите в первый ряд ранее установленных пробирок.
- Возьмите 50 мкл образца элюата ТЭВ для анализа вестерн-блоттинга.
- Установите термоблочный инкубатор на 37 °C. Ко второму ряду пробирок добавляют 1 мкл коктейля РНКазы (разведение 1:50).
- Возьмите 550 мкл элюата ТЭВ из первого ряда пробирок и добавьте во второй ряд (содержащий коктейль РНКазы). Энергично используйте пипетку, чтобы обеспечить перемешивание.
- Выполнив это для первого образца, сразу же поместите пробирку в термоблок и запустите таймер. Переходите к последующим образцам, чтобы каждый из них располагался в шахматном порядке.
- Выдерживать ровно 5 мин. После завершения извлеките первый образец из термоблока и перенесите раствор в третий ряд пробирок (содержащих порошок гидрохлорида гуанидия).
ПРИМЕЧАНИЕ: 5-минутная инкубация с разведением коктейля РНКазы в соотношении 1:50 обычно подходит для большинства белков, но этот шаг необходимо тщательно оптимизировать с различным временем инкубации или концентрациями для каждого белка, чтобы убедиться, что сшитые РНК имеют правильный размер (30–100 нт). - Немедленно покрутите пару секунд на полной скорости, чтобы растворить порошок гуанидия, а затем переходите к следующему образцу.
- После того, как все образцы были перенесены в порошок гуанидия, снова перемешайте, чтобы убедиться, что весь порошок полностью растворился.
- Добавьте 100 мкл промытых никелевых шариков и вращайте в течение ночи при 4 ° C. Эту инкубацию можно сократить до 2 ч.
4. Обработка щелочной фосфатазой на шариках
- Установите термоблок на 37 °C.
- Поместите спиновую колонку очистки в пробирку объемом 2 мл, по одной для каждого образца. Перенесите никелевые шарики в колонны и дайте надосадочной жидкости стечь. После этого убедитесь, что все никелевые шарики были удалены из пробирки объемом 1,5 мл, промыв WB I и приложив к колонке.
- Установите 2 пробирки объемом 2 мл, по шесть на образец (по одной для сбора при каждой промывке). Держите колонны снаружи сухими, чтобы поддерживать поток. Промойте шарики 3 раза 500 мкл WB I, а затем 3 раза 500 мкл NP-PNK.
- Закройте крышку вращающейся колонны и ненадолго покрутите шарики, чтобы удалить лишний буфер.
- Наденьте пробку на колонку, поместите колонки в пробирки объемом 1,5 мл и добавьте 60 мкл реакционной смеси, показанной в таблице 2.
| Компонент | 1x | В 7,5 раза |
| 5 буферов PNK | 12 | 90 |
| Щелочная фосфатаза | 4 | 30 |
| Ингибитор РНКазы | 2 | 15 |
| H2O | 42 | 315 |
| Заключительный том | 60 мкл | 450 мкл |
Таблица 2: Реакционная смесь щелочной фосфатазы.
- Инкубируйте шарики в течение 1 часа при 37 °C.
- Промойте шарики 1 раз 500 мкл WB I для инактивации щелочной фосфатазы, а затем 3 раза 500 мкл буфера NP-PNK. Обязательно тщательно промойте внутреннюю часть колонки буфером NP-PNK, чтобы удалить следы гуанидия.
5. Лигирование линкера App-PE на шарике к 3'-концу РНК
- Открутите оставшийся буфер и добавьте 60 мкл смеси, указанной в таблице 3 (см. Таблицу 4 для последовательности App-PE) в столбцы. Инкубировать реакцию в течение 6 ч при 25 °C.
| Компонент | 1x | В 7,5 раза |
| 5 буферов PNK | 12 | 90 |
| Адаптер App-PE (100 мкМ) | 0.6 | 4.5 |
| Т4 РНК-лигаза 2 усеченная K227Q | 3 | 22.5 |
| Ингибитор РНКазы | 1.5 | 11.25 |
| 50% ПЭГ 8000 | 12 | 90 |
| H2O | 30.9 | 231.75 |
| Заключительный том | 60 мкл | 450 мкл |
Таблица 3: Реакционная смесь линкера App-PE.
| Название олигонуклеотида | Последовательность (5'-3') | |||
| Л5Аа | invddT-ACACrGrArCrGrCrCrUrCrUrUrCrCrArUrCrUrNrNrNrNrUrArArGrN-OH | |||
| Л5Аб | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrArUrCrUrNrNrNrRArUrUrArGrCrN-OH | |||
| L5AC | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrArUrCrUrNrNrNrGrGrCrGrGrCrGrCrN-OH | |||
| Л5Ад | invddT-ACACrGrArCrGrCrCrUrCrUrUrCrCrGrArUrCrUrNrNrNrNrCrGrCrUrUrArGrCrN-OH | |||
| Л5Ба | invddT-ACACrGrArCrGrCrUrCrUrUrCrCrArUrCrUrNrNrNrArGrNrN-OH | |||
| Л5Бб | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrGrArUrCrUrNrNrNrGrGrUrGrArGrN-OH | |||
| L5Bc | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrCrАрUrCrUrNrNrNrNrCrArCrCrUrArGrCrN-OH | |||
| L5Bd | invddT-ACACrGrArCrGrCrUrCrUrCrCrCrGrArUrCrUrNrNrNrNrUrCrUrCrUrCrUrArCrN-OH | |||
| L5Ca | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrGrArUrCrUrNrNrNrNrCrCrUrArGrN-OH | |||
| L5Cb | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrGrArUrCrUrNrNrNrNrUrGrGrArGrN-OH | |||
| L5Cc | invddT-ACACrGrArCrGrCrCrUrCrUrCrCrCrArUrCrUrNrNrNrArCrUrCrArGrCrN-OH | |||
| L5Cd | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrGrArUrCrUrNrNrNrGrArCrUrUrArGrCrN-OH | |||
| Л5Да | invddT-ACACrGrArCrGrCrUrCrUrCrCrCrGrArUrCrUrNrNrNrNrCrGrGrUrGrArUrN-OH | |||
| Л5ДБ | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrCrArUrCrUrNrNrNrGrCrArCrUrArN-OH | |||
| L5Dc | invddT-ACACrGrArCrGrCrCrUrCrUrUrCrCrArUrCrUrNrNrNrNrUrArGrUrGrCrN-OH | |||
| Л5Дд | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrGrArUrCrUrUrNrNrNrArUrCrArCrGrN-OH | |||
| L5Ea | invddT-ACACrGrArCrGrCrUrCrUrCrCrCrGrArUrCrUrNrNrNrNrCrArCrUrGrUrN-OH | |||
| Л5Эб | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrGrArUrCrUrNrNrNrGrUrGrArCrArN-OH | |||
| L5Ec | invddT-ACACrGrArCrGrCrUrCrUrCrUrCrCrGrArUrCrUrNrNrNrNrUrGrUrUrCrArCrN-OH | |||
| L5Ed | invddT-ACACrGrArCrGrCrUrCrUrUrCrCrArUrCrUrNrNrNrArCrArGrUrGrN-OH | |||
| App_PE | App-NAGATCGGAAGAGCACACGTCTG-ddC | |||
Таблица 4: Последовательности адаптеров ДНК и РНК, необходимые для лигирования на 5'- и 3'-концах захваченных РНК. Они были очищены с помощью ВЭЖХ без РНКазы.
- Промывайте шарики 1x с 500 мкл WB I и 3x с 500 мкл буфера NP-PNK. Поместите колонку в новую трубку и раскрутите оставшийся буфер.
6. Фосфорилирование 5'-концов РНК на шариках
- Добавьте в колонки 80 мкл смеси, указанной в таблице 5 . Инкубировать реакцию в течение 40 мин при 37 °C.
ПРИМЕЧАНИЕ: Образцы теперь будут очень радиоактивными. Таким образом, все последующие работы должны выполняться за защитным экраном, а отходы должны утилизироваться в соответствии с местными правилами охраны труда и техники безопасности.
| Компонент | 1x | В 7,5 раза |
| 5 буферов PNK | 16 | 120 |
| 32 См.P-ɣATP (10 мкКи/мкл) | 3 | 22.5 |
| Т4 ПНК | 3 | 22.5 |
| H2O | 58 | 435 |
| Заключительный том | 80 мкл | 600 мкл |
Таблица 5: Смесь реакций фосфорилирования.
- Добавьте 1 мкл 100 мМ АТФ и дайте реакции продолжаться еще 20 минут. Это гарантирует, что почти все 5'-концы содержат фосфаты для облегчения лигирования 5'-линкера.
- Установите пробирки по 2 мл, по пять на образец.
- Промывайте шарики 1x с 500 мкл WB I и 3x с 500 мкл буфера NP-PNK. Обратите внимание, что эти элюции будут очень радиоактивными и поэтому должны быть утилизированы надлежащим образом.
- Переместите столбец в последнюю трубку и раскрутите оставшийся буфер.
7. Лигирование по валику 5'-линкера
ПРИМЕЧАНИЕ: 5'-линкеры содержат штрих-код РНК, который используется для идентификации каждого образца после секвенирования. Таким образом, крайне важно отметить, какой компоновщик используется для какой выборки.
- Добавить в колонки 78 мкл смеси, описанной в таблице 6 . Добавьте 2 мкл 5'-адаптера (100 мкМ; см. Таблицу 4) в каждую пробирку и выдерживайте в течение ночи при 18 °C.
| Компонент | 1x | В 7,5 раза |
| 5 буферов PNK | 16 | 120 |
| АТФ (10 мМ) | 8 | 60 |
| Ингибитор РНКазы | 2 | 15 |
| Т4 РНК-лигаза | 4 | 30 |
| H2O | 48 | 360 |
| Заключительный том | 78 мкл | 585 мкл |
Таблица 6: 5' смесь реакции линкерного лигирования.
- На следующий день промойте шарики 1x 500 мкл WB I и 3x 500 мкл WB II и перенесите колонки в новую пробирку объемом 2 мл.
8. Элюция, SDS-PAGE и экстракция РНК
- Установите центрифугу на 4 °C. Подготовьте два ряда пробирок по 1,5 мл на образец для элюирования.
- Раскрутите пустотный объем колонн никелевыми шариками с быстрым вращением. Поместите столбцы в первый ряд элюирующих трубок и добавьте 200 мкл элюирующего буфера. Подождите 2 минуты, затем принудительно пропустите буфер через столбец с помощью быстрого отжима.
- Переместите столбцы во второй ряд трубок и повторите шаг 8.2. Каждый образец теперь будет содержать в общей сложности 400 мкл элюата, разделенного на две пробирки по 1,5 мл.
- Возьмите все элюаты и перенесите их вместе в пробирку объемом 5 мл. Добавьте 2 мкл 20 мг/мл гликогена. Таким образом, при использовании семи образцов в пробирке объемом 5 мл теперь будет 2,8 мл объединенного элюата.
- Добавьте 100 мкл трихлоруксусной кислоты (ТСА) на образец [например, 700 мкл ТСА для 7 образцов (2,8 мл объединенного элюата)] в пробирку объемом 5 мл и вихритите скважину в течение 30 с.
- Инкубировать на льду 20 мин.
- Центрифуга в течение 30 мин при 17 000 x g, 4 °C, в настольной центрифуге.
- Осторожно извлеките надосадочную жидкость из конической трубки, проверив пипетку счетчиком Гейгера, чтобы убедиться, что гранулы не были удалены случайно. Если это так, верните надосадочную жидкость в пробирку и центрифугу еще на 10 минут.
ПРИМЕЧАНИЕ: Надосадочная жидкость все еще может быть очень радиоактивной. Убедитесь, что вы используете правильное экранирование. - Полностью ресуспендируйте гранулу в 2 мл ледяного ацетона.
- Центрифуга в течение 15 мин при 17 000 x g, 4 °C.
- Удалите как можно больше ацетона с помощью пипетки P1000. После этого ненадолго раскрутите трубку, чтобы собрать маленькие капельки ацетона, а затем удалите пипеткой P10. Сушить в течение 2 минут в вытяжном шкафу.
ПРИМЕЧАНИЕ: Надосадочная жидкость ацетона все еще может быть радиоактивной. Убедитесь, что вы используете правильное экранирование. - Ресуспендируют образец в 30 мкл 1x буфера загрузки белка. Чтобы убедиться, что гранула должным образом ресуспендирована, убедитесь, что подавляющее большинство радиоактивности теперь присутствует в загрузочном буфере, а не оставлено в пробирке объемом 1,5 мл, удалив раствор в пипетке P200 и измерив активность, оставшуюся в пробирке объемом 1,5 мл, с помощью счетчика Гейгера.
- Нагрейте образец в течение 10 мин при 65 °C. Загрузите 1 мм, 4–12% сборный гель Bis-Tris и работайте в течение 1,5 ч при напряжении 125 В в буфере MOPS.
- После того, как гель закончит течь, откройте гелевую кассету. Гель должен удерживаться на нижней пластине. Утилизируйте верхушку.
- Заверните гель в пищевую пленку, а затем закрепите его с помощью скотча на внутренней стороне светонепроницаемой кассеты. Убедитесь, что кассета оснащена усилительным экраном для улучшения сигнала.
- Подвергните воздействию геля авторадиографическую пленку и храните кассету при температуре -80 °C во время экспозиции. Время воздействия будет варьироваться между белками с разной эффективностью сшивания.
- При размещении пленки должен быть способ выравнивания ее по кассете, чтобы вырезать интересующую полосу на последующем этапе. Чтобы убедиться в этом, используйте флуоресцентную линейку, а также убедитесь, что гель находится в углу кассеты, который затем покрывается пленкой, также помещенной в крайний угол.
ПРИМЕЧАНИЕ: Как правило, элюаты в загрузочном буфере, которые дают показания не менее ~ 250 cps при отображении на счетчике Гейгера, дают достаточный сигнал для экспозиции в течение 3 часов. В противном случае выполняется ночная выдержка.
- При размещении пленки должен быть способ выравнивания ее по кассете, чтобы вырезать интересующую полосу на последующем этапе. Чтобы убедиться в этом, используйте флуоресцентную линейку, а также убедитесь, что гель находится в углу кассеты, который затем покрывается пленкой, также помещенной в крайний угол.
- Проявите пленку. Срежьте пищевую пленку, покрывающую гель, но не перемещайте гель. В противном случае изображение будет смещено от геля.
ПРИМЕЧАНИЕ: Гель, скорее всего, будет очень радиоактивным. Убедитесь, что вы используете надлежащее экранирование при вырезании кусочка геля. - Поместите пленку поверх геля и иссеките интересующую ленту. Поместите кусочек геля в тюбик объемом 2 мл.
- Разбейте кусочек геля с помощью наконечника пипетки P1000 и добавьте 600 мкл буфера протеиназы К плюс 200 мкг протеиназы К (в этом протоколе используется 10 мкл раствора протеиназы К 20 мг/мл). Инкубировать в течение 2 ч при 55 °C при энергичном встряхивании.
- После этого отрежьте конец наконечника P1000 чистым скальпелем и перенесите кусочки надосадочной жидкости и геля в спиновую колонку, помещенную в пробирку объемом 2 мл.
- Вращайте колонку в течение 1 мин при 17 000 x g при RT. Соберите проточную трубку, которая содержит радиоактивные изолированные РНК.
- Выполните экстракцию фенола: хлороформа.
- Добавьте 50 мкл 3 М ацетата натрия, pH = 5,2, и 500 мкл фенола: хлороформа и встряхните лунку. Отжим в течение 5 минут при 17 000 г. Удалите водный верхний слой и поместите в новую пробирку объемом 1,5 мл.
- Добавьте 500 мкл хлороформа и энергично встряхните в течение 10–15 с. Отжим в течение 5 мин при 17 000 x g при RT. Удалите водный слой и поместите в новую пробирку объемом 1,5 мл.
- Добавьте 1 мкл 20 мг/мл гликогена и 1 мл ледяного 96% этанола. Осаждайте в течение 30 минут при -80 °C или на ночь при -20 °C.
- Центрифуга в течение 30 мин при 4 °C, 17 000 x g. Удалите надосадочную жидкость, добавьте 500 мкл 70% этанола и центрифугу в течение 5 мин, 4 ° C при 17 000 x g. Удалите весь этанол, выполните быстрое вращение, чтобы собрать остатки и удалить излишки с помощью пипетки P10.
- Высушите гранулы в течение ~3 мин в вытяжном шкафу. Ресуспендировать в 20 мкл воды, обработанной DEPC.
- Храните РНК при температуре -80 °C в течение ночи или сразу переходите к этапу обратной транскрипции.
9. Обратная транскрипция
- Добавьте 2 мкл 10 мкМ олиго RT (PE_reverse; см. Таблицу 7) и 4 мкл 5 мМ dNTP к 20 мкл РНК.
| Название олигонуклеотида | Последовательность (5'-3') | |||
| P5 вперед | AATGATACGGCGACCACCGAGATCTACACTCTTTCCCACACGACGCTCTTCCGATCT | |||
| БК1 | CAAGCAGAACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGCTCTTCCGATCT | |||
| БК3 | CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGAGTTCAGACGTGCTCTTCCGATCT | |||
| БК4 | CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGCTCTTCCGATCT | |||
| БК5 | CAAGGAAGACGGCATACGAGATCACTGTGTGACTGGAGTTCAGACGTGCTCTTCCGATCT | |||
| БК7 | CAAGCAGAACGGCATACGAGATCAGATCGTGACTGGAGTTCAGACGTGCTCTTCCGATCT | |||
| БК8 | CAAGCAGAACGGCATACGAGATTAGCTTGTGACTGGAGTTCAGACGTGCTCTTCCGATCT | |||
| БК9 | CAAGCAGAAGACGGCATACGAGATGATCAGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| БК10 | CAAGGAAGACGGCATACGAGATATCACGGTGACTGGAGTTCAGACGTGCTCTTCCGATCT | |||
| PE_reverse | CAGACGTGCTCTTCCGATCT | |||
Таблица 7: Праймеры ПЦР (включая последовательности штрих-кода) и праймер обратной транскрипции.
- Переложить в предварительно нагретый термоблок при 85 °C на 3 мин, затем охладить на льду в течение 5 мин. Соберите содержимое пробирки путем кратковременного центрифугирования, а затем добавьте 8 мкл 5-кратного буфера обратной транскриптазы, 2 мкл 100 мМ DTT и 2 мкл ингибитора РНКазы.
- Смесь инкубируют при 50 °C в течение 3 мин, затем добавляют 2 мкл обратной транскриптазы и инкубируют в течение 1 ч при 50 °C.
- Инактивируйте обратную транскриптазу путем инкубации при 65 ° C в течение 15 мин.
- Переложите пробирки в предварительно нагретый термоблок при 37 °C и оставьте на 3 минуты для акклиматизации.
- Добавьте 2 мкл РНКазы H и инкубируйте в течение 30 мин при 37 ° C.
- Изолируйте кДНК с помощью шариков SPRI.
- Добавьте два объема шариков по 84 мкл. Выдерживать 15 мин. Положите бусины на магнитную решетку и оставьте на 1 минуту, чтобы собрать бусины.
- Удалите и утилизируйте надосадочную жидкость и добавьте 200 мкл 70% этанола. Не снимайте бусины с магнитной стойки. Инкубируйте шарики с этанолом в течение 30 с.
- Удалите этанол и повторите этап стирки. Удалите все остатки этанола с помощью наконечника P10.
- Поместите шарики в вытяжной шкаф на 2 минуты, чтобы они высохли. Снимите шарики со стойки, ресуспендируйте их в 12 мкл воды, а затем положите шарики обратно на решетку. Удалите 11 мкл надосадочной жидкости.
- Заморозьте кДНК при -20 °C или сразу приступайте к этапу ПЦР.
10. Реакция кПЦР
- Перед окончательной ПЦР для амплификации кДНК проводят количественную полимеразную цепную реакцию (кПЦР) для определения оптимального количества циклов амплификации кДНК для предотвращения гиперамплификации библиотеки.
- Настройте реакцию кПЦР на льду в соответствии с таблицей 8. Все грунтовки см. в таблице 7 .
| Компонент | 1x |
| 2x мастермикс реакции кПЦР | 5 |
| Праймер P5 0,1 мкМ (прямой) | 0.8 |
| Праймер 0,1 мкМ BC (обратный) | 0.8 |
| кДНК (или вода как отрицательный контроль) | 1 |
| H2O | 2.4 |
| Заключительный том | 10 мкл |
Таблица 8: Реакционная смесь для кПЦР.
- Для правильной количественной оценки циклов, необходимых для амплификации, используйте три технические реплики для кДНК и три отрицательных (т.е. водных) контроля.
- Запечатайте планшеты оптически прозрачной пленкой и запустите qPCR в соответствии с инструкциями производителя набора.
- Проанализируйте образцы с помощью метода абсолютного количественного определения, чтобы определить количество циклов (n), при которых достигается колено экспоненциального роста (см. Рисунок 4C для примера). Это количество циклов затем используется для окончательной амплификации остальной части кДНК.
11. ПЦР-реакция и гелевая экстракция
- Настройте реакцию ПЦР на льду в соответствии с таблицей 9. Все грунтовки см. в таблице 7 .
ПРИМЕЧАНИЕ: Используется только 5 мкл библиотеки кДНК.
| Компонент | 1x |
| 10 полимеразных буферов для корректуры | 5 |
| Праймер P5 10 мкМ (вперед) | 1 |
| Праймер BC 10 мкМ (обратный) | 1 |
| 5 мМ dNTP | 2.5 |
| Корректура фермента полимеразы | 1 |
| кДНК | 5 |
| H2O | 34.5 |
| Заключительный том | 50 мкл |
Таблица 9: Реакционная смесь ПЦР.
- Запустите ПЦР следующим образом: 95 °C в течение 2 мин; n циклов 98 °C в течение 20 с, 52 °C в течение 30 с и 72 °C в течение 1 мин; и 72 °C в течение 5 мин. Количество (n) циклов амплификации библиотеки χCRAC определяется методом кПЦР, описанным в разделе 10.
- Добавьте 1 мкл экзонуклеазы I и инкубируйте при 37 ° C в течение 60 мин.
- Очистите амплифицированную кДНК с помощью шариков SPRI, как описано выше, используя два объема шариков (т.е. 100 мкл). Элют в 11 мкл.
- Добавьте 3 мкл красителя с 6-кратной загрузкой и нанесите на сборный 6% гель КЭ при 100 В в течение 1 ч в буфер с 1x КЭ. Используйте лестницу, подходящую для количественного определения коротких фрагментов ДНК.
- После завершения извлеките гель из кассеты и поместите в подходящий герметичный контейнер с достаточным количеством 1x КЭ, чтобы покрыть гель (например, ~ 50 мл). Добавьте соответствующее количество безопасного красителя SYBR (например, для 50 мл используйте 5 мкл 10 000-кратного красителя)
- Дайте гелю испачкаться путем осторожного перемешивания в течение 15 минут при ЛТ. Слейте 1x TBE, содержащий SYBR, и замените его чистым 1x TBE. Смойте гель в течение 10 минут, слегка встряхивая при RT.
- Слейте 1x TBE и поместите гель в прозрачную папку. Обрежьте папку до соответствующего размера.
- Изображение геля с помощью соответствующего средства, такого как люминограф. Иссекают фрагменты ДНК от ~175.н. до ~400.н. Поместите кусочек геля в тюбик объемом 1,5 мл.
- Тщательно разбейте срез геля наконечником P1000 и добавьте 400 мкл H2O. Выдержать при 37 °C при встряхивании в течение 1 ч в термоблоке.
- Заморозьте образец на сухом льду на 10 мин, затем поместите обратно в термоблок при 37 °C при встряхивании на 1 ч.
- Создайте фильтрующий блок, взяв фильтрующую колонну и вставив внутрь два стеклянных фильтра из микрофибры. Поместите устройство в пробирку объемом 1.5 мл.
- Отрежьте конец наконечника P1000 чистым скальпелем и впитайте раздавленную суспензию геля КЭ, затем распределите в фильтрующий блок, созданный на шаге 11.12. Отжим при 17 000 x g в течение 30 с.
- Добавьте 1 мкл гликогена в надосадочную жидкость вместе с 40 мкл ацетата натрия, рН = 5,2, и 1 мл 96% этанола. Инкубировать при -80 °C в течение 30 мин.
- Центрифуга в течение 30 мин при 17 000 x g, 4 °C. Выбросьте надосадочную жидкость и промойте 500 мкл 70% этанола.
- Отжимайте в течение 5 минут, полностью удалите этанол, а затем высушите гранулы в вытяжном шкафу в течение 3 минут.
- Ресуспендировать в 10 мкл H2O и измерить концентрацию ДНК.
Результаты
Чтобы продемонстрировать эффективность метода χCRAC, был проведен эксперимент с дрожжами, экспрессирующими белок Nrd1, меченный HTP. Подробное схематическое изображение, описывающее, как работает метод, представлено на рисунке 1. Как и Nab3, Nrd1 участвует в распаде ядерной РНК различных транскриптовРНК 37. Предыдущая работа лаборатории Кордена показала, что связывание Nrd1 со своими мишенями РНК значительно изменяется, когда клетки подвергаются глюкозному голоданию28,38. Таким образом, клетки, растущие экспоненциально в среде, содержащей глюкозу (SD-TRP), были перенесены в ту же среду без глюкозы (S-TRP) в течение определенного периода времени для мониторинга динамических изменений во взаимодействиях Nrd1-РНК. Образцы отбирали и сшивали в камере Vari-X-linker (рис. 3A) перед сменой, а затем через 1, 2, 4, 8, 14 и 20 минут. Среда, используемая для роста клеток, намеренно не содержала триптофана, чтобы уменьшить поглощение ультрафиолета этой ароматической аминокислотой. Обратите внимание, что лучше всего использовать синтетическую среду, которая стерилизована фильтром, потому что автоклавирование среды может привести к карамелизации сахаров. Это снижает эффективность сшивания.
На рисунке 4A показана репрезентативная авторентгенограмма из эксперимента χCRAC. Обратите внимание, что в этом примере образцы не были объединены вместе. Вместо этого каждый из них был проведен индивидуально на геле. Это рекомендуется для первоначальных экспериментальных испытаний, чтобы показать, что белок эффективно сшивается с РНК во все тестируемые моменты времени. Особенно интенсивный сигнал наблюдался при ожидаемой молекулярной массе RBP, представляющей собой белок, связанный с очень короткими, меченными радиоактивным изобилием РНК, не поддающимися секвенированию. Таким образом, был выделен сигнал смаза над этой полосой, который представляет собой белок, сшитый с более длинными фрагментами РНК. Фрагмент был вырезан чуть выше белковой полосы плюс около 30 кДа. На рисунке 4B показана авторадиограмма после иссечения, при этом белок сшит с короткими РНК, оставшимися в геле, и ранее смазанным сигналом, теперь иссеченным.
После обратной транскрипции библиотека кДНК должна быть амплифицирована с помощью ПЦР. Однако следует избегать чрезмерной амплификации библиотеки, поскольку это может привести к смещению в сторону последовательностей, преимущественно амплифицируемых полимеразой, и генерировать артефакты ПЦР. Чрезмерно усиленные библиотеки также содержат большое количество повторяющихся последовательностей, которые тратят впустую чтение на секвенсоре. Чтобы рассчитать идеальное количество циклов ПЦР для амплификации конечной библиотеки, аликвоту кДНК амплифицировали с помощью кПЦР с использованием олигонуклеотидов P5 и BC. Первый цикл, при котором библиотека достигла пика флуоресценции, был выбран в качестве подсчета циклов ПЦР. На рисунке 4C приведен пример кПЦР из типичной библиотеки кДНК, которая дала пиковое количество циклов 16. Затем это значение использовалось для окончательной χCRAC PCR. Для обработки секвенированных данных мы использовали программное обеспечение, ранее разработанное в нашей лаборатории (pyCRAC), и соответствующий конвейер для анализа кинетических данных CRAC (Nues et al., 2017; https://git.ecdf.ed.ac.uk/sgrannem/pycrac, https://bitbucket.org/sgrann/kinetic_crac_pipeline/src/default/). Эти программные средства с открытым исходным кодом позволяют демультиплексировать и обрезать данные, удалять дубликаты ПЦР, идентифицировать статистически значимые пики, считывать кластеры в непрерывные последовательности и идентифицировать мотивы связывания39. Более подробную информацию о том, как работают эти инструменты, можно найти на соответствующих веб-страницах.
Мы также приступили к разработке протокола χCRAC для клеток млекопитающих. Большинство клеточных линий млекопитающих выращиваются в виде монослоя, и лоток в нашем сшивающем агенте с УФ-проницаемым мешком не подходит для экспериментов с адгезивными клетками. Чтобы преодолеть эту проблему, мы разработали стадию, на которой пользователи могут облучать ультрафиолетом 1–2 чашки Петри (диаметром 150 мм и глубиной 25 мм) с адгезивными ячейками (рис. 3B). В качестве первого теста эффективность сшивающего агента для клеток млекопитающих была измерена путем сшивания и захвата стабильно меченного GFP-RBM7 с использованием антител против GFP и традиционной очистки на основе CLIP. Как показано на рисунке 5A, сшивающий агент смог восстановить комплексы белок-РНК из клеток млекопитающих, выращенных в виде монослоя, с использованием УФ-облучения 254 нм с эффективностью, сравнимой с широко используемым устройством УФ-облучения. Однако стандартная пластиковая посуда для клеточных культур, обычно используемая для экспериментов по сшиванию УФ-излучения, непроницаема для ультрафиолетового излучения 254 нм. Следовательно, в нашем сшивающем агенте клетки будут получать облучение только от верхнего ряда ультрафиолетовых ламп. Чтобы преодолеть это, мы разработали проницаемую для ультрафиолета кварцевую чашку Петри для роста клеток и сшивания. Использование кварцевой посуды показало надежное восстановление белково-РНК-комплексов всего за 2 с УФ-облучения (рис. 5B). В сочетании с методами захвата RBP для клеток млекопитающих, такими как технологии CLIP, эти короткие периоды сшивания поддаются временным курсам для восстановления пространственно-временных РНК-связывающих профилей RBP в ответ на генотоксические стрессы или быстрое истощение белковых факторов или параллельно с транскрипцией или синхронизацией клеточного цикла.
На рисунке 6 показано несколько примеров данных Nrd1, обрабатываемых конвейером χCRAC. Этот рисунок был подготовлен с использованием файлов bedgraph, сгенерированных конвейером, и пакета python GenomeBrowser (https://pypi.org/project/GenomeBrowser/1.6.3/), который мы разработали для упрощения создания изображений данных в браузере генома публикационного качества. Серые прямоугольники представляют собой геномные области, которые экспрессируют некодирующие РНК, такие как загадочный нестабильный транскрипт (CUT), стабильные неохарактеризованные транскрипты (SUTs)40 и Xrn1-чувствительные нестабильные транскрипты (XUT)41. Данные на рисунке 6 показывают, что Nrd1 связывается со многими из этих некодирующих транскриптов РНК, что согласуется с идеей о том, что этот белок участвует в деградации этого класса транскриптов42. На рисунке 6A показана область ~15 кб на хромосоме IV. Здесь наблюдалось значительное увеличение связывания Nrd1 с транскриптами, кодирующими высокоаффинные переносчики глюкозы HXT6 и HXT7, оба из которых активируются во время голодания глюкозы. Вполне вероятно, что прекращение транскрипции комплексом NNS может влиять на кинетику индукции этих генов во время глюкозного голодания. На рисунке 6B показан пример сшивания Nrd1 с транскриптом Imd3, который, как известно, регулируется Nab343. В этом случае данные продемонстрировали значительное снижение связывания при глюкозном голодании. Предыдущая работа показала снижение связывания Nab3 с транскриптом Tye7 во время голодания глюкозой44. В соответствии с этим наблюдением данные χCRAC свидетельствуют о том, что связывание Nrd1 уменьшалось во время голодания глюкозой, а сшивание Nrd1 с Tye7 было самым низким после 8 минут стресса (рис. 4C). Однако, по-видимому, этот эффект был лишь временным, потому что после 14 минут голодания глюкозой связывание Nrd1 вернулось к исходным уровням.
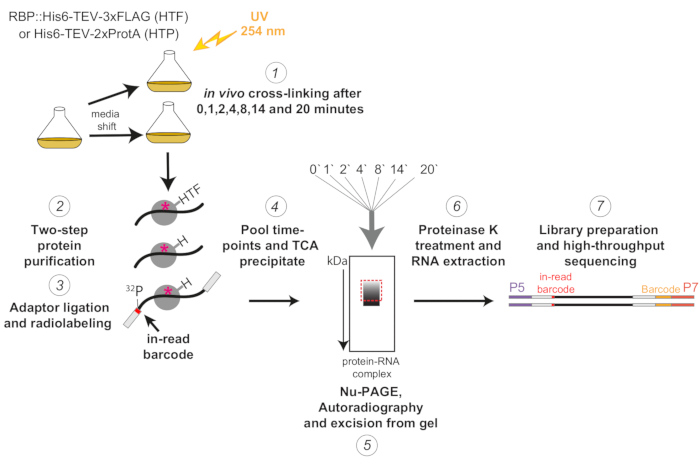
Рисунок 1: Схематическое изображение протокола χCRAC. Меченые штаммы выращивали до желаемой густоты. RBP указывает на РНК-связывающий белок. После этого был взят эталонный образец, который был сшит с ультрафиолетовым светом 254 нм. Оставшиеся клетки были собраны путем фильтрации, а затем быстро перемещены в среду, вызывающую стресс. Для описанного здесь эксперимента χCRAC были взяты пробы и сшиты через 1, 2, 4, 8, 14 и 20 минут после сдвига (1). Затем интересующий RBP очищали с использованием очень строгой двухступенчатой аффинной очистки (2). Затем захваченные сшитые РНК частично расщепляли РНКазами, радиоактивно меченными на 5'-конце, и на них лигировали адаптеры (3). 5'-адаптеры содержали уникальные «считываемые» последовательности штрих-кодов, так что отдельные образцы могли быть разделены биоинформационно после секвенирования. Затем комплексы RBP-РНК элюировали, объединяли и осаждали вместе (4), разрешали с помощью SDS-PAGE и визуализировали с помощью авторадиографии (5). Впоследствии из геля был вырезан один гелевый срез, содержащий радиоактивный сигнал чуть выше основной полосы, проиллюстрированный пунктирной красной рамкой на изображении авторадиографии (5). Срезы геля обрабатывали протеазой К, а затем экстрагировали РНК (6), превращали в кДНК и амплифицировали с помощью ПЦР (7). На этапе ПЦР были введены дополнительные штрих-коды (желтый блок, введенный олиго P7), так что многие библиотеки могут быть объединены в одну полосу. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 2: Сшивание и вакуумная фильтрация. а) Сшивающий агент. Клеточная суспензия выливается в воронку, расположенную в правом верхнем углу машины (также см. Рисунок 3A для крупного плана) и удерживается в УФ-прозрачном пакете, расположенном в среднем лотке. Этот мешок окружен двумя жалюзи, которые остаются закрытыми до тех пор, пока пользователь не даст указание машине начать этап облучения. Клетки облучаются ультрафиолетовым светом из лотков как сверху, так и снизу. Аппарат поставляется с УФ-лампами с длиной волны 254 и 365 нм, причем последняя применима для экспериментов PAR-CLIP. Машина управляется через сенсорную панель, расположенную в правом верхнем углу, которая позволяет контролировать дозировку ультрафиолета или время воздействия. (B) После сшивания ячейки сливаются с левой стороны машины. Клеточные суспензии извлекаются через вакуум и сливаются в стеклянную колбу, где их впоследствии можно вылить в вакуумное фильтрационное устройство для сбора. (C) Вакуумные фильтрационные устройства. Они открываются и закрываются с помощью зажима, и между ними вставляется фильтр. Четыре фильтрующих устройства использовались параллельно в течение очень коротких временных рядов, чтобы не терять время в результате смены фильтров. (D) После фильтрации надосадочная жидкость среды сливалась в колбы для последующего удаления. Клапаны были установлены под вакуумными фильтрующими устройствами для поддержания вакуума в системе при снятии фильтра. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 3: Сшивание свешенных и адгезивных клеток. А) Сшивающий агент с камерой Vari-X-linker для суспензионных элементов. Клеточная культура выливается во входное отверстие для образца (воронку), расположенное в правом верхнем углу лотка. (B) Лоток, который может содержать пластиковые или кварцевые чашки Петри для сшивания адгезивных клеток или небольших объемов суспензионных ячеек. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 4: Подготовка библиотеки. (A) Пример авторадиограммы из эксперимента Nrd1-HTP χCRAC. Сильный, концентрированный сигнал представляет собой белок, сшитый с очень короткими РНК, в то время как мазок выше представляет собой белок, сшитый с РНК достаточной длины для секвенирования. (B) Мазок был иссечен, как показано на авторадиограмме, сделанной после иссечения геля. (C) Репрезентативная кПЦР из библиотеки кДНК χCRAC. В этом примере максимальная амплификация кДНК была достигнута за 16 циклов. Таким образом, для окончательного усиления было использовано 16 циклов. Полоса погрешностей представляет собой стандартное отклонение трех технических реплик qPCR. (D) Пример люминофорного изображения из библиотеки кДНК на 6% геле КЭ. (E) анализ длины и качества кДНК с помощью капиллярного электрофореза на основе чипа. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
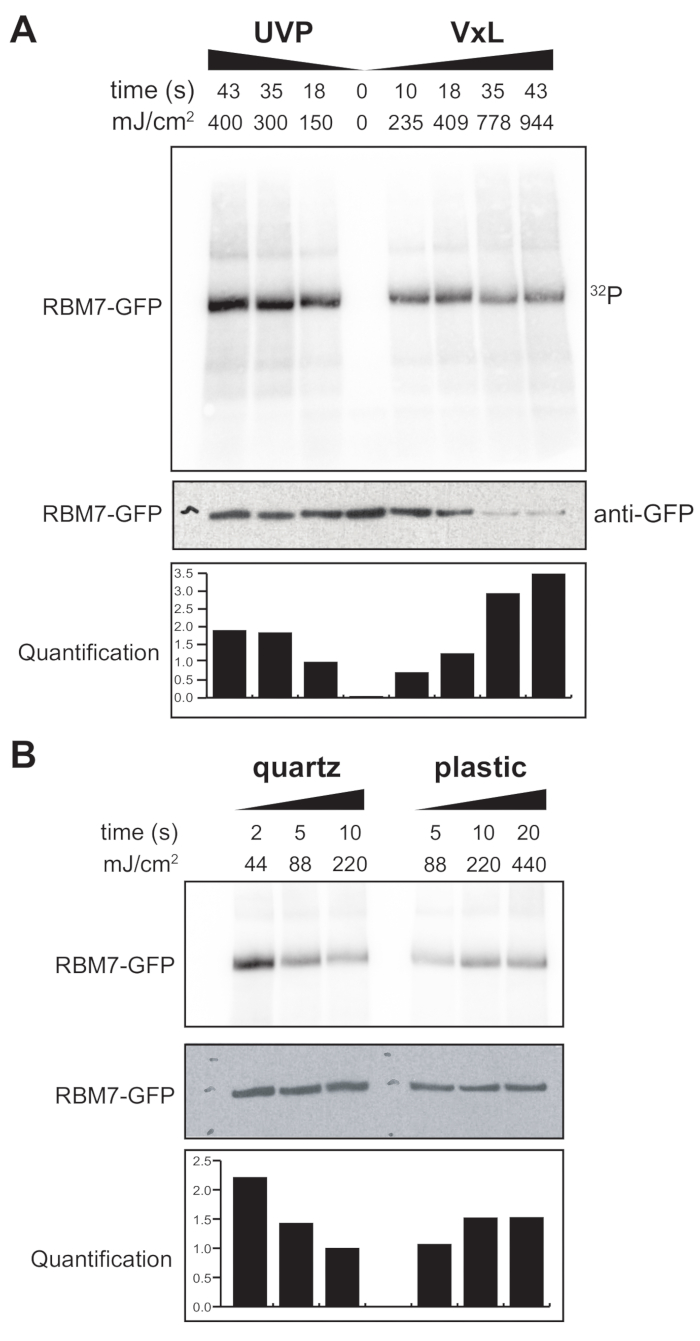
Рисунок 5: Эксперимент iCLIP с тестом с высоким уровнем РНКазы для тестирования сшивания в клетках млекопитающих. Показаны авторадиограммы из экспериментов GFP-RBM7 iCLIP, в которых проверялась эффективность восстановления RNP при различных энергиях сшивания. Иммунопреципитацию проводили с использованием антител против GFP, связанных с магнитными шариками на сшитых клетках, которые стабильно экспрессировали GFP-RBM7. Иммунопреципитаты инкубировали с высокими концентрациями РНКазы I, чтобы обрезать ассоциированные РНК до коротких однородных длин. RNP визуализировались с помощью мечения 32P и SDS-PAGE и мигрировали в виде определенной полосы, близкой к миграции несшитого белка. Количественная оценка указывает на результаты денситометрического анализа радиоактивно меченного сигнала RBM7-РНК, нормализованного к сигналу вестерн-блоттинга анти-GFP. (A) Ход времени сшивания обычно используемого сшивающего агента UVP по сравнению с нашим сшивающим агентом (Vari-X-linker; VxL). (B) Сшивание времени нашего сшивающего агента на кварцевой (слева) и пластиковой (справа) посуде. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
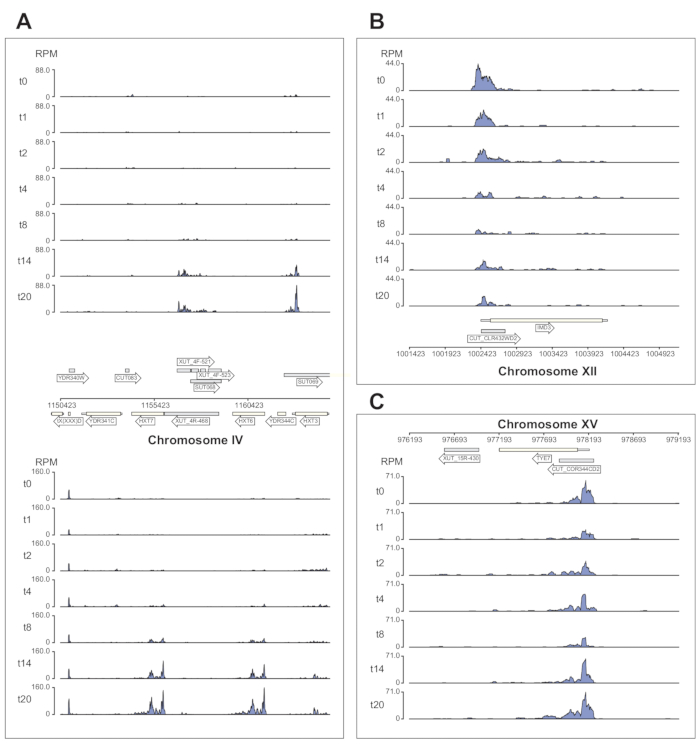
Рисунок 6: Пример графиков браузера генома, показывающих способность χCRAC показывать дифференциальное, временное связывание Nrd1 с его мишенями. В каждой коробке показаны графики для отдельных областей генома. Стрелки указывают, на какой цепи закодированы гены (стрелка слева = минус цепь; стрелка вправо = плюс цепь). Временные точки (мин) обозначаются t0, t1, t2 и т. д. на осях Y каждого подучастка. Показаны римские цифры, обозначающие хромосомы и координаты. (A) При депривации глюкозы Nrd1 связывает два высокоаффинных переносчика глюкозы, HXT6 и HXT7, которые в этом состоянии активируются. (B) Наблюдается, что Nrd1 связывается с Imd3, уже проверенной мишенью Nab344, со снижением интенсивности после голодания глюкозой. (C) Связывание Nrd1 Tye7 проявляет динамический и преходящий характер, снижаясь после голодания глюкозы до минимума после 8 минут стресса. Однако впоследствии связывание возвращается к базальным уровням через 14 мин. Показания были нормализованы до «чтений на миллион» (RPM; ось y). Серыми прямоугольниками обозначены области, кодирующие некодирующие РНК. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Обсуждение
Метод χCRAC в сочетании с новыми устройствами для сшивания и сбора клеток имеет большой потенциал, поскольку он применим к широкому кругу модельных организмов и, следовательно, должен представлять общий интерес для области РНК. Есть много областей, в которых χCRAC может быть использован. Например, метод может быть использован для измерения иерархической сборки белков в большие макромолекулярные комплексы, такие как сплайсосома и рибосома, которая часто включает динамические взаимодействия между белками и молекулами РНК. В настоящее время мы также регулярно используем его для мониторинга взаимодействия между факторами распада РНК и их субстратами, когда клетки подвергаются различным видам стрессов. Это позволяет определить, на какой стадии адаптивного ответа эти факторы наиболее активны, с какими субстратами они связываются и насколько динамичны эти взаимодействия. Такие данные должны позволить исследователям определить относительный вклад каждого фактора в адаптацию к изменениям окружающей среды.
χCRAC использует метки очистки с двойным сродством (HTF или HTP) для очистки белка в очень строгих и денатурирующих условиях. Это гарантирует, что коочищенная РНК высоко обогащена для РНК, которые были ковалентно сшиты с интересующим белком. Однако использование тегов сходства имеет недостатки. Например, метка может мешать функции белка, что может дать искаженное считывание его РНК-связывающего интерактома. Кроме того, для некоторых модельных организмов не всегда возможно использовать метки, потому что генетические инструменты для интеграции фрагментов ДНК в геном или для преобразования экспрессионных плазмид еще не доступны. Тем не менее, легко изменить некоторые части протокола χCRAC, чтобы сделать его совместимым с протоколами на основе CLIP, которые полагаются на антитела для очистки RBP. Действительно, это исследование показало, что можно комбинировать очистки на основе iCLIP с нашим сшивающим агентом. В настоящее время мы находимся в процессе разработки протоколов CLIP для изучения временной ассоциации РНК-связывающих белков человека с зарождающимися транскриптами РНК.
При выполнении χCRAC на новом белке необходимо оптимизировать воздействие ультрафиолета, чтобы вызвать максимальное сшивание. Это важно, потому что высокое воздействие ультрафиолета может уменьшить восстановление РНК на этапе очистки. Клетки, экспрессирующие рекомбинантный RBP, подвергались воздействию различных доз ультрафиолета: 100 мДж/см2, 250 мДж/см2, 500 мДж/см2 и 1 Дж/см2. Затем РНП были захвачены, а РНК были фрагментированы и радиоактивно мечены. После этого RNP были разрешены с помощью SDS-PAGE, и была сделана авторадиограмма, чтобы определить, какое воздействие дало наиболее интенсивный сигнал (т.е. максимальное сшивание).
После оптимизации условий эксперимента рекомендуется провести несколько контрольных экспериментов при выполнении χCRAC. Во-первых, облученный ультрафиолетом, немаркированный образец можно использовать для контроля фонового связывания с очищающими шариками. Во-вторых, при применении χCRAC во время эксперимента со сдвигом второй временной ряд, в котором клетки смещаются обратно в исходную среду, позволяет исследовать, вызывает ли сама фильтрация клеток изменения уровней РНК или взаимодействия белок-РНК.
Как упоминалось во введении, многочисленные недавно опубликованные статьи предлагают ряд оптимизаций протокола CLIP. Это включает в себя использование флуоресцентно меченных адаптеров для детектирования комплекса белок-РНК с помощью инфракрасного сканирования10, а также оптимизацию различных этапов очистки нуклеиновых кислот и выбора размера, которые, как показано, увеличивают сложность результирующих библиотек12,45. В настоящее время мы внедряем некоторые из этих улучшений для дальнейшего совершенствования протокола χCRAC. Представленный здесь протокол уже содержит ряд улучшений по сравнению с первоначальными протоколами CRAC и χCRAC, которые увеличивают сложность данных. Например, ранее после разрешения сшитых радиоактивных белково-РНК-комплексов на гелях SDS-PAGE их переносили на нитроцеллюлозную мембрану и выделяли сшитую РНК из блоттинга. Однако перенос РНП и последующая экстракция РНК могут быть очень неэффективными, особенно при работе с большими RBP, такими как субъединицы РНК-полимеразы. Это может привести к значительному снижению восстановления сшитой РНК. В текущем протоколе сшитая РНК извлекается непосредственно из срезов геля SDS-PAGE, как показано на рисунке 1. Это увеличило восстановление сшитых РНК. Кроме того, после ПЦР-амплификации кДНК продукт первоначально растворяли на 3%, низкотемпературных агарозных гелях, а затем из геля извлекали продукты ПЦР с содержанием 175–300.н. Однако эти гели могут быть легко перегружены, что приводит к очень плохому разделению ДНК. Замена агарозных гелей на сборные гели КЭ привела к более равномерному разделению по размерам и лучшему извлечению продуктов ПЦР.
Раскрытие информации
А. Лэнгфорд и У. Уорбойс связаны с коммерческой компанией UVO3. Они не играли никакой роли в дизайне исследования, сборе и интерпретации данных или принятии решения о представлении работы для публикации.
Благодарности
Эта работа была поддержана грантами Wellcome Trust (091549 для S.G и 109093/Z/15/A для S.M.), основным грантом Wellcome Trust Centre for Cell Biology (092076) и Советом по медицинским исследованиям для неклинических старших научных сотрудников (MR / R008205 / 1 для S.G.), Европейской организации молекулярной биологии в рамках долгосрочной постдокторской стипендии (ALTF 1070-2017 для R.A.C.), и Независимый исследовательский фонд Дании (T.H.J.).
Материалы
| Name | Company | Catalog Number | Comments |
| 1,4-dithioreitol | Merck | 10708984001 | Buffer component in mammalian cell lysis |
| 1.5 mL tubes | Eppendorf | 0030 120.086 | General reaction tube |
| 2 mL tubes | Eppendorf | 0030 123.344 | For holding columns and collection of waste |
| 32P-yATP | Perkin Elmer | NEG502Z-250 | For radiolabelling the 5' end of the RNA |
| 4-12% Bis-Tris gel | Invitrogen | NP0321BOX | SDS-PAGE gel |
| 4X loading buffer | Novex | NP0008 | Protein loading dye concentrate |
| 50 bp ladder | New England Biolabs | N3236 | Reference ladder for excising region of interest from the amplified cDNA library |
| 50% PEG | NEB | B100045 | For the L5 linker ligation |
| 6% TBE gel | Invitrogen | EC6265BOX | For separation and purification of the cDNA library |
| Acetone | ACROS Organics | 423245000 | Washing of TCA-precipitated proteins |
| anti-FLAG beads | Sigma Aldrich | M8823-1ML | For purifcation of FLAG-tagged RBPs |
| ATP (100 mM) | Thermo Fisher Scientific | R0441 | For ligation of the L5 linker onto the 5' end of captured RNAs |
| Beta-mercaptoethanol | Sigma Aldrich | M3148-100ML | Buffer component |
| Biomax MS intensifying screen | Sigma Aldrich | Z363162-1EA | For intensifying the autoradiogram signal |
| Chloroform | Thermo Fisher Scientific | 1010219 | For phenol-chloroform extraction following RNA purification |
| cOmplete EDTA-free protease inhibitor cocktail | Roche | 11873580001 | For inhibition of cellular proteases after lysis |
| Complete supplement mixture -TRP | Formedium | DCS0149 | For preparation of synthetic defined medium |
| Costar Spin-X 0.22 µm filters | Sigma Aldrich | CLS8160 | For isolating the excised cDNAs following gel extraction |
| DNase RQ1 | Promega | M6101 | For DNA digest following cell lysis |
| dNTPs (10 mM) | Sigma Aldrich | 4638956001 | For reverse transcription and PCR |
| Ethanol | Thermo Fisher Scientific | 10041814 | For phenol-chloroform extraction following RNA purification and DNA precipitation |
| Ethylenediaminetetraacetic acid | Invitrogen | AM9261 | For protease K buffer |
| Exonuclease I | New England Biolabs | M0293 | For degradation of primers following PCR |
| Glass microfiber filters | Whatman | 1823-010 | For isolating the excised cDNAs following gel extraction |
| Glucose | Formedium | GLU03 | For preparation of glucose-containing, synthetic defined medium |
| Glycogen (20 mg/mL) | Roche | 10901393001 | Precipitation of proteins, RNA and DNA |
| GST-TEV | Homemade | Construct and purification protocol is available upon request | |
| Guanidium hydrochloroide | Thermo Fisher Scientific | 10071503 | Required for pulldown denaturing conditions and washing buffer |
| IgG beads | GE Healthcare | 17-0969-01 | For purification of protein A-tagged RBPs |
| Imidazole | Sigma Aldrich | I2399-100G | For elution of captured proteins from Nickel beads |
| Isoamyl alcohol | Thermo Fisher Scientific | A393-500 | For phenol-chloroform extraction following RNA purification |
| Luna Universal One-Step RT-qPCR | NEB | E3005S | For qPCR of the cDNA in order to calculate required number of PCR cycles |
| Magnesium chloride | Fluka Analytical | 63020-1L | For PNK buffer |
| Membrane filters | Millipore | AAWP09000 for yeast or HAWP09000 for bacteria | For vacuum filtration of cells |
| Micro bio-spin columns | Biorad | 732-6204 | For collecting eluate after gel extraction |
| Ni-NTA beads | Qiagen | 30210 | For secondary protein capture |
| NP-40 | Sigma Aldrich | I8896-100ML | Buffer component |
| Pfu polymerase | Promega | M7741 | For amplification of the cDNA library |
| Phenol | Sigma Aldrich | P4682-400ML | For phenol-chloroform extraction following RNA purification |
| Pierce spin columns | Thermo Fisher Scientific | 69725 | For on-column enzymatic reactions |
| Protease K | Roche | 3115887001 | For degradation of the RBP following gel extraction |
| Quartz Petri dish | UVO3 | N/A | For cross-linking of adherent cells. Available from https://www.vari-x-link.com for 400 GBP |
| Radiography films | Amersham | 28906843 | For autoradiography visualisation |
| RNAClean XP beads | Beckmann | A63987 | SPRI beads for clean up of RNAs and cDNAs |
| RNase H | New England Biolabs | M0297 | For degradation of RNAs following reverse transcription |
| RNase-It | Agilent | 400720 | For RNA digestion |
| rRNasin | Promega | N2511 | For inhibition of any contaminating RNases during enzymatic reaction |
| Sodium acetate | Sigma Aldrich | S2889-1KG | For phenol-chloroform extraction following RNA purification and DNA precipitation |
| Sodium chloride | Thermo Fisher Scientific | 7647-14-5 | Buffer component |
| Sodium deoxycholate | Sigma Aldrich | D6750-100G | Buffer component in mammalian cell lysis |
| Sodium dodecylsulfate | Sigma Aldrich | L3771-1KG | For protease K buffer |
| SUPERase-In | Invitrogen | AM2694 | For inhibition of cellular RNases after lysis |
| SuperScript IV | Thermo Fisher Scientific | 18090010 | For reverse transcription |
| T4 PNK | New England Biolabs | M0201 | For radiolabelling the 5' end of the RNA |
| T4 RNA ligase 1 | New England Biolabs | M0204 | For ligation of the L5 adaptor onto the RNA 5' end |
| T4 RNase ligase 2, truncated K222Q | NEB | M0351S | For ligation of the App_PE linker onto the 3' end of captured RNAs |
| TBE buffer (10X) | Invitrogen | 15581-028 | For running TBE gels |
| TEV protease | Homemade | For eluting captured proteins following FLAG capture | |
| Thermosensitive alkaline phosphatase | Promega | M9910 | For 5' and 3' dephosphorylation of RNAs |
| Trichloroacetic acid (100%) | Sigma Aldrich | T0699-100ML | For precipitation of RBP-RNA complexes |
| Tris hydrochloride | Invitrogen | 15504-020 | Buffer component |
| Triton X-100 | Sigma Aldrich | T8787-100ML | Buffer component in mammalian cell lysis |
| Vari Filter | UVO3 | N/A | Device for vacuum harvesting cells. Available from https://www.vari-x-link.com for 100 GBP |
| Vari-X-Linker | UVO3 | N/A | Cross-linker for cross-linking cells. Available from https://www.vari-x-link.com for 16,000 GBP |
| Yeast nitrogen base | Formedium | CYN0410 | For preparation of synthetic defined medium |
| Zirconia beads | Thistle | 11079105Z for yeast or 11079101Z for bacteria | For cell lysis via bead beating |
Ссылки
- Ule, J., et al. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Granneman, S., Kudla, G., Petfalski, E., Tollervey, D. Identification of protein binding sites on U3 snoRNA and pre-rRNA by UV cross-linking and high-throughput analysis of cDNAs. Proceedings of the National Academy of Sciences. 106 (24), 9613-9618 (2009).
- Licatalosi, D. D., et al. HITS-CLIP yields genome-wide insights into brain alternative RNA processing. Nature. 456 (7221), 464-469 (2008).
- König, J., et al. iCLIP reveals the function of hnRNP particles in splicing at individual nucleotide resolution. Nature Structural & Molecular Biology. 17 (7), 909-915 (2010).
- Hafner, M., et al. Transcriptome-wide Identification of RNA-Binding Protein and MicroRNA Target Sites by PAR-CLIP. Cell. 141 (1), 129-141 (2010).
- Aktaş, T., et al. DHX9 suppresses RNA processing defects originating from the Alu invasion of the human genome. Nature. 544 (7648), 115-119 (2017).
- Huppertz, I., et al. iCLIP: Protein–RNA interactions at nucleotide resolution. Methods. 65 (3), 274-287 (2014).
- Li, X., et al. Comprehensive in vivo RNA-binding site analyses reveal a role of Prp8 in spliceosomal assembly. Nucleic Acids Research. 41 (6), 3805-3818 (2013).
- Rosenberg, M., et al. Denaturing CLIP, dCLIP, Pipeline Identifies Discrete RNA Footprints on Chromatin-Associated Proteins and Reveals that CBX7 Targets 3′ UTRs to Regulate mRNA Expression. Cell Systems. 5 (4), 368-385 (2017).
- Zarnegar, B. J., et al. irCLIP platform for efficient characterization of protein–RNA interactions. Nature Methods. 13 (6), 489-492 (2016).
- Kargapolova, Y., Levin, M., Lackner, K., Danckwardt, S. sCLIP—an integrated platform to study RNA–protein interactomes in biomedical research: identification of CSTF2tau in alternative processing of small nuclear RNAs. Nucleic Acids Research. 45 (10), 6074-6086 (2017).
- Van Nostrand, E. L., et al. Robust transcriptome-wide discovery of RNA-binding protein binding sites with enhanced CLIP (eCLIP). Nature Methods. 13 (6), 508-514 (2016).
- Flynn, R. A., et al. Dissecting noncoding and pathogen RNA–protein interactomes. RNA. 21 (1), 135-143 (2015).
- Brugiolo, M., Botti, V., Liu, N., Müller-McNicoll, M., Neugebauer, K. M. Fractionation iCLIP detects persistent SR protein binding to conserved, retained introns in chromatin, nucleoplasm and cytoplasm. Nucleic Acids Research. 45 (18), 10452-10465 (2017).
- Sanford, J. R., et al. Identification of Nuclear and Cytoplasmic mRNA Targets for the Shuttling Protein SF2/ASF. PLOS ONE. 3 (10), e3369 (2008).
- Garzia, A., Meyer, C., Morozov, P., Sajek, M., Tuschl, T. Optimization of PAR-CLIP for transcriptome-wide identification of binding sites of RNA-binding proteins. Methods. 118-119, 24-40 (2017).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Research. 22 (10), 2031-2042 (2012).
- Chen, K., et al. High-Resolution N6-Methyladenosine (m6A) Map Using Photo-Crosslinking-Assisted m6A Sequencing. Angewandte Chemie International Edition. 54 (5), 1587-1590 (2015).
- Ke, S., et al. A majority of m6A residues are in the last exons, allowing the potential for 3′ UTR regulation. Genes & Development. 29 (19), 2037-2053 (2015).
- Linder, B., et al. Single-nucleotide-resolution mapping of m6A and m6Am throughout the transcriptome. Nature Methods. 12 (8), 767-772 (2015).
- Kudla, G., Granneman, S., Hahn, D., Beggs, J. D., Tollervey, D. Cross-linking, ligation, and sequencing of hybrids reveals RNA–RNA interactions in yeast. Proceedings of the National Academy of Sciences. 108 (24), 10010-10015 (2011).
- Sugimoto, Y., et al. hiCLIP reveals the in vivo atlas of mRNA secondary structures recognized by Staufen 1. Nature. 519 (7544), 491-494 (2015).
- Hwang, H. W., et al. cTag-PAPERCLIP Reveals Alternative Polyadenylation Promotes Cell-Type Specific Protein Diversity and Shifts Araf Isoforms with Microglia Activation. Neuron. 95 (6), 1334-1349 (2017).
- Hwang, H. W., et al. PAPERCLIP Identifies MicroRNA Targets and a Role of CstF64/64tau in Promoting Non-canonical poly(A) Site Usage. Cell Reports. 15 (2), 423-435 (2016).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Beckmann, B. M. RNA interactome capture in yeast. Methods. 118-119, 82-92 (2017).
- Granneman, S., Petfalski, E., Tollervey, D. A cluster of ribosome synthesis factors regulate pre-rRNA folding and 5.8S rRNA maturation by the Rat1 exonuclease. The EMBO Journal. 30 (19), 4006-4019 (2011).
- Schaughency, P., Merran, J., Corden, J. L. Genome-Wide Mapping of Yeast RNA Polymerase II Termination. PLOS Genetics. 10 (10), e1004632 (2014).
- Bernstein, J. A., Khodursky, A. B., Lin, P. H., Lin-Chao, S., Cohen, S. N. Global analysis of mRNA decay and abundance in Escherichia coli at single-gene resolution using two-color fluorescent DNA microarrays. Proceedings of the National Academy of Sciences. 99 (15), 9697-9702 (2002).
- Kresnowati, M. T. A. P., et al. When transcriptome meets metabolome: fast cellular responses of yeast to sudden relief of glucose limitation. Molecular Systems Biology. 2, 49 (2006).
- Marguerat, S., Lawler, K., Brazma, A., Bähler, J. Contributions of transcription and mRNA decay to gene expression dynamics of fission yeast in response to oxidative stress. RNA Biology. 11 (6), 702-714 (2014).
- van Nues, R., et al. Kinetic CRAC uncovers a role for Nab3 in determining gene expression profiles during stress. Nature Communications. 8 (1), 12 (2017).
- Selinger, D. W., Saxena, R. M., Cheung, K. J., Church, G. M., Rosenow, C. Global RNA Half-Life Analysis in Escherichia coli Reveals Positional Patterns of Transcript Degradation. Genome Research. 13 (2), 216-223 (2003).
- Tudek, A., Candelli, T., Libri, D. Non-coding transcription by RNA polymerase II in yeast: Hasard or nécessité?. Biochimie. 117, 28-36 (2015).
- Lingaraju, M., et al. The MTR4 helicase recruits nuclear adaptors of the human RNA exosome using distinct arch-interacting motifs. Nature Communications. 10 (1), 1-11 (2019).
- Lubas, M., et al. Interaction Profiling Identifies the Human Nuclear Exosome Targeting Complex. Molecular Cell. 43 (4), 624-637 (2011).
- Conrad, N. K., et al. A yeast heterogeneous nuclear ribonucleoprotein complex associated with RNA polymerase II. Genetics. 154 (2), 557-571 (2000).
- Darby, M. M., Serebreni, L., Pan, X., Boeke, J. D., Corden, J. L. The Saccharomyces cerevisiae Nrd1-Nab3 Transcription Termination Pathway Acts in Opposition to Ras Signaling and Mediates Response to Nutrient Depletion. Molecular and Cellular Biology. 32 (10), 1762-1775 (2012).
- Webb, S., Hector, R. D., Kudla, G., Granneman, S. PAR-CLIP data indicate that Nrd1-Nab3-dependent transcription termination regulates expression of hundreds of protein coding genes in yeast. Genome Biology. 15 (1), R8 (2014).
- Jensen, T. H., Jacquier, A., Libri, D. Dealing with Pervasive Transcription. Molecular Cell. 52 (4), 473-484 (2013).
- van Dijk, E. L., et al. XUTs are a class of Xrn1-sensitive antisense regulatory non-coding RNA in yeast. Nature. 475 (7354), 114-117 (2011).
- Thiebaut, M., et al. Futile Cycle of Transcription Initiation and Termination Modulates the Response to Nucleotide Shortage in S. cerevisiae. Molecular Cell. 31 (5), 671-682 (2008).
- Merran, J., Corden, J. L. Yeast RNA-Binding Protein Nab3 Regulates Genes Involved in Nitrogen Metabolism. Molecular and Cellular Biology. 37 (18), e00154-e00117 (2017).
- Bresson, S., Tuck, A., Staneva, D., Tollervey, D. Nuclear RNA Decay Pathways Aid Rapid Remodeling of Gene Expression in Yeast. Molecular Cell. 65 (5), 787-800 (2017).
- Buchbender, A., et al. Improved library preparation with the new iCLIP2 protocol. Methods. , (2019).
Erratum
Formal Correction: Erratum: Monitoring Protein-RNA Interaction Dynamics in vivo at High Temporal Resolution using χCRAC
Posted by JoVE Editors on 2/17/2023. Citeable Link.
An erratum was issued for: Monitoring Protein-RNA Interaction Dynamics in vivo at High Temporal Resolution using χCRAC. The Authors section was updated from:
Stuart W. McKellar1
Ivayla Ivanova1
Robert W. van Nues2
Ross A. Cordiner3
Will Worboys4
Andrew Langford4
Torben Heick Jensen3
Sander Granneman1
1Centre for Synthetic and Systems Biology, University of Edinburgh
2Institute of Cell Biology, University of Edinburgh
3Department of Molecular Biology and Genetics, Aarhus University, 4UVO3 Ltd.
to:
Stuart W. McKellar1
Ivayla Ivanova1
Robert W. van Nues2
Ross A. Cordiner3
Mehak Chauhan1
Niki Christopoulou1
Will Worboys4
Andrew Langford4
Torben Heick Jensen3
Sander Granneman1
1Centre for Engineering Biology, University of Edinburgh
2Institute of Cell Biology, University of Edinburgh
3Department of Molecular Biology and Genetics, Aarhus University, 4UVO3 Ltd.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены