Method Article
וילונות DNA שופכים אור על מערכות מולקולריות מורכבות במהלך רקומבינציה הומולוגית
* These authors contributed equally
In This Article
Summary
וילונות DNA מציגים שיטה חדשה להמחשת מאות או אפילו אלפי חלבונים קושרי DNA בזמן אמת כשהם מקיימים אינטראקציה עם מולקולות DNA מיושרות על פני תא דגימה מיקרופלואידי.
Abstract
רקומבינציה הומולוגית (HR) חשובה לתיקון שברי DNA דו-גדיליים (DSBs) ומזלגות שכפול תקועים בכל האורגניזמים. פגמים ב-HR קשורים קשר הדוק לאובדן שלמות הגנום וטרנספורמציה אונקוגנית בתאים אנושיים. HR כרוך בפעולות מתואמות של קבוצה מורכבת של חלבונים, שרבים מהם עדיין לא מובנים היטב. ההיבט המרכזי של המחקר המתואר כאן הוא טכנולוגיה הנקראת "וילונות DNA", טכניקה המאפשרת הרכבה של מולקולות DNA מיושרות על פני תא דגימה מיקרופלואידי. לאחר מכן ניתן לדמיין אותם על ידי מיקרוסקופ פלואורסצנטי השתקפות פנימית כוללת (TIRFM). וילונות DNA היו חלוצים על ידי המעבדה שלנו ומאפשרים גישה ישירה למידע מרחבי-זמני בקנה מידה של אלפית שנייה ורזולוציה בקנה מידה ננומטרי, שלא ניתן לחשוף בקלות באמצעות מתודולוגיות אחרות. יתרון מרכזי של וילונות DNA הוא שהוא מפשט את איסוף הנתונים הרלוונטיים מבחינה סטטיסטית מניסויים במולקולה בודדת. מחקר זה ממשיך להניב תובנות חדשות לגבי האופן שבו תאים מווסתים ושומרים על שלמות הגנום.
Introduction
שמירה על שלמות הגנום חיונית לתפקוד תקין של כל התאים החיים1. פגמים בשלמות הגנום עלולים להוביל למצבים בריאותיים חמורים, כולל סוגים שונים של סרטן ומחלות ניווניות הקשורות לגיל2. רקומבינציה הומולוגית (HR) משתמשת בסינתזת DNA תלויה בתבנית כדי לתקן שברים דו-גדיליים של DNA (DSB), פערי DNA חד-גדיליים (ssDNA) וקישורים צולבים של DNA בין-גדיליים3. משאבי אנוש נחוצים גם לשחזור מזלגות שכפול תקועים וקורסים 3,4. יתר על כן, HR חיוני להפרדת הכרומוזומים המדויקת במהלך מיוזה 5,6.
HR כרוך בפעולות מתואמות של קבוצה מורכבת של חלבונים, שרבים מהם אינם מובנים היטב1. דוגמאות כוללות חלבון שכפול A (RPA), Rad51 ו-Rad54, בין רבים אחרים7. תגובות HR הן בתאים פרוקריוטיים והן בתאים אוקריוטיים מערבות מתווך ssDNA, המצופה במהירות על ידי חלבונים קושרי ssDNA (SSB בפרוקריוטים ו-RPA באיקריוטים)8. חלבונים אלה מגנים על ה-ssDNA מפני נוקלאזות, מבטלים מבנה משני ומקדמים גיוס של גורמים במורד הזרם 8,9. Rad51 הוא חבר במשפחת Rad51/RecA התלויה ב-ATP של רקומבינאזות DNA, הקיימות בכל האורגניזמים החיים1. Rad51 מקדם פלישת גדיל DNA לתורם ה-dsDNA ההומולוגי. בהתחשב בחשיבותו, Rad51 מוסדר מאוד, ופגמים בתהליכי רגולציה אלה קשורים בדרך כלל לאובדן שלמות הגנום וטרנספורמציה אונקוגנית7. Rad54 הוא חבר במשפחת Swi2/Snf2 של טרנסלוקזים dsDNA ומשפצי כרומטין10,11. חלבונים אלה משמשים כגורמי ויסות חיוניים של Rad51. חשוב לציין, Rad54 מסיר את Rad51 מתוצר ה-dsDNA של פלישת הגדילים והוא הכרחי גם כדי למנוע הצטברות שגויה של Rad51 על כרומטין11. פעילויות מולקולריות של חלבונים המעורבים ב-HR הן בתאי שמרים והן בתאי חיידקים שפכו אור על תפקודם ב-HR, אך כיצד בדיוק פעילותם תורמת ל-HR עדיין לא מובנת היטב 12.
וילונות DNA התגלו כפלטפורמה ייחודית המספקת גישה ישירה למנגנונים מולקולריים ודינמיקה מקרו-מולקולרית שאחרת היו נשארים בלתי נגישים13,14. כדי להכין וילונות DNA, פני השטח של תא מיקרופלואידית מצופים בשכבה דו-שכבתית של שומנים, ומולקולות DNA קשורות לשכבה הדו-שכבתית באמצעות קישור ביוטין-סטרפטווידין. השכבה הדו-שכבתית הופכת את פני השטח לאינרטיים על ידי חיקוי ממברנות תאים טבעיות. כוח הידרודינמי מיישר את ה-DNA לאורך מחסומים ננו-מפוברקים, ומאפשר הדמיה של מאות מולקולות בשדה ראייה יחיד על ידי מיקרוסקופ פלואורסצנטי של השתקפות פנימית כוללת (TIRFM). המחסומים מיוצרים על ידי ליתוגרפיה של אלומת אלקטרונים, וריאציות בתכנון המחסום מאפשרות שליטה מדויקת על גיאומטריית ההתפלגות והקשירה של ה-DNA. גישות אלו ישימות בקלות עם ssDNA או dsDNA 13,14,15,16,17,18. כנים עשויים להיות גם ננו-מיוצרים (יחד עם המחסומים) כדי לאפשר לקשור את שני קצוות ה-DNA למשטח תא הדגימה, כך שניתן יהיה לבצע ניסויים במצב יציב בהיעדר זרימת חיץ.
שינויים תלויי זמן בקומפלקסים בודדים של חלבון-חומצות גרעין מתגלים על ידי בדיקה של סרטונים בזמן אמת ומיוצגים בדפוס באמצעות קימוגרפים, המציגים את המיקום המשתנה של חלבונים על ה-DNA לאורך זמן. היבט חשוב של גישת וילונות ה-DNA הוא שהיא אינה דורשת בהכרח מודלים אפריוריים או הנחות לגבי מנגנונים מולקולריים, מכיוון שניתן לצפות בהתנהגות של רכיבי תגובה בודדים בזמן אמת. זה מאפשר התבוננות ישירה בהתנהגויות מולקולריות. כאן, פרוטוקול זה מתאר כיצד להכין וילונות DNA עם מצעי dsDNA וכן את יישומו לחקר חומרי ביניים ברקומבינציה הומולוגית.
Protocol
1. הכנת ציר שומנים
- יש להמיס 1 גרם של 18:1 (Δ9-Cis) PC, 100 מ"ג של 18:1 PEG2000 PE ו-5 מ"ג של 18:1 ביוטיניל פקק PE ב-10 מ"ל של כלורופורם.
הערה: גם DOPC וגם DPPE מאופיינים היטב ונבחרים (יחד עם שינוי PEG) בשל יכולתם למזער ספיחה לא ספציפית לפני השטח של תא הזרימה. ניתן לאכול תערובת מאסטר ליפידים מומסים ולאחסן בטמפרטורה של -20 מעלות צלזיוס עד 12 חודשים. - השתמש בכלורופורם כדי לנקות מזרק זכוכית ממס אורגני, ולאחר מכן העבר 200 מיקרוליטר (10% מהנפח הרצוי הסופי) של תערובת מאסטר השומנים לבקבוקון הזכוכית.
- אידוי הכלורופורם מבקבוקון הזכוכית באמצעות זרם עדין מאוד של חנקן דחוס. הגדל מעט את הלחץ לקראת סוף האידוי כדי להבטיח שכל עקבות הכלורופורם יוסרו.
הערה: הטה וסובב את הבקבוקון במהלך תהליך האידוי כך שהליפידים יתייבשו כשכבה אחידה של סרט לבן על הקיר. - הנח את הבקבוקון ללא מכסה תחת ואקום למשך הלילה.
- הוסף 2 מ"ל של מאגר שומנים (10 מ"מ Tris-HCl [pH 8.0], 100 מ"מ NaCl) לסרט השומנים המיובש וכסה את הבקבוקון. יש לדגור בטמפרטורת החדר (RT) למשך שעה אחת לפחות ומערבולת עד להמסה.
- העבירו את התמיסה לצינור תחתון עגול מפוליסטירן בנפח 5 מ"ל וסוניקט על קרח באמצעות מיקרוטיפ עם הפרמטרים הבאים: משרעת = 50, זמן תהליך = 1.5 דקות, דופק = 15 שניות, זמן הפסקה = 2 דקות. הפתרון יהיה ברור לאחר סוניקציה (אנרגיה כוללת = 1,500-2,000 J).
- סנן את התמיסה דרך מסנן מזרק PVDF של 0.22 מיקרומטר ואחסן את מלאי השומנים ב-4 מעלות צלזיוס למשך עד חודש.
2. הכנת מצע dsDNA
- מחממים את מלאי ה-λ-DNA (500 מיקרוגרם/מ"ל) ל-65 מעלות צלזיוס.
- הכן תערובת חישול/קשירה המכילה: 1.6 pM של λ-DNA, עודף מולרי של 100x (160 pM) של כל אוליגונוקלאוטיד (BioL: 5'Phos-AGG TCG CCG CCC-3Bio ו-DigR: 5'Phos-GGG CGG CGA CCT-3Dig_N) ו-60 מיקרוליטר של מאגר תגובה 10x T4 ligase (בנפח כולל של 600 מיקרוליטר).
הערה: אוליגונוקלאוטידים של BioL ו-DigR עוברים ביוטינילציה (Bio) ומשתנים עם דיגוקסיגנין (Dig), בהתאמה. ביוטינילציה מאפשרת הצמדה של מולקולות DNA לליפידים ביוטיניליים באמצעות קשרי סטרפטווידין. שינוי ה-Dig מאפשר קשירה כפולה באמצעות קשירה של נוגדנים נגד Dig המצופים על כנים לחלקי Dig. - חישול האוליגונוקלאוטידים לקצוות λ-DNA על ידי דגירה של התגובה ב-65 מעלות צלזיוס למשך 10 דקות, ואז אפשר לה להתקרר לאט ל-RT.
- הוסף 5 מיקרוליטר של T4 DNA ligase ודגירה ב-RT למשך הלילה.
הערה: שלבי חישול וקשירה עשויים להתבצע גם במחזור תרמי. - לזרז את ה-λ-DNA על ידי הוספת 300 מיקרוליטר של תמיסת PEG-8000 (30% w/v PEG-8000 ו-10 mM MgCl2). דגירה בטמפרטורה של 4 מעלות צלזיוס למשך שעה לפחות.
- צנטריפוגה ב-14,000 x גרם למשך 5 דקות ב-RT. הסר בזהירות את הסופרנטנט מבלי להפריע לכדור. לחלופין, שטפו את הגלולה פי 1 עם 500 מיקרוליטר של אתנול 70% צונן.
- השעו מחדש את הגלולה ב-40 מיקרוליטר של מאגר TE (10 מ"מ Tris-HCl, 1 מ"מ EDTA [pH 8.0]).
3. ננו-ייצור של דפוסי כרום
- קדחו שני חורים במרכז שקופית מיקרוסקופ קוורץ בגודל 1 אינץ' x 3 אינץ' (~3 ס"מ זה מזה) באמצעות מקדח מצופה יהלום בגודל 1.4 מ"מ על מקדחה שולחנית (ראה איור 1 לגיאומטריה של מיקום הקידוח).
- נקה את המגלשות הקדוחות על ידי טבילתן ב-200 מ"ל של תמיסת פיראנה (75% חומצה גופרתית [97%] ו-25% מי חמצן). דגירה למשך 30 דקות.
זהירות: יש לנקוט משנה זהירות כדי להימנע מכל מגע עם עור חשוף. הכן את התמיסה על ידי ערבוב איטי מאוד של מי חמצן קר לחומצה גופרתית. השלך פסולת פיראנה כראוי למיכל פסולת ייעודי. - שטפו את השקופיות על ידי טבילה ב-200 מ"ל מים מזוקקים כפולים. חזור על הפעולה פעמיים כדי לחסל את כל הפיראנה שנותרה. יבש את השקופיות בזרם עדין של חנקן דחוס.
- בעזרת ציפוי מסתובב (4,000 סל"ד למשך 45 שניות, קצב רמפה של 300 סל"ד לשנייה), מצפים את השקופיות הנקיות בפולימר פוטו-רזיסט ומוליך באופן הבא:
- שקופיות ציפוי בשכבה של 3% (w/v) פולימתיל-מתאקרילט (PMMA; 24.3K MW; מומס באניסול).
- שקופיות ציפוי עם שכבה של 1.5% (w/v) פולימתיל-מתאקרילט (PMMA; 495K MW; מומס באניסול).
- שקופיות ציפוי בשכבה של חומר אנטי סטטי לליתוגרפיה של קרן אלקטרונים.
- כתוב דפוסים מותאמים אישית עבור מחסומים וכנים על השקופיות המצופות באמצעות ליתוגרפיה של קרן אלקטרונים עם מיקרוסקופ אלקטרונים סורק המצויד בתוכנת מערכת ליצירת ננו-תבניות.
- פתח את השקופיות על ידי שטיפה תחילה של החומר האנטי סטטי עבור ליתוגרפיית קרן האלקטרונים במים מזוקקים כפולים. לאחר מכן, הנח אותם בצינורות של 50 מ"ל המכילים את התמיסה המתפתחת (75% מתיל איזובוטיל קטון [MIBK] ו-25% איזופרופנול; שמור מקורר ב-20 מעלות צלזיוס) וסוניקט באמבט קרח בעוצמה נמוכה למשך דקה אחת.
- שוטפים את התמיסה המתפתחת עם איזופרופנול ומייבשים בזרם עדין של חנקן דחוס.
- בעזרת מאייד קרן אלקטרונים, הפקידו שכבת כרום בעובי 250 Å על המשטח המעוצב ב-0.5 Å/s.
- הרם את הכרום ואת ה-PMMA הנותר באמצעות אצטון מבקבוק שפריץ, ולאחר מכן סוניקציה של השקופיות באמבט אצטון למשך 10 דקות. שוטפים שוב עם אצטון מבקבוק השפריץ.
- כדי למנוע משקעים מהאצטון שנותר, יש לשטוף את השקופיות באלכוהול איזופרופיל ולייבש בזרם עדין של חנקן דחוס.
4. הרכבת תא זרימה
- הנח תבנית נייר מלבנית בגודל 35 מ"מ על 5 מ"מ (המכסה את תבנית הכרום כמו גם את חורי הכניסה/יציאה שנקדחו מראש) מעל שקופית הקוורץ, כשהצד המעוצב פונה כלפי מעלה ( איור 1A).
- החל חתיכת סרט דו צדדי (עובי 1 מ"מ) על שקופית הקוורץ, מכסה את תבנית הנייר, ולאחר מכן השתמש בסכין גילוח כדי לגזור את תבנית הנייר המלבנית ( איור 1A).
- הנח כיסוי זכוכית מעל הסרט הדו-צדדי והפעל לחץ עדין כדי לאטום את כיסוי הכיסוי לסרט ( איור 1A). הרווח בין החלקת הכיסוי למגלשה, אטום על ידי סרט דו צדדי, יוצר את החדר המיקרופלואידי.
הערה: חיוני שהאיטום בין החלקת הכיסוי להחלקה יהיה מאובטח מספיק כדי למנוע דליפה במהלך הניסוי. - הנח את כריך ההחלקה בין שקופיות זכוכית והצמד תפסי קלסר מכל ארבעת הצדדים כדי לפזר את הלחץ באופן שווה. הכניסו את המכלול לתנור ואקום למשך 60 דקות בטמפרטורה של 135 מעלות צלזיוס כדי להמיס את הסרט הדו-צדדי ולאטום את החדר.
- הוציאו אותו מהתנור ולאחר מכן שחררו את תפסי הקלסר ושקופיות הזכוכית.
- חבר יציאות מיקרופלואידיות לחורי הכניסה/יציאה שנקדחו מראש עם אקדח דבק חם על ידי הנחת המכלול המודבק על בלוק חימום (220 מעלות צלזיוס) למשך דקה אחת, הסרתו והפעלת לחץ עדין בזהירות כדי לאטום כראוי את היציאות המיקרופלואידיות. אפשר לדבק להתרפא לחלוטין לפני שתמשיך.
הערה: ניתן למקם תאי זרימה מורכבים בצינורות של 50 מ"ל ולאחסן אותם בוואקום.
5. הרכבת וילון dsDNA
- חבר צינורות כניסה/יציאה ליציאות המיקרופלואידיות של תא הזרימה ושטוף את התא ב-20 מ"ל מים מזוקקים כפולים באמצעות שני מזרקים של 10 מ"ל.
- עם מזרק של 10 מ"ל מלא במים מזוקקים כפולים המחוברים לשני צידי תא הזרימה, הפעל כוח כדי לדחוף/למשוך את המים בין מזרקי הכניסה והיציאה. שלב זה מסיר את כל בועות האוויר מתא הזרימה.
- שטפו את החדר עם 3 מ"ל של מאגר שומנים (10 מ"מ Tris-HCl [pH 8.0], 100 מ"מ NaCl).
הערה: ודא שכל החיבורים לתאי הזרימה הם טיפה לטיפה כדי למנוע הכנסת בועות אוויר לתא הזרימה. - מערבבים 40 מיקרוליטר של תמיסת ליפוזום עם 1 מ"ל של מאגר שומנים. הזרקו 300 מיקרוליטר מתמיסת השומנים לתא הזרימה ודגרו למשך 5 דקות. חזור על ההזרקה והדגירה פעמיים.
הערה: החלף את צד הכניסה/יציאה להזרקה בכל פעם שמזרק מחובר לתא הזרימה. - שוטפים את החדר עם 3 מ"ל מאגר שומנים ודוגרים למשך 15 דקות.
הערה: תקופת דגירה זו יכולה לנוע בין 5 דקות לשעתיים, בהתאם לתזמון הניסויים, ללא הבדלים ניכרים בהרכבת הווילון הסופית. - מערבבים 40 מיקרוליטר של תמיסת מלאי אנטי-דיגוקסיגנין של 1 מ"ג/מ"ל עם 200 מיקרוליטר של מאגר שומנים. מזריקים 100 מיקרוליטר מהתמיסה ודוגרים למשך 10 דקות. חזור על ההזרקה והדגירה 1x לפני הכביסה עם 3 מ"ל של מאגר שומנים.
הערה: שלב זה נחוץ רק עבור וילונות dsDNA עם קשירה כפולה. דלג על שלב זה עבור וילונות dsDNA עם קשירה יחידה. - שטפו את החדר עם 3 מ"ל של מאגר BSA (40 מ"מ Tris-HCl [pH 8.0], 2 מ"מ MgCl2, 1 מ"מ DTT ו-0.2 מ"ג/מ"ל BSA).
- מערבבים 10 מיקרוליטר של תמיסת מלאי סטרפטווידין של 1 מ"ג/מ"ל עם 990 מיקרוליטר של מאגר BSA. יש להזריק כשתי זריקות נפרדות של 400 מיקרוליטר עם דגירה של 10 דקות לאחר כל זריקה.
- שטפו את החדר עם 3 מ"ל של מאגר BSA כדי להסיר עודפי סטרפטווידין.
- לדלל 25 ננוגרם של מלאי מצע dsDNA במאגר BSA של 1 מ"ל. הזרקו לאט את תמיסת ה-DNA 200 מיקרוליטר בכל פעם, ולאחר מכן דגירה של 5 דקות לאחר כל זריקה. כמות ה-DNA הדרושה עשויה להיות אופטימלית עבור סוגים ספציפיים של ניסויים.
- התקן את תא הזרימה על שלב המיקרוסקופ והתאם את המיקוד לפי הצורך.
- חבר את יציאות הקלט והפלט למערכת הזרקה לדוגמה המורכבת ממשאבת מזרק ושסתום מתג בלחץ גבוה.
- קשר פעמיים את ה-DNA לבסיסים (אורך קשירה של 12 מיקרומטר) על ידי שטיפה רציפה של החדר עם מאגר BSA במהירות של 0.8 מ"ל/דקה למשך 5 דקות.
הערה: ייתכן שיהיה צורך להתאים את קצבי הזרימה והזמנים כדי לבצע אופטימיזציה לאורכי קשירה כפולים שונים. שלב זה נחוץ רק עבור וילונות DNA עם קשירה כפולה. דלג על שלב זה עבור וילונות dsDNA עם קשירה יחידה. - הווילונות מוכנים כעת לניסויים.
6. מיחזור תאי זרימה
- טבלו את השקופיות באתנול למשך 30 דקות לפחות לפני השימוש בסכין גילוח כדי להסיר בזהירות את היציאות המיקרופלואידיות ואת החלקת הכיסוי. השלך כראוי את הכיסוי ונקה את כל הדבק שנותר מיציאות המיקרופלואידיות כדי שניתן יהיה לעשות בהם שימוש חוזר. קרצף את כל שאריות הדבק בשקופית.
הערה: הקפד לא לשרוט את האזור המעוצב במרכז השקופית. - שוטפים היטב את השקופיות במים מזוקקים כפולים וטובלים בתרכיז ניקוי קוורץ נוזלי אלקליין 2% למשך הלילה, תוך ערבוב.
- שטפו היטב את השקופיות במים מזוקקים כפולים וטבלו בתמיסת תרכיז ניקוי קוורץ נוזלי אלקליין 2% למשך שעה אחת תוך ערבוב ב-80 מעלות צלזיוס, ולאחר מכן סוניקציה למשך 15 דקות ב-45 מעלות צלזיוס.
- שוטפים היטב את השקופיות במים מזוקקים כפולים וטובלים ב-1 M NaOH למשך 30-60 דקות, תוך ערבוב.
- שטפו היטב את השקופיות במים מזוקקים כפולים, ולאחר מכן טבלו באלכוהול איזופרופיל וסוניקט למשך 15 דקות בטמפרטורה של 45 מעלות צלזיוס.
- שוטפים היטב את השקופיות באתנול וטובלים באתנול למשך שעה, תוך ערבוב.
- מכניסים את השקופיות לתנור ואקום למשך שעה לייבוש בחום של 135 מעלות צלזיוס. השקופיות מוכנות כעת להרכבה מחדש לתאי זרימה.
תוצאות
המתוארים לעיל הם הכנה, הרכבה והדמיה של וילונות dsDNA חד-קשורים וכפולים בהקשר של חקר אינטראקציות חלבון-DNA בחומרי ביניים לתיקון DNA. איור 1A מציג את כל הרכיבים בתא זרימה, בשכבות לפי סדר הרכבתם. איור 1B מתאר את הסכימה של וילון DNA עם קשר יחיד או כפול. שכבת שומנים דו-שכבתית משמשת לפסיבציה של פני תא הזרימה. וילון ה-DNA מורכב ממערך מקביל של מולקולות DNA הקשורות בקצה אחד לשומן ומיושרות במחסום הכרום, בכיוון מאונך לכיוון הזרימה. עבור וילונות DNA עם קשירה כפולה, הקצה השני של ה-DNA קשור לכן באמצעות אינטראקציות דיגוקסיגנין ואנטי-דיגוקסיגנין, כך שניתן לבצע הדמיה בהיעדר זרימה בזמן שה-DNA נשאר מורחב. ניתן לצבוע dsDNA בצבע פלואורסצנטי YoYo1 ולהמחיש אותו על ידי מיקרוסקופ TIRF. איור 2A מציג תמונה מייצגת של שדה רחב של וילון DNA כפול מוכתם ב-YoYo1.
סדרות תמונות זמן-lapse שנאספו מניסויי וילון DNA מנותחות בדרך כלל על ידי יצירת קימוגרף, המתווה תחילה את המיקום לאורך מולקולת ה-DNA על הציר האנכי לעומת הזמן על הציר האופקי עבור כל מולקולת DNA מעניינת ב-ImageJ. איור 2B הם קימוגרפים מייצגים המציגים את הכריתה של λ-DNA חד-קשור מוכתם YoYo1 (ירוק) על ידי מכונות כריתת השמרים GFP-Sgs1 (לא נראה), Top3-Rmi1 ו-Dna2 בנוכחות חלבון קושר DNA חד-גדילי RPA-mCherry (מג'נטה) ו-ATP. אות YoYo1 הולך לאיבוד עם הזמן כאשר dsDNA נכרת מהקצה החופשי על ידי Sgs1-Dna2. במקביל, אות mCherry מתמקם עם קצוות ה-DNA שבהם נוצר ssDNA כתוצאה מהכריתה. ניתן לחלץ מאפיינים רלוונטיים ביופיזיקליים של הכריתה, כגון מהירות ותהליכיות, על ידי כימות השיפועים של מסלולי אובדן האות YoYo1. איור 2C,D מציג את התפלגות המהירויות והתהליכיות, בהתאמה, של הכריתה על ידי Sgs1-Dna2.
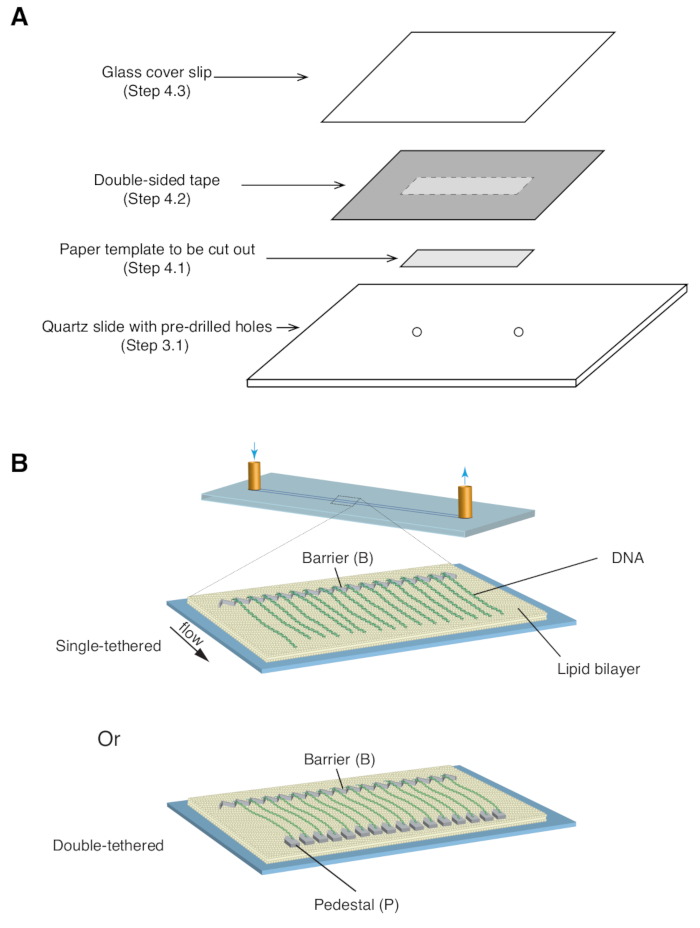
איור 1: סכמות של מכלול תאי זרימה ווילונות DNA. (A) המחשה מדורגת עבור הרכבת תאי זרימה. סרט דו צדדי מונח על גבי שקופית הקוורץ, ותבנית נייר משמשת לחיתוך תעלה מלבנית מהמרכז, האטומה בהחלקת כיסוי מלמעלה ליצירת תא. (B) סכמות של וילונות דנ"א מורכבים במלואם. הפאנל האמצעי מציג תא זרימה קשור-יחיד, בעוד שהפאנל התחתון מציג תא זרימה עם קשירה כפולה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
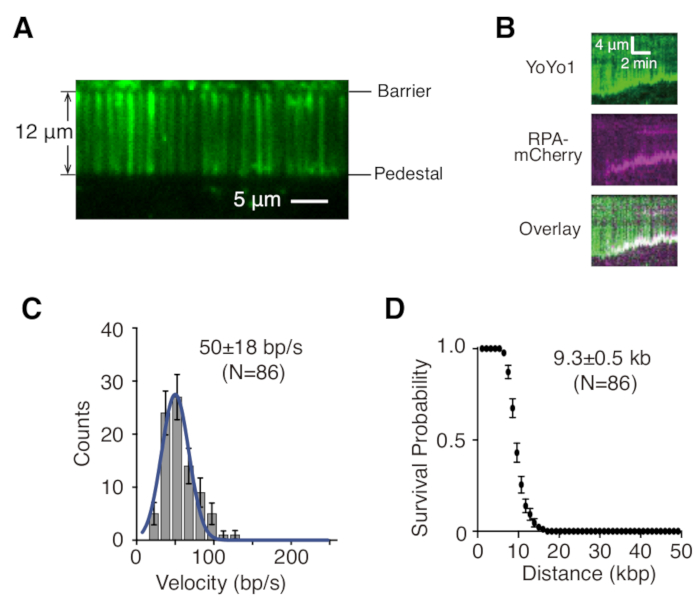
איור 2: הדמיה וניתוח של וילון DNA של מולקולה בודדת. (A) תמונה מייצגת בשדה רחב של וילון λ-DNA עם קשירה כפולה צבוע ב-YoYo1 (ירוק). (B) קימוגרפים מייצגים שמראים כריתה של dsDNA מוכתם ב-YoYo1 (ירוק) בנוכחות GFP-Sgs1 קשור מראש (לא נראה) כאשר רודפים אחריו עם Dna2, בנוכחות RPA-mCherry (מג'נטה) ו-2 מילימטר ATP. התפלגות מהירות (C) ותרשים תהליכי (D) של כריתת dsDNA על ידי Sgs1-Dna2, המתקבלים על ידי כימות הקצב וההיקף של אובדן האות YoYo1 לאורך זמן. לוחות (B), (C) ו-(D) מותאמים מ-Xue et al.19. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Discussion
וילונות DNA הוכיחו את עצמם כפלטפורמה רב-תכליתית לחקר תהליכי תיקון DNA שונים 13,14,15,16. מולקולות DNA שנשארות מורחבות ליד פני השטח לאורך כל הניסוי, באמצעות זרימת חיץ רציפה או קשירה כפולה לכן, מאפשרות הדמיית TIRFM של אינטראקציות חלבון-DNA על מתווכי תיקון ברמת המולקולה הבודדת. השיטה מספקת שיפור בתפוקה הניסויית וביכולת החיזוי, מכיוון שמאות מולקולות DNA מיושרות בצורה מסודרת במחסומים ננו-מיוצרים ולא מחוברות באופן לא ספציפי לפני השטח, מה שמועיל עוד יותר לעיבוד של מערכי נתונים גדולים.
ניתוח זהיר של סדרת התמונות המתקבלת מספק מדידות כמותיות של אינטראקציות חלבון-DNA (כלומר, אורך חיים של קשירה, מיקומים, סטוכיומטריה, קינטיקה של דיסוציאציה, שיעורי טרנסלוקציה, תהליכיות של חלבונים מוטוריים וקולוקליזציות ואינטראקציות בין חלבונים ו/או DNA). בהשוואה לטכניקות הדמיה אחרות של מולקולה בודדת הקושרות מולקולות DNA ישירות לפני השטח או משלבות הדמיה קונפוקלית עם מניפולציה בכוח במכשירים מסחריים, הפשרה העיקרית של וילונות DNA לתפוקה גבוהה יותר היא ביסוס הטכניקות הדרושות להגדרה ראשונית 13,14,15,16. עם זאת, לאחר הקמתה, היכולת לאסוף נתונים על מאות מולקולות בו זמנית בתא זרימה אחד מובילה לחיסכון משמעותי בזמן המושקע באיסוף נתונים.
כמו בשיטות הדמיה רבות אחרות של מולקולה בודדת, היכולת להכין משטח נקי מכתיבה במידה רבה את יכולתו של המשתמש להרכיב וילונות DNA בצורה אמינה. במקרה זה, משטחים נקיים דורשים לא רק להתחיל עם מגלשה נקייה אלא גם להקפיד לא להכניס בועות אוויר מיקרוסקופיות לתא תוך כדי ביצוע כל אחד מהחיבורים הנוזליים. ראשית, לעניין ניקיון המגלשות, השלבים המפורטים לעיל בסעיף 6 אמורים להסתפק כתהליך ניקוי שגרתי. מומלץ גם להשתמש בתמיסות ומאגרים מסוננים. שנית, יצירת חיבורי טיפה לטיפה במהלך הצמדת תא הזרימה למערכת הזרקת המאגר חיונית כדי למנוע הכנסת בועות. זה עשוי להיות מועיל לנקות את המים המזוקקים הכפולים המשמשים להכנת חוצצים. כמו כן, מומלץ להקפיד על כל תהליך הזרקה כדי לוודא שאין בועות. אם בועה מיקרוסקופית תוכנס לפני שהיא מגיעה לאזור המעוצב במרכז, ניתן לתקן אותה על ידי הזרקת מאגר מהיציאה המיקרופלואידית בצד הנגדי כדי לדחוף את הבועה בחזרה החוצה.
נקודת שיקול חשובה נוספת בכל ניסוי מבוסס פלואורסצנטי היא אות לרעש. מפתח להתבוננות בחלבונים פלואורסצנטיים בודדים (כלומר, GFP) שוכן גם במשטחים נקיים. לאחר שימוש ממושך, אגרגטים של חלבון מצטברים בדרך כלל במחסומי הכרום ובכנים. לכן, אם נצפה אות פלואורסצנטי חזק במחסומים לפני הוספת חלבונים פלואורסצנטיים, מומלץ לטפל במגלשות עם טבילה של 10 דקות בתמיסת פיראנה. פרק הזמן נשמר קצר כדי למנוע שחיקה של תכונות הכרום. לאחר שטיפת הפיראנה יש לבצע את הליך הניקוי הרגיל.
ננו-ייצור באמצעות ליתוגרפיה של קרן אלקטרונים יכול להיות מאתגר, בשל דרישותיו לציוד ומתקנים כמו גם המומחיות הקיימת במדעי החומרים. כחלופה, פותחה שיטה מבוססת ליתוגרפיה UV לייצור תכונות כרום עבור וילונות DNA20. כמו כן מסופק עם פרוטוקול זה דפוס בסיסי לקשירה כפולה של DNA למבדה ב-12 מיקרומטר (ראה מידע משלים). ניתן להתאים מספר היבטים של העיצוב של תבנית זו כך שיתאימו לצרכים ניסיוניים שונים. אלה כוללים את המרחק בין מחסומים לכנים, מרווח בין בארות סמוכות במחסום כדי לשלוט בצפיפות ה-DNA, ואת הצורה והגודל של הכן כדי לייעל את יעילות הקשירה הכפולה ולמזער את אי הוודאות באורכי קשירה כפולה. אתגרים מעשיים אחרים עשויים לכלול בחירה באסטרטגיות תיוג פלואורסצנטיות לחלבונים, מזעור הטיה בניתוח נתונים ידני ופיתוח סקריפטים לעיבוד נתונים יעיל יותר.
בעוד שהפרוטוקול המתואר כאן כולל מצעי dsDNA, נעשה שימוש נרחב גם בווילונות DNA המבוססים על ssDNA 13,14,15,16. הכנת מצעי ssDNA משתמשת בשכפול מעגל מתגלגל של פלסמיד M13 ssDNA עם פריימרים ביוטיניליים13. הרכבת וילון ssDNA דורשת גם שימוש באוריאה וחלבון קושר ssDNA RPA כדי לחסל מבנה משני ולהרחיב את מצע ssDNA13. ייתכן גם שניתן יהיה להרחיב את וילונות ה-DNA לשימוש בהכלאות RNA או DNA/RNA למחקרים של חלבונים המקיימים אינטראקציה עם RNA. כפי שזה נראה כרגע, פלטפורמת וילון ה-DNA יכולה להפיק תועלת מאוטומציה בתהליך ההרכבה כמפורט בסעיף 5, שרובו הזרקות חוזרות ונשנות של סט סטנדרטי של מאגרים. זה עשוי להוביל ליעילות משופרת בביצוע ניסויים מרובים בו זמנית. בנוסף, חבילה מאוחדת ומבוססת למידת מכונה של סקריפטים לניתוח תאיץ עוד יותר את עיבוד הנתונים עם הטיה מינימלית.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
המחברים מודים לחברים בעבר ובהווה במעבדת גרין על פיתוח, אופטימיזציה ודיון בפרוטוקול וילונות הדנ"א, כמו גם על הערות על כתב היד הזה. עבודה זו נתמכה על ידי מענקים מהקרן הלאומית למדע (MCB1154511 ל-E.C.G) והמכונים הלאומיים לבריאות (R01CA217973 ל-E.C.G).
Materials
| Name | Company | Catalog Number | Comments |
| nano pattern generation system software | JC Nabity Lithography Systems | N/A | NPGS |
| PFA Tubing, Natural 1/16" OD x .020" ID | IDEX | 1512L | |
| Poly(Methyl Methacrylate), Atactic (24.3K MW) | Polymer Source Inc. | P9790-MMA | |
| Quartz Microscope Slide | G. Finkenbeiner Inc. | N/A | 1" x 3" x 1 mm thick |
| Scanning Electron Microscope | FEI | N/A | Nova Nano SEM 450 |
| Scotch Double Sided Tape, 3/4 x 300 inches | 3M | N/A | |
| Sonicator | Misonix | S-4000 | For preparation of lipids |
| Sonicator | Branson | 1800 | For sonicating slides |
| Streptavidin from Streptomyces avidinii | Sigma-Aldrich | S4762 | Dissolved in 1X PBS to a final concentration of 1 mg/mL |
| Sulfuric Acid | Avantor - J.T.Baker | 9681-01 | |
| Syringe Pump | KD Scientific | 78-0200 | |
| Syringe, 500 µL, Model 750 RN SYR, Large Removable NDL, 22 ga, 2 in, point style 2 | Hamilton | 80830 | Reserve for use only with lipids |
| T4 DNA Ligase (400,000 units/ml) | NEB | M0202S |
References
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor Perspectives in Biology. 7, 016410(2015).
- Ferlay, J., et al. Estimating the global cancer incidence and mortality in 2018: globocan sources and methods. International Journal of Cancer. 144, 1941-1953 (2018).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Research. 18, 99-113 (2008).
- Michel, B., Baoubakri, H., Baharoglu, Z., LeMasson, M., Lestini, R. Recombination proteins and rescue of arrested replication forks. DNA Repair. 6, 967-980 (2007).
- Paques, F., Haber, J. E. Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 63, 349-404 (1999).
- Heyer, W. D., Ehmsen, K. T., Liu, J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 44, 113-139 (2010).
- Mason, J. M., et al. RAD54 family translocases counter genotoxic effects of RAD51 in human tumor cells. Nucleic Acids Research. 43, 3180-3196 (2015).
- Wold, M. S. Replication protein A: A heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annual Review of Biochemistry. 66, 61-92 (1997).
- Bianco, P. R. The tale of SSB. Progress in Biophysics and Molecular Biology. 127, 111-118 (2017).
- Hopfner, K. P., Gerhold, C. B., Lakomek, K., Wollmann, P. Swi2/Snf2 remodelers: hybrid views on hybrid molecular machines. Current Opinion in Structural Biology. 22, 225-233 (2012).
- Shah, P. P., et al. Swi2/Snf2-related translocases prevent accumulation of toxic Rad51 complexes during mitotic growth. Molecular Cell. 39, 862-872 (2010).
- Wright, W. D., Heyer, W. D. Rad54 functions as a heteroduplex DNA pump modulated by its DNA substrates and Rad51 during D loop formation. Molecular Cell. 53, 420-432 (2014).
- Ma, C. J., Steinfeld, J. B., Greene, E. C. Single-Stranded DNA Curtains for Studying Homologous Recombination. Methods in Enzymology. 582, 193-219 (2017).
- Qi, Z., et al. DNA sequence alignment by microhomology sampling during homologous recombination. Cell. 160, 856-869 (2015).
- Visnapuu, M. L., Greene, E. C. Single-molecule imaging of DNA curtains reveals intrinsic energy landscapes for nucleosome deposition. Nature Structural Molecular Biology. 16, 1056-1062 (2009).
- Kaniecki, K., De Tullio, L., Greene, E. C. A change of view: homologous recombination at single-molecule resolution. Nature Reviews Genetics. 19, 191-207 (2018).
- Redding, S., et al. Surveillance and Processing of Foreign DNA by the Escherichia coli CRISPR-Cas System. Cell. 163, 854-865 (2015).
- Lee, J. Y. Base triplet stepping by the Rad51/RecA family of recombinases. Science. 349, 977-981 (2015).
- Xue, C., et al. Regulatory control of Sgs1 and Dna2 during eukaryotic DNA end resection. Proceedings of the National Academy of Sciences USA. 116 (13), 6091-6100 (2019).
- Gallardo, I. F., Finkelstein, I. J. High-Throughput Universal DNA Curtain Arrays for Single-Molecule Fluorescence Imaging. Langmuir. 31 (37), 10310-10317 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved