Method Article
עריכת אפיגנום CRISPR בתאים אנושיים באמצעות טרנספקציה של DNA פלסמיד והעברת נוקלאופקציה של mRNA
In This Article
Summary
הפרוטוקול מתאר שיטות לעריכת אפיגנום מבוססת Clustered Regular Interspaced Short Palindromic Repeats (CRISPR) בקווי תאים אנושיים באמצעות טרנספקציה של DNA פלסמיד ונוקלאופקציה של mRNA.
Abstract
אפיגנטיקה מתייחסת לשינויים כימיים של חלבוני היסטון ו-DNA שיכולים לווסת את ביטוי הגנים. האפיגנום האנושי משתנה באופן דינמי במהלך התמיינות התאים והזדקנותם, ומחלות רבות קשורות לדפוס אפיגנום חריג. ההתקדמות האחרונה בקריספר הובילה לפיתוח כלים ניתנים לתכנות לעריכת שינויים אפיגנטיים במיקומים גנומיים ממוקדים, המאפשרים שכתוב מדויק של שינויים אפיגנטיים בתאים אנושיים. עורכי אפיגנום מבוססי CRISPR מסתמכים על Cas9 מת קטליטית יחד עם שינויים אפיגנטיים שבסופו של דבר מביאים לדיכוי מתוכנת או הפעלה של גנים ממוקדים בגנום של יונקים. בניגוד לשיטות עריכת גנום מסורתיות, עריכת אפיגנום אינה דורשת הפסקות DNA או שינויים ברצף הגנום האנושי ולכן משמשת כחלופה בטוחה יותר לשליטה בביטוי הגנים. בפרוטוקול זה, אנו מדגישים שתי שיטות שונות לביצוע עריכת אפיגנום בתיווך dCas9 בקווי תאים אנושיים באמצעות טרנספקציות DNA פלסמיד ונוקלאופקציה של mRNAs המקודדים עורכי אפיגנום CRISPR. אנו מדגימים עריכת אפיגנום ניתנת לתכנות לדיכוי חולף של גנים באמצעות הפרעות CRISPR (CRISPRi) ולהשתקת גנים לאורך זמן במשך שבועות רבים באמצעות CRISPRoff, מיזוג של dCas9 לתחום KRAB וקומפלקס דה נובו DNA methyltransferase. אנו מספקים גם הדרכה לגבי שיטות כמותיות למדידת עריכה מוצלחת של אפיגנום של גני מטרה ושיקולים מרכזיים באיזה כלי עריכת אפיגנום להשתמש, בהתאם לקריטריונים ניסיוניים.
Introduction
למרות שהתוכן הגנומי של כל תא בגופנו כמעט זהה, פרופיל השעתוק של כל סוג תא שונה מאוד. שינויים אפיגנטיים ב-DNA ובחלבוני היסטון הם מווסתים מרכזיים של ביטוי שעתוק. אוכרומטין פעיל שעתוק מסומן על ידי סימנים אפיגנטיים מובהקים בהשוואה להטרוכרומטין קומפקטי שאינו פעיל מבחינה שעתוק. לדוגמה, אזורים הטרוכרומטיים מוגדרים על ידי שינויים מדכאים בהיסטון, כולל טרימתילציה על ליזין 9 של היסטון 3 (H3K9me3), טרימתילציה על ליזין 27 של היסטון H3 (H3K27me3), ומתילציה של DNA על ציטוזינים ליד גואנינים (CpG) במקדמי גנים1. אזורים גנומיים של ביטוי גנים פעילים מוגדרים בדרך כלל על ידי אצטילציה של היסטון וטרימתילציה על ליזין 4 של היסטון 3 (H3K4me3)1.
מהפכת ה-Clustered Regular Interspaced Short Palindromic Repeats (CRISPR) יצרה שפע של כלים המאפשרים שינוי מתוכנת של רצפים גנומיים. טכנולוגיית CRISPR מבוססת על מנגנון הגנה פרוקריוטי המסוגל לבקע חומצות גרעין ברצפי מטרה הניתנים לתכנות. נוקלאזות CRISPR 2,3,4, עורכי בסיס 5,6 ועורכים ראשיים 7,8 יכולים לשנות את רצף ה-DNA של גנומים של יונקים באמצעות חיתוך DNA ותיקון של שברים אלה. למרות יעילותן, אסטרטגיות אלה יכולות לגרום לשבירות DNA באתרים מחוץ למטרה 9,10 ולמוטציות מבניות גנומיות בקנה מידה גדול 11,12,13,14,15. כלים חלופיים מבוססי CRISPR מאפשרים אפנון ניתן למעקב של הפעלה ודיכוי גנים מבלי לשנות את רצף ה-DNA הבסיסי. כלים אלה רותמים Cas9 חסר נוקלאז (dCas9), המאפשר קשירת DNA באתרי יעד המוכתבים על ידי רצף sgRNA, בשילוב עם חלבוני אפקטור המשנים את נוף הכרומטין 16,17,18. חלבוני אפקטור, כגון כותבים אפיגנטיים, קוראים ומחקים, יכולים להתמזג ישירות ל-dCas9 או לגייס אותם על ידי פיגום פפטיד המותך ל-dCas9, כגון SunTag, או פיגום RNA על ה-sgRNA, כגון מערכת MS2-MCP 16,17,18. דוגמאות לכלי בקרת שעתוק הניתנים לתכנות כוללות הפעלת CRISPR (CRISPRa)19,20,21 והפרעות CRISPR (CRISPRi)22,23. CRISPRa מתפקד על ידי גיוס ישיר של מנגנון השעתוק, הגדלת ביטוי הגנים של שעתוק היעד19. לעומת זאת, CRISPRi מדכא שעתוק על ידי יצירת H3K9me3, סימן אפיגנטי מדכא22.
ההתקדמות בעריכת אפיגנום אפשרה שימוש נרחב בכלים אלה בתחומים מדעיים. היתוך של תחומי אפקטור וחלבונים שונים הרחיבו את ערכת הכלים של עורכי אפיגנום זמינים 18,24,25,26,27,28,29,30. בנוסף, עורכי אפיגנום משמשים לפענוח התפקידים של שינויים אפיגנטיים 31,32,33,34,35,36, אפקטורים 37,38,39 ומוטציות אפקטור 28,40,41 בוויסות גנים. באופן ספציפי, CRISPRi ו-CRISPRa משמשים במסכי גנומיקה פונקציונליים למגוון תהליכים ביולוגיים, כולל הישרדות תאים42 וגורל תא 43,44,45,46,47. יתר על כן, עריכת אפיגנום טומנת בחובה פוטנציאל טיפולי להנדסת תאים ex vivo וטיפולים in vivo 18.
כאן, אנו מתארים שיטות ליישום שני עורכי אפיגנום מבוססי dCas9 לדיכוי שעתוק הניתן לתכנות בשורות תאים אנושיים: CRISPRi22 ו-CRISPRoff48. CRISPRi הוא היתוך של dCas9 לתחום ה-KRAB המדכא מחלבון אצבע אבץ כגון ZNF10 (KOX1) ו-ZIM322,23. כאשר CRISPRi מכוון למקדם גן מסוים, תחום ה-KRAB מגייס כותב H3K9me3, SETDB1, כדי לדכא את גן המטרה (איור 1A). כאשר CRISPRi מתבטא באופן ארעי, ה-H3K9me3 המבוסס בלוקוס המטרה אינו נשמר, וביטוי הגנים משוחזר לאורך זמן32,48. כדי לבצע נוקדאונים יציבים באמצעות CRISPRi כמו ביישומי גנומיקה פונקציונליים, ביטוי מכונן של CRISPRi בתאים יחד עם ה-sgRNA הוא חיוני. לאחרונה, CRISPRoff תוכנן לתכנת עריכת אפיגנום תורשתית48. CRISPRoff הוא היתוך חלבון יחיד של dCas9 לתחום KRAB וקומפלקס דה נובו DNA methyltransferase, DNMT3A ו-DNMT3L. פולס חולף של CRISPRoff בתאים אנושיים מתכנת שקיעה של H3K9me3 ומתילציה של DNA בגנים הממוקדים, מה שמוביל לדיכוי ארוך טווח של גני מטרה על ידי שמירה על מתילציה של DNA ו-H3K9me3 (איור 1B)48. יתר על כן, ניתן לבטל עריכות אפיגנום. לדוגמה, גן שהושתק ביציבות על ידי CRISPRoff יכול להיות מופעל מחדש על ידי TET1-dCas9 שיכול להסיר אנזימטית את סימני המתילציה של ה-DNA במיקומי מטרה49.
פרוטוקול זה יפרט שתי שיטות אספקה לביטוי חולף של עורכי אפיגנום: טרנספקציה של DNA פלסמיד ונוקלאופקציה של mRNA. בנוסף, אנו מתארים כיצד להשתמש בזרימה ציטומטרית כדי להעריך את יעילות עריכת האפיגנום בשני גנים אנדוגניים, CLTA ו-CD55. ניתן להתאים שיטות אלה וליישם אותן בניסויי עריכת אפיגנום אחרים באמצעות עורכים נוספים או להשתמש בהן למיקוד גנים שונים.
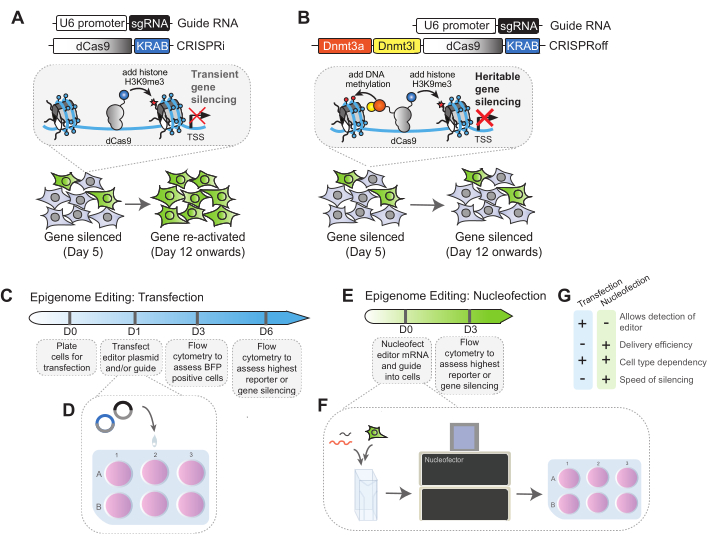
איור 1: סכמטי של מנגנון העריכה וזרימת העבודה של אפיגנום CRISPRi ו-CRISPRoff. (A) סכמות ליניאריות של הטרנסגן sgRNA ועורך האפיגנום CRISPRi. התוספת של CRISPRi ו-sgRNA מאפשרת להוסיף את סימן ההיסטון המדכא H3K9me3 כדי להשתיק את מיקום המטרה. רמת ההשתקה הגבוהה ביותר מושגת בשלב מוקדם לאחר הוספת CRISPRi, ומטרת הגן מופעלת מחדש בדרך כלל לאחר מספר מעברים. (B) סכמות ליניאריות של הטרנסגן sgRNA ועורך האפיגנום של CRISPRoff. התוספת של CRISPRoff לתאים המכוונים לגן מעניין מובילה לתוספת של H3K9me3 מדכא יחד עם מתילציה של DNA באתרי CpG כדי להשתיק את גן המטרה. השתקה על ידי CRISPRoff היא תורשתית - רמת ההשתקה הגבוהה מושגת בשלב מוקדם במהלך הטרנספקציה ונמשכת על פני חלוקות תאים מרובות. (C) סקירה כללית של ציר הזמן לעריכת אפיגנום בשיטת טרנספקציה. ביום 0, התאים מצופים לטרנספקציה. ביום הראשון, ניתן להכניס את העורך והמדריך פלסמיד לתאים באמצעות טרנספקציה. ביום השלישי, התאים יוערכו לביטוי BFP באמצעות ציטומטריית זרימה. אחוז ה-BFP משמש כגורם מנרמל לקביעת יעילות ההשתקה הסופית של הניסוי עבור כל מצב. ביום השישי ואילך, התאים מנותחים להשתקת המדווח המעניין מכיוון שרמת ההשתקה הגבוהה ביותר תושג ביום זה. (D) סקירה כללית של שיטת טרנספקציה שבה עורך אפיגנום ופלסמידים sgRNA מתווספים לתאים בצורה טיפתית. (E) סקירה כללית של ציר הזמן לעריכת אפיגנום באמצעות שיטות נוקלאופקציה. בפרוטוקול זה, mRNA הוא נוקלאופקציה לתאים ביום 0. התאים מוערכים להשתקה ביום השלישי לאחר נוקלאופקציה באמצעות ניתוח ציטומטריית זרימה. (F) סקירה כללית של פרוטוקול הנוקלאופקציה. כמויות מתאימות של תאים ו-mRNA מעורבבות ומתווספות לקובטות נוקלאופקטור. אם ה-sgRNA מוכנס על ידי נוקלאופקציה, ניתן להוסיף אותו גם לתערובת זו. הקובטות מוכנסות לתוך הגרעין וקודי דופק מתאימים משמשים להחדרת ה-mRNA לתאים. לאחר נוקלאופקציה, התאים מצופים ומועברים לניתוח בימים מאוחרים יותר. (G) השוואה בין טרנספקציה של פלסמיד לאסטרטגיות נוקלאופקציה של mRNA לעריכת אפיגנום. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Protocol
הערה: משלים File 1 מכיל פרטים על עיצוב sgRNA, שיבוט ויצירת קו תאים של הנתונים המייצגים שלנו. מקטע התוצאות המייצגות מפרט גם הצעות לפקדים.
1. טרנספקציה של פלסמידים המבטאים עורך אפיגנום לתאים HEK293T
הערה: פרוטוקול זה מתאר אספקה של פלסמידים מקודדי CRISPR לתאי HEK293T. הנדסנו תאים לבטא sgRNA (Addgene 217306) המכוון לאזור המקדם של CLTA, גן לא חיוני, המתויג באופן אנדוגני עם GFP. תאי CLTA-GFP HEK 293T מקורם במחקר קודם50. בדוגמה זו, עורכי האפיגנום מתמזגים ישירות לחלבון פלואורסצנטי כחול (BFP), המאפשר לנו לכמת את יעילות הטרנספקציה ומבטיח הערכה מדויקת של תנאי הניסוי. יעילות הגישה מודגמת על ידי השתקה של CLTA-GFP, אותה ניתן למדוד כמותית ברמת החלבון בתאים בודדים באמצעות ציטומטריית זרימה.
- תרבית תאים
- שמור על תאי HEK293T ב-DMEM עם גלוקוז גבוה עם 10% FBS ו-1x פניצילין-סטרפטומיצין-גלוטמין. מעבר התאים כל 2-3 ימים, וודא שהם נשארים במפגש של 60%-70%.
הערה: כדי להבטיח יעילות טרנספקציה אופטימלית, קריטי לשמור על מפגש תאים מתחת ל-70%, אותו ניתן לנטר מדי יום במיקרוסקופ.
- שמור על תאי HEK293T ב-DMEM עם גלוקוז גבוה עם 10% FBS ו-1x פניצילין-סטרפטומיצין-גלוטמין. מעבר התאים כל 2-3 ימים, וודא שהם נשארים במפגש של 60%-70%.
- יום 0: זריעת תאים HEK293T לטרנספקציה
- יום לפני הטרנספקציה, ספור תאים עם מונה תאים אוטומטי באמצעות צביעה חיה/מתה כחולה של טריפן וזרע ~ 1.5 x 104 תאים חיים לבאר בצלחת של 96 בארות. שמור על הנפח הסופי של כל באר על 200 מיקרוליטר. התאים צריכים להגיע למפגש של 60%-70% למחרת.
הערה: ניתן לבצע זאת גם בצלחות של 24 בארות או 6 בארות ביעילות. ראה טבלה 1 למספרי תאים וכמות DNA עבור טרנספקציה.
- יום לפני הטרנספקציה, ספור תאים עם מונה תאים אוטומטי באמצעות צביעה חיה/מתה כחולה של טריפן וזרע ~ 1.5 x 104 תאים חיים לבאר בצלחת של 96 בארות. שמור על הנפח הסופי של כל באר על 200 מיקרוליטר. התאים צריכים להגיע למפגש של 60%-70% למחרת.
- יום 1: טרנספקציה של פלסמידים CRISPRoff ו-CRISPRi לתאי HEK29T
- 150 ננוגרם של פלסמיד עורך האפיגנום (Addgene 167981) עבור כל באר בנפח כולל של 3 מיקרוליטר. ניתן להכין דילול פלסמיד זה בצינורות רצועת PCR.
הערה: אם עורך האפיגנום והפלסמיד sgRNA עוברים טרנספקט משותף, אנו ממליצים לנסות יחס מולארי של 3:4 של פלסמידים של עורך:sgRNA. ייתכן שיהיה צורך לבצע אופטימיזציה של היחס כדי להניב את היעילות הגבוהה ביותר של עריכת אפיגנום. - יש לחמם מראש את מגיב הטרנספקציה ואת המדיום החיוני המינימלי לטמפרטורת החדר כ-30-60 דקות לפני הטרנספקציה. מערבבים 0.6 מיקרוליטר של מגיב טרנספקציה ו -20 מיקרוליטר של מדיום חיוני מינימלי לבאר ודוגרים בטמפרטורת החדר למשך 15 דקות.
הערה: ניתן ליצור תערובת מאסטר של מגיב טרנספקציה ומדיום חיוני מינימלי אם מבצעים טרנספקציות מרובות. - הוסף 20.6 מיקרוליטר של מגיב הטרנספקציה ותערובת בינונית חיונית מינימלית לכל צינור רצועת PCR המכיל 3 מיקרוליטר של DNA פלסמיד. מניחים לשבת בטמפרטורת החדר למשך 15 דקות.
- מוסיפים את התערובת לאט לצלחת התאים הזרעים ומקישים בעדינות על הצלחת כדי לפזר את התערובת. כלול בקרה לא מועברת כדי לספק בסיס מתאים להשוואה בניתוחים במורד הזרם.
הערה: בעת הוספת תערובת הטרנספקציה לתאים זרעים, חיוני לעשות זאת בצורה טיפתית ובעדינות כדי למזער את ההפרעה, מכיוון שהדבר עלול להשפיע לרעה על יעילות ההעברה.
- 150 ננוגרם של פלסמיד עורך האפיגנום (Addgene 167981) עבור כל באר בנפח כולל של 3 מיקרוליטר. ניתן להכין דילול פלסמיד זה בצינורות רצועת PCR.
- יום 2: בדיקת הצלחת הטרנספקציה
- בדוק תאים במיקרוסקופ בהגדלה של פי 10 כדי לקבוע אם הם חיוביים ל-BFP, מכיוון שהדבר מעיד על טרנספקציה מוצלחת. ודא שהתאים אינם מופרעים במהלך תהליך התצפית.
- יום 3: פיצול צלחת 96 הבארות והערכת יעילות הטרנספקציה באמצעות סמן BFP
הערה: טרנספקציה מוצלחת אמורה להניב לפחות 30% מהתאים כחיוביים ל-BFP. עם זאת, עם dCas9, CRISPRi ו-CRISPRoff, אנו בדרך כלל מזהים יותר מ-60% תאים חיוביים ל-BFP יומיים לאחר הטרנספקציה. יעילות הטרנספקציה נמצאת בקורלציה שלילית עם גודל הפלסמיד, כך שעורכי אפיגנום שונים עשויים להניב יעילות טרנספקציה שונה.- יש לחמם מראש טריפסין ו-DMEM עם 10% FBS ו-1x פניצילין-סטרפטומיצין-גלוטמין לטמפרטורת החדר למשך כ-30-60 דקות לפני השימוש.
- שאיבת מדיה מהבארות; היזהר לא להסיר תאים המחוברים לתחתית. הוסף 50 מיקרוליטר טריפסין לבאר. יש לדגור במשך 2-3 דקות בחום של 37 מעלות צלזיוס.
הערה: זה לא יוצא דופן לצפות בכמה תאים צפים, במיוחד לאחר טרנספקציה עם CRISPRoff. אנו משערים שמוות תאים זה נובע מרעילות של ביטוי יתר של DNMT3A-3L. - מעכב טריפסין על ידי הוספת 150 מיקרוליטר של DMEM מלא לכל באר. פיפטה למעלה ולמטה כדי להשעות מחדש תאים ולפצל תאים למפגש ביום 5 (~20 מיקרוליטר של תערובת תאים ב-200 מיקרוליטר של מדיה של צלחת של 96 בארות).
- השתמש ב-50 מיקרוליטר של תאים תלויים עבור ציטומטריית זרימה כדי להעריך את יעילות הטרנספקציה.
2. נוקלאופקציה של mRNA של עורך אפיגנום לתאי K-562
הערה: סעיף זה מפרט את תהליך הגרעין של CRISPRoff mRNA לתאי K-562. לשם הפשטות, הנדסנו מראש את תאי K-562 כדי לבטא באופן קונסטיטוטיבי sgRNA המכוון למקדם הגן האנדוגני CD55 (Addgene 217306). להעברת mRNA של CRISPRoff ישירות לתאים יש פוטנציאל להפחית את הרעילות התאית המלווה גישות מבוססות DNA פלסמיד תוך השגת יעילות השתקת גנים דומה. בנוסף, ניתן להשתמש בנוקלאופקציה כדי להציג מבני עורך אפיגנום בקווי תאים שמאתגרים להעביר ביעילות, כגון K-562s.
- תרבית תאים
- לפני הנוקלאופקציה, שמור על תאי K-562 בבקבוק עם RPMI עם 10% FBS ו-1x פניצילין-סטרפטומיצין-גלוטמין. לעבור את התאים מדי יום, ולוודא שהם יישארו במפגש של 60%-70%.
- ביום הגרעין יש להפשיר ולערבב בעדינות את ה-CRISPRoff mRNA בצינור מיקרו-צנטריפוגה סטרילי נטול RNase ולאחסן את ה-mRNA על קרח. השתמש ב-2 מיקרוגרם של mRNA לכל 2.0 x 105 תאים בכל באר של נוקלאוקובט רצועה. ודא שנפח ה-mRNA אינו עולה על 10% מנפח הנוקלאופקציה הכולל (22 מיקרוליטר).
הערה: ניתן לבצע נוקלאופקציה גם בקובטות גדולות יותר עם יותר תאים ו-mRNA. אנו מציעים לשנות את קנה המידה של כמות ה-mRNA באופן פרופורציונלי למספר התאים שתוסיפו. לדוגמה, בעת ביצוע נוקלאופקציה בקובט גדול על 1 x 106 תאים, אנו מציעים להשתמש ב-10 מיקרוגרם של mRNA, עם נפח נוקלאופקציה כולל של 100 מיקרוליטר. בנוסף, ניתן לייעל את המינון של ה-mRNA כדי להבטיח יעילות עריכה גבוהה. לעתים קרובות אנו משתמשים ב-2-5 מיקרוגרם של mRNA עורך עבור כל 2.0 x 105 תאים לקבלת תוצאות יעילות. יש להסיר נוקלאוטידים חופשיים שלא שולבו במהלך סינתזת mRNA לפני הכמות. - הכן את תמיסת הנוקלאופקטציה על פי הוראות היצרן וחמם לטמפרטורת החדר למשך 15 דקות לפני הנוקלאופקטיה.
הערה: תמיסת הנוקלאופקטור עשויה להיות שונה או שיש לבצע אופטימיזציה לסוגי תאים שונים. - קצור וספור תאי K-562 באמצעות מונה תאים אוטומטי עם צביעה חיה/מתה כחולה טריפאן. עבור נוקלאופקציה בקובט רצועה, aliquot ~ 2.0 x 105 תאים לדגימה לתוך צינור מיקרו-צנטריפוגה סטרילי.
- צנטריפוגה של התאים ב-500 x גרם למשך 5 דקות בטמפרטורת החדר, ואז השליכו את הסופרנטנט. שטפו את התאים 1x עם PBS בטמפרטורת החדר ב-500 x גרם למשך 5 דקות, ואז השליכו את הסופרנטנט.
- חשב את כמות תמיסת הנוקלאופקטור להשעיית תאים על ידי הפחתת נפח ה-mRNA מ-22 מיקרוליטר.
- הוסף תמיסת תא ל-2 מיקרוגרם של CRISPRoff mRNA (נפח נקבע בשלב 2.1.2) והעבר את התמיסה לקובטה, תוך היזהרות לא ליצור בועות, מכיוון שהדבר עלול לפגוע ביעילות הנוקלאופציה. הקש בעדינות על הנוקלאוקובטה כדי לוודא שהתאים נמצאים בתחתית.
- גרעין את התאים באמצעות מערכת 4D-Nucleofector עם קוד הדופק המתאים. קוד FF-120 מומלץ לתאי K-562.
הערה: ייתכן שיהיה צורך לבצע אופטימיזציה של קוד הדופק עבור סוגי תאים שונים. התייעץ עם היצרן אם יש צורך באופטימיזציה נוספת. - לאחר נוקלאופקציה, הוסף 80 מיקרוליטר של מדיה RPMI לכל באר נוקלאוקובט קטנה ותן לתאים לשבת ב-37 מעלות צלזיוס למשך 15 דקות.
- העבירו את תרחיף התא לבאר של צלחת בת 24 בארות המכילה 400 מיקרוליטר של מדיה RPMI מחוממת מראש (נוקלאוקובט קטן).
הערה: לאחר נוקלאופקציה, התאים שבירים במיוחד ויש להעבירם מהנוקלאוקובט לבארות תרבית בעדינות. - לאחר היום השני לאחר נוקלאופקציה: עקוב אחר פרוטוקול צביעת סמן פני השטח עם נוגדן CD55 כמתואר בסעיף 3 ומדוד את הקרינה של CD55 APC על ציטומטר זרימה כדי לבחון את יעילות עריכת האפיגנום.
הערה: כדי להעריך את הצלחת הנוקלאופקציה, אנו ממליצים לבצע נוקלאופקציה עם GFP או mCherry mRNA. יומיים לאחר הנוקלאופקציה, בצע ציטומטריית זרימה של תאים אלה כדי לאשר נוקלאופקציה מוצלחת. בדרך כלל אנו מזהים 90% עד 100% מהתאים הללו המבטאים GFP או mCherry. חלופה היא לבצע נוקלאופקציה עם CRISPRi mRNA, הידוע כמשתיק חזק גני מטרה רבים. בעת ביצוע זרימה ציטומטרית ביום השני, דגימות שטופלו ב-CRISPRi אמורות להראות השתקת גן מטרה בלמעלה מ-90% מהתאים.
3. מכתים סמן משטח
הערה: סעיף זה מפרט את כימות רמות חלבון CD55 לאחר עריכת אפיגנום בתאי K-562. אנו מכמתים את הירידה בביטוי CD55 בתאים בודדים באמצעות צביעת נוגדנים וזרימה ציטומטרית (ראה סעיף 4 להלן) כדי להעריך את יעילות הנוקדאון בתיווך CRISPRoff. ניתן להשתמש בטכניקות נוספות, כולל PCR כמותי של שעתוק הפוך או כתמים מערביים, כדי לאשר את רמת הנוקדאון הן ברמת התעתיק והן ברמת החלבון.
- ספרו תאים באמצעות מונה תאים אוטומטי והוסיפו כ-5.0 x 105 עד 1.0 x 106 תאים לתוך צינור מיקרו-צנטריפוגה ותאי צנטריפוגה ב-500 x גרם למשך 5 דקות.
הערה: ניתן להשתמש ביותר או פחות תאים לצביעה, אולם ייתכן שיהיה צורך להתאים את ריכוז הנוגדנים בהתאם. - השליכו את ה-supernatant תוך הקפדה לא להפריע לגלולה והוסיפו 500 מיקרוליטר PBS והשעו מחדש את הגלולה בעדינות.
- צנטריפוגה את התאים שוב ב -500 x גרם למשך 5 דקות. השליכו את הסופרנטנט והוסיפו את הנוגדן בריכוז הרצוי המדולל ב-PBS. עבור צביעת CD55, השתמש ב-1 מיקרוליטר של CD55 אנטי-אנושי מטוהר (מלאי 0.5 מ"ג/מ"ל) ב-49 מיקרוליטר של PBS.
- פיפטה בעדינות את התאים ואפשר להם לדגור במשך 25 דקות במקום חשוך בטמפרטורת החדר. לאחר הדגירה, סובבו את התאים שוב ב-500 x גרם למשך 5 דקות והשליכו את הסופרנטנט.
- השעו מחדש את הגלולה עם 500 מיקרוליטר PBS כדי לשטוף עודפי נוגדנים. צנטריפוגה של התאים ב-500 x g למשך 5 דקות והשליכו את ה-PBS supernatant. השעו מחדש ב-100 מיקרוליטר עד 200 מיקרוליטר של PBS טרי והוסיפו לצלחת של 96 בארות לניתוח ציטומטריית זרימה.
4. ציטומטריית זרימה
הערה: פרוטוקול זה נכתב לשימוש במנתח תאים BD FACSymphony A1. הפרטים עשויים להשתנות בהתאם לציטומטרי הזרימה שבו אתה משתמש. אנו ממליצים לעיין במדריך למשתמש של המכשיר שבו אתה משתמש לקבלת פרטים.
- לפני הפעלת הציטומטריה, בדוק שמיכל הפסולת אינו מלא ושיש הרבה נוזל נדן. הפעל את מתג ההפעלה הראשי (ירוק) שנמצא ליד נוזל הנדן.
- הפעל את קורא הלוחות באמצעות המתג האחורי. לאחר מכן, הפעל את המכונה באמצעות הכפתור הירוק בצד ימין וודא שהמכונה מוגדרת לפונקציית קורא הצלחות של 96 בארות.
- הפעל את המחשב והיכנס. פתח את התוכנה והיכנס באמצעות שם המשתמש והסיסמה שלך.
- הגדר את הניסוי. צור ניסוי חדש על-ידי בחירה באפשרות ניסוי > ניסוי חדש. שנה את שם הניסוי על ידי לחיצה כפולה על השם הנוכחי.
- הוסף צלחת על-ידי לחיצה על Experiment > New Plate. בחר את פורמט הבאר לשימוש. כדי להוסיף בארות ספציפיות להפעלה, לחץ על מיקום הבאר בצלחת ולאחר מכן לחץ על המזרק הכחול (כעת יש להדגיש את הבאר בכחול). בחר מספר בארות בו-זמנית להוספה, במידת הצורך.
- הוסף שמות ספציפיים לכל באר על ידי לחיצה ימנית על דגימה בצד ימין של תצוגת הצלחת. לאחר מכן בחר פריסה ניסיונית. פעולה זו תפתח דף המפרט את כל הבארות שבחרת. כדי לשנות את שמות הבארות, לחץ פעם אחת והקלד את השם הרצוי. לחץ על אישור בסיום.
- הוסף עלילות ושערים מתאימים לניסוי בגיליון העבודה הגלובלי. אנו ממליצים תחילה לשרטט אזור פיזור קדימה (FSC-A) לעומת אזור פיזור צדדי (SSC-A). לאחר מכן צור שער שרירותי לתאים חיים כדי לחדד מאוחר יותר על ידי לחיצה על הכלי Polygon.
- לחץ לחיצה ימנית על שער זה ולחץ על הסתעפות. זה ייצור תרשים חדש המציג רק תאים מאותו שער תא חי.
- שנה את הציר של תרשים חדש זה ל- FSC-A לעומת גובה פיזור קדימה (FSC-H) וצור שער עבור תאים בודדים.
- אם מבטאים באופן מכונן את ה-sgRNA בתאים, וה-lentivirus מקודד גם לחלבון פלואורסצנטי, אז קדחו את שער התא הבודד הזה ובצעו שער נוסף לביטוי של אותו חלבון כפרוקסי לביטוי sgRNA (דוגמה: PE-CF594-A לעומת FSC-A).
- בשער ההתקנה הסופי (עבור תאים בודדים או עבור תאים המבטאים sgRNA), לחץ באמצעות לחצן העכבר הימני ובחר הסתעפות. צור שער לבדיקת הביטוי של הגן המדווח (לדוגמה: BB515-A לעומת FSC-A).
- אם אתה משתמש בתאים שעברו טרנספיקציה ויש לך שיטה לבדיקת יעילות טרנספקציה כגון ביטוי BFP, לחץ לחיצה ימנית על עלילת ביטוי המדווח ולחץ על שכפל. שנה את הצירים ל-BV421-A לעומת FSC-A. צור שער שרירותי עבור תאים חיוביים ל- BFP.
- הסר את כל הפרמטרים שאינם בשימוש בכרטיסייה פרמטרים בתצוגת הציטומטר.
- כדי להגדיר את המכונה ולהכין אותה להפעלה, לחץ על הפעל בלוח הבקרה של ציטומטר הזרימה. הכפתור אמור להופיע בירוק. בתוכנה, עבור אל HTS בחלונית העליונה ולאחר מכן Prime. לאחר שתסיים, הוסף את הצלחת, וודא שהיא ממוקמת היטב במכונה והמכסה כבוי.
- כדי לפעול, בחר את כל הבארות להפעלה וודא שהפקדים מוגדרים כראוי. בחר היטב על ידי לחיצה כפולה, שנה את נפח הדגימה ל-10 מיקרוליטר ולחץ על השגת נתונים בלוח המחוונים של הרכישה. פעולה זו תפעיל את הדוגמה אך לא תתעד את הנתונים. כאשר דגימה זו פועלת, כוונן את מתחי הלייזר לפי הצורך. בנוסף, החלף את השערים.
הערה: אנו ממליצים להריץ דגימה שלילית לכל הצבעים הנמדדים וחיובית לכל הצבעים כדי לוודא שפרמטרי הלייזר והשער טובים. - לאחר בדיקת הגדרות הלייזר, לחץ על הדגימות בלוח הצלחת וודא שערכי הריצה מוגדרים כרצונך.
- לחץ על לוח הפעלה כדי לרוץ בלוח המחוונים של הרכישה ולתעד נתונים מכל הבארות. בסיום, נקה את המכונה על ידי הסרת הצלחת.
- לצלחת חדשה של 96 בארות, הוסף 10% אקונומיקה לארבע השורות העליונות ו-H2O לארבע הבארות שמתחת. עבור אל HTS > Clean. לאחר שתסיים, סגור את התוכנית וכבה את הכפתורים בסדר הפוך שבו הם הופעלו בכפתור צד ירוק > קורא צלחות > מתג ירוק על ידי בזבוז.
- מוסיפים 20% אקונומיקה לצלחת 96 בארות עם תאים וזורקים.
5. ניתוח נתונים
הערה: שיטה זו מתארת אסטרטגיות שער ועיבוד נתונים כדי לכמת עריכת אפיגנום על ידי ציטומטריית זרימה. אסטרטגיית השער מיוצגת חזותית לצד תרשימים לדוגמה שנוצרו מניתוח הנתונים באיור 2 ובאיור 3.
- הגדרת שער FlowJo
- טען את כל קבצי FSC ל-FlowJo על ידי גרירה ושחרור שלהם לגליון עבודה חדש.
- לחץ על כל הדוגמאות כדי להתחיל ליצור שערים. זה יבטיח שניתן יהיה ליישם את השערים בקלות על כל הדגימות.
- פתח דגימת בקרה (כגון לא מועבר או לא גרעיני) על ידי לחיצה כפולה על קובץ זה.
- צור שער עבור תאים חיים בתרשים FSC-A לעומת SSC-A באמצעות כלי המצולע. ברשימה של כל הדגימות, הכרטיסיה תאים חיים אמורה להופיע כעת מתחת לשם המדגם. לחץ לחיצה ימנית על שער זה ובחר העתק ניתוח לקבוצה כדי להחיל שער זה על כל הדגימות (איור 2A, איור 3A).
- לחץ פעמיים על שער התאים החיים כדי להסתעף לתאים החיים. שנה את הצירים ל-FSC-A לעומת FSC-H (איור 2B, איור 3B).
- צייר מצולע לשער עבור תאים בודדים בלבד. החל שער זה על כל הדגימות כפי שנעשה בסעיף 4 (איור 2B, איור 3B).
הערה: אם מבטאים באופן קונסטרוקטיבי מבנה sgRNA עם מדווח פלואורסצנטי, בצע שער נוסף על צבע זה כך שהניתוח יתמקד רק בתאים המבטאים את ה-sgRNA. דוגמה לכך תהיה יצירת שער עבור תאים חיוביים ל-mCherry (PE-CF594-A לעומת FSC-A) אם התאים מכילים sgRNA מעמוד השדרה של pLG1 (איור 2C, איור 3C). - לחץ פעמיים על שער ההתקנה האחרון (תאים בודדים או תאים המבטאים מדריך).
- אם מבצעים טרנספקציה של פלסמיד ועורך האפיגנום מקודד עבור היתוך BFP, צור שער עבור תאים חיוביים של BFP (דוגמה: BV421-A לעומת FSC-A) עבור דגימות היום השני (איור 2D).
- צור שער עבור ביטוי מדווח (דוגמה: BB515-A לעומת FSC-A לשער עבור תאים שליליים GFP) והחל שער זה על כל הדגימות (איור 2E, איור 3D).
- ניתוח טרנספקציה
- לאחר ביצוע הגדרת השער, לחץ על עורך טבלאות בחלונית העליונה. גרור אוכלוסיות לניתוח (דוגמאות: BFP חיובי, GFP שלילי).
- בחלונית עורך הטבלאות, לחץ על צור טבלה. העתק נתונים אלה לגיליון אלקטרוני כדי לבצע חישובי נורמליזציה.
- עיבוד נתונים
- הפחיתו את מספר התאים השליליים של המדווח (לדוגמה: GFP שלילי) בדגימת הבקרה מכל הדגימות האחרות. ייתכן שיש השתקה מסוימת של הרקע של המדווח, כך שזה מסיר את רעשי הרקע האלה מהנתונים.
- כל הערכים ינורמלו לאחוז התאים החיוביים ל-BFP (תאים שהועברו בהצלחה) שנמדדו ביום השני. כדי לבצע נורמליזציה זו, קח את הערך, חלק באחוז התאים החיוביים ל- BFP ביום השני והכפל ב- 100. חישוב זה מניב את הערך המנורמל של יעילות הטרנספקציה של התאים הערוכים הנתונים מוכנים כעת לשרטוט. אנו ממליצים על גרפים קוויים לאורך מהלך זמן עריכת אפיגנום כפי שמתואר באיור 2F.
- ניתוח נוקלאופקציה
- לאחר ביצוע הגדרת השער, לחץ על עורך טבלאות בחלונית העליונה. גרור אוכלוסיות לניתוח (דוגמאות: APC שלילי).
- בחלונית עורך הטבלאות, לחץ על צור טבלה. העתק נתונים אלה לעורך גיליונות אלקטרוניים כדי לבצע חישובי נורמליזציה.
- עיבוד נתונים
- הפחיתו את מספר התאים השליליים של המדווח (לדוגמה: APC שלילי) בדגימת הבקרה מכל הדגימות האחרות. ייתכן שיש השתקה מסוימת של הרקע של המדווח, כך שזה מסיר את רעשי הרקע האלה מהנתונים. הנתונים מוכנים כעת לשרטוט. אנו ממליצים על גרפים קוויים על פני קורס זמן עריכת אפיגנום.
הערה: ניתן להשתמש בסטטיסטיקה כדי לכמת את ההבדלים בהשתקה בין שני עורכי אפיגנום שונים. אנו ממליצים לבצע את כל ניסויי הטרנספקציה והנוקלאופקציה בשלישיות טכניות לביצוע ניתוחים סטטיסטיים. כאשר מתארים גרפים קוויים של השתקה לאורך זמן, שרטטו סטיית תקן של שכפולים טכניים. ניתן לבצע מבחני t של תלמידים בנקודות זמן ספציפיות כדי להשוות השתקה בין שני עורכי אפיגנום. יש להתייחס לדגימות כלא מזווגות עם שונות לא שווה.
- הפחיתו את מספר התאים השליליים של המדווח (לדוגמה: APC שלילי) בדגימת הבקרה מכל הדגימות האחרות. ייתכן שיש השתקה מסוימת של הרקע של המדווח, כך שזה מסיר את רעשי הרקע האלה מהנתונים. הנתונים מוכנים כעת לשרטוט. אנו ממליצים על גרפים קוויים על פני קורס זמן עריכת אפיגנום.
תוצאות
עבור כל ניסויי עריכת האפיגנום, בקרות מתאימות הן קריטיות להערכת יעילות עריכת האפיגנום. אנו ממליצים להשתמש ב-sgRNA בקרה, שאינו מכוון לשום רצף בגנום האנושי. שימוש בבקרת מדריך שאינה ממוקדת ייתן ביטחון שהשינויים במיקומי היעד מונעים על ידי עורך האפיגנום המופנה לאתר זה ולא רק מביטוי יתר של עורך האפיגנום או כריכה לא ספציפית. בנוסף, עבור ניסויים מבוססי גנים של מדווחים, אנו מציעים להשתמש בבקרה של dCas9 בלבד כדי להבטיח שהשינויים בביטוי המדווח נובעים ממיזוג עורך אפיגנום ולא מעיכוב סטרי של קשירת dCas9 למיקום המטרה ומעכב זמנית את השעתוק (איור 2F).
עבור ניסויי טרנספקציה, אנו ממליצים להשתמש בעורך אפיגנום עם היתוך חלבון פלואורסצנטי נוסף, כגון BFP. היתוך זה מאפשר הדמיה של תאים שהועברו בהצלחה עם עורך האפיגנום באמצעות מיקרוסקופיה וזרימה ציטומטרית. תאים שעברו טרנספטציה מוצלחת יבטאו רמות גבוהות של BFP יומיים לאחר הטרנספקציה (איור 2D). הכימות של תאים שהומרו בהצלחה משמש לנורמליזציה של יעילות עריכת אפיגנום בימים מאוחרים יותר (איור 2F).
גם CRISPRoff וגם CRISPRi מראים השתקת שיא ביום 5 לאחר הטרנספקציה (איור 2E-F). לעורכי אפיגנומים שונים יש צירי זמן שונים של עריכת אפיגנום, כגון השתקה תורשתית עם CRISPRoff והשתקה חולפת עם CRISPRi (איור 2F). איור 2F מציג גם את השימוש ב-dCas9 רק כבקרה חשובה לניסויי עריכת אפיגנום. בניסויי נוקלאופקציה של mRNA, תאים שנערכו בהצלחה עם CRISPRoff יראו השתקה חזקה של גן המטרה עד היום השלישי לאחר הנוקלאופקציה (איור 3E-F).

איור 2: אסטרטגיית שער ונתונים מייצגים להעברת עורך אפיגנום על ידי טרנספקציה של פלסמיד. (א-ג) עלילות זרימה מייצגות להצגת אסטרטגיית שער לניסויי טרנספקציה של פלסמיד. עלילות הזרימה המוצגות הן של תאים לא עוברים טרנספקציה יומיים לאחר הטרנספקציה. כל נקודה בעלילות מייצגת תא אחד. (A) עלילת זרימה של אזור פיזור קדימה (FSC-A) ואזור פיזור צדדי (SSC-A) עם שער לתאים חיים. (B) תרשים זרימה של תאים חיים המציג FSC-A וגובה פיזור קדימה (FSC-H) עם שער לתאים בודדים. (C) תרשים זרימה של תאים בודדים בגרף PE-CF594-A (ביטוי mCherry ) ו-FSC-A. שער לתאים חיוביים של mCherry כפרוקסי לביטוי sgRNA. (D) אסטרטגיית שער מייצגת לביטוי עורך אפיגנום (BFP+) ביום השני לאחר הטרנספקציה. אוכלוסיית ההורים היא תאים המבטאים מדריך מגודרים ב-(C). (E) אסטרטגיית שער מייצגת לבדיקת השתקה של גן מדווח CLTA-GFP. אוכלוסיית ההורים היא תאים המבטאים מדריך מגודרים בפאנל C. (F) השתקה של CLTA-GFP במשך ימים לאחר טרנספקציה לאחר אספקת פלסמיד של dCas9, CRISPRi ו-CRISPRoff. אחוז CLTA-GFP מושתק מנורמל ליעילות טרנספקציה הנמדדת כתאים חיוביים ל-BFP יומיים לאחר הטרנספקציה. נקודות הן ממוצעים של ארבעה שכפולים של טרנספקציה. קווי השגיאה מייצגים את סטיית התקן. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
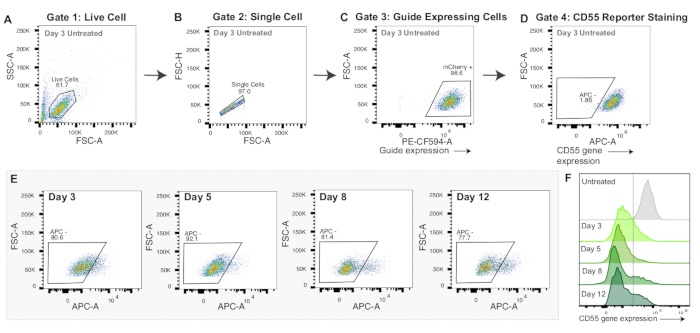
איור 3: אסטרטגיית שער ונתונים מייצגים עבור נוקלאופקציה של CRISPRoff mRNA. (א-ד) עלילות זרימה מייצגות להצגת אסטרטגיית שער לניסויי נוקלאופקציה של mRNA. עלילות הזרימה המוצגות הן של תאי ביקורת 3 ימים לאחר נוקלאופקציה מוכתמים בנוגדני CD55 אנטי-אנושיים APC. כל נקודה בעלילות מייצגת תא אחד. (A) תרשים זרימה של עוצמת שטח פיזור קדימה (FSC-A) ועוצמת אזור פיזור צדדי (SSC-A) עם שער לתאים חיים. (B) תרשים זרימה של תאים חיים המציג FSC-A ועוצמת גובה פיזור קדימה (FSC-H) עם שער לתאים בודדים. (C) תרשים זרימה של תאים בודדים בגרף PE-CF594-A (ביטוי mCherry ) ו-FSC-A. שער לתאים חיוביים של mCherry כפרוקסי לביטוי sgRNA. (D) תרשים זרימה של APC-A לעומת FSC-A. שער שצויר עבור תאים שליליים של APC, מה שיצביע על השתקת גן מדווח CD55. (E) עלילות זרימה של השתקת CD55 (APC - שער) במשך 3, 5, 8 ו-12 ימים לאחר נוקלאופקציה עם CRISPRoff mRNA. (F) היסטוגרמות של ביטוי חלבון CD55 (APC-A) במשך 3, 5, 8 ו-12 ימים לאחר נוקלאופקציה עם CRISPRoff mRNA בהשוואה לבקרה מוכתמת אך לא גרעינית. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| צלחת | צפיפות זריעה (תאים לבאר) | כמות פלסמיד |
| 96 באר | 15,000 | 150 נ"ג |
| 24 בארות | 90,000 | 500 נ"ג |
| 6 בארות | 400,000 | 2 מיקרוגרם |
טבלה 1: כמויות קנה מידה של טרנספקציה. צפיפות זריעה וכמויות DNA פלסמיד להעברת עורך אפיגנום לתאי HEK293T בקנה מידה שונה.
טבלה משלימה 1: רצפי ספייסר sgRNA לניסויי עריכת אפיגנום. רצפי sgRNA למיקוד עורכי אפיגנום ל-CLTA ו-CD55 יחד עם רצף למדריך בקרה שאינו ממוקד. בנוסף, מופיעים אוליגוס לשיבוט בעמוד השדרה של pLG1. אנא לחץ כאן להורדת קובץ זה.
קובץ משלים 1. אנא לחץ כאן להורדת קובץ זה.
Discussion
פרוטוקול זה מפרט שתי שיטות אספקה חולפות שונות עבור עורכי אפיגנום CRISPR: טרנספקציה של DNA פלסמיד ונוקלאופקציה של mRNA. לשתי הטכניקות יש יתרונות, חסרונות ושיקולים כלליים ייחודיים (איור 1F).
טרנספקציה של DNA פלסמיד מובילה לביטוי חזק של עורך אפיגנום, ואנו כוללים היתוך BFP בתוך מבני עורך האפיגנום המאפשר זיהוי וכימות של יעילות טרנספקציה באמצעות ציטומטריית זרימה. בנוסף, ניתן להשתמש בביטוי BFP כדי למיין תאים המבטאים את עורך האפיגנום או שניתן לכמת אותו כדי לנרמל נתוני השתקה בנקודות זמן מאוחרות יותר, כמפורט בפרוטוקול זה. עם זאת, חשוב לציין שיעילות הטרנספקציה היא בדרך כלל לא 100%, ולכן האוכלוסייה תהיה הטרוגנית עבור אלה שקיבלו את העורך ואלה שלא, אלא אם כן התאים ממוינים. יתר על כן, אספקת DNA פלסמיד יכולה לעורר תגובה חיסונית מ-DNA דו-גדילי ציטופלזמי, מה שמוביל להפעלת מסלול חיסוני וציטוטוקסיות. לבסוף, טרנספקציה של DNA פלסמיד אינה ניתנת לכל סוגי התאים, וייתכן שיהיה צורך בנוקלאופקציה של הפלסמיד.
בניגוד לטרנספקציה של DNA פלסמיד, נוקלאופקציה של mRNA ישימה עבור סוגי תאים רבים ולעתים קרובות מביאה ליעילות אספקה גבוהה. ניתן לסנתז את ה-mRNA של העורך באמצעות ערכות סינתזה של mRNA במבחנה הזמינות מסחרית, שאחת מהן פורטה בעבר51. לחלופין, ניתן לסנתז mRNA מ-Aldevron או Trilink. עם זאת, אזהרה אחת של נוקלאופקציה של mRNA היא שהיא אינה מייצרת מספיק חלבון עורך כדי לבדוק אילו תאים קיבלו את עורכי האפיגנום. הניסויים לעיל מציעים שכמעט כל תא קיבל את ה-mRNA כאשר אנו מזהים ~90% מהשתקת גן המטרה ביום החמישי (איור 3F). עם זאת, אנו ממליצים לבצע אופטימיזציה של קודי הדופק ויחס ה-mRNA לתא עבור כל סוג תא.
שיקול נוסף הוא שלעריכת אפיגנום המופעלת על ידי טרנספקציה של DNA פלסמיד או גרעין mRNA יש לוחות זמנים שונים להשתקה. עם טרנספקציה של דנ"א פלסמיד, השתקת שיא עם CRISPRoff ו-CRISPRi נצפית ביום החמישי לאחר הטרנספקציה (איור 2F). לשם השוואה, השתקה מקסימלית עם CRISPRoff ו-CRISPRi המועברת על ידי נוקלאופקציה של mRNA נראית כבר ביום השלישי לאחר נוקלאופקציה (איור 3F). בנוסף, DNA פלסמיד נמשך בתאים בהשוואה ל-mRNA, וכתוצאה מכך משך זמן ארוך יותר של ביטוי העורך. בהתאם למטרות הניסוי, ניתן להעדיף ציר זמן ומשך ביטוי ארוכים או קצרים יותר.
חשוב גם לציין שלא ניתן ליישם עריכת אפיגנום באופן אוניברסלי על כל הגנים או סוגי התאים. הבדלים אינהרנטיים ברצף הגנום או במצבי הכרומטין של גני המטרה עשויים להשפיע על יעילות העורך, אפילו עבור אותו גן בסוגי תאים שונים. לדוגמה, גנים שחסרים איי CpG מבוארים יכולים להיות קשים להשתקה יציבה על ידי מתילציה של DNA בתיווך CRISPRoff48. לכן, ייתכן שיהיה צורך לבדוק עורכי אפיגנומים שונים כדי להשתיק גנים כאלה. בנוסף, אנו ממליצים גם לבדוק לפחות את שלושת ה-CRISPRi gRNAs המובילים כדי לזהות את המדריך הטוב ביותר ליעילות גבוהה יותר של עריכת אפיגנום42. עם זאת, בהתחשב בארגז הכלים המוגבל שלנו של עורכי אפיגנום חזקים, ייתכן שיהיה יעיל יותר לבצע נוקאאוט של גנים או להשתמש באסטרטגיות נוק-דאון אחרות בהתאם לגן המעניין.
פרוטוקול זה מתמקד ב-CRISPRi ו-CRISPRoff, שניים מבין עורכי האפיגנום הרבים הזמינים מבוססי CRISPR. מחקרי גילוי בקנה מידה גדול לאחרונה פיתחו כלים חדשים לשכתוב האפיגנום האנושי 29,30,52. לעורכי אפיגנום יש יישומים במחקר ביו-רפואי וטיפולים. לדוגמה, מחקרים אחרונים השתמשו במתילציה של DNA ועריכת אפיגנום מבוססת H3K9me3 במודלים של עכברים ופרימטים לא אנושיים, וכתוצאה מכך דיכוי תורשתי של גן הקשור למחלה 53,54,55. אנו צופים כי אופני אספקה עתידיים לעורכי אפיגנום יפתחו אפיקים חדשים ליישום נרחב של עריכת אפיגנום.
Disclosures
J.K.N. הוא ממציא פטנטים הקשורים לטכנולוגיות CRISPRoff/on, שהוגשו על ידי The Regents of the University of California.
Acknowledgements
אנו מודים לחברי מעבדת נונייז, במיוחד ל-Rithu Pattali ו-Izaiah Ornelas, על פיתוח ואופטימיזציה של הפרוטוקולים המתוארים בכתב היד הזה.
Materials
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
References
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -H., Tee, L. Y., Wang, X. -G., Huang, Q. -S., Yang, S. -H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264(2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136(2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -Y., Gingras, A. -C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284(2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26(2019).
- Kim, J. -M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485(2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760(2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460(2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009(2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082(2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved