È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Editing dell'epigenoma CRISPR nelle cellule umane mediante trasfezione del DNA plasmidico e consegna della nucleofecazione dell'mRNA
In questo articolo
Riepilogo
Il protocollo descrive i metodi per l'editing dell'epigenoma basato su CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) in linee cellulari umane utilizzando la trasfezione del DNA plasmidico e la nucleofecazione dell'mRNA.
Abstract
L'epigenetica si riferisce alle modifiche chimiche delle proteine istoniche e del DNA che possono regolare l'espressione dei geni. L'epigenoma umano viene alterato dinamicamente durante la differenziazione cellulare e l'invecchiamento e molte malattie sono associate a un pattern aberrante dell'epigenoma. I recenti progressi nel CRISPR hanno portato allo sviluppo di strumenti programmabili per modificare le modifiche epigenetiche in loci genomici mirati, consentendo una riscrittura precisa delle modifiche epigenetiche nelle cellule umane. Gli editor di epigenomi basati su CRISPR si basano su Cas9 cataliticamente morto accoppiato con modificatori epigenetici che alla fine provocano la repressione programmata o l'attivazione di geni mirati nei genomi dei mammiferi. A differenza dei metodi tradizionali di editing del genoma, l'editing dell'epigenoma non richiede rotture del DNA o cambiamenti nella sequenza del genoma umano e quindi funge da alternativa più sicura per controllare l'espressione genica. In questo protocollo, evidenziamo due diversi metodi per eseguire l'editing dell'epigenoma mediato da dCas9 in linee cellulari umane utilizzando trasfezioni di DNA plasmidico e nucleofeczione di mRNA codificanti editor di epigenoma CRISPR. Dimostriamo l'editing programmabile dell'epigenoma per reprimere transitoriamente i geni utilizzando l'interferenza CRISPR (CRISPRi) e per silenziare i geni in modo duraturo per molte settimane utilizzando CRISPRoff, una fusione di dCas9 con il dominio KRAB e il complesso de novo DNA metiltransferasi. Forniamo anche indicazioni sui metodi quantitativi per misurare il successo dell'editing dell'epigenoma dei geni bersaglio e considerazioni chiave su quale strumento di editing dell'epigenoma utilizzare, a seconda dei criteri sperimentali.
Introduzione
Sebbene il contenuto genomico di ogni cellula del nostro corpo sia quasi identico, il profilo trascrizionale di ciascun tipo di cellula differisce notevolmente. Le modificazioni epigenetiche del DNA e delle proteine istoniche sono regolatori chiave dell'espressione trascrizionale. L'eucromatina trascrizionalmente attiva è caratterizzata da segni epigenetici distinti rispetto all'eterocromatina compatta e trascrizionalmente inattiva. Ad esempio, le regioni eterocromatiche sono definite da modificazioni repressive degli istoni, tra cui la trimetilazione sulla lisina 9 dell'istone 3 (H3K9me3), la trimetilazione sulla lisina 27 dell'istone H3 (H3K27me3) e la metilazione del DNA sulle citosine accanto alle guanine (CpG) ai promotori del gene1. Le regioni genomiche di espressione genica attiva sono definite tipicamente dall'acetilazione e dalla trimetilazione degli istoni sulla lisina 4 dell'istone 3 (H3K4me3)1.
La rivoluzione CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) ha generato una vasta gamma di strumenti che consentono l'alterazione programmata delle sequenze genomiche. La tecnologia CRISPR si basa su un meccanismo di difesa procariotico in grado di scindere gli acidi nucleici in sequenze bersaglio programmabili. Le nucleasi CRISPR 2,3,4, gli editor di base 5,6 e gli editor principali 7,8 possono modificare la sequenza del DNA dei genomi dei mammiferi attraverso il taglio del DNA e la riparazione di queste rotture. Sebbene efficaci, queste strategie possono causare rotture del DNA in siti fuori bersaglio 9,10 e mutazioni strutturali genomiche su larga scala 11,12,13,14,15. Strumenti alternativi basati su CRISPR consentono una modulazione trattabile dell'attivazione e della repressione genica senza modificare la sequenza di DNA sottostante. Questi strumenti sfruttano un Cas9 carente di nucleasi (dCas9), consentendo il legame del DNA nei siti bersaglio dettati dalla sequenza di sgRNA, in combinazione con proteine effettrici che alterano il panorama della cromatina 16,17,18. Le proteine effettrici, come gli scrittori epigenetici, i lettori e le gomme, possono essere fuse direttamente con dCas9 o reclutate da un'impalcatura peptidica fusa con dCas9, come SunTag, o da un'impalcatura di RNA sull'sgRNA, come il sistema MS2-MCP 16,17,18. Esempi di strumenti programmabili per il controllo della trascrizione includono l'attivazione CRISPR (CRISPRa)19,20,21 e l'interferenza CRISPR (CRISPRi)22,23. CRISPRa funziona reclutando direttamente il meccanismo di trascrizione, aumentando l'espressione genica trascrizionale target19. Al contrario, CRISPRi reprime la trascrizione stabilendo H3K9me3, un segno epigenetico repressivo22.
I progressi nell'editing dell'epigenoma hanno consentito un ampio uso di questi strumenti in tutti i campi scientifici. Le fusioni di diversi domini effettori e proteine hanno ampliato il kit di strumenti degli editor di epigenomi disponibili 18,24,25,26,27,28,29,30. Inoltre, gli editor di epigenomi vengono utilizzati per decifrare i ruoli delle modificazioni epigenetiche 31,32,33,34,35,36, degli effettori 37,38,39 e delle mutazioni effettrici 28,40,41 nella regolazione genica. In particolare, CRISPRi e CRISPRa sono utilizzati negli screening di genomica funzionale per una varietà di processi biologici, tra cui la sopravvivenza cellulare42 e il destino cellulare 43,44,45,46,47. Inoltre, l'editing dell'epigenoma ha un potenziale terapeutico per l'ingegneria cellulare ex vivo e le terapie in vivo 18.
Qui, descriviamo i metodi per l'applicazione di due editor di epigenoma basati su dCas9 per la repressione trascrizionale programmabile in linee cellulari umane: CRISPRi22 e CRISPRoff48. CRISPRi è una fusione di dCas9 nel dominio repressivo KRAB da una proteina a dita di zinco come ZNF10 (KOX1) e ZIM322,23. Quando CRISPRi è mirato a uno specifico promotore genico, il dominio KRAB recluta uno scrittore H3K9me3, SETDB1, per reprimere il gene bersaglio (Figura 1A). Quando CRISPRi è espresso transitoriamente, l'H3K9me3 stabilito nel locus bersaglio non viene mantenuto e l'espressione genica viene ripristinata nel tempo 32,48. Per condurre knockdown stabili utilizzando CRISPRi, come nelle applicazioni di genomica funzionale, l'espressione costitutiva di CRISPRi nelle cellule insieme all'sgRNA è essenziale. Recentemente, CRISPRoff è stato progettato per programmare l'editing ereditario dell'epigenoma48. CRISPRoff è una fusione di una singola proteina di dCas9 con il dominio KRAB e il complesso de novo DNA metiltransferasi, DNMT3A e DNMT3L. Un impulso transitorio di CRISPRoff nelle cellule umane programma la deposizione di H3K9me3 e la metilazione del DNA nei geni bersaglio, che porta alla repressione a lungo termine dei geni bersaglio mediante il mantenimento della metilazione del DNA e di H3K9me3 (Figura 1B)48. Inoltre, le modifiche all'epigenoma possono essere annullate. Ad esempio, un gene che è stabilmente silenziato da CRISPRoff può essere riattivato da TET1-dCas9 che può rimuovere enzimaticamente i segni di metilazione del DNA nei loci bersaglio49.
Questo protocollo descriverà in dettaglio due metodi di somministrazione per l'espressione transitoria degli editor di epigenomi: la trasfezione del DNA plasmidico e la nucleofecazione dell'mRNA. Inoltre, descriviamo come utilizzare la citometria a flusso per valutare l'efficacia dell'editing dell'epigenoma in due geni endogeni, CLTA e CD55. Questi metodi possono essere adattati e applicati ad altri esperimenti di editing dell'epigenoma utilizzando editor aggiuntivi o possono essere utilizzati per indirizzare geni diversi.
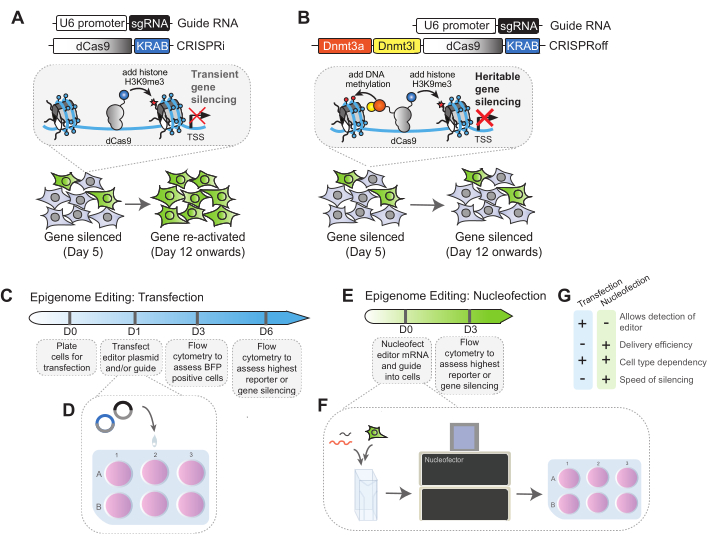
Figura 1: Schema del meccanismo e del flusso di lavoro di editing dell'epigenoma CRISPRi e CRISPRoff. (A) Schemi lineari del transgene sgRNA e dell'editor di epigenoma CRISPRi. L'aggiunta di CRISPRi e sgRNA consente l'aggiunta del marcatore istonico repressivo H3K9me3 per silenziare il locus bersaglio. Il livello più alto di silenziamento viene raggiunto precocemente dopo l'aggiunta di CRISPRi e il gene bersaglio viene generalmente riattivato dopo pochi passaggi. (B) Schemi lineari del transgene sgRNA e dell'editor di epigenoma CRISPRoff. L'aggiunta di CRISPRoff alle cellule che hanno come bersaglio un gene di interesse porta all'aggiunta di H3K9me3 repressivo insieme alla metilazione del DNA nei siti CpG per silenziare il gene bersaglio. Il silenziamento mediante CRISPRoff è ereditabile: l'alto livello di silenziamento viene raggiunto precocemente durante la trasfezione e persiste su più divisioni cellulari. (C) Panoramica della cronologia per l'editing dell'epigenoma tramite il metodo della trasfezione. Il giorno 0, le cellule vengono piastrate per la trasfezione. Il giorno 1, il plasmide editor e guida può essere introdotto nelle cellule tramite trasfezione. Il giorno 3, le cellule saranno valutate per l'espressione di BFP tramite citometria a flusso. La percentuale di BFP viene utilizzata come fattore normalizzante per determinare l'efficacia finale del silenziamento dell'esperimento per ciascuna condizione. Dal giorno 6 in poi, le cellule vengono analizzate per silenziare il reporter di interesse, poiché il livello più alto di silenziamento sarà raggiunto in questo giorno. (D) Panoramica del metodo di trasfezione in cui l'editor di epigenoma e i plasmidi sgRNA vengono aggiunti alle cellule in modo graduale. (E) Panoramica della cronologia per l'editing dell'epigenoma attraverso i metodi della nucleofezione. In questo protocollo, l'mRNA è la nucleofezione nelle cellule il giorno 0. Le cellule vengono valutate per il silenziamento il giorno 3 dopo la nucleofecazione utilizzando l'analisi della citometria a flusso. (F) Panoramica del protocollo di nucleofezione. Quantità appropriate di cellule e mRNA vengono mescolate e aggiunte in cuvette di nucleolettori. Se l'sgRNA viene introdotto per nucleofezione, può anche essere aggiunto a questa miscela. Le cuvette vengono inserite nel nucleocongelatore e vengono utilizzati codici di impulsi appropriati per introdurre l'mRNA nelle cellule. Dopo la nucleofezione, le cellule vengono piastrate e fatte passare per l'analisi nei giorni successivi. (G) Confronto tra la trasfezione plasmidica e le strategie di nucleofezione dell'mRNA per l'editing dell'epigenoma. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
NOTA: Il file supplementare 1 contiene dettagli sulla progettazione dell'sgRNA, il clonaggio e la generazione di linee cellulari dei nostri dati rappresentativi. La sezione dei risultati rappresentativi fornisce anche suggerimenti per i controlli.
1. Trasfezione di plasmidi che esprimono l'editor dell'epigenoma in cellule HEK293T
NOTA: Questo protocollo descrive la somministrazione di plasmidi codificanti CRISPR in cellule HEK293T. Abbiamo ingegnerizzato le cellule per esprimere un sgRNA (Addgene 217306) che ha come bersaglio la regione del promotore di CLTA, un gene non essenziale, che è marcato endogenamente con GFP. Le cellule CLTA-GFP HEK 293T hanno avuto origine da uno studio precedente50. In questo esempio, gli editor dell'epigenoma sono fusi direttamente con una proteina fluorescente blu (BFP), che ci consente di quantificare l'efficienza della trasfezione e garantisce una valutazione accurata delle condizioni sperimentali. L'efficacia dell'approccio è dimostrata dal silenziamento di CLTA-GFP, che può essere misurato quantitativamente a livello proteico in singole cellule utilizzando la citometria a flusso.
- Coltura cellulare
- Mantenere HEK293T cellule in DMEM ad alto contenuto di glucosio con il 10% di FBS e 1x penicillina-streptomicina-glutammina. Far passare le celle ogni 2-3 giorni, assicurandosi che rimangano a una confluenza del 60%-70%.
NOTA: Per garantire un'efficienza di trasfezione ottimale, è fondamentale mantenere la confluenza cellulare al di sotto del 70%, che può essere monitorata quotidianamente al microscopio.
- Mantenere HEK293T cellule in DMEM ad alto contenuto di glucosio con il 10% di FBS e 1x penicillina-streptomicina-glutammina. Far passare le celle ogni 2-3 giorni, assicurandosi che rimangano a una confluenza del 60%-70%.
- Giorno 0: Semina HEK293T cellule per la trasfezione
- Il giorno prima della trasfezione, contare le cellule con un contatore di cellule automatizzato utilizzando la colorazione viva/morta con blu di tripano e seminare ~1,5 x 104 cellule vive per pozzetto in una piastra da 96 pozzetti. Mantenere il volume finale di ciascun pozzetto a 200 μL. Le cellule dovrebbero raggiungere il 60%-70% di confluenza il giorno successivo.
NOTA: Questa operazione può essere eseguita in modo efficiente anche in piastre a 24 o 6 pozzetti. Vedere la Tabella 1 per il numero di cellule e la quantità di DNA per la trasfezione.
- Il giorno prima della trasfezione, contare le cellule con un contatore di cellule automatizzato utilizzando la colorazione viva/morta con blu di tripano e seminare ~1,5 x 104 cellule vive per pozzetto in una piastra da 96 pozzetti. Mantenere il volume finale di ciascun pozzetto a 200 μL. Le cellule dovrebbero raggiungere il 60%-70% di confluenza il giorno successivo.
- Giorno 1: Trasfezione dei plasmidi CRISPRoff e CRISPRi in cellule HEK29T
- Aliquotare 150 ng del plasmide editor dell'epigenoma (Addgene 167981) per ciascun pozzetto in un volume totale di 3 μl. Queste diluizioni plasmidiche possono essere preparate in provette per PCR.
NOTA: Se l'editor di epigenomi e il plasmide sgRNA vengono co-trasfettati, si consiglia di provare un rapporto molare 3:4 di plasmidi editor:sgRNA. Potrebbe essere necessario ottimizzare il rapporto per ottenere la massima efficienza dell'editing dell'epigenoma. - Preriscaldare il reagente di trasfezione e il mezzo minimo essenziale a temperatura ambiente circa 30-60 minuti prima della trasfezione. Miscelare 0,6 μl di reagente di trasfezione e 20 μl di terreno essenziale minimo per pozzetto e incubare a temperatura ambiente per 15 minuti.
NOTA: Se si eseguono più trasfezioni, è possibile realizzare una miscela master di reagente di trasfezione e di un terreno essenziale minimo. - Aggiungere 20,6 μl del reagente di trasfezione e una miscela minima di terreno essenziale a ciascuna provetta per PCR contenente 3 μl di DNA plasmidico. Lasciare riposare a temperatura ambiente per 15 min.
- Aggiungere lentamente il composto alla piastra delle cellule seminate e picchiettare delicatamente la piastra per disperdere il composto. Includere un controllo non trasfettato per fornire una base adeguata per il confronto nelle analisi a valle.
NOTA: Quando si aggiunge la miscela di trasfezione alle cellule seminate, è essenziale farlo goccia a goccia e delicatamente per ridurre al minimo i disturbi, poiché ciò può influire negativamente sull'efficienza della trasfezione.
- Aliquotare 150 ng del plasmide editor dell'epigenoma (Addgene 167981) per ciascun pozzetto in un volume totale di 3 μl. Queste diluizioni plasmidiche possono essere preparate in provette per PCR.
- Giorno 2: Verifica il successo della trasfezione
- Esamina le cellule al microscopio con un ingrandimento 10X per determinare se sono BFP positive, poiché ciò indica che la trasfezione è riuscita. Assicurarsi che le celle non vengano disturbate durante il processo di osservazione.
- Giorno 3: Dividere la piastra a 96 pozzetti e valutare l'efficienza della trasfezione utilizzando il marcatore BFP
NOTA: Una trasfezione riuscita dovrebbe produrre almeno il 30% delle cellule come BFP positive. Tuttavia, con dCas9, CRISPRi e CRISPRoff, in genere rileviamo più del 60% di cellule BFP positive due giorni dopo la trasfezione. L'efficienza di trasfezione è correlata negativamente con le dimensioni del plasmide, quindi diversi editor di epigenomi possono produrre diverse efficienze di trasfezione.- Preriscaldare la tripsina e il DMEM con il 10% di FBS e 1x penicillina-streptomicina-glutammina a temperatura ambiente per circa 30-60 minuti prima dell'uso.
- Aspirare i mezzi dai pozzi; Fare attenzione a non rimuovere le celle attaccate al fondo. Aggiungere 50 μL di tripsina per pozzetto. Incubare per 2-3 minuti a 37 °C.
NOTA: Non è insolito osservare alcune celle galleggianti, soprattutto dopo la trasfezione con CRISPRoff. Ipotizziamo che questa morte cellulare sia dovuta alla tossicità della sovraespressione di DNMT3A-3L. - Inibire la tripsina aggiungendo 150 μL di DMEM completo per pozzetto. Pipettare su e giù per risospendere le cellule e dividere le cellule per la confluenza il giorno 5 (~20 μL di miscela cellulare in 200 μL di terreno di una piastra a 96 pozzetti).
- Utilizzare 50 μl di cellule risospese per la citometria a flusso per valutare l'efficienza della trasfezione.
2. Nucleofezione dell'mRNA dell'editor dell'epigenoma in cellule K-562
NOTA: Questa sezione descrive in dettaglio il processo di nucleofecting dell'mRNA CRISPRoff nelle cellule K-562. Per semplicità, abbiamo pre-ingegnerizzato le cellule K-562 per esprimere costitutivamente un sgRNA che ha come bersaglio il promotore del gene endogeno CD55 (Addgene 217306). La somministrazione dell'mRNA di CRISPRoff direttamente alle cellule ha il potenziale per ridurre la tossicità cellulare che accompagna gli approcci basati sul DNA plasmidico, pur ottenendo un'efficacia simile nel silenziamento genico. Inoltre, la nucleofezione può essere utilizzata per introdurre costrutti dell'editor di epigenomi in linee cellulari che sono difficili da trasfettare in modo efficiente, come i K-562.
- Coltura cellulare
- Prima della nucleofezione, mantenere le cellule K-562 in un pallone con RPMI con il 10% di FBS e 1x penicillina-streptomicina-glutammina. Far passare le celle ogni giorno, assicurandosi che rimangano a una confluenza del 60%-70%.
- Il giorno della nucleofezione, scongelare e agitare delicatamente l'mRNA CRISPRoff in una provetta da microcentrifuga sterile priva di RNasi e conservare l'mRNA sul ghiaccio. Utilizzare 2 μg di mRNA per 2,0 x 105 cellule in ciascun pozzetto di una nucleocuvette a striscia. Assicurarsi che il volume dell'mRNA non superi il 10% del volume totale della nucleofezione (22 μL).
NOTA: La nucleofezione può essere eseguita anche in cuvette più grandi con più cellule e mRNA. Suggeriamo di scalare la quantità di mRNA proporzionalmente al numero di cellule che si aggiungeranno. Ad esempio, quando si esegue una nucleofecazione in una cuvetta grande su 1 x 106 cellule, si consiglia di utilizzare 10 μg di mRNA, con un volume totale di nucleofezione di 100 μL. Inoltre, il dosaggio dell'mRNA può essere ottimizzato per garantire un'elevata efficacia di editing. Usiamo spesso 2-5 μg di mRNA editor per ogni 2,0 x 105 cellule per ottenere risultati efficienti. I nucleotidi liberi che non sono stati incorporati durante la sintesi dell'mRNA devono essere rimossi prima della quantificazione. - Preparare la soluzione di nucleofezione secondo le istruzioni del produttore e scaldare a temperatura ambiente per 15 minuti prima della nucleofezione.
NOTA: La soluzione del nucleolettore può essere diversa o deve essere ottimizzata per diversi tipi di cellule. - Raccogliete e contate le cellule K-562 utilizzando un contatore di cellule automatizzato con colorazione viva/morta in blu di tripano. Per la nucleofecazione in una cuvetta a strisce, aliquotare ~2,0 x 105 cellule per campione in una provetta sterile per microcentrifuga.
- Centrifugare le cellule a 500 x g per 5 minuti a temperatura ambiente, quindi scartare il surnatante. Lavare le celle 1 volta con PBS a temperatura ambiente a 500 x g per 5 minuti, quindi scartare il surnatante.
- Calcolare la quantità di soluzione di nucleofector in cui risospendere le cellule sottraendo il volume di mRNA da 22 μL. Risospendere le cellule nella quantità appropriata di soluzione di nucleofector.
- Aggiungere la soluzione cellulare a 2 μg di mRNA CRISPRoff (volume determinato al punto 2.1.2) e trasferire la soluzione in una cuvetta, facendo attenzione a non formare bolle, in quanto ciò potrebbe compromettere l'efficienza della nucleofezione. Picchiettare delicatamente la nucleocuvetta per assicurarsi che le cellule siano sul fondo.
- Nucleofect le cellule utilizzando il sistema 4D-Nucleofector con il codice di impulso appropriato. Il codice FF-120 è consigliato per le celle K-562.
NOTA: Potrebbe essere necessario ottimizzare il codice dell'impulso per diversi tipi di celle. Consultare il produttore se è necessaria un'ulteriore ottimizzazione. - Dopo la nucleofezione, aggiungere 80 μL di terreno RPMI a ciascun pozzetto della nucleocuvetta piccola e lasciare riposare le cellule a 37 °C per 15 minuti.
- Trasferire la sospensione cellulare in un pozzetto di una piastra a 24 pozzetti contenente 400 μl di terreno RPMI preriscaldato (piccola nucleocuvetta).
NOTA: Dopo la nucleofezione, le cellule sono particolarmente fragili e devono essere trasferite delicatamente dalla nucleocuvetta ai pozzetti di coltura. - Dopo il giorno 2 post-nucleofezione: seguire il protocollo di colorazione del marcatore di superficie con l'anticorpo CD55 come descritto nella sezione 3 e misurare la fluorescenza di CD55 APC su un citometro a flusso per valutare l'efficacia dell'editing epigenomico.
NOTA: Per valutare il successo della nucleofezione, si consiglia di eseguire una nucleofezione con GFP o mCherry mRNA. Due giorni dopo la nucleofezione, eseguire la citometria a flusso di queste cellule per confermare l'avvenuta nucleofecazione. Generalmente rileviamo dal 90% al 100% di queste cellule che esprimono GFP o mCherry. Un'alternativa consiste nell'eseguire una nucleofezione con l'mRNA CRISPRi, che è noto per silenziare in modo robusto molti geni bersaglio. Quando si esegue la citometria a flusso il giorno 2, i campioni trattati con CRISPRi devono mostrare il silenziamento del gene target in oltre il 90% delle cellule.
3. Colorazione del marcatore superficiale
NOTA: Questa sezione descrive in dettaglio la quantificazione dei livelli di proteina CD55 dopo l'editing dell'epigenoma nelle cellule K-562. Quantifichiamo la diminuzione dell'espressione di CD55 nelle singole cellule utilizzando la colorazione degli anticorpi e la citometria a flusso (vedere la sezione 4 di seguito) per valutare l'efficienza del knockdown mediato da CRISPRoff. Ulteriori tecniche, tra cui la PCR quantitativa a trascrizione inversa o il western blotting, possono essere utilizzate per confermare il livello di knockdown sia a livello di trascritto che di proteina.
- Contare le cellule utilizzando un contatore di cellule automatizzato e aggiungere da 5,0 x 105 a 1,0 x 106 celle in una provetta da microcentrifuga e centrifugare le celle a 500 x g per 5 minuti.
NOTA: È possibile utilizzare più o meno cellule per la colorazione, tuttavia potrebbe essere necessario ridimensionare di conseguenza la concentrazione di anticorpi. - Scartare il surnatante assicurandosi di non disturbare il pellet e aggiungere 500 μl di PBS e risospendere delicatamente il pellet.
- Centrifugare nuovamente le celle a 500 x g per 5 min. Scartare il surnatante e aggiungere l'anticorpo alla concentrazione desiderata diluito in PBS. Per la colorazione con CD55, utilizzare 1 μL di CD55 anti-umano purificato (0,5 mg/mL di stock) in 49μL di PBS.
- Pipettare delicatamente le cellule e lasciarle incubare per 25 minuti in un luogo buio a temperatura ambiente. Dopo l'incubazione, centrifugare nuovamente le cellule a 500 x g per 5 minuti e scartare il surnatante.
- Risospendere il pellet con 500 μL di PBS per eliminare l'anticorpo in eccesso. Centrifugare le cellule a 500 x g per 5 minuti ed eliminare il surnatante PBS. Risospendere in 100 μl - 200 μl di PBS fresco e aggiungere a una piastra a 96 pozzetti per l'analisi della citometria a flusso.
4. Citometria a flusso
NOTA: Questo protocollo è stato scritto per l'utilizzo di un analizzatore cellulare BD FACSymphony A1. Le specifiche possono variare a seconda del citometro a flusso che si sta utilizzando. Si consiglia di fare riferimento al manuale utente della macchina che si sta utilizzando per le specifiche.
- Prima di attivare la citometria, verificare che il contenitore dei rifiuti non sia pieno e che ci sia molto liquido della guaina. Accendere l'interruttore di alimentazione principale (verde) che si trova vicino al fluido della guaina.
- Accendere il lettore di piastre utilizzando l'interruttore posteriore. Quindi, accendere la macchina utilizzando il pulsante verde sul lato destro e assicurarsi che la macchina sia impostata sulla funzione di lettore di piastre a 96 pozzetti.
- Accendi il computer e accedi. Apri il software e accedi utilizzando il tuo nome utente e password.
- Configura l'esperimento. Per creare un nuovo esperimento, selezionare Esperimento > Nuovo esperimento. Rinomina l'esperimento facendo doppio clic sul nome corrente.
- Aggiungi una piastra facendo clic su Sperimenta > nuova piastra. Selezionare il formato del pozzetto da utilizzare. Per aggiungere pozzetti specifici da eseguire, fare clic sulla posizione del pozzetto nella piastra e quindi fare clic sulla siringa blu (il pozzetto dovrebbe ora essere evidenziato in blu). Selezionare più pozzetti contemporaneamente da aggiungere, se necessario.
- Aggiungere nomi specifici a ciascun pozzetto facendo clic con il pulsante destro del mouse su Campione a destra del display della piastra. Quindi seleziona Layout sperimentale. Si aprirà una pagina che elenca tutti i pozzetti selezionati. Per rinominare i pozzetti, fare clic una volta e digitare il nome desiderato. Al termine, fare clic su OK .
- Aggiungere i grafici e i gate appropriati all'esperimento nel foglio di lavoro globale. Si consiglia di tracciare prima l'area di dispersione diretta (FSC-A) rispetto all'area di dispersione laterale (SSC-A). Quindi crea un gate arbitrario per le celle live da perfezionare successivamente facendo clic sullo strumento Poligono.
- Fare clic con il pulsante destro del mouse su questo cancello e fare clic su Drill Down. Questo creerà un nuovo grafico che mostra solo le celle di quel cancello di celle attive.
- Cambia l'asse di questo nuovo grafico in FSC-A rispetto all'altezza di dispersione in avanti (FSC-H) e crea un gate per le singole celle.
- Se l'sgRNA viene espresso costitutivamente nelle cellule, e il lentivirus codifica anche per una proteina fluorescente, allora perforare questa singola porta cellulare ed eseguire un ulteriore gating per l'espressione di quella proteina come proxy per l'espressione dell'sgRNA (esempio: PE-CF594-A vs FSC-A).
- Sulla porta di configurazione finale (per singole cellule o per cellule che esprimono sgRNA), fare clic con il pulsante destro del mouse e selezionare Drill Down. Creare un cancello per il test dell'espressione del gene reporter (esempio: BB515-A vs FSC-A).
- Se si utilizzano cellule trasfettate e si dispone di un metodo per misurare l'efficienza della trasfezione come l'espressione BFP, fare clic con il pulsante destro del mouse sul grafico dell'espressione reporter e fare clic su Duplica. Cambia gli assi in BV421-A vs FSC-A. Creare un gate arbitrario per le celle BFP-positive.
- Rimuovere tutti i parametri che non sono in uso nella scheda Parametri del display del citometro.
- Per configurare la macchina e prepararla per l'esecuzione, fare clic su Esegui sul pannello di controllo del citometro a flusso. Il pulsante dovrebbe apparire verde. Nel software, vai su HTS nel pannello superiore e poi su Prime. Una volta terminato, aggiungere il piatto, assicurandosi che sia ben posizionato nella macchina e che il coperchio sia spento.
- Per eseguire, selezionare tutti i pozzetti da eseguire e assicurarsi che i controlli siano impostati correttamente. Selezionare il pozzetto facendo doppio clic, modificare il volume del campione a 10 μL e fare clic su Acquisisci dati nel dashboard di acquisizione. In questo modo verrà eseguito il campione, ma non verranno registrati i dati. Mentre questo campione è in esecuzione, regolare le tensioni del laser secondo necessità. Inoltre, cambia i cancelli.
NOTA: Si consiglia di eseguire un campione negativo per tutti i colori misurati e positivo per tutti i colori per assicurarsi che i parametri del laser e il gating siano buoni. - Dopo aver controllato le impostazioni del laser, fare clic su Campioni nel pannello della piastra e verificare che i valori di funzionamento siano impostati come desiderato.
- Fare clic su Esegui piastra per eseguire l'esecuzione nel dashboard di acquisizione e registrare i dati da tutti i pozzetti. Una volta terminato, pulire la macchina rimuovendo la piastra.
- A una nuova piastra da 96 pozzetti, aggiungere il 10% di candeggina alle prime quattro file e H2O ai quattro pozzetti sottostanti. Vai a HTS > Clean. Una volta terminato, chiudere il programma e spegnere i pulsanti nell'ordine inverso a quello in cui erano accesi il pulsante laterale verde > il lettore di piastre > l'interruttore verde da spreco.
- Aggiungere il 20% di candeggina alla piastra a 96 pozzetti con le celle e scartare.
5. Analisi dei dati
NOTA: Questo metodo delinea le strategie di gating e l'elaborazione dei dati per quantificare l'editing dell'epigenoma mediante citometria a flusso. La strategia di gating è rappresentata visivamente insieme a grafici di esempio generati dall'analisi dei dati nella Figura 2 e nella Figura 3.
- Configurazione del gating FlowJo
- Carica tutti i file FSC su FlowJo trascinandoli e rilasciandoli in un nuovo foglio di lavoro.
- Clicca su Tutti i campioni per iniziare a creare cancelli. Ciò garantirà che i cancelli possano essere facilmente applicati a tutti i campioni.
- Aprire un campione di controllo (ad esempio non trasfettato o non nucleofettato) facendo doppio clic su tale file.
- Crea un gate per le Live Cells in un grafico FSC-A vs SSC-A utilizzando lo strumento poligono. Nell'elenco di tutti i campioni, dovrebbe ora apparire una scheda Celle Live sotto il nome del campione. Fare clic con il pulsante destro del mouse su questo gate e selezionare Copy Analysis to Group (Copia analisi per raggruppare per applicare questo gate a tutti i campioni (Figura 2A, Figura 3A).
- Fare doppio clic sul cancello Celle attive per eseguire il drill-down sulle celle attive. Modificare gli assi in FSC-A e FSC-H (Figura 2B, Figura 3B).
- Disegna un poligono per il cancello solo per singole celle. Applicare questo gate a tutti i campioni come fatto nella sezione 4 (Figura 2B, Figura 3B).
NOTA: Se si esprime costitutivamente un costrutto di sgRNA con un reporter fluorescente, eseguire un ulteriore gating su questo colore in modo che l'analisi si concentri solo sulle cellule che esprimono l'sgRNA. Un esempio di ciò potrebbe essere la creazione di un gate per le cellule mCherry positive (PE-CF594-A vs FSC-A) se le cellule contengono un sgRNA dalla spina dorsale pLG1 (Figura 2C, Figura 3C). - Fare doppio clic sull'ultimo gate di configurazione (celle singole o celle che esprimono guide).
- Se si esegue una trasfezione plasmidico e l'editor dell'epigenoma codifica per una fusione BFP, creare un gate per le cellule BFP positive (esempio: BV421-A vs FSC-A) per i campioni del Giorno 2 (Figura 2D).
- Creare un gate per l'espressione del reporter (esempio: BB515-A vs FSC-A per il gate per le cellule GFP negative) e applicare questo gate a tutti i campioni (Figura 2E, Figura 3D).
- Analisi di trasfezione
- Dopo aver eseguito la configurazione del cancello, fare clic su Editor tabelle nel pannello superiore. Popolazioni di trascinamento per l'analisi (esempi: BFP positivo, GFP negativo).
- Nel pannello dell'editor di tabelle, fare clic su Crea tabella. Copiare questi dati in un foglio di calcolo per eseguire calcoli di normalizzazione.
- Elaborazione dati
- Sottrarre il numero di cellule reporter negative (ad esempio: GFP negative) nel campione di controllo da tutti gli altri campioni. Potrebbe esserci un silenziamento di fondo del reporter, quindi questo rimuove questo rumore di fondo dai dati.
- Tutti i valori saranno normalizzati alla percentuale di cellule BFP positive (cellule trasfettate con successo) misurata il giorno 2. Per eseguire questa normalizzazione, prendere il valore, dividere per la percentuale di celle positive al BFP il giorno 2 e moltiplicare per 100. Questo calcolo produce il valore normalizzato dell'efficienza di trasfezione delle celle modificate. I dati sono ora pronti per essere tracciati. Si consiglia di utilizzare grafici a linee lungo un corso temporale di modifica dell'epigenoma, come illustrato nella Figura 2F.
- Analisi della nucleofezione
- Dopo aver eseguito la configurazione del cancello, fare clic su Editor tabelle nel pannello superiore. Popolazioni di trascinamento per l'analisi (esempi: APC negativo).
- Nel pannello dell'editor di tabelle, fare clic su Crea tabella. Copia questi dati in un editor di fogli di calcolo per eseguire calcoli di normalizzazione.
- Elaborazione dati
- Sottrarre il numero di cellule negative del reporter (ad esempio: APC negative) nel campione di controllo da tutti gli altri campioni. Potrebbe esserci un silenziamento di fondo del reporter, quindi questo rimuove questo rumore di fondo dai dati. I dati sono ora pronti per essere tracciati. Si consiglia di utilizzare grafici a linee in un corso temporale di modifica dell'epigenoma.
NOTA: Le statistiche possono essere utilizzate per quantificare le differenze nel silenziamento tra due diversi editor di epigenomi. Si consiglia di eseguire tutti gli esperimenti di trasfezione e nucleofezione in triplicati tecnici per eseguire analisi statistiche. Quando si rappresentano grafici a linee del silenziamento nel tempo, tracciare la deviazione standard delle repliche tecniche. I t-test degli studenti in momenti specifici potrebbero essere eseguiti per confrontare il silenziamento tra due editor di epigenoma. I campioni devono essere trattati come non appaiati con varianza disuguale.
- Sottrarre il numero di cellule negative del reporter (ad esempio: APC negative) nel campione di controllo da tutti gli altri campioni. Potrebbe esserci un silenziamento di fondo del reporter, quindi questo rimuove questo rumore di fondo dai dati. I dati sono ora pronti per essere tracciati. Si consiglia di utilizzare grafici a linee in un corso temporale di modifica dell'epigenoma.
Risultati
Per tutti gli esperimenti di editing dell'epigenoma, controlli adeguati sono fondamentali per valutare l'efficienza dell'editing dell'epigenoma. Si consiglia di utilizzare un sgRNA di controllo, che non prende di mira alcuna sequenza nel genoma umano. L'utilizzo di un controllo guida non mirato darà la certezza che i cambiamenti nei loci target siano guidati dall'editor dell'epigenoma che viene indirizzato a quel sito piuttosto che solo dalla sovraespressione dell'editor dell'epigenoma ...
Discussione
Questo protocollo descrive in dettaglio due diversi metodi di somministrazione transitoria per gli editor di epigenomi CRISPR: trasfezione del DNA plasmidico e nucleofecazione dell'mRNA. Entrambe le tecniche presentano vantaggi, svantaggi e considerazioni generali (Figura 1F).
La trasfezione del DNA plasmidico porta a una robusta espressione dell'editor dell'epigenoma e includiamo una fusione BFP all'interno dei costrutti dell'edi...
Divulgazioni
J.K.N. è un inventore di brevetti relativi alle tecnologie CRISPRoff/on, depositati da The Regents dell'Università della California.
Riconoscimenti
Ringraziamo i membri del laboratorio Nuñez, in particolare Rithu Pattali e Izaiah Ornelas, per lo sviluppo e l'ottimizzazione dei protocolli descritti in questo manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
Riferimenti
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -. H., Tee, L. Y., Wang, X. -. G., Huang, Q. -. S., Yang, S. -. H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264 (2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136 (2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -. Y., Gingras, A. -. C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. . Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26 (2019).
- Kim, J. -. M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485 (2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. . Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760 (2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460 (2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009 (2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. . CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. . A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082 (2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon