Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
CRISPR-Epigenom-Editierung in menschlichen Zellen mittels Plasmid-DNA-Transfektion und mRNA-Nukleofektion
In diesem Artikel
Zusammenfassung
Das Protokoll beschreibt Methoden zur CRISPR-basierten Epigenom-Editierung (Clustered Regularly Interspaced Short Palindromic Repeats) in humanen Zelllinien unter Verwendung von Plasmid-DNA-Transfektion und mRNA-Nukleofektion.
Zusammenfassung
Epigenetik bezieht sich auf chemische Modifikationen von Histonproteinen und DNA, die die Expression von Genen regulieren können. Das menschliche Epigenom verändert sich dynamisch während der Zelldifferenzierung und des Alterns, und viele Krankheiten sind mit einer abweichenden Epigenommusterung verbunden. Jüngste Fortschritte bei CRISPR haben zur Entwicklung programmierbarer Werkzeuge geführt, um epigenetische Modifikationen an gezielten genomischen Loci zu editieren, die ein präzises Umschreiben epigenetischer Modifikationen in menschlichen Zellen ermöglichen. CRISPR-basierte Epigenom-Editoren stützen sich auf katalytisch totes Cas9 in Verbindung mit epigenetischen Modifikatoren, die letztendlich zu einer programmierten Repression oder Aktivierung von Zielgenen in Säugetiergenomen führen. Im Gegensatz zu herkömmlichen Genom-Editing-Methoden erfordert das Epigenom-Editing keine DNA-Brüche oder Veränderungen in der menschlichen Genomsequenz und dient somit als sicherere Alternative zur Kontrolle der Genexpression. In diesem Protokoll stellen wir zwei verschiedene Methoden zur Durchführung von dCas9-vermittelter Epigenom-Editierung in menschlichen Zelllinien unter Verwendung von Plasmid-DNA-Transfektionen und Nukleofektion von mRNAs vor, die für CRISPR-Epigenom-Editoren kodieren. Wir demonstrieren programmierbare Epigenom-Editierung zur vorübergehenden Unterdrückung von Genen mittels CRISPR-Interferenz (CRISPRi) und zur dauerhaften Stummschaltung von Genen über viele Wochen mit CRISPRoff, einer Fusion von dCas9 mit der KRAB-Domäne und dem de novo DNA-Methyltransferase-Komplex. Wir bieten auch Anleitungen zu quantitativen Methoden zur Messung des erfolgreichen Epigenom-Editings von Zielgenen und wichtige Überlegungen dazu, welches Epigenom-Editing-Tool in Abhängigkeit von experimentellen Kriterien verwendet werden sollte.
Einleitung
Obwohl der genomische Gehalt jeder Zelle in unserem Körper nahezu identisch ist, unterscheidet sich das Transkriptionsprofil der einzelnen Zelltypen stark. Epigenetische Modifikationen an DNA- und Histonproteinen sind Schlüsselregulatoren der transkriptionellen Expression. Transkriptionell aktives Euchromatin zeichnet sich durch ausgeprägte epigenetische Markierungen im Vergleich zu kompaktem, transkriptionell inaktivem Heterochromatin aus. Zum Beispiel werden heterochromatische Regionen durch repressive Histonmodifikationen definiert, einschließlich Trimethylierung an Lysin 9 von Histon 3 (H3K9me3), Trimethylierung an Lysin 27 von Histon H3 (H3K27me3) und DNA-Methylierung an Cytosinen neben Guaninen (CpG) an Genpromotoren1. Genomische Regionen der aktiven Genexpression werden typischerweise durch Histonacetylierung und Trimethylierung an Lysin 4 von Histon 3 (H3K4me3)1 definiert.
Die CRISPR-Revolution (Clustered Regularly Interspaced Short Palindromic Repeats) hat eine Fülle von Werkzeugen hervorgebracht, die eine programmierte Veränderung genomischer Sequenzen ermöglichen. Die CRISPR-Technologie basiert auf einem prokaryotischen Abwehrmechanismus, der in der Lage ist, Nukleinsäuren an programmierbaren Zielsequenzen zu spalten. Die CRISPR-Nukleasen 2,3,4, die Baseneditoren 5,6 und die Prime-Editoren 7,8 können die DNA-Sequenz von Säugetiergenomen durch DNA-Schnitt und Reparatur dieser Brüche verändern. Obwohl diese Strategien effektiv sind, können sie DNA-Brüche an Off-Target-Stellen 9,10 und großflächige genomische Strukturmutationenverursachen 11,12,13,14,15. Alternative CRISPR-basierte Werkzeuge ermöglichen eine steuerbare Modulation der Genaktivierung und -repression, ohne die zugrunde liegende DNA-Sequenz zu verändern. Diese Werkzeuge nutzen ein Nuklease-defizientes Cas9 (dCas9), das eine DNA-Bindung an Zielstellen ermöglicht, die von der sgRNA-Sequenz vorgegeben werden, in Kombination mit Effektorproteinen, die die Chromatinlandschaft verändern 16,17,18. Effektorproteine, wie epigenetische Schreiber, Reader und Radiergummis, können direkt mit dCas9 fusioniert oder durch ein mit dCas9 fusioniertes Peptidgerüst wie SunTag oder ein RNA-Gerüst auf der sgRNA, wie das MS2-MCP-System 16,17,18, rekrutiert werden. Beispiele für programmierbare Transkriptionskontrollwerkzeuge sind die CRISPR-Aktivierung (CRISPRa)19,20,21 und die CRISPR-Interferenz (CRISPRi)22,23. CRISPRa funktioniert, indem es die Transkriptionsmaschinerie direkt rekrutiert und so die Transkriptionsgenexpression des Ziels erhöht19. Im Gegensatz dazu unterdrückt CRISPRi die Transkription, indem es H3K9me3, eine repressive epigenetische Markierung, etabliert22.
Fortschritte in der Epigenom-Editierung haben einen breiten Einsatz dieser Werkzeuge in allen wissenschaftlichen Bereichen ermöglicht. Fusionen verschiedener Effektordomänen und Proteine haben das Toolkit der verfügbaren Epigenom-Editorenerweitert 18,24,25,26,27,28,29,30. Darüber hinaus werden Epigenom-Editoren verwendet, um die Rolle von epigenetischen Modifikationen 31,32,33,34,35,36, Effektoren 37,38,39 und Effektormutationen 28,40,41 zu entschlüsseln in der Genregulation. Insbesondere werden CRISPRi und CRISPRa in funktionellen Genomik-Screenings für eine Vielzahl biologischer Prozesse verwendet, einschließlich des Zellüberlebens42 und des Zellschicksals 43,44,45,46,47. Darüber hinaus birgt die Epigenom-Editierung therapeutisches Potenzial für ex vivo Cell Engineering und in vivo Therapien18.
Hier beschreiben wir Methoden zur Anwendung von zwei dCas9-basierten Epigenom-Editoren für die programmierbare transkriptionelle Repression in humanen Zelllinien: CRISPRi22 und CRISPRoff48. CRISPRi ist eine Fusion von dCas9 mit der repressiven KRAB-Domäne aus einem Zinkfingerprotein wie ZNF10 (KOX1) und ZIM322,23. Wenn CRISPRi auf einen bestimmten Genpromotor abzielt, rekrutiert die KRAB-Domäne einen H3K9me3-Schreiber, SETDB1, um das Zielgen zu unterdrücken (Abbildung 1A). Wenn CRISPRi transient exprimiert wird, wird das etablierte H3K9me3 am Ziellocus nicht aufrechterhalten und die Genexpression wird im Laufe der Zeit wiederhergestellt32,48. Um stabile Knockdowns mit CRISPRi durchzuführen, z. B. in der funktionellen Genomik, ist die konstitutive Expression von CRISPRi in den Zellen zusammen mit der sgRNA unerlässlich. Vor kurzem wurde CRISPRoff entwickelt, um die Bearbeitung von vererbbaren Epigenomen zu programmieren48. CRISPRoff ist eine Einzelproteinfusion von dCas9 mit der KRAB-Domäne und dem de novo DNA-Methyltransferase-Komplex, DNMT3A und DNMT3L. Ein transienter Impuls von CRISPRoff in menschlichen Zellen programmiert die Ablagerung von H3K9me3 und die DNA-Methylierung an den Zielgenen, was zu einer langfristigen Repression der Zielgene durch die Aufrechterhaltung der DNA-Methylierung und von H3K9me3 führt (Abbildung 1B)48. Darüber hinaus können Epigenom-Bearbeitungen rückgängig gemacht werden. Zum Beispiel kann ein Gen, das durch CRISPRoff stabil zum Schweigen gebracht wird, durch TET1-dCas9 reaktiviert werden, das die DNA-Methylierungsmarkierungen an den Zielloci49 enzymatisch entfernen kann.
In diesem Protokoll werden zwei Verabreichungsmethoden für die transiente Expression von Epigenom-Editoren beschrieben: die Plasmid-DNA-Transfektion und die mRNA-Nukleofektion. Darüber hinaus skizzieren wir, wie die Durchflusszytometrie zur Beurteilung der Wirksamkeit der Epigenom-Editierung an zwei endogenen Genen, CLTA und CD55, verwendet werden kann. Diese Methoden können mit zusätzlichen Editoren angepasst und auf andere Epigenom-Editing-Experimente angewendet werden oder für das Targeting verschiedener Gene verwendet werden.
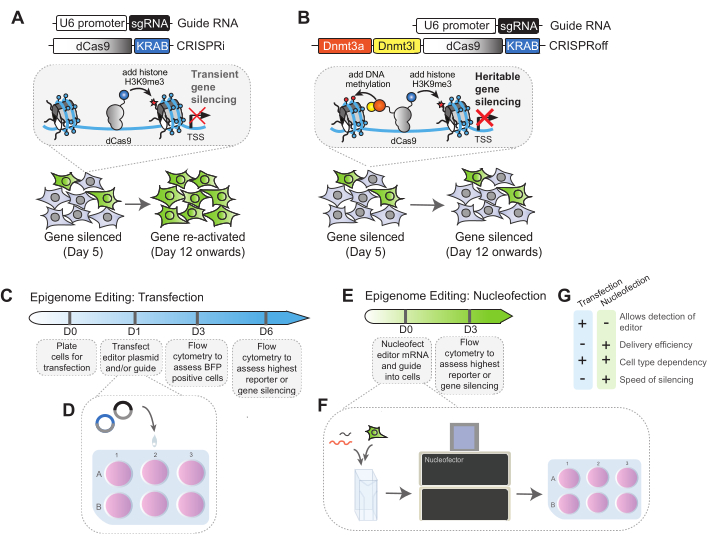
Abbildung 1: Schematische Darstellung des Mechanismus und des Arbeitsablaufs der Epigenom-Editierung von CRISPRi und CRISPRoff. (A) Lineare Schemata des sgRNA-Transgens und des CRISPRi-Epigenom-Editors. Die Zugabe von CRISPRi und sgRNA ermöglicht die Zugabe der repressiven H3K9me3-Histonmarkierung, um den Ziellocus zum Schweigen zu bringen. Der höchste Grad an Silencing wird früh nach der CRISPRi-Zugabe erreicht, und das Genziel wird in der Regel nach einigen Passagen reaktiviert. (B) Lineare Schemata des sgRNA-Transgens und des CRISPRoff-Epigenom-Editors. Die Zugabe von CRISPRoff zu Zellen, die auf ein Gen von Interesse abzielen, führt zur Zugabe von repressivem H3K9me3 zusammen mit DNA-Methylierung an CpG-Stellen, um das Zielgen zum Schweigen zu bringen. Das Silencing durch CRISPRoff ist vererbbar - das hohe Silencing wird früh während der Transfektion erreicht und bleibt über mehrere Zellteilungen bestehen. (C) Überblick über den Zeitplan für die Epigenom-Editierung mittels Transfektionsmethode. Am Tag 0 werden die Zellen für die Transfektion plattiert. An Tag 1 kann das Editor- und Guide-Plasmid per Transfektion in die Zellen eingebracht werden. An Tag 3 werden die Zellen mittels Durchflusszytometrie auf BFP-Expression untersucht. Der Prozentsatz der BFP wird als Normalisierungsfaktor verwendet, um die endgültige Stummschaltungswirksamkeit des Experiments für jede Bedingung zu bestimmen. Ab Tag 6 werden die Zellen auf Stummschaltung des interessierenden Reporters analysiert, da an diesem Tag der höchste Grad an Stummschaltung erreicht wird. (D) Überblick über die Transfektionsmethode, wobei Epigenom-Editor und sgRNA-Plasmide den Zellen tropfenweise zugesetzt werden. (E) Überblick über den zeitlichen Ablauf der Epigenom-Editierung mit Hilfe der Nukleofektionsmethoden. In diesem Protokoll ist mRNA die Nukleofektion in die Zellen am Tag 0. Die Zellen werden am Tag 3 nach der Nukleofektion mittels Durchflusszytometrie auf Silencing untersucht. (F) Überblick über das Nukleofektionsprotokoll. Entsprechende Mengen an Zellen und mRNA werden gemischt und in Nukleofektorküvetten gegeben. Wird die sgRNA durch Nukleofektion eingebracht, kann sie auch zu diesem Gemisch hinzugefügt werden. Die Küvetten werden in den Nukleofektor gegeben, und geeignete Pulscodes werden verwendet, um die mRNA in die Zellen einzuführen. Nach der Nukleofektion werden die Zellen plattiert und für die Analyse in späteren Tagen durchgelassen. (G) Vergleich zwischen Plasmidtransfektion und mRNA-Nukleofektionsstrategien für die Epigenom-Editierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
HINWEIS: Die ergänzende Datei 1 enthält Details über das sgRNA-Design, die Klonierung und die Zellliniengenerierung unserer repräsentativen Daten. Im Abschnitt "Repräsentative Ergebnisse" finden Sie auch Vorschläge für Steuerelemente.
1. Transfektion von Epigenom-Editor-exprimierenden Plasmiden in HEK293T Zellen
HINWEIS: Dieses Protokoll beschreibt die Verabreichung von CRISPR-kodierenden Plasmiden in HEK293T Zellen. Wir haben Zellen so verändert, dass sie eine sgRNA (Addgene 217306) exprimieren, die auf die Promotorregion von CLTA abzielt, einem nicht-essentiellen Gen, das endogen mit GFP markiert ist. Die CLTA-GFP HEK 293T-Zellen stammen aus einer früheren Studie50. In diesem Beispiel sind die Epigenom-Editoren direkt mit einem blau fluoreszierenden Protein (BFP) fusioniert, was es uns ermöglicht, die Transfektionseffizienz zu quantifizieren und eine genaue Bewertung der experimentellen Bedingungen zu gewährleisten. Die Wirksamkeit des Ansatzes wird durch das Silencing von CLTA-GFP demonstriert, das mittels Durchflusszytometrie quantitativ auf Proteinebene in Einzelzellen gemessen werden kann.
- Zellkultur
- Halten Sie HEK293T Zellen in hohem Glukose-DMEM mit 10% FBS und 1x Penicillin-Streptomycin-Glutamin. Passieren Sie die Zellen alle 2-3 Tage und stellen Sie sicher, dass sie bei einer Konfluenz von 60%-70% bleiben.
HINWEIS: Um eine optimale Transfektionseffizienz zu gewährleisten, ist es wichtig, die Zellkonfluenz unter 70 % zu halten, die täglich unter einem Mikroskop überwacht werden kann.
- Halten Sie HEK293T Zellen in hohem Glukose-DMEM mit 10% FBS und 1x Penicillin-Streptomycin-Glutamin. Passieren Sie die Zellen alle 2-3 Tage und stellen Sie sicher, dass sie bei einer Konfluenz von 60%-70% bleiben.
- Tag 0: Aussaat HEK293T Zellen für die Transfektion
- Zählen Sie am Tag vor der Transfektion Zellen mit einem automatisierten Zellzähler unter Verwendung von Trypanblau-Lebend-/Totfärbung und säen Sie ~1,5 x 104 lebende Zellen pro Well in einer 96-Well-Platte aus. Halten Sie das endgültige Volumen jeder Vertiefung bei 200 μl. Die Zellen sollten am nächsten Tag eine Konfluenz von 60 % bis 70 % erreichen.
HINWEIS: Dies kann auch in 24-Well- oder 6-Well-Platten effizient durchgeführt werden. Siehe Tabelle 1 für die Zellzahl und die DNA-Menge für die Transfektion.
- Zählen Sie am Tag vor der Transfektion Zellen mit einem automatisierten Zellzähler unter Verwendung von Trypanblau-Lebend-/Totfärbung und säen Sie ~1,5 x 104 lebende Zellen pro Well in einer 96-Well-Platte aus. Halten Sie das endgültige Volumen jeder Vertiefung bei 200 μl. Die Zellen sollten am nächsten Tag eine Konfluenz von 60 % bis 70 % erreichen.
- Tag 1: Transfektion von CRISPRoff- und CRISPRi-Plasmiden in HEK29T-Zellen
- Aliquotieren Sie 150 ng des Epigenom-Editor-Plasmids (Addgene 167981) für jede Vertiefung in 3 μL Gesamtvolumen. Diese Plasmidverdünnungen können in PCR-Streifenröhrchen hergestellt werden.
HINWEIS: Wenn der Epigenom-Editor und das sgRNA-Plasmid gemeinsam transfiziert werden, empfehlen wir, ein molares Verhältnis von editor:sgRNA-Plasmiden von 3:4 zu versuchen. Das Verhältnis muss möglicherweise optimiert werden, um die höchste Effizienz der Epigenom-Editierung zu erzielen. - Das Transfektionsreagenz und das minimal essentielle Medium ca. 30-60 min vor der Transfektion auf Raumtemperatur vorwärmen. Mischen Sie 0,6 μl Transfektionsreagenz und 20 μl minimal essentielles Medium pro Vertiefung und inkubieren Sie 15 Minuten lang bei Raumtemperatur.
HINWEIS: Ein Mastermix aus Transfektionsreagenz und minimalem essentiellem Medium kann hergestellt werden, wenn mehrere Transfektionen durchgeführt werden. - Geben Sie 20,6 μl des Transfektionsreagenzes und einer minimalen Mischung aus essentiellem Medium in jedes PCR-Streifenröhrchen, das 3 μl Plasmid-DNA enthält. 15 Minuten bei Raumtemperatur ziehen lassen.
- Geben Sie die Mischung langsam auf die Platte mit den ausgesäten Zellen und klopfen Sie vorsichtig auf die Platte, um die Mischung zu verteilen. Fügen Sie eine nicht transfizierte Kontrolle hinzu, um eine geeignete Grundlage für den Vergleich in nachgelagerten Analysen zu schaffen.
HINWEIS: Wenn Sie die Transfektionsmischung zu ausgesäten Zellen hinzufügen, ist es wichtig, dies tropfenweise und sanft zu tun, um Störungen zu minimieren, da dies die Transfektionseffizienz beeinträchtigen kann.
- Aliquotieren Sie 150 ng des Epigenom-Editor-Plasmids (Addgene 167981) für jede Vertiefung in 3 μL Gesamtvolumen. Diese Plasmidverdünnungen können in PCR-Streifenröhrchen hergestellt werden.
- Tag 2: Überprüfen Sie den Erfolg der Transfektion
- Untersuchen Sie Zellen unter einem Mikroskop mit 10-facher Vergrößerung, um festzustellen, ob sie BFP-positiv sind, da dies auf eine erfolgreiche Transfektion hinweist. Stellen Sie sicher, dass die Zellen während des Beobachtungsprozesses nicht gestört werden.
- Tag 3: Teilen Sie die 96-Well-Platte und beurteilen Sie die Transfektionseffizienz mit dem BFP-Marker
HINWEIS: Eine erfolgreiche Transfektion sollte mindestens 30% der Zellen als BFP-positiv ergeben. Mit dCas9, CRISPRi und CRISPRoff weisen wir jedoch in der Regel zwei Tage nach der Transfektion mehr als 60 % BFP-positive Zellen nach. Die Transfektionseffizienz korreliert negativ mit der Plasmidgröße, so dass verschiedene Epigenom-Editoren unterschiedliche Transfektionseffizienzen erzielen können.- Trypsin und DMEM mit 10% FBS und 1x Penicillin-Streptomycin-Glutamin ca. 30-60 min vor der Anwendung auf Raumtemperatur erwärmen.
- Saugen Sie Medien aus den Vertiefungen an; Achten Sie darauf, keine Zellen zu entfernen, die an der Unterseite befestigt sind. Fügen Sie 50 μl Trypsin pro Vertiefung hinzu. 2-3 min bei 37 °C inkubieren.
HINWEIS: Es ist nicht ungewöhnlich, einige schwebende Zellen zu beobachten, insbesondere nach der Transfektion mit CRISPRoff. Wir vermuten, dass dieser Zelltod auf die Toxizität der Überexpression von DNMT3A-3L zurückzuführen ist. - Hemmen Sie Trypsin, indem Sie 150 μl vollständiges DMEM pro Vertiefung hinzufügen. Pipettieren Sie auf und ab, um Zellen zu resuspendieren, und teilen Sie Zellen für die Konfluenz an Tag 5 (~20 μl Zellmischung in 200 μl Medium einer 96-Well-Platte).
- Verwenden Sie 50 μl resuspendierte Zellen für die Durchflusszytometrie, um die Transfektionseffizienz zu beurteilen.
2. Nukleofektion von Epigenom-Editor-mRNA in K-562-Zellen
HINWEIS: In diesem Abschnitt wird der Prozess der Nukleofektion von CRISPRoff-mRNA in K-562-Zellen beschrieben. Der Einfachheit halber haben wir die K-562-Zellen so konstruiert, dass sie konstitutiv eine sgRNA exprimieren, die auf den Promotor des endogenen CD55-Gens (Addgene 217306) abzielt. Die direkte Verabreichung von CRISPRoff-mRNA an die Zellen hat das Potenzial, die zelluläre Toxizität zu verringern, die mit Plasmid-DNA-basierten Ansätzen einhergeht, während gleichzeitig eine ähnliche Wirksamkeit beim Gen-Silencing erreicht wird. Darüber hinaus kann die Nukleofektion verwendet werden, um Epigenom-Editor-Konstrukte in Zelllinien einzuführen, die nur schwer effizient transfiziert werden können, wie z. B. K-562s.
- Zellkultur
- Vor der Nukleofektion werden K-562-Zellen in einem Kolben mit RPMI mit 10 % FBS und 1x Penicillin-Streptomycin-Glutamin aufbewahrt. Passieren Sie die Zellen jeden Tag und stellen Sie sicher, dass sie bei einer Konfluenz von 60 % bis 70 % bleiben.
- Tauen Sie die CRISPRoff-mRNA am Tag der Nukleofektion in einem sterilen RNase-freien Mikrozentrifugenröhrchen auf und lagern Sie sie auf Eis. Verwenden Sie 2 μg mRNA pro 2,0 x 105 Zellen in jeder Vertiefung einer Streifennukleokuvette. Stellen Sie sicher, dass das Volumen der mRNA 10 % des gesamten Nukleofektionsvolumens (22 μl) nicht überschreitet.
HINWEIS: Die Nukleofektion kann auch in größeren Küvetten mit mehr Zellen und mRNA durchgeführt werden. Wir empfehlen, die Menge an mRNA proportional zur Anzahl der Zellen zu skalieren, die Sie hinzufügen werden. Wenn Sie beispielsweise eine Nukleofektion in einer großen Küvette an 1 x 106 Zellen durchführen, empfehlen wir die Verwendung von 10 μg mRNA mit einem Gesamtnukleofektionsvolumen von 100 μl. Darüber hinaus kann die Dosierung der mRNA optimiert werden, um eine hohe Editierungseffizienz zu gewährleisten. Wir verwenden oft 2-5 μg Editor-mRNA pro 2,0 x 105 Zellen, um effiziente Ergebnisse zu erzielen. Freie Nukleotide, die während der mRNA-Synthese nicht eingebaut wurden, sollten vor der Quantifizierung entfernt werden. - Bereiten Sie die Nukleofektionslösung gemäß den Anweisungen des Herstellers vor und erwärmen Sie sie vor der Nukleofektion 15 Minuten lang auf Raumtemperatur.
HINWEIS: Die Nukleofektorlösung kann unterschiedlich sein oder muss für verschiedene Zelltypen optimiert werden. - Ernten und zählen Sie K-562-Zellen mit einem automatisierten Zellzähler mit Trypanblau-Lebend-/Totfärbung. Für die Nukleofektion in einer Streifenküvette aliquotieren Sie ~2,0 x 105 Zellen pro Probe in ein steriles Mikrozentrifugenröhrchen.
- Die Zellen werden bei 500 x g 5 min bei Raumtemperatur zentrifugiert, dann wird der Überstand verworfen. Waschen Sie die Zellen 1x mit Raumtemperatur PBS bei 500 x g für 5 min, dann entsorgen Sie den Überstand.
- Berechnen Sie die Menge an Nukleofektorlösung, in der die Zellen resuspendiert werden sollen, indem Sie das mRNA-Volumen von 22 μl subtrahieren. Resuspendieren Sie die Zellen in der entsprechenden Menge der Nukleofektorlösung.
- Geben Sie Zelllösung zu 2 μg CRISPRoff-mRNA (Volumen bestimmt in Schritt 2.1.2) und geben Sie die Lösung in eine Küvette, wobei Sie darauf achten müssen, dass sich keine Blasen bilden, da dies die Effizienz der Nukleofektion beeinträchtigen kann. Klopfen Sie vorsichtig auf die Nukleocuvette, um sicherzustellen, dass sich die Zellen unten befinden.
- Nukleofectieren Sie die Zellen mit dem 4D-Nucleofector-System mit dem entsprechenden Pulscode. Für K-562-Zellen wird der FF-120-Code empfohlen.
HINWEIS: Der Impulscode muss möglicherweise für verschiedene Zelltypen optimiert werden. Wenden Sie sich an den Hersteller, wenn eine zusätzliche Optimierung erforderlich ist. - Geben Sie nach der Nukleofektion 80 μl RPMI-Medium in jede kleine Nukleocuvetten-Vertiefung und lassen Sie die Zellen 15 Minuten lang bei 37 °C ruhen.
- Die Zellsuspension wird in eine Vertiefung einer 24-Well-Platte mit 400 μl vorgewärmtem RPMI-Medium (kleine Nukleokuvette) überführt.
HINWEIS: Nach der Nukleofektion sind die Zellen besonders zerbrechlich und sollten vorsichtig von der Nukleocuvette in die Kulturvertiefungen überführt werden. - Nach Tag 2 nach der Nukleofektion: Befolgen Sie das Oberflächenmarker-Färbeprotokoll mit CD55-Antikörpern, wie in Abschnitt 3 beschrieben, und messen Sie die Fluoreszenz von CD55 APC auf einem Durchflusszytometer, um die Wirksamkeit der Epigenom-Editierung zu testen.
HINWEIS: Um den Erfolg der Nukleofektion zu testen, empfehlen wir, eine Nukleofektion mit GFP oder mCherry mRNA durchzuführen. Führen Sie zwei Tage nach der Nukleofektion eine Durchflusszytometrie dieser Zellen durch, um eine erfolgreiche Nukleofektion zu bestätigen. Wir weisen in der Regel 90% bis 100% dieser Zellen nach, die GFP oder mCherry exprimieren. Eine Alternative besteht darin, eine Nukleofektion mit CRISPRi-mRNA durchzuführen, von der bekannt ist, dass sie viele Zielgene robust zum Schweigen bringt. Bei der Durchführung der Durchflusszytometrie an Tag 2 sollten CRISPRi-behandelte Proben in über 90 % der Zellen ein Zielgen-Silencing zeigen.
3. Färben von Oberflächenmarkern
HINWEIS: In diesem Abschnitt wird die Quantifizierung der CD55-Proteinspiegel nach Epigenom-Editierung in K-562-Zellen beschrieben. Wir quantifizieren die Abnahme der CD55-Expression in Einzelzellen mit Hilfe von Antikörperfärbung und Durchflusszytometrie (siehe Abschnitt 4 unten), um die CRISPRoff-vermittelte Knockdown-Effizienz zu bewerten. Zusätzliche Techniken, einschließlich quantitativer PCR der reversen Transkription oder Western Blotting, können ebenfalls verwendet werden, um den Grad des Knockdowns sowohl auf Transkript- als auch auf Proteinebene zu bestätigen.
- Zählen Sie die Zellen mit einem automatisierten Zellzähler und geben Sie etwa 5,0 x 105 bis 1,0 x 106 Zellen in ein Mikrozentrifugenröhrchen und zentrifugieren Sie die Zellen bei 500 x g für 5 min.
HINWEIS: Es können mehr oder weniger Zellen für die Färbung verwendet werden, jedoch muss die Konzentration der Antikörper möglicherweise entsprechend skaliert werden. - Entsorgen Sie den Überstand, achten Sie darauf, das Pellet nicht zu stören, fügen Sie 500 μl PBS hinzu und resuspendieren Sie das Pellet vorsichtig.
- Zentrifugieren Sie die Zellen erneut bei 500 x g für 5 min. Den Überstand verwerfen und den Antikörper in der gewünschten Konzentration in PBS verdünnt hinzufügen. Für die CD55-Färbung verwenden Sie 1 μl gereinigtes Anti-Human-CD55 (0,5 mg/ml) in 49 μl PBS.
- Pipettieren Sie die Zellen vorsichtig und lassen Sie sie 25 Minuten lang an einem dunklen Ort bei Raumtemperatur inkubieren. Nach der Inkubation die Zellen erneut bei 500 x g für 5 min drehen und den Überstand verwerfen.
- Resuspendieren Sie das Pellet mit 500 μl PBS, um überschüssige Antikörper abzuwaschen. Die Zellen werden 5 min lang bei 500 x g zentrifugiert und der PBS-Überstand verworfen. In 100 μl bis 200 μl frischem PBS resuspendieren und für die Durchflusszytometrie-Analyse auf eine 96-Well-Platte geben.
4. Durchflusszytometrie
HINWEIS: Dieses Protokoll wurde für die Verwendung eines BD FACSymphony A1 Cell Analyzers geschrieben. Die Einzelheiten können je nach verwendetem Durchflusszytometer variieren. Wir empfehlen Ihnen, sich für Einzelheiten auf das Benutzerhandbuch der Maschine zu beziehen, die Sie verwenden.
- Vergewissern Sie sich vor dem Einschalten der Zytometrie, dass der Abfallbehälter nicht voll ist und ob genügend Scheidenflüssigkeit vorhanden ist. Schalten Sie den Hauptschalter (grün) ein, der sich in der Nähe der Mantelflüssigkeit befindet.
- Schalten Sie den Plattenleser mit dem Rückschalter ein. Schalten Sie dann das Gerät mit der grünen Taste auf der rechten Seite ein und stellen Sie sicher, dass das Gerät auf die 96-Well-Plattenlesefunktion eingestellt ist.
- Schalten Sie den Computer ein und melden Sie sich an. Öffnen Sie die Software und melden Sie sich mit Ihrem Benutzernamen und Passwort an.
- Richten Sie das Experiment ein. Erstellen Sie ein neues Experiment, indem Sie Experiment > Neues Experiment auswählen. Benennen Sie das Experiment um, indem Sie auf den aktuellen Namen doppelklicken.
- Fügen Sie eine Platte hinzu, indem Sie auf Experiment > Neue Platte klicken. Wählen Sie das zu verwendende Well-Format aus. Um bestimmte Vertiefungen zum Ausführen hinzuzufügen, klicken Sie auf die Position der Vertiefung in der Platte und dann auf die blaue Spritze (die Vertiefung sollte jetzt blau hervorgehoben sein). Wählen Sie bei Bedarf mehrere Wells gleichzeitig aus, um sie hinzuzufügen.
- Fügen Sie jeder Vertiefung spezifische Namen hinzu, indem Sie mit der rechten Maustaste auf Probe auf der rechten Seite des Plattendisplays klicken. Wählen Sie dann Experimentelles Layout aus. Daraufhin wird eine Seite geöffnet, auf der alle von Ihnen ausgewählten Vertiefungen aufgelistet sind. Um die Vertiefungen umzubenennen, klicken Sie einmal und geben Sie den gewünschten Namen ein. Klicken Sie auf OK , wenn Sie fertig sind.
- Fügen Sie dem Experiment auf dem globalen Arbeitsblatt Diagramme und entsprechende Gates hinzu. Es wird empfohlen, zuerst die Vorwärtsstreufläche (FSC-A) und die Seitenstreufläche (SSC-A) darzustellen. Erstellen Sie dann ein beliebiges Gate für aktive Zellen, um es später zu verfeinern, indem Sie auf das Polygon-Werkzeug klicken.
- Klicken Sie mit der rechten Maustaste auf dieses Tor, und klicken Sie auf Drilldown. Dadurch wird ein neues Diagramm erstellt, in dem nur Zellen aus diesem Live-Zellen-Gate angezeigt werden.
- Ändern Sie die Achse dieses neuen Diagramms in FSC-A vs. Vorwärtsstreuungshöhe (FSC-H), und erstellen Sie ein Gate für einzelne Zellen.
- Wenn die sgRNA konstitutiv in Zellen exprimiert wird und das Lentivirus auch für ein fluoreszierendes Protein kodiert, dann bohren Sie dieses einzelne Zellgatter auf und führen Sie ein weiteres Gating für die Expression dieses Proteins als Proxy für die sgRNA-Expression durch (Beispiel: PE-CF594-A vs. FSC-A).
- Klicken Sie auf dem endgültigen Setup-Gate (entweder für einzelne Zellen oder für sgRNA-exprimierende Zellen) mit der rechten Maustaste, und wählen Sie Drilldown aus. Erstellen Sie ein Gate, um die Expression des Reportergens zu testen (Beispiel: BB515-A vs. FSC-A).
- Wenn Sie transfizierte Zellen verwenden und über eine Methode zum Testen der Transfektionseffizienz verfügen, z. B. die BFP-Expression, klicken Sie mit der rechten Maustaste auf das Reporter-Expressionsdiagramm , und klicken Sie auf Duplizieren. Ändern Sie die Achsen in BV421-A und FSC-A. Erstellen Sie ein beliebiges Gate für BFP-positive Zellen.
- Entfernen Sie alle Parameter, die nicht auf der Registerkarte "Parameter" des Zytometer-Displays verwendet werden.
- Um das Gerät einzurichten und für den Betrieb vorzubereiten, klicken Sie auf dem Bedienfeld des Durchflusszytometers auf Ausführen . Die Schaltfläche sollte grün erscheinen. Gehen Sie in der Software im oberen Bereich auf HTS und dann auf Prime. Wenn Sie fertig sind, fügen Sie die Platte hinzu und stellen Sie sicher, dass sie gut in der Maschine positioniert ist und der Deckel ausgeschaltet ist.
- Wählen Sie zum Ausführen alle Wells aus, die ausgeführt werden sollen, und stellen Sie sicher, dass die Steuerelemente ordnungsgemäß festgelegt sind. Wählen Sie die Vertiefung durch Doppelklick aus, ändern Sie das Probenvolumen auf 10 μl und klicken Sie im Erfassungs-Dashboard auf Daten erfassen . Dadurch wird das Beispiel ausgeführt, aber die Daten werden nicht aufgezeichnet. Während dieses Muster läuft, passen Sie die Laserspannungen nach Bedarf an. Ändern Sie außerdem die Tore.
HINWEIS: Wir empfehlen, eine Stichprobe durchzuführen, die für alle gemessenen Farben negativ und für alle Farben positiv ist, um sicherzustellen, dass die Laserparameter und das Gating gut sind. - Nachdem Sie die Lasereinstellungen überprüft haben, klicken Sie auf die Proben im Plate Panel und überprüfen Sie, ob die Laufwerte wie gewünscht eingestellt sind.
- Klicken Sie auf Platte ausführen , um im Erfassungs-Dashboard auszuführen und Daten aus allen Bohrlöchern aufzuzeichnen. Wenn Sie fertig sind, reinigen Sie die Maschine, indem Sie die Platte entfernen.
- Zu einer neuen 96-Well-Platte fügen Sie 10 % Bleiche in den oberen vier Reihen und H2O in den vier Wells darunter hinzu. Gehen Sie zu HTS > Clean. Wenn Sie fertig sind, schließen Sie das Programm und schalten Sie die Tasten in der umgekehrten Reihenfolge aus, in der sie eingeschaltet wurden: grüne Seitentaste > Tellerleser > grüner Schalter durch Abfall.
- Geben Sie 20 % Bleichmittel in die 96-Well-Platte mit den Zellen und entsorgen Sie es.
5. Datenanalyse
HINWEIS: Diese Methode beschreibt Gating-Strategien und Datenverarbeitung zur Quantifizierung der Epigenom-Editierung durch Durchflusszytometrie. Die Gating-Strategie wird visuell zusammen mit Beispieldiagrammen dargestellt, die aus der Datenanalyse in Abbildung 2 und Abbildung 3 generiert wurden.
- Transfektionsanalyse
- Nachdem Sie die Gating-Einrichtung durchgeführt haben, klicken Sie im oberen Bereich auf Tabelleneditor . Drag Populationen für die Analyse (Beispiele: BFP positiv, GFP negativ).
- Klicken Sie im Bereich des Tabelleneditors auf Tabelle erstellen. Kopieren Sie diese Daten in eine Tabelle, um Normalisierungsberechnungen durchzuführen.
- Datenverarbeitung
- Subtrahieren Sie die Anzahl der negativen Reporterzellen (Beispiel: GFP-negativ) in der Kontrollstichprobe von allen anderen Proben. Möglicherweise kommt es zu einer Hintergrundstummschaltung des Reporters, sodass dieses Hintergrundrauschen aus den Daten entfernt wird.
- Alle Werte werden auf den Prozentsatz der BFP-positiven Zellen (erfolgreich transfizierte Zellen) normalisiert, der an Tag 2 gemessen wurde. Um diese Normalisierung durchzuführen, nehmen Sie den Wert, dividieren Sie ihn durch den Prozentsatz der BFP-positiven Zellen an Tag 2 und multiplizieren Sie ihn mit 100. Diese Berechnung ergibt den normierten Wert der Transfektionseffizienz der editierten Zellen. Die Daten können nun geplottet werden. Wir empfehlen Liniendiagramme über einen Zeitverlauf der Epigenom-Editierung, wie in Abbildung 2F dargestellt.
- Nukleofektions-Analyse
- Nachdem Sie die Gating-Einrichtung durchgeführt haben, klicken Sie im oberen Bereich auf Tabelleneditor . Drag Populationen für die Analyse (Beispiele: APC negativ).
- Klicken Sie im Bereich des Tabelleneditors auf Tabelle erstellen. Kopieren Sie diese Daten in einen Tabellenkalkulationseditor, um Normalisierungsberechnungen durchzuführen.
- Datenverarbeitung
- Subtrahieren Sie die Anzahl der negativen Reporterzellen (Beispiel: APC-negativ) in der Kontrollstichprobe von allen anderen Stichproben. Möglicherweise kommt es zu einer Hintergrundstummschaltung des Reporters, sodass dieses Hintergrundrauschen aus den Daten entfernt wird. Die Daten können nun geplottet werden. Wir empfehlen Liniendiagramme über einen Zeitverlauf der Epigenom-Editierung.
HINWEIS: Statistiken können verwendet werden, um Unterschiede im Silencing zwischen zwei verschiedenen Epigenom-Editoren zu quantifizieren. Wir empfehlen, alle Transfektions- und Nukleofektionsexperimente in technischer Ausfertigung durchzuführen, um statistische Analysen durchführen zu können. Bei der Darstellung von Liniendiagrammen der Stummschaltung im Zeitverlauf ist die Standardabweichung technischer Replikate darzustellen. Studentische t-Tests zu bestimmten Zeitpunkten könnten durchgeführt werden, um das Silencing zwischen zwei Epigenom-Editoren zu vergleichen. Stichproben sollten als ungepaart mit ungleicher Varianz behandelt werden.
- Subtrahieren Sie die Anzahl der negativen Reporterzellen (Beispiel: APC-negativ) in der Kontrollstichprobe von allen anderen Stichproben. Möglicherweise kommt es zu einer Hintergrundstummschaltung des Reporters, sodass dieses Hintergrundrauschen aus den Daten entfernt wird. Die Daten können nun geplottet werden. Wir empfehlen Liniendiagramme über einen Zeitverlauf der Epigenom-Editierung.
Ergebnisse
Bei allen Epigenom-Editing-Experimenten sind geeignete Kontrollen entscheidend, um die Effizienz der Epigenom-Editierung zu bewerten. Wir empfehlen die Verwendung einer Kontroll-sgRNA, die auf keine Sequenz im menschlichen Genom abzielt. Die Verwendung einer Nicht-Targeting-Guide-Kontrolle gibt die Gewissheit, dass Änderungen an den Zielloci durch den Epigenom-Editor verursacht werden, der an diese Stelle geleitet wird, und nicht nur durch Überexpression oder unspezifische Bindung des ...
Diskussion
Dieses Protokoll beschreibt zwei verschiedene transiente Verabreichungsmethoden für CRISPR-Epigenom-Editoren: Plasmid-DNA-Transfektion und mRNA-Nukleofektion. Beide Techniken haben einzigartige Vor- und Nachteile und allgemeine Überlegungen (Abbildung 1F).
Die Transfektion von Plasmid-DNA führt zu einer robusten Expression des Epigenomeditors, und wir schließen eine BFP-Fusion in die Konstrukte des Epigenomeditors ein, die den...
Offenlegungen
J.K.N. ist ein Erfinder von Patenten im Zusammenhang mit den CRISPRoff/on-Technologien, die von den Regenten der University of California eingereicht wurden.
Danksagungen
Wir danken den Mitgliedern des Nuñez-Labors, insbesondere Rithu Pattali und Izaiah Ornelas, für die Entwicklung und Optimierung der in diesem Manuskript beschriebenen Protokolle.
Materialien
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
Referenzen
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -. H., Tee, L. Y., Wang, X. -. G., Huang, Q. -. S., Yang, S. -. H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264 (2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136 (2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -. Y., Gingras, A. -. C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. . Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26 (2019).
- Kim, J. -. M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485 (2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. . Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760 (2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460 (2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009 (2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. . CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. . A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082 (2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten