Method Article
Edição de Epigenoma CRISPR em Células Humanas usando Transfecção de DNA de Plasmídeo e Entrega de Nucleofecção de mRNA
Neste Artigo
Resumo
O protocolo descreve métodos para edição de epigenoma baseada em Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR) em linhagens de células humanas usando transfecção de DNA de plasmídeo e nucleofecção de mRNA.
Resumo
Epigenética refere-se a modificações químicas de proteínas histonas e DNA que podem regular a expressão de genes. O epigenoma humano é alterado dinamicamente durante a diferenciação celular e o envelhecimento, e muitas doenças estão associadas a padrões epigenomas aberrantes. Avanços recentes no CRISPR levaram ao desenvolvimento de ferramentas programáveis para editar modificações epigenéticas em loci genômicos direcionados, permitindo a reescrita precisa de modificações epigenéticas em células humanas. Os editores de epigenoma baseados em CRISPR dependem de Cas9 cataliticamente morto, juntamente com modificadores epigenéticos que, em última análise, resultam em repressão programada ou ativação de genes-alvo em genomas de mamíferos. Ao contrário dos métodos tradicionais de edição do genoma, a edição do epigenoma não requer quebras de DNA ou alterações na sequência do genoma humano e, portanto, serve como uma alternativa mais segura para controlar a expressão gênica. Neste protocolo, destacamos dois métodos diferentes para realizar a edição do epigenoma mediada por dCas9 em linhagens de células humanas usando transfecções de DNA de plasmídeo e nucleofecção de mRNAs que codificam editores de epigenoma CRISPR. Demonstramos a edição programável do epigenoma para reprimir transitoriamente genes usando interferência CRISPR (CRISPRi) e para silenciar genes de forma duradoura por muitas semanas usando CRISPRoff, uma fusão de dCas9 com o domínio KRAB e complexo metiltransferase de DNA de novo . Também fornecemos orientação sobre métodos quantitativos para medir a edição bem-sucedida do epigenoma de genes-alvo e as principais considerações sobre qual ferramenta de edição de epigenoma usar, dependendo dos critérios experimentais.
Introdução
Embora o conteúdo genômico de todas as células do nosso corpo seja quase idêntico, o perfil transcricional de cada tipo de célula difere muito. Modificações epigenéticas no DNA e nas proteínas histonas são os principais reguladores da expressão transcricional. A eucromatina transcricionalmente ativa é marcada por marcas epigenéticas distintas em comparação com a heterocromatina compacta e transcricionalmente inativa. Por exemplo, as regiões heterocromáticas são definidas por modificações repressivas de histonas, incluindo trimetilação na lisina 9 da histona 3 (H3K9me3), trimetilação na lisina 27 da histona H3 (H3K27me3) e metilação do DNA em citosinas próximas a guaninas (CpG) nos promotores do gene1. As regiões genômicas da expressão gênica ativa são definidas tipicamente pela acetilação e trimetilação de histonas na lisina 4 da histona 3 (H3K4me3)1.
A revolução das repetições palindrômicas curtas agrupadas regularmente interespaçadas (CRISPR) gerou uma grande variedade de ferramentas que permitem a alteração programada de sequências genômicas. A tecnologia CRISPR é baseada em um mecanismo de defesa procariótico capaz de clivar ácidos nucléicos em sequências-alvo programáveis. As nucleases CRISPR 2,3,4, os editores de base 5,6 e os editores principais 7,8 podem alterar a sequência de DNA dos genomas de mamíferos por meio do corte de DNA e reparo dessas quebras. Embora eficazes, essas estratégias podem causar quebras de DNA em locais fora do alvo 9,10 e mutações estruturais genômicas em larga escala11,12,13,14,15. Ferramentas alternativas baseadas em CRISPR permitem a modulação tratável da ativação e repressão gênica sem alterar a sequência de DNA subjacente. Essas ferramentas aproveitam um Cas9 deficiente em nuclease (dCas9), permitindo a ligação ao DNA em locais-alvo ditados pela sequência de sgRNA, em combinação com proteínas efetoras que alteram a paisagem da cromatina16 , 17 , 18 . Proteínas efetoras, como escritores, leitores e borrachas epigenéticas, podem ser diretamente fundidas a dCas9 ou recrutadas por um andaime peptídico fundido a dCas9, como SunTag, ou um andaime de RNA no sgRNA, como o sistema MS2-MCP16 , 17 , 18 . Exemplos de ferramentas de controle de transcrição programáveis incluem ativação CRISPR (CRISPRa)19,20,21 e interferência CRISPR (CRISPRi)22,23. O CRISPRa funciona recrutando diretamente a maquinaria de transcrição, aumentando a expressão gênica transcricional alvo19. Em contraste, o CRISPRi reprime a transcrição estabelecendo H3K9me3, uma marca epigenética repressiva22.
Os avanços na edição do epigenoma permitiram o amplo uso dessas ferramentas em campos científicos. Fusões de diferentes domínios efetores e proteínas expandiram o kit de ferramentas de editores de epigenoma disponíveis 18,24,25,26,27,28,29,30. Além disso, os editores de epigenoma são usados para decifrar os papéis das modificações epigenéticas 31,32,33,34,35,36, efetores 37,38,39 e mutações efetoras 28,40,41 na regulação gênica. Especificamente, CRISPRi e CRISPRa são usados em telas genômicas funcionais para uma variedade de processos biológicos, incluindo sobrevivência celular42 e destino celular 43,44,45,46,47. Além disso, a edição do epigenoma possui potencial terapêutico para engenharia celular ex vivo e terapias in vivo 18.
Aqui, descrevemos métodos para aplicar dois editores de epigenoma baseados em dCas9 para repressão transcricional programável em linhagens celulares humanas: CRISPRi22 e CRISPRoff48. CRISPRi é uma fusão de dCas9 para o domínio repressivo KRAB de uma proteína de dedo de zinco, como ZNF10 (KOX1) e ZIM322,23. Quando o CRISPRi é direcionado a um promotor de gene específico, o domínio KRAB recruta um escritor H3K9me3, SETDB1, para reprimir o gene alvo (Figura 1A). Quando o CRISPRi é expresso transitoriamente, o H3K9me3 estabelecido no locus alvo não é mantido e a expressão gênica é restaurada ao longo do tempo32,48. Para conduzir knockdowns estáveis usando CRISPRi, como em aplicações de genômica funcional, a expressão constitutiva de CRISPRi nas células junto com o sgRNA é essencial. Recentemente, o CRISPRoff foi projetado para programar a edição hereditária do epigenoma48. CRISPRoff é uma fusão de proteína única de dCas9 para o domínio KRAB e complexo metiltransferase de DNA de novo, DNMT3A e DNMT3L. Um pulso transitório de CRISPRoff em células humanas programa a deposição de H3K9me3 e metilação do DNA nos genes-alvo, o que leva à repressão de longo prazo dos genes-alvo pela manutenção da metilação do DNA e H3K9me3 (Figura 1B) 48 . Além disso, as edições do epigenoma podem ser revertidas. Por exemplo, um gene que é silenciado de forma estável por CRISPRoff pode ser reativado por TET1-dCas9, que pode remover enzimaticamente as marcas de metilação do DNA nos loci alvo49.
Este protocolo detalhará dois métodos de entrega para a expressão transitória de editores de epigenoma: transfecção de DNA de plasmídeo e nucleofecção de mRNA. Além disso, descrevemos como usar a citometria de fluxo para avaliar a eficácia da edição do epigenoma em dois genes endógenos, CLTA e CD55. Esses métodos podem ser adaptados e aplicados a outros experimentos de edição de epigenoma usando editores adicionais ou podem ser usados para direcionar diferentes genes.
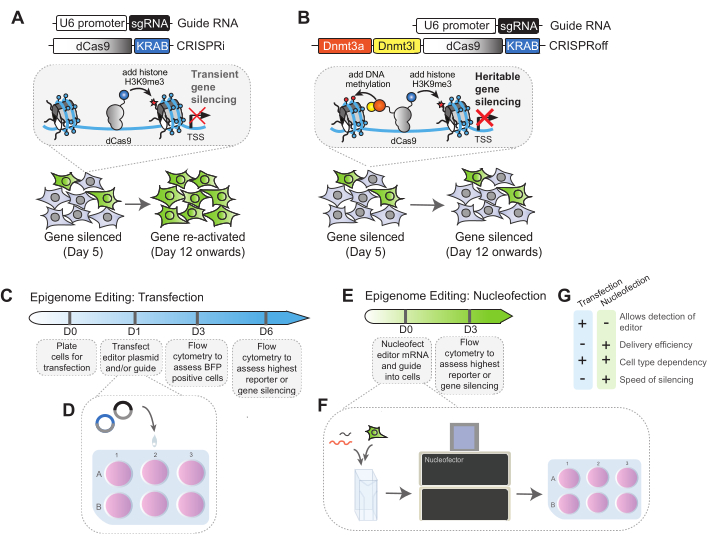
Figura 1: Esquema do mecanismo de edição do epigenoma CRISPRi e CRISPRoff e fluxo de trabalho. (A) Esquemas lineares do transgene sgRNA e editor de epigenoma CRISPRi. A adição de CRISPRi e sgRNA permite a adição da marca de histona repressiva H3K9me3 para silenciar o locus alvo. O nível mais alto de silenciamento é alcançado logo após a adição de CRISPRi, e o alvo do gene geralmente é reativado após algumas passagens. (B) Esquemas lineares do transgene sgRNA e editor de epigenoma CRISPRoff. A adição de CRISPRoff a células direcionadas a um gene de interesse leva à adição de H3K9me3 repressivo junto com a metilação do DNA em locais CpG para silenciar o gene alvo. O silenciamento por CRISPRoff é hereditário - o alto nível de silenciamento é alcançado no início da transfecção e persiste em várias divisões celulares. (C) Visão geral da linha do tempo para edição do epigenoma via método de transfecção. No dia 0, as células são plaqueadas para transfecção. No dia 1, o plasmídeo editor e guia pode ser introduzido nas células por meio de transfecção. No dia 3, as células serão avaliadas quanto à expressão de BFP por citometria de fluxo. A porcentagem de BFP é usada como um fator de normalização para determinar a eficácia final de silenciamento do experimento para cada condição. No dia 6 em diante, as células são analisadas para silenciar o repórter de interesse, pois o nível mais alto de silenciamento será alcançado neste dia. (D) Visão geral do método de transfecção em que o editor de epigenoma e os plasmídeos sgRNA são adicionados às células de maneira gota a gota. (E) Visão geral da linha do tempo para edição do epigenoma por meio dos métodos de nucleofecção. Neste protocolo, o mRNA é nucleofecção nas células no dia 0. As células são avaliadas quanto ao silenciamento no dia 3 pós-nucleofecção usando análise de citometria de fluxo. (F) Visão geral do protocolo de nucleofecção. Quantidades apropriadas de células e mRNA são misturadas e adicionadas em cubetas nucleofectoras. Se o sgRNA for introduzido por nucleofecção, ele também pode ser adicionado a essa mistura. As cubetas são colocadas no nucleofector e os códigos de pulso apropriados são usados para introduzir o mRNA nas células. Após a nucleofecção, as células são plaqueadas e passadas para análise em dias posteriores. (G) Comparação entre transfecção de plasmídeo e estratégias de nucleofecção de mRNA para edição de epigenoma. Clique aqui para ver uma versão maior desta figura.
Protocolo
NOTA: O Arquivo Suplementar 1 contém detalhes sobre o design do sgRNA, clonagem e geração de linha celular de nossos dados representativos. A seção de resultados representativos também detalha sugestões de controles.
1. Transfecção de plasmídeos que expressam o editor de epigenoma em células HEK293T
NOTA: Este protocolo descreve a entrega de plasmídeos codificadores de CRISPR em células HEK293T. Projetamos células para expressar um sgRNA (Addgene 217306) direcionado à região promotora de CLTA, um gene não essencial, que é marcado endogenamente com GFP. As células CLTA-GFP HEK 293T originaram-se de um estudo anterior50. Neste exemplo, os editores de epigenoma são fundidos diretamente a uma proteína fluorescente azul (BFP), o que nos permite quantificar a eficiência da transfecção e garante uma avaliação precisa das condições experimentais. A eficácia da abordagem é demonstrada pelo silenciamento do CLTA-GFP, que pode ser medido quantitativamente no nível da proteína em células individuais usando citometria de fluxo.
- Cultura de células
- Mantenha HEK293T células em DMEM com alto teor de glicose com 10% de FBS e 1x Penicilina-Estreptomicina-Glutamina. Passe as células a cada 2-3 dias, garantindo que permaneçam em uma confluência de 60% a 70%.
NOTA: Para garantir a eficiência ideal da transfecção, é fundamental manter a confluência celular abaixo de 70%, o que pode ser monitorado diariamente ao microscópio.
- Mantenha HEK293T células em DMEM com alto teor de glicose com 10% de FBS e 1x Penicilina-Estreptomicina-Glutamina. Passe as células a cada 2-3 dias, garantindo que permaneçam em uma confluência de 60% a 70%.
- Dia 0: Semeando células HEK293T para transfecção
- No dia anterior à transfecção, conte as células com um contador de células automatizado usando coloração de azul tripano vivo/morto e semeie ~ 1,5 x 104 células vivas por poço em uma placa de 96 poços. Mantenha o volume final de cada poço em 200 μL. As células devem atingir 60%-70% de confluência no dia seguinte.
NOTA: Isso também pode ser realizado em placas de 24 ou 6 poços de forma eficiente. Consulte a Tabela 1 para obter o número de células e a quantidade de DNA para transfecção.
- No dia anterior à transfecção, conte as células com um contador de células automatizado usando coloração de azul tripano vivo/morto e semeie ~ 1,5 x 104 células vivas por poço em uma placa de 96 poços. Mantenha o volume final de cada poço em 200 μL. As células devem atingir 60%-70% de confluência no dia seguinte.
- Dia 1: Transfecção de plasmídeos CRISPRoff e CRISPRi em células HEK29T
- Alíquota de 150 ng do plasmídeo editor de epigenoma (Addgene 167981) para cada poço em volume total de 3 μL. Essas diluições de plasmídeo podem ser preparadas em tubos de tiras de PCR.
NOTA: Se o editor de epigenoma e o plasmídeo sgRNA estiverem sendo co-transfectados, recomendamos tentar uma proporção molar de 3:4 de plasmídeos editor:sgRNA. A proporção pode precisar ser otimizada para produzir a mais alta eficiência da edição do epigenoma. - Pré-aqueça o reagente de transfecção e o meio essencial mínimo à temperatura ambiente cerca de 30-60 minutos antes da transfecção. Misture 0,6 μL de reagente de transfecção e 20 μL de meio essencial mínimo por poço e incube em temperatura ambiente por 15 min.
NOTA: Uma mistura mestre de reagente de transfecção e meio essencial mínimo pode ser feita se realizar várias transfecções. - Adicione 20,6 μL do reagente de transfecção e mistura mínima de meio essencial a cada tubo de tira de PCR contendo 3 μL de DNA de plasmídeo. Deixe descansar em temperatura ambiente por 15 min.
- Adicione a mistura lentamente ao prato de células semeadas e bata suavemente no prato para dispersar a mistura. Inclua um controle não transfectado para fornecer uma linha de base adequada para comparação em análises posteriores.
NOTA: Ao adicionar a mistura de transfecção às células semeadas, é essencial fazê-lo gota a gota e suavemente para minimizar a perturbação, pois isso pode afetar adversamente a eficiência da transfecção.
- Alíquota de 150 ng do plasmídeo editor de epigenoma (Addgene 167981) para cada poço em volume total de 3 μL. Essas diluições de plasmídeo podem ser preparadas em tubos de tiras de PCR.
- Dia 2: Verifique o sucesso da transfecção
- Examine as células ao microscópio com ampliação de 10X para determinar se elas são positivas para BFP, pois isso indica uma transfecção bem-sucedida. Certifique-se de que as células não sejam perturbadas durante o processo de observação.
- Dia 3: Divida a placa de 96 poços e avalie a eficiência da transfecção usando o marcador BFP
NOTA: Uma transfecção bem-sucedida deve produzir pelo menos 30% de células como BFP positivas. No entanto, com dCas9, CRISPRi e CRISPRoff, normalmente detectamos mais de 60% de células positivas para BFP dois dias após a transfecção. A eficiência da transfecção está negativamente correlacionada com o tamanho do plasmídeo, portanto, diferentes editores de epigenoma podem produzir diferentes eficiências de transfecção.- Pré-aqueça tripsina e DMEM com 10% de FBS e 1x Penicilina-Estreptomicina-Glutamina à temperatura ambiente por cerca de 30-60 min antes do uso.
- Aspirar meios dos poços; Tenha cuidado para não remover as células presas à parte inferior. Adicione 50 μL de tripsina por poço. Incube por 2-3 min a 37 °C.
NOTA: Não é incomum observar algumas células flutuantes, especialmente após a transfecção com CRISPRoff. Nossa hipótese é que essa morte celular é devido à toxicidade da superexpressão de DNMT3A-3L. - Inibir a tripsina adicionando 150 μL de DMEM completo por poço. Pipete para cima e para baixo para ressuspender as células e dividir as células para confluência no dia 5 (~ 20 μL de mistura de células em 200 μL de meio de uma placa de 96 poços).
- Use 50 μL de células ressuspensas para citometria de fluxo para avaliar a eficiência da transfecção.
2. Nucleofecção do mRNA do editor de epigenoma em células K-562
NOTA: Esta seção detalha o processo de nucleofecção do mRNA CRISPRoff em células K-562. Para simplificar, pré-projetamos as células K-562 para expressar constitutivamente um sgRNA que tem como alvo o promotor do gene endógeno CD55 (Addgene 217306). A entrega de mRNA CRISPRoff diretamente às células tem o potencial de diminuir a toxicidade celular que acompanha as abordagens baseadas em DNA de plasmídeo, ao mesmo tempo em que alcança eficácia semelhante de silenciamento de genes. Além disso, a nucleofecção pode ser usada para introduzir construções de editores de epigenoma em linhagens celulares que são difíceis de transfectar com eficiência, como K-562s.
- Cultura de células
- Antes da nucleofecção, mantenha as células K-562 em um frasco com RPMI com 10% de FBS e 1x Penicilina-Estreptomicina-Glutamina. Passe as células todos os dias, garantindo que elas permaneçam em uma confluência de 60% a 70%.
- No dia da nucleofecção, descongele e vore suavemente o mRNA CRISPRoff em um tubo de microcentrífuga estéril sem RNase e armazene o mRNA no gelo. Use 2 μg de mRNA por 2,0 x 105 células em cada poço de uma tira nucleocuvette. Certifique-se de que o volume de mRNA não exceda 10% do volume total de nucleofecção (22 μL).
NOTA: A nucleofecção também pode ser realizada em cubetas maiores com mais células e mRNA. Sugerimos dimensionar a quantidade de mRNA proporcionalmente ao número de células que você adicionará. Por exemplo, ao realizar uma nucleofecção em uma cubeta grande em 1 x 106 células, sugerimos o uso de 10 μg de mRNA, com um volume total de nucleofecção de 100 μL. Além disso, a dosagem do mRNA pode ser otimizada para garantir alta eficácia de edição. Costumamos usar 2-5 μg de mRNA editor para cada 2,0 x 105 células para resultados eficientes. Os nucleotídeos livres que não foram incorporados durante a síntese de mRNA devem ser removidos antes da quantificação. - Prepare a solução de nucleofecção de acordo com as instruções do fabricante e aqueça à temperatura ambiente por 15 minutos antes da nucleofecção.
NOTA: A solução do nucleofector pode ser diferente ou precisa ser otimizada para diferentes tipos de células. - Colha e conte células K-562 usando um contador de células automatizado com coloração viva/morta de azul tripano. Para nucleofecção em uma cubeta de tira, alíquota ~ 2,0 x 105 células por amostra em um tubo de microcentrífuga estéril.
- Centrifugue as células a 500 x g durante 5 min à temperatura ambiente e, em seguida, elimine o sobrenadante. Lave as células 1x com PBS em temperatura ambiente a 500 x g por 5 min e descarte o sobrenadante.
- Calcule a quantidade de solução de nucleofector para ressuspender as células subtraindo o volume de mRNA de 22 μL. Ressuspenda as células na quantidade apropriada de solução de nucleofector
- Adicione a solução celular a 2 μg de mRNA CRISPRoff (volume determinado na etapa 2.1.2) e transfira a solução para uma cubeta, tomando cuidado para não formar bolhas, pois isso pode comprometer a eficiência da nucleofecção. Bata suavemente na nucleocubeta para garantir que as células estejam na parte inferior.
- Nucleofecte as células usando o sistema 4D-Nucleofector com o código de pulso apropriado. O código FF-120 é recomendado para células K-562.
NOTA: O código de pulso pode precisar ser otimizado para diferentes tipos de células. Consulte o fabricante se for necessária uma otimização adicional. - Após a nucleofecção, adicione 80 μL de meio RPMI a cada pequeno poço de nucleocubeta e deixe as células assentarem a 37 °C por 15 min.
- Transfira a suspensão celular para um poço de uma placa de 24 poços contendo 400 μL de meio RPMI pré-aquecido (pequena nucleocuvette).
NOTA: Após a nucleofecção, as células são particularmente frágeis e devem ser transferidas da nucleocuvette para os poços de cultura suavemente. - Após o dia 2 pós-nucleofecção: siga o protocolo de coloração do marcador de superfície com o anticorpo CD55 conforme descrito na seção 3 e meça a fluorescência do CD55 APC em um citômetro de fluxo para testar a eficácia da edição do epigenoma.
NOTA: Para testar o sucesso da nucleofecção, recomendamos realizar uma nucleofecção com GFP ou mCherry mRNA. Dois dias após a nucleofecção, realizar citometria de fluxo dessas células para confirmar o sucesso da nucleofecção. Geralmente detectamos 90% a 100% dessas células expressando GFP ou mCherry. Uma alternativa é realizar uma nucleofecção com mRNA CRISPRi, que é conhecido por silenciar de forma robusta muitos genes-alvo. Ao realizar citometria de fluxo no dia 2, as amostras tratadas com CRISPRi devem mostrar silenciamento do gene alvo em mais de 90% das células.
3. Coloração do marcador de superfície
NOTA: Esta seção detalha a quantificação dos níveis da proteína CD55 após a edição do epigenoma em células K-562. Quantificamos a diminuição da expressão de CD55 em células únicas usando coloração de anticorpos e citometria de fluxo (consulte a seção 4 abaixo) para avaliar a eficiência de knockdown mediada por CRISPRoff. Técnicas adicionais, incluindo PCR quantitativo de transcrição reversa ou western blotting, também podem ser utilizadas para confirmar o nível de knockdown nos níveis de transcrição e proteína.
- Conte as células usando um contador de células automatizado e adicione cerca de 5,0 x 105 a 1,0 x 106 células em um tubo de microcentrífuga e centrifugue as células a 500 x g por 5 min.
NOTA: Mais ou menos células podem ser usadas para coloração, no entanto, a concentração de anticorpos pode precisar ser dimensionada de acordo. - Rejeitar o sobrenadante, certificando-se de não perturbar o sedimento, adicionar 500 μL de PBS e ressuspender o sedimento suavemente.
- Centrifugue as células novamente a 500 x g por 5 min. Rejeitar o sobrenadante e adicionar o anticorpo à concentração desejada diluída em PBS. Para a coloração de CD55, use 1 μL de CD55 anti-humano purificado (estoque de 0,5 mg / mL) em 49 μL de PBS.
- Pipete suavemente as células e deixe-as incubar por 25 minutos em um local escuro em temperatura ambiente. Após a incubação, gire as células novamente a 500 x g por 5 min e descarte o sobrenadante.
- Ressuspenda o pellet com 500 μL de PBS para lavar o excesso de anticorpos. Centrifugue as células a 500 x g durante 5 min e elimine o sobrenadante PBS. Ressuspenda em 100 μL a 200 μL de PBS fresco e adicione a uma placa de 96 poços para análise de citometria de fluxo.
4. Citometria de fluxo
NOTA: Este protocolo foi escrito para usar um BD FACSymphony A1 Cell Analyzer. As especificidades podem variar dependendo do citômetro de fluxo que você está usando. Sugerimos consultar o manual do usuário da máquina que você está usando para obter detalhes.
- Antes de ligar a citometria, verifique se o recipiente de resíduos não está cheio e se há bastante fluido da bainha. Ligue o interruptor de alimentação principal (verde) que está próximo ao fluido da bainha.
- Ligue o leitor de placas usando o interruptor traseiro. Em seguida, ligue a máquina usando o botão verde no lado direito e certifique-se de que a máquina esteja configurada para a função de leitor de placas de 96 poços.
- Ligue o computador e faça login. Abra o software e faça login usando seu nome de usuário e senha.
- Configure o experimento. Crie um novo experimento selecionando Experimento > Novo Experimento. Renomeie o experimento clicando duas vezes no nome atual.
- Adicione uma placa clicando em Experimentar > Nova placa. Selecione o formato de poço a ser usado. Para adicionar poços específicos para executar, clique na localização do poço na placa e, em seguida, clique na Seringa Azul (o poço agora deve ser destacado em azul). Selecione vários poços ao mesmo tempo para adicionar, se necessário.
- Adicione nomes específicos a cada poço clicando com o botão direito do mouse em Espécime à direita da exibição da placa. Em seguida, selecione Layout experimental. Isso abrirá uma página listando todos os poços que você selecionou. Para renomear os poços, clique uma vez e digite o nome desejado. Clique em OK quando terminar.
- Adicione gráficos e portões apropriados ao experimento na Planilha Global. Recomendamos primeiro plotar a área de dispersão direta (FSC-A) versus a área de dispersão lateral (SSC-A). Em seguida, crie uma porta arbitrária para células ativas refinarem posteriormente clicando na ferramenta Polígono.
- Clique com o botão direito do mouse neste portão e clique em Drill Down. Isso criará um novo gráfico exibindo apenas as células dessa porta de célula ativa.
- Altere o eixo deste novo gráfico para FSC-A vs altura de dispersão direta (FSC-H) e crie uma porta para células únicas.
- Se estiver expressando constitutivamente o sgRNA nas células, e o lentivírus também codificar uma proteína fluorescente, então perfure essa única porta celular e execute um gating adicional para a expressão dessa proteína como um proxy para a expressão de sgRNA (exemplo: PE-CF594-A vs FSC-A).
- Na porta de configuração final (para células únicas ou para células que expressam sgRNA), clique com o botão direito do mouse e selecione Detalhar. Crie uma porta para testar a expressão do gene repórter (exemplo: BB515-A vs FSC-A).
- Se estiver usando células transfectadas e tiver um método para testar a eficiência da transfecção, como a expressão BFP, clique com o botão direito do mouse no gráfico de expressão do repórter e clique em Duplicar. Altere os eixos para BV421-A vs FSC-A. Crie uma porta arbitrária para células BFP-positivas.
- Remova todos os parâmetros que não estão em uso na guia Parâmetros do visor do citômetro.
- Para configurar a máquina e prepará-la para funcionar, clique em Executar no painel de controle do citômetro de fluxo. O botão deve aparecer em verde. No software, vá para HTS no painel superior e, em seguida, Prime. Quando terminar, adicione o prato, certificando-se de que esteja bem posicionado na máquina e que a tampa esteja DESLIGADA.
- Para executar, selecione todos os poços a serem executados e verifique se os controles estão definidos adequadamente. Selecione o poço clicando duas vezes, altere o volume da amostra para 10 μL e clique em Adquirir dados no Painel de aquisição. Isso executará o exemplo, mas não registrará os dados. Enquanto esta amostra está em execução, ajuste as tensões do laser conforme necessário. Além disso, troque os portões.
NOTA: Recomendamos executar uma amostra que seja negativa para todas as cores que estão sendo medidas e uma que seja positiva para todas as cores para garantir que os parâmetros do laser e o gating estejam bons. - Depois de verificar as configurações do laser, clique em Amostras no Painel de Placas e verifique se os valores de execução estão definidos conforme desejado.
- Clique em Executar placa para executar no Painel de aquisição e registrar dados de todos os poços. Quando terminar, limpe a máquina removendo a placa.
- Para uma nova placa de 96 poços, adicione 10% de alvejante nas quatro fileiras superiores e H2O nos quatro poços abaixo. Vá para HTS > Clean. Uma vez terminado, feche o programa e desligue os botões na ordem inversa em que foram ligados botão lateral verde > leitor de placas > interruptor verde por desperdício.
- Adicione 20% de água sanitária à placa de 96 poços com células e descarte.
5. Análise dos dados
NOTA: Este método descreve estratégias de gating e processamento de dados para quantificar a edição do epigenoma por citometria de fluxo. A estratégia de gating é representada visualmente ao lado de gráficos de exemplo gerados a partir da análise de dados na Figura 2 e na Figura 3.
- Configuração de bloqueio FlowJo
- Carregue todos os arquivos FSC no FlowJo arrastando e soltando-os em uma nova planilha.
- Clique em Todas as amostras para começar a fazer portões. Isso garantirá que os portões possam ser facilmente aplicados a todas as amostras.
- Abra uma amostra de controle (como não transfectada ou não nucleofectada
- Crie uma porta para células vivas em um gráfico FSC-A vs SSC-A usando a ferramenta de polígono. Na lista de todos os exemplos, uma guia Células Vivas agora deve aparecer abaixo do nome do exemplo. Clique com o botão direito do mouse nessa porta e selecione Copiar análise para o grupo para aplicar essa porta a todas as amostras (Figura 2A, Figura 3A).
- Clique duas vezes no portão Células vivas para detalhar as células ativas. Altere os eixos para FSC-A vs FSC-H (Figura 2B, Figura 3B).
- Desenhe um polígono para portar apenas células únicas. Aplique esta porta a todas as amostras conforme feito na seção 4 (Figura 2B, Figura 3B).
NOTA: Se expressar constitutivamente uma construção de sgRNA com um repórter fluorescente, execute mais gating nessa cor para que a análise se concentre apenas nas células que estão expressando o sgRNA. Um exemplo disso seria a criação de uma porta para células positivas para mCherry (PE-CF594-A vs FSC-A) se as células contiverem um sgRNA do backbone pLG1 ( Figura 2C , Figura 3C ). - Clique duas vezes no Last Setup Gate (células únicas ou células que expressam guia).
- Se estiver realizando uma transfecção de plasmídeo e o editor de epigenoma codificar para uma fusão BFP, crie uma porta para células BFP positivas (exemplo: BV421-A vs FSC-A) para as amostras do Dia 2 (Figura 2D).
- Crie uma porta para a expressão do repórter (exemplo: BB515-A vs FSC-A para porta para células GFP negativas) e aplique essa porta a todas as amostras (Figura 2E, Figura 3D).
- Análise de transfecção
- Depois de realizar a configuração do gating, clique em Editor de tabelas no painel superior. Arraste populações para análise (exemplos: BFP positivo, GFP negativo).
- No painel do editor de tabelas, clique em Criar tabela. Copie esses dados em uma planilha para executar cálculos de normalização.
- Processamento de dados
- Subtraia o número de células repórter negativas (exemplo: GFP negativo) na amostra de controle de todas as outras amostras. Pode haver algum silenciamento de fundo do repórter, portanto, isso remove esse ruído de fundo dos dados.
- Todos os valores serão normalizados para a porcentagem de células positivas para BFP (células transfectadas com sucesso) medidas no dia 2. Para fazer essa normalização, pegue o valor, divida pela porcentagem de células positivas para BFP no dia 2 e multiplique por 100. Esse cálculo produz o valor normalizado da eficiência de transfecção das células editadas. Os dados agora estão prontos para serem plotados. Recomendamos gráficos de linha em um curso de tempo de edição de epigenoma, conforme ilustrado na Figura 2F.
- Análise de nucleofecção
- Depois de realizar a configuração do gating, clique em Editor de tabelas no painel superior. Arraste populações para análise (exemplos: APC negativo).
- No painel do editor de tabelas, clique em Criar tabela. Copie esses dados em um editor de planilhas para realizar cálculos de normalização.
- Processamento de dados
- Subtraia o número de células repórter negativas (exemplo: APC negativo) na amostra de controle de todas as outras amostras. Pode haver algum silenciamento de fundo do repórter, portanto, isso remove esse ruído de fundo dos dados. Os dados agora estão prontos para serem plotados. Recomendamos gráficos de linhas em um curso de tempo de edição de epigenoma.
NOTA: As estatísticas podem ser usadas para quantificar as diferenças no silenciamento entre dois editores de epigenoma diferentes. Recomendamos a realização de todos os experimentos de transfecção e nucleofecção em triplicatas técnicas para realizar análises estatísticas. Ao representar gráficos de linha de silenciamento ao longo do tempo, plote o desvio padrão das réplicas técnicas. Testes t de Student em pontos de tempo específicos podem ser realizados para comparar o silenciamento entre dois editores de epigenoma. As amostras devem ser tratadas como não pareadas com variância desigual.
- Subtraia o número de células repórter negativas (exemplo: APC negativo) na amostra de controle de todas as outras amostras. Pode haver algum silenciamento de fundo do repórter, portanto, isso remove esse ruído de fundo dos dados. Os dados agora estão prontos para serem plotados. Recomendamos gráficos de linhas em um curso de tempo de edição de epigenoma.
Resultados
Para todos os experimentos de edição de epigenoma, controles adequados são essenciais para avaliar as eficiências de edição de epigenoma. Recomendamos o uso de um sgRNA de controle, que não tem como alvo nenhuma sequência no genoma humano. O uso de um controle de guia sem direcionamento dará confiança de que as alterações nos loci de destino são conduzidas pelo editor de epigenoma sendo direcionado para esse local, e não apenas pela superexpressão do editor de epigenoma ou ligação não específica. Além disso, para experimentos baseados em genes repórteres, sugerimos o uso de um controle somente dCas9 para garantir que as mudanças na expressão do repórter sejam devidas a fusões do editor de epigenoma, em vez de impedimento estérico da ligação do dCas9 ao locus alvo e impedindo temporariamente a transcrição ( Figura 2F ).
Para experimentos de transfecção, recomendamos o uso de um editor de epigenoma com uma fusão de proteína fluorescente adicional, como BFP. Essa fusão permite a visualização de células transfectadas com sucesso com o editor de epigenoma por meio de microscopia e citometria de fluxo. As células transfectadas com sucesso expressarão altos níveis de BFP dois dias após a transfecção (Figura 2D). A quantificação de células transduzidas com sucesso é usada para normalizar a eficácia da edição do epigenoma em dias posteriores (Figura 2F).
Tanto o CRISPRoff quanto o CRISPRi mostram o pico de silenciamento no dia 5 após a transfecção (Figura 2E-F). Diferentes editores de epigenoma têm cronogramas distintos de edição de epigenoma, como silenciamento hereditário com CRISPRoff e silenciamento transitório com CRISPRi (Figura 2F). A Figura 2F também exibe o uso de dCas9 apenas como um controle importante para experimentos de edição de epigenoma. Em experimentos de nucleofecção de mRNA, as células editadas com sucesso com CRISPRoff mostrarão forte silenciamento do gene alvo no dia 3 pós-nucleofecção (Figura 3E-F).

Figura 2: Estratégia de bloqueio e dados representativos para entrega do editor de epigenoma por transfecção de plasmídeo. (AC) Gráficos de fluxo representativos para exibir a estratégia de gating para experimentos de transfecção de plasmídeo. Os gráficos de fluxo exibidos são de células não transfectadas 2 dias após a transfecção. Cada ponto nos gráficos representa uma célula. (A) Gráfico de fluxo da área de dispersão direta (FSC-A) e área de dispersão lateral (SSC-A) com uma porta para células vivas. (B) Gráfico de fluxo de células vivas exibindo FSC-A e altura de dispersão direta (FSC-H) com uma porta para células individuais. (C) Gráfico de fluxo de células individuais representando PE-CF594-A (expressão mCherry) e FSC-A. Gating para células positivas para mCherry como um proxy para a expressão de sgRNA. (D) Estratégia de gating representativa para expressão do editor de epigenoma (BFP+) no dia 2 pós-transfecção. A população parental é composta por células que expressam guia fechadas em (C). (E) Estratégia de bloqueio representativa para o silenciamento do gene repórter CLTA-GFP. A população parental é composta por células que expressam guia no painel C. (F) Silenciamento de CLTA-GFP ao longo de dias após a transfecção após a entrega de plasmídeo de dCas9, CRISPRi e CRISPRoff. A porcentagem de CLTA-GFP silenciada é normalizada para a eficiência de transfecção medida como células positivas para BFP 2 dias após a transfecção. Os pontos são médias de quatro réplicas de transfecção. As barras de erro representam o desvio padrão. Clique aqui para ver uma versão maior desta figura.
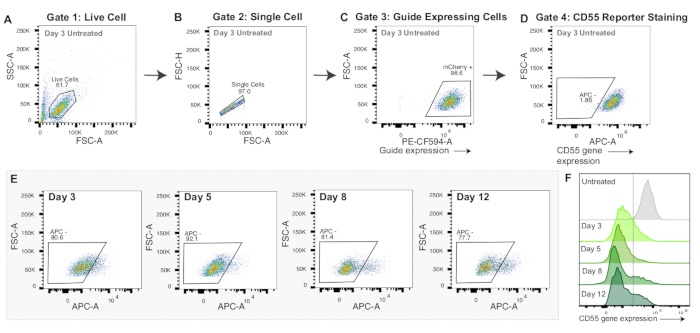
Figura 3: Estratégia de bloqueio e dados representativos para nucleofecção de mRNA CRISPRoff. (A-D) Gráficos de fluxo representativos para exibir a estratégia de gating para experimentos de nucleofecção de mRNA. Os gráficos de fluxo exibidos são de células de controle 3 dias após a nucleofecção coradas com o anticorpo anti-CD55 humano APC. Cada ponto nos gráficos representa uma célula. (A) Gráfico de fluxo de intensidade da área de dispersão direta (FSC-A) e intensidade da área de dispersão lateral (SSC-A) com uma porta para células vivas. (B) Gráfico de fluxo de células vivas exibindo FSC-A e intensidade de altura de dispersão direta (FSC-H) com uma porta para células individuais. (C) Gráfico de fluxo de células individuais representando PE-CF594-A (expressão mCherry) e FSC-A. Gating para células positivas para mCherry como um proxy para a expressão de sgRNA. (D) Gráfico de fluxo de APC-A versus FSC-A. Portão desenhado para células negativas para APC, o que indicaria silenciamento do gene repórter CD55. (E) Gráficos de fluxo de silenciamento de CD55 (APC - gate) ao longo de 3, 5, 8 e 12 dias após a nucleofecção com mRNA CRISPRoff. (F) Histogramas sobrepostos da expressão da proteína CD55 (APC-A) ao longo de 3, 5, 8 e 12 dias após a nucleofecção com mRNA CRISPRoff em comparação com um controle corado, mas não nucleofectado. Clique aqui para ver uma versão maior desta figura.
| Prato | Densidade de semeadura (células por poço) | Quantidade de plasmídeo |
| com 96 poços | 15,000 | 150 ng |
| com 24 poços | 90,000 | 500 ng |
| com 6 poços | 400,000 | 2 μg |
Tabela 1: Quantidades de escala de transfecção. Densidades de semeadura e quantidades de DNA de plasmídeo para transfecção do editor de epigenoma em células HEK293T em diferentes escalas.
Tabela suplementar 1: sequências espaçadoras de sgRNA para experimentos de edição de epigenoma. sequências de sgRNA para direcionar editores de epigenoma para CLTA e CD55, juntamente com uma sequência para um guia de controle sem direcionamento. Além disso, oligos para clonagem no backbone pLG1 são listados. Clique aqui para baixar este arquivo.
Arquivo suplementar 1. Clique aqui para baixar este arquivo.
Discussão
Este protocolo detalha dois métodos diferentes de entrega transitória para editores de epigenoma CRISPR: transfecção de DNA de plasmídeo e nucleofecção de mRNA. Ambas as técnicas têm vantagens, desvantagens e considerações gerais únicas (Figura 1F).
A transfecção de DNA de plasmídeo leva à expressão robusta do editor de epigenoma, e incluímos uma fusão BFP nas construções do editor de epigenoma que permite a detecção e quantificação da eficácia da transfecção usando citometria de fluxo. Além disso, a expressão de BFP pode ser usada para classificar células que expressam o editor de epigenoma ou pode ser quantificada para normalizar os dados de silenciamento em pontos de tempo posteriores, conforme detalhado neste protocolo. No entanto, é importante notar que a eficiência da transfecção geralmente não é de 100% e, portanto, a população será heterogênea para aqueles que receberam o editor e aqueles que não receberam, a menos que as células sejam classificadas. Além disso, a entrega de DNA de plasmídeo pode desencadear uma resposta imune do DNA citoplasmático de fita dupla, levando à ativação da via imune e citotoxicidade. Finalmente, a transfecção de DNA do plasmídeo não é passível de todos os tipos de células, e a nucleofecção do plasmídeo pode ser necessária.
Ao contrário da transfecção de DNA de plasmídeo, a nucleofecção de mRNA é aplicável para muitos tipos de células e geralmente resulta em alta eficácia de entrega. Os mRNAs do editor podem ser sintetizados usando kits de síntese de mRNA in vitro disponíveis comercialmente, um dos quais foi detalhado anteriormente51. Alternativamente, os mRNAs podem ser sintetizados a partir de Aldevron ou Trilink. No entanto, uma ressalva da nucleofecção de mRNA é que ela não produz proteína editora suficiente para testar quais células receberam os editores de epigenoma. Os experimentos acima sugerem que quase todas as células receberam o mRNA quando detectamos ~ 90% do silenciamento do gene alvo no dia 5 (Figura 3F). No entanto, recomendamos otimizar os códigos de pulso e a proporção de mRNA para célula para cada tipo de célula.
Outra consideração é que a edição do epigenoma iniciada por transfecção de DNA de plasmídeo ou nucleofecção de mRNA tem diferentes cronogramas de silenciamento. Com a transfecção de DNA de plasmídeo, o pico de silenciamento com CRISPRoff e CRISPRi é observado no dia 5 pós-transfecção (Figura 2F). Em comparação, o silenciamento máximo com CRISPRoff e CRISPRi entregue pela nucleofecção de mRNA é visto já no dia 3 após a nucleofecção (Figura 3F). Além disso, o DNA do plasmídeo persiste nas células em comparação com o mRNA, resultando em uma duração mais longa da expressão do editor. Dependendo dos objetivos experimentais, uma linha do tempo de silenciamento mais longa ou mais curta e a duração da expressão podem ser preferidas.
Também é importante notar que a edição do epigenoma não pode ser aplicada universalmente a todos os genes ou tipos de células. Diferenças inerentes na sequência do genoma ou nos estados de cromatina dos genes-alvo podem afetar a eficácia do editor, mesmo para o mesmo gene em diferentes tipos de células. Por exemplo, genes que não possuem ilhas CpG anotadas podem ser difíceis de silenciar de forma estável pela metilação do DNA mediada por CRISPRoff48. Portanto, pode ser necessário testar diferentes editores de epigenoma para silenciar esses genes. Além disso, também recomendamos testar pelo menos os três principais gRNAs CRISPRi para identificar o melhor guia para maior eficiência de edição de epigenoma42. No entanto, dada a nossa caixa de ferramentas limitada de editores robustos de epigenoma, pode ser mais eficaz conduzir o nocaute genético ou usar outras estratégias de nocaute, dependendo do gene de interesse.
Este protocolo é focado em CRISPRi e CRISPRoff, dois dos muitos editores de epigenoma baseados em CRISPR disponíveis. Estudos recentes de descoberta em larga escala desenvolveram novas ferramentas para reescrever o epigenoma humano 29,30,52. Os editores de epigenoma têm aplicações em pesquisa biomédica e terapêutica. Por exemplo, estudos recentes usaram metilação de DNA e edição de epigenoma baseada em H3K9me3 em modelos de camundongos e primatas não humanos, resultando em repressão hereditária do gene associado à doença 53,54,55. Prevemos que as futuras modalidades de entrega para editores de epigenoma abrirão novos caminhos para a ampla aplicação da edição de epigenoma.
Divulgações
J.K.N. é um inventor de patentes relacionadas às tecnologias CRISPRoff/on, registradas pelos Regentes da Universidade da Califórnia.
Agradecimentos
Agradecemos aos membros do laboratório Nuñez, especialmente Rithu Pattali e Izaiah Ornelas, por desenvolver e otimizar os protocolos descritos neste manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| 4D-Nucleofector | Lonza | AAF-1003 | |
| 96-well tissue culture plates | Corning | 3596 | |
| 96-well U-bottom Plate | Corning | 351177 | |
| APC anti-human CD55 Antibody | BioLegend | 311312 | |
| BD FACSymphony A1 Flow Cytometer | BD Biosciences | ||
| Bleach | Waxie | 11003428432 | |
| Centrifuge | Eppendorf | 5425 | |
| Countess Automated Cell Counter | Thermo Scientific | Countess 3 | |
| CRISPRoff transfection plasmid | Addgene | 167981 | |
| Diluent 2 Hematology Reagent for Flow Cytometry (Sheath fluid) | Thermo Scientific | 23-029-361 | |
| DMEM, High Glucose | Thermo Scientific | 11965118 | |
| DPBS | Gibco | 14-190-250 | |
| Eppendorf tubes | Thomas scientific | 1159M35 | |
| FBS | Avantor Seradigm | 89510-186 | |
| Lonza Walkersville SF Cell Line 4D-Nucleofector X Kit L | Fisher Scientific | NC0281111 | |
| mMESSAGE mMACHINE™ T7 ULTRA Transcription Kit | Thermo Fisher | AM1345 | |
| Opti-MEM | Gibco | 31985070 | |
| PCR strip tubes | USA Scientific | 1402-4700 | |
| Penicillin-Streptomycin-Glutamine | Gibco | 10378016 | |
| pLG1 sgRNA expression plasmid | Addgene | 217306 | |
| RPMI 1640 | Gibco | 22-400-105 | |
| SF Cell Line 96-well Nucleofector® Kit | Lonza | V4SC-2096 | |
| Tissue culture incubator | PHCbi | MCO-170AICUVDL-PA | |
| TransIT-LTI transfection reagent | Mirus | MIR 2306 | |
| Trypsin-EDTA (0.25%) | Gibco | 25200114 |
Referências
- Kimura, H. Histone modifications for human epigenome analysis. Journal of Human Genetics. 58 (7), 439-445 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Esvelt, K. M., Mali, P., Braff, J. L., Moosburner, M., Yaung, S. J., Church, G. M. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences. 110 (39), 15644-15649 (2013).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Gaudelli, N. M., et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Anzalone, A. V., et al. Programmable deletion, replacement, integration and inversion of large DNA sequences with twin prime editing. Nature Biotechnology. 40 (5), 731-740 (2022).
- Zhang, X. -H., Tee, L. Y., Wang, X. -G., Huang, Q. -S., Yang, S. -H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4, e264(2015).
- Lin, Y., et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Research. 42 (11), 7473-7485 (2014).
- Adikusuma, F., et al. Large deletions induced by Cas9 cleavage. Nature. 560 (7717), E8-E9 (2018).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36 (8), 765-771 (2018).
- Cullot, G., et al. CRISPR-Cas9 genome editing induces megabase-scale chromosomal truncations. Nature Communications. 10 (1), 1136(2019).
- Fiumara, M., et al. Genotoxic effects of base and prime editing in human hematopoietic stem cells. Nature Biotechnology. 42 (6), 877-891 (2024).
- Tsuchida, C. A., et al. Mitigation of chromosome loss in clinical CRISPR-Cas9-engineered T cells. Cell. 186 (21), 4567-4582.e20 (2023).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Villiger, L., Joung, J., Koblan, L., Weissman, J., Abudayyeh, O. O., Gootenberg, J. S. CRISPR technologies for genome, epigenome and transcriptome editing. Nature Reviews Molecular Cell Biology. 25 (6), 464-487 (2024).
- McCutcheon, S. R., Rohm, D., Iglesias, N., Gersbach, C. A. Epigenome editing technologies for discovery and medicine. Nature Biotechnology. 42 (8), 1199-1217 (2024).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Maeder, M. L., Linder, S. J., Cascio, V. M., Fu, Y., Ho, Q. H., Joung, J. K. CRISPR RNA-guided activation of endogenous human genes. Nature Methods. 10 (10), 977-979 (2013).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A Protein-Tagging System for Signal Amplification in Gene Expression and Fluorescence Imaging. Cell. 159 (3), 635-646 (2014).
- Gilbert, L. A., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Alerasool, N., Segal, D., Lee, H., Taipale, M. An efficient KRAB domain for CRISPRi applications in human cells. Nature Methods. 17 (11), 1093-1096 (2020).
- Nakamura, M., Gao, Y., Dominguez, A. A., Qi, L. S. CRISPR technologies for precise epigenome editing. Nature Cell Biology. 23 (1), 11-22 (2021).
- Tycko, J., et al. High-Throughput Discovery and Characterization of Human Transcriptional Effectors. Cell. 183 (7), 2020-2035.e16 (2020).
- Alerasool, N., Leng, H., Lin, Z. -Y., Gingras, A. -C., Taipale, M. Identification and functional characterization of transcriptional activators in human cells. Molecular Cell. 82 (3), 677-695.e7 (2022).
- Ludwig, C. H., et al. High-throughput discovery and characterization of viral transcriptional effectors in human cells. Cell Systems. 14 (6), 482-500.e8 (2023).
- DelRosso, N., et al. Large-scale mapping and mutagenesis of human transcriptional effector domains. Nature. 616 (7956), 365-372 (2023).
- Tycko, J., et al. Development of compact transcriptional effectors using high-throughput measurements in diverse contexts. Nature Biotechnology. , (2024).
- Wilson, C. M., et al. Combinatorial effector targeting (COMET) for transcriptional modulation and locus-specific biochemistry. , (2024).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247.e17 (2016).
- Bintu, L., et al. Dynamics of epigenetic regulation at the single-cell level. Science. 351 (6274), 720-724 (2016).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nature Biotechnology. 33 (5), 510-517 (2015).
- Cano-Rodriguez, D., et al. Writing of H3K4Me3 overcomes epigenetic silencing in a sustained but context-dependent manner. Nature Communications. 7 (1), 12284(2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Research. 45 (17), 9901-9916 (2017).
- O'Geen, H., et al. Ezh2-dCas9 and KRAB-dCas9 enable engineering of epigenetic memory in a context-dependent manner. Epigenetics & Chromatin. 12 (1), 26(2019).
- Kim, J. -M., et al. Cooperation between SMYD3 and PC4 drives a distinct transcriptional program in cancer cells. Nucleic Acids Research. 43 (18), 8868-8883 (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nature Methods. 12 (5), 401-403 (2015).
- Li, K., et al. Interrogation of enhancer function by enhancer-targeting CRISPR epigenetic editing. Nature Communications. 11 (1), 485(2020).
- Stepper, P., et al. Efficient targeted DNA methylation with chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase. Nucleic Acids Research. 45 (4), 1703-1713 (2017).
- Valbuena, R., et al. Prediction and design of transcriptional repressor domains with large-scale mutational scans and deep learning. , (2024).
- Horlbeck, M. A., et al. Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. eLife. 5, e19760(2016).
- Liu, Y., et al. CRISPR Activation Screens Systematically Identify Factors that Drive Neuronal Fate and Reprogramming. Cell Stem Cell. 23 (5), 758-771.e8 (2018).
- Black, J. B., et al. Master Regulators and Cofactors of Human Neuronal Cell Fate Specification Identified by CRISPR Gene Activation Screens. Cell Reports. 33 (9), 108460(2020).
- Yang, J., et al. Genome-Scale CRISPRa Screen Identifies Novel Factors for Cellular Reprogramming. Stem Cell Reports. 12 (4), 757-771 (2019).
- Chakraborty, S., Ji, H., Kabadi, A. M., Gersbach, C. A., Christoforou, N., Leong, K. W. A CRISPR/Cas9-Based System for Reprogramming Cell Lineage Specification. Stem Cell Reports. 3 (6), 940-947 (2014).
- Black, J. B., et al. Targeted Epigenetic Remodeling of Endogenous Loci by CRISPR/Cas9-Based Transcriptional Activators Directly Converts Fibroblasts to Neuronal Cells. Cell Stem Cell. 19 (3), 406-414 (2016).
- Nuñez, J. K., et al. Genome-wide programmable transcriptional memory by CRISPR-based epigenome editing. Cell. 184 (9), 2503-2519.e17 (2021).
- Xu, X., et al. A CRISPR-based approach for targeted DNA demethylation. Cell Discovery. 2 (1), 16009(2016).
- Leonetti, M. D., Sekine, S., Kamiyama, D., Weissman, J. S., Huang, B. A scalable strategy for high-throughput GFP tagging of endogenous human proteins. Proceedings of the National Academy of Sciences. 113 (25), (2016).
- Pattali, R. K., Ornelas, I. J., Nguyen, C. D., Xu, D., Divekar, N. S., Nuñez, J. K. CRISPRoff epigenetic editing for programmable gene silencing in human cells without DNA breaks. , (2024).
- Moon, H. C., Herschl, M. H., Pawluk, A., Konermann, S., Hsu, P. D. A combinatorial domain screening platform reveals epigenetic effector interactions for transcriptional perturbation. , (2024).
- Cappelluti, M. A., et al. Durable and efficient gene silencing in vivo by hit-and-run epigenome editing. Nature. 627 (8003), 416-423 (2024).
- Naumann, E. N., et al. rainwide silencing of prion protein by AAV-mediated delivery of an engineered compact epigenetic editor. Science. 384 (6703), ado7082(2024).
- Tremblay, F., et al. A potent epigenetic editor targeting human PCSK9 for durable reduction of low-density lipoprotein cholesterol levels. Nature Medicine. 31 (4), 1329-1338 (2025).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados