È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Dimostrare l'Usi del romanzo Spectrometer forza gravitazionale su Stretch e misura proteine fibrose
In questo articolo
Riepilogo
Questo è un passo-passo guida che mostra la finalità, il funzionamento ei risultati rappresentante del spettrometro romanzo forza gravitazionale.
Abstract
Lo studio della struttura macromolecolare è diventato fondamentale per il chiarimento dei meccanismi molecolari e funzione. Ci sono diversi bioinstruments limitato, ma importante, in grado di testare la dipendenza forza delle caratteristiche strutturali di proteine. Scala è stata un parametro che limita la quantità accuratamente i ricercatori possono scrutare il mondo nanomeccaniche di molecole, come gli acidi nucleici, enzimi e proteine motrici che eseguono sostiene la vita di lavoro. Microscopia a forza atomica (AFM) è ben sintonizzato per determinare le strutture native delle proteine fibrose con una risoluzione distanza alla pari con microscopia elettronica. Tuttavia, in studi AFM vigore, le forze sono in genere molto più alto di una singola molecola che si verifichino 1, 2. Trappole ottiche (OT) sono molto bravi a determinare la distanza relativa tra le perle intrappolati e possono impartire piccole forze 3. Tuttavia, essi non producono accurata lunghezze assolute delle molecole oggetto di studio. Simulazione molecolare fornire informazioni di supporto a tali esperimenti, ma sono limitate nella capacità di gestire le stesse grandi dimensioni molecolari, tempi lunghi, e convincere alcuni ricercatori, in assenza di altre prove 2, 4.
La forza gravitazionale spettrometro (GFS), riempie una nicchia critico nell'arsenale di un investigatore, fornendo una combinazione unica di capacità. Questo strumento è in grado di generare forze di solito con il 98% o maggiore precisione della gamma femtonewton alla gamma nanonewton. Le misure di distanza attualmente sono in grado di risolvere la lunghezza assoluta molecolare fino a cinque nanometri, e relative distanze di perline paio di separazione con una precisione simile a una trappola ottica. Inoltre, il GFS può determinare stretching o srotolare in cui la forza è vicino all'equilibrio o fornire una forza graduata a giustapporre contro qualsiasi cambiamento strutturale misurata. E 'anche possibile determinare il numero di residui di aminoacidi sono coinvolti in eventi di srotolare sotto carichi fisiologici forza 2. A differenza di altri metodi dove si trova la forza di calibrazione estesa che deve precedere qualsiasi test, il GFS non richiede alcuna calibrazione tale forza 5. Integrando i punti di forza di altri metodi, il GFS colmerà le lacune nella comprensione dei nanomeccanica delle proteine vitali e di altre macromolecole.
Protocollo
Introduzione alla configurazione di GFS Novel
La GFS è costituito da diversi componenti essenziali: un microscopio ottico normale, una montatura equatoriale, una telecamera e un computer [Figura 1]. La tenuta del flusso delle cellule da camera che contiene il campione è anche indispensabile secondo il disegno GFS. Il microscopio a luce è montato sulla montatura equatoriale in modo che il campo può essere ruotato in diversi orientamenti nello spazio. Questa capacità consente al vettore di gravità statica da sfruttare in modo che i campioni possono essere dinamicamente orientato in relazione al vettore così la forza di gravità può dare piconewton raggio carichi di forza ai campioni. La fotocamera sostituisce lente oculare del microscopio ottico in modo che possa registrare i cambiamenti nell'orientamento del campione. Questi dati grezzi è digitalizzato e manipolato dal computer di interpretare i dati in forza effettiva e misure di distanza. La tenuta di flusso-camera è stata progettata per consentire a tutti i gradi di libertà nello spazio senza perdita di campione. Nella camera trova la molecola campione che è legato vicino a una capolinea di un "ancorato" perla che è incollata alla superficie della camera. Il capolinea opposto è legato ad un "mobile" perla che è libero dalla superficie della camera. E 'questo cellulare tallone, libero nel tampone che possono essere attuate dai forza gravitazionale scandagliando la molecola legato a così carichi di forza bassa [Figura 2]. Il campione appare semplicemente come un paio di microsfere al microscopio, anche se ci vuole una certa esperienza a discernere il bene coppie utilizzabile accoppiato con il loro attaccamento ad una molecola da coppie in cui entrambi i talloni sono seduti sulla superficie del flusso-camera. Una modifica al sistema è l'aggiunta di una piattaforma galleggiante che tiene il GFS ed è sospeso da molle. In questa configurazione, una volta che il campione è stato ruotato in una posizione dove la forza gravitazionale può agire sul campione, la piattaforma e tutti i suoi componenti possono essere caduto contro la costante della molla. Vicino a caduta libera, la forza che agisce sul tallone mobile è prossimo allo zero e alla massima estensione delle molle ', la forza gravitazionale è moltiplicato per ben due volte. In questo modo, un graduato forza / distanza risposta può essere tracciata per misurare il comportamento di una singola molecola a carichi di forza diversi.
1. Microsfere di preparazione
- Immergere circa 10 mg di perline di vetro o silice nel 0,04% 3-amminopropiltrietossisilano (taglio con acetone) per due minuti.
- Risciacquare con due cambi di acqua bidistillata e centrifugare a pellet a 2000 xg per 5 minuti. Gettare il surnatante.
- Aggiungere 5 ml di tampone di accoppiamento (0,01 M piridina tagliato con acqua bidistillata e aggiustare il pH a 6,0), e agitare questa miscela vigorosamente. Centrifuga come sopra descritto. Ripetere questa operazione tre volte.
- Per torta umida di perline, aggiungere 2 ml di soluzione gluteraldehyde 5% (taglio gluteraldehyde con buffer di accoppiamento). Agitare vigorosamente.
- Sotto una cappa, ruotare tallone miscela / gluteraldehyde per 3 ore a temperatura ambiente.
- Centrifuga come sopra e aspirare il surnatante.
- Lavare le perline in 5 ml di buffer di accoppiamento agitando vigorosamente e centrifugare e aspirare il surnatante. Ripetere questa operazione altre tre volte.
- Aggiungere circa 15 ml di anticorpi desiderata per perline e agitare vigorosamente. Le sfere dovrebbero essere ruotato per 16-24 ore.
- Sotto il cofano, aggiungere 5 ml di soluzione quenching 1 M glicina (taglio glicina con doppio acqua distillata e aggiustare il pH a 7,0). Agitare vigorosamente questa miscela e ruotare per 30 minuti.
- Centrifuga e aspirare il surnatante.
- Aggiungere 5 ml di tampone di lavaggio (0,01 M Tris, pH 7,0, sodio azide allo 0,1%, 0,1% di BSA, 0,15 M di NaCl, e 0.001 M EDTA). Agitare vigorosamente questo, centrifuga e aspirare il surnatante. Ripetere questa operazione tre volte.
- Cambia il buffer a basso sale tampone (0,1 M KCl; imidazolo 0,02 M, 5 mM MgCl 2; regolare a pH 7,0). Ripetete tre volte.
2. Esempio di attaccamento al Microsfere
- Prendete una piccola quantità di perle di preparato (circa 2 microlitri di ogni torta di perline tuttavia, se c'è una discrepanza di diametro tra i lotti, è vantaggioso utilizzare intorno ad un rapporto 8:1 di grandi perle di piccole dimensioni) e aggiungerli alla una provetta con il tampone. Ridurre la concentrazione delle proteine a circa 5 micron, utilizzando il tampone. Preparare almeno un volume totale di 400 microlitri tra cui il buffer, la proteina, e le perle.
- Ruota intorno a questa miscela a 1 RPM per 3 ore (se la proteina è agitato troppo, sarà aggregato e diventano inutili).
3. Far scorrere Camera di preparazione
- Cappotto un vetrino da microscopio di spessore con 0,01% nitrocellulosa (in acetato di amile). Lasciate che questa diapositiva asciugare per circa 10 minuti.
- Con una fresa di vetro affilato, tagliate coperchio scorrevole in modo da fare una camera. Ciò richiede quattro strisce di vetro.
- Utilizzare il fattoORY bordo del vetro e rastrello che attraverso una macchia di grasso per vuoto su entrambi i lati del bordo.
- Strisce di grasso stampa patinata sulla nitrocellulosa secca coperto diapositiva per creare una casella sulla superficie del vetrino.
- Pipettare circa 2 ml di miscela tallone / proteine toccando pipetter sulla superficie di vetro all'interno della scatola.
- Aggiungere circa 20-400 ml di buffer di sale basso a seconda di come la camera è grande.
- Premere un slittamento di copertura sulla parte superiore della scatola che è già ricoperto di grasso vuoto per terminare la camera stagna e tamponata.
- Lasciate scorrere sedersi in un luogo pianeggiante così le perle hanno il tempo per ancorarsi alla nitrocellulosa.
4. GFS Acquisizione Dati
- Scivolo montare sul palco GFS
- Monitorare quello che la macchina GFS è la registrazione e la ricerca di legittimo "coppie di perline," in cui il microsfere piccolo è attaccato vicino all'equatore del microsfere di grandi dimensioni.
- Quando una coppia potenziale tallone viene trovato, passare attraverso la profondità di campo per determinare se il "mobile" tallone non è appoggiata sulla superficie del vetrino.
- Una volta che una coppia adatta viene identificato, si noti l'angolo mobili è lontano dal tallone d max (d max = distanza massima tra i centroidi delle microsfere accoppiato). Spostare il campo in grado di acquisire video.
- L'angolo di corsa del GFS dovrebbe essere sufficiente per registrare d min, d max e min d che possa rendere necessario 25-90 gradi a seconda della lunghezza della molecola.
- RECORD come la coppia viaggia da d a d min max e torna a d min.
- E 'una buona idea di girare anche un film dello sfondo in modo che possa essere sottratta in seguito per l'analisi.
- Se si esegue una goccia GFS, spostare la portata indietro d max e registrare la caduta di almeno 60 fotogrammi al secondo. La parte critica è l'oscillazione prima, ma i tempi di lunga può anche essere utilizzato per lo studio dinamico.
5. GFS Analisi dei dati
- Trasforma il video prima in immagine digitale "thresholded" ed eseguire macro in ImageJ per determinare la posizione centroide di ogni perlina in ogni fotogramma del video. Questo è anche per il video goccia.
- Dump X, Y e dati zona da ImageJ in Excel e tracciare i punti.
- Se una coppia proprio tallone è stata acquisita da video, una gobba evidente nel grafico è indicativo delle perle di essere a loro più vicino (d min) e al vertice del grafico è la posizione di d max.
- Utilizzando questi dati, il raggio di ciascuna microsfera deve essere determinato con precisione in ImageJ e tutte queste informazioni è messo nell'equazione nidificate:
d = [(g sin α) 2 + (g cos α + d max - g) 2] 0,5
g = [d min 2 r + b 2 - (r a + r b) 2] 0,5 r = b sin β
c = max d - r A - G
(D = distanza tra centroidi; g = forza di gravità; α = l'angolo in gradi parallelo alla lente dell'obiettivo; r b = raggio di mobili tallone, r a = raggio di ancorate tallone; β = angolo di attacco fuori l'asse del l'equatore del ancorati tallone. - Utilizzando il raggio dotati del cellulare tallone che dà anche il suo volume, e data la densità del tallone, la forza il tallone mobili impartisce sulla molecola può essere calcolato in piconewtons dopo la galleggiabilità del solvente viene sottratto. Questo metodo misura la forza sulla molecola legato in piconewtons e calcola la lunghezza assoluta molecola tra gli allegati degli anticorpi in nanometri. F = V (db) a (F = forza, V = volume, d = densità del microsfere di vetro, a = accelerazione dovuta alla gravità, b = la densità dell'acqua spostata).
6. Rappresentante dei risultati:
Se la preparazione tallone è fatto correttamente, ci saranno minimo aggregazione perla anche se ci possono ancora essere un gruppo occasionale tallone. Quando viene visto attraverso la portata, ci dovrebbe essere una distribuzione ragionevole di perline se accoppiati o meno nella camera.
E 'importante ridurre al minimo le vibrazioni quanto più possibile, per fare questo sia un tavolo d'aria, urti speciale assorbente piedi per un treppiede che contiene un montaggio EQ, o il sistema che utilizza sorgenti possono essere utilizzati per l'isolamento delle vibrazioni.
Un altro suggerimento utile riguardante la camera stagna flusso è di lasciarlo riposare per circa cinque minuti su un tavolo di livello dopo che è stato completamente costruito. Questo consente a qualsiasi perle più distaccato a stare a galla attraverso i buffer e resto nello strato di nitrocellulosa. Se la diapositiva sono stati invece montati direttamente dopo il completamento, l'investigatore dovrebbe continuamente fare i conti con perline letteralmente volando attraverso il campo di vista, e se questo accade durante l'acquisizione video che possono danneggiare °e sperimentare. Se questo è fatto correttamente, il volo tallone è significativamente ridotta al minimo e risultati video più pulito.
Quando una coppia potenziale tallone è identificato dall'operatore GFS, è utile per dirla con una rotazione preliminare per monitorare il comportamento della coppia. Raramente, il grande tallone non è saldamente fissata alla diapositiva. In questo caso, non serve ad utilizzare la coppia, perché è fondamentale che il più grande tallone rimanere ancorata in una posizione fissa per tutta la durata dell'acquisizione. Se la coppia è stabile e non presenta "ancorato rotolo perla", quindi è adatto per la sperimentazione.
Un grafico delle distanze di separazione tallone contro il cambiamento in angolo relativo alla forza di gravità può essere utilizzato per valutare i dati acquisiti. Un risultato di buon rappresentante avrebbe mostrato separazione, adagiata su un altopiano e come il tallone mobili si libera il tallone ancorato a causa della influenza della gravità, il grafico mostrerà più separazione. Questo continua fino a quando un bel picco è raggiunto che si chiama d max e la curva inizia a tornare di nuovo verso il basso la linea di base d min [Figura 1]. In condizioni ideali, questa firma è simmetrico. Un non-rappresentativo risultato avrebbe mostrato un grafico con i modelli incoherent mostrando non distinte d min-max d-d modello min; o sarebbe mostrano separazioni che sono ordini di grandezza troppo alto per una singola molecola indicating forse c'era un pezzo di polvere tra le perline, o forse che il tallone del mobile è stato semplicemente non collegato a tutti. Il processo di trovare la coppia, tiro esso, l'elaborazione, e l'analisi ha molte lacune fermare dove sono abbattuti improprio coppie tallone fuori. Quindi, se si arriva alla fine di tutto il processo e si dispone di una lunghezza molecola che è coerente con i risultati precedenti, si può essere molto fiducioso che la coppia originaria tallone è rappresentativo e possono essere inclusi nei risultati. In una giornata buona, circa la metà della ripresa tallone coppie possono essere prese attraverso di diventare un punto legittima dati. Per esempio, la lunghezza della bobina a spirale della miosina tra MF20 e MF30 anticorpi, si sa sulla base di EM dati e AFM, i dati di avvicinarsi 100 nm 2,7,8,9. Se il risultato è più volte questa lunghezza, il campione si è aggregato. I risultati presentati qui sono da standard esperimenti di rotazione GFS e dimostrare una distanza di 96 nm ± 5 nm, [Figura 2] che concorda a stretto contatto con le misure delle MF30 (che si lega a N-terminale della miosina subfragment-2) e MF20 (che si lega alla meromyosin luce) sulla distanza di separazione anticorpi miosina deduce dai valori di letteratura [Figura 3].
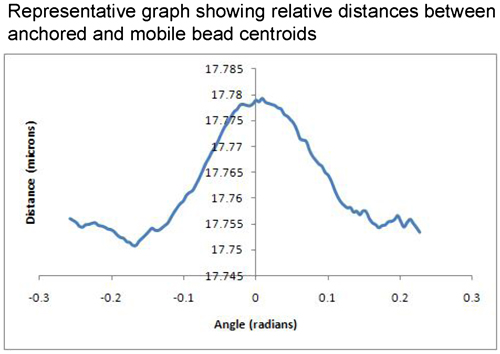
Figura 1. GFS configurazione. Parti principali del GFS sono etichettati.
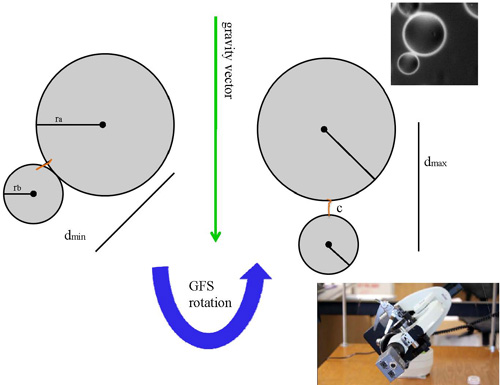
Figura 2. Schema di principio GFS. Lato sinistro mostra baricentro mobili tallone ad una distanza minima dal baricentro del tallone ancorata. Come GFS viene ruotato, mobile tallone allinea con vettore di gravità che è anche parallela con l'asse della molecola legato. In questa posizione, la distanza tra i centroidi delle perline mobili e fissi è al massimo. In alto a destra mostra una fetta rappresentante di un film GFS. In basso a destra è un'immagine della rotazione GFS subendo.
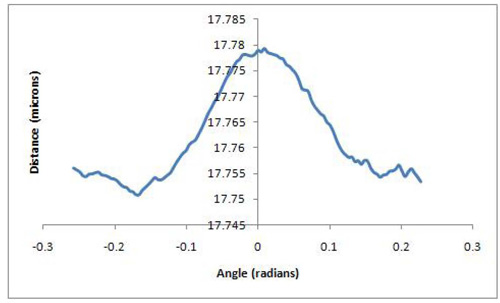
Figura 3 risultato Rappresentante mostrando min d sulla sinistra del grafico;. D max a una separazione relativa di 17,78 micron, e un ritorno al min d intorno alla linea di base di circa 17,75 micron di separazione relativa tra le perle.
Figura 4. Rappresentante risultato dell'esperimento di rotazione GFS mostrano distanza tra MF20 e MF30 anticorpi mediamente 96 nm. Questo rappresenta la durata approssimativa di S2.
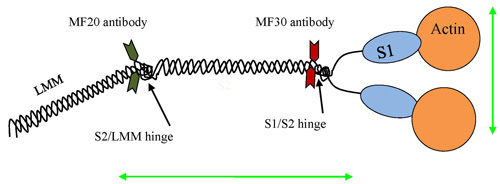
Figura 5. Miosina II dimero utilizzato per mostrare eventuali allegati da utilizzare con il GFS tra anticorpi e / o gli allegati di actina. Possibilità di attacco differenti per le diverse strategie consente GFS per misurare le diverse regioni, o di applicare forza perpendicolare o parallela al dominio asta del dimero miosina.
Access restricted. Please log in or start a trial to view this content.
Discussione
Durante la conversione di un filmato a una rappresentazione digitale thresholded, è fondamentale per l'immagine thresholded a mantenere la stessa area in ogni fotogramma del video. Perché le perle di una coppia cordone si muovono indipendentemente l'uno dall'altro, la sua deriva nelle aree thresholded può anche causare le distanze relative tra i centroidi delle perline alla deriva e introdurre errori significativi. Il controllo della zona ha ridotto la soglia di errore di cinque volte nelle misure da 26 n...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Riconoscimenti
Questo materiale si basa su un lavoro sostenuto dal National Science Foundation sotto Grant No. 0842736.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 3-Aminopropyltriethoxysilane | Polysciences, Inc. | 919-30-2 | |
| Acetone | Fisher Scientific | A18P-4 | |
| Pyridine | Sigma-Aldrich | 110-86-1 | |
| Glutaraldehyde | Fisher Scientific | G7776 | |
| Glycine | Research Organics | BP381-1 | |
| Tris | Sigma-Aldrich | 9682T | |
| Sodium azide | Amresco | 71289 | |
| BSA | Sigma-Aldrich | AMR-0332-100G | |
| NaCl | Sigma-Aldrich | S7653 | |
| EDTA | MSI | E9884 | |
| Nitrocellulose | Sigma-Aldrich | 60443 | |
| N-N Dimethyl Formamide | Extracted from Large New | D4254 | |
| Rabbit skeletal myosin II | Zealand White Rabbits (7-8) | NA | |
| MF30 antibody (9-10) | Developmental Studies Hybridoma Bank | MF30 | |
| MF20 antibody (6) | Hybridoma Bank | MF20 | |
| Lab microscope | Boreal | WW57905M00 | |
| Equatorial mount | Celestron | CG-5 | |
| Digital video cam | Sony Corporation | XCDV60 | |
| Caliper release | Cabelas | IA-415482 | |
| Compression spring | Jones Spring Co. | 723 | |
| Extension spring | Jones Spring Co. | 770 | |
| ImageJ | National Institutes of Health | NA | |
| Fire-i drivers & application | Unibrain | 3.80 | |
| Excel | Microsoft | NA |
Riferimenti
- Schwaiger, I., Sattler, C., Hostetter, D. R., Rief, M. The myosin coiled-coil is a truly elastic protein structure. Nat. Mater. 1, 232-235 (2002).
- Root, D. D., Yadavalli, V. M., Forbes, J. G., Wang, K. Coiled-coil nanomechanics and uncoiling and unfolding of the superhelix and alpha-helices of myosin. Biophysical Journal. 90, 2852-2866 (2006).
- Nishizaka, T., Miyata, H., Yoshikawa, H., Ishiwata, S., Kinosita, K. Unbinding force of a single motor molecule of muscle measured using optical tweezers. Nature. 377, 251-254 (1995).
- Gawalapu, R. K., Root, D. D. Fluorescence labeling and computational analysis of the strut of myosin's 50 kDa cleft. Arch. Biochem. Biophys. 456, 102-111 (2006).
- Kellermayer, M. S. Z. Visualizing and manipulating individual protein. Molecules Physiol. Meas. 26, R119-R153 (2005).
- Shimizu, T., Dennis, J. E., Masaki, T., Fischman, D. A. Axial arrangement of the myosin rod in vertebrate thick filaments: immunoelectron microscopy with a monoclonal antibody to light meromyosin. J. Cell Biol. 101, 1115-1123 (1985).
- Godfrey, J. E., Harrington, W. F. Self-association in the myosin system at high ionic strength. I. Sensitivity of the interaction to pH and ionic environment. Biochemistry. 9, 886-893 (1970).
- Root, D. D., Stewart, S., Xu, J. Dynamic docking of myosin and actin observed with resonance energy transfer. Biochemistry. 41, 1786-1794 (2002).
- Xu, J., Root, D. D. Conformational Selection during Weak Binding at the Actin and Myosin Interface. Biophys. J. 79, 1498-1510 (2000).
- Sattin, B. D., Pelling, A. E., Goh, M. C. DNA base pair resolution by single molecule force spectroscopy. Nucleic Acids Res. 32, 4876-4883 (2004).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon