È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Test di suscettibilità agli antibiotici indotti dallo stress su un chip
In questo articolo
Riepilogo
Abbiamo sviluppato una piattaforma microfluidica per test di suscettibilità agli antibiotici rapidi. Il fluido viene passato ad alta velocità sui batteri immobilizzati sul fondo di un canale microfluidico. In presenza di stress e antibiotici, i ceppi sensibili di batteri muoiono rapidamente. Tuttavia, i batteri resistenti possono sopravvivere a queste condizioni stressanti.
Abstract
Abbiamo sviluppato un metodo microfluidico rapido per i test di suscettibilità agli antibiotici in un ambiente basato sullo stress. Il fluido viene passato ad alta velocità sui batteri immobilizzati sul fondo di un canale microfluidico. In presenza di stress e antibiotici, i ceppi sensibili di batteri muoiono rapidamente. Tuttavia, i batteri resistenti sopravvivono a queste condizioni stressanti. L'ipotesi alla base di questo metodo è nuova: l'attivazione dello stress delle vie biochimiche, che sono obiettivi di antibiotici, può accelerare i test di suscettibilità agli antibiotici. Rispetto ai metodi standard di test di suscettibilità agli antibiotici, il passaggio di limitazione del tasso - crescita batterica - viene omesso durante l'applicazione di antibiotici. L'implementazione tecnica del metodo è in una combinazione di tecniche standard e approcci innovativi. Le parti standard del metodo includono protocolli di coltura batterica, definendo canali microfluidici in polidimetilsassano (PDMS), monitoraggio della vitalità cellulare con fluorescenza e elaborazione delle immagini batch per il conteggio dei batteri. Parti innovative del metodo sono nell'uso del flusso dei mezzi di coltura per l'applicazione di sollecitazioni meccaniche, nell'uso di enzimi per danneggiare ma non uccidere i batteri e nell'uso di substrati microarray per l'attacco batterico. La piattaforma sviluppata può essere utilizzata nello sviluppo e nei test di farmaci correlati agli antibiotici e non antibiotici. Rispetto agli esperimenti standard di sospensione batterica, l'effetto del farmaco può essere acceso e disattivato ripetutamente durante i periodi di tempo controllati. L'osservazione ripetitiva della stessa popolazione batterica è possibile nel corso dello stesso esperimento.
Introduzione
L'aumento della resistenza batterica intensifica la necessità di test di suscettibilità agli antibiotici a base di fenotipo veloce al fine di salvaguardare i nostri farmaci di ultima istanza. I test di suscettibilità standard si basano sull'inibizione della crescita batterica in presenza di antibiotici che prendono più (8-24) ore per essere completati. Abbiamo sviluppato un nuovo test di suscettibilità agli antibiotici su una piattaforma microfluidica che si basa sull'attivazione dello stress delle vie biosintetiche per accelerare l'azione degli antibiotici.
I test di suscettibilità agli antibiotici su scala microfluidico comportano il vantaggio di un uso efficace del campione, poiché richiedono un piccolo numero di batteri. Inoltre, i dispositivi microfluidici possono essere multiplexati per testare più campioni in più condizioni1,2. Recentemente, sono stati riportati diversi metodi microfluidici per i test di suscettibilità agli antibiotici3-9. In questi metodi, i batteri vengono coltivati all'interno di goccioline nano e picoliter3,7, nell'intero volume del canale microfluidico4-6,8, o come singoli batteri localizzati elettricamente sulla superficie inferiore del canale9. Sebbene questi test siano eseguiti in canali microfluidici, tutti monitorano la crescita microbica in presenza e assenza di antibiotici simili ai metodi tradizionali. Le misurazioni della crescita vengono effettuate tramite densità ottica, coloranti sensibili al pH o immagini a contrasto campo/fase luminoso o fluorescenza. Sebbene alcuni di questi test siano più veloci dei metodi tradizionali, ciascuno rileva passivamente la resistenza agli antibiotici. In altre parole, questi metodi richiedono ancora all'utente di attendere la crescita batterica come lettura finale.
Al contrario, abbiamo sviluppato un metodo che utilizza una combinazione di taglio e stress enzimatico per attivare vie biochimiche sensibili agli antibiotici10. Sfidare i batteri stressati con quegli antibiotici crea un test di suscettibilità più rapido. I batteri resistenti all'antibiotico sono in grado di resistere alle condizioni stressanti. I batteri sensibili, d'altra parte, vengono rapidamente uccisi dalle sollecitazioni combinate. La percentuale di morte cellulare dopo un'ora, misurata per microscopia utilizzando una macchia fluorescente a cellule morte, definisce il fenotipo dei batteri (resistente vs suscettibile).
Per un'implementazione riuscita del nostro metodo, i batteri devono essere immobilizzati sulla superficie inferiore del canale microfluidico. In questo modo, i batteri possono essere sottoposti a varie sollecitazioni e simultaneamente immagini al microscopio in un unico piano. Un vetrino al microscopio rivestito viene utilizzato per l'immobilizzazione dei batteri. Lo scivolo è prerivestito dal produttore con gruppi di epossossido per il legame proteico non specifico. Il legame non specifico di questi epossidi alle proteine superficiali batteriche attacca covalentemente i batteri alla superficie dello scivolo.
I ceppi sono testati in condizioni identiche (taglio + stress enzimatico) in assenza (controllo) e presenza (esperimento) di antibiotico. Il contrasto di fase e le immagini al microscopio a fluorescenza di ciascun canale vengono scattate automaticamente ogni due minuti per un'ora. Le designazioni di resistenza vengono quindi effettuate confrontando la percentuale di batteri morti nel canale sperimentale con quelli presenti nel canale di controllo. Dopo un'ora, un campione con una percentuale di morte cellulare superiore all'1% è considerato suscettibile, mentre meno dello 0,5% della morte è indicativo di resistenza. Le percentuali che rientrano tra questi due cut-off sono considerate indeterminate e il campione deve essere nuovamente testato.
I canali microfluidici sono definiti in PDMS, che è un materiale di scelta per i dispositivi microfluidici11. Il PDMS è otticamente trasparente in un'ampia gamma di lunghezze d'onda, biocompatibile, inerte, permeabile ai gas e ha una bassa permeabilità ai liquidi; pertanto è adatto per questi esperimenti.
La sollecitazione meccanica/di taglio è creata dal flusso di mezzi a temperatura ambiente sui batteri immobilizzati. (Nota: riscaldare il supporto a 37 °C non ha alcun effetto significativo sull'esito del saggio.) Le pompe automatiche per siringhe forzano i mezzi (contenenti macchie cellulari morte +/- antibiotico, nonché fattori di stress enzimatici opzionali) attraverso i canali microfluidici (200 μm x 400 μm) ad una portata di 1 ml/min per dare 6,25 kPa di forza di taglio o una velocità di taglio di 6.000 sec-1. Questo tasso è uguale o superiore alle sollecitazioni di taglio precedentemente studiate sugli Staphylococci.
L'enzima, lisostafina, è stato selezionato per esperimenti preliminari perché causa danni diretti alla parete cellulare dello Stafilococco. La concentrazione di licostafina (0,7 ng/ml) è stata sufficiente a causare danni alle pareti cellulari batteriche, ma non sufficiente a causare la morte delle cellule batteriche senza antibiotico nei tempi dell'esperimento. La licostaphin non è necessaria per la corretta designazione della suscettibilità batterica, ma aumenta il risultato, portando ad un aumento della morte cellulare nei ceppi sensibili. Al contrario, la sollecitazione di taglio è fondamentale per la funzione di dosaggio. Quando i ceppi di Staphylococcus aureus sensibili alla meticillina vengono trattati con lisostafina ed oxacillina in assenza di flusso, non viene registrata alcuna morte cellulare nel corso dell'esperimento.
La vitalità cellulare è monitorata con una macchia fluorescente a cellulemorte 12. La selezione del colorante era basata sulla sua capacità di macchiare selettivamente solo le cellule danneggiate, la sua non tossicità per le cellule vive e la sua bassa fluorescenza di fondo, che ne consentiva l'aggiunta diretta al supporto cellulare senza ulteriori passaggi. La scelta di una concentrazione di colorante fluorescente di 0,25 μM doveva raggiungere livelli di segnale accettabili durante un tempo di esposizione di 1,6 secondi alla luce di eccitazione a fluorescenza.
Il beta-lattame, oxacillina, è stato utilizzato nei nostri studi preliminari. Le specie S. aureus (MRSA) resistenti alla meticillina sono resistenti all'oxacillina e non mostreranno alcuna morte cellulare apprezzabile nel periodo di tempo dell'esperimento. La concentrazione di 50 μg/ml è stata determinata negli studi preliminari. Concentrazioni più basse di antibiotici hanno dato meno separazione tra ceppi resistenti e sensibili, mentre concentrazioni più elevate non hanno causato una differenza apprezzabile nei risultati sperimentali.
In precedenza abbiamo riferito del successo dello sviluppo di un test che combina sollecitazioni meccaniche ed enzimatiche che influenzano direttamente la parete cellularebatterica 13 con un antibiotico che inibisce la biosintesi della paretecellulare 14,15. Questi esperimenti di prova di principio sono stati effettuati su un gruppo di MRSA e S. aureus (MSSA) sensibili alla meticillina. Tuttavia, con la selezione di parametri sperimentali adeguati, il nostro metodo dovrebbe essere applicabile a più specie di batteri e più classi di antibiotici.
Protocollo
1. Creare il livello PDMS (Figura 1)
- Mescolare vigorosamente PDMS e agente polimerizzante in un rapporto 10:1. Per rimuovere le bolle, degasare la miscela viscosa in una camera a vuoto per 1 ora a temperatura ambiente.
- Su una scala, versare lentamente il PDMS sullo stampo in alluminio. Versare dal centro e mantenere livellato lo stampo. Assicurati di lasciare i perni scoperti. Smettere di versare una volta raggiunto il peso target.
Il nostro stampo richiede 4 g di PDMS e 0,4 g di reagente di polimerizzazione. - Livellare lo stampo all'interno di un forno e polimerigliare a 37 °C durante la notte.
I tempi di polimerizzazione alternativi sono di 2 ore a 60 °C o 1 ora a 90 °C. - Sezionare lo strato PDMS polimeri lungo il bordo dello stampo e staccarlo con cura dalla superficie dello stampo con un paio di forcep. Pulire la superficie dello stampo con il 70% di etanolo e una punta Q.
2. Assemblare la cella di flusso secondo la figura 2
L'assemblaggio standard di PDMS con vetrini viene eseguito attraverso il trattamento al plasma di ossigeno di entrambe le superfici, che garantisce un legame senza perdite tra il PDMS e lo scivolo di vetro al microscopio. Nel protocollo presentato, il trattamento al plasma distruggerebbe il rivestimento chimico sul vetrino. Pertanto lo scivolo è sigillato a pressione piuttosto che trattato al plasma.
- Posizionare la finestra di vetro nella tasca della cella di flusso.
- Disporre uno scivolo di vetro rivestito sopra la finestra di vetro all'interno della tasca della cella di flusso con il lato attivo verso l'alto e posizionare lo strato PDMS con i canali rivolti verso il basso sopra di esso. Posizionare la diapositiva PDMS in modo che gli ingressi del canale si allineino con i fori di scorrimento nella piastra metallica. Spingere delicatamente l'aria fuori tra gli strati.
- Capovolgere l'assieme PDMS/vetro in modo che il PDMS sia affacciato sulla finestra di vetro. Sovrapporre gli ingressi del canale PDMS ai fori di through nella piastra metallica.
- Posizionare la piastra di pressione sopra e stringere le viti.
- Posizionare la cella di flusso assemblata al microscopio. Impostare l'ingrandimento del microscopio su 60X e preallineare le posizioni del canale.
3. Preparare i batteri della fase di log
- Giorno prima dell'esperimento: Inoculare 50 ml di brodo di Mueller Hinton contenente il 2% di NaCl (MH2) con una colonia batterica. Agitare a 250 giri/min durante la notte a 37 °C.
Uno o due ceppi batterici possono essere studiati in un esperimento per l'insieme descritto. - Prima dell'esperimento: Mescolare 50 μl di coltura batterica durante la notte in 50 ml di mezzi MH2. Agitare a 250 giri/min per 3 ore a 37 °C per assicurarsi che i batteri siano in fase di log.
4. Riscaldare i componenti sperimentali della soluzione almeno 10 minuti prima che siano necessari
- Scongelare il colorante fluorescente (5 mM) e la licostafina (10 μg/ml) a temperatura ambiente.
- Riscaldare la polvere di oxacillina a temperatura ambiente.
5. Preparare e caricare la sospensione batterica
- Dopo la fine della sottocultura di 3 ore: Assumere 10 ml di coltura batterica e centrifuga a 1.650 x g per 2 min.
- Rimuovere i batteri supernatanti e resuspend in 1 ml di mezzi MH2 freschi.
- Fissare una breve lunghezza del tubo a una siringa di blocco Luer da 1 ml. Lavare il tubo della siringa con 1 ml di supporto. Lasciare un po 'di supporto nei tubi per evitare bolle d'aria quando si disegna la sospensione batterica.
- Caricare 0,7 ml di batteri di tipo 1 nella siringa. Riempire due canali della cella di flusso con batteri di tipo 1. Guarda che il liquido appaia sull'altro lato del canale dopo ca. 150 μl.
La trasparenza del canale cambia man mano che viene riempito di batteri. - Se si sperimentano più tipi di batteri, ripetere la procedura di caricamento per i batteri di tipo 2 nei due canali rimanenti della cella di flusso.
- Posizionare la cella di flusso all'interno dell'incubatore a 37 °C per 45 minuti per consentire la sedimentazione batterica e l'attacco alla superficie dello scivolo.
6. Preparare e caricare le soluzioni sperimentali
- Preparare 140 μl di soluzione di colorante fluorescente da 0,5 mM mescolando 14 μl di materiale colorante fluorescente (5 mM) e 126 μl di mezzi MH2.
- Diluire 10 mg di oxacillina in 40 ml di mezzi MH2 per ottenere una concentrazione finale di 250 μg/ml di ossacillina.
- Preparare 130 ml di soluzione di controllo con concentrazioni finali di colorante fluorescente da 0,25 μM e 0,7 ng/ml di lysostaphin. Per fare ciò, mescolare 65 μl di colorante fluorescente (0,5 mM), 9,12 μl di lysostaphin stock (10 μg/ml) e 130 ml di mezzi MH2.
- Preparare 130 ml di soluzione antibiotica con concentrazioni finali di colorante fluorescente da 0,25 μM, 0,7 ng/ml di licostafina e 50 μg/ml di ossacillina. A tal fine, mescolare 65 μl di colorante fluorescente (0,5 mM), 9,12 μl di lysostaphin stock (10 μg/ml), 26 ml di oxacillina (250 μg/ml) e 104 ml di mezzi MH2.
- Riempire due siringhe da 60 ml con soluzione di controllo e due siringhe da 60 ml con soluzione antibiotica. Conservare le soluzioni avvolte in un foglio di alluminio per evitare la degradazione indotta dalla luce dei reagenti.
Riempire e troppo le siringhe per tenere conto della perdita dovuta al lavaggio dei tubi. - Rimuovere le bolle d'aria dalle siringhe svolazzando. Collegare e riempire i tubi di ingresso alla punta con la soluzione sperimentale.
- Montare le siringhe sulla pompa. Posizionare prima la siringa con il volume più piccolo, quindi bloccare la posizione dello stantuffo. Montare il resto delle siringhe sulla pompa, spremendo i pistoni se necessario.
- Impostare la velocità della pompa su 1 ml/min e il volume della pompa su 60 ml. Fare un lavaggio con la pompa fino a quando non viene visto un flusso costante di liquido da tutte le siringhe.
7. Impostare la cella di flusso al microscopio
- Rimuovere la cella di flusso dalla centrifuga e montarla sullo stadio del microscopio (Figura 3).
- Collegare tubi di ingresso/uscita a ciascuno dei canali delle celle di flusso (un ingresso/un uscita per canale).
La raccolta dell'output in quattro diversi contenitori consente la misurazione dei singoli volumi di uscita del canale.
8. Esegui l'esperimento di 60 minuti
- Controllare le posizioni preallineate dal passaggio 2.5. Se il campo visivo del microscopio non è centrato sul canale e/o è fuori fuoco, regolare le impostazioni e salvare le nuove posizioni.
Una messa a fuoco precisa potrebbe non essere possibile prima dell'inizio del flusso a causa dell'alta densità di batteri caricati. - Impostare il tempo di acquisizione del contrasto di fase su 10 msec e il tempo di acquisizione della fluorescenza su 1.600 msec.
- Ottenere immagini di contrasto di fase e fluorescenza per ogni posizione prima di iniziare il flusso.
Questo fornisce una stima qualitativa della densità batterica caricata. - Avviare il flusso di liquido e verificare immediatamente che il microscopio sia focalizzato sul fondo dei canali.
- Scatta immagini di contrasto di fase e fluorescenza delle aree di destinazione entro il primo minuto di flusso.
- Acquisire immagini ogni 2 minuti dopo il primo set di immagini fino a quando non si sono verificati 60 minuti di flusso. Rifocalizzare se necessario.
9. Disinfettare la cella di flusso
- Fare una soluzione di candeggina al 10% in un bicchiere (100 ml). Riempire 4 siringhe da 20 ml con 10 ml della miscela. Debubble le siringhe e attaccarle alla cella di flusso.
Ci ranno ~ 1-2 minuti perché i canali siano chiari dai batteri. - Impostare la velocità della pompa su 1 ml/min e il volume della pompa su 3 ml. Corri per 3 minuti.
- Riempire 4 siringhe da 60 ml con 60 ml di acqua DI. Debubble le siringhe e attaccarle alla cella di flusso.
- Impostare la velocità della pompa su 1 ml/min e il volume della pompa a 30 ml. Corri per 30 minuti.
- Monitorare la pulizia del canale al microscopio.
- Smontare la cella di flusso. Scartare la diapositiva epossidica utilizzata. Immergere i componenti delle celle di flusso in acqua DI per 20 min.
10. Analizzare le immagini e generare dati
- Contare il numero di batteri in ogni immagine.
Il software di accesso aperto CellProfiler viene utilizzato per eseguire l'elaborazione delle immagini batch16. Nella tabella 1 viene riepilogata una struttura di alto livello della routine CellProfiler. Il numero di batteri presenti nell'immagine di contrasto di fase (Np) fornisce la conta batterica totale. Il numero di batteri visibili nell'immagine di fluorescenza (Nf) fornisce il numero di batteri morti. - Calcolare la percentuale di morte delle cellule batteriche normalizzate in funzione del tempo.
- Importare Nf e Np per singole immagini nel software di analisi dei dati.
- Calcolare la frazione di batteri morti in ogni canale in un punto di tempo specifico dato dalla frazione (Nf / Np) a t = T.
- Sottrarre la frazione di batteri morti presenti all'inizio dell'esperimento (t = 1 min) sia per il controllo che per i canali sperimentali.
- Sottrarre la frazione di batteri morti presenti nel canale di controllo da quella presente nel canale sperimentale in ogni punto del tempo con la seguente equazione:
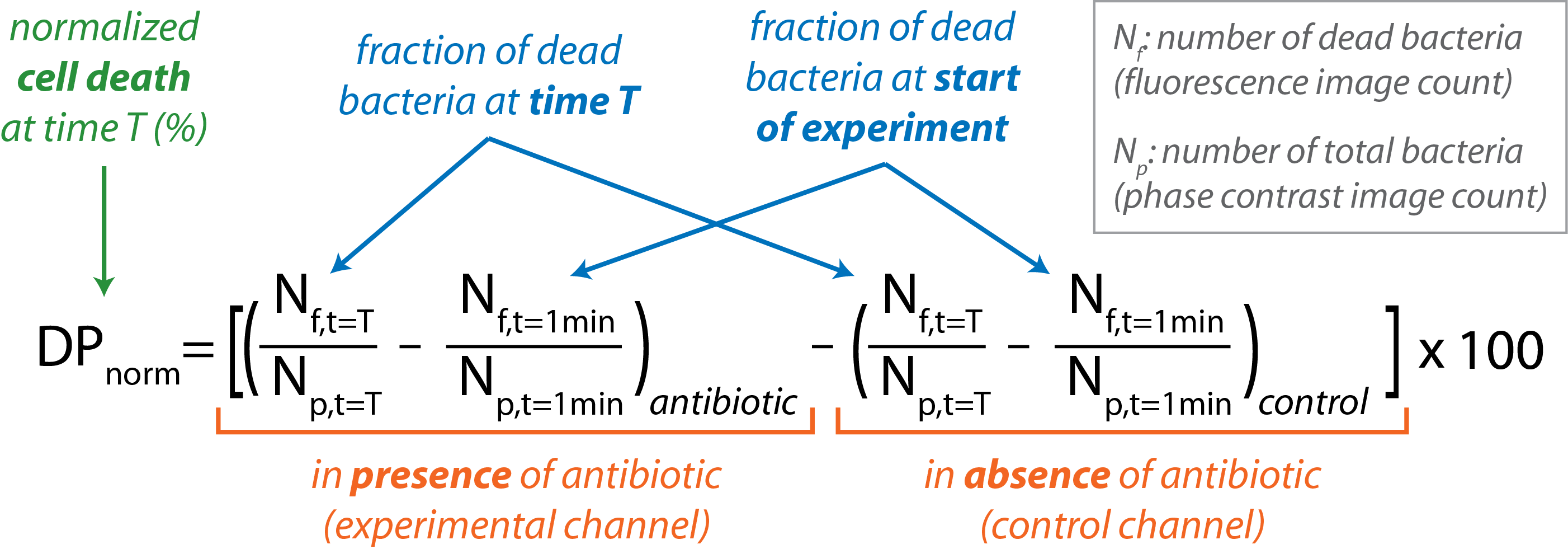
- Grafico DPnorm vs.
Si noti che un campione con una percentuale di morte cellulare superiore all'1% è considerato suscettibile, mentre meno dello 0,5% di morte è indicativo di resistenza. Le percentuali che rientrano tra questi due cut-off sono considerate indeterminate e il campione deve essere nuovamente testato. - Utilizzare un foglio di calcolo per riepilogare e analizzare i risultati di diversi esperimenti.
Risultati
I dati presentati nella figura 4 mostrano la risposta di un ceppo di Staphylococcus aureus sensibile nel tempo in un canale microfluidico contenente antibiotici. Le immagini di contrasto di fase acquisite a 1 minuto e alla fine dell'esperimento di 1 ora sono mostrate nelle figure 4A e B. I dati analizzati di 1 ora sono mostrati nella figura 4C con i batteri evidenziati in rosso (5.828 totali). Le corrispondenti immagini a fluorescenza ...
Discussione
Il protocollo presentato è stato convalidato e ottimizzato in una serie di esperimenti con ceppi stafilococco aureo sensibili alla meticillina e resistenti alla meticillina10. Pertanto, questo protocollo senza modifiche dovrebbe essere direttamente applicabile ad altri ceppi di S. aureus e altri antibiotici con meccanismi d'azione che influenzano la biosintesi della parete cellulare batterica. Tipi di batteri diversi da S. aureus possono richiedere variazioni nei parametri di sollec...
Divulgazioni
Il metodo microfluidico è in attesa di brevetto: Sauer-Budge A, Sharon A, Kalashnikov M, Wirz H, inventori; Metodo e dispositivo per il rilevamento rapido del brevetto di resistenza/suscettibilità agli antibiotici batterici PCT/US10/33523.
Riconoscimenti
Ringraziamo gli ingegneri e gli studenti del Fraunhofer Center for Manufacturing Innovation. Per aver contribuito alla progettazione, lavorazione e automazione del sistema sperimentale, ringraziamo Andreas Prinzen, Holger Wirz, Doug Foss, David Chargin e il Dr. Sudong Shu. Ringraziamo Julia Kuckartz, Melanie Zimmermann, Niko Kraetzmar, Tim Gumbel, Josh Villanueva, Minori Shimizu e Katarzyna Kuliga per l'aiuto nel test dei protocolli sperimentali e nella raccolta dei dati. Prendiamo atto della dott.ssa Anne E. Carpenter e di Mark-Anthony Bray della piattaforma di imaging presso il Broad Institute di Harvard e il MIT per l'aiuto nello sviluppo della routine di analisi delle immagini in CellProfiler. Il progetto descritto è stato sostenuto in parte dai Premi R21AI079474 e 1R01AI101446 del National Institute of Allergy and Infectious Diseases. Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente le opinioni ufficiali dell'Istituto Nazionale di Allergia e Malattie Infettive o degli Istituti Nazionali di Salute. Il progetto è stato sostenuto anche da Fraunhofer USA.
Materiali
| Name | Company | Catalog Number | Comments |
| SYTOX Green | Invitrogen Corporation | S7020 | Dead cell fluorescence stain |
| Bovine Serum Albumin (BSA) | Sigma Aldrich, Inc | A9418-5G | Used for lysostaphin storage |
| Sodium Acetate | Sigma Aldrich, Inc | S8750-500G | Used for lysostaphin storage |
| Lysostaphin | Cell Sciences | CRL309A | Arrives as 1 mg solid. For storage: Dissolve in 20 mM sodium acetate. Mix with BSA solution to final concentration of 1% BSA and 100 µg/ml lysostaphin for storage |
| Oxacillin salt | Sigma Aldrich, Inc | 28221-1G | Antibiotic |
| Mueller Hinton Broth | Fisher Scientific | DF0757-17-6 | |
| Sodium chloride | Sigma Aldrixh | S3014-500G | 2% added to Mueller-Hilton broth prior to autoclaving |
| 1 ml, Luer-lock syringe | BD (Beckton, Dickinson and Comp.) | 14-823-2F | |
| 2 oz, Luer-lock syringe | BD (Beckton, Dickinson and Comp.) | 309653 - 60 mL | Overfill to ~65 ml |
| Microscope | |||
| Inverted Fluoresccence Microscope Olympus IX-70 | Cambridge Scientific | 9349 | |
| 60X, Fluorescence/Phase contrast objective | Olympus Corp. | LCPlan F1 60x/0.70 Ph2 | |
| Retiga 12-bit monochrome CCD camera | QImaging | RET-4000R-F-M-12-C | |
| Microscope automation | |||
| Shutters phase contrast/fluorescence | PRIOR Scientific | H204/H202 | |
| X/Y Stage | PRIOR Scientific | H107AENN | |
| Focus motor | PRIOR Scientific | H122 | |
| Joystick for XYZ control | PRIOR Scientific | CS152EF | |
| Proscan Controller | PRIOR Scientific | H3-XY2 | |
| Image Acquisition Software | Fraunhofer CMI | ||
| Flow Cell Assembly and PDMS | |||
| Flow Cell | BU Scientific Instruments Facility/Fraunhofer CMI | 3333-1044 | Engineering drawings were produced by Fraunhofer CMI |
| Glass window | Fraunhofer CMI | 3333-1054 | Glass window was cut to the proper size at Fraunhofer CMI |
| BOROFLOAT Window 50 mm x 50 mm | Edmund Optics Inc. | NT48-543 | |
| Sealing plate | BU Scientific Instruments Facility | 3333-1045 | |
| Epoxide glass slide | Arrayit Corporation | SuperEpoxy 2 | |
| PDMS master | Fraunhofer CMI | 3333-1053 | Master machined in aluminum or brass with UPM-0005 (ultrapresicion fly-cutting machine) |
| PDMS slide design | Fraunhofer CMI | 3333-1053 | |
| Tubing | |||
| Nut, Super flangeless Tinytight, headless, 1/16 in, PEEK, green | IDEX Health Science | M-644-03 | Flow cell inputs/outputs are tapped for this ferrule |
| Ferrule, Tinytight, 1/16 in, 6-40, .030 in TH, PEEK w/ SS lock ring, black | IDEX Health Science | M-657 | |
| Nut, Super flangeless Tinytight, headless, 1/16-1/32 in, 1/4-28, PEEK, natural | IDEX Health Science | P-255 | |
| Ferrule, Super Falngeless, 1/16 in, Tefzel (ETFE), yellow | IDEX Health Science | P-259 | Fits Luer-lock adapter |
| Tubing, Teflon FEP, .030 in x 1/16 in x 20 ft, green | IDEX Health Science | 1520G | |
| Adapter, quick connect female Luer to female 1/4-28, PEEK, red | IDEX Health Science | P-658 |
Riferimenti
- Mairhofer, J., Roppert, K., Ertl, P. Microfluidic systems for pathogen sensing: a review. Sensors. 9, 4804-4823 (2009).
- Yager, P., et al. Microfluidic diagnostic technologies for global public health. Nature. 442, 412-418 (2006).
- Boedicker, J. Q., Li, L., Kline, T. R., Ismagilov, R. F. Detecting bacteria and determining their susceptibility to antibiotics by stochastic confinement in nanoliter droplets using plug-based microfluidics. Lab Chip. 8, 1265-1272 (2008).
- Cao, J., et al. Uncovering toxicological complexity by multi-dimensional screenings in microsegmented flow: modulation of antibiotic interference by nanoparticles. Lab Chip. 12, 474-484 (2012).
- Chen, C. H., et al. Antimicrobial susceptibility testing using high surface-to-volume ratio microchannels. Anal. Chem. 82, 1012-1019 (2010).
- Churski, K., et al. Rapid screening of antibiotic toxicity in an automated microdroplet system. Lab Chip. 12, 1629-1637 (2012).
- Eun, Y. J., Utada, A. S., Copeland, M. F., Takeuchi, S., Weibel, D. B. Encapsulating bacteria in agarose microparticles using microfluidics for high-throughput cell analysis and isolation. ACS Chem. Biol. 6, 260-266 (2011).
- Kim, K. P., et al. In situ monitoring of antibiotic susceptibility of bacterial biofilms in a microfluidic device. Lab Chip. 10, 3296-3299 (2010).
- Peitz, I., van Leeuwen, R. Single-cell bacteria growth monitoring by automated DEP-facilitated image analysis. Lab Chip. 10, 2944-2951 (2010).
- Kalashnikov, M., Lee, J. C., Campbell, J., Sharon, A., Sauer-Budge, A. F. A microfluidic platform for rapid, stress-induced antibiotic susceptibility testing of Staphylococcus aureus. Lab Chip. 12, 4523-4532 (2012).
- McDonald, J. C., Whitesides, G. M. Poly(dimethylsiloxane) as a material for fabricating microfluidic devices. Acc. Chem. Res. 35, 491-499 (2002).
- Roth, B. L., Poot, M., Yue, S. T., Millard, P. J. Bacterial viability and antibiotic susceptibility testing with SYTOX green nucleic acid stain. Appl. Environ. Microbiol. 63, 2421-2431 (1997).
- Francius, G., Domenech, O., Mingeot-Leclercq, M. P., Dufrene, Y. F. Direct observation of Staphylococcus aureus cell wall digestion by lysostaphin. J. Bacteriol. 190, 7904-7909 (2008).
- Jordan, S., Hutchings, M. I., Mascher, T. Cell envelope stress response in Gram-positive bacteria. FEMS Microbiol. Rev. 32, 107-146 (2008).
- Koch, A. L. Bacterial wall as target for attack: past, present, and future research. Clin. Microbiol. Rev. 16, 673-687 (2003).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7, R100 (2006).
- Kohanski, M. A., Dwyer, D. J., Collins, J. J. How antibiotics kill bacteria: from targets to networks. Nat. Rev. Microbiol. 8, 423-435 (2010).
- Kohanski, M. A., Dwyer, D. J., Hayete, B., Lawrence, C. A., Collins, J. J. A common mechanism of cellular death induced by bactericidal antibiotics. Cell. 130, 797-810 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon