Method Article
Uso di
In questo articolo
Riepilogo
Per contrastare la diffusione degli agenti patogeni, le cellule ospiti riorganizzano il loro citoscheletro a compartimenti stagni batteri e indurre l'autofagia. Utilizzando Shigella infezione di cellule di coltura di tessuti, di accoglienza e di determinanti patogeni alla base di tale processo sono identificati e caratterizzati. Utilizzando modelli di zebrafish di infezione Shigella, il ruolo di molecole e meccanismi scoperti sono indagato in vivo.
Abstract
Shigella flexneri è un patogeno intracellulare che può sfuggire da phagosomes per raggiungere il citosol, e polimerizzare l'host citoscheletro di actina per promuovere la sua motilità e la diffusione. Nuovo lavoro ha dimostrato che le proteine coinvolte nella motilità base di actina-sono anche legati ad autofagia, un processo di degradazione intracellulare cruciale per l'immunità delle cellule autonome. Sorprendentemente, cellule ospiti possono impedire a base actina-motilità di S. flexneri da compartimenti batteri all'interno Septin 'gabbie' e mira a autofagia. Queste osservazioni indicano che una comprensione più completa di septins, una famiglia di proteine leganti il GTP filamentosi, fornirà nuove intuizioni nel processo di autofagia. Questo rapporto descrive i protocolli per monitorare le interazioni autofagia-citoscheletro causate da S. flexneri in vitro utilizzando cellule di coltura di tessuti e in vivo utilizzando larve di zebrafish. Questi protocolli consentono indagini di mechanis intracellularims che controllano la diffusione batterica a livello dell'organismo molecolare, cellulare, e tutto.
Introduzione
Shigella flexneri, un batterio enteropatogena invasivo Gram-negativi, può sfuggire phagosomes al citosol, e polimerizzare il citoscheletro di actina host per eludere le risposte immunitarie citosoliche e promuovere intra e intercellulare movimento 1,2. Nonostante la comprensione della motilità actina-base in vitro 3,4, i meccanismi che limitano la diffusione dei batteri in vivo non sono state completamente definite. Questo è fondamentale per una comprensione più completa di immunità innata e difesa ospite.
Septins, una famiglia altamente conservata di proteine tra i metazoi, sono guanosina trifosfato (GTP) -binding proteine che si assemblano in complessi etero-oligomeriche e formano filamenti polari che si associano con le membrane cellulari e il citoscheletro 5,6. Studi recenti hanno scoperto che le cellule ospiti infette possono impedire Shigella motilità actina base da batteri compartimentazione mirati a autophFOXG dentro "gabbie Septin", rivelando il primo meccanismo cellulare che contrasta la motilità actina base 7,8. Un campo spalancato di indagine ora si trova in 'biologia Septin e infezioni'. Montaggio Septin, indotta da una varietà di agenti patogeni (ad esempio, Listeria monocytogenes 7,9,10, Mycobacterium marinum 7,8, Candida albicans 11), potrebbe emergere come una questione chiave in difesa ospite 5,12.
L'autofagia, un processo di degradazione intracellulare altamente conservata, è visto come una componente chiave della immunità cellulo-autonoma a causa della sua capacità di fornire i batteri citosol al lisosoma 13,14. Tuttavia, il ruolo dell'autofagia batterica in vivo per limitare o promuovere la replicazione batterica rimane poco compresa 15,16. Il pesce zebra (Danio rerio) è emerso come un modello vertebrato per lo studio delle infezioni perché è otticamente accessibilinelle fasi larvali quando il sistema immunitario innato è già funzionante 17,18. Un recente lavoro ha caratterizzato la suscettibilità delle larve di zebrafish a S. flexneri, un paradigma per autofagia batterica 15, ed ha utilizzato il modello di infezione -zebrafish Shigella per studiare la manipolazione di autofagia per la terapia antibatterica in vivo 19.
Questo rapporto fornisce nuovi strumenti e saggi per studiare S. interazioni flexneri con autofagia e il citoscheletro. In una prima fase, i protocolli per monitorare le interazioni autofagia-citoscheletro vengono descritte utilizzando infezione Shigella della linea di cellule epiteliali umane HeLa. Per valutare il ruolo delle interazioni autofagia-citoscheletro sul processo di infezione Shigella in vitro, i metodi per manipolare l'autofagia e componenti del citoscheletro (utilizzando siRNA o reagenti farmacologici) sono forniti. Nuovo lavoro ha dimostrato che utilizzando Shigella infezione of larve zebrafish, saggi simili può essere applicato per studiare la biologia cellulare di infezione in vivo. Protocolli per la preparazione e infettare zebrafish larve sono dettagliati, e di valutare la risposta dell'ospite alle infezioni Shigella in vivo, i protocolli per determinare la sopravvivenza host e carica batterica delle larve infette sono forniti. Metodi per monitorare l'assunzione di Septin e autofagia marcatori per Shigella (utilizzando larve di zebrafish fisso o vivente) e metodi per testare il ruolo di questi processi in vivo [utilizzando oligonucleotidi morpholino (iniettati in 1-4 embrioni stadio delle cellule) o reagenti farmacologici ( aggiunto direttamente all'acqua del bagno zebrafish)] vengono anche discussi. Questo programma di lavoro dovrebbe fornire informazioni sui meccanismi necessari per il controllo dell'infezione da risposte ospitanti citosolici.
Protocollo
1 Monitoraggio autofagia e il citoscheletro in vitro utilizzando colture cellulari
- Preparare S. flexneri
- Piastra S. flexneri M90T (wild-type) da -80 ° C stock di glicerolo su un Congo Red trittico di caseina di soia (TCS) piastra di agar. Incubare per una notte a 37 ° C. La stessa piastra può essere utilizzata per diversi esperimenti.
- Scegli un individuo colonia e crescere in 8 ml di mezzi TCS in uno shaker notte a 37 ° C.
NOTA: Congo-rosso vincolante indica che il plasmide di virulenza è stata mantenuta. - Per sottocultura batteri per la crescita esponenziale, inoculare TCS fresco con la coltura batterica pernottamento in 1/80 di diluizione e crescere in uno shaker a 37 ° C a OD 600 = 0,3-0,6.
- Gira la sottocultura batterica a 1.000 xg per 5 min. Lavare il pellet con il MEM e centrifugare a 1000 xg per 5 min. Ricostituire il pellet in MEM a OD 600 = 0,3-0,6.
- Preparare HeLa Cells per infezione
- Grow cellule HeLa in 'terreno completo', cioè., MEM più L-alanil-L-glutammina integrate con 1 mM di piruvato di sodio, soluzione 0,1 mM aminoacido non essenziale, e siero di vitello fetale del 10%.
- Piatto 1-1,5 x 10 5 cellule in piastre da 6 pozzetti 24 o 48 ore prima dell'inizio dell'esperimento. Piatto su vetrini in 6 pozzetti per la microscopia, o la piastra di 35 millimetri di vetro piatti inferiori a prepararsi per l'imaging dal vivo.
NOTA: (ad es., Code di actina, gabbie Septin) Seguire autofagia (ad esempio, ATG8 / LC3 + ve autophagosomes) e citoscheletro dinamiche in tempo reale durante l'infezione da Shigella utilizzando l'imaging dal vivo, le cellule di coltura tissutale possono essere trasfettate con GFP, RFP- o costrutti CFP-targhetta (vedi discussione).
- Infezione
- Infettare le cellule con 100: 1 molteplicità di infezione (MOI) di Shigella (OD 600 = 0,3-0,6) diluito in MEM; aggiungere direttamente alle cellule HeLa placcati in 6-wpiastre ELL 24 - 48 ore prima di infezione (come descritto nella sezione 1.2) in 2 ml di MEM (siero Starved).
- Per massimizzare l'adesione batterica alle cellule ospiti, batteri e cellule centrifugare a 700 xg per 10 min a temperatura ambiente. Dopo centrifugazione, incubare per 30 min a 37 ° C, 5% di CO 2 e consentire infezione per procedere.
- Lavare le cellule infettate due volte con MEM fresca e incubare con gentamicina contenenti terreno completo (50 mg / ml, per eliminare i batteri extracellulari) per 1-4 ore a seconda dell'esperimento.
- Fissaggio e Etichettatura cellule HeLa infette per microscopia
- Lavare le cellule infettate due volte con PBS 1x, e fissare per 15 minuti a 4% paraformaldeide in PBS 1x a temperatura ambiente. Per rimuovere paraformaldeide, Lavare le cellule con PBS 1x 2x.
- Incubare le cellule fissate in 50 mM di cloruro di ammonio per 10 minuti a temperatura ambiente. Lavare una volta con PBS 1x, e permeabilize cellule per 4 min con 0,1% di ossido di etilene ottilfenolo condensa in camera temperatura.
NOTA: Alternative a ottilfenolo etilene ossido di condensa per permeabilizzazione, come la saponina o metanolo, può essere applicato per diversi conservazione delle strutture cellulari 20. - Lavare le cellule in 1x PBS e incubare in camera umida con anticorpi primari contro i componenti critici autofagia (ad esempio, p62 / SQSTM1) o il citoscheletro Septin (SEPT2, SEPT6, SEPT7, SEPT9, e SEPT11 sono espressi in cellule HeLa) per 30 min (a temperatura ambiente) per tutta la notte (a 4 ° C).
- Lavare le cellule due volte con PBS 1x e incubare in camera umida con anticorpi secondari, ed etichettare actina filamentosa (F-actina) con falloidina, per 30 minuti (a temperatura ambiente) per tutta la notte (a 4 ° C). Per la colorazione dei nuclei delle cellule ospite aggiungono 4 ", 6-diamidino-2-fenilindolo (DAPI).
- Lavare le cellule in PBS 1x e montare i vetrini su vetrini con mezzo di montaggio.
- Imaging al microscopio di cellule HeLa infette
- Per l'immagine infette cellule utilizzano un epifluorescenza o microscopio confocale e un obiettivo 63X o 100X per identificare DAPI-marcato Shigella.
NOTA: Come mostrato nelle figure 1A - 1C, gabbie Septin possono essere visualizzati come anello come strutture, ~ 0,6 micron di diametro, che circonda i batteri citosolici polimerizzazione actina e reclutamento marcatori autofagia (ad esempio, p62 e LC3) 7,8. Al contrario, i batteri polimerizzazione code di actina non saranno compartimenti stagni da gabbie Septin e non saranno finalizzate ad autofagia 7,8. - Utilizzando un epifluorescenza o microscopio confocale e un obiettivo 63X o 100X, quantificare il numero di batteri intracellulari per campo microscopico. Di quantificare anche il numero di batteri intrappolati in gabbie Septin e mirate per l'autofagia, o polimerizzazione code di actina e fuga autofagia.
- Per determinare la percentuale di batteri intrappolati in gabbie Septin o polimerizzazione code di actina, scattare una serie di immagini Z-stack di infeCTED cellule, processo le immagini e contare almeno 500-1.000 batteri per esperimento di almeno 3 esperimenti indipendenti.
- Per l'immagine infette cellule utilizzano un epifluorescenza o microscopio confocale e un obiettivo 63X o 100X per identificare DAPI-marcato Shigella.
2 Analisi funzionale della autofagia e citoscheletro In Vitro
NOTA: Sia la genetica e approcci farmacologici possono essere usati per perturbare l'autofagia nelle cellule di coltura dei tessuti infetti, e l'impatto di questi trattamenti in corso di infezione possono essere monitorati.
- silenziamento siRNA-mediata
- Piatto 0,8 x 10 5 cellule HeLa su vetrini in 6 pozzetti in terreno completo.
- Trasfezione il giorno seguente con un reagente di trasfezione base lipidica con siRNA contro autofagia e / o marcatori citoscheletro.
- Dopo il periodo desiderato di incubazione, di infettare le cellule con Shigella come descritto nella sezione 1.3.
- Fissare ed etichettare le cellule come descritto nella sezione 1.4.
- Manipolazione farmacologica
NOTA: Il citoscheletro può essere manipolato farmacologicamente, ad esempio, per depolimerizzano l'actina uso citoscheletro citocalasina D o latrunculin B, a depolimerizzano microtubuli utilizzare nocodazole, per bloccare l'attività actomiosina uso blebbistatin, o di distruggere Septin uso assemblaggio forchlorfeneuron. L'autofagia può essere stimolata mediante rapamicina o bloccato tramite bafilomycin.- Per manipolare il citoscheletro durante l'infezione Shigella, prima di infettare le cellule con Shigella come descritto nella sezione 1.3 e lasciare il tempo sufficiente per i batteri di entrare nelle cellule e la fuga dal phagosome al citosol (ad es.,> 1,5 infezione post hr).
- Diluire la droga dalla soluzione di [soluzione madre sospesi in dimetilsolfossido (DMSO)] in MEM ad una concentrazione finale di 5 M (citocalasina D, latrunculin B, nocodazole), 20 M (forchlorfeneuron) o 50 M (blebbistatin), e il trattamento di cellule di 30 minuti a 37 ° C. La quantità totale di farmaco (volume di DMSO / dmiscela tappeto) aggiunto per piastra / flacone di cellule è di 1-5 ml (a seconda della soluzione stock) per 2 ml di media. Trattare cellule con una dose simile di DMSO diluita in MEM come controllo negativo.
- Fissare ed etichettare le cellule come descritto nella sezione 1.4.
NOTA: Per la manipolazione del flusso autofagica (nelle cellule infette o non infette), estendere il trattamento farmacologico con rapamicina (20 nM) o bafilomycin (160 nM) dal 4 al 12 hr.
- Western Blot
NOTA: l'attività autofagica può essere quantificato misurando il livello di proteina di marcatori autofagia, come p62 e LC3.- Dopo il periodo desiderato di incubazione, raccogliere e lisare le cellule per immunoblotting. Estratti proteici eseguito su 8, 10, o 14% gel di acrilamide.
- Flusso autophagic, vale a dire. il tasso di autofagia, può essere analizzato come descritto in 21,22.
- Imaging microscopica e quantificazione
- Utilizzando un epifluorescenza o microscopio confocale e un 100X oggetto 63X oive, l'effetto di siRNA o trattamento farmacologico sulla Shigella infezione può essere valutata mediante microscopia quantitativa (cioè, contando di autofagosomi, gabbie Septin e code di actina), come descritto nella sezione 1.5 e illustrato nelle figure 2A e 2B.
3. imaging in vivo di S. flexneri Interazioni con autofagia e il citoscheletro
NOTA: Il modello di zebrafish di infezione da Shigella possono essere utilizzati per studiare Septin ingabbiamento e dell'autofagia in vivo 19.
- Preparare S. flexneri
- Cultura S. flexneri come descritto nella sezione 1.1.
- A OD 600 = 0,3-0,6, rotazione 8 ml sottocultura batterica a 1000 xg per 10 min. Lavare il pellet con 1x PBS e centrifugare a 1000 xg per 10 min.
- Risospendere il pellet in 80 ml di 0,1% rosso fenolo 1x PBS per ottenere ~ 2.000 batteri / nl. Tenere il BacTpreparazione erial sul ghiaccio per rallentare la crescita.
NOTA: L'aggiunta di rosso fenolo aiuterà a visualizzare l'inoculo quando si inietta in larve.
- Preparare Zebrafish larve per iniezione
NOTA: Zebrafish sono definiti come le uova e sono identificati come gli embrioni fino a 72 ore dopo la fecondazione, quando sono chiamati larve.- Razza zebrafish adulto come descritto in Westerfield 23 mettendo 4 maschi e 8 femmine (di solito un rapporto di 2: 1) in un acquario separato con il fondo ricoperto di marmi (che impedirà gli adulti di mangiare le uova deposte). In alternativa, posizionare cestini raccolta delle uova all'interno delle vasche di allevamento la sera prima.
NOTA: Le uova vengono fecondate ~ 30 minuti dopo che le luci si accendono nella struttura zebrafish 23, e devono essere raccolti il prima possibile per evitare la crescita di muffe. Cestini di raccolta delle uova servono a raccogliere le uova in modo che possano essere facilmente raccolti e anche proteggere le uova dagli adulti. - Raccogliere l'embriones e pulirli con il lavaggio in mezzi embrione (E2) con il 0,003% di candeggina per 10 min. Rimuovere E2 con candeggina, lavare gli embrioni 5x in E2 media, e far crescere gli embrioni a 10 centimetri scatola di Petri (100 embrioni / 50 ml E2 medio) a 28 ° C.
- Se saranno utilizzati embrioni o larve per gli studi di microscopia, a 24 ore dopo la fecondazione aggiungere 0,003% N-phenylthiourea al mezzo E2 per evitare melanizzazione. Mantenere gli embrioni a 28 ° C per il normale sviluppo.
NOTA: larve Zebrafish sono pronti per l'infezione a 72 ore dopo la fecondazione. - Per le procedure di infezione e di microscopia, anestetizzare larve di zebrafish in 200 g / ml tricaine in E2.
- Razza zebrafish adulto come descritto in Westerfield 23 mettendo 4 maschi e 8 femmine (di solito un rapporto di 2: 1) in un acquario separato con il fondo ricoperto di marmi (che impedirà gli adulti di mangiare le uova deposte). In alternativa, posizionare cestini raccolta delle uova all'interno delle vasche di allevamento la sera prima.
- Preparazione di Zebrafish larve per via endovenosa e infezione locale
NOTA: Per valutare la sopravvivenza zebrafish durante l'infezione Shigella, effettuare iniezioni endovenose caudale. Per visualizzare il reclutamento di Septin e autofagia marcatori per Shigella, effettuare infezione in siti localizzati, come il muscolo coda.- Per un'iniezione endovenosa caudale, posizionare le larve anestetizzato lateralmente dal lato dorsale rivolta verso l'ago. Come mostrato in Figura 3A, posizionare la stretta punta dell'ago (posteriore) per l'apertura urogenitale, puntando la vena caudale, e perforare la pelle e fornire la dose batterica desiderata (volume di iniezione 1-5 nl).
NOTA: l'infezione per via endovenosa è difficile da eseguire e vorranno diverse settimane di formazione per acquisire familiarità con questa procedura. Iniezione di rosso fenolo (senza batteri) per la formazione contribuirà a valutare il sito di iniezione correttamente.
NOTA: Nel caso di Shigella, esperimenti dose-dipendente hanno dimostrato che una infezione dose bassa (<1.000 CFU) viene cancellata entro 48 ore, e una infezione dose elevata (> 4.000 CFU) porta ad ospitare la mortalità entro 48 ore 19. - Per una infezione muscolare coda, posizionare le larve anestetizzato come descritto nella sezione 3.3.1. Come mostrato in Figura 3A, posizionare la attenzione agoy su somiti muscolari (ad esempio, segmenti di muscolo scheletrico) e iniettare un piccolo volume (ad esempio., 1 nl) di preparazione batterica.
- Per un'iniezione endovenosa caudale, posizionare le larve anestetizzato lateralmente dal lato dorsale rivolta verso l'ago. Come mostrato in Figura 3A, posizionare la stretta punta dell'ago (posteriore) per l'apertura urogenitale, puntando la vena caudale, e perforare la pelle e fornire la dose batterica desiderata (volume di iniezione 1-5 nl).
- Iniezione endovenosa di batteri in larve
- Tirare microcapillari di vetro borosilicato, come descritto in 24.
- Collegare l'ago al titolare del tridimensionale grossolana manipolatore manuale e rompere la punta dell'ago con le pinzette sottili.
- Per caricare l'ago, una goccia di coltura batterica su un vetrino coprioggetti. Accendere il cilindro microinjector e del gas, leggermente immergere la punta dell'ago nella goccia, e riempire l'ago con la quantità desiderata di preparazione batterica.
- Per calibrare il volume di iniezione, una goccia di olio minerale su un vetrino e iniettare il preparato batterico. Misurare il diametro della goccia utilizzando un micrometro e calcolare il volume iniettato [V = (4/3) πr 3].
NOTA: Utilizzando le impostazioni di iniezione di 40 psi e 50 msec con un bacterial preparato come descritto nella sezione 3.1 darà ~ 2.000 CFU / nl. - Preparare la piastra di iniezione utilizzando uno stampo di plastica, come descritto in Westerfield 23.
- Trasferire le larve alla piastra di iniezione e allinearli con un pennello fine. Orientare e iniettare le larve come descritto nella sezione 3.3.1.
- Per la valutazione della sopravvivenza zebrafish, trasferimento infetti larve singolarmente in piastre da 24 pozzetti in 1 ml di E2 / pozzetto e incubare a 28 ° C. Monitorare le larve infette al giorno per i prossimi 2-5 giorni e la sopravvivenza trama nel tempo (Figura 3B).
- Placcatura Zebrafish larve per la quantificazione batterica
NOTA: Lavorare sotto una cappa sterile per evitare contaminazioni.- Per valutare il numero di batteri iniettati nel pesce (al tempo 0 ore dopo l'infezione) e per la quantificazione batterica in momenti desiderati, sacrificare larve di zebrafish con una dose eccessiva di tricaine (200-500 mg / L). Mettere larve individuo in 1,5 ml polprovetta ypropylene con 200 ml di 0,1% ottilfenolo ossido di etilene condensa 1x PBS e omogeneizzare meccanicamente utilizzando un pestello.
NOTA: Per verificare la carica batterica del volume di iniezione, pompa una dose pari in una sterile 1x PBS goccia e piatto fuori. - Effettuare una diluizione seriale dei omogenati larve di zebrafish in acqua sterile e piastra su lisogenia brodo (LB) piastre di agar. Le larve può essere placcato in alternativa su piastre Congo Red TCS di discriminare tra Shigella aver mantenuto il plasmide virulento o meno.
NOTA: Tavola 3 o più pesci non infetto come controllo per verificare lo stato delle larve di zebrafish utilizzato per l'infezione. - Dopo una notte di incubazione delle piastre a 37 ° C, contare le colonie batteriche. Come mostrato nella Figura 3C, rappresentare utilizzando una scala logaritmica.
NOTA: carica batterica durante l'infezione delle larve di zebrafish può essere visualizzato utilizzando fluorescente Shigella ed i microscopicamante come descritto nella sezione 3.7 o 3.8 (Figura 3D).
- Per valutare il numero di batteri iniettati nel pesce (al tempo 0 ore dopo l'infezione) e per la quantificazione batterica in momenti desiderati, sacrificare larve di zebrafish con una dose eccessiva di tricaine (200-500 mg / L). Mettere larve individuo in 1,5 ml polprovetta ypropylene con 200 ml di 0,1% ottilfenolo ossido di etilene condensa 1x PBS e omogeneizzare meccanicamente utilizzando un pestello.
- Zebrafish larve Immunostaining
- A intervalli di tempo desiderati, sacrificare le larve con una dose eccessiva di tricaine. Raccogliere il pesce in provette da 1,5 ml in polipropilene per microcentrifuga (10 a 20 larve / tubo).
- Fissare le larve con paraformaldeide al 4% con il 0,4% ottilfenolo ossido di etilene condensa in 1x PBS e incubare in un agitatore orbitale (per evitare il clustering larvale) per 2 ore (a temperatura ambiente) o durante la notte (a 4 ° C).
NOTA: La microscopia elettronica può essere utilizzata per le analisi ultrastrutturali di infetti larve zebrafish. In questo caso, gli embrioni anestetizzati devono essere fissati e trattati secondo Mostowy et al 19. - Lavare 3x in PBS 1x 0,4% ottilfenolo etilene ossido condensa per 5 min, quindi bloccare in una soluzione bloccante (siero bovino fetale al 10%, 1% DMSO, 0,1% polyoxyethylenesorbitan monolaurato in PBS 1x) per 1 ora a temperatura ambiente.
- Diliuto l'anticorpo primario nel bloccare soluzione. Aggiungere larve di anticorpo primario diluito e incubare una notte a 4 ° C in un agitatore orbitale. Utilizzare anticorpi primari come sopra descritto nella sezione 1.4.3.
- Lavare i 4x larve per 15 min a 0,1% polyoxyethylenesorbitan monolaurato 1x PBS a temperatura ambiente.
- Diluire l'anticorpo secondario nel bloccare soluzione. Aggiungere larve di anticorpo secondario diluito e incubare una notte a 4 ° C in un agitatore orbitale. Utilizzare gli stessi anticorpi secondari e falloidina come descritto nella sezione 1.4.4.
- Lavare 4 volte per 15 min a 0,1% monolaurato polyoxyethylenesorbitan 1x PBS a temperatura ambiente. Per la colorazione dei nuclei delle cellule ospitanti aggiungere DAPI (150 nM concentrazione finale) durante il primo di questi 15 min lavaggi.
- Per la conservazione delle larve fluorescenza marcata, li incubare progressivamente in un gradiente di glicerolo di 15, 30, 60, e il 80% diluito in PBS 1x e 0,1% polyoxyethylenesorbitan monolaurato per 2 ore (a camera temperamentoatura) per tutta la notte (a 4 ° C).
NOTA: Stained larve possono essere conservati per lunghi periodi di tempo in 80% glicerolo a 4 ° C (ad esempio, 4 mesi.).
- Imaging microscopico di Zebrafish fisso larve
- Trasferimento glicerolo fisso larve incorporato per una piccola goccia di 80% glicerolo in un piatto 35 millimetri Petri (per stereomicroscopia) o un piatto fondo di vetro pieno (per miscopy confocale).
- Prendere Z-stack immagine serie di larve infette ed elaborare le immagini come richiesto.
- Utilizzare un epifluorescenza o microscopio confocale e un obiettivo 10X o 20X per l'imaging dell'intero organismo. Quindi utilizzare un microscopio confocale e un obiettivo 40X, 63X, 100X o visualizzare singole celle e il reclutamento di autofagia e citoscheletro marcatori a singoli batteri in vivo 19 (Figure 4A e 4B).
NOTA: Infettare pesci nel muscolo coda e montare in glicerolo pianeggiante lungo il fondo del piatto fondo di vetro per consentire eamessa a fuoco SY.
- Vivere microscopica Imaging di Infected Zebrafish larve
NOTA: Le larve sono otticamente accessibili, quindi in autophagosomes vivo può essere osservata con la GFP-Lc3 zebrafish linea transgenica 25. Infettare larve di zebrafish come descritto nella sezione 3.3 e montare come descritto in questa sezione.- Preparare basso punto di fusione 1% agarosio (LMA) in E2 e lasciare raffreddare a 35-37 ° C per evitare danni larve / uccisione. Distribuire gocce di LMA in un piatto 35 millimetri Petri (per stereomicroscopia) o un piatto fondo di vetro pieno (per microscopia confocale).
- Trasferimento anestetizzato zebrafish larve singolarmente (con meno acqua possibile) per la LMA scende. Larve Orientare nella posizione desiderata con un pennello e attendere che il agarosio solidificare.
- Coprire tutta la superficie piatto con LMA, e overlay con E2 contenente 200 mg / ml tricaine per evitare la preparazione si secchi e per consentire ai pesci di scambiare ossigeno dall'acqua.
- Utilizzare un epifluorescenza o microscopio confocale e un obiettivo 10X o 20X per l'imaging dell'intero larve di zebrafish. Utilizzare un microscopio confocale e un obiettivo 40X, 63X, 100X o visualizzare authopagosomes batteriche (ad esempio, GFP-Lc3 + ve vacuoli circostanti Shigella) in vivo.
- Dai Z-pila di larve infetto nel tempo (ad es., Ogni 2 min su più hr) per visualizzare autophagosomes, e la loro dinamica, in tempo reale.
4 Analisi funzionale di autofagia e citoscheletro In Vivo
NOTA: L'impatto delle perturbazioni farmacologici e genetici del autofagia sul decorso dell'infezione possono essere monitorati a livello di intero animale, ed a livello di singola cellula.
- L'autofagia manipolazione da Morpholino Injection
- Ricostituire morpholino oligonucleotidi in acqua sterile ad una soluzione stock di 1 mM riscaldando a 65 ° C per 10 min. Negozioa temperatura ambiente.
NOTA: morpholino iniezioni oligonucleotidi devono essere eseguite in 1-4 embrioni stadio delle cellule. - Preparare la soluzione di lavoro morpholino oligonucleotide con sterile allo 0,1% di rosso fenolo in tampone fosfato di Dulbecco. Caricare l'ago come descritto nella sezione 3.4.2., Morpholino volume di iniezione oligonucleotide può essere calibrato come descritto nel paragrafo 3.4.3.
NOTA: rosso fenolo aiuterà a visualizzare il volume iniettato. - Preparare una camera di embrione-posizionamento (ad esempio., Un vetrino da microscopio incollato con cianoacrilato su un Petri coperchio 10 centimetri piatto con bordi di fronte l'ago parzialmente rimosso). Trasferire 1-4 embrioni stadio cella alla camera con una piccola quantità di acqua e di allinearle con un pennello fine.
- Penetrare il corion e il tuorlo senza intoppi. Una volta all'interno, premere il pedale per iniettare il volume desiderato di soluzione morfolino oligonucleotidi.
NOTA: Ridurre al minimo il volume di iniezione per 0,5-2 nl; maggiori volumi THAn 5 nl possono causare difetti di sviluppo e aumentare la mortalità delle uova. - Dopo la microiniezione, embrioni puliti (di sbianca come descritto nella sezione 3.2) e incubare in una capsula di Petri con E2 a 28 ° C.
- Infettare 72 ore di controllo dopo la fecondazione (cioè, larve di zebrafish iniettato con controllo morfolino oligonucleotide) o morphants p62 (es., Larve di zebrafish iniettato con p62 morfolino oligonucleotide) con Shigella, come descritto nella sezione 3.4. Valutare la sopravvivenza e la carica batterica per i prossimi 2-5 giorni, come descritto nella sezione 3.5. Immagine fissa larve di zebrafish come descritto nella sezione 3.7 ed evidenziato in Figura 4C, o immagine vivente larve di zebrafish come descritto nella sezione 3.8.
NOTA: La dose efficace morpholino oligonucleotide può essere valutata in base alla sua efficienza di inibire la trascrizione splicing o la traduzione di proteine (vedi discussione).
- Ricostituire morpholino oligonucleotidi in acqua sterile ad una soluzione stock di 1 mM riscaldando a 65 ° C per 10 min. Negozioa temperatura ambiente.
Risultati
Su infezione di cellule di coltura dei tessuti in vitro, S. flexneri può fuoriuscire dalla phagosome e invadere il citosol. Nel citosol, cellule ospiti possono impedire la motilità actina-base di Shigella da compartimenti batteri all'interno gabbie Septin (Figura 1A). I batteri intrappolati da gabbie Septin possono anche essere etichettati con marcatori autofagia p62 (Figura 1B) e LC3 (Figura 1C). Queste osservazioni mettono in luce un nuovo meccanismo di difesa dell'ospite che limita la diffusione di agenti patogeni invasivi, e rivelano anche nuovi collegamenti tra autofagia e il citoscheletro. Sorprendentemente, l'esaurimento dei marcatori autofagia riduce significativamente Septin ingabbiamento di batteri (Figura 2A), e il lavoro ha anche dimostrato che la deplezione di Septin ingabbiamento riduce significativamente l'assunzione di marcatori di autofagia 8. Così, almeno nel caso di Shigella, Septin gruppo telaio e formatio dell'autofagosoman può essere visto come processi interdipendenti. Altri requisiti cellulari per compartimentalizzazione di Shigella da gabbie Septin includono polimerizzazione e l'attività actomiosina (Figura 2B).
Non esiste un modello naturale topo di shigellosi, e ricerca di Shigella patogenesi, Septin biologia e autofagia batterica in vivo possono beneficiare di un nuovo modello animale di infezione, le larve zebrafish 19. E 'possibile infettare larve zebrafish iniettando batteri in varie sedi anatomiche come iniezioni endovenose caudale per esperimenti di sopravvivenza, e iniezioni muscolari coda per microscopia in vivo (Figura 3A). A seconda della dose, S. flexneri iniettato in larve di zebrafish può o essere cancellata entro 48 ore dopo l'infezione, o può provocare una infezione progressiva e infine fatale (Figure 3B - 3D). Shigella virulenza factors sono espresse a 28 ° C, la temperatura di crescita ottimale di zebrafish, e l'infezione da Shigella zebrafish è strettamente dipendente il proprio sistema di secrezione di tipo III (T3SS) 19, un determinante virulenza essenziale nella malattia umana. Nel loro insieme, queste osservazioni indicano che la larva zebrafish rappresenta un nuovo host valido per l'analisi in vivo di infezione da Shigella.
L'accessibilità ottica di larve di zebrafish permette la visualizzazione di Septin ingabbiamento in vivo (Figura 4A), un risultato che non è mai stato realizzato utilizzando modelli ospitanti mammiferi. Per completare la prova che le gabbie Septin intrappolano i batteri mirati per autofagia in vivo, si può infettare larve di zebrafish transgenici che esprimono GFP-Lc3 e osservare marcatore autofagia reclutamento Shigella (Figura 4B). Per l'analisi ultrastrucutral di autofagosomi Shigella in vivo, ELmicroscopia ECTRON può essere usato per mostrare chiaramente il sequestro citosolica dei batteri da vacuoli doppia membrana 19. L'autofagia è visto come una componente chiave della immunità cellulo-autonomo e un meccanismo fondamentale di difesa contro i batteri intracytosolic 14-16. Per caratterizzare la funzione autofagia in vivo, P62 morfolino trattati con larve di zebrafish può essere utilizzato. A differenza delle macchine nucleo autofagia [ad es., 36 autofagia relative proteine (ATGS) 26], P62 non è essenziale per lo sviluppo dei vertebrati 27 e quindi larve di zebrafish può svilupparsi normalmente prima infezione. Sorprendentemente, larve P62-impoverito inoculati con S. risultato flexneri in modo significativo aumento della mortalità e aumento della carica batterica 19. In accordo con il lavoro in vitro dimostrano che l'assemblaggio della gabbia Septin è interdipendente con la formazione dell'autofagosoma 7,8, Septin reclutamento di Shigella è chiaramente ridotta in larve p62-impoverito ( Figura 4C). Questi dati dimostrano che la sopravvivenza zebrafish dipende da p62-mediata autofagia per controllare l'infezione batterica intracellulare.
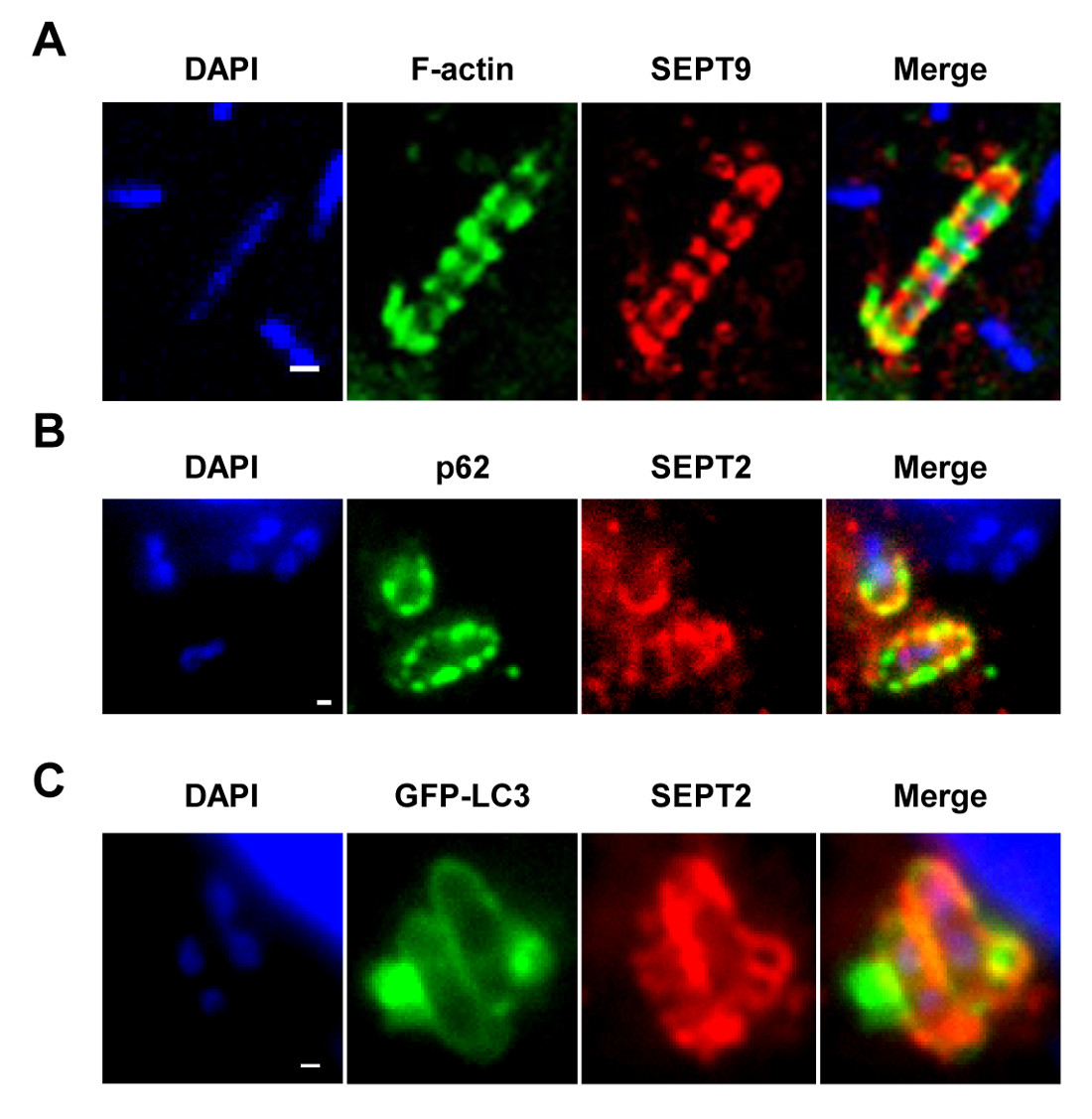
Figura 1 La gabbia Septin in vitro. (A) cellule HeLa sono state infettate con S. flexneri per 4 ore 40 min, fisso, etichettato con anticorpi anti SEPT9 e falloidina, e ripreso da microcopy confocale. Bar Scala, 1 micron. (B), le cellule HeLa sono state infettate con S. flexneri per 4 ore 40 min, fisso, etichettato con anticorpi anti-p62 e SEPT2, e ripreso al microscopio ottico a fluorescenza. Bar Scala, 1 micron. Cellule (C) HeLa sono state trasfettate con GFP-ATG8 / LC3, infettati con S. flexneri per 4 ore 40 min, fisso, marcato con anticorpi anti SEPT2, e ripreso da Mi fluorescentimicroscopia. Bar Scala, 1 micron. Questi dati sono stati modificati da Mostowy et al 7.
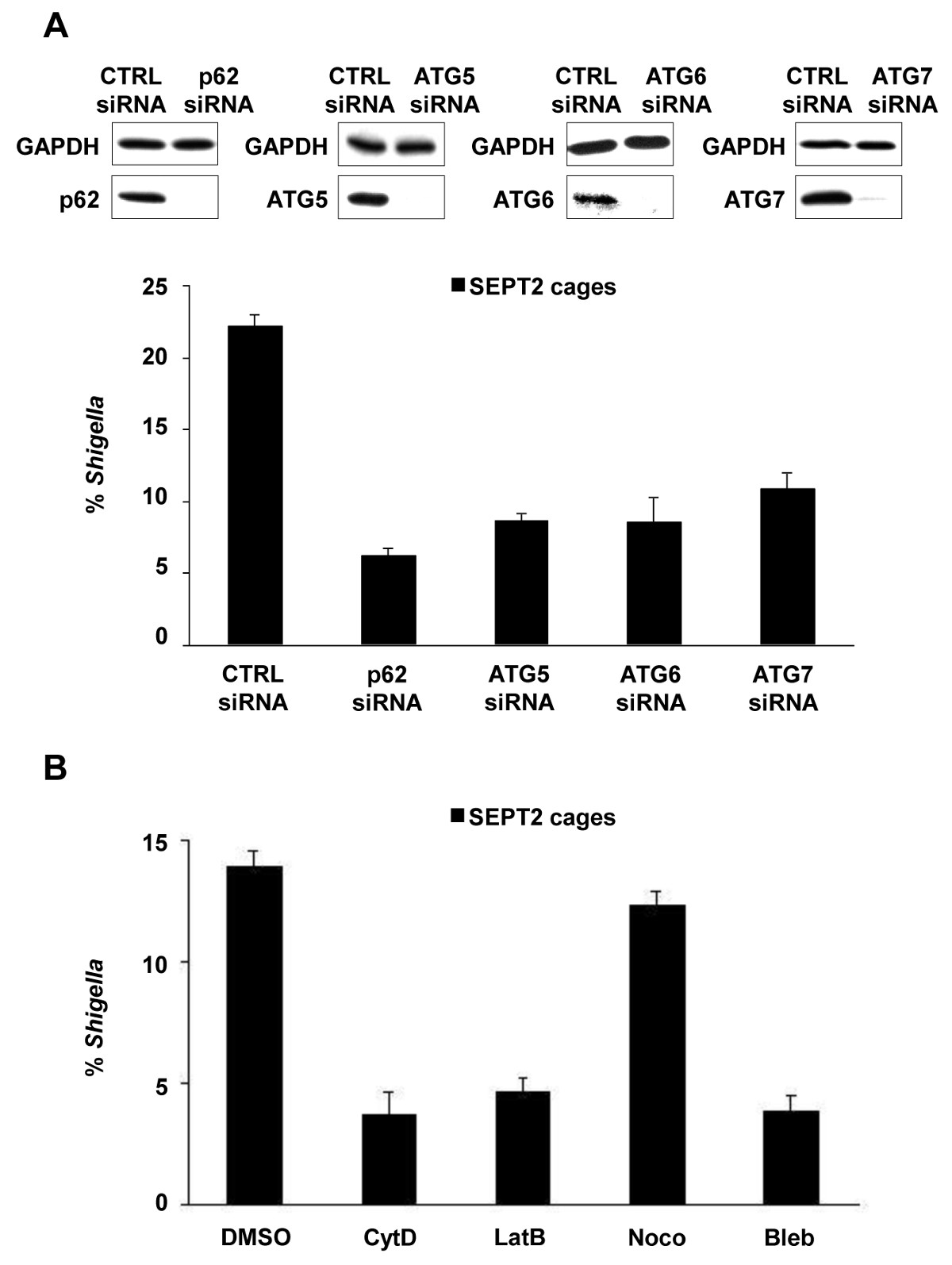
Figura 2. requisiti cellulari per Shigella formazione gabbia -septin. (A) cellule HeLa sono stati trattati con il controllo (CTRL), p62, ATG5, ATG6 o ATG7 siRNA. Lisati Whole-cellulari di siRNA-trattate sono state immunoblotted per GAPDH, p62, ATG5, ATG6, o ATG7 per mostrare l'efficienza dei siRNA esaurimento (in alto). cellule trattate con siRNA sono stati infettati con S. flexneri per 4 ore 40 min, fisso, ed etichettati per la microscopia quantitativa. Grafici (in basso) rappresentano la media ± SEM% di Shigella dentro gabbie SEPT2 da n ≥3 esperimenti per il trattamento. (B) cellule HeLa sono state infettate con S. flexneri, trattati con DMSO, citocalasina D (CytD), latrunculin B (LATB), nocodazole (Noco), o blebbistatin (Bleb) e dopo 4 ore 40 minuti sono stati fissati ed etichettati per la microscopia quantitativa. I grafici rappresentano la media ± SEM% di Shigella dentro gabbie SEPT2 da due esperimenti indipendenti per ogni trattamento. Questi dati sono stati modificati da Mostowy et al 7.
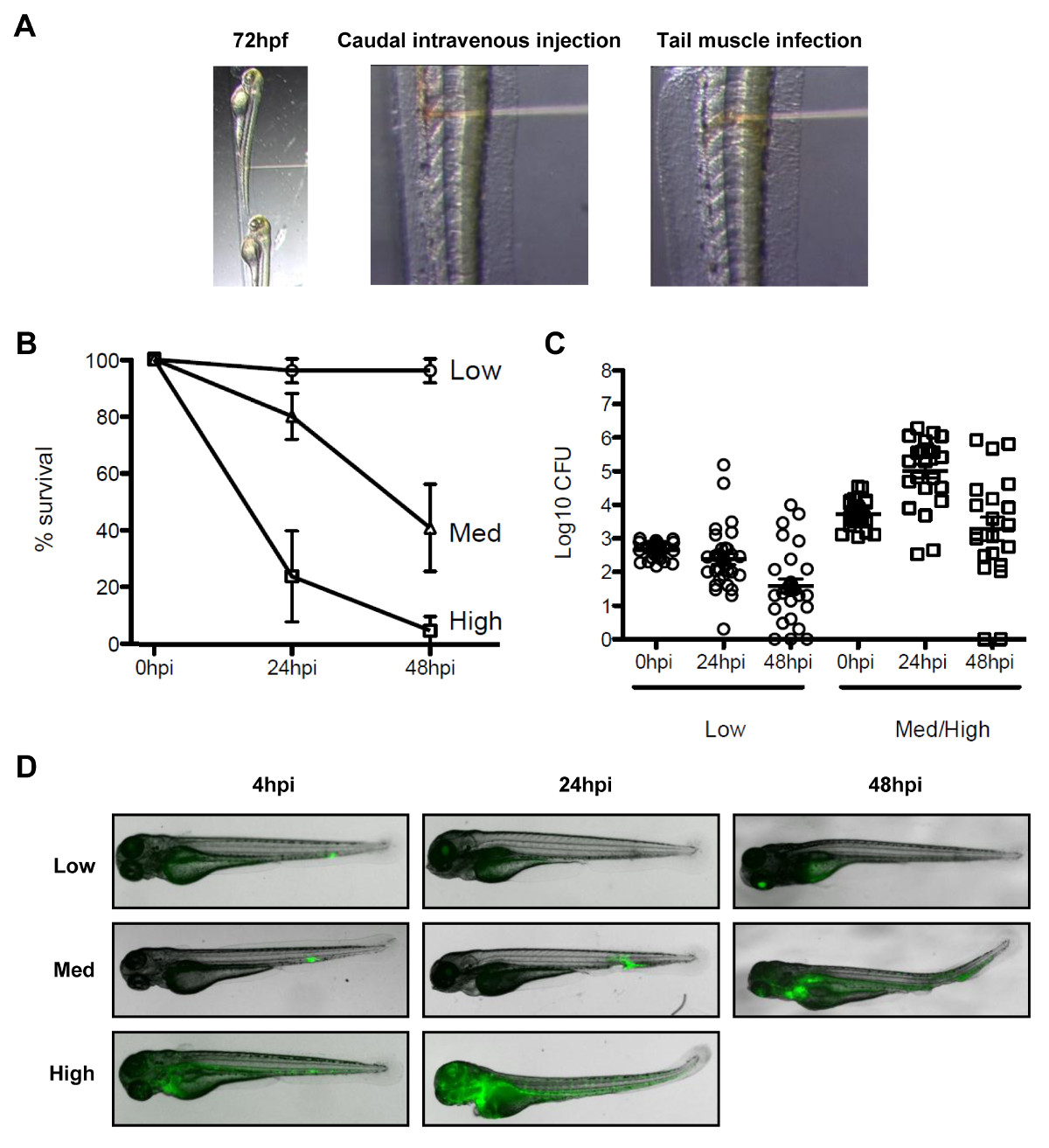
Figura 3 Il modello di zebrafish di infezione Shigella. (A) Immagini per illustrare l'orientamento della larva di zebrafish allo stereomicroscopio. (Pannello di sinistra) Zebrafish larve 72 ore dopo la fecondazione sono stati posizionati lateralmente nella piastra di iniezione con il loro lato dorsale rivolta verso l'ago di iniezione. Infezione (pannello centrale) Flusso sanguigno è stata eseguita da injette i batteri (soluzione rossa) nella vena caudale, posteriormente all'apertura urogenitale. (Pannello di destra) Infezione nel muscolo coda è stata eseguita iniettando i batteri (soluzione rossa) nel corso di un somite. (B) Le curve di sopravvivenza di 72 ore dopo la fecondazione larve iniettati con varie dosi di S. flexneri e incubato a 28 ° C per 48 ore dopo l'infezione. L'inoculo efficace è stato classificato come basso (<10 3 CFU, cerchi aperti), medio (~ 4 x 10 3 CFU, triangoli aperti) o alto (~ 10 4 CFU, piazze). Media% ± SEM (barre orizzontali) da n ≥3 esperimenti per classe inoculo. (C) Enumerazione di batteri vivi in omogenati da larve individuo in tempi diversi dopo l'infezione rilevata dalla placcatura su LB. Nota, solo le larve sopravvissute l'infezione sono inclusi nell'analisi di enumerazione. Media ± SEM (barre orizzontali) anche mostrato. (D) Distribuzione di GFP Shigella determinato daimmagini dal vivo utilizzando uno stereomicroscopio a fluorescenza in vari momenti dopo l'infezione con un basso, medio, o inoculo ad alte dosi (iniezioni endovenose caudale). Sovrapposizione immagine di trasmissione (grigio) e fluorescenza GFP (verde) (B) di -. (D) Questi dati sono stati modificati da Mostowy et al 19.
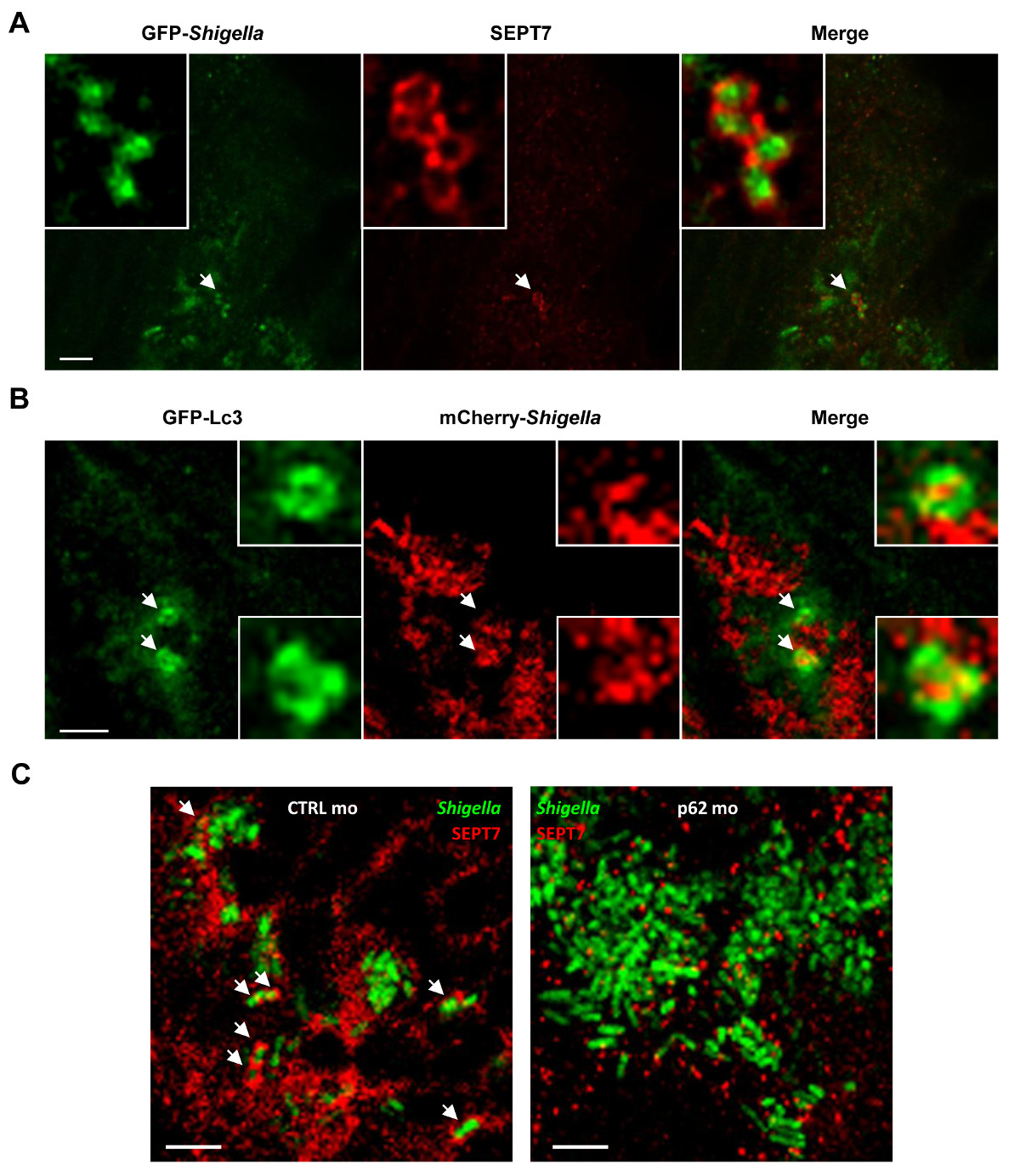
Figura 4 La biologia cellulare di infezione Shigella in vivo. (A) larve Zebrafish sono stati infettati nel muscolo coda con GFP Shigella (basso dosaggio) per 24 ore, fissa, marcato con anticorpi contro SEPT7 (rosso) e di GFP (green ), e ripreso al microscopio confocale. Bar Scala, 5 micron. (B) GFP-Lc3 larve di zebrafish sono stati infettati con mCherry- Shigella (media della dose) per4 ore, fissa, marcato con anticorpi contro mCherry (rosso) e di GFP (verde), e ripreso al microscopio confocale. . Bar Scala, 1.5 micron (C) larve Zebrafish trattati sia con controllo (CTRL; immagine a sinistra) o p62 (immagine a destra) Morpholinos sono stati infettati con GFP Shigella per 4 ore (media della dose), fissa, marcato con anticorpi contro SEPT7 ( rosso) e per GFP (verde), e ripreso al microscopio confocale. Le frecce evidenziano alcuni esempi di Shigella intrappolate in gabbie Septin (CTRL) o non (p62 esaurita) un post infezione 4 ore. Bar Scala, 5 micron. Questi dati sono stati modificati da Mostowy et al 19.
Discussione
Durante il monitoraggio autofagia e il citoscheletro in vitro utilizzando cellule di coltura tissutale, i protocolli descritti nelle sezioni 1 e 2 possono essere applicati a una vasta gamma di tipi di cellule di coltura di tessuti. Inoltre, per seguire autofagia (ad esempio, ATG8 / LC3 + ve autophagosomes) e citoscheletro (ad es., Le code di actina, gabbie Septin) dinamiche in tempo reale durante l'infezione da Shigella utilizzando l'imaging dal vivo, le cellule di coltura tissutale possono essere trasfettate con GFP, costrutti RFP- o CFP-targhette come descritto in precedenza 7,8. Per aumentare la percentuale di cellule infettate da Shigella (vale a dire, in genere auspicabile per l'analisi in tempo reale se si considera che Shigella può invadere 5-30% delle cellule HeLa a 100:. 1 MOI), aggiungere direttamente 400 ml di Shigella (OD 600 = 0.3 -0.6) alle cellule in 2 ml di MEM (siero-fame) e attendere almeno 1,5 ore dopo l'infezione batterica sufficiente per l'ingresso, la fuga dalla phagosome, replica, auriconoscimento tophagy e Septin ingabbiamento. In alternativa, si può usare il ceppo Shigella M90T Afai che esprime la adesina AfaE e sono molto più alte capacità di invasione nelle cellule epiteliali rispetto al ceppo M90T 28. Da segnalare, il ceppo M90T Afai non è ancora stato testato in vivo utilizzando zebrafish. Lastre di colonie Shigella possono essere mantenuti a 4 ° C per 2-3 giorni e utilizzati per diversi esperimenti. Tuttavia, nel corso del tempo, colonie di Shigella che hanno perso il plasmide di virulenza può anche assorbire il Congo rosso e sembra aver mantenuto il loro plasmide di virulenza. Per questo motivo si consiglia di utilizzare ceppi batterici freschi quando possibile.
Quando il monitoraggio della biologia cellulare di infezione in vivo, i protocolli descritti nelle sezioni 3 e 4, uso la linea di tipo selvatico AB zebrafish. Per monitorare Shigella interazioni -leukocyte, linee di zebrafish transgenici possono essere utilizzate, ad esempio, mpx: GFP o lyz: DsRed a Visualize neutrofili 19,29,30 o mpeg1: mCherry per visualizzare macrofagi 19,31. Per visualizzare l'autofagia in vivo, la GFP-Lc3 zebrafish linea transgenica 19,24 può essere utilizzato come descritto nella sezione 3.8.
Per perturbare l'autofagia in vivo, la dose efficace morfolino oligonucleotidi deve essere valutata sperimentalmente in base alla sua efficienza di inibire la trascrizione splicing o la traduzione della proteina. Si consiglia di eseguire un esperimento di titolazione e per confermare l'esaurimento mediante RT-PCR (per splice morfolino oligonucleotidi) o mediante SDS-PAGE (per traslazionale morfolino oligonucleotidi) 32. RNA isolamento da embrioni di zebrafish o larve può essere effettuata utilizzando l'estrazione guanidina tiocianato-fenolo-cloroformio. Per estrarre proteine da larve di zebrafish (da 8 a 15 larve / tubo), omogeneizzare meccanicamente con un pestello in 200 microlitri tampone di lisi (1 M Tris, NaCl 5 M, 0,5 M EDTA, 0,01% ottilfenolo ossido di etilene condensate, e inibitore della proteasi). Tubi centrifugare a 19.000 xg a 4 ° C per 15 minuti e trasferire il surnatante in una nuova provetta. Aggiungere tampone Laemmli e riscaldare il campione a 95 ° C per 15 min. Lisati possono essere conservati a -80 ° C fino al momento, e possono essere valutati mediante Western blotting come descritto nella sezione 2.3.
Il pesce zebra è un modello eccellente per applicazioni in vivo di droga. Analisi mediante oligonucleotidi morpholino può essere integrata con farmaci stabiliti per manipolare autofagia (ad esempio, rapamicina e bafilomycin). Larve non infette e / o infetti può essere trattata con rapamicina (1,5 mM) o bafilomycin (80 nM) diluito in E2 e flusso autofagica può essere valutata mediante Western blotting come descritto in 25,33. La conseguenza della manipolazione autofagia sul risultato dell'infezione e la sopravvivenza delle larve infetto può essere valutata come descritto nella sezione 3.5.
Oltre allo studio della cellula ospitedeterminanti, in vitro ed in vivo protocolli possono essere applicati per valutare i determinanti batterici necessari per il riconoscimento autofagia, utilizzando ceppi mutanti batterici che sono differenzialmente riconosciute da autofagia, ad es., Shigella ΔicsA (la proteina Shigella IcsA recluta N-WASP e poi Arp2 / 3 per actina coda e formazione gabbia Septin, in sua assenza, non ci possono essere code di actina, senza gabbie Septin) e ShigellaΔicsB (Shigella evita la risposta autofagica tramite la proteina batterica ICSB effettrici, che impedisce l'assunzione di macchinari autofagia per IcsA, in sua assenza ci possono essere più gabbie Septin, più autofagia) 7,8.
Shigella non è un patogeno naturale di zebrafish e cresce in modo ottimale a 37 ° C. Tuttavia, il lavoro ha dimostrato che i fattori di virulenza necessari per Shigella invasione, fuggire dal vacuolo di fagocitosi e la replica nel cytosol può essere espresso e sono funzionali in larve di zebrafish a 28 ° C 19. 28 ° C è la temperatura più comunemente usato per l'allevamento zebrafish e temperatura standard per garantire lo sviluppo di zebrafish normale 23. Sorprendentemente, i principali eventi patogenetici che portano alla shigellosi negli esseri umani (cioè, la morte cellulare dei macrofagi, invasione e moltiplicazione all'interno delle cellule epiteliali, la diffusione cellula-cellula, distruzione infiammatoria del dell'epitelio ospitante) sono fedelmente riprodotti nel modello di zebrafish di infezione da Shigella 19.
Autofagia e citoscheletro geni sono espressi ubiquitariamente e hanno una vasta gamma di funzioni biologiche. Studi hanno dimostrato che mouse knockout di autofagia essenziali 26 o Septin geni 5 sono embrionale letale, ed è probabile che alcuni di questi geni sarà anche essenziale per lo sviluppo di zebrafish (anche se questo problema può essere ridotto dal fatto che il pesce zebra hanno molteplicigeni paraloghi 33). Se è così, ci sono diverse alternative per superare questo problema, tra cui (i) l'uso di reagenti farmacologiche per regolare l'autofagia e il citoscheletro, (ii) Morpholinos possono essere titolati in basso, (iii) knockout di geni può essere progettato solo per specifica cella tipi, e / o (iv) geni coinvolti nel riconoscimento autofagica che non sono essenziali per lo sviluppo degli animali (ad es., P62) possono essere mirati.
Mentre il pesce zebra è un sistema modello ideale per studiare l'autofagia e il citoscheletro durante l'infezione Shigella, strumenti molecolari attualmente mancano. Il campo ha bisogno di generare nuovi strumenti e cellulare in auto specifica espressione delle proteine di interesse. Per abbattere l'espressione di geni dell'autofagia / citoscheletro, nuove sequenze morpholino sono necessari, e di nuovi metodi per l'ingegneria del genoma (ad esempio, TALEN, CRISPR / Cas9) possono anche essere utilizzati. Nel frattempo, diversi strumenti precedentemente generati per gli studi umani o del mousepossono ugualmente funzionare per zebrafish.
I batteri intracellulari S. flexneri è emerso come un organismo modello eccezionale per affrontare le questioni fondamentali in biologia, tra cui la capacità dei batteri di essere riconosciuto dal 1,2 del sistema immunitario. La cellula ospite impiega septins per limitare la motilità di S. flexneri e la loro destinazione ad autofagia, una componente critica di cellule dell'immunità autonoma 7,8. Queste osservazioni suggeriscono un nuovo quadro molecolare per studiare l'autofagia e la sua capacità di degradare batteri citosolici. Una questione importante è ora quello di decifrare pienamente gli eventi molecolari e cellulari alla base, e per convalidare questi eventi analizzati in vitro durante l'infezione batterica in vivo su modelli animali pertinenti. A tal fine, il pesce zebra è stato stabilito come un nuovo ospite prezioso per l'analisi di S. infezione flexneri 19. Le interazioni tra batteri e cellule ospiti possono essere esposte ad alta risoluzione, eil modello di zebrafish dovrebbe rivelarsi utile per comprendere la biologia cellulare di infezione Shigella in vivo. Larve Zebrafish può essere utilizzato per studiare il ruolo dell'autofagia batterica in difesa ospite, e il lavoro ha dimostrato che la perturbazione di autofagia può influenzare negativamente la sopravvivenza di accoglienza in risposta alle infezioni Shigella 19.
Le osservazioni generate dallo studio di Shigella, Septin ingabbiamento e autofagia in vitro utilizzando colture cellulari e in vivo utilizzando larve di zebrafish potrebbe fornire progressi fondamentali nella comprensione difesa ospite. Si potrebbe anche suggerire lo sviluppo di nuove strategie volte a combattere le malattie infettive.
Un obiettivo fondamentale di questo rapporto è quello di dare un senso degli eventi molecolari e cellulari analizzati in vitro (cioè, l'autofagia, code di actina, Septin ingabbiamento) durante l'infezione batterica in vivo, nel contesto di un entorganismo ire, utilizzando larve di zebrafish. Se non ha familiarità con la biologia e la manipolazione zebrafish, si può fare riferimento ai protocolli di profondità per un corretto allevamento zebrafish 23 e in analisi in vivo di infezione zebrafish 19,35.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari in competizione.
Riconoscimenti
Il lavoro in laboratorio SM è supportato da un Wellcome Trust Research Career Development Fellowship [WT097411MA].
Materiali
| Name | Company | Catalog Number | Comments |
| Bafilomycin A1 | Sigma-Aldrich | B1793 | |
| Blebbistatin | Sigma-Aldrich | B0560 | |
| Borosilicate glass microcapillars | Harvard Apparatus | 30038 | |
| Coarse manual manipulator | Narishige | M-152 | |
| Cytochalasin D | Sigma-Aldrich | C6762 | |
| 4',6-diamidino-2-phenylindole (DAPI) | Molecular Probes | D1306 | |
| Dumont #5 fine tweezers | Fine Science Tools | 11254-20 | |
| Forchlorfeneuron | Sigma-Aldrich | 32974 | |
| Goat anti-mouse IgG (H+L) antibody | Molecular Probes | N/A | |
| Goat ant-rabbit IgG (H+L) antibody | Molecular Probes | N/A | |
| JetPEI transfection reagent | Polyplus transfection | 101-01N | |
| Latrunculin B | Sigma-Aldrich | L5288 | |
| LC3 antibody | Novus Biologicals | NB100-2220 | |
| Low melting agarose | Promega | V2111 | |
| MatTek glass bottom dish | MatTek corporation | P35G-1.0-14 | |
| MEM plus L-alanyl-L-glutamine | GIBCO | 41090028 | |
| MEM non-essential amino acids solution | GIBCO | 11140-035 | |
| Microinjector | Narishige | IM-300 | |
| Micropipette puller device | Sutter Instrument Co., Novato, | P-87 | |
| Mineral oil | Sigma-Aldrich | P35G-1.0-14 | |
| Monoclonal anti-tubulin, acetylated antibody | Sigma-Aldrich | T6793 | |
| Nocodazole | Sigma-Aldrich | M1404 | |
| N-phenylthiourea | Sigma-Aldrich | P7629 | |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Phalloidin | Molecular Probes | A12379 | |
| Phenol red solution | Sigma-Aldrich | P0290 | |
| Protease inhibitor cocktail | Roche | 4693116001 | |
| p62/SQSTM1 antibody | Cliniscience | PM045 | |
| Rapamycin | Sigma-Aldrich | R8781 | |
| Sodium pyruvate | GIBCO | 11360039 | |
| Transfection reagent | Life Technologies | 12252-011 | |
| Vectashield hard set mounting medium containing DAPI | Vector Laboratories | H-1500 |
Riferimenti
- Ashida, H., et al. Shigella are versatile mucosal pathogens that circumvent the host innate immune system. Curr Opin Immunol. 23, 448-455 (2011).
- Phalipon, A., Sansonetti, P. J. Shigella's ways of manipulating the host intestinal innate and adaptive immune system: a tool box for survival. Immunol Cell Biol. 85, 119-129 (2007).
- Welch, M. D., Way, M. Arp2/3-mediated actin-based motility: A tail of pathogen abuse. Cell Host Microbe. 14, 242-255 (2013).
- Haglund, C. M., Welch, M. D. Pathogens and polymers: microbe-host interactions illuminate the cytoskeleton. J Cell Biol. 195, 7-17 (2011).
- Mostowy, S., Cossart, P. Septins: the fourth component of the cytoskeleton. Nat Rev Mol Cell Biol. 13, 183-194 (2012).
- Saarikangas, J., Barral, Y. The emerging functions of septins in metazoans. EMBO Rep. 12, 1118-1126 (2011).
- Mostowy, S., et al. Entrapment of intracytosolic bacteria by septin cage-like structures. Cell Host Microbe. 8, 433-444 (2010).
- Mostowy, S., et al. p62 and NDP52 proteins target intracytosolic Shigella and Listeria to different autophagy pathways. J Biol Chem. 286, 26987-26995 (2011).
- Mostowy, S., et al. Septins regulate bacterial entry into host cells. PLoS One. 4 (15), (2009).
- Mostowy, S., et al. Septin 11 restricts InlB-mediated invasion by Listeria. J Biol Chem. 284, 11613-11621 (2009).
- Phan, Q. T., et al. Role of endothelial cell Septin 7 in the endocytosis of Candida albicans. mBio. 4 (e00542-13), (2013).
- Mostowy, S., Cossart, P. Septins as key regulators of actin based processes in bacterial infection. Biol Chem. 392, 831-835 (2011).
- Levine, B., Mizushima, N., Virgin, H. W. Autophagy in immunity and inflammation. Nature. 469, 323-335 (2011).
- Randow, F., MacMicking, J. D., James, L. C. Cellular self-defense: how cell-autonomous immunity protects against pathogens. Science. 340, 701-706 (2013).
- Mostowy, S. Autophagy and bacterial clearance: a not so clear picture. Cell Microbiol. 2 (12063), (2012).
- Mostowy, S., Cossart, P. Bacterial autophagy: restriction or promotion of bacterial replication. Trends Cell Biol. 22, 283-291 (2012).
- Kanther, M., Rawls, J. F. Host-microbe interactions in the developing zebrafish. Curr Opin Immunol. 22, 10-19 (2010).
- Renshaw, S. A., Trede, N. S. A model 450 million years in the making: zebrafish and vertebrate immunity. Dis Model Mech. 5, 38-47 (2012).
- Mostowy, S., et al. The zebrafish as a new model for the in vivo study of Shigella flexneri interaction with phagocytes and bacterial autophagy. Plos Path. 9, e1003588 (2013).
- Fischer, A. H., Jacobson, K. A., Rose, J., Zeller, R. Fixation and permeabilization of cells and tissues. Cold Spring Harb Protoc. , (2008).
- Klionsky, D. J., et al. Guidelines for the use and interpretation of assays for monitoring autophagy. Autophagy. 8, 445-544 (2012).
- Mizushima, N., Yoshimori, T., Levine, B. Methods in mammalian autophagy research. Cell. 140, 313-326 (2010).
- Westerfield, M. A guide for the laboratory use of zebrafish (Danio rerio). The Zebrafish Book. , (2000).
- Benard, E. L., et al. Infection of zebrafish embryos with intracellular bacterial pathogens. J Vis Exp. 61 (e3781), (2012).
- He, C., Bartholomew, C. R., Zhou, W., Klionsky, D. J. Assaying autophagic activity in transgenic GFP-Lc3 and GFP-Gabarap zebrafish embryos. Autophagy. 5 (4), 520-526 (2009).
- Mizushima, N., Komatsu, M. Autophagy: renovation of cells and tissues. Cell. 147, 728-741 (2011).
- Komatsu, M., et al. Homeostatic levels of p62 control cytoplasmic inclusion body formation in autophagy-deficient mice. Cell. 131, 1149-1163 (2007).
- Nowicki, B., Coyne, K. E., Lublin, D. M., Nowicki, S., Hart, A. Short consensus repeat-3 domain of recombinant decay-accelerating factor is recognized by Escherichia coli recombinant Dr adhesin in a model of a cell-cell interaction. J Exp Med. 178, 2115-2121 (1993).
- Hall, C., Flores, M., Storm, T., Crosier, K., Crosier, P. The zebrafish lysozyme C promoter drives myeloid-specific expression in transgenic fish. BMC Developmental Biology. 7 (42), (2007).
- Renshaw, S. A., et al. A transgenic zebrafish model of neutrophilic inflammation. Blood. 108, 3976-3978 (2006).
- Ellett, F., Pase, L., Hayman, J. W., Andrianopoulos, A., Lieschke, G. J. mpeg1 promoter transgenes direct macrophage-lineage expression in zebrafish. Blood. 117, 2010-2010 (2011).
- Bill, B. R., Petzold, A. M., Clark, K. J., Schimmenti, L. A., Ekker, S. C. A primer for morpholino use in zebrafish. Zebrafish. 6 (9), (2009).
- He, C., Klionsky, D. J. Analyzing autophagy in zebrafish. Autophagy. 6, 642-644 (2010).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 25 (496 (7446)), 498-503 (2013).
- Levraud, J. P., Colucci-Guyon, E., Redd, M. J., Lutfalla, G., Herbomel, P. In vivo analysis of zebrafish innate immunity. Methods Mol Biol. 415, 337-363 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon