È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
A High Throughput, Multiplexer e mirata Proteomica CSF test per quantificare neurodegenerative Biomarkers e apolipoproteina E Isoforme Stato
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Descriviamo un alto rendimento, multiplex, e il dosaggio mirato liquido cerebrospinale proteomica (CSF) sviluppata con un potenziale per la traduzione clinica. Il test può quantificare potenziali marcatori e fattori di rischio per la neurodegenerazione, come le varianti apolipoproteina E (E2, E3 e E4), e misurarne l'espressione allelica.
Abstract
Molte malattie neurodegenerative mancano ancora trattamenti efficaci. biomarcatori affidabili per l'identificazione e la classificazione di queste malattie saranno importanti per lo sviluppo di future terapie innovative. Spesso i potenziali nuovi biomarcatori non rendono in clinica a causa di limitazioni nel loro sviluppo e costi elevati. Tuttavia, la proteomica mirati che utilizzano reazione multipla Monitoraggio cromatografia liquida-tandem / Spettrometria di Massa (MRM LC-MS / MS), in particolare utilizzando triple spettrometri di massa a quadrupolo, è un metodo che può essere utilizzato per valutare rapidamente e validare biomarcatori per la traduzione clinica in laboratori diagnostici . Tradizionalmente, questa piattaforma è stato ampiamente utilizzato per la misurazione di piccole molecole in laboratori clinici, ma è la possibilità di analizzare le proteine, che lo rende una valida alternativa ai ELISA (Enzyme-Linked Immunosorpiegato Assay) a base di metodi. Descriviamo qui come proteomica mirata può essere utilizzato per misurare i marcatori multiplex di demenza, tra cui il rilevamento e la quantificazione del noto fattore di rischio apolipoproteina E isoforma 4 (ApoE4).
Al fine di rendere il saggio adatto per definizione, è progettato per essere rapido, semplice, altamente specifico e redditizio. Per raggiungere questo obiettivo, ogni passo nello sviluppo del dosaggio deve essere ottimizzato per le singole proteine e dei tessuti che sono analizzate in. Il metodo descrive un flusso di lavoro tipico tra cui vari suggerimenti e trucchi per lo sviluppo di una proteomica mirata MRM LC-MS / MS per la traduzione .
Lo sviluppo del metodo è ottimizzato utilizzando le versioni sintetizzate personalizzato di peptidi quantotypic tryptic, che calibrare la MS per il rilevamento e poi a spillo in CSF per determinare la corretta identificazione del peptide endogena nella separazione cromatografica prima dell'analisi nel MS. Per raggiungere absoluTE quantificazione, versioni standard interni marcati con isotopi stabili dei peptidi con breve aminoacidi tag sequenza di acido e che contiene un sito di taglio tripsina, sono inclusi nel saggio.
Introduzione
L'impatto crescente delle malattie neurodegenerative come il morbo di Alzheimer, demenza Lewy e la malattia di Parkinson, sta diventando un problema socio-economico in molti paesi 1. Vi è la necessità di biomarker aggiuntivi che possono essere utilizzati per identificare e classificare i pazienti nei primi stadi della malattia, e per monitorare eventuali nuovi trattamenti. L'obiettivo generale di questo metodo è quello di creare una pipeline generico per un modo semplificato, economico e più veloce di validare potenziali marcatori CSF di neurodegenerazione. La logica è quella di utilizzare la proteomica mirati o peptide MRM LC-MS / MS come un metodo facilmente modificabili per valutare molteplici potenziali biomarcatori proteici da esperimenti di scoperta. Questi possono essere ulteriormente multiplexing su una separazione rapida cromatografica (<10 min) e valutati. All'interno di questa schermata multiplex per biomarcatori neurodegenerative, abbiamo incluso la demenza noto fattore di rischio apolipoproteina E4 isoforma (ApoE4) in modo che CAn determinare contemporaneamente il suo stato e livello di espressione, da tale eliminando la necessità di test di genotipizzazione separati 2. LC MS / MS è abitualmente utilizzato come metodo di scelta per quantificazione con precisione piccole molecole rispetto ad altri metodi, quali ELISA o radioimmunologico (RIA). Questo cambiamento nell'uso della tecnologia MS alle proteine analisi, è stata guidata principalmente da questioni con le tecnologie immuno-based. Questi includono cross-specificità, variazione tra le partite, durata limitata, e il costo elevato. Pertanto, la proteomica mirati sta rapidamente diventando una crescente alternativa ai metodi di anticorpi based come Western blotting, RIA ed ELISA. Tuttavia, la capacità di multiplex molti marcatori in un saggio è il principale vantaggio di questa tecnica rispetto ai metodi immuno-base 3. La tecnica è applicabile a molti tessuti ed è stato usato come una strategia di convalida per studi molti proteomica compreso plasma 4 e nelle urine 5,6.
Il technique può essere applicato a qualsiasi laboratorio che ha accesso e la competenza nell'uso triple spettrometri di massa quadrupolo. disegno Peptide è relativamente semplice con il crescente utilizzo di database open source. Il mercato competitivo della sintesi peptidi personalizzati li rende molto più conveniente. peptidi pesanti, però, sono costosi e, pertanto, gli indicatori dovrebbero essere valutati su una piccola coorte prima di passare ad una scala più ampia. Vi è un crescente potenziale per la tecnica da utilizzare nelle impostazioni di diagnostica clinica, con la maggior parte dei grandi ospedali con le piattaforme basate triplo quadrupolo che possono essere facilmente adattati per eseguire analisi proteomica mirati. Una tale applicazione del metodo, rendendo nel setting diagnostici di routine, è la sua recente richiesta di proiezione macchia di sangue neonatale per l'anemia falciforme 7.
Protocollo
NOTA:. Uno schema del protocollo generale qui descritto è dato in figura 1 Tutti i campioni utilizzati per lo sviluppo di questo metodo sono campioni diagnostici clinici in eccesso e hanno l'approvazione etica dal Comitato Etico London Bloomsbury.
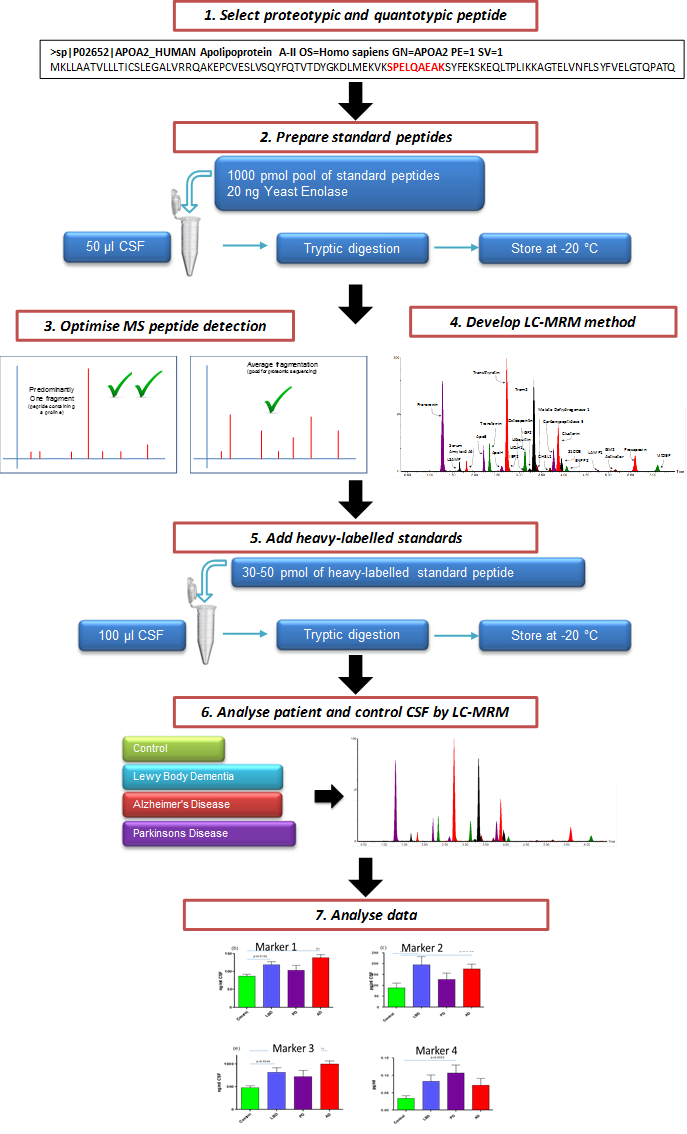
Figura 1. Schema Illustrando il processo globale di creare un mirate CSF MRM peptidi LC-MS / MS dosaggio. Marcatori candidato per la valutazione vengono selezionati dai bersagli proteici. Attraverso l'uso di peptidi sintetizzati personalizzato, viene creato un metodo multiplex LC-MS / MS mirato. Dopo la valutazione, il test può essere utilizzato per valutare l'efficacia di potenziali marcatori di neurodegenerazione.
1. Peptide Selezione e design
NOTA: I criteri per un peptide marcatore è che deve essere univoco (proteotypic ) e che rappresenta l'abbondanza quantitativa della proteina (quantotypic). Per determinare se un peptide è unico o meno, lo strumento 'esplosione' di ricerca sul sito web UniProt (http://www.uniprot.org/blast/) può essere utilizzato.
- Definire un elenco di proteine bersaglio (marcatori), vale a dire, ApoE (vedi tabella 2).
NOTA: se il marcatore è stato identificato da precedenti esperimenti di proteomica profiling 8, quindi selezionare il peptide che dà la migliore risposta da questo insieme di dati. - Se questa informazione non è disponibile, utilizzare i siti web open source, come ad esempio in silico tripsina digestione delle proteine bersaglio, utilizzando uno strumento software come MS-Digest.
- Scegliere il peptide che è trittico e non suscettibili di modificazione post traduzionale.
NOTA: Questa informazione può essere verificata sul sito UniProt www.uniprot.org. Evitare di peptidi soggette a modificazione chimica durante la preparazione dei campioni LC-MS. - Ordine sintesi personalizzata dei peptidi scelti.
NOTA: peptidi marker possono essere personalizzati sintetizzati dalle varie aziende commerciali. Per ApoE il peptide quantotypic utilizzato per misurare i livelli totali ApoE è stato determinato per essere AATVGSLAGQPLQER. Le sequenze peptidiche proteotypic utilizzati per determinare le varianti E2 ed E4 sono i peptidi trittico per la posizione 158 (RLAVYQAGAR e CLAVYQAGAR) e la posizione 112 (LGADMEDVCGR e LGADMEDVR), rispettivamente.
2. Preparazione dei peptidi standard
NOTA: Al fine di selezionare i migliori transizioni quantitativi, la rilevazione della matrice (CSF) deve essere ottimizzato. Il modo più efficace di ottimizzare peptidi multiplati è creare pool di peptidi a concentrazioni note. Queste piscine possono poi essere utilizzati per lo sviluppo del metodo e curve standard.
- Risospendere peptidi sintetici (dettagli peptide sono riportati nella tabella 2) a 1 mg / ml magazzinola concentrazione in base alle istruzioni produce. Per impostazione predefinita, se le istruzioni non sono disponibili, i peptidi risospendere in 50:50 (v / v) acetonitrile (ACN) / H 2 O.
- Preparare le diluizioni 1:10 del peptide dalla concentrazione magazzino e piscina 1.000 pmol di ciascun peptide in un tubo a bassa microcentrifuga vincolante. Asciugare giù in un concentratore di velocità-vac la piscina finale e conservare a -20 ° C. Preparare diverse piscine per uso futuro.
- Aliquota 100 ml di liquido cerebrospinale in basso tubi vincolanti. Liofilizzare il CSF.
- Risospendere un'aliquota di aggregati 1.000 pmol peptidi in tampone di digestione (100 mM Tris HCl, pH 7,8, 6 M urea, 2 M tiourea, 2% ASB14) per ottenere concentrazioni di 10 e 1 pmol / ml.
- Spike i peptidi raggruppati nelle liofilizzati aliquote di 100 microlitri di CSF a 0, 1, 2, 5, 10 e 15 le concentrazioni di PM. Aggiungere 20 ng di proteine non correlate intatto come il lievito enolasi di agire come standard interno e controllo per l'efficienza digestione del trypsin.
- Rabboccare le aliquote del QSC con digerire buffer per un importo finale di 20 ml. Vortice.
- Aggiungere 1,5 ml di ditiotreitolo (30 mg in 1 ml di 100 mM Tris HCl, pH 7.8) e agitare a temperatura ambiente per 1 ora.
- Aggiungere 3 ml di iodoacetamide (35 mg in 1 ml di 100 mM Tris HCl, pH 7.8) e agitare a temperatura ambiente per 45 minuti al buio.
- Aggiungere 165 ml di DDH 2 O.
- Aggiungere 10 ml di 0,1 mg / mL sequenziamento soluzione di tripsina grado modificata risospese in 50 mM di tampone di bicarbonato di ammonio, pH 7.8. Incubare a bagnomaria a 37 ° CO / N e fermare la digestione mediante congelamento campioni. Conservare digerisce a -20 ° C fino al momento di analizzare.
3. Ottimizzazione della SM Peptide Detection
- Copiare e incollare la sequenza del peptide di ottimizzare, in un software appropriato, ad esempio, Skyline 9. Clicca sulla sequenza peptidica per ottenere le informazionidi ioni di massa di ioni precursori e dei prodotti attesi.
- Diluire i peptidi a magazzino concentrazione (vedi paragrafo 2.1) a seguito di 1 ng / ml concentrazione per lo sviluppo del metodo MS.
- Direttamente infondere peptidi nello spettrometro di massa a portata ottimizzata (solitamente 0,1 - 0,8 ml / min) e sul 0 - 5% energia di collisione.
- Acquisire spettro MS al fine di individuare le sperimentali ioni precursori moltiplicano pagano 8. Scegli lo ione precursore (m / z) che da maggior intensità (doubly- o sono invitati ioni precursori triplamente-cariche).
- Reinfuse il peptide e applicare energia di collisione di frammentare il peptide dalla collisione indotta dissociazione (CID). Ottimizzare le energie cono e di collisione per ottenere il miglior modello di frammentazione (frammenti di ioni singolarmente o doppiamente cariche, e la massa dei frammenti di ioni preferibilmente più grande di ioni genitore, si consiglia).
- Verificare che le transizioni sperimentalmente ottenuti corrispondono transizioni generate in silico </ Em> (come descritto al punto 3.1). Salvare l'elenco di transizione nel file metodo MRM con almeno 2 passaggi più intensi per ione precursore. Include 2 transizioni: 1 per la quantificazione e 1 per la conferma nel test finale.
NOTA: Alcuni produttori SM hanno una funzione (ad esempio, acque IntelliStart) per MRM automatizzato o di ottimizzazione analisi SRM. Se possibile infondere peptidi con fasi mobili combinati al 50 - 70% ACN con il 0,1% FA.
Lo sviluppo del metodo 4. LC-MRM
NOTA: Analizzare la miscela di peptidi sintetici da un sistema di cromatografia liquida (UPLC) accoppiato ad uno spettrometro di massa a triplo quadrupolo Ultra Performance. Assicurarsi che la fonte è pulita. Solvente A è DDH 2 0 con 0,1% FA; Solvente B è ACN, con 0,1% FA.
- Utilizzare il sistema LC-MS dotato di una colonna UPLC ricco di C-18 fase (1,6 micron di diametro, 90 pori Å 2,1 mm x 50 mm di lunghezza) e collegato a una precolonna della stessa fase.
- Sbrinamento digerisce CSF su ghiaccio, centrifugare a 16.000 g per 10 minuti e trasferire 60 ml in 300 ml fiale inserto in vetro e conservare il resto torna a -20 ° C.
- Iniettare la più alta concentrazione dal punto curva standard utilizzando 10 min 1 - 40% di pendenza ACN lineare (vedi Tabella 1 per le impostazioni di pendenza)
- Aprire il cromatogramma e ritenzione nota tempo risultante e primi due più intense transizioni (quantitative) per peptide con tutte le transizioni create al punto 3.
- Sulla base di queste informazioni, aggiornare un metodo MRM 10 min (creato nel passo 3.6) con canali a tempo per misurare peptidi (vedi figura 2 per un esempio). Per mantenere la sensibilità, mantenere ogni canale punti per picco superiore a 8 e tempi maggiori di 0,01 sec per almeno una transizione per ogni peptide.
- Includono 'ritardi solvent' nel metodo MRM: una all'inizio fino a 10 secondi prima della prima eluizione di picco eun'altra alla fine del metodo, 20 sec dopo l'ultimo picco di eluizione. A tale scopo, selezionando "ritardi solvente" in eventi di metodo nel file metodo MS.
- Eseguire la curva standard attraverso il metodo MRM cronometrato e garantire non ci sono picchi di interferenza non specifici con transizioni (generate nel passaggio 3.6) controllando per la linearità.
- Determinare se i peptidi sono rilevabili mediante l'esecuzione non a spillo di controllo in pool e la malattia CSF attraverso il metodo.
- Rimuovere i peptidi che si trovano al di sotto del limite di rilevazione del test.
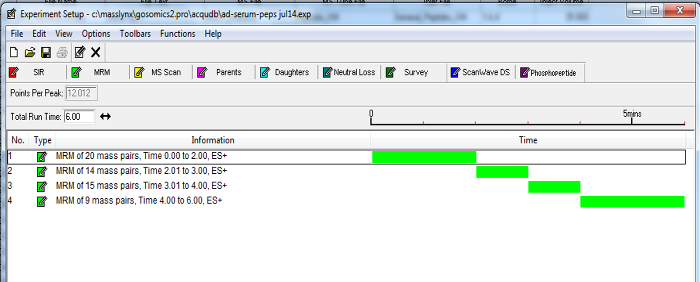
Figura 2. Esempio di una dinamica MRM MS Metodo. Timed canali di transizioni peptidici possono essere raggruppati in base a tempi di ritenzione stabiliti. L'attivazione di MRM per essere incorporato in modo temporizzato come i peptidi marker selezionato eluiscano dalla cromatografia su colonna, riduce al minimo il numero di Transitions per un periodo di tempo e aumenta la sensibilità del test. Clicca qui per vedere una versione più grande di questa figura.
5. L'aggiunta di standard interni
NOTA: Come descritto in precedenza 10, stabili standard interni marcati con isotopi possono essere inclusi nel test. A causa della spesa di questi standard, si consiglia di valutare prima peptidi nella matrice.
- Determinare i peptidi ideali ottimizzati per il rilevamento in CSF: scegliere i peptidi che sono i più ricorrenti, con la massima intensità e senza picchi di interferenza.
- Disegno peptidi corrispondenti a includere pesanti 13 C 15 N etichette aminoacidi che aumentano la massa del peptide di almeno 6 Da rispetto al peptide endogeno. Inoltre, aggiungere un tag di 4 - 6 aminoacidi aggiuntivi per sia la N- o C-terminale del Pep pesantemarea di controllare per la digestione trittico.
- Diluire stabili standard interni marcati con isotopi nel buffer di digestione (vedi punto 2.4). Determinare la quantità di standard interno marcati con isotopi stabili ideale che sarà a spillo nel liquido cerebrospinale di chiodare in vari livelli a seconda delle abundancies precedentemente osservati durante lo sviluppo. Mirano a raggiungere circa il rapporto 1: 1 di stabilità di serie marcati con isotopi di peptide endogeno.
NOTA: Stabile standard interni marcati con isotopi possono essere sintetizzati da varie società commerciali.
6. LC-MRM Assay del CSF campioni di pazienti
- Spike quantità di standard marcati con isotopi stabili ottimizzato in 100 ml di CSF e liofilizzare il composto.
- Risospendere il CSF in 20 ml di digest tampone ed eseguire la digestione di cui ai punti 2.7 - 2.10.
- Analizzare i campioni utilizzando il metodo LC-MRM sviluppato al punto 4.
- Per l'analisi quantitativa, eseguire il pep di seriemaree a concentrazioni di 0 -15 pmol in una curva standard. Vedere il punto 2.5 per la preparazione di curva standard.
Analisi 7. I dati
NOTA: quantitativa dei dati si basa sul rapporto di intensità della linea di base standard interni di picco (peptidi pesante etichettati). Informazioni dettagliate per quanto riguarda l'analisi dei dati SRM / MRM è stato descritto in precedenza 10. dati di rapporto possono essere usati in una curva standard per determinare i livelli assoluti o calcolare dalla concentrazione aggiunta del peptide pesanti marcato.
- Analizzare i dati LC-MRM utilizzando software standard spettrometri di massa costruttori 10. In alternativa, utilizzare il software Skyline per analizzare i dati quantitativi MRM 10.
- Controllare la sensibilità della corsa controllando la risposta di uno standard interno come l'enolasi lievito spillo o uno standard stabile isotopo marcato in ogni seduta. Assicurarsi che il coefficiente di variazione (CV) non è maggiore di25%.
- rivedere manualmente l'annotazione dei dati per garantire la precisione. Analizzare ogni peptide e il rapporto di uno standard interni marcati con isotopi stabili del caso, cioè, usare la versione stabile marcati con isotopi del peptide, se disponibile.
- Per ottenere valori assoluti di pmol per 100 ml di CSF, eseguire i dati rapporto attraverso opportuni curve standard che sono stati eseguiti allo stesso tempo.
- Calcolare il coefficiente di variazione (CV). CV per ogni peptide dovrebbe essere inferiore a 25% e inferiore al 15% per alte peptidi abbondanti.
NOTA: Valore assoluto pmol per 100 valori CSF microlitri può utilizzato nella successiva analisi statistica a valle.
8. Apolipoproteina E Isoform Stato
NOTA: per determinare lo stato ApoE isoforma, la presenza dei peptidi corrispondenti può essere effettuata determinando la presenza di ogni isoforma.
- Si consideri il ApoE in 100 ml di soglia CSF di> 1.000 segnale-to-rumore come positivo per questo peptide. Vedere la Figura 5 per i peptidi richiesti / assenti per determinare lo stato isoforma di un paziente. Determinare l'espressione allelica calcolando la% di ogni isoforma al totale espressione ApoE.
Risultati
Utilizzando il metodo sopra descritto, un saggio multiplex high throughput 10 min composta da 74 peptidi 54 proteine è stato sviluppato, come dosaggio per i marcatori di disturbi neurodegenerativi morbo di Alzheimer e demenza Lewy (LBD) 8. La figura 3 mostra un cromatogramma multiplex pubblicato in precedenza 8 dei marcatori peptidici significative dal test. I peptidi incluse nel dosaggio e loro transizioni quantitative sono riportati nella
Discussione
Come con tutti i test basati MS, le fasi critiche del metodo sono la determinazione degli importi appropriate ed esatte degli standard interni. Se si utilizza la quantificazione assoluta, quindi la corretta quantità di peptidi a spillo nella curva standard sono anche critici.
Il nostro test non richiede la precipitazione del CSF o l'uso di qualsiasi tipo di puliti gradini o dissalazione prima dell'analisi MS - è un metodo di reazione one-pot interamente. A causa del piccolo volume ...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This work was funded and facilitated through the GOSomics research initiative by the National Institute for Health Research, Biomedical Research Centre at Great Ormond Street Hospital and the UCL Biological Mass Spectrometry Centre at the UCL Institute of Child Health with kind donations from the Szeban Peto Foundation. The Dementia Research Centre is an Alzheimer's Research UK coordinating centre. The authors acknowledge the support of Alzheimer's Research UK, the NIHR Queen Square Dementia Biomedical Research Unit, UCL/H Biomedical Research Centre, and Leonard Wolfson Experimental Neurology Centre.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetonitrile (ACN), LC-MS grade | Fisher | A955-1 | |
| Formic acid, LC-MS grade, | Fisher | A117-50 | |
| Dithiothreitol (DTT) | Sigma | D5545-5G | |
| Hydrochloric acid, 37% w/w | VWR | BDH3028-2.5LG | |
| Iodoacetamide | Sigma | I1149-5G | |
| Sodium hydroxide (NaOH) | Fisher | S318-500 | |
| Trypsin, sequencing grade, modified | Promega | V5113 | |
| Trifluoroacetic acid (TFA), LC-MS grade | Fisher | A116-50 | |
| Urea | Sigma | U0631-500g | |
| Water, LC-MS ULTRA Chromasolv | Fluka | 14263 | |
| Custom synthesised peptides desalted 1-4mg | Genscript | custom | |
| heavy labelled amino acid [C13 N15] custom peptides | Genscript | custom | |
| ASB 14 | Merck Millipore | 182750-25gm | |
| Thiourea | Sigma | T7875-500G | |
| Tris base | Sigma | T6066 | |
| VanGuard precolumn | Waters | 186007125 | |
| Cortecs UPLC C18+ 1.6um 2.1 x50mm column | Waters | 186007114 | |
| Yeast Enolase | Sigma | E6126 | |
| 300ul clear screw top glass vials | Fisher scientific | 03-FISV | |
| Y slit screw caps | Fisher scientific | 9SCK-(B)-ST1X | |
| Freeze dryer | Edwards Mudulyo | Mudulyo system | |
| Concentrator/Speed vaccum | Eppendof | concentrator plus 5301 | |
| Xevo -TQ-S mass spectrometer | Waters | ||
| Acquity UPLC system | Waters |
Riferimenti
- Prince, P. M., Guerchet, D. M., Prina, D. M., et al. . Policy Brief: The Global Impact of Dementia 2013-2050. , (2013).
- Hirtz, C., et al. Development of new quantitative mass spectrometry and semi-automatic isofocusing methods for the determination of Apolipoprotein E typing. Clin Chim Acta. 454, 33-38 (2015).
- Marx, V. Targeted proteomics. Nat Methods. 10 (1), 19-22 (2013).
- Heywood, W., et al. The development of a peptide SRM-based tandem mass spectrometry assay for prenatal screening of Down syndrome. J Proteomics. 75 (11), 3248-3257 (2012).
- Heywood, W. E., et al. Proteomic Discovery and Development of a Multiplexed Targeted MRM-LC-MS/MS Assay for Urine Biomarkers of Extracellular Matrix Disruption in Mucopolysaccharidoses I, II, and VI. Anal Chem. , (2015).
- Manwaring, V., et al. The identification of new biomarkers for identifying and monitoring kidney disease and their translation into a rapid mass spectrometry-based test: evidence of presymptomatic kidney disease in pediatric Fabry and type-I diabetic patients. J Proteome Res. 12 (5), 2013-2021 (2013).
- Moat, S. J., et al. Newborn blood spot screening for sickle cell disease by using tandem mass spectrometry: implementation of a protocol to identify only the disease states of sickle cell disease. Clin Chem. 60 (2), 373-380 (2014).
- Heywood, W. E., et al. Identification of novel CSF biomarkers for neurodegeneration and their validation by a high-throughput multiplexed targeted proteomic assay. Mol Neurodegener. 10, 64 (2015).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26 (7), 966-968 (2010).
- Manes, N. P., Mann, J. M., Nita-Lazar, A. Selected Reaction Monitoring Mass Spectrometry for Absolute Protein Quantification. J Vis Exp. (102), e52959 (2015).
- Craig-Schapiro, R., et al. YKL-40: a novel prognostic fluid biomarker for preclinical Alzheimer's disease. Biol Psychiatry. 68 (10), 903-912 (2010).
- Perrin, R. J., et al. Identification and validation of novel cerebrospinal fluid biomarkers for staging early Alzheimer's disease. PLoS One. 6 (1), e16032 (2011).
- Liguori, C., et al. Orexinergic system dysregulation, sleep impairment, and cognitive decline in Alzheimer disease. JAMA Neurol. 71 (12), 1498-1505 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon