È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Generazione di fusioni proteina fluorescente in
In questo articolo
Riepilogo
Modificazione genetica PCR-mediata può essere utilizzato per generare fusioni di proteine fluorescenti a Candida specie, che facilita la visualizzazione e la quantificazione delle cellule di lievito e proteine. Qui, vi presentiamo una strategia per la costruzione di una fusione proteina fluorescente (Eno1-FP) in Candida parapsilosis.
Abstract
Specie di Candida, colonizzatori prevalente dei tratti intestinali e genito-urinario, sono la causa della maggior parte delle infezioni fungine invasive sugli esseri umani. Pertanto, sono necessari strumenti molecolari e genetici per facilitare lo studio dei loro meccanismi patogeni. PCR-mediata modificazione genetica è un metodo semplice e rapido per generare proteine epitopi-tag per facilitare la loro individuazione. In particolare, la proteina fluorescente (FP) fusioni sono potenti strumenti che consentono la visualizzazione e la quantificazione di entrambe le cellule di lievito e proteine da microscopia a fluorescenza e immunoblotting, rispettivamente. I plasmidi contenenti FP sequenze di codifica, insieme con geni marcatori nutrizionali che facilitano la trasformazione delle specie Candida, sono stati generati al fine di costruzione FP ed espressione in Candida. Qui, vi presentiamo una strategia per la costruzione di una fusione FP in una specie di Candida. I plasmidi contenenti il impermeabi nourseothricinnce gene marcatore di trasformazione (NAT1) insieme a sequenze per uno verde, giallo, o ciliegio PQ (GFP, YFP, mCherry) sono utilizzati insieme con gli iniettori che comprendono sequenze gene-specifici in una reazione a catena della polimerasi (PCR) per generare una cassetta FP . Questa cassetta-specific gene ha la capacità di integrarsi nel 3'-end del corrispondente locus genico mediante ricombinazione omologa. Successful fusione in-frame della sequenza FP nel locus gene di interesse viene verificato geneticamente, seguita da analisi dell'espressione della proteina di fusione mediante microscopia e / o metodi immuno-rilevazione. Inoltre, nel caso di proteine altamente espressi, fusioni successo possono essere esaminati per principalmente da tecniche di imaging di fluorescenza.
Introduzione
Specie di Candida sono funghi commensali che colonizzano i tratti intestinali e genito-urinario di tutti gli esseri umani. In condizioni di immunodeficienza, come ad esempio che si verificano con parto prematuro o effetti immunosoppressivi di trattamenti per il cancro, specie Candida possono diventare patogeni opportunisti. Tra le specie di Candida, Candida albicans è il colonizzatore fungina più diffusa e provoca la maggior parte delle infezioni fungine invasive. Altre specie di Candida come C. glabrata, C. parapsilosis, C. tropicalis e C. kruseii anche causare gravi infezioni nei pazienti immunocompromessi, con qualche esibendo resistenza intrinseca alla usati comunemente antibiotici anti-fungine quali fluconazolo e amfotericina B. Quindi, infezioni con alcune di queste specie sono osservati più frequentemente, soprattutto nei pazienti in trattamento profilattico con farmaci anti-fungini. Anche con una appropriata e tempestivatrattamento NTI-fungine, infezioni da Candida invasive continuano ad essere associata a morbidità e mortalità 1 significativa. A causa del significato di specie Candida nella salute umana, vi è l'esigenza di strumenti molecolari prontamente disponibili che permettono lo studio e la spiegazione dei loro meccanismi patogeni.
Uno strumento importante che permette ai ricercatori di visualizzare e quantificare le cellule microbiche e le proteine che essi esprimono è tecnologia della fusione FP. Reazione a catena della polimerasi (PCR) mediata modificazione genetica, come descritto in questo documento, permette la costruzione di fusioni, tra le sequenze FP ed una proteina Candida sequenza codificante di interesse al suo locus genomico. Integrazione stabile del costrutto facilita l'analisi dell'espressione della proteina e dinamiche localizzazione della proteina. I plasmidi contenenti sequenze FP, ottimizzati per l'espressione in Candida albicans e che possono essere utilizzati nella g PCR-mediatastrategia modifica ene, è stato costruito in precedenza 2, 3, 4, 5. I plasmidi contengono FP trasformazione "cassette": una sequenza FP legato ad un gene marcatore nutrizionale che facilita la trasformazione di C. albicans e C. parapsilosis 2, 3, 4, 5, 6, 7. Attualmente plasmidi disponibili contengono una varietà di geni marcatori selezionabili nutrizionali (URA3, HIS1, Arg4) per la trasformazione di ceppi auxotrofi così come marcatore resistenza ai farmaci dominante (NAT1), che facilita la trasformazione di ceppi clinici privi auxotrophies. Inoltre, plasmidi contengono opzioni per un massimo di quattro differenti sequenze FP (verde [GFP], Yellow [YFP], ciano [CFP], e la ciliegia [mCherry]) e sia una sequenza di terminazione ADH1 per la costruzione di fusioni di proteine carbossi-terminale, o di una sequenza promotrice per la costruzione di fusioni di proteine amino-terminale. Primer sono progettati con omologia al DNA plasmidico che circonda la cassetta FP. Inoltre, i primer contengono anche sequenze 5'-estensione recanti omologia con il gene di lievito di interesse per essere etichettato, che facilita l'integrazione della cassetta nel locus genomico tramite ricombinazione omologa (Figura 1). Gene-specifici cassette FP sono generati mediante PCR e poi trasformati in cellule di Candida rese competenti per l'assorbimento di DNA mediante trattamento con acetato di litio.

Figura 1: Diagramma di come FP fusioni sequenza sono generati in specie di Candida. (A) inclusa, plasmidi DNAes una sequenza FP e una codifica sequenza nourseothricin resistenza (NAT1). posizioni relative di Forward (FWD) e reverse primer (REV) sono mostrati, con porzioni nere dei primer che indica la regione di omologia alla sequenza plasmidi e le porzioni viola denotano la regione di omologia gene-specifica o estensione di primer. Cassette (B) FP si trasformano in Candida e si integrano all'interno del ENO1 locus genomico mediante ricombinazione omologa (linee tratteggiate). (C) risultante FP sequenza di fusione presso il 3'end di ENO1. Clicca qui per vedere una versione più grande di questa figura.
Qui, vi presentiamo un esempio di fusione di proteine (Eno1-FP) costruzioni in specie di Candida. Usiamo codifica plasmidi contenenti il gene marcatore NAT1 trasformazione insieme a sequenze codificanti GFP, YFP, omCherry (Figura 2). Questi plasmidi sono utilizzati insieme con primer in PCR per generare cassette gene-specifici che facilitano fusione PQ alla estremità 3 'del ENO1, con conseguente espressione di Eno1 fusa PQ al suo carbossi-terminale.
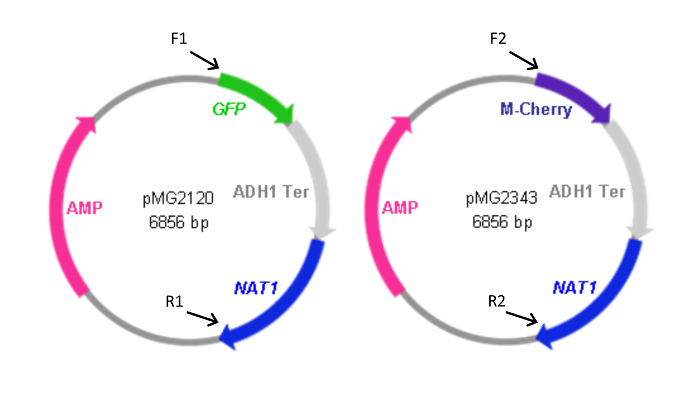
Figura 2: Mappe di FP plasmidi cassette contenenti. Forward (F) e reverse primer (R) utilizzati per generare le cassette dei plasmidi sono indicati nonché la posizione relativa della loro omologia con i plasmidi. Sequenze primer sono elencati nella tabella 1. F1 e R1 sono stati utilizzati anche per generare la cassetta pYFP- NAT1. Il plasmide contenente la cassetta YFP- NAT1 (pMG2263) è identica a pMG2120 con l'eccezione di YFP al posto della sequenza GFP. Dimensioni cassette: GFP-NAT1, 3.7 KBP; mCherry- NAT1, 3,2 kbp; YFP- NAT1, 3.7 kbp. Questo dato è stato modificato da Gerami-Nejad, et al. 4 Clicca qui per vedere una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Isolare Template plasmidi da E. coli
- Grow E. coli contenenti il plasmide modello durante la notte in 10 ml di brodo lisogenia (LB) + 200 mg / L ampicillina (AMP) a 37 ° C con agitazione.
- Celle di raccolta per centrifugazione a 6.000 xg per 2 min.
- Decantare liquido, isolare e purificare il DNA da E. coli cellule con un metodo standard, come descritto in precedenza in Ausubel et al. 8.
- Risospendere DNA in Tris-EDTA (TE; 10 mM Tris, pH 8,0, 1 mM EDTA, pH 8,0) ad una concentrazione di lavoro di 50-100 ng / ml.
2. Primer design
| Primer | Sequenza Primer |
| F1 | 5 ' GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTTCTAAAGGTGAAGAATTATT 3 ' |
| R1 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC GTAAAACGACGGCCAGTGAATTC 3 ' |
| F2 | 5 'GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTGTTTCAAAAGGTGAAGAAGATAAT 3 ' |
| R2 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC ACTGGATGGCGGCGTTAGTATC 3 ' |
Tabella 1: sequenze di primer utilizzati in questo studio. Testo in corsivo grassetto indica omologia al ENO1 locus genomico, normali le regioni di font sono omologhi di DNA plasmidico.
- primer design per essere omologo al plasmide sequenze confinanti la cassetta da amplificare nonché alla estremità 3 'del gene bersaglio di interesse (ad es ENO1) agevolare ricombinazione nel locus genomico del gene (Figura 2 e Tabella 1) .
- Assicurarsi che le sequenze dei primer forward corrispondono ultimi 70 coppie di basi (bp) del gene di interesse, 5'-3 ', meno il codone di stop, per mantenere il telaio codifica, più la prima di circa 30 bp della sequenza plasmide siano amplificato. Come una nota, il GGTGGTGGTT in ogni primer è un linker poli-glicina senza FP omologia. Da segnalare, in assenza di un linker, non ci può essere la fusione diretta dei domini funzionali, che possono teoricamente portare a misfolding, bassa resa nella produzione di proteine, o bioattività compromessa.
- Assicurarsi che le sequenze primer reverse sono 70 bp solo a valle del gene, 3'5 ', ad esclusione di qualsiasi sequenza genica, più l'ultima approximately 30 bp del marcatore nutrizionale o resistenza ai farmaci utilizzati nel plasmide.
- Usare F1 e R1 per generare GFP NAT1 e YFP- NAT1 cassette con rispettivamente pMG2120 e pMG2263, e F2 e R2 per generare mCherry- NAT1 con pMG2343.
3. Generare FP cassette mediante PCR (1 ° giorno)
- Preparare i reagenti per la PCR. Fare un master mix (500 volume finale mL) aggiungendo i seguenti volumi e concentrazioni in una provetta da 1,5 ml: 50 microlitri di buffer PCR (500 mm cloruro di potassio, 100 mM Tris pH 8,0 in acqua), deoxynucleotides 20 microlitri (dNTP; magazzino miscela di nucleotidi a 10 mm ciascuno), 40 ml di 25 mM cloruro di magnesio, 20 l purificati plasmide (da ~ 50-100 ng / ml di soluzione), 10 ml ciascuno in avanti e di primer (da 10 soluzioni madre mM inversa), 30 ml di Taq polimerasi (generica, 5.000 unità / ml), e 320 ml di acqua.
- Aliquota 50 ml di master mix in ciascuno dei 10 PCR-compatibLe 0,5 ml tubi.
- Mettere provette PCR in termociclatore ed eseguire le seguenti operazioni: 1 ciclo di 5 min a 94 ° C per denaturare dsDNA; 40 cicli in sequenza di 45 secondi a 94 ° C, 30 sec a 55 ° C per consentire primers per temprare di plasmide DNA stampo, e 4 minuti a 68 ° C per l'estensione dei prodotti DNA; e 1 ciclo estensione finale di 15 min a 72 ° C.
possono avere bisogno di essere modificato in base alla particolare Taq polimerasi utilizzata master mix PCR e in bicicletta parametri: NOTA. - Pool tutti i prodotti provenienti dai 10 reazioni PCR in una provetta da 1,5 ml.
- Oggetto 5 ml di prodotto di PCR in pool a elettroforesi su gel di agarosio per verificare dimensioni amplicone ed ottenere una stima della concentrazione del prodotto, basata sul confronto di una scaletta DNA. In generale, usare ~ 250 mg di DNA cassette in ogni successiva mix di trasformazione.
- Precipitare DNA aggiungendo acetato di 50 microlitri 3 M di sodio seguito da 750 microlitri 95% etanolo per i prodotti e incubare almeno 30 mina -20 ° C.
- Raccogliere i prodotti PCR mediante centrifugazione del tubo a 16.000 xg per 10 min. Rimuovere con cautela e scartare il surnatante e asciugare il pellet durante la notte. Risospendere il DNA cassette pellet essiccati in 40 microlitri TE pH 8,0 e conservare a temperatura ambiente fino al momento dell'uso.
4. Trasformare cellule di Candida con FP DNA Cassette
- Il giorno 1, recuperare ceppo di lievito da trasformare da un glicerolo 15% congelati (-80 ° C) stock strisciando alcuni cristalli raschiati Onto lievito peptone destrosio con adenina (YPAD) agar e incubare a 30 ° C. Dopo il recupero della crescita delle colonie, inoculare una singola colonia in 2 ml di mezzo YPAD liquido in una provetta di vetro con tappo traspirante e incubare una notte a 30 ° C con agitazione.
- Il giorno 2, diluire 300 ml di coltura di lievito durante la notte in 50 ml YPAD fresco (ad un OD finale 600 ~ 0.2) in un 125 ml beuta con un tappo traspirante. Agitare a 30 ° Cper ~ 3 ore (per un diametro finale 600 ~ 0,6-0,8).
- Versare il cultura durante la notte in una provetta conica da 50 ml e agglomerare le cellule mediante centrifugazione per 5 min a 1 500 xg in una centrifuga da tavolo.
- Eliminare e scartare correttamente il surnatante. Risospendere il pellet di cellule in acqua 5 ml. Re-agglomerare le cellule per centrifugazione ancora per 5 min a 1500 xg in una centrifuga da tavolo.
- Eliminare e scartare correttamente il surnatante. Risospendere le cellule in 500 microlitri TELiAc (TE litio acetato: 10 mM Tris, pH 8,0, 1 mM EDTA, pH 8,0, 0,1 M di litio acetato) durante il trasferimento in una provetta da 1,5 ml. Centrifugare la provetta per 2 minuti a 3000 xg in una microcentrifuga.
- Risospendere le cellule in 250 microlitri TELiAc. Il volume totale compreso il pellet dovrebbe essere ~ 300 microlitri.
- Per un (provetta da microcentrifuga diverso in 4.2.4) pulito, aggiungere 5 microlitri vettore DNA (10 mg / ml) e 150 ml di cellule di Candida preparati (da 4.2.4). Questo è il controllo negativo per donnaransformation.
- Per un secondo tubo da microcentrifuga pulita, aggiungere 5 ml di DNA denaturato (carrier che è stata bollita a 90 ° C per 10 min e raffreddato a 4 ° C), tutti i 40 microlitri del prodotto PCR preparato (da 3.3.1) e 150 pl di cellule di Candida preparati (da 4.2.4).
- Incubare le due miscele di trasformazione per 30 min a temperatura ambiente.
- Per ogni tubo mix trasformazione, aggiungere 700 microlitri della miscela PLATE (10 mM Tris, pH 8,0, 1 mM EDTA, pH 8,0, 0,1 M di litio acetato in 50% di polietilene glicole 3350). Capovolgere i tubi per miscelare e incubare una notte a temperatura ambiente.
- Il giorno 3, incubare la trasformazione si mescola a 42 ° C per 1 ora (shock termico).
- Centrifugare la trasformazione miscele per 30 secondi a 16.000 xg in una microcentrifuga. Rimuovere e gettare correttamente il surnatante. Risospendere ciascuno dei pellet cellulari in 150 microlitri di acqua delicatamente pipettando su e giù in modo da non danneggiare le cellule.
- Per le trasformazioni utilizing geni marcatori auxotrofi (ad esempio URA3), piastra ogni intera miscela pipettando le soluzioni sul appropriata agar terreni selettivi (ad es uridina manca) e diffondere la miscela in modo uniforme utilizzando microsfere di vetro sterili.
- Per trasformazioni utilizzando il gene marcatore di resistenza nourseothricin (NAT1), come descritto qui, piastra di trasformazione mescola prima sul agar YPAD non selettiva e incubare a 30 ° C per 6-12 ore. Questo passo aiuti nel recupero delle cellule, shock post di calore, prima nourseothricin lo stress è applicato.
- Dopo il recupero della crescita parziale, piastra replica le cellule Candida Onto YPAD contenente 400 mg / ml nourseothricin. Per le trasformazioni che utilizzano geni marcatori nutrizionali (per esempio URA3), questa fase intermedia la placcatura non è necessario e le cellule può essere placcato direttamente su supporti di lievito selettivi (ad esempio YPAD manca uridina), come descritto in 4.4.2.
NOTA: Se la trasformazione è successo, colOnies dovrebbe apparire da uno a tre giorni (potenzialmente fino a cinque giorni di conseguenza per la selezione su agar nourseothricin contenente). Non ci sono colonie dovrebbero apparire su piastre diffuse con miscele di trasformazione che contengono DNA vettore da solo (controllo negativo).
- Per la selezione marcatore auxotrophic e nourseothricin, striscia trasformanti putativi come singole colonie per piastre di agar terreni selettivi freschi e incubare a 30 ° C per propagare cellule di lievito che possono essere sottoposti a screening per la costruzione di successo di fusioni FP.
- Trasformanti dello schermo per la corretta integrazione della cassetta di tagging (vedi Rappresentante dei risultati per un esempio dettagliato). Se il gene di interesse viene espresso in quantità sufficiente, tutta la colonia fluorescenza può verificare tale che è possibile rilevare potenziali candidati integrants utilizzando un sistema di imaging piastra con capacità di rilevamento di fluorescenza.
- Controllare integrants putativi mediante PCR utilizzando primer omologa a sequenze outside della regione di integrazione per confermare fusione al gene bersaglio.
- Inoltre, in considerazione analisi Western blot per determinare l'espressione e dimensioni della proteina di fusione, come anche per microscopia a fluorescenza di singole cellule per la conferma visiva della localizzazione delle proteine, se noto.
Access restricted. Please log in or start a trial to view this content.
Risultati
A titolo di esempio, abbiamo usato il protocollo sopra descritto per la costruzione di GFP e mCherry fusioni di Eno1 in un ceppo di laboratorio C. parapsilosis. Ogni transformant putativo è stato inizialmente restreaked per la crescita. In questo esempio, dal momento che la proteina di fusione risultante è altamente espresso (enolasi) e le PQ sono luminose, siamo stati in grado di schermare trasformanti di microscopia a fluorescenza prima di eseguire una diagnosi mediante PCR ...
Access restricted. Please log in or start a trial to view this content.
Discussione
Costruzione di epitopo etichettato sequenze in specie Candida utilizzando la strategia di modificazione genetica PCR-mediata sopra descritto possono essere riassunti come un processo in tre fasi. Innanzitutto, una cassetta è fatta da PCR che codifica sia la sequenza desiderata di integrazione e regioni omologa al locus di inserimento nel genoma di lievito. In secondo luogo, le cellule di lievito da trasformare sono chimicamente competenti con acetato di litio e co-incubate con la cassetta. Terzo, le cellule ve...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo N. Dean per fornire la sequenza originale mCherry FP, M. Gerami-Nejad per la costruzione di plasmidi, B. Larson per l'assistenza tecnica, e T. Heisel per consigli utili durante lo sviluppo di questo progetto. JB è stato sostenuto dal premio Consiglio europeo della ricerca avanzata 340.087 (RAPLODAPT). sistemi di microscopia e di imaging sono stati forniti dalla University of Minnesota Pediatria Foundation e l'Università del Minnesota Imaging Center.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 100 W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50 ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5 ml, 1.5 ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125 ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75 mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2 ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4 °C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
Riferimenti
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. , Wiley. New York. (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon