Method Article
Un emoderivato di Test per la rilevazione di ROS1 e RET trascrizioni di fusione da acido ribonucleico usando reazione a catena della polimerasi digitale di circolazione
In questo articolo
Riepilogo
Rilevamento di acido ribonucleico (cRNA) dal sangue di circolazione è un bisogno insoddisfatto in diagnostica clinica. Qui descriviamo i metodi che caratterizzano cRNA dai malati di cancro del polmone non a piccole cellule usando reazione a catena della polimerasi digitale sensibile e specifico. Le prove di soddisfano requisiti di progettazione per individuare varianti di fusione entro 72 ore.
Abstract
Abbiamo sviluppato metodi innovativi per l'isolamento e la caratterizzazione del tumore-derivati circolo acido ribonucleico (cRNA) per biopsia liquida a base di sangue. Rilevamento affidabile di cRNA recuperato dal sangue rappresenta una soluzione per un bisogno insoddisfatto critico nella diagnostica clinica. Il test inizia con la raccolta di sangue intero nelle provette di raccolta di sangue contenente conservanti che stabilizzano il cRNA. In questo sistema di test è isolata da plasma senza cellula, exosomal e RNA piastrina-collegato. Il cRNA è inverso trascritto in DNA complementare (cDNA) e amplificato usando reazione a catena della polimerasi digitale (dPCR). I campioni vengono valutati per il biomarcatore di destinazione così come un gene di controllo. Convalida del test incluso limite di rilevamento, precisione e robustezza studi con campioni analitici. Il metodo sviluppato a seguito di questi studi riproducibile rilevare le varianti multiple di fusione per ROS1 (proto-oncogene C-Ros 1; 8 varianti) e RET (riarrangiato durante il proto-oncogene transfezione; 8 varianti). Il flusso di lavoro di elaborazione del campione è stato ottimizzato in modo che i risultati dei test possono essere generati costantemente entro 72 ore dal ricevimento del campione.
Introduzione
Fino al 25% del carcinoma polmonare non a piccole cellule (NSCLC) pazienti non possono avere tessuto sufficiente disponibile per il test al momento della diagnosi. Anche nei casi in cui il tessuto è disponibile, può non essere sufficiente quantità o qualità di eseguire raccomandato test molecolari1,2. In casi dove c'è abbastanza tessuto da una biopsia per l'analisi molecolare, pazienti potrebbero essere necessario attendere diverse settimane o più per ottenere risultati, o iniziare il trattamento senza risultati molecolari3,4. Tuttavia, è fondamentale che la diagnosi molecolare informativa essere disponibile dato l'avvento di opzioni multiple di trattamento mirato per i pazienti diagnosticati con NSCLC. Test del DNA libero circolante (cfDNA) da biopsia liquida è una soluzione per le sfide del tradizionale tessuto test4,5,6. Opzioni di test corrente per actionable mutazioni nel NSCLC utilizzando cfDNA e un flusso di lavoro basato su dPCR simile per generazione risultato rapido, includono il recettore del fattore di crescita epidermico (EGFR) sensibilizzante mutazioni ΔE746-A750 e L858R, mutazione di EGFR resistenza T790M , Varianti del proto-oncogene KRAS (KRAS) e B-Raf proto-oncogene (BRAF) variante V600E. Anche se non quanto più ampiamente adottata dal campo, circolanti tumore-derivato di RNA messaggero (mRNA) isolato da liquida biopsia può anche fornire importanti informazioni cliniche7,8,9. In precedenza abbiamo sviluppato e segnalato sui metodi di rilevazione multiplex delle Echinoderm Microtubule Associated Protein come 4-Anaplastic linfoma recettore tirosina chinasi (EML4-ALK) fusione varianti da plasma sanguigno10. In questo studio, abbiamo esteso questi metodi per includere ordine superiore multiplex RNA target per ROS1 e RET, che copre otto varianti di fusione all'interno di ogni dosaggio. L'obiettivo era di sviluppare una tecnica rapida, sensibile, specifica e riproducibile per la rilevazione di queste varianti di fusione dal plasma di pazienti precedentemente diagnosticati con NSCLC.
Il processo di test viene avviato nell'ufficio di un medico utilizzando RNA sangue raccolta tubi11di stabilizzazione. Questi tubi contengono una cella conservante così come inibitori della RNAsi. I campioni sono spediti priorità durante la notte agli centralizzata College of American patologi CAP-accreditato/Clinical Laboratory miglioramento emendamenti (CLIA)-laboratorio certificato (laboratorio clinico) per l'elaborazione da parte di personale competente. Una volta ricevuto dal laboratorio clinico, ogni fase della lavorazione è condotta sotto approvato procedure operative Standard (SOP). Sangue intero viene centrifugata per recuperare il plasma, che viene quindi utilizzato per isolare il RNA che può essere di circolazione libera nel sangue o all'interno di incapsulamento moiety, ad esempio esosomi e piastrine7,8,9. Per isolare il RNA da questi compartimenti, abbiamo selezionato il sistema per il recupero di RNA basato sul confronto di diversi metodi di estrazione. il RNA isolato è concentrato e inverso trascritto a cDNA. Parecchi enzimi trascrittasi inversa e gli iniettori di gene-specific sono stati valutati durante l'ottimizzazione del metodo di sintesi del cDNA per massimizzare ROS1 e RET destinazione trascrizione conversione10. Questo è fondamentale per abbondanza bassa circolazione trascrizioni, quali varianti di fusione tumore-derivato. Infine, abbiamo ottimizzato dPCR concentrazioni di primer e sonde per consentire il rilevamento multiplex di RET o ROS1 varianti di fusione ed il gene di controllo, glucuronidase-β (GUSB). Quindi abbiamo combinato le migliori condizioni da ciascuno degli studi ottimizzazione in un protocollo finale bloccato prima di eseguire il convalida analitica studi descritti in questo rapporto. Questo protocollo e questi risultati forniscono la base per un flusso di lavoro rapido e sensibile per la rilevazione sistematica delle varianti di rara fusione in circolazione.
Protocollo
Le istruzioni dei fabbricanti sono seguite per i reagenti elencati di seguito, se non diversamente specificato. L'analisi di PCR sono prodotti disponibili in commercio progettati per rilevare le fusioni ROS1 e RET.
1. lavorare con RNA in preparazione per trascrizione inversa (RT)-dPCR: laboratorio consigliate
- Creare un ambiente RNAsi-libera quando si lavora con RNA.
- Utilizzare spray disponibili in commercio progettato per inattivare le RNasi contaminanti.
- Uso certificato RNAsi-libera i reagenti, punte e tubi. Utilizzare puntali per pipettatori per impedire l'introduzione delle RNasi o contaminazione dei campioni.
- Indossare sempre un camice da laboratorio per impedire la caduta dall'abbigliamento in vostro campione di particolato. Designare un laboratorio cappotto specifico da utilizzare con elaborazione del RNA.
- Indossare guanti per prevenire la contaminazione del campione da RNasi in pelle. Cambiare spesso i guanti.
Nota: Si supponga di superfici di laboratorio sono contaminate con RNAsi, poiché sono esposti all'ambiente. Guanti che contattare capelli, pelle, ecc. , maniglie delle porte, maniglie del congelatore, penne/marcatori vengono considerati per non più essere RNAsi-libera. - Decontaminare pipettatori, benchtops, centrifughe e altre superfici di lavoro con una RNasi inattivazione spray prima dell'uso.
- Se possibile, mantenere un set di attrezzatura per utilizzare solo con RNA.
- Minimizzare le interruzioni del flusso d'aria nelle aree di laboratorio quando si lavora con campioni di RNA per evitare che particelle da cadere in campioni o contaminare l'area di lavoro.
- Archivio purificato RNA a-80 ˚ c.
- Evitare più gelo-disgelo dei campioni del RNA, in quanto ciò può causare la degradazione.
2. generazione di analitica RNA materiale per i controlli positivi
- Progettare il DNA sintetico utilizzando sequenze di mRNA pubblicati per varianti di fusione di interesse10.
- Per una variante di fusione specificato, selezionare una sequenza di fusione mRNA che include il sito fusion plus lunghezza sufficiente che fiancheggiano su ciascun lato per coprire l'amplicone PCR.
- Selezionare sequenze nucleotidiche tra 50-250 nt per imitare la dimensione del RNA catturato usando plasma arricchito di piastrine di circolazione.
- Aggiungere una sequenza del promotore T7 (5'-CAGAGATGCATAATACGACTCACTATAGGGAGA-3') all'estremità 5' della sequenza di destinazione.
- Ordinare sequenze sintetiche come frammenti incagliato doppio dell'acido deossiribonucleico (DNA).
- Ricostituire il DNA sintetico in tampone Tris-EDTA (TE) ad una concentrazione finale di 10 ng / µ l.
- Convertire 60 ng DNA sintetico di RNA mediante trascrizione in vitro .
- Purificare i trascritti di RNA mediante reagente fenolo/guanidina-base12.
- Includono la dnasi I, RNAsi-libera per rimuovere residui modello DNA.
- Misurare la concentrazione di purificato in vitro RNA usando un fluorometro commercialmente disponibile con standard e RNA specifici coloranti. Garantire che RNA è all'interno della gamma accettabile per gli standard selezionati. Diluizione può essere richiesto.
- Confermare successo trascrizione mediante elettroforesi in gel utilizzando un gel di agarosio al 2% mescolato con RNA gel macchia e una gamma alta scaletta di RNA compreso intervallo di grandezza di nt 50-250.
- Carico 500 ng di ogni in vitro RNA in un gel.
- Esegua il gel a 5 V/cm.
- Visualizzate le singole fasce usando l'illuminazione e documentare i risultati.
- Confermare dimensioni di trascrizione previsto per ciascuna delle varianti fusione (basate sul progetto al punto 2.1.2).
- Confermare il rilevamento di ogni in vitro RNA da RT-dPCR usando analisi PCR specifica variante abbinato (vedere passaggi 5-8 del presente protocollo).
- Optional: Preparare una miscela equimolare di in vitro RNA che contiene tutte le varianti di fusione e il gene di controllo GUSB.
- Se viene eseguita passo 2.9: confermare il rilevamento di ognuna delle varianti fusione incluse nella miscela di controllo da dPCR usando le analisi PCR di variante specifica (vedere i passaggi 5-8 del presente protocollo).
- Determinare la concentrazione desiderata ingresso per analitici controlli positivi testando concentrazioni che vanno da 0,25 a 2,5 fg10. Scegliere concentrazione basate sull'output di numero di copie richiesto.
- Dopo la conferma, è possibile preparare le aliquote monouso a 10 µ l di RNA analitica per il controllo positivo (punto 4.4) e conservare a-80 ° c.
3. donatore esemplari
- Raccogliere campioni di sangue umano intero 10 mL in provette da 10 mL (BCT) contenente un conservante di RNA senza cellula.
Nota: Tutti i donatori umani consentiranno a uso di ricerca e nessuna informazione di identificazione del donatore-specific dovrà essere raccolti o utilizzate durante i test. - Processare i campioni di sangue intero entro il periodo indicato dal costruttore del BCT.
- Pool di plasma umano normale può essere acquistato da una fonte commerciale per l'utilizzo all'interno del controllo analitico positivo. Preparare monouso, aliquote di 1 mL del pool di plasma umano normale e conservare a-80 ° c da utilizzare con il controllo positivo (punto 4.4).
4. il recupero di RNA dal Plasma di circolazione
Nota: È importante lavorare rapidamente durante questa procedura.
- Centrifugare le provette di sangue intero a 200 x g per 20 min.
- Raccogliere fino a 4 mL di plasma dal tubo di raccolta sangue centrifugato utilizzando una pipetta sierologica. Fare attenzione a non disturbare o aspirare lo strato del buffy-coat.
- Isolare la circolazione RNA usando un kit disponibile in commercio che possono catturare senza cellula di RNA dal plasma, piastrine ed esosomi. Isolare gli RNA da campione di controllo positivo a fianco di ogni lotto.
-
Preparare il controllo positivo per ogni batch di campioni clinici come segue:
- Scongelare 1 mL in pool plasma umano normale aliquota (punto 3.3).
- Scongelare aliquota di 10 µ l analitica RNA (passo 2.12).
- Preparare controllo positivo aggiungendo 10 µ l analitica RNA nel campione di plasma umano normale una volta che l'etanolo è stato aggiunto al plasma lisato.
- Eluire campioni con acqua priva di nucleasi di 100 µ l. Procedere immediatamente con RNA ripulire e concentrazione.
- Campioni possono essere conservati nel ghiaccio bagnato e coperto, fino a un'ora.
- Concentrarsi RNA usando il metodo della colonna basata ed eluire in 9 µ l di acqua RNAsi-libera.
- Procedere immediatamente al passaggio 5, o mantenere i campioni su ghiaccio bagnato fino a un'ora.
5. retrotrascrizione di RNA in cDNA
- Convertire concentrato circolo campione del RNA in cDNA utilizzando un kit di reazione di trascrizione inversa commercialmente disponibili, tra cui primer casuale (vedere tabella 1 per i componenti).
Nota: Gene primers specifici sono facoltativi e possono essere progettati per le varianti di test. Gli iniettori sono progettati in base sul bersaglio sequenza di RNA. Utilizzare sequenze variante di fusione dal punto 2.1.- Nessun campione di controllo di trascrittasi inversa e nessun campione di controllo di RNA sono (Vedi tabella 1).
- Isolare il cDNA da reazione di retrotrascrizione usando una colonna di spin di concentratore DNA commercialmente disponibile.
Nota: Questo passaggio facilita la rimozione di enzimi, primer e gratuito deossinucleotidi trifosfati (dNTPs). - Utilizzare immediatamente cDNA in reazioni di PCR o conservare a-80 ° c.
6. digital PCR
Nota: La PCR è specifico di gocciolina digitale PCR (Vedi Tabella materiali).
-
Precauzioni di mix PCR.
- Indossare un cappotto e nitrile guanti di laboratorio monouso.
- Uso PCR mescolare reagenti in una zona di preparazione di reagenti dedicati. Non maneggiare cDNA nell'area preparazione solo reagente.
- Coprire i sonde mentre lavorava per proteggerli dalla luce. Luce eccessiva può foto candeggina il colorante fluorescente collegato alla sonda.
- Trasporto mescola, coperto e al riparo dalla luce, in una zona di pre-amplificazione separata prima del cDNA deve essere aggiunto.
- Aggiungere cDNA da sottoporre al mix PCR in un cappuccio PCR pulito situato nella zona di pre-amplificazione.
- Preparare miscele PCR per un volume di reazione finale di 20 µ l secondo la tabella 2.
- Distribuire le miscele PCR + cDNA per piastre PCR.
Nota: Uso di un layout di piatto come una guida è consigliato. - Coprire la piastra con un foglio sigillante rimovibile.
- Centrifugare le piastre brevemente per raccogliere campioni presso il fondo dei pozzetti.
- Mix su agitatore per piastre su un'impostazione bassa per 10 s.
- Centrifugare le piastre brevemente per raccogliere il campione nella parte inferiore del pozzo.
- Rimuovere il foglio protettivo. Eseguire la generazione di goccia per mix di PCR-cDNA sia con un sistema di generazione di gocciolina manuali o automatizzati.
- Per la generazione manuale gocciolina, trasferimento miscela PCR 20 µ l a pozzetti dei campioni della cartuccia di generazione della gocciolina. Aggiungere 70 µ l di olio di generazione della gocciolina. Coprire con cartuccia di guarnizione e trasferimento di gomma al generatore di gocciolina manuale per inizializzare la generazione della gocciolina. Dopo generazione gocciolina, trasferire le goccioline di un fresco piatto PCR utilizzando suggerimenti raccomandati dal produttore. Aspirare e dispensare le goccioline lentamente, nel corso di 5 a 6 s ciascuno, senza toccare l'apertura della punta per la cartuccia di goccia o la piastra.
- Per generazione automatizzata della gocciolina, sigillare la piastra con un foglio di sigillatura e trasferimento al generatore della gocciolina. Garantire tutti i suggerimenti, le cartucce, e piastre sono a posto prima di iniziare la generazione della gocciolina.
- A seguito di generazione gocciolina e trasferimento di goccioline in un fresco piatto PCR, sigillare con un foglio sigillante lamina e le piastre di ciclo termico utilizzando le impostazioni nella tabella 3.
- Dopo il termociclatore run è completo, leggere la piastra utilizzando un lettore di goccia. Creare un layout di piastra per software reader che identifica la posizione di controlli, campioni, ecc.e di carico in software per iniziare la lettura.
7. analisi dei dati e la revisione e la generazione di risultati
- Analizzare la piastra leggere risultati utilizzando il software disponibile in commercio.
- Passare al menu Analyze per visualizzare grafici bidimensionali (2D) ampiezza.
- Valutare la qualità complessiva dei dati esaminando i dati di goccia.
- Valutare i dati per i numeri di evento accettato totale utilizzando il menu eventi. Se ci sono meno di 10.000 eventi per pozzetto, valutare con attenzione i dati per ulteriori problemi.
- Verifica dati per ampiezze di fluorescenza aberrante. Differenze di ampiezza significativa e le differenze di concentrazione tra campioni replicati indicano scarsa manipolazione o miscelazione di campioni.
- Rendere note di cluster gocciolina con modelli di spruzzo su un asse di 45 gradi, che è indicativo di goccioline di scarsa qualità o campioni problematici.
- Esaminare i dati di controllo positivo, No Reverse Transcriptase (RT No) e nessun RNA controllo (NRC) prima. Selezionare tutti i campioni di controllo ed esaminare qualità cluster di trama 2D. Per soglia adeguata, una netta separazione tra i cluster gocciolina dovrebbe essere evidente.
- Per ogni variante di analisi, impostare la soglia basata su pozzetti di controllo.
- Impostare le soglie su trame 2D utilizzando lo strumento mirino per separare la popolazione di gocciolina di doppia negazione dalla popolazione del gene di controllo (etichettato con 5'-esacloro-fluorescina-CE phosphoramidite sonda), asse y e popolazione variante genica, se presentano ( etichettato con fluorescina amidite sonda 6-carboxyfluorescein OR), asse x.
- Copie di somma da ogni pozzetto replica per un singolo campione.
- Esprimere i risultati di test come il numero di copie variante rilevato.
Nota: Per determinare il valore di cut-off analitico per la chiamata di un campione positivo o negativo, eseguire un normale campione di donatore sano impostare (almeno 10 singoli campioni) attraverso il processo finalizzato e stabilire la frequenza di taglio sopra qualsiasi segnale di fondo rilevabile per la mutazione di interesse. Inoltre, stabilire il numero di copie del gene di controllo necessari per chiamare un risultato positivo o negativo. Questo cut-off del gene di controllo funziona come un controllo interno di qualità (QC) per valutare la quantità e la qualità di ciascun campione di RNA che viene elaborato.
8. Verifica delle condizioni di reazione di RT-dPCR utilizzando linee cellulari (opzionale)
- Per verificare il rilevamento delle varianti di fusione, utilizzare commercialmente disponibili linee cellulari che esprimono la fusione ROS1 o RET mRNA di interesse. Procedere come segue:
- Omogeneizzare celle flash-congelato in una soluzione di base di guanidinium Lisi direttamente allo stato congelato. Anche breve lo scongelamento prima dell'omogeneizzazione può causare perdita e degradazione del RNA.
- Isolare il RNA utilizzando colonne di spin di silice-membrana progettate per RNA.
- Misurare la concentrazione di campioni di RNA usando un fluorometro con standard e reagenti specifici RNA.
- Diluire il RNA isolato in uno sfondo di selvaggio-tipo RNA da plasma o un'altra origine commerciale.
- Eseguire i punti da trascrizione inversa di RNA di cDNA, PCR digitale e l'analisi dei dati e revisione, e generazione di risultati elencati nel presente protocollo per confermare il rilevamento della variante desiderata.
Risultati
Questo protocollo descrive un sistema di test sviluppato per il rilevamento di varianti di fusione di RNA per uso nella misura di mutazioni driver all'interno del plasma di pazienti con NSCLC (Figura 1A). MRNA di fusione prodotti dall'espressione dei riarrangiamenti RET e ROS1 più comuni nella popolazione NSCLC sono stati identificati13,14,15,16,17. Analisi di PCR multiplexate quindi sono state progettate per rilevare le otto varianti di trascrizione più comuni per ogni destinazione in NSCLC all'interno di una singola reazione. Le traslocazioni più comuni a livello del locus ROS1 generano associazioni con le 5' porzioni di CD74, SDC4, SLC34A2, EZR o TPM3 geni (Figura 1B). Le traslocazioni più comuni a livello del locus RET portano a giustapposizione con KIF5B, per cui il dosaggio copre sei giunzioni esone. Ulteriori partner RET che vengono analizzati sono quelli con CCDC6 e TRIM33 (Figura 1C). In totale, i saggi coprono circa l'88% di ROS1 e il 99% delle alterazioni di RET notoriamente che si verifica nella popolazione di pazienti NSCLC17.
La specificità dei componenti analisi valutata per prime utilizzando otto singoli in vitro RNA che contengono la sequenza di mRNA per le trascrizioni di fusione coperte dalla ROS1 o RET multiplexed saggi. Ogni specie di RNA è stato testato contro ogni singolo dosaggio variante che comprende le versioni multiplex. Non c'era nessuna reattività crociata di questi saggi, dimostrando in tal modo 100% specificità analitica all'interno il progettato multiplexed saggi (dati non mostrati). Per determinare il limite inferiore di rilevamento del test di protocollo, derivato da linee cellulari che esprimono una variante di fusione inclusa nell'analisi di RNA totale sono stati mescolati in uno sfondo di RNA normale alle concentrazioni di 5%, 1%, 0,2% e 0,04%. Il multiplex RET e ROS1 variante analisi PCR rilevate poco come variante di fusione di 0,2% (Figura 2A-B). Inoltre, una preparazione di 5% fuori bersaglio linea cellulare derivata RNA (che esprimono una trascrizione di fusione di EML4-ALK) non è stato rilevato con le analisi multiplexate ROS1 e RET, dimostrando ulteriormente la specificità (Figura 2A-B).
Test di precisione del processo di RT-dPCR è stata eseguita per entrambi ROS1 e RET. Analytic materiale di controllo composto da equimolare in vitro RNAs è stato elaborato a tre concentrazioni (alta, media e bassa) attraverso la trascrizione inversa e dPCR su tre diverse occasioni entro il giorno stesso (intra-day), per tre giorni consecutivi (Inter-day) e con due operatori (inter-operatore). Risultati da test di precisione hanno dimostrato un rilevamento accurato di sia la trascrizione di fusione di interesse, nonché un gene di controllo, GUSB, che è incluso come una metrica interna di QC (Figura 2-D).
Oltre al controllo interno GUSB, ogni batch di campioni clinici è stato eseguito con un insieme di comandi batch. Un controllo positivo è stato sviluppato da una miscela di analitica in vitro RNA che rappresentate ognuna delle varianti fusione testate in RT-dPCR, nonché analitica in vitro RNA per GUSB. Questo RNA è stata aggiunta in plasma umano normale lisato durante l'estrazione di RNA ed è stato elaborato insieme agli esempi clinici in tutto il protocollo. Nessun controllo della trascrittasi inversa (RT No) è stato un controllo negativo per confermare l'assenza di contaminante materiale del workflow di estrazione di RNA e dimostrare la specificità dei primer di RNA. Il controllo di n RT è stato generato utilizzando lo stesso materiale come controllo positivo, ma non include degli enzimi all'interno della reazione di sintesi del cDNA. Il nessun controllo di RNA (NRC) è un controllo negativo per confermare l'assenza di contaminanti trascrizioni nei componenti di reazione di trascrizione inversa. Questo controllo è stato introdotto nel flusso di lavoro nella fase di sintesi del cDNA e acqua è stato aggiunto nella reazione invece di un modello di RNA. I controlli No RT e NRC devono essere negativi in entrambi i canali, se risultati accurati sono da consegnare. La tabella 1 elenca i componenti di reazione di trascrizione inversa per ogni controllo. Sono riportati alcuni esempi delle trame 2D per ognuno di questi controlli per il ROS1 (Figura 3 A-C) e saggi multiplex RET (Figura 3 E-G). Varianti di fusione sono stati rilevati usando una sonda di amidite (FAM) della fluorescina e sono rappresentati lungo l'asse y, mentre il gene di controllo, GUSB, è stato rilevato usando una sonda 5'-esacloro-fluorescina-CE phosphoramidite (HEX) e sull'asse x. Questi controlli batch sono stati valutati nel corso di 21 giorni per determinare il dosaggio robustezza. GUSB controllo gene gocce e goccioline positive di fusione sono stati osservati per ROS1 e RET in tutti i 21 piste eseguite nel corso dello studio (Figura 3S, H). Tutti i controlli negativi (No RT e NRC) ha dato i risultati negativi attraverso l'intero 21 giorni (dati non mostrati).
La capacità di risolvere i problemi è una componente fondamentale di qualsiasi protocollo di test da eseguire nell'impostazione del laboratorio clinico. Qui, forniamo esempi reali di risultati non ottimali utilizzando il protocollo RT-dPCR. Il primo è un esempio di grafico 2D che dimostra l'importanza del non controllo della trascrittasi inversa (Figura 4A). In questo esempio, mutante goccioline positive erano presenti anche se non c'era nessuna conversione di cDNA dovuta alla mancanza dell'enzima. Questo risultato era probabilmente dovuto dPCR primer di amplificazione del DNA genomico fuori bersaglio. In questo caso, progettazione di un test che misurano introne impedirà l'amplificazione del DNA genomico. In alternativa, un enzima dnasi RNAsi-libera può essere utilizzato per eliminare il DNA contaminante, ma questo non è consigliato per la rilevazione degli obiettivi rari, come qualche degradazione del RNA può verificarsi durante l'incubazione con l'enzima. Il prossimo esempio grafico 2D era un NRC con le goccioline di positive in entrambi i canali (Figura 4B). Ciò ha indicato la contaminazione a un certo punto nel setup del RT-dPCR. In questo caso, la raccomandazione è di scartare qualsiasi potenzialmente contaminati i reagenti utilizzati nel test, accuratamente decontaminare tutte le attrezzature e ripetere il test con componenti di reazione fresco. Il terzo esempio 2D grafico presentato come uno spray di goccioline lungo una linea di 45° (Figura 4C). Questo è spesso causato da tosatura e la coalescenza delle goccioline. Gocciolina attenta gestione prima del ciclo termico è essenziale, poichè le goccioline sono soggette a danni. Si consiglia l'uso di generazione automatizzata della gocciolina, quando disponibile. Se trasferimento manuale generato goccioline, essere certi di scegliere i suggerimenti consigliati di wide-bore e impiegano un'attenta tecnica di dispensazione. Gocciolina trasferimento richiede lenta aspirazione e erogazione, con ciascuna che si svolgono in 5-6 secondi, ed è essenziale che l'apertura di punta della pipetta non toccare la cartuccia di goccia o bene. Durante la dispensazione, mantenere la punta della pipetta al livello liquido e sollevare lentamente come le goccioline sono dispensati (Vedi video per dimostrazione). Nell'esempio di trama 2D finale dimostra una mancanza di separazione tra le popolazioni di positivi e negativi della gocciolina (Figura 4). Questo può avere diverse cause. Inibitori della PCR di forte, ad esempio i detergenti utilizzati in buffer di lisi e un eccesso di DNA altamente degradato, può causare la perdita di separazione. In questo caso, prendere in considerazione l'aggiunta di un passaggio di pulizia tra sintesi di cDNA e dPCR (come descritto nel punto 5 del presente protocollo). Infine, mancanza di separazione può anche essere a causa di condizioni di amplificazione sub-ottimale e ottimizzazione del passaggio del PCR dovrebbe anche essere considerata.
Dati all'interno del paziente di Figura 5 rappresentano 984 mondo reale campione tempi di turn-around e dimostra la natura rapida di questo flusso di lavoro di prova. Risultati sono stati segnalati per il medico curante come presto come entro 48 ore (79% dei casi) dal ricevimento del campione e nel 95% dei casi, entro 72 ore. In conclusione, l'uso di stabilizzato circolanti provette per prelievo ematico RNA, ottimizzato le procedure di estrazione di RNA da sangue e RT-dPCR eseguire secondo un protocollo ottimizzato con l'appropriato interno e controlli batch, può fornire un sistema di test rapido per la rilevazione accurata delle varianti di RNA fusione pertinenti in NSCLC.
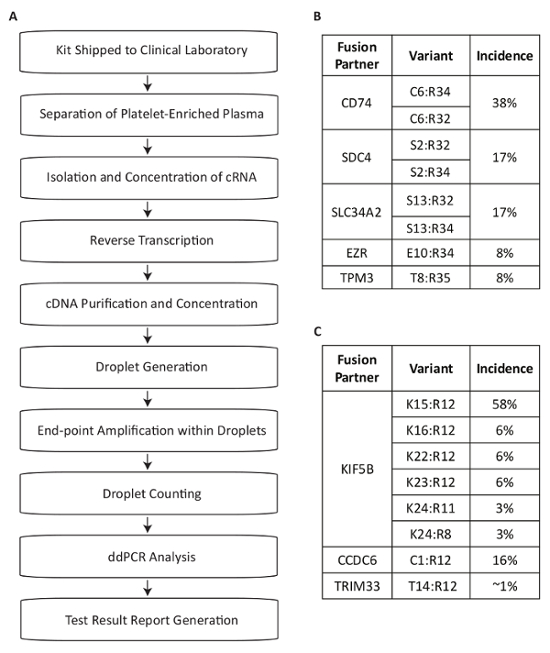
Figura 1 : Panoramica di sangue campione passaggi di elaborazione per rilevazione variante Fusion utilizzando dosaggi specifici per la più prevalente RET e ROS1 varianti in NSCLC. (A) campione di prova viene avviato quando viene disegnato il sangue intero e una BCT è spedito entro il kit di raccolta campioni per il laboratorio clinico. RNA di circolazione viene recuperato da più fonti entro il plasma arricchito di piastrine, reverse trascritto con innesco specifico gene e purificata per uso in dPCR. I campioni vengono elaborati utilizzando un sistema disponibile in commercio che consiste della generazione della gocciolina (emulsione), amplificazione e gocciolina di conteggio. Dati vengono analizzati utilizzando il software disponibile in commercio. I risultati del test sono quindi documentati e segnalati al medico richiesta di prova. Il processo è progettato per funzionare entro un lasso di tempo di 72 ore dal ricevimento del campione a rilascio di risultato. Otto varianti per ROS1 (B) e (C) RET sono contemplati i saggi multiplexati. Tratto dal sito Web di Biodesix con permesso. Clicca qui per visualizzare una versione più grande di questa figura.
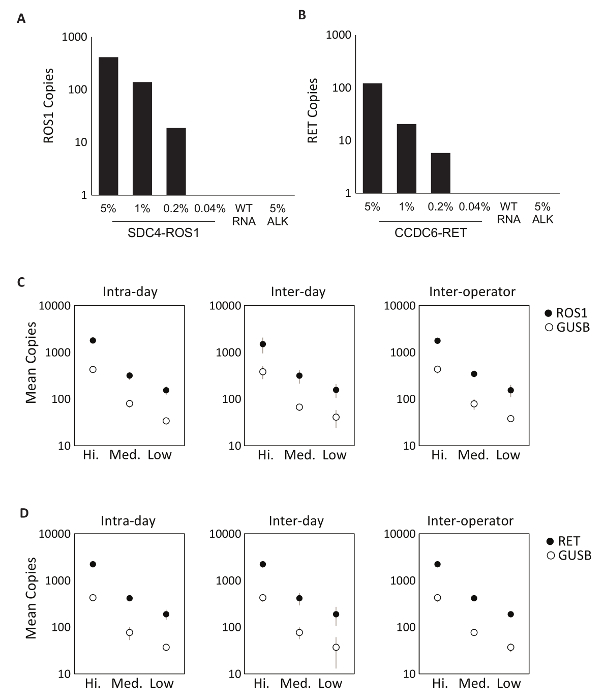
Figura 2 : Convalida analitica. Linee cellulari che esprimono (A) SDC4-ROS1 fusione e fusione (B) CCDC6-RET sono stati diluiti in una priorità bassa di RNA totale umano a wild type (WT RNA). Con ogni variante di fusione, il limite di rilevazione è stato stabilito a 0,2% frequenza variante utilizzando criteri pre-definiti per ogni dosaggio variante. Tutti i campioni sopra questa soglia contenevano anche almeno 21 copie del gene di controllo. Standard di EML4-ALK (ALK) 5% in una priorità bassa di selvaggio-tipo RNA è stato testato per dimostrare la specificità dell'analisi, che è stata confermata da un risultato negativo. Analitico standard multiplex di RNA sono stati misurati ad alto, medio, e concentrazioni basse per ROS1 (C) e (D) RET. precisione è stata valutata in tre esecuzioni nello stesso giorno (Intra-day), tre piste per tre giorni consecutivi (Inter-day) e con due operatori indipendenti (inter-operatore). I mezzi di numero di copie e le deviazioni standard sono indicati. Tratto dal sito Web di Biodesix con permesso. Clicca qui per visualizzare una versione più grande di questa figura.
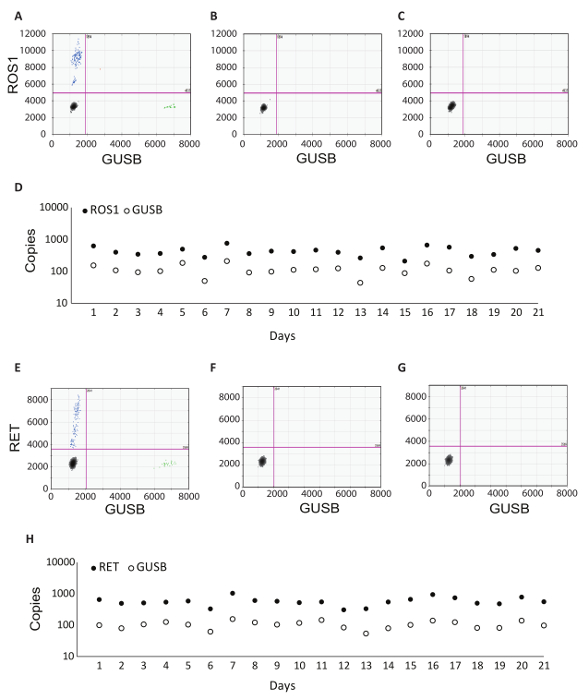
Figura 3 : Elaborazione dati di robustezza e degli esempi dei controlli in batch. Trama 2D del dPCR di dosaggio ROS1 multiplexata non risultati per controllo positivo (A), (B) nessun controllo della trascrittasi inversa e (C) nessun controllo del modello di RNA. (D) i controlli sono stati eseguiti su 21 giorni consecutivi (esclusi i weekend e le festività). Copia numero + /-deviazioni standard medio per ROS1 positivo controllo erano 439 + /-141. Nessun trascrittasi inversa e nessun controllo di modello sono stati anche eseguito ogni giorno, e questi erano tutti negativi (dati non mostrati). 2D trama della RET multiplexata dosaggio dPCR non risultati per controllo positivo (E), (F) nessun controllo della trascrittasi inversa e (G) nessun controllo del modello di RNA. (H) i controlli sono stati eseguiti su 21 giorni consecutivi (esclusi i weekend e le festività). Dire copie + /-deviazioni standard per RET positivi di controllo erano 586 + /-182. Vengono mostrati non nessun trascrittasi inversa e senza controlli del modello che sono stati anche eseguiti ogni giorno ed erano tutti negativi. Tratto dal sito Web di Biodesix con permesso. Clicca qui per visualizzare una versione più grande di questa figura.
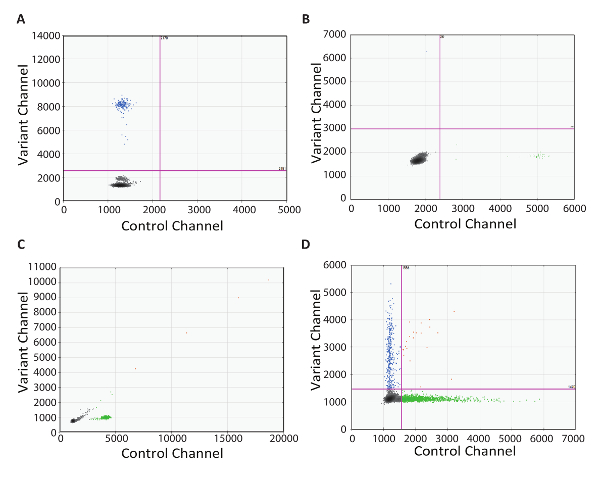
Figura 4 : Risoluzione dei problemi RT-dPCR. 2D grafici che rappresentano dPCR sub-ottimali risultati ottenuti quando c'è (A) contaminazione all'interno di nessun controllo della trascrittasi inversa, (B) contaminazione all'interno di nessun controllo di RNA, (C) tosatura e la coalescenza delle goccioline e (D ) scarsamente ottimizzato condizioni di PCR o inibizione di PCR. Clicca qui per visualizzare una versione più grande di questa figura.
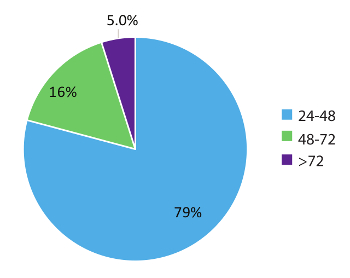
Figura 5 : Tempo di consegna (TAT). TAT (in ore) è stato compilato per test richiede una variante di RNA (n = 984). Dati escludono i fine settimana, festivi e campioni conservati per > 24 h incompletezza dei dati clinici su moduli di richiesta di Test di laboratorio. Clicca qui per visualizzare una versione più grande di questa figura.
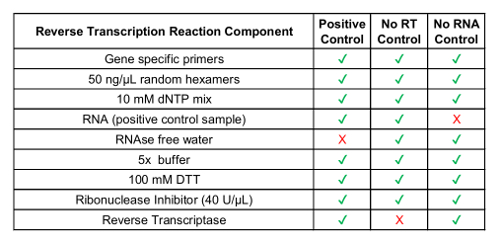
Tabella 1: Preparazione dei reagenti di trascrizione inversa per controlli di processo.
| Componente | Volume |
| 2 x dPCR supermix per sonde (no 2'-deoxyuridine 5'-trifosfato) | 10 Μ l |
| 20 x destinazione variante Primer/sonde set (450 nmol/L gli iniettori, sonda di FAM 250 nmol/L) | 1 Μ l |
| set di primer/sonda di controllo destinazione: 20x (450 nmol/L gli iniettori, sonda HEX 250 nmol/L) | 1 Μ l |
| acqua priva di nucleasi | 1 Μ l |
| cDNA | 7 Μ l |
Tabella 2: Preparazione del master mix per dPCR.
| Passaggio in bicicletta | Temperatura | Tempo | # Cicli | Velocità di rampa |
| Attivazione degli enzimi | 95 ° C | 10 min | 1 | ~ 2 oC/s |
| Denaturazione | 94 ° C | 30 s | 40 | |
| Ricottura/estensione | 55 ° C | 1 min | ||
| Disattivazione degli enzimi | 98 ° C | 10 min | 1 | |
| Tenere premuto (opzionale) | 4 ° C | infinito | 1 | ~ 1 oC/s |
Tabella 3: Condizioni termiche in bicicletta.
Discussione
Riarrangiamenti di RET e ROS1 compongono insieme ~ 3% delle mutazioni driver all'interno della popolazione di NSCLC18. Anche se rara, la rilevazione di queste alterazioni genetiche è vitale. Pazienti con NSCLC con queste alterazioni possono beneficiare di terapie mirate che specificamente inibiscono l'attività della chinasi aberrante che deriva dalla onco-proteine13. Alcuni tali terapie già approvate dal FDA per l'uso in ROS1 NSCLC positivo, mentre gli altri sono stati dimostrati per essere efficace contro RET in studi clinici19.
Tecnologia digitale di PCR fornisce la sensibilità che è ideale per applicazioni di biopsia liquida20. C'è stato significativo adozione di questa tecnologia per l'utilizzo con circolazione DNA libero per la misurazione delle mutazioni del tumore in pazienti con NSCLC4,6,21,22,23 . Oltre a cfDNA, abbiamo sviluppato un protocollo progettato per misura robusta delle varianti fusione più prevalente in pazienti con NSCLC da circolanti tumore RNA (Figura 1A)10.
Il nostro protocollo stabilito consente per i limiti analitici di rilevazione fino a 0,2% (Figura 2). Mentre la RT-dPCR è eccezionalmente sensibile e specifico, i dosaggi sono limitati al pannello delle varianti di fusione conosciuto che sono scelti e multiplex per il rilevamento nell'analisi di PCR. Così, fusioni da includere nelle analisi multiplexate devono essere accuratamente selezionate per garantire una copertura adeguata all'interno della popolazione dei pazienti con NSCLC. Abbiamo progettato correttamente le analisi per RET e ROS1 contemporaneamente rilevare otto risultante di varianti di fusione da riarrangiamenti di RET o ROS1 loci e coprire il 99% e l'88% della popolazione positiva RET e ROS1, rispettivamente (Figura 1B-C )17.
Il flusso di lavoro di prova finale come descritto in questo studio include controlli batch per garantire la coerenza dei risultati. Tali controlli includono sia positivo standard analitici, nonché due controlli negativi, che insieme garantiscono non c'è nessuna contaminazione o inibizione di PCR che si verificano all'interno del batch (Figura 3). Per garantire la robustezza del dosaggio, uno studio è stato effettuato usando i comandi batch su un periodo di 21 giorni (Figura 3D, H). Questi dati dimostrano la coerenza del processo RNA come stabilito nell'ambito di questo protocollo.
Buone pratiche di laboratorio e gestione adeguata di RNA sono componenti chiave di garantire risultati affidabili e accurati. Spazio-laboratorio e attrezzature dedicate per utilizzare con RNA, pulizia delle attrezzature dopo ogni uso, utilizzando materiali di consumo e reagenti RNAsi-libera e l'applicazione di uno spray di inattivazione RNasi per lo spazio di lavoro tutti contribuire a ridurre la contaminazione RNasi. Coscienzioso manipolazione dei campioni di RNA dai tecnici, tra cui un camice da laboratorio dedicato, frequenti cambiamenti di guanto, lavorando rapidamente attraverso la procedura di estrazione di RNA, e mantenere i campioni su ghiaccio sono della massima importanza per preservare l'integrità del campione. Una volta che il RNA è stato inverso trascritto in cDNA, il campione è in una forma più stabile che è meno soggetta a degradazione. Oltre alle pratiche che supportano l'integrità RNA, campioni e componenti PCR dovrebbero essere mantenuti in zone segregate per evitare contaminazioni che potrebbero portare a risultati falsi positivi. Il magazzino reagenti PCR e la preparazione di miscele master PCR dovrebbero essere tenuti separati dai modelli di PCR e dovrebbe prestare molta attenzione a separare il modello amplificato (post-PCR) da tutti i materiali pre-amplificati, inclusi campioni di cDNA, RNA e reagenti. Infine, corretta generazione e manipolazione di miscele PCR emulsionate prima dell'amplificazione è fondamentale per mantenere l'integrità della gocciolina e dPCR ottimali condizioni. Precauzioni come questi sono fondamentali durante l'esecuzione del presente protocollo per ottenere risultati coerenti e accurati. Tutti i dati dovrebbero essere esaminati da personale addestrato prima del rilascio di risultati per essere sicuri che sono state rispettate tutte le metriche di QC. Nel caso di risultati non ottimali (Figura 4), il batch deve essere esaminato da personale tecnico e il direttore del laboratorio e può richiedere la ri-elaborazione.
RT-dPCR risultati possono essere prodotte più presto 24 ore dal ricevimento del campione e il 95% dei risultati dei campioni all'interno del test set usati in questo studio (n = 984) sono stati segnalati al medico di ordinazione in meno di 72 ore dal momento della ricezione (Figura 5). Questo tempo fornisce al medico con tanto bisogno di informazioni molecolari in un lasso di tempo che permette l'inizio della terapia appropriata. Questi risultati sono in genere disponibili prima di quelli ottenuti con una biopsia del tessuto convenzionale. Ulteriori biomarcatori per NSCLC e altri tipi di cancro potrebbero essere sviluppati utilizzando approcci simili circolanti RNA-ha basato e beneficerebbero gli stessi tempo-per-risultati rapidi. Ad esempio, misura della trascrizione programmata morte Ligand 1 (PD-L1) mRNA mediante RT-dPCR potrebbe informare i medici sulle opzioni di immunoterapia. C'è anche un crescente interesse per l'utilità della biopsia liquida e dPCR nel monitoraggio efficacia terapeutica. Indicazioni precedenti della ricomparsa del tumore usando test per specifiche varianti genomiche potrebbero consentire ai medici di regolare i regimi terapeutici prima di pazienti sono sintomatici di standard di misure di cura ad esempio imaging24. Protocolli come quello riportato in questo studio sono ideali per il monitoraggio a causa loro non invasività, sensibilità, tempo di ritorno rapido e rapporto costo-efficacia. Il dosaggio descritto nel presente documento fornisce risultati entro 72 ore dal ricevimento del campione, con tassi di rilevamento minimo di falsi positivi, che facilita le decisioni di trattamento rapido e aggira alcune limitazioni sperimentati con tissutali test4.
Il nostro protocollo e i dati dimostrano un sistema robusto per identificare varianti di RNA di abbondanza bassa così come il potenziale per mutazione emoderivato test nella pratica clinica. Per quei pazienti che non hanno un driver actionable mutazione identificata dalla rapida biopsia mirata liquida si avvicina come questo, l'aggiunta di più vasto genoma e proteoma test da tessuto e di sangue può fornire informazioni cliniche ancora più ampie per supportare la pianificazione del trattamento.
Divulgazioni
H.M., L.J., K.A. e G.A.P sono dipendenti di e detenere partecipazioni in H.M Biodesix, Inc., L.J. e G.A.P sono co-inventori su una domanda di brevetto depositata da Biodesix, un sistema di test diagnostico per l'individuazione di varianti genetiche in non a piccole cellule di circolazione di copertura cancro del polmone.
Riconoscimenti
Ringraziamo i nostri collaboratori, Stephen Jones, Nia Charrington, Dr. Dianna Maar e Dr. Samantha Cooper dal centro di biologia di Digital (Bio-Rad Inc. CA) per il loro design di analisi supportano; Nezar Rghei e Dr. Moemen Abdalla (Norgen Biotek, Canada) per la consulenza critica quando si ottimizza il protocollo di estrazione di RNA; e Shannon Campbell, Scott Thurston, Jeff Fensterer, Shannon Martello e Joellyn Enos per assistenza con test requisiti e monitoraggio commerciale.
Materiali
| Name | Company | Catalog Number | Comments |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1604071 |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1809353 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1604071 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1606077 |
| Phosphate Buffered Saline 1X, Sterile | Amresco | K812-500mL | 1446C189 |
| Phosphate Buffered Saline 1X, Sterile – 500 mL | Invitrogen | 10010023 | 1916C092 |
| RNase Zap (Life Tech) (250 mL) | Ambion | AM9780 | 353952 |
| Beta-Mercaptoethanol (BME) (250 mL) | CalbioChem | 6050 | W105B |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56054611 |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56238638 |
| Isopropyl Alcohol | VWR | 0918-4L | 2116C416 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 403648 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 288461 |
| DNase I | Thermo | K0441 | 371299 |
| QIAzol Lysis Reagent | Qiagen | 79306 | 54809699 |
| 20x TE buffer pH 8.0 | Alfa Aesar | J62388 | R13C548 |
| UltraPure Agarose | Invitrogen | 16500-100 | 552730 |
| 10x TBE buffer | Invitrogen | AM9863 | 353065 |
| Cell-Free RNA BCT | Streck | 218976 | 60110327 |
| Cell-Free RNA BCT | Streck | 218976 | 61900327 |
| Cell-Free RNA BCT | Streck | 218976 | 61480327 |
| Cell-Free RNA BCT | Streck | 218976 | 62320327 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 585849 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 588308 |
| Lysis Buffer | Norgen | 21205 | A5F61E |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 585848 |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 588309 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC186976 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188077 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188413 |
| Collection Tubes 500 pack | Zymo | C1001-500 | N/A |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 391657 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 392504 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 448001 |
| SuperScript IV Reverse Transcriptase | Life Technologies | 18090200 | 451702 |
| Qubit HS RNA Assay Kit (500) | Life Technologies | Q32854 | 1745264 |
| Qubit assay tubes (500) | Life Technologies | Q32856 | 13416Q311 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64031651 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64063941 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065740 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065741 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64079083 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64025320 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64052358 |
| gBlock KIF5B-RET K15:R12 | IDT | 151004172 | 4-Oct-16 |
| gBlock KIF5B-RET K16:R12 | IDT | 151004173 | 4-Oct-16 |
| gBlock KIF5B-RET K22:R12 | IDT | 151004174 | 4-Oct-16 |
| gBlock KIF5B-RET K23:R12 | IDT | 151004175 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R11 | IDT | 151004176 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R8 | IDT | 151004177 | 4-Oct-16 |
| gBlock CCDC6-RET C1:R12 | IDT | 151004178 | 4-Oct-16 |
| gBlock TRIM33-RET T14:R12 | IDT | 151004179 | 4-Oct-16 |
| RET exon 8 RT Gene Specific Primer | IDT | 150554385 | 28-Sep-16 |
| 5’-CTCCACTCACACCTG-3’ | IDT | 150554385 | 28-Sep-16 |
| RET exon 11 RT Gene Specific Primer | IDT | 150554384 | 28-Sep-16 |
| 5’-GCAAACTTGTGGTAGCAG-3’ | IDT | 150554384 | 28-Sep-16 |
| RET exon 12 RT Gene Specific Primer | IDT | 150554383 | 28-Sep-16 |
| 5’-CTGCCTTTCAGATGGAAG-3’ | IDT | 150554383 | 28-Sep-16 |
| gBlock CD74-ROS1 C6:R34 | IDT | 152324366 | 15-Nov-16 |
| gBlock CD74-ROS1 C6:R32 | IDT | 152324367 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R32 | IDT | 152324368 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R34 | IDT | 152324369 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R32 | IDT | 152324370 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R34 | IDT | 152324371 | 15-Nov-16 |
| gBlock EZR-ROS1 E10:R34 | IDT | 152324372 | 15-Nov-16 |
| gBlock TPM3-ROS1 T8:R35 | IDT | 152324373 | 15-Nov-16 |
| ROS1 exon 34 RT Gene Specific Primer | IDT | 152704983 | 21-Nov-16 |
| 5’-CCTTCCTTGGCACTTT-3’ | IDT | 152704983 | 21-Nov-16 |
| ROS1 exon 35 RT Gene Specific Primer | IDT | 152704985 | 21-Nov-16 |
| 5’-CTCTTGGGTTGGAAGAGTATG-3’ | IDT | 152704985 | 21-Nov-16 |
| ALK Gene Specific Primer | IDT | 140035422 | 26-Aug-16 |
| 5’-CAGTAGTTGGGGTTGTAGTCG-3’ | IDT | 140035422 | 26-Aug-16 |
| EML4-ALK Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| SLC34A2-ROS1 Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| CCDC6-RET Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| Human Brain Total RNA | Ambion | AM7962 | 1703548 |
| PrimePCR ddPCR Expert Design Assay: K15:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K16:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K22:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K23:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R11 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R8 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C1:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: T14:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: E10:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: T8:R35 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| (version 2) | Bio-Rad | 12003909 | 213939881 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 13-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 20170112v3.2 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851151 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 207383915 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 195995635 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851152 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 213949301 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 20160914 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 211383227 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 1065C220 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052953 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052358 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 1065C320 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64052952 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64064127 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000065883 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084276 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000079928 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084395 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084634 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20160627 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161107 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161206 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161216 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20170125 |
| Pipet Tips for Automated Droplet Generator | Bio-Rad | 1864120 | PR125340 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206894 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206893 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 1409850 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 100402 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 145851 |
| Microseal 'B' seals | Bio-Rad | MSB1001 | BR00428490 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64039089 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049253 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049255 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64081870 |
| DNA Lo Bind Tube 0.5 mL | Eppendorf | 22431005 | E1629620 |
| DNA Lo Bind Tube 1.5 mL | Eppendorf | 22431021 | F16698K |
| DNA Lo Bind Tube 2 mL | Eppendorf | 22431048 | E160610I |
| 50 mL Conicals, Polypropylene (25) | Thermo | 339652 | G5ZF5W8118 |
| TempAssure PCR 8-Strips, Optical Caps, Natural, polypropylene (120) | USA Scientific | 1402-4700 | 16202 |
| For Rainin LTS Pipettors 0.5-20 µL tips | Pipette.com | LF-20 | 40155-642C4-642C |
| For Rainin LTS Pipettors 5-200 µL tips | Pipette.com | LF-250 | 40154-642C4-642B |
| Tips LTS 200 ul Filter 960/10 RT-L200F (10 boxes) | Rainin | 17002927 | 1635 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F1175551-1108 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F118054L-1720 |
| Pipet Tips, 100 ul TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1180-1740 | 0014961Q-2501 |
| Pipet Tips, 200 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1180-8710 | E116684P-1540 |
| Pipet Tips, 1000 ul XL TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1182-1730 | F118815P |
| 5 mL Standard Racked Gilson-fit Reference Tips | Scientific Specialties | 4411-00 | 14312 |
| Combitips advanced, 0.1 mL Biopur | Eppendorf | 003 008 9618 | F165414H |
| Combitips advanced, 0.2 mL Biopur | Eppendorf | 0030 089.626 | F166689J |
| Combitips advanced, 5 mL Biopur | Eppendorf | 0030.089 669 | F166054J |
| Combitips advanced, 50 mL Biopur | Eppendorf | 003.008.9693 | F166055I |
| Reagent Reservoir | VWR | 89094-680 | 141500 |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165029I |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165028G |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Green | Eppendorf | 951020346 | F166183K |
| Equipment Type | Equipment ID | ||
| Analytical Balance | EQP0125 | ||
| Cryogenic Freezer 1, -80oC | EQP0095 | ||
| Refrigerator 6.1 cu ft GP06W1AREF | EQP0139 | ||
| -20oC Freezer | EQP0140 | ||
| Beckman Coulter Microfuge 22R | EQP0025 | ||
| Beckman Coulter Microfuge 22R | EQP0124 | ||
| Thermo Scientific Hereaus Megafuge 8 | EQP0104 | ||
| Mini Centrifuge | EQP0131 | ||
| Mini Centrifuge | EQP0136 | ||
| Mini Centrifuge | EQP0134 | ||
| Mini Centrifuge | EQP0235 | ||
| Mini Centrifuge | EQP0216 | ||
| Thermo Scientific HeraTherm Incubator | EQP0105 | ||
| Pipette 0.1 - 2.5 μL | EQP0182 | ||
| Pipette 0.1 - 2.5 μL | EQP0072 | ||
| Pipette 0.1 - 2.5 μL | EQP0070 | ||
| Pipette 0.5-10 μL | EQP0218 | ||
| Pipette 0.5-10 μL | EQP0075 | ||
| Pipette 0.5-10 μL | EQP0169 | ||
| Pipette 0.5-10 μL | EQP0074 | ||
| Pipette 0.5-10 μL | EQP0147 | ||
| Pipette 2 - 20 μL | EQP0128 | ||
| Pipette 2 - 20 μL | EQP0160 | ||
| Pipette 2 - 20 μL | EQP0018 | ||
| Pipette 2 - 20 μL | EQP0146 | ||
| Pipette 10 - 100 μL | EQP0079 | ||
| Pipette 10 - 100 μL | EQP0181 | ||
| Pipette 10 - 100 μL | EQP0085 | ||
| Pipette 10 - 100 μL | EQP0077 | ||
| Pipette 20 - 200 μL | EQP0088 | ||
| Pipette 20 - 200 μL | EQP0087 | ||
| Pipette 20 - 200 μL | EQP0231 | ||
| Pipette 100 - 1000 μL | EQP0050 | ||
| Pipette 100 - 1000 μL | EQP0158 | ||
| Pipette 100 - 1000 μL | EQP0217 | ||
| Pipette 100 - 1000 μL | EQP0082 | ||
| Pipette 100 - 1000 μL | EQP0183 | ||
| Pipette 100 - 1000 μL | EQP0083 | ||
| Pipette 5 mL | EQP0153 | ||
| Timer | S/N 140623950 | ||
| Hamilton SafeAire VAV Fume Hood | EQP0206 | ||
| Biosafety Cabinet | EQP0205 | ||
| Biosafety Cabinet | EQP0204 | ||
| Qubit 3.0 | EQP0102 | ||
| Benchmark Digital Heat Block | EQP0108 | ||
| Benchmark Digital Heat Block | EQP0231 | ||
| Polaroid Z2300 Instant Print Digital Gel Camera with WiFi and 16GB SDHC memory card | EQP0111 | ||
| Electrophoresis Power Unit | EQP0113 | ||
| Electrophoresis Small Gel Box | EQP0116 | ||
| Maestro Transilluminator | EQP0118 | ||
| Microwave | EQP0215 | ||
| Multichannel 8-well Pipette 2 - 20 μL | EQP0207 | ||
| Multichannel 8-well Pipette 10 - 100 μL | EQP0090 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0094 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0161 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0162 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0163 | ||
| Vortex Genie 2 | EQP0052 | ||
| Vortex Genie 2 | EQP0007 | ||
| Vortex Genie 2 | EQP0132 | ||
| Vortex Genie 2 | EQP0137 | ||
| Vortex Genie 2 | EQP0135 | ||
| Air Clean PCR Workstation | EQP0203 | ||
| Air Clean PCR Workstation | EQP0096 | ||
| Air Clean PCR Workstation | EQP0148 | ||
| Air Clean PCR Workstation | EQP0097 | ||
| QX200 Droplet Generator | EQP0202 | ||
| QX200 Droplet Generator | EQP0121 | ||
| Automated Droplet Generator | EQP0179 | ||
| PX1 PCR Plate Sealer | EQP0123 | ||
| PX1 PCR Plate Sealer | EQP0186 | ||
| C1000 Touch Cycler w/96W FS RM | EQP0120 | ||
| S1000 Cycler w/96W FS RM | EQP0174 | ||
| S1000 Cycler w/96W FS RM | EQP0173 | ||
| T100 Thermal Cycler | EQP0180 | ||
| T100 Thermal Cycler | EQP0175 | ||
| QX200 Droplet Reader | EQP0194 | ||
| QX200 Droplet Reader | EQP0122 |
Riferimenti
- Ignatiadis, M., Lee, M., Jeffrey, S. S. Circulating Tumor Cells and Circulating Tumor DNA: Challenges and Opportunities on the Path to Clinical Utility. Clin Cancer Res. 21 (21), 4786-4800 (2015).
- Alix-Panabieres, C., Pantel, K. Clinical Applications of Circulating Tumor Cells and Circulating Tumor DNA as Liquid Biopsy. Cancer Discov. , (2016).
- Paxton, A. Is Molecular AP testing in sync with guidelines. CAP Today. , (2014).
- Sacher, A. G., et al. Prospective Validation of Rapid Plasma Genotyping for the Detection of EGFR and KRAS Mutations in Advanced Lung Cancer. JAMA Oncol. , (2016).
- Sozzi, G., et al. Quantification of free circulating DNA as a diagnostic marker in lung cancer. J Clin Oncol. 21 (21), 3902-3908 (2003).
- Oxnard, G. R., et al. Noninvasive detection of response and resistance in EGFR-mutant lung cancer using quantitative next-generation genotyping of cell-free plasma DNA. Clin Cancer Res. 20 (6), 1698-1705 (2014).
- Best, M. G., et al. RNA-Seq of Tumor-Educated Platelets Enables Blood-Based Pan-Cancer, Multiclass, and Molecular Pathway Cancer Diagnostics. Cancer Cell. 28 (5), 666-676 (2015).
- Rodriguez, M., et al. Different exosome cargo from plasma/bronchoalveolar lavage in non-small-cell lung cancer. Genes Chromosomes Cancer. 53 (9), 713-724 (2014).
- Kalluri, R. The biology and function of exosomes in cancer. J Clin Invest. 126 (4), 1208-1215 (2016).
- Mellert, H., et al. Development and Clinical Utility of a Blood-Based Test Service for the Rapid Identification of Actionable Mutations in Non-Small Cell Lung Carcinoma. J Mol Diagn. 19 (3), 404-416 (2017).
- Qin, J., Williams, T. L., Fernando, M. R. A novel blood collection device stabilizes cell-free RNA in blood during sample shipping and storage. BMC Res Notes. 6, 380 (2013).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. Biotechniques. 15 (3), 532-537 (1993).
- Kohno, T., et al. Beyond ALK-RET, ROS1 and other oncogene fusions in lung cancer. Transl Lung Cancer Res. 4 (2), 156-164 (2015).
- Takeuchi, K., et al. ROS1 and ALK fusions in lung cancer. Nat Med. 18 (3), 378-381 (2012).
- Rimkunas, V. M., et al. Analysis of receptor tyrosine kinase ROS1-positive tumors in non-small cell lung cancer: identification of a FIG-ROS1 fusion. Clin Cancer Res. 18 (16), 4449-4457 (2012).
- Tsuta, K., et al. RET-rearranged non-small-cell lung carcinoma: a clinicopathological and molecular analysis. Br J Cancer. 110 (6), 1571-1578 (2014).
- Forbes, S. A., et al. COSMIC: somatic cancer genetics at high-resolution. Nucleic Acids Res. 45 (1), 777-783 (2017).
- Salgia, R. Diagnostic challenges in non-small-cell lung cancer: an integrated medicine approach. Future Oncol. 11 (3), 489-500 (2015).
- Cagle, P. T., Raparia, K., Portier, B. P. Emerging Biomarkers in Personalized Therapy of Lung Cancer. Adv Exp Med Biol. 890, 25-36 (2016).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci U S A. 96 (16), 9236-9241 (1999).
- Oxnard, G. R., et al. Association Between Plasma Genotyping and Outcomes of Treatment With Osimertinib (AZD9291) in Advanced Non-Small-Cell Lung Cancer. J Clin Oncol. 34 (28), 3375-3382 (2016).
- Reckamp, K. L., et al. A Highly Sensitive and Quantitative Test Platform for Detection of NSCLC EGFR Mutations in Urine and Plasma. J Thorac Oncol. 11 (10), 1690-1700 (2016).
- Yanagita, M., et al. A prospective evaluation of circulating tumor cells and cell-free DNA in EGFR mutant non-small cell lung cancer patients treated with erlotinib on a phase II trial. Clin Cancer Res. , (2016).
- Abbosh, C., et al. Phylogenetic ctDNA analysis depicts early-stage lung cancer evolution. Nature. 545 (7655), 446-451 (2017).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-534 (1993).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon