Method Article
На основе крови тест для обнаружения ROS1 и ОТСТАВКЕ стенограммы Fusion от циркулирующих рибонуклеиновой кислоты с использованием цифровой полимеразной цепной реакции
В этой статье
Резюме
Обнаружение циркулирующих рибонуклеиновой кислоты (cRNA) от крови является неудовлетворенная потребность в клинической диагностике. Здесь мы описываем методы, которые характеризуют cRNA от немелкоклеточного больных раком легкого с использованием чувствительных и конкретные цифровые полимеразной цепной реакции. Тесты требованиям дизайн для обнаружения fusion вариантов в течение 72 часов.
Аннотация
Мы разработали новые методы для изоляции и характеристика опухоли производные циркулирующего рибонуклеиновой кислоты (cRNA) для жидких биопсии, на основе крови. Надежное обнаружение cRNA оправился от крови представляет собой решение важнейших неудовлетворенных потребностей в клинической диагностике. Тест начинается с коллекцией цельной крови в пробирки, содержащие консерванты, которые стабилизировать cRNA. Ячейки бесплатно, exosomal и тромбоцитов связанные РНК изолирован от плазмы в этой тестовой системе. CRNA обратной транскрипции к комплементарной ДНК — (cDNA кДНК) и усиливается с помощью цифровой полимеразной цепной реакции (dPCR). Образцы оцениваются как биомаркер целевой, так и управления гена. Тест проверки включены предел обнаружения, точности и надежности исследований с аналитической пробы. Метод разработан в результате этих исследований можно воспроизвести обнаружить несколько вариантов Фьюжн для ROS1 (C-рос протоонкоген 1; 8 варианты) и RET (переставить во время трансфекции протоонкоген; 8 вариантов). Образец рабочего процесса обработки был оптимизирован таким образом, чтобы результаты тестирования могут создаваться последовательно в течение 72 часов с момента получения образца.
Введение
До 25%-немелкоклеточного рака легкого (НМРЛ) пациенты могут не иметь достаточно ткани доступны для тестирования на момент постановки диагноза. Даже в тех случаях, когда ткани она может не быть достаточного количества или качества для выполнения рекомендуется молекулярные тесты1,2. В тех случаях, где есть достаточно ткани от биопсии для молекулярной профилирования, пациенты могут иметь ждать несколько недель или дольше для достижения результатов, или начать лечение без молекулярной результаты3,4. Однако важно, что информативный молекулярной диагностики иметься ввиду появления нескольких вариантов целенаправленного лечения для пациентов с диагнозом НМРЛ. Тестирование циркулирующих клеток бесплатные ДНК (cfDNA) от жидких биопсия является решение проблем традиционной ткани тестирование4,5,6. Текущие параметры тестирования для действия мутации в НМРЛ с помощью cfDNA и аналогичных рабочих процессов на основе dPCR для быстрого результата поколения, включают в себя информирование мутации ΔE746-A750 и L858R, EGFR сопротивления мутации T790M рецептор эпидермального фактора роста (EGFR) , Протоонкоген (КАРСТ крас) варианты и B-Raf протоонкоген (BRAF) вариант V600E. Хотя не принято как широко по полю, циркулирующих опухоли производные матричная РНК (мРНК), изолированы от жидких биопсия может также обеспечить важной клинической информации7,,8,9. Ранее мы разработали и сообщили о методах мультиплексированных обнаружения иглокожие микротрубочек связанные белком как 4-анапластическая лимфома рецептор тирозинкиназы (EML4-ALK) фьюжн вариантов из плазмы крови10. В этом исследовании мы расширили эти методы, чтобы включить мультиплексированных РНК целей более высокого порядка ROS1 и RET, охватывающих восемь фьюжн варианты в рамках каждого пробирного. Цель заключалась в разработке быстрое, чувствительных, конкретные и воспроизводимые техника для обнаружения этих вариантов фьюжн от плазме пациентов, ранее диагнозом НМРЛ.
В амбулаторных условиях с использованием РНК, стабилизации крови коллекции трубок11инициируется процесс тестирования. Эти трубы содержат консервант а также битор РНКазы ячейки. Образцы являются поставляется приоритет на ночь для централизованного аккредитованный колледж американских патологов CAP/клиническая лаборатория улучшение поправки (CLIA)-Аттестованная лаборатория (клиническая лаборатория) для обработки компетентным персоналом. После того, как получил клинической лабораторией, каждый шаг обработки проводится согласно утвержденных стандартных оперативных процедур (СОП). Цельной крови центрифугируется восстановить плазмы, которая затем используется для изоляции, циркулирующих РНК, которая либо бесплатно в крови или в пределах инкапсуляции постановление, таких как exosomes и тромбоцитов7,8,9. Чтобы изолировать РНК из этих отсеков, мы выбрали систему для восстановления РНК, основанный на сравнении нескольких методов извлечения. Изолированные РНК концентрированной и обратной транскрипции cDNA. Несколько обратной транскриптазы ферментов и Джин специфические праймеры были оценены во время оптимизации метода синтеза cDNA максимально ROS1 и RET целевой Стенограмма преобразования10. Это критически важно для низких изобилия, циркулирующих стенограммы, например варианты опухоли производные фьюжн. Наконец мы оптимизировали концентрации dPCR грунт и зонд для мультиплексных обнаружения RET или ROS1 фьюжн вариантов и управления гена, глюкуронидазы β (GUSB). Затем мы объединили лучшие условия от каждого из исследований оптимизации в Заключительный протокол блокировки перед выполнением исследования аналитической проверки, описанные в настоящем докладе. Этот протокол и эти результаты обеспечивают основу для быстрого и чувствительной рабочего процесса для рутинной выявления редких фьюжн вариантов в обращении.
протокол
Производителей инструкций для реагентов, перечисленных ниже, если не указано иное. Анализы ПЦР, коммерчески доступных продуктов, предназначенных для обнаружения ROS1 и RET сплавливания.
1. Работа с РНК в рамках подготовки к обратной транскрипции (RT)-dPCR: лучшие практики лаборатории
- Создание свободной РНКазы среды при работе с РНК.
- Использование коммерчески доступных аэрозоли, предназначенные для инактивации загрязняющих полиадениновый.
- Использование сертифицированных бесплатно РНКазы реагентов, советы и трубы. Используйте подсказки барьера для дозаторов для предотвращения введения полиадениновый или перекрестное загрязнение образцов.
- Всегда носите пальто лаборатории для предотвращения падения от одежды в ваш образец частиц. Назначьте конкретный пальто лаборатории для использования с обработкой РНК.
- Надевайте перчатки для предотвращения контаминации образцов от полиадениновый в коже. Часто менять перчатки.
Примечание: Предположим, что Лаборатория поверхности загрязнены РНКазы, так как они подвергаются воздействию окружающей среды. Перчатки, которые контакт кожи, волос, дверные ручки, Морозильная камера ручки, Ручки/маркеры и т.д. считаются больше не РНКазы бесплатно. - Обеззараживанию дозаторов, benchtops, центрифуги и других рабочих поверхностей с РНКазы инактивации спрей до использования.
- Если это возможно поддерживать набор оборудования для использования только с РНК.
- При работе с образцами РНК, чтобы предотвратить частицы от попадания образцов или заражение рабочей области, сведения к минимуму перебоев воздушного потока в лаборатории районах.
- Магазин очищенного РНК в-80 градусов.
- Избегайте несколько замораживания оттепели образцов РНК, как это может привести к деградации.
2. поколение аналитического материала РНК для положительных элементов управления
- Дизайн синтетических ДНК с помощью опубликованных последовательностями мРНК для синтеза вариантов интерес10.
- Для варианта данного слияния выберите последовательность фьюжн мРНК, которая включает в себя сайт фьюжн плюс достаточной длины, окаймляющие на каждой стороне для покрытия Ампликон ПЦР.
- Выберите нуклеотидные последовательности между 50-250 nt имитировать размер оборотных РНК, захвачен с помощью плазмы обогащенной тромбоцитов.
- Добавить последовательность промоутер T7 (5'-CAGAGATGCATAATACGACTCACTATAGGGAGA-3') к 5' концу последовательности целевого объекта.
- Заказать синтетических последовательности, как фрагменты двойной мель дезоксирибонуклеиновой кислоты (ДНК).
- Воссоздания синтетических ДНК в буфер Tris-ЭДТА (TE) до конечной концентрации 10 нг/мкл.
- Преобразуйте 60 нг синтетических ДНК с помощью в vitro транскрипция РНК.
- Очищайте стенограммы РНК с помощью реагентов на основе фенола/гуанидина12.
- Включать DNase I, RNase бесплатно для удаления остаточного шаблон ДНК.
- Измерить концентрацию очищенный в vitro РНК, коммерчески доступных флюорометр с помощью РНК конкретных красители и стандартов. Убедитесь, что РНК находится в пределах диапазона приемлемых для выбранной стандартов. Может потребоваться разрежения.
- Подтверждение успешного транскрипции электрофорезом геля, используя 2% агарозном геле, смешанного с пятно гель РНК и высокий диапазон РНК лестница, включая 50-250 nt размер диапазона.
- Нагрузки 500 нг каждого в vitro РНК на геле.
- Побегите гель на 5 V/см.
- Визуализировать одной полосы, используя освещение и документирования результатов.
- Подтвердите ожидаемое Стенограмма размер для каждого из вариантов Фьюжн (на основе дизайна в шаге 2.1.2).
- Подтвердить обнаружение каждого в vitro РНК, используя соответствующий вариант конкретного ПЦР пробирного RT-dPCR (см. шаги 5-8 настоящего Протокола).
- Дополнительно: Подготовьте эквимолярных смесь в vitro РНК, которая содержит каждый из вариантов фьюжн и управления гена GUSB.
- Если выполняется шаг 2.9: подтвердить обнаружения каждого из вариантов фьюжн, включенных в смеси управления по dPCR, используя вариант конкретные анализы ПЦР (см. шаги 5-8 настоящего Протокола).
- Определите желаемый ввода концентрацию для аналитически положительных элементов управления путем тестирования концентрациях от 0,25 до 2,5 ФГ10. Выберите концентрации, основанные на номер желаемого копирование вывода.
- После подтверждения Подготовьте 10 мкл одноразовые аликвоты аналитически РНК для использования в положительный контроль (шаг 4.4) и хранить в-80 градусов.
3. доноров образцы
- Собирайте 10 мл крови человека целом образцов в 10 мл пробирки (ВСТ) содержащий консерванта РНК клетки бесплатно.
Примечание: Все человеческие доноры должны дать согласие на исследования использования и не идентифицирующую конкретных доноров должна собирать или используется во время тестирования. - Обработка образцов цельной крови в сроки, указанные производителем BCT.
- Обобщённые нормальной человеческой плазмы можно приобрести от коммерческого источника для использования в рамках аналитической положительный контроль. Подготовьте одноразового использования, 1 мл аликвоты пуле нормальной человеческой плазмы и в магазине в-80 ° c для использования с позитивного управления (шаг 4.4).
4. Восстановление циркулирующих РНК из плазмы
Примечание: Важно работать быстро во время этой процедуры.
- Центрифуга трубы цельной крови на 200 g x 20 мин.
- Соберите до 4 мл плазмы от центрифугировали пробирка с помощью Серологические Пипетки. Будьте осторожны, чтобы не нарушить или аспирационная Баффи слой.
- Изолирование циркулирующих РНК, использование коммерчески доступных комплект, который может захватить exosomes, тромбоциты и свободной ячейки РНК из плазмы. Изолируйте РНК из образца позитивного управления наряду с каждой партии.
-
Готовят позитивного управления для каждого пакета клинических образцов следующим образом:
- Размораживание 1 мл пуле нормальной человеческой плазмы аликвота (шаг 3.3).
- Оттепель 10 мкл аналитического РНК Алиготе (шаг 2.12).
- Подготовьте положительный контроль путем добавления 10 мкл аналитического РНК в нормальной человеческой плазмы образца после того, как был добавлен этанола в lysate плазмы.
- Элюировать образцы с 100 мкл нуклеиназы свободной воды. Немедленно с РНК очистку и концентрации.
- Образцы могут храниться на мокрый лед и охватывает, до одного часа.
- Концентрат РНК с помощью столбца на основе метода и элюировать в 9 мкл РНКазы свободной воды.
- Сразу перейти к шагу 5, или хранить пробы на мокрый лед на срок до одного часа.
5. Обратная транскрипция РНК в cDNA
- Конвертировать концентрации циркулирующего РНК образца в cDNA, используя набор реакции коммерчески доступных обратной транскрипции, включая случайных праймеров (см. таблицу 1 для компонентов).
Примечание: Джин конкретных грунтовки являются необязательными и могут быть разработаны для тестирования вариантов. Грунты разрабатываются на основании целевом РНК последовательности. Используйте сплавливание вариант последовательности от шаг 2.1.- Включать не примеры управления обратной транскриптазы и не управления РНК (см. таблицу 1).
- Изолируйте cDNA от реакции обратной транскрипции, использование коммерчески доступных столбцов спин концентратор ДНК.
Примечание: Этот шаг облегчает удаление ферментов, грунтовки и бесплатные deoxynucleotide трифосфаты (дНТФ). - Сразу использовать cDNA в реакциях PCR или хранить на-80 градусов.
6. цифровой PCR
Примечание: Этот ПЦР для капли цифровой ПЦР (см. Таблицу материалов).
-
Меры предосторожности смесь ПЦР.
- Используйте одноразовые лаборатории пальто и нитриловые перчатки.
- Использование ПЦР смешивать реагенты в области подготовки специального реагента. Не обрабатывать cDNA в районе реагент только Подготовка.
- Обложка зондов во время работы, чтобы защитить их от света. Чрезмерная свет может фото отбеливателем флуоресцентного красителя, придает зонд.
- Транспортировки смеси, охватывает и защищенном от света, в отдельную область предварительного усиления до cDNA добавляется.
- Добавить cDNA испытываемого ПЦР смесь в чистый капот ПЦР расположен в зоне предварительного усиления.
- Подготовка смеси ПЦР для окончательной реакции объем 20 мкл согласно таблице 2.
- Распространите смеси ПЦР + cDNA для ПЦР планшетов.
Примечание: Использовать макет пластины как руководство рекомендуется. - Крышка с помощью съемного листа герметик.
- Центрифуга пластины кратко для сбора образцов в нижней части скважины.
- Mix на пластине шейкер на низкое значение для 10 s.
- Центрифуга пластины кратко для сбора образцов в нижней части скважины.
- Снять пластину герметик. Выполните капелька поколения для ПЦР cDNA смешивают с либо систему поколения ручных или автоматических капли.
- Для ручной капелька поколения передаче 20 мкл ПЦР смеси образца скважин на поколения картриджа капли. 70 мкл поколения капля масла. Крышка с резиновой прокладки и передачи картридж ручной капелька генератор инициировать капелька поколения. После формирования капли передачи капельки свежие ПЦР пластину, используя советы, рекомендованные производителем. Аспирационная и распределить капли медленно, через 5-6 s каждый, не касаясь открытия наконечник картриджа капли или плиты.
- Для поколения автоматизированных капелька уплотнение пластины с фольги печати и передачи капелька генератор. Обеспечить все советы, картриджи, и пластины в место перед началом капелька поколения.
- Следующие поколения капли и передачи капель свежего ПЦР-планшете, печать с sealer пластины фольги и тепловой цикл пластины с использованием параметров в таблице 3.
- После теплового циклователь run является полным, читать пластину с помощью дроплета читателя. Создать макет пластины для читателя программного обеспечения, которое определяет расположение элементов управления, образцы и т.д.и нагрузки в программное обеспечение, чтобы начать читать.
7. анализ данных и обзор и генерации результатов
- Анализируйте пластины, читать результаты, с использованием имеющегося программного обеспечения.
- Перейдите к меню Analyze для просмотра двухмерные (2D) амплитуды участков.
- Оценивают общее качество данных путем изучения данных капли.
- Оцените данные для чисел всего признанных событий, с помощью меню «события». Если есть меньше, чем 10 000 событий в колодец, тщательно оцените данные для дополнительных проблем.
- Проверьте данные для аберрантных флуоресценции амплитуд. Значительную амплитуду различия и концентрации различия между реплицировать образцы свидетельствуют о плохой обработки или перемешивания образцов.
- Делайте заметки капелька кластеров с распыления на 45-градусный оси, которая является показателем низкого качества капель или проблематичным образцов.
- Сначала изучите положительный контроль, нет обратной транскриптазы (No RT) и нет контроля РНК (СРН) данных. Выберите все контрольные образцы и анализировать качество кластера по 2D сюжет. Для правильного Бинаризация четкое разделение между кластерами капелька должно быть очевидно.
- Для каждого варианта пробирного установите порог, на основе контроля скважин.
- Установить пороги на 2D участках, используя инструмент перекрестья для разделения населения двойное отрицание капли от управления гена населения (помечены с 5'-Гексахлор флуоресцеин CE фосфорамидит зонд), ось y и вариант гена населения, если представить) помечены флуоресцеин amidite или 6-Карбоксифлуоресцеина зонд), ось x.
- Сумма копируется из каждого реплицировать хорошо для одного образца.
- Экспресс результаты теста как количество обнаруженных вариант копий.
Примечание: Определить аналитические пороговое значение для вызова положительный или отрицательный пример, запуск нормального здорового донора образец набор (по крайней мере 10 отдельных образцов) через процесс окончательно и установить отсечки выше любых обнаруживаемых фонового сигнала для мутации интерес. Кроме того установить количество копий гена управления, необходимого для вызова положительный или отрицательный результат. Этот элемент управления гена отключения функции внутреннего контроля качества (QC) для оценки количества и качества каждого образца РНК, которая обрабатывается.
8. Проверка условий реакции RT-dPCR, с помощью линий клетки (опционально)
- Чтобы проверить обнаружение fusion вариантов, используйте коммерчески доступных линий клетки, выражая ROS1 или RET фьюжн mRNA интереса. Выполните следующие действия:
- Однородный флэш замороженные клетки в раствором на основе Гуанидиновые лизис непосредственно из замороженного состояния. Даже краткое оттаивания до гомогенизации может вызвать РНК деградации и потери.
- Изолируйте РНК с помощью кремния мембраны спин столбцы, предназначенные для РНК.
- Измерение концентрации РНК образцов флюорометр с помощью РНК специфические реагенты и стандартов.
- Разбавьте изолированных РНК в фоне одичал тип РНК из плазмы или другого коммерческого источника.
- Выполнить шаги от обратного транскрипции РНК cDNA, цифровой PCR и анализа данных и обзор, и поколения результаты перечислены в настоящем Протоколе, для подтверждения обнаружения нужного варианта.
Результаты
Этот протокол описывает тест системы, разработанной для обнаружения РНК синтеза варианты для использования в измерении драйвер мутаций в плазме больных НМРЛ (рис. 1А). Синтез мРНК, продукты из выражения, наиболее распространенных RET и ROS1 перестановки в НМРЛ населения были определены13,14,,1516,17. Мультиплексной ПЦР-анализов были затем предназначены для обнаружения восемь наиболее распространенные варианты Стенограмма для каждого целевого объекта в НМРЛ в пределах одной реакции. Наиболее распространенными транслокации в локусе ROS1 создавать ассоциации с 5' части CD74, SDC4, SLC34A2, EZR или TPM3 генов (рис. 1Б). Наиболее распространенными транслокации в локусе RET приводят к соседство с KIF5B, для которого assay охватывает шесть экзона развязок. Дополнительные партнеры RET , которые охвачены включают те с CCDC6 и TRIM33 (рис. 1C). В общей сложности, анализы охватывают приблизительно 88% из ROS1 и 99% изменений RET, знаны, что происходит в популяции пациентов НМРЛ17.
Специфика анализа компонентов был сначала оцениваются с помощью восьми отдельных в vitro РНК, которые содержат последовательность мРНК для фьюжн стенограммы, охватываемых ROS1 или RET мультиплексированных анализов. Каждый вид РНК был проверяться каждый индивидуальный вариант assay, который состоит из мультиплексированных версии. Существовал нет перекрестной реактивности этих анализов, продемонстрировав 100% аналитически специфики в рамках разработан мультиплексированных анализов (данные не показаны). Чтобы определить нижний предел обнаружения протокол испытаний, Общая РНК, производные от клеточных линий, выражая вариант слияния включены в assay были смешаны в на фоне нормальной РНК в концентрации 5%, 1%, 0,2% и 0,04%. Мультиплексированных RET и ROS1 вариант ПЦР анализов обнаружены всего лишь 0,2% фьюжн вариант (Рисунок 2A-B). Кроме того подготовка 5% мимо линии клеток, производные РНК (выражая EML4-ALK фьюжн транскрипт) не было обнаружено с мультиплексной ROS1 и RET анализов, далее демонстрируют специфику (A-BРисунок 2).
Точность тестирования процесса RT-dPCR была выполнена для обеих ROS1 и отставке аналитического управления материал состоит из эквимолярных в vitro РНК была обработана в трех концентрациях (высокий, средний и низкий) через обратной транскрипции и dPCR на три раз в тот же день (intra-day), на три дня подряд (между день) и с двумя операторами (Интер оператор). Результаты тестирования точности продемонстрировал точное обнаружение fusion Стенограмма интерес, а также управления гена, GUSB, который входит в качестве внутренней метрики КК (рис. 2 c-D).
Помимо внутреннего контроля GUSB каждая партия клинических образцов выполнялся с набором элементов пакета управления. Положительный контроль был разработан из смеси аналитической пробирке в РНК, которые представляют каждый из вариантов фьюжн, протестированы в RT-dPCR, а также аналитические в vitro РНК для GUSB. Эта РНК шипами в нормальной человеческой плазмы lysate во время извлечения РНК и было обработано наряду с клинических образцов на протяжении всего протокола. Никакого контроля обратной транскриптазы (No RT) был отрицательный контроль для подтверждения отсутствия загрязняющих материалов в процессе извлечения РНК и демонстрируют специфику Праймеры для РНК. No RT управления был создан с помощью того же материала как позитивный элемент управления, но он не включает фермента в реакции синтеза cDNA. Нет управления РНК (СРН) является отрицательным для подтверждения отсутствия загрязнения стенограммы в реакции компонентов обратной транскрипции. Этот элемент управления был введен в рабочем процессе на этапе синтеза cDNA, и вода была добавлена в реакции вместо шаблона РНК. No RT и НСБ контроля должна быть отрицательной, в обоих каналах, если точные результаты должны быть доставлены. В таблице 1 перечислены компоненты реакции обратной транскрипции для каждого элемента управления. На ROS1 приведены примеры 2D участков для каждого из этих элементов управления (рис. 3 A-C) и мультиплексной анализов RET (Рисунок 3 E-G). Фьюжн варианты были обнаружены с помощью зонда флуоресцеин amidite (FAM) и представлены по оси y, в то время как элемент управления гена, GUSB, был обнаружен с помощью зонда фосфорамидит 5'-Гексахлор флуоресцеин CE (HEX) и на оси x. Эти элементы управления партии были оценены в течение 21 дня для определения надежности анализа. Для ROS1 и RET в всех 21 работает, выполнены в течение этого исследования (рис. 3D, H) наблюдались позитивные капельки фьюжн и GUSB управления гена капельки. Все отрицательные элементы (No RT и НСБ) дала негативные результаты через весь 21 дня (данные не показаны).
Возможность устранения неполадок является критически важным компонентом любой протокол испытания должны выполняться в клинических лабораторных условиях. Здесь мы предоставляем реальные примеры югу оптимальных результатов, используя протокол RT-dPCR. Во-первых, пример 2D сюжет, демонстрируя важность не контроля обратной транскриптазы (рис. 4A). В этом примере мутант позитивные капельки присутствовали, хотя было не cDNA преобразования из-за отсутствия фермента. Этот результат был вероятно из-за dPCR грунты, усиливая пробить геномной ДНК. В этом случае дизайн Интрон охватывающих assay предотвратит амплификацию геномных ДНК. Кроме того бесплатно РНКазы DNase фермента может использоваться для ликвидации загрязнения ДНК, но это не рекомендуется для обнаружения редких целей, как некоторые РНК деградации могут произойти во время инкубации с ферментом. Следующий пример 2D сюжет был СРН с позитивным капельки в обоих каналах (Рисунок 4B). Это свидетельствует загрязнение в некоторый момент в RT-dPCR установки. В этом случае рекомендуется отказаться от любых потенциально загрязненных реагентов, используемых в тестировании, тщательно деконтаминации все оборудование и повторного тестирования с свежими реакции компонентов. Третий пример 2D участок представлен как спрей капель вдоль линии 45° (рис. 4C). Это часто вызвано стрижка и сливающихся капель. Тщательно капелька обработки до тепловой Велоспорт имеет важное значение, как капельки подвержены повреждениям. Мы рекомендуем использовать поколения автоматизированных капли, когда они становятся доступными. Если передача вручную капельки, обязательно выбрать рекомендуемый советы масштабах родила и использовать тщательно дозирования технику. Капельная передача требует медленно аспирации и дозирования, с каждой проходит более 5-6 секунд, и важно, что открытие наконечник пипетки не прикасайтесь картриджа капли или хорошо. Когда дозирования, держите кончик пипетки на уровне жидкости и поднять его медленно, как капельки обойтись (Просмотр видео для демонстрации). Пример окончательной 2D график демонстрирует отсутствие разделения между населением положительные и отрицательные капли (рис. 4 d). Это может быть вызвано несколькими причинами. ПЦР сильные ингибиторы, таких как моющие средства, используемые в буферах lysis и избыток сильно деградированных ДНК, может вызвать потерю разделения. В этом случае рассмотрим добавление шага очистки между синтеза cDNA и dPCR (такие, как описано в шаге 5 настоящего Протокола). Наконец отсутствие разделения также может быть из-за условий субоптимальных амплификация, и оптимизации ПЦР шага следует также рассматривать.
Данные внутри Figure 5 представляют 984 реальном мире пациента образец оборачиваемости раз и демонстрирует быстрый характер этого рабочего процесса тестирования. Результаты были доведены до лечащего врача уже как в течение 48 часов (79% случаев) получения образца и в 95% случаев, в течение 72 часов. В заключение использование стабилизировалась циркулирующие РНК пробирки, оптимизированные процедуры извлечения РНК из крови и запустить по словам оптимизированный протокол с соответствующим внутренним и пакет управления, RT-dPCR может обеспечить быстрый тест-системы для точное обнаружение синтеза РНК варианты соответствующих НМРЛ.
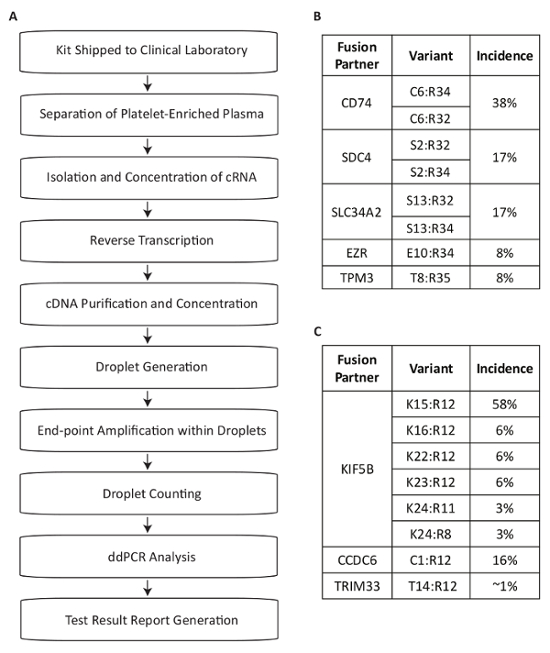
Рисунок 1 : Обзор шагов обработки крови образца для варианта обнаружения Fusion с использованием конкретных анализов на наиболее распространенных RET и ROS1 варианты НМРЛ. (A) образец тестирования инициируется, когда рисуется цельной крови и BCT поставляется в комплект для сбора образцов для клинической лаборатории. Циркулирующих РНК восстанавливается из нескольких источников в тромбоцитов обогащенный плазмы, обратной транскрипции гена конкретных грунтовки и очищенный для использования в dPCR. Образцы обрабатываются с использованием коммерчески доступные системы, которая состоит из капли поколения (эмульсия), усилители и капелька подсчета. Данные анализируются с использованием имеющегося программного обеспечения. Затем результаты испытаний документированы и доложил тест просить врача. Этот процесс предназначен для работы в течение 72 часов с момента получения образца результат выпуска. Восемь вариантов для ROS1 (B) и (C) RET, охватываются мультиплексированных анализов. Адаптировано из Biodesix сайта с разрешения. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
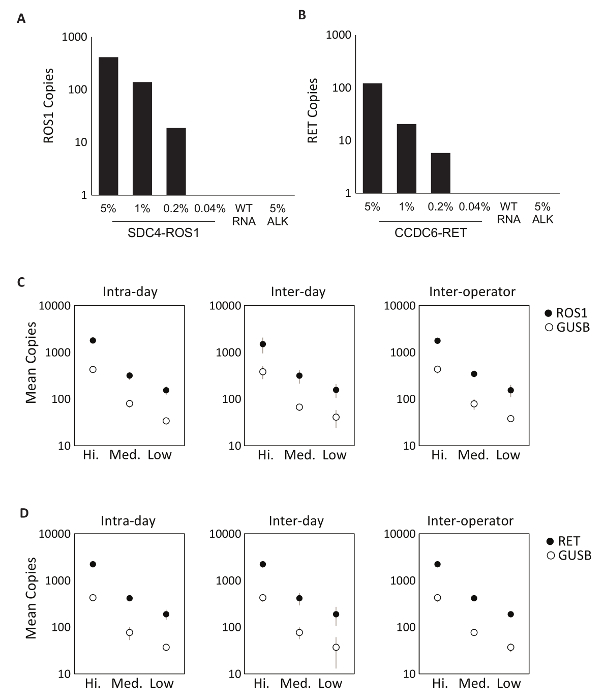
Рисунок 2 : Аналитической проверки. Линии клетки выражения (A) SDC4-ROS1 fusion и (B) CCDC6-RET фьюжн разводили на фоне общего человека одичал тип РНК (РНК WT). С каждым синтеза вариант предел обнаружения был создан вариант частотой 0,2%, используя предварительно определенные критерии для каждого варианта assay. Все образцы выше этого порога также содержится по крайней мере 21 копии гена управления. Стандарт EML4-ALK (ALK) 5% на фоне одичал тип РНК был протестирован для демонстрации пробирного специфичностью, которая была подтверждена отрицательный результат. Аналитически мультиплексированных РНК стандартов были измерены на высокий, средний, и низкой концентрации ROS1 (C) и (D) в отставке точности оценивалась над 3 работает в тот же день (Intra-day), три работает на три дня подряд (между день) и с два независимых операторов (Интер оператор). Показаны средства копирования номер и стандартных отклонений. Адаптировано из Biodesix сайта с разрешения. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
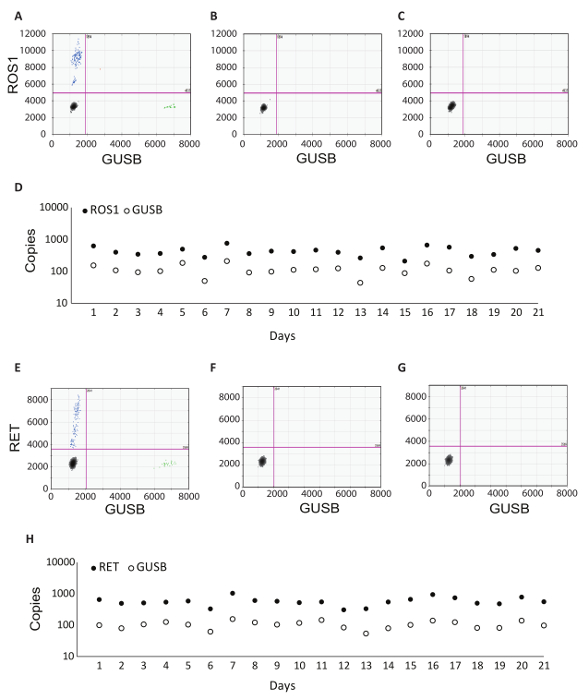
Рисунок 3 : Пакетная обработка примеров элементов управления и надежности данных. 2D график dPCR ROS1 мультиплексированных пробирного для позитивного управления (A), (B) не управления обратной транскриптазы и результаты (C) не РНК шаблон элемента управления. (D) контроль были запущены на 21 дней подряд (исключая выходные и праздничные дни). Означает копию номер + / стандартные отклонения для ROS1 позитивного управления были 439 + / 141. Нет обратной транскриптазы и без шаблона элементов управления также были запущены на каждый день, и они были все отрицательные (данные не показаны). 2D участок dPCR пробирного RET мультиплексированных результаты для позитивного управления (E), (F) не управления обратной транскриптазы и (G) не РНК шаблон элемента управления. (H) элементы управления были запущены на 21 дней подряд (исключая выходные и праздничные дни). Означать копии + / стандартные отклонения для RET позитивного управления были 586 + / 182. Не изображены не обратной транскриптазы и не шаблона элементов управления, которые также были запущены на каждый день и были отрицательными. Адаптировано из Biodesix сайта с разрешения. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
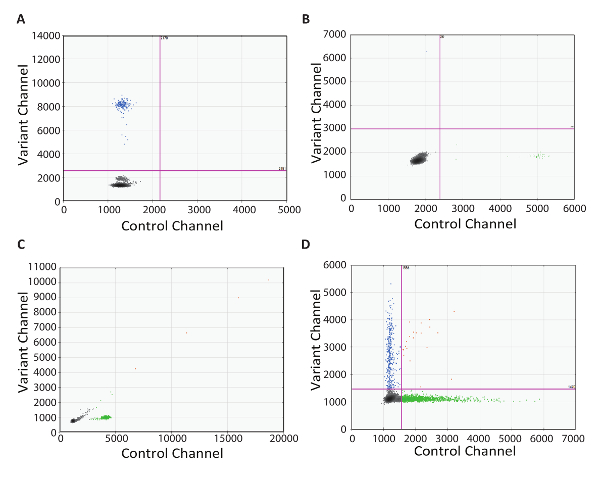
Рисунок 4 : Устранение неполадок RT-dPCR. 2D участков представляющих субоптимальных dPCR полученные результаты когда есть (A) загрязнения в пределах никакого контроля обратной транскриптазы, (B) загрязнения в пределах никакого контроля РНК, (C) стрижка и сливающихся капель, и (D ) плохо оптимизированы условия PCR или ингибитированием ПЦР. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
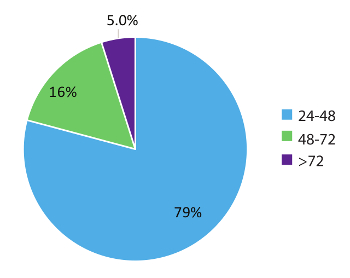
Рисунок 5 : Время оборота (ТАТ). ТАТ (в часах) был составлен для тестов с просьбой вариант РНК (n = 984). Исключает данные, выходные, праздники и образцы для > 24 часа из-за неполной клинической информации о лабораторных испытаний запрос формы. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
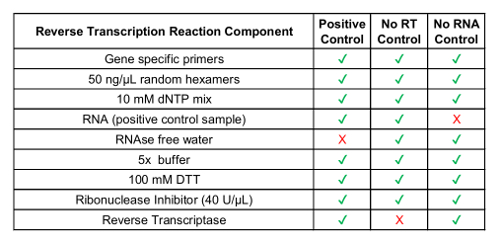
Таблица 1: Подготовка реагентов для обратной транскрипции для управления производственным процессом.
| Компонент | Объем |
| 2 x dPCR Супермикс для датчиков (не 2'-дезоксиуридина 5'-трифосфата) | 10 МКЛ |
| 20 x вариант целевой грунты/зонды набор (450 нмоль/Л грунтовки, 250 нмоль/Л FAM зонд) | 1 МКЛ |
| 20 x управления поставленной грунты/зонд (450 нмоль/Л грунтовки, 250 нмоль/Л HEX зонд) | 1 МКЛ |
| Нуклеаза свободной воды | 1 МКЛ |
| cDNA | 7 МКЛ |
Таблица 2: Подготовка мастер смесь для dPCR.
| Велоспорт шаг | Температура | Время | # Циклов | Рамп курс |
| Активация энзимов | 95 ° C | 10 мин | 1 | ~ 2 oC/s |
| Денатурация | 94 ° C | 30 s | 40 | |
| Отжиг/расширение | 55 ° C | 1 мин | ||
| Деактивация, фермент | 98 ° C | 10 мин | 1 | |
| Провести (опционально) | 4 ° C | бесконечные | 1 | ~ 1 oC/s |
Таблица 3: Термических условий Велоспорт.
Обсуждение
RET и ROS1 перестановки вместе составляют ~ 3% мутаций водителя в пределах НМРЛ населения18. Хотя редко, обнаружение этих генетических изменений является жизненно важным. Больных НМРЛ с этими изменениями могут воспользоваться целенаправленной терапии, которая специально ингибировать активность аберрантных киназы, что результаты от онко белка13. Некоторые такие методы лечения уже утвержденных FDA для использования в ROS1 положительные НМРЛ, в то время как другие были продемонстрированы быть эффективным против RET в клинических испытаниях19.
Цифровая технология ПЦР обеспечивает чувствительность, которая идеально подходит для жидких биопсии приложений20. Наблюдается значительный принятия этой технологии для использования с циркулирующих клеток свободной ДНК для измерения опухоли мутаций у больных НМРЛ4,6,21,,2223 . В дополнение к cfDNA мы разработали протокол, разработанный для надежного измерения наиболее распространенных вариантов Фьюжн у больных НМРЛ от циркулирующих опухоли РНК (рис. 1A)10.
Наш протокол позволяет аналитически пределы обнаружения до 0,2% (рис. 2). В то время как RT-dPCR исключительно конкретные и чувствительной, анализы ограничены группа известных фьюжн вариантов, которые отбираются и мультиплексированных для обнаружения в assay ПЦР. Таким образом слияния должны быть включены в мультиплексированном анализы должны быть тщательно отобраны для обеспечения надлежащего охвата в рамках населения больных НМРЛ. Мы успешно разработали анализов для RET и ROS1, которые одновременно обнаруживать восемь вариантов результирующей фьюжн от перестановки RET или ROS1 локусов и охватывают 99% и 88% населения RET и ROS1 положительных, соответственно (рис. 1Б )17.
Заключительный тест рабочего процесса, как описано в настоящем исследовании включает в себя пакет управления для обеспечения согласованности результатов. Эти элементы управления включают в себя позитивные аналитического стандарта, а также два отрицательных элементов управления, которые вместе убедитесь, что нет загрязнения или ПЦР ингибирование, происходящих в пределах пакета (рис. 3). Для обеспечения надежности assay, исследование было проведено с помощью пакета управления над 21-дневный период (рис. 3D, H). Эти данные показывают последовательности процесса РНК как в рамках настоящего Протокола.
Надлежащей лабораторной практики и соответствующей обработки РНК являются ключевыми компонентами обеспечения надежных и точных результатов. Лаборатория пространство и оборудование выделенных для использования с РНК, очистке оборудования после каждого использования, используя бесплатные РНКазы реагентов и расходных материалов и применения РНКазы инактивации спрей рабочее пространство, все помогают уменьшить загрязняющих полиадениновый. По соображениям обработки образцов РНК, техников, в том числе посвященных лаборатории пальто, частые изменения перчатка, работая быстро через процедуру извлечения РНК, и поддержанию образцы на льду имеют крайне важное значение для сохранения целостности образца. После того как РНК был обратной транскрипции cDNA, образец находится в более стабильной форме, которая меньше подвержены деградации. Помимо практики, поддерживающие целостность РНК ПЦР компоненты и образцы должны поддерживаться в сегрегированных районах для предотвращения перекрестного загрязнения, которые могут привести к ложным положительным результатам. Складе ПЦР реактивы и подготовка мастер смеси ПЦР должны храниться отдельно от PCR шаблоны и большой осторожностью следует отделить усиливается шаблон (пост ПЦР) от всех предварительно усиленный материалов, реагентов, РНК и cDNA образцы. Наконец правильной генерации и обработке эмульсионные смеси ПЦР до амплификации центральное значение для сохранения целостности капли и условий оптимального dPCR. Меры предосторожности, как они важны во время выполнения этого протокола для получения соответствующих и точных результатов. Все данные должны рассматриваться обученным персоналом перед публикацией результатов чтобы убедиться что были выполнены все метрики КК. В случае субоптимальные результаты (рис. 4) пакет должен быть рассмотрен технический персонал и директор лаборатории и могут потребовать повторной обработки.
RT-dPCR результаты могут быть изготовлены как 24 часа от получения образца и 95% результаты образца в тесте, используемую в этом исследовании (n = 984) сообщалось заказов врача в менее чем за 72 часов с момента получения (рис. 5). Этот поворот вокруг времени предоставляет врачам с гораздо необходимые молекулярных информацию в сроки, что позволяет начало соответствующей терапии. Эти результаты обычно доступны раньше, чем те, которые получены с помощью биопсии обычных тканей. Дополнительные биомаркеров для НМРЛ и другие виды рака могут быть разработаны с использованием аналогичных циркулирующих подходы, основанные на РНК и выиграют от же быстрое время к результаты. Например измерение Стенограмма запрограммированных смерти лигандом 1 (PD-L1) мРНК, используя RT-dPCR мог бы информировать врачей о вариантах иммунотерапии. Существует также растущий интерес к утилите жидкого биопсии и dPCR в мониторинге терапевтической эффективности. Более ранние признаки возрождения опухоли с помощью геномной тестирования для конкретных вариантов может позволить врачей для настройки схемы лечения, прежде чем пациенты являются симптомами стандартом мер помощи таких изображений24. Протоколы, такие как одна, сообщили в этом исследовании идеально подходит для наблюдения за их не инвазивность, чувствительность, быстрый поворот вокруг времени и эффективности затрат. Assay описаного предоставляет результаты в течение 72 часов после получения образца, с минимальным ложных детектирование, который облегчает быстрое лечение решений и обходит некоторые ограничения, опыт работы с тестирования на основе ткани4.
Наши протокол и данные демонстрируют надежный тест системы для выявления низкого залегания РНК варианты, а также потенциал на основе крови Мутация тестирование в клинической практике. Для тех пациентов, которые не имеют действия водителя мутации, выявленные быстрого целевых жидкого биопсии подходы, как это одно, более обширные генома и протеом тестирования из ткани и крови может обеспечить даже более широкой клинической информации для поддержки планирования лечения.
Раскрытие информации
Его Величество, L.J., к.а. и G.A.P. являются сотрудниками и держать акции в Biodesix, Inc. H.M. и L.J., G.A.P. являются Сопредседатель изобретателей на патент, поданных Biodesix, охватывающих диагностический тест системы для обнаружения циркулирующих генетических вариантов в немелкоклеточного Рак легких.
Благодарности
Мы благодарим наших партнеров, Стивен Джонс, Ниа Charrington, д-р Диана Maar и доктор Купер Саманта из центра биологии цифровой (Inc. CA Био-Rad) для их анализа дизайн поддержки; Незар Rghei и доктор Moemen Абдалла (Norgen Biotek, Канада) для критических рекомендаций при оптимизации РНК добыча протокол; и Шеннон Кэмпбелл, Скотт Тёрстон, Джефф Fensterer, Шеннон Martello и Энос Joellyn для помощи с тестирования требований и коммерческого мониторинга.
Материалы
| Name | Company | Catalog Number | Comments |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1604071 |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1809353 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1604071 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1606077 |
| Phosphate Buffered Saline 1X, Sterile | Amresco | K812-500mL | 1446C189 |
| Phosphate Buffered Saline 1X, Sterile – 500 mL | Invitrogen | 10010023 | 1916C092 |
| RNase Zap (Life Tech) (250 mL) | Ambion | AM9780 | 353952 |
| Beta-Mercaptoethanol (BME) (250 mL) | CalbioChem | 6050 | W105B |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56054611 |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56238638 |
| Isopropyl Alcohol | VWR | 0918-4L | 2116C416 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 403648 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 288461 |
| DNase I | Thermo | K0441 | 371299 |
| QIAzol Lysis Reagent | Qiagen | 79306 | 54809699 |
| 20x TE buffer pH 8.0 | Alfa Aesar | J62388 | R13C548 |
| UltraPure Agarose | Invitrogen | 16500-100 | 552730 |
| 10x TBE buffer | Invitrogen | AM9863 | 353065 |
| Cell-Free RNA BCT | Streck | 218976 | 60110327 |
| Cell-Free RNA BCT | Streck | 218976 | 61900327 |
| Cell-Free RNA BCT | Streck | 218976 | 61480327 |
| Cell-Free RNA BCT | Streck | 218976 | 62320327 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 585849 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 588308 |
| Lysis Buffer | Norgen | 21205 | A5F61E |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 585848 |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 588309 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC186976 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188077 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188413 |
| Collection Tubes 500 pack | Zymo | C1001-500 | N/A |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 391657 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 392504 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 448001 |
| SuperScript IV Reverse Transcriptase | Life Technologies | 18090200 | 451702 |
| Qubit HS RNA Assay Kit (500) | Life Technologies | Q32854 | 1745264 |
| Qubit assay tubes (500) | Life Technologies | Q32856 | 13416Q311 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64031651 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64063941 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065740 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065741 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64079083 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64025320 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64052358 |
| gBlock KIF5B-RET K15:R12 | IDT | 151004172 | 4-Oct-16 |
| gBlock KIF5B-RET K16:R12 | IDT | 151004173 | 4-Oct-16 |
| gBlock KIF5B-RET K22:R12 | IDT | 151004174 | 4-Oct-16 |
| gBlock KIF5B-RET K23:R12 | IDT | 151004175 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R11 | IDT | 151004176 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R8 | IDT | 151004177 | 4-Oct-16 |
| gBlock CCDC6-RET C1:R12 | IDT | 151004178 | 4-Oct-16 |
| gBlock TRIM33-RET T14:R12 | IDT | 151004179 | 4-Oct-16 |
| RET exon 8 RT Gene Specific Primer | IDT | 150554385 | 28-Sep-16 |
| 5’-CTCCACTCACACCTG-3’ | IDT | 150554385 | 28-Sep-16 |
| RET exon 11 RT Gene Specific Primer | IDT | 150554384 | 28-Sep-16 |
| 5’-GCAAACTTGTGGTAGCAG-3’ | IDT | 150554384 | 28-Sep-16 |
| RET exon 12 RT Gene Specific Primer | IDT | 150554383 | 28-Sep-16 |
| 5’-CTGCCTTTCAGATGGAAG-3’ | IDT | 150554383 | 28-Sep-16 |
| gBlock CD74-ROS1 C6:R34 | IDT | 152324366 | 15-Nov-16 |
| gBlock CD74-ROS1 C6:R32 | IDT | 152324367 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R32 | IDT | 152324368 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R34 | IDT | 152324369 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R32 | IDT | 152324370 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R34 | IDT | 152324371 | 15-Nov-16 |
| gBlock EZR-ROS1 E10:R34 | IDT | 152324372 | 15-Nov-16 |
| gBlock TPM3-ROS1 T8:R35 | IDT | 152324373 | 15-Nov-16 |
| ROS1 exon 34 RT Gene Specific Primer | IDT | 152704983 | 21-Nov-16 |
| 5’-CCTTCCTTGGCACTTT-3’ | IDT | 152704983 | 21-Nov-16 |
| ROS1 exon 35 RT Gene Specific Primer | IDT | 152704985 | 21-Nov-16 |
| 5’-CTCTTGGGTTGGAAGAGTATG-3’ | IDT | 152704985 | 21-Nov-16 |
| ALK Gene Specific Primer | IDT | 140035422 | 26-Aug-16 |
| 5’-CAGTAGTTGGGGTTGTAGTCG-3’ | IDT | 140035422 | 26-Aug-16 |
| EML4-ALK Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| SLC34A2-ROS1 Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| CCDC6-RET Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| Human Brain Total RNA | Ambion | AM7962 | 1703548 |
| PrimePCR ddPCR Expert Design Assay: K15:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K16:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K22:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K23:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R11 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R8 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C1:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: T14:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: E10:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: T8:R35 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| (version 2) | Bio-Rad | 12003909 | 213939881 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 13-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 20170112v3.2 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851151 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 207383915 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 195995635 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851152 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 213949301 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 20160914 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 211383227 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 1065C220 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052953 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052358 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 1065C320 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64052952 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64064127 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000065883 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084276 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000079928 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084395 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084634 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20160627 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161107 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161206 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161216 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20170125 |
| Pipet Tips for Automated Droplet Generator | Bio-Rad | 1864120 | PR125340 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206894 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206893 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 1409850 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 100402 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 145851 |
| Microseal 'B' seals | Bio-Rad | MSB1001 | BR00428490 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64039089 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049253 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049255 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64081870 |
| DNA Lo Bind Tube 0.5 mL | Eppendorf | 22431005 | E1629620 |
| DNA Lo Bind Tube 1.5 mL | Eppendorf | 22431021 | F16698K |
| DNA Lo Bind Tube 2 mL | Eppendorf | 22431048 | E160610I |
| 50 mL Conicals, Polypropylene (25) | Thermo | 339652 | G5ZF5W8118 |
| TempAssure PCR 8-Strips, Optical Caps, Natural, polypropylene (120) | USA Scientific | 1402-4700 | 16202 |
| For Rainin LTS Pipettors 0.5-20 µL tips | Pipette.com | LF-20 | 40155-642C4-642C |
| For Rainin LTS Pipettors 5-200 µL tips | Pipette.com | LF-250 | 40154-642C4-642B |
| Tips LTS 200 ul Filter 960/10 RT-L200F (10 boxes) | Rainin | 17002927 | 1635 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F1175551-1108 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F118054L-1720 |
| Pipet Tips, 100 ul TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1180-1740 | 0014961Q-2501 |
| Pipet Tips, 200 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1180-8710 | E116684P-1540 |
| Pipet Tips, 1000 ul XL TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1182-1730 | F118815P |
| 5 mL Standard Racked Gilson-fit Reference Tips | Scientific Specialties | 4411-00 | 14312 |
| Combitips advanced, 0.1 mL Biopur | Eppendorf | 003 008 9618 | F165414H |
| Combitips advanced, 0.2 mL Biopur | Eppendorf | 0030 089.626 | F166689J |
| Combitips advanced, 5 mL Biopur | Eppendorf | 0030.089 669 | F166054J |
| Combitips advanced, 50 mL Biopur | Eppendorf | 003.008.9693 | F166055I |
| Reagent Reservoir | VWR | 89094-680 | 141500 |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165029I |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165028G |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Green | Eppendorf | 951020346 | F166183K |
| Equipment Type | Equipment ID | ||
| Analytical Balance | EQP0125 | ||
| Cryogenic Freezer 1, -80oC | EQP0095 | ||
| Refrigerator 6.1 cu ft GP06W1AREF | EQP0139 | ||
| -20oC Freezer | EQP0140 | ||
| Beckman Coulter Microfuge 22R | EQP0025 | ||
| Beckman Coulter Microfuge 22R | EQP0124 | ||
| Thermo Scientific Hereaus Megafuge 8 | EQP0104 | ||
| Mini Centrifuge | EQP0131 | ||
| Mini Centrifuge | EQP0136 | ||
| Mini Centrifuge | EQP0134 | ||
| Mini Centrifuge | EQP0235 | ||
| Mini Centrifuge | EQP0216 | ||
| Thermo Scientific HeraTherm Incubator | EQP0105 | ||
| Pipette 0.1 - 2.5 μL | EQP0182 | ||
| Pipette 0.1 - 2.5 μL | EQP0072 | ||
| Pipette 0.1 - 2.5 μL | EQP0070 | ||
| Pipette 0.5-10 μL | EQP0218 | ||
| Pipette 0.5-10 μL | EQP0075 | ||
| Pipette 0.5-10 μL | EQP0169 | ||
| Pipette 0.5-10 μL | EQP0074 | ||
| Pipette 0.5-10 μL | EQP0147 | ||
| Pipette 2 - 20 μL | EQP0128 | ||
| Pipette 2 - 20 μL | EQP0160 | ||
| Pipette 2 - 20 μL | EQP0018 | ||
| Pipette 2 - 20 μL | EQP0146 | ||
| Pipette 10 - 100 μL | EQP0079 | ||
| Pipette 10 - 100 μL | EQP0181 | ||
| Pipette 10 - 100 μL | EQP0085 | ||
| Pipette 10 - 100 μL | EQP0077 | ||
| Pipette 20 - 200 μL | EQP0088 | ||
| Pipette 20 - 200 μL | EQP0087 | ||
| Pipette 20 - 200 μL | EQP0231 | ||
| Pipette 100 - 1000 μL | EQP0050 | ||
| Pipette 100 - 1000 μL | EQP0158 | ||
| Pipette 100 - 1000 μL | EQP0217 | ||
| Pipette 100 - 1000 μL | EQP0082 | ||
| Pipette 100 - 1000 μL | EQP0183 | ||
| Pipette 100 - 1000 μL | EQP0083 | ||
| Pipette 5 mL | EQP0153 | ||
| Timer | S/N 140623950 | ||
| Hamilton SafeAire VAV Fume Hood | EQP0206 | ||
| Biosafety Cabinet | EQP0205 | ||
| Biosafety Cabinet | EQP0204 | ||
| Qubit 3.0 | EQP0102 | ||
| Benchmark Digital Heat Block | EQP0108 | ||
| Benchmark Digital Heat Block | EQP0231 | ||
| Polaroid Z2300 Instant Print Digital Gel Camera with WiFi and 16GB SDHC memory card | EQP0111 | ||
| Electrophoresis Power Unit | EQP0113 | ||
| Electrophoresis Small Gel Box | EQP0116 | ||
| Maestro Transilluminator | EQP0118 | ||
| Microwave | EQP0215 | ||
| Multichannel 8-well Pipette 2 - 20 μL | EQP0207 | ||
| Multichannel 8-well Pipette 10 - 100 μL | EQP0090 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0094 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0161 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0162 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0163 | ||
| Vortex Genie 2 | EQP0052 | ||
| Vortex Genie 2 | EQP0007 | ||
| Vortex Genie 2 | EQP0132 | ||
| Vortex Genie 2 | EQP0137 | ||
| Vortex Genie 2 | EQP0135 | ||
| Air Clean PCR Workstation | EQP0203 | ||
| Air Clean PCR Workstation | EQP0096 | ||
| Air Clean PCR Workstation | EQP0148 | ||
| Air Clean PCR Workstation | EQP0097 | ||
| QX200 Droplet Generator | EQP0202 | ||
| QX200 Droplet Generator | EQP0121 | ||
| Automated Droplet Generator | EQP0179 | ||
| PX1 PCR Plate Sealer | EQP0123 | ||
| PX1 PCR Plate Sealer | EQP0186 | ||
| C1000 Touch Cycler w/96W FS RM | EQP0120 | ||
| S1000 Cycler w/96W FS RM | EQP0174 | ||
| S1000 Cycler w/96W FS RM | EQP0173 | ||
| T100 Thermal Cycler | EQP0180 | ||
| T100 Thermal Cycler | EQP0175 | ||
| QX200 Droplet Reader | EQP0194 | ||
| QX200 Droplet Reader | EQP0122 |
Ссылки
- Ignatiadis, M., Lee, M., Jeffrey, S. S. Circulating Tumor Cells and Circulating Tumor DNA: Challenges and Opportunities on the Path to Clinical Utility. Clin Cancer Res. 21 (21), 4786-4800 (2015).
- Alix-Panabieres, C., Pantel, K. Clinical Applications of Circulating Tumor Cells and Circulating Tumor DNA as Liquid Biopsy. Cancer Discov. , (2016).
- Paxton, A. Is Molecular AP testing in sync with guidelines. CAP Today. , (2014).
- Sacher, A. G., et al. Prospective Validation of Rapid Plasma Genotyping for the Detection of EGFR and KRAS Mutations in Advanced Lung Cancer. JAMA Oncol. , (2016).
- Sozzi, G., et al. Quantification of free circulating DNA as a diagnostic marker in lung cancer. J Clin Oncol. 21 (21), 3902-3908 (2003).
- Oxnard, G. R., et al. Noninvasive detection of response and resistance in EGFR-mutant lung cancer using quantitative next-generation genotyping of cell-free plasma DNA. Clin Cancer Res. 20 (6), 1698-1705 (2014).
- Best, M. G., et al. RNA-Seq of Tumor-Educated Platelets Enables Blood-Based Pan-Cancer, Multiclass, and Molecular Pathway Cancer Diagnostics. Cancer Cell. 28 (5), 666-676 (2015).
- Rodriguez, M., et al. Different exosome cargo from plasma/bronchoalveolar lavage in non-small-cell lung cancer. Genes Chromosomes Cancer. 53 (9), 713-724 (2014).
- Kalluri, R. The biology and function of exosomes in cancer. J Clin Invest. 126 (4), 1208-1215 (2016).
- Mellert, H., et al. Development and Clinical Utility of a Blood-Based Test Service for the Rapid Identification of Actionable Mutations in Non-Small Cell Lung Carcinoma. J Mol Diagn. 19 (3), 404-416 (2017).
- Qin, J., Williams, T. L., Fernando, M. R. A novel blood collection device stabilizes cell-free RNA in blood during sample shipping and storage. BMC Res Notes. 6, 380(2013).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. Biotechniques. 15 (3), 532-537 (1993).
- Kohno, T., et al. Beyond ALK-RET, ROS1 and other oncogene fusions in lung cancer. Transl Lung Cancer Res. 4 (2), 156-164 (2015).
- Takeuchi, K., et al. ROS1 and ALK fusions in lung cancer. Nat Med. 18 (3), 378-381 (2012).
- Rimkunas, V. M., et al. Analysis of receptor tyrosine kinase ROS1-positive tumors in non-small cell lung cancer: identification of a FIG-ROS1 fusion. Clin Cancer Res. 18 (16), 4449-4457 (2012).
- Tsuta, K., et al. RET-rearranged non-small-cell lung carcinoma: a clinicopathological and molecular analysis. Br J Cancer. 110 (6), 1571-1578 (2014).
- Forbes, S. A., et al. COSMIC: somatic cancer genetics at high-resolution. Nucleic Acids Res. 45 (1), 777-783 (2017).
- Salgia, R. Diagnostic challenges in non-small-cell lung cancer: an integrated medicine approach. Future Oncol. 11 (3), 489-500 (2015).
- Cagle, P. T., Raparia, K., Portier, B. P. Emerging Biomarkers in Personalized Therapy of Lung Cancer. Adv Exp Med Biol. 890, 25-36 (2016).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci U S A. 96 (16), 9236-9241 (1999).
- Oxnard, G. R., et al. Association Between Plasma Genotyping and Outcomes of Treatment With Osimertinib (AZD9291) in Advanced Non-Small-Cell Lung Cancer. J Clin Oncol. 34 (28), 3375-3382 (2016).
- Reckamp, K. L., et al. A Highly Sensitive and Quantitative Test Platform for Detection of NSCLC EGFR Mutations in Urine and Plasma. J Thorac Oncol. 11 (10), 1690-1700 (2016).
- Yanagita, M., et al. A prospective evaluation of circulating tumor cells and cell-free DNA in EGFR mutant non-small cell lung cancer patients treated with erlotinib on a phase II trial. Clin Cancer Res. , (2016).
- Abbosh, C., et al. Phylogenetic ctDNA analysis depicts early-stage lung cancer evolution. Nature. 545 (7655), 446-451 (2017).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-534 (1993).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены