Method Article
Isolamento ed analisi di comunità microbiche nel suolo, rizosfera e radici negli esperimenti di erba perenne
In questo articolo
Riepilogo
Scavo delle radici delle piante dal campo e trattamento dei campioni in endosphere, rizosfera e suolo sono descritte in dettaglio, compresi i metodi di analisi di dati e di estrazione del DNA. Questa carta è progettata per consentire altri laboratori di utilizzare queste tecniche per lo studio del suolo, endosphere e rizosfera microbiomi.
Abstract
Pianta e terreno microbiome studi stanno diventando sempre più importanti per la comprensione che i microrganismi ruoli giocare nella produttività agricola. Lo scopo di questo manoscritto è quello di fornire dettagli su come rapidamente del campione di suolo, rizosfera ed endosphere di replicata prove sul campo e analizzare i cambiamenti che possono verificarsi nelle comunità microbica a causa del tipo di campione, il trattamento e pianta genotipo. L'esperimento usato per dimostrare questi metodi consiste di trame di campo replicata contenente due, puro, erbe di stagione calda (Panicum virgatum e Andropogon gerardii) e una miscela di erba bassa-diversità (a. gerardii, Sorghastrum nutans, e Bouteloua curtipendula). Brevemente, piante sono scavati, una varietà di radici tagliati e inseriti in tampone fosfato e poi scosso per raccogliere nella rizosfera. Le radici sono portate al laboratorio su ghiaccio e superficie sterilizzato con candeggina ed etanolo (EtOH). La rizosfera è filtrata e concentrata per centrifugazione. Terreno scavato da intorno alla sfera della radice è messo in sacchetti di plastica e portato al laboratorio dove si è preso una piccola quantità di terreno per le estrazioni di DNA. Il DNA è estratta dalle radici, terreno e rizosfera e poi amplificato con primers per la regione di V4 del gene del rRNA 16S. Ampliconi sono sequenziati, poi analizzati con strumenti di bioinformatica di accesso aperto. Questi metodi permettono ai ricercatori di testare come la diversità della comunità microbica e la composizione varia a causa del tipo di campione, trattamento e genotipo della pianta. Utilizzando questi metodi insieme ai modelli statistici, i risultati rappresentativi dimostrano ci sono differenze significative nelle comunità microbiche di radici, rizosfera e suolo. Metodi presentati qui forniscono un set completo di procedura raccogliere campioni di campo, isolare, estrarre, quantificare, amplificare e sequenza del DNA e analizzano la diversità della comunità microbica e composizione in replicati prove sul campo.
Introduzione
Microbioma ricerca ha importanti implicazioni per la comprensione e la manipolazione di processi ecosistemici quali nutrienti ciclismo, fatturato di materia organica e sviluppo o inibizione di agenti patogeni del terreno1,2. Questa area di ricerca inoltre ha un grande potenziale per comprendere l'impatto dei microbi del suolo sulla produttività delle comunità vegetali naturali e agroecosistemi. Mentre ci sono molti studi che hanno messo a fuoco sul microbioma del suolo negli ecosistemi naturali, meno si sono concentrati sui microbi di rizosfera ed endosphere pianta in agroecosistemi3. In Nebraska, agricoltura domina il paesaggio attraverso grandi parti dello stato, che gli studi di questi terreni coltivati un argomento vitale per la ricerca sul piano agricolo importanti colture. Lo scopo di questa carta di metodi è quello di fornire ai ricercatori con un set standard di protocolli per descrivere i microbi presenti negli agroecosistemi, per determinare come le radici delle piante modificare le comunità microbiche nella rizosfera ed endosphere e alla fine comprendere le funzioni di che questi microbi giocare nella produttività del suolo e fitosanitarie.
Il metodo presentato qui differisce leggermente dalle tecniche usate da altri4,5 a che il presente documento è finalizzato all'apprendimento quali microbi sono esclusivamente all'interno della radice e come si differenziano dai microbi immediatamente di fuori della radice nella rizosfera. Il sequenziamento degli ampliconi utilizzato in questo studio identifica i taxa microbici trovati il campione di DNA e permette ai ricercatori di determinare come le comunità cambiano a seconda del tipo di campione o trattamento. Una delle differenze chiave tra il presente protocollo e un protocollo molto simile utilizzato da Lundberg et al. 6 è che invece di sonicazione, questo protocollo utilizza sterilizzazione superficiale con candeggina ed etanolo per rimuovere la rizosfera dalle radici. Altri hanno anche utilizzato la sterilizzazione superficiale efficacemente7,8,9,10. Questi metodi non sono più vantaggiosi rispetto ad altri metodi, ma leggermente diversa. Questi metodi sono particolarmente adatti per esperimenti sul campo grande perché con abbastanza gente è possibile elaborare oltre 150 trame di campo al giorno, che aggiunge fino a circa 450 campioni quando partizionato in endosphere, rizosfera e suolo. Questo manoscritto descrive in dettaglio i metodi utilizzati per esempio nel campo, elaborare il materiale in laboratorio, estrarre e il DNA di sequenza e fornisce una breve panoramica dei passaggi per analizzare i dati di sequenziamento risultanti.
Protocollo
1. campo Descrizione del sito
- Descrivere siti campo sperimentale durante i periodi di raccolta. Determinare la posizione del campo (Latitudine, Longitudine e altitudine) utilizzando un GPS.
- Descrivere la profondità di campionamento, tempo di campionamento e struttura del terreno.
- Fattori ambientali svolgono un ruolo importante nel plasmare le comunità microbiche. Registrare informazioni climatiche come temperatura media annuale, precipitazioni annuali, rotazione delle colture precedenti esercizi, pratiche di coltivazione, metodo di fecondazione e la storia del sito campo. Stazioni meteo automatiche o altri dispositivi sono utili per registrare la pioggia giornaliera e la temperatura su una stagione di crescita.
2. raccolta e trattamento del suolo, rizosfera e radice campo campioni
- Scavo delle piante.
- Etichettare un lavaggio pan e secchio con una nota di Sticky Notes contenente informazioni su materiale vegetale da campionare. Includere informazioni quali numero di trama, piantare il genotipo e specie vegetali. Trasportare il secchio con etichettato per la trama e la vaschetta di lavaggio di lasciare la workstation affermata nel campo.
- Perforare del suolo con una pala per una profondità di 30 cm per tagliare qualsiasi delle radici laterali che tengono la pianta nel terreno. Il volume approssimativo è di 18 cm3. In modo casuale scegliere e raccogliere due piante al complotto da diverse aree all'interno della trama.
- Scavare le radici della pianta sfruttando la pala e posizionare la sfera di radice nel secchio con etichettato. Portare il secchio con etichettato con la sfera di radice scavata alla workstation nel campo. Tagliare ed eliminare la biomassa aboveground della pianta.
- Rimozione del terreno dalle radici e raccolta del suolo alla rinfusa.
- Scuotere le radici per rimuovere lo sporco o utilizzare una vanga o un attrezzo portatile per rimuovere lo sporco dalle radici manualmente. Scuotendo le radici è sufficiente per rimuovere lo sporco in terreni molto sabbiosi. Indossare i guanti e mettere radici nei pressi della stazione di elaborazione.
- Dopo l'agitazione le radici, la maggior parte del terreno sarà in padella lavare. Mescolare il terreno nella padella lavare e spezzare qualsiasi zolle di terreno con un tiller tenuto in mano. Posizionare un campione di suolo che sia libero da detriti in un 17,7 x 19.5 cm cerniera custodia etichettati e posto in un luogo fresco o su ghiaccio.
- Raccolta di radici e rizosfera.
- Utilizzando le Forbici potatura sterilizzate in 70% EtOH, asportare una varietà delle radici, circa 4 a 6 radici per pianta e ogni radice circa 9-12 cm di lunghezza. Posizionare le radici asportate (taglio come necessarie per adattarsi a) in un tubo da 50ml con etichetta contenente 35 mL di tampone fosfato in autoclave, (6,33 g/L NaH2PO4, 8,5 g/L Na2HPO4 anidro, pH = 6.5, tensioattivo di 200 µ l/L).
Nota: Il tensioattivo (Vedi Tabella materiali) è stato aggiunto dopo la sterilizzazione in autoclave il tampone fosfato. Il volume della sfera di radice e lunghezze di radice variano a seconda di età della pianta e specie di piante. - Agitare le provette per 2 min rilasciare la rizosfera dalla superficie delle radici. Con la pinzetta sterilizzati in 70% EtOH, Rimuovi le radici dal tubo, macchia brevemente su asciugamani di carta e posto in una nuova, con etichetta provetta da 50 mL. Posto sia nella provetta contenente la rizosfera e quello contenente le radici sul ghiaccio.
- Utilizzando le Forbici potatura sterilizzate in 70% EtOH, asportare una varietà delle radici, circa 4 a 6 radici per pianta e ogni radice circa 9-12 cm di lunghezza. Posizionare le radici asportate (taglio come necessarie per adattarsi a) in un tubo da 50ml con etichetta contenente 35 mL di tampone fosfato in autoclave, (6,33 g/L NaH2PO4, 8,5 g/L Na2HPO4 anidro, pH = 6.5, tensioattivo di 200 µ l/L).
3. elaborazione del campo di campioni in laboratorio
- Sterilizzazione superficiale delle radici dopo la raccolta di campi.
Nota: Eseguire la sterilizzazione superficiale appena possibile dopo il ritorno dal campo. Se non è possibile alla superficie sterilizzare le radici nello stesso giorno, memorizzare le radici a 4 ° C fino a quando non elaborati.- Aggiungere circa 35 mL di candeggina 50% + 0.01% Tween 20 per provette da 50 mL radice raccolti sul campo. Agitare provette da 50 mL per 30 a 60 secondi. Versare 50% candeggina e aggiungere 35 mL di 70% EtOH. Agitare per un altro 30 a 60 secondi.
- Versi fuori 70% EtOH e aggiungere 35 mL di acqua distillata sterile. Agitare per 1 minuto. Ripetere il lavaggio con acqua due volte.
Nota: Per garantire che il nostro trattamento di sterilizzazione superficiale è sufficiente, abbiamo placcato in campioni di acqua dall'ultimo risciacquo e non osservato alcuna crescita di batteri (P. Wang, dati non pubblicati, 2014). Altri ricercatori hanno testato per efficienza di sterilizzazione di radice usando simili metodi9,10. - Asciugare le radici sui tovaglioli di carta pulita. Utilizzare un panno di carta per ogni campione.
Nota: Asciugamani di carta possono essere sterilizzati prima dell'uso. Noi non sterilizzare i nostri asciugamani di carta. Tuttavia, usiamo 1 tovagliolo di carta per campione e tenere asciugamani avvolti fino a quando non utilizzato. - Utilizzando pinze sterili e forbici da pota, tagliare le radici a circa 5 mm pezzi e luogo taglia le radici in un tubo conico di pulito, con etichetta 15 mL. Conservare i campioni a-80 ° C fino a quando ulteriormente elaborati.
- Trattamento dei campioni di rizosfera.
- Agitare le provette da 50 mL contenente campioni di rizosfera dal campo per risospendere l'intero campione. Utilizzando un colino di cellule sterili, 100-µm-maglia filtro (Vedi Tabella materiali), il campione di sedimento in una nuova provetta da 50 mL.
- Centrifugare le provette a 3000 x g per 5 min a temperatura ambiente. Immediatamente versare fuori e scartare il surnatante.
- Posto pellet rizosfera nei tubi 50 mL su ghiaccio. Aggiungere 1,5 mL di tampone fosfato sterile (senza tensioattivo) alla rizosfera pellet e vortice di sospendere.
- Dispensare il liquido sospeso in un tubo di microfuge pulito, con etichetta, 2 mL. Gira tubi a 15.871 x g per 2 minuti a temperatura ambiente. Immediatamente versare il sovranatante e svuotare i tubi sui tovaglioli di carta pulita.
- Conservare il pellet a-20 ° C fino a ulteriormente elaborati.
Nota: Passaggi 3.2.2 - 3.2.4 vengono eseguiti per ridurre la dimensione di tubo del campione per l'archiviazione. È più spazio efficiente per memorizzare i più piccoli tubi di 2 mL rispetto ai tubi 50 mL.
- L'elaborazione di campioni di suolo per l'analisi del DNA estrazione e del suolo.
- Con una spatola metallica sterile, riempire una provetta pulita, con etichetta 2 mL con circa 3 g di suolo per l'estrazione del DNA. Evitare qualsiasi radice piccoli pezzi e detriti. Conservare questo campione di suolo a-20 ° C. Spatola di metallo di risciacquo in 70% EtOH tra ogni campione.
- In una padella pulita lavare, svuotare il sacchetto del suolo in setacci in pila (Vedi Tabella materiali), più grande setaccio sopra il setaccio più piccolo e manualmente passare al setaccio il terreno attraverso entrambi setacci. Utilizzare una spazzola per pulire accuratamente i setacci tra campioni.
- Mettere da parte 100-125 g di terreno setacciato in un sacchetto con cerniera 17,7 x 19,5 cm per futuro terreno fisico-chimico e analisi della tessitura. Posizionare i sacchetti del suolo a 4 ° C per la conservazione a breve termine.
- Trattamento dei campioni di suolo per l'umidità del terreno.
- Tare un sacchetto di carta marrone con etichetta su una scala. Misura 40-45 g di terreno setacciato nel sacchetto di carta marrone. Registrare il peso del sacchetto di carta marrone e terreno in un foglio dati e borse posto in un forno di essiccazione impostata a 55-60 ° C.
- Dopo 72 ore, è necessario rimuovere i sacchetti da forno di essiccazione. Consentire le borse di terreno a raffreddare per almeno 30 min e poi pesare.
- Per registrare il peso, tarare la bilancia a zero e mettete i sacchetti di carta marrone sulla bilancia. Calcolare la percentuale di umidità del suolo per ciascun campione utilizzando la formula:
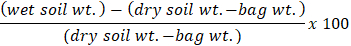
Nota: Il metodo di umidità del suolo è da Kellogg Biological Station Long Term Ecological Research (KBS LTER). KBS LTER fornisce una vasta gamma di protocolli stabiliti per i ricercatori sul loro sito Web (https://Lter.kbs.msu.edu/).
4. preparazione della radice trasformati campioni per estrazioni di DNA.
- Macinare materiale congelato radice affinché il campione è omogeneo.
- Versare azoto liquido in un bicchiere in plastica con spatole pulite e in un pulito mortaio e pestello per mantenere i campioni congelati durante tutto il processo di macinazione.
- Posizionare il tessuto congelato nel mortaio e macinare con il pestello in una polvere fine. Continuamente aggiungere azoto liquido durante la macinazione per mantenere i campioni congelati.
- Utilizzare una spatola per collocare terra tessuto in una provetta pulita, con etichetta 2 mL. Conservare a-80 ° C.
5. estrazione del DNA dal suolo e della rizosfera campioni in formato da 96 pozzetti
- Caricamento campioni di terreno in piastre da 96 pozzetti.
- Pulire l'area di lavoro con 70% EtOH e 1% di candeggina. Indossare guanti di laboratorio durante questi passaggi. Rimuovere i campioni di suolo da stoccaggio-20 ° C e lasciar per scongelare in un secchio di ghiaccio.
- Rimuovere il coperchio opaco tenuto da una piastra 96 pozzetti estrazione che viene fornita con il kit di estrazione del DNA. Posizionare il coperchio opaco tenuto tra 2 salviette di carta per mantenere pulito mentre non in uso. Per evitare la contaminazione, usare adesivo 8 pozzetti PCR strisce per coprire le 12 colonne della piastra 96 pozzetti estrazione.
- Tare un sterile pesare-imbuto (dimensione SM) su una scala e pesare 200-250 mg del suolo.
Nota: Questi imbuti sterili sono appiattiti su un lato, quindi l'imbuto appoggiato in piano su una scala. L'imbuto è riempito con terreno e collocato direttamente in un pozzo. Questa tecnica evita la perdita di campioni, Elimina le perdite e previene la contaminazione incrociata. - Per scoprire il primo pozzetto della piastra di estrazione, sollevare attentamente la striscia adesiva, posto il collo dell'imbuto-pesare riempito negli appositi ben e dolcemente guidare il campione di terreno nel pozzetto corrispondente. Sostituire il nastro adesivo per coprire il pozzo.
- Ripetere questo processo per ogni pozzetto della piastra, utilizzando un imbuto nuovo, sterile per ogni campione fino a quando la piastra è pieno. Lasciare uno ben vuota come un controllo vuoto di estrazione.
Nota: Uno ben viene lasciato vuoto su ogni piastra di estrazione per servire come controllo negativo (in bianco). Controlla di contaminanti che possono essere presenti nel kit di reagenti11. - Ricollocare il coperchio opaco tenuto sulla piastra di estrazione e memorizzare la piastra a-20 ° C fino al momento di estrazione del DNA.
- Caricamento campioni rizosfera in piastre da 96 pozzetti.
- Rimuovere i campioni di rizosfera da stoccaggio-20 ° C e lasciar per scongelare in un secchio di ghiaccio.
- Seguire il passaggio 5.1.2 sopra per preparare la piastra di estrazione del DNA.
- Posizionare una salvietta di carta pulita su una scala, poi una spatola di metallo sterile sulla scala della veccia. Usare la spatola per raccogliere con attenzione alcuni del pellet rizosfera da una provetta. Restituire la spatola sulla scala e pesano fra 200 e 250 mg del campione rizosfera.
- Sollevare delicatamente la striscia adesiva per scoprire il primo pozzetto della piastra di estrazione, inclinare la spatola riempita nel pozzo e raschiare il materiale rizosfera nel pozzo appropriato con uno stuzzicadenti sterile.
- Sciacquare la spatola di metallo in acqua, seguita da 70% EtOH tra campioni. Ripetere questo processo per ogni pozzetto della piastra fino a quando la piastra è pieno, lasciando uno ben vuota come un controllo vuoto di estrazione.
- Estrazione del DNA dai campioni di rizosfera e terreno in formato da 96 pozzetti.
- Estratto di suolo e rizosfera DNA utilizzando un kit ottimizzato per suolo (Vedi Tabella materiali), seguendo il protocollo del produttore.
Nota: Usiamo questo kit specifico per isolare il suolo e della rizosfera del DNA a causa della capacità dei reagenti proprietarie per rimuovere l'acido umico e altri potenti inibitori PCR trovati nel suolo.
- Estratto di suolo e rizosfera DNA utilizzando un kit ottimizzato per suolo (Vedi Tabella materiali), seguendo il protocollo del produttore.
- Quantificazione del DNA.
- Quantificare 92 campioni e 4 concentrazioni degli standard utilizzando un kit (Vedi Tabella materiali; standard sono inclusi), secondo il protocollo del produttore.
- Quantificare i rimanenti 4 campioni che sono stati rimossi dalla piastra per ospitare pozzi per le quattro norme utilizzando un kit (Vedi Tabella materiali), secondo il protocollo del produttore.
6. estrazione di DNA da radice campioni in formato da 96 pozzetti.
- Caricamento campioni radice in piastre da 96 pozzetti.
- Pulire l'area di lavoro con 70% EtOH e 1% di candeggina. Indossare guanti durante questi passaggi.
- Mantenere i campioni di radice di terreno congelati a tutte le volte inserendo i campioni in un secchiello con ghiaccio secco.
- Riempire un bicchiere in plastica con azoto liquido e microspatulas antistatico e sterili pesare-imbuti (dimensione XSM) nel becher plastica raffreddare.
- Rimuovere il coperchio opaco tenuto dalla piastra 96 pozzetti estrazione perlina che viene fornita con il kit e posizionarlo tra 2 salviette di carta per mantenere pulito mentre non in uso.
Nota: La versione più recente di questo kit, rilasciato a seguito del tempo che abbiamo effettuato queste estrazioni di DNA, l'utente deve fornire le piastre di perlina di estrazione. Nella Tabella materiali, abbiamo elencato le informazioni di catalogo fornitore necessarie per ordinare questi articoli. - Per evitare la contaminazione, usare adesivo 8 pozzetti PCR strisce per coprire le 12 colonne della piastra 96 pozzetti estrazione.
- Posizionare la piastra di perlina di estrazione su ghiaccio secco per mantenere i campioni nei pozzetti per surgelati.
- Sollevare con attenzione la striscia adesiva per scoprire il primo pozzetto della piastra di estrazione, posizionare il collo dell'imbuto-pesare riempito nel pozzetto appropriato e aggiungere 3 misurini di spatola del tessuto di radice di terra. Sostituire il nastro adesivo per coprire il pozzo.
Nota: Il terreno e la rizosfera vengono scongelati su ghiaccio prima della pesata, considerando che il materiale vegetale viene pesato congelati. Tessuti vegetali surgelati, soprattutto piccole quantità, sono difficili valutare su una scala senza scongelamento. Pesare prove sono state effettuate sul tessuto di radice di terra per determinare quanti misurini di spatola erano sufficienti. Nota che il produttore del kit non richiede una quantità esatta di tessuto ma raccomanda circa 50 mg. tipi differenti della pianta del campione variano e l'utente sarà necessario determinare la quantità appropriata. - Ripetere questo processo per ogni pozzetto della piastra fino a quando la piastra è pieno.
- Conservare piastra a-20 ° C fino al momento di estrazione del DNA.
- Estrazione del DNA del tessuto principale.
- Estrarre il DNA utilizzando un kit ottimizzato per le piante (Vedi Tabella materiali) seguendo il protocollo del produttore.
Nota: Usiamo questo kit che è stato progettato specificamente per il tessuto di pianta raggiungere tenori massimi dai campioni di radice. A differenza del suolo e rizosfera, l'acido umico e altri contaminanti sono meno di un problema per il tessuto principale.
- Estrarre il DNA utilizzando un kit ottimizzato per le piante (Vedi Tabella materiali) seguendo il protocollo del produttore.
- Quantificare il DNA come descritto al punto 5.4.
7. amplificazione e sequenziamento del DNA isolato.
- Amplificare la regione V4 del gene 16S con una correzione di bozze della polimerasi (Vedi Tabella materiali), come descritto in Gohl et al. 12 campioni di codice a barre con primer indicizzazione diversi e piscina prima di sequenziamento. Sequenza utilizzando i metodi descritti da Gohl et al12 utilizzando un processo in due fasi PCR con primer V4 (Supplemental File 1).
Nota: Alcuni dei metodi per questi passaggi vengono descritti in dettaglio altrove12,13,14 e pertanto non saranno descritti qui. Per gli esempi di radice, bloccanti dei PNA sono stati aggiunti per ridurre la quantità di DNA plastidiale amplificato dal tessuto della pianta, che precedentemente è stato descritto completamente15. Due controlli sono stati utilizzati in sequenza, un controllo negativo che comprendeva i controlli vuoto piastra di estrazione (Vedi nota dopo il passo 5.1.5), e una comunità finta di una popolazione più conosciuta del DNA batterico (Vedi Tabella materiali) che ha servito come un positivo controllo.
Nota: Nella maggior parte dei casi, il kit di reagenti MiSeq v3 sono utilizzati in modalità base fine accoppiati 2 X 300. Per gli esempi in questo manoscritto, il 2500 HiSeq Illumina è stato utilizzato in modalità rapida con 250 Paired-fine (2 x 250) modalità. Tutto il campione sono stati sequenziati nella stessa corsia. - Elaborare i dati di sequenziamento attraverso una pipeline per le analisi di comunità microbica (USEARCH v9.2.64, QIIME v1.9.1 e RStudio v3.4.3 16).
- Preparare i dati di sequenziamento utilizzando USEARCH17.
Nota: USEARCH è disponibile online con le istruzioni complete (https://www.drive5.com/usearch/). - Demultiplexing dei dati di sequenziamento utilizzando indice letture o codici a barre per assegnare Illumina legge ai campioni.
- Unire la fine accoppiato letture per ottenere sequenze consenso. Utilizzare il comando: usearch-fastq_mergepairs * R1*.fastq-rietichettatura @ - fastq_maxdiffs 10 - fastq_minmergelen 230 - fastq_maxmergelen 320 - fastq_pctid 80 - fastqout merged.fq.
Nota: I parametri sono impostati tramite il manuale di istruzioni USEARCH di riferimento. - Rimuovere il primer dai dati di sequenziamento per evitare sostituzioni nelle sequenze dell'iniettore, che possono essere causate tramite la reazione di PCR. Utilizzare il comando: usearch-fastx_truncate merge.fq - stripleft 19 - stripright 20 - fastqout stripped.fq.
- Filtrare i dati di sequenziamento per rimuovere le letture di bassa qualità e mantenere sequenze operative unità tassonomica (OTU) di alta qualità. Utilizzare il comando: usearch-fastq_filter stripped.fq-filtered.fa - fastaout fastq_maxee 1.0.
- Preparare i dati di sequenziamento utilizzando USEARCH17.
- Generare OTUs in USEARCH.
- Eseguire il dereplication per identificare il set di sequenze uniche OTU. Utilizzare il comando: usearch-fastx_uniques filtered.fa - fastaout uniques.fa - sizeout-rietichettare Uniq.
- OTUs di cluster con somiglianza di sequenza 97 – 100% per designare l'unico OTUs. Utilizzare il comando: usearch-cluster_otus uniques.fa - minsize 2 - otus otus.fa-rietichettare Otu.
Nota: Questo passaggio include anche la rimozione di singleton dal cluster OTUs e rimozione delle chimere da dati di sequenziamento. - Creare una tabella OTU in USEARCH. Utilizzare il comando: usearch-usearch_global stripped.fq - db otus.fa-filo plus - id 0.97 - otutabout otutable.txt.
Nota: Questo comando genera una tabella con il numero di letture (conteggi) di OTUs tutti per ogni campione. La tabella OTU viene utilizzata per la procedura a valle, tra cui analisi differenziale abbondanza e diversità microbica analisi. Esempio di tabella OTU è mostrato in figura supplementare 2. - Condurre un'analisi di rarefazione in QIIME v1.9.1 18.
Nota: La curva di rarefazione viene calcolata utilizzando la tabella OTU per determinare se la profondità della sequenziazione campioni correttamente la comunità microbica. Esempio di rarefazione curve sono illustrate in Figura 3 supplementare. - Condurre un' analisi di alfa diversità18. Utilizzare alpha_rarefaction.py in QIIME v1.9.1 per calcolare la diversità della comunità microbica all'interno di ogni campione.
Nota: Questa analisi calcola gli indici di diversità come Shannon19, Simpson20e Chao121. - Condurre un beta diversità analisi18,22. Utilizzare lo script di Python: beta_diversity_through_plots.py nella matrice di dissimilarità v1.9.1 Bray – Curtis QIIME.
Nota: Questa analisi confronta la composizione della comunità microbica tra campioni. - Condurre analisi statistiche tra i gruppi. Utilizzare le matrici di distanza calcolate per PERMONOVA utilizzando la funzione di adonis e anova nel vegano pacchetto23 v2.4.5 RStudio16. Condurre analisi canonica di analisi principali coordinate (CAP) utilizzando la funzione di capscale nel pacchetto vegano. Visualizzare i dati utilizzando ggplot2 pacchetto24 v 2.2.1 in RStudio.
Risultati
I risultati rappresentativi presentati in questo manoscritto provengono da un sito di campo fondato nel 2012 presso l'University of Nebraska-Lincoln agricoltura ricerca divisione azienda agricola vicino a Mead, Nefi prima dell'esperimento, il sito era stato gestito come una rotazione mais-soia . Il sito di studio era situato su tre diversi tipi di suoli, ma i dati sono stati analizzati come se tutte le modifiche in proprietà del suolo misurati erano dovuto i trattamenti imposti.
Il campo sito conteneva due, puro, basamenti di switchgrass (p. virgatum cv Liberty) e grande bluestem (a. gerardii) così come una miscela di erba bassa-diversità contenente bluestem grande, indiangrass (S. nutans) e 'Butte' sideoats grama ( B. curtipendula). I tre appezzamenti di erba stagione calda erano in un disegno randomizzato blocco completo che è stato replicato tre volte. Annidate in tre appezzamenti di erba differenti erano due trattamenti di fecondazione di azoto (N), che erano 56 (N1) e 112 (N2) kg N ha-1 di urea applicata. Al momento del campionamento microbiome alla fine del periodo della crescita, il nitrato di ppm (media ± SD) 8,0 ± 1.1 suolo contenuto nelle trame fertilizzate con 112 kg N ha-1 e 6,8 ± 0,7 (media + SD) nitrato ppm nelle trame fertilizzate con 56 kg N ha-1. Le trame erano state fecondate una volta all'anno. L'erba di stagione calda appezzamenti sono stati designati come le trame principali (8000 m2) e N trattamenti erano le trame di Spalato (4000 m2). Bluestem grande è stato seminato come una miscela di 50: 50 di 'Bonanza' e 'Goldmine' e Indiangrass è stato seminato come una miscela di 50: 50 di 'Scout' e 'Guerriero'. Le trame sono state piantate nel 2012, e la prima applicazione di N si è verificato nella primavera del 2013.
Il terreno e il campionamento di radice è stato condotto su 15 settembre 2014. Il lavoro descritto di seguito è stato condotto su un campo che è stato istituito come un disegno randomizzato di dividere-trama con tre repliche (Figura 1). La profondità media sequenziamento di tutti i campioni per endosphere erano come segue: 4871 ± 5711 (media ± SD), rizosfera: 40726 ± 14684, terreno: 38184 ± 9043. Una delle maggiori fonti di variazione in questi esperimenti, utilizzando i metodi descritti, è la differenza nella comunità microbiche trovata fra tipi di campione (Figura 2). In questo set di dati rappresentativo, la rizosfera e terreno sembrano essere più simile in composizione a vicenda che il endosphere (Figura 2A). Tuttavia, c'erano anche altamente significativa (p = 0,001) differenze nella composizione della comunità microbica rizosfera e terreno (Figura 2B). La variazione totale hanno rappresentata in questi esperimenti analizzati dal tipo di campione era 26%.
Alfa diversità l'analisi ha mostrato che le comunità microbiche nella endosphere erano più bassi nella diversità del campione rispetto al suolo e della rizosfera (Figura 3). Le uniche differenze significative nella diversità tra le specie di erba in qualsiasi vano erano tra i campioni di endosphere di grande bluestem e switchgrass, con switchgrass avendo significativamente più alta diversità di specie microbiche (Figura 3). L'analisi relativa abbondanza (Figura 4) evidenzia la dominanza di Proteobacteria seguita da Actinobacteria in tutti i tipi di campioni. Suolo e della rizosfera inoltre sono dominati da Acidobacteria e Chloroflexi mentre il endosphere ha avuto una più grande abbondanza relativa di Bacteriodetes.
In questo esperimento, piante sono state coltivate con due diverse quantità di fertilizzante N e pertanto abbiamo analizzato i dati per determinare se c'erano gli effetti del trattamento. Gli effetti del trattamento rappresentavano il 12% della variazione totale ma non erano significativamente differenti anche se nell'ordinazione i due trattamenti aspetto diverso (Figura 5). Ciò evidenzia l'importanza delle analisi statistiche per questi DataSet anziché ispezione visiva o giudizi qualitativi.
Pianta-influenzato differenze nel microbioma di tessuti vegetali e di terreno sono state visualizzate utilizzando un metodo vincolato dell'ordinazione. Le differenze statistiche sono state determinate mediante un'analisi PERMANOVA per verificare se variabili specifiche, come specie, provocare la composizione della comunità microbica significativamente differente tra i campioni. Quando tutti i tipi di campioni sono stati analizzati insieme, una differenza altamente significativa è stata trovata nella composizione della comunità microbica dovuto la specie di pianta (Figura 6). In questo esperimento, la quantità di variazione rappresentata dalle specie vegetali era 6,7%. Infine, ogni tipo di campione è stato analizzato singolarmente per determinare quale dei tipi del campione potrebbe essere guida l'effetto di specie di pianta significativo. Solo nella endosphere c'era una differenza altamente significativa (p = 0,001) tra le composizioni di comunità microbica di diverse specie vegetali (Figura 7). In altri tipi di campione, l'effetto di specie non era significativa quando analizzate singolarmente. In endosphere, la variazione percentuale dovuto la specie era 27%, mentre era più basso nella rizosfera (18%) e terreno (15%). Questo evidenzia ulteriormente l'importanza di analizzare singolarmente ogni tipo di tessuto.
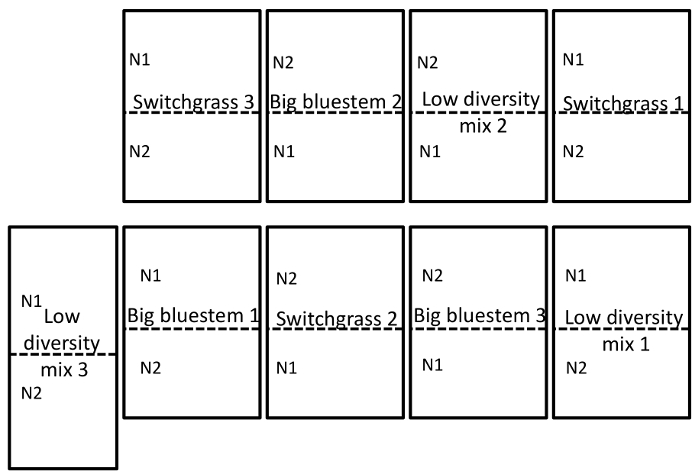
Figura 1: esempio di progettazione campo sperimentale. Progettazione campo sperimentale che illustra un disegno randomizzato blocco completo in triplice copia del sito campo situato presso la University of Nebraska-Lincoln orientale Nebraska ricerca ed Extension Center vicino a Mead, NE. Per la descrizione del sito completo vedere la sezione di risultati. N1 è il basso (56 kg N ha-1 urea) e N2 (112 kg N ha-1 urea) è il più alto tasso di azoto che è stato applicato. Clicca qui per visualizzare una versione più grande di questa figura.
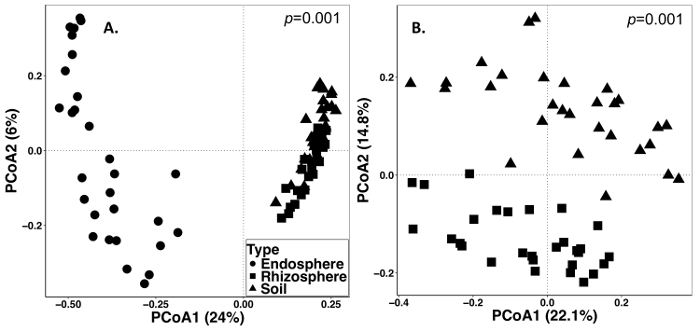
Figura 2: analisi di Beta diversità confrontando la composizione microbica nei tipi differenti del campione tra cui endosphere, rizosfera e suolo dal campionamento erba perenne nel 2014. L'analisi è stata effettuata utilizzando uno script di Python in QIIME1.9.1 per produrre la matrice di dissimilarità di Bray-Curtis. Analisi di coordinate principali (PCoA) basato sulla matrice di dissimilarità di Bray-Curtis è stata visualizzata in RStudio. PCoA1 e PCoA2 indicano la prima e la seconda più grande varianza spiegata dall'analisi PCoA. PERMANOVA analisi statistica è stata eseguita per determinare il significato tra tipi di campioni, e il valore di p è visualizzato nell'angolo superiore destro. Ogni simbolo nelle figure rappresentano l'intera comunità microbica per ciascun campione. (A) Endosphere, rizosfera e suolo tipi di campioni sono stati analizzati insieme. Tutti i 87 campioni erano rarefatto a 486 sequenze per campione. (B) rizosfera e suolo campioni sono stati analizzati insieme. Tutti i 59 campioni erano rarefatte con 8231 sequenze. Clicca qui per visualizzare una versione più grande di questa figura.
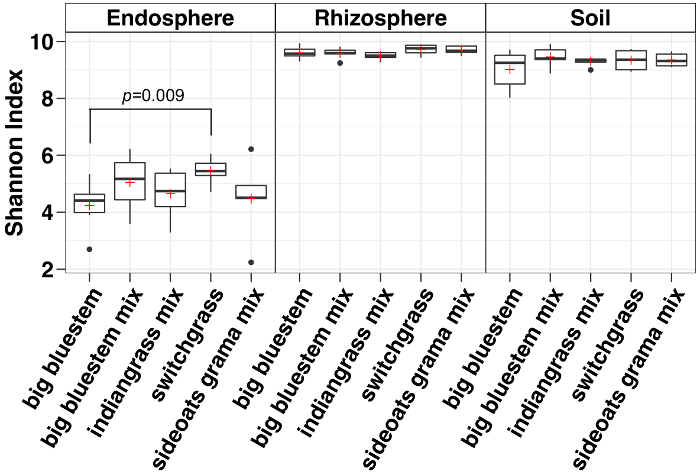
Figura 3: analisi di diversità alfa utilizzando indice di Shannon per ciascuna specie nell'endosphere, rizosfera e nel terreno. L'analisi è stata effettuata utilizzando uno script di Python nella QIIME1.9.1. Rarefazione è stato fatto per l'endosphere, rizosfera e tipi di campione di suolo rispettivamente con sequenze 486, 17154 e 8231 per campione. Le caselle indicano i percentili 25 e 75 ° (primi e terzi quartili). La linea orizzontale all'interno della casella denota la mediana e il rosso più Mostra la media. Baffi Visualizza l'intervallo di dati escluse outlier (che appaiono come puntini neri) che è caduto più di 1,5 volte lo scarto interquartile (n = 6 per ogni campione tranne per sideoats grama mix dove n = 5). L'indice di Shannon di tutte le cinque specie del endosphere erano più bassi di rizosfera e il suolo. Non-parametrico Wilcoxon rank sum test è stato utilizzato per determinare il significato tra le specie e solo le differenze significative tra le specie sono state mostrate in cima le caselle. Clicca qui per visualizzare una versione più grande di questa figura.
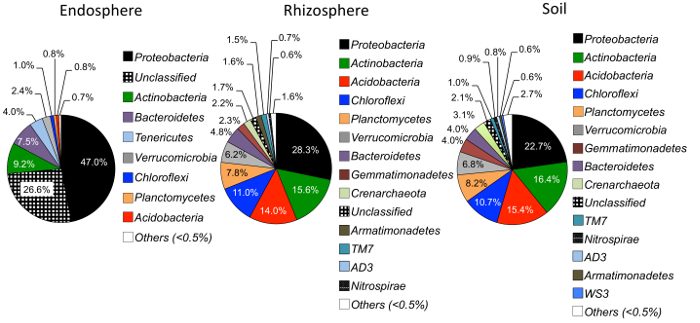
Figura 4: abbondanza relativa al livello di phylum endosphere, rizosfera, e suolo. I campioni sono stati analizzati per confrontare l'abbondanza dei phyla microbica tra tipi diversi campioni (n = 29 per ogni tipo di campione). L'analisi è stata effettuata utilizzando uno script di Python in QIIME1.9.1 dalla tabella OTU. I diversi colori all'interno del grafico a torta denotano i phyla. La percentuale indica l'abbondanza relativa di ogni phylum in ogni tipo di campione. Le informazioni di phylum è state annotate usando il progetto di Database Ribosomal classificatore (RDP)25. Clicca qui per visualizzare una versione più grande di questa figura.
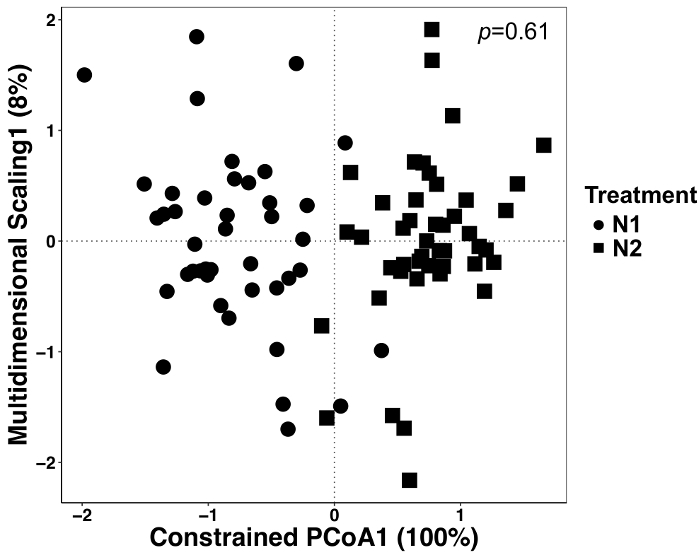
Figura 5: analisi mediante trattamento come fattore vincolante tra tutti i tipi di campioni. Analisi canonica di analisi principali coordinate (CAP) è stato effettuato per determinare se c'erano differenze nella composizione della comunità microbica fra i trattamenti. Per ogni trattamento di N, n = 42 per N1 (56 kg N ha-1) e n = 45 per N2 (112 kg N ha-1). La matrice di dissimilarità di Bray-Curtis è stata generata utilizzando uno script python nella QIIME1.9.1. Analisi CAP basato sulla matrice di dissimilarità di Bray-Curtis è stato fatto vincolando il trattamento come il fattore RStudio. PERMANOVA analisi è stata effettuata per determinare se le differenze di trattamento erano significative, e il valore di p è visualizzato nell'angolo superiore destro. Clicca qui per visualizzare una versione più grande di questa figura.
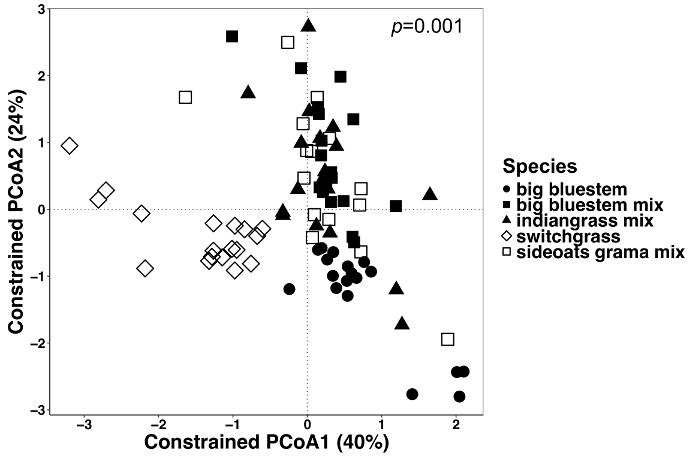
Figura 6: analisi utilizzando specie vegetali come fattore vincolante tra tutti i tipi di campioni. Analisi è stata condotta per determinare se c'erano differenze nella composizione comunità microbica tra specie di piante in tutti i tipi di campioni. Ordinazione di coordinate principali e analisi di CAP di tutti i tipi di campione (endosphere, rizosfera e suolo) sono stati fatti utilizzando una matrice di dissimilarità di Bray-Curtis. La matrice di dissimilarità di Bray-Curtis è stata generata utilizzando lo script Python in QIIME1.9.1. Analisi CAP basato sulla matrice di dissimilarità di Bray-Curtis è stato fatto vincolando le specie vegetali come il fattore RStudio. PERMANOVA analisi statistica è stata eseguita per determinare il significato tra specie vegetali, e il valore di P è visualizzato nell'angolo superiore destro. Ogni simbolo nelle figure rappresenta l'intera comunità microbica per quel campione. n = 18 per ciascuna specie in tutti i tipi di campione tranne n = 15 per il mix di grama sideoats. Clicca qui per visualizzare una versione più grande di questa figura.
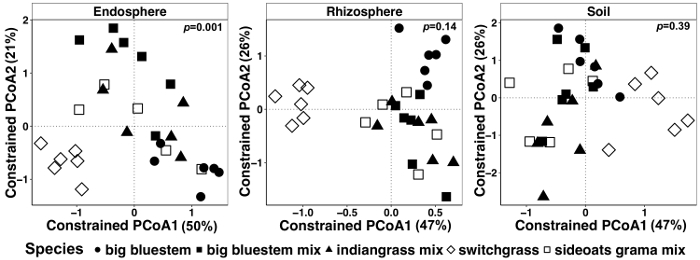
Figura 7: esempio di analisi di CAP utilizzando specie come fattore vincolante per ogni tipo di campione individualmente. Ordinazione delle coordinate principali e l'analisi di CAP di ogni tipo di campione (endosphere, rizosfera e suolo) utilizzando la matrice di dissimilarità di Bray-Curtis. Ogni tipo di campione era rarefatto a 486, 17154 e 8231 letture per campione rispettivamente in endosphere, rizosfera e nel terreno. Specie è stato utilizzato come il fattore per vincolare l'ordinazione. PERMANOVA analisi statistica è stata eseguita per determinare il significato tra specie di piante di ogni tipo di campione, e il valore di p è visualizzato nell'angolo superiore destro. Ogni simbolo di figura rappresenta l'intera comunità microbica per ciascun campione. Dimensione del campione è n = 29 per ogni tipo di campione, n = 6 per ogni specie di piante di ogni tipo di campione tranne che per il mix di grama sideoats (n = 5). Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
I metodi descritti in questo manoscritto dovrebbero consentire agli scienziati di entrare facilmente nel campo della metagenomica del suolo e pianta. Nel corso degli anni, abbiamo affinato i nostri metodi dal condurre l'esperimento descritto in questo manoscritto. Un cambiamento è che abbiamo ora pre-Etichettare provette prima di andare al campo per esempio. Il nostro laboratorio utilizza un sistema di codici a barre e stampante di etichette. La stampante di etichette non solo di risparmiare tempo quando etichettatura tubi, ma anche rende tutto più facile per tenere traccia e identificare correttamente i campioni senza i capricci di scrittura della mano umana. Un altro punto critico è che cerchiamo di elaborare il materiale dopo portarlo indietro dal campo appena possibile. Miriamo a congelare il terreno utilizzato per l'analisi del DNA, sterilizzare e congelare le radici e filtrare e bloccare la rizosfera entro 12-36 ore dopo il ritorno dal campo. Le procedure di estrazione del DNA sono lunghe con molti passaggi, specialmente per suolo e della rizosfera, così abbiamo acquistato un robot (Kingfisher Flex, ThermoFisher) che minimizza le mani in tempo per i protocolli di estrazione del DNA, riduce l'errore umano che può essere introdotte, e migliora l'uniformità in modo diversi lotti di terreno, radici o rizosfera vengono elaborati. Quando si lavora con materiale vegetale che è importante decidere il tipo di radice per essere studiato o prendere una varietà di tipi di radice per ottenere un "campione rappresentativo". Mantenimento di radici e foglie in stato congelato quando conducendo le estrazioni di DNA è importante, in quanto consiste nel garantire non c'è nessuna contaminazione tra campioni durante la compilazione di piastre da 96 pozzetti DNA estrazione. Un altro fattore importante da considerare è il numero delle ripetizioni da utilizzare durante la progettazione di esperimenti sul campo e utilizzando un completo randomizzati progettazione ove possibile26. A causa della variabilità di alto campo può essere necessario disporre di un gran numero di repliche per rilevare piccole differenze. Infine, dalla nostra esperienza è essenziale per assicurarsi che i suoli non sono troppo bagnati quando le radici di scavo. Se i terreni sono saturi d'acqua non è solo disordinato a lavorare con, ma è anche molto difficile definire la rizosfera e togliere la terra dalle radici.
Una modifica che è stata fatta presto durante lo sviluppo di questi metodi è stato invece scuotendo i tubi a mano per rilasciare la rizosfera che ci siamo trasferiti in vortexers alimentato da un generatore di gas per rendere più facile il lavoro in campo e più standardizzata in termini di tempi e nei modi che ogni tubo era agitato. Una limitazione dell'approccio sequenziamento degli ampliconi è che la risoluzione tassonomica dei risultati è spesso limitata e molti OTUs sono conosciuti sconosciuti o solo a livello di famiglia o genere. Questo campo di ricerca è in rapida evoluzione quindi è importante essere consapevoli di approcci nuovi e in via di sviluppo, in particolare per l'analisi di dati che possono migliorare la risoluzione dei risultati.
Questi protocolli sono solo per lo studio di batteri e archaea, non funghi. L'uso di primer diversi per amplificazione permetterà per lo studio delle Comunità fungine utilizzando la stessa DNA campioni27,,28. Questi metodi non richiedono l'acquisto di grandi quantità di attrezzature, perché i metodi possono essere semplificati. I metodi che descriviamo qui sono principalmente per la determinazione di "chi è", ma il campo è in rapida evoluzione in domande importanti sulla funzione, che può essere risolto utilizzando metodi di sequenziamento shotgun, isolamento e controllo della funzionalità dei microbi o genomi interi microbici di sequenziamento.
I risultati rappresentativi evidenziano le differenze nelle comunità microbiche che possono essere identificate utilizzando i metodi descritti. Utilizzando un approccio di beta-diversità per l' analisi di dati22, differenze composizionali sono state indicate tra tipi di campioni. Queste differenze sono state osservate chiaramente nella maggior parte dei altri studi dove endosphere, rizosfera e suolo contengono unica comunità microbiche3. L'indice di diversità di Shannon è stata calcolata per determinare l'abbondanza e la regolarità delle specie microbiche presenti all'interno di ogni specie di pianta endosphere, rizosfera, e suolo. Come mostrato in questo studio e in molti altri, alfa diversità è più alta nel terreno, diminuendo leggermente nella rizosfera e quindi diminuendo significativamente nel endosphere3,5,29. Questi risultati indicano che i metodi descritti qui sono adatti per l'identificazione di cambiamenti composizionali endosphere, rizosfera, e suolo.
La dominanza di Proteobacteria è un'individuazione comune negli studi endosphere e terreno30,31,32. Endosphere ha generalmente una bassa diversità di specie microbiche con una maggiore abbondanza relativa di Proteobacteria. Questo sottolinea ancora una volta che i risultati qui sono rappresentante di altri risultati nella letteratura. Gli effetti del trattamento in questo studio non erano significativamente differenti e due ragioni principali per cui potrebbero essere che le differenze imposte dai trattamenti non erano abbastanza grandi per generare variazione sufficiente per rilevare e che il campionamento è stato fatto alla fine della stagione, quando i campi possono avere avuto tempo sufficiente per trarre verso il basso l'azoto a simili livelli, che è quello che è stata misurata alla fine della stagione di crescita. In un altro studio utilizzando simili tassi di fertilizzazione su un periodo più lungo di tempo, solo relativamente piccoli cambiamenti nella composizione del microbioma sono stati misurati33. Altri studi hanno mostrato cambiamenti nelle comunità fungine e batteriche a causa di azoto fertilizzante34,35.
Specie di piante sono notoriamente giocano un ruolo nel determinare la loro microbiomi3,32,36 e anche piccole differenze nella variazione di comunità microbica sono state dimostrate tra genotipi differenti della pianta all'interno di un singola specie37. In questo studio, una differenza significativa nella composizione della comunità microbica è stata trovata fra specie vegetali. In tutti i tipi di campione sembrava che switchgrass aveva la composizione microbica più distinta, ma le differenze tra le specie erano solo statisticamente significative nella endosphere. Composizione della Comunità rizosfera può diventare significativo se ulteriori repliche erano disponibili per l'analisi.
Il campo combinato, laboratorio e protocolli analitici descritti qui offrono un metodo efficace per studiare come i diversi fattori influenzano la composizione delle comunità microbiche nel suolo, rizosfera e la endosphere di radici36. C'è una grande quantità di lavoro da fare nella zona di studiare microbiomi, specialmente in campi agricoli. Domande importanti su come i rendimenti sono alterati dal microbioma del suolo hanno ancora completamente essere delucidato. Anche le più elementari domande relative a come rotazioni delle colture influenzare il microbioma del suolo, come tempi altera il microbioma, stress abiotici come altera il microbioma, come tipo di suolo interagisce con questi fattori per alterare il microbioma e se ci sono i microbi universali in determinate colture o le regioni degli Stati Uniti sono tutte le questioni aperte. Questi metodi sarà anche utili per studi epidemiologici per identificare la presenza e la persistenza di batteri patogeni e benefici. Un altro orizzonte futuro per questi metodi sarà per iniziare l'integrazione di metodi DNA descritti qui con pianta e microbo RNA e metabolita dati. Ulteriore miglioramento e test di più variabili saranno importanti per un'ulteriore ottimizzazione di questi protocolli.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Lo sviluppo di questo manoscritto è sostenuto dal National Science Foundation EPSCoR Center per Root e Rhizobiome Innovation Award OIA-1557417. La raccolta dei dati è stata sostenuta dai fondi dalla University of Nebraska-Lincoln, ricerca agricola e lo sviluppo e da una sovvenzione di tratteggio dall'USDA. Riconosciamo anche il supporto dal USDA-ARS e il supporto è stato fornito dall'agricoltura e cibo ricerca iniziativa concorrenziale Grant n ° 2011-68005-30411 dal USDA National Institute di cibo e l'agricoltura a stabilire e gestire questi campi.
Materiali
| Name | Company | Catalog Number | Comments |
| Dneasy PowerSoil HTP 96 Kit | Qiagen/MoBio | 12955-4 | Extraction kit for soil and rhizosphere |
| Dneasy PowerPlant HTP 96 Kit | Qiagen/MoBio | 13496-4 | Extraction kit for roots |
| D-Handle Digging shovel, 101 cm L | Fiskars | 9669 | |
| Rapid Tiller, 40 cm L | Truper | 34316 | |
| Ziploc Bags, 17.7 cm x 19.5 cm | Ziploc | NA | |
| Cooler | Any | NA | |
| Wash pan | Any | NA | |
| Plastic bucket | Any | NA | |
| Gloves (work and lab) | Any | NA | |
| 20 cm diameter Soil sieve #8, 2360 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-8FS | |
| 20 cm diameter Soil Sieve #4, 4750 μm mesh size | Dual Manufacturing Co., Chicago IL | US8-4FS | |
| portable generator | Honda brand works well | NA | |
| Sterile cell strainers 100 μm mesh size | Fisher Scientific | 22-363-549 | |
| NaH2PO4·H2O | VWR | 0823 | |
| Na2HPO4 | VWR | 0404 | |
| Silwet L-77 | Lehman Seeds | VIS-30 | Surfactant |
| Autoclaves | Any | NA | |
| Drying Oven | Any | NA | |
| Scale | Any | Any | |
| Bleach | CLOROX - household strength | NA | |
| Tween 20 | Any | NA | |
| Liquid Nitrogen | Any | NA | |
| Dry Ice pellets | Any | NA | |
| Ethanol | Any | NA | |
| 11 cm precision fine point tweezers | Fisher | 17456209 | |
| 18 cm Straight point specimen forceps | VWR | 82027-436 | |
| 13.5 cm Pruning Scissors | Fiskars | 9921 | |
| 2 mL tube | Any | NA | |
| 15 mL PP conical tube | MIDSCI | C15B | |
| 50 mL PP conical tube | MIDSCI | C50B | |
| Ultrapure water | Millipore-sigma | Milli-Q Integral, Q-POD | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32854 | For DNA quantification of removed samples |
| QuantiFluor dsDNA System | Promega | E2670 | For DNA quantification |
| 96-Well Black with Clear Flat Bottom Plates | Corning | 3631 | |
| pPNA PCR Blocker | PNA Bio | PP01-50 | |
| mPNA PCR Blocker | PNA Bio | MP01-50 | |
| Genomic DNA from Microbial Mock Community B (Even Low Concentration) v5.1L, for 16s rRNA Gene sequencing | BEI Resources | HM782D | |
| Adhesive 8 well-strips for plates | VWR | 89134-434 | |
| Stainless steel beads, 3.2 mm dia | Next Advance | SSB32 | |
| 1 ml Assay block (DNA extraction plate for the Qiagen/MoBio Dneasy PowerPlant HTP Kit) | CoStar | 3959 | |
| antistatic PP weighing funnel, size small for soil/rhizosphere | TWD Tradewinds, INC | ASWF1SPK | |
| antistatic PP weighing funnel, size x-small for root/leaf | TWD Tradewinds, INC | ASWFXSCS | |
| Genie 2 Digital Vortex | Scientific Industries | SI-0236 | |
| Vortex adapter for 50 mL tubes | Scientific Industries | SI-H506 | |
| Mortar (100 mL) and pestle | Any | NA | |
| Metal micro-spatula | VWR | 80071-672 | |
| Disposable antistatic microspatulas | VWR | 231-0106 | |
| Brown Paper bag 2# (10.95 cm x 6.19 cm x 20 cm) | Duro | 18402 | |
| 5424 Centrifuge for 2 mL tube | Eppendorf | 22620461 | |
| Centrifuge for 96-well plate | Sigma4-16S | 81510 | |
| Centrifuge rotor for 50 mL tubes | Sigma4-16S | 12269 - Biosafe | |
| KAPA HiFi DNA polymerase | Kapa Biosystems |
Riferimenti
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: the microbial ecology of the rhizosphere. Nature Reviews Microbiology. 11, 789-799 (2013).
- Fierer, N. Embracing the unknown: disentangling the complexities of the soil microbiome. Nature Review Microbiology. 15 (10), 579-590 (2017).
- Wang, P., et al. Shifts in microbial communities in soil, rhizosphere and roots of two major crop systems under elevated CO2 and O3. Scientific Reports. 7 (1), 15019(2017).
- White, L. J., Jothibasu, K., Reese, R. N., Brözel, V. S., Subramanian, S. Spatio Temporal influence of isoflavonoids on bacterial diversity in the soybean Rhizosphere. Molecular Plant-Microbe Interactions. 28 (1), 22-29 (2015).
- Edwards, J., et al. Structure, variation, and assembly of the root-associated microbiomes of rice. Proceedings of the National Academy of Science of the United States of America. 112 (8), E911-E920 (2015).
- Lundberg, D. S., et al. Defining the core Arabidopsis thaliana root microbiome. Nature. 488, 86-90 (2012).
- Bougoure, D. S., Cairney, J. W. Assemblages of ericoid mycorrhizal and other root-associated fungi from Epacris pulchella (Ericaceae) as determined by culturing and direct DNA extraction from roots. Environmental Microbiology. 7 (6), 819-827 (2005).
- Doty, S. L., et al. Diazotrophic endophytes of native black cottonwood and willow. Symbiosis. 47 (1), 23-33 (2009).
- Gottel, N. R., et al. Distinct microbial communities within the endosphere and rhizosphere of populus deltoides roots across contrasting soil types. Applied and Environmental Microbiology. 77 (17), 5934-5944 (2011).
- Xin, G., Glawe, D., Doty, S. L. Characterization of three endophytic, indole-3-acetic acid-producing yeasts occurring in Populus trees. Mycological Research. 113, 973-980 (2009).
- Salter, S. J., et al. Reagent and laboratory contamination can critically impact sequence-based microbiome analyses. BMC Biolology. 12, 87(2014).
- Gohl, D. M., et al. Systematic improvement of amplicon marker gene methods for increased accuracy in microbiome studies. Nature Biotechnology. 34 (9), 942-949 (2016).
- Kozich, J. J., Westcott, S. L., Baxter, N. T., Highlander, S. K., Schloss, P. D. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform. Applied Environmental Microbiology. 79 (17), 5112-5120 (2013).
- Caporaso, J. G., et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. ISME Journal. 6 (8), 1621-1624 (2012).
- Lundberg, D. S., Yourstone, S., Mieczkowski, P., Jones, C. D., Dangl, J. L. Practical innovations for high-throughput amplicon sequencing. Nature Methods. 10 (10), 999-1002 (2013).
- RStudio, T. RStudio: Integrated Development for R. RStudio, Inc. , Available from: http://www.rstudio.com/ (2015).
- Edgar, R. C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods. 10 (10), 996-998 (2013).
- Caporaso, J. G., et al. QIIME allows analysis of high-throughput community sequencing data. Nature Methods. 7 (5), 335-336 (2010).
- Shannon, C. A mathematical theory of communication. Bell System Technical Journal. 27, 379-423 (1948).
- Simpson, E. H. Measurement of diversity. Nature. 163, 688(1949).
- Chao, A. Non-parametric estimation of the number of classes in a population. Scandanavian Journal of Statistics. 11, 265-270 (1984).
- Legendre, P. Studying beta diversity: Ecological variation partitioning by multiple regression and canonical analysis. Journal of Plant Ecology. 1 (1), 3-8 (2008).
- Oksanen, J., et al. The vegan package. Community Ecology Package. 10, 631-637 (2007).
- Wickham, H. ggplot2: Elegant graphics for data analysis. Ggplot2: Elegant Graphics for Data Analysis. , Springer Nature. Switzerland. 1-212 (2009).
- Cole, J. R., et al. The Ribosomal Database Project (RDP-II): sequences and tools for high-throughput rRNA analysis. Nucleic Acids Research. 33, D294-D296 (2006).
- Wagner, M. R., et al. Host genotype and age shape the leaf and root microbiomes of a wild perennial plant. Nature Communications. 7, 12151(2016).
- O'Brien, H. E., Parrent, J. L., Jackson, J. A., Moncalvo, J. M., Vilgalys, R. Fungal community analysis by large-scale sequencing of environmental samples. Applied and Environmental Microbiology. 71 (9), 5544-5550 (2005).
- Taylor, D. L., et al. A first comprehensive census of fungi in soil reveals both hyperdiversity and fine-scale niche partitioning. Ecological Monographs. 84 (1), 3-20 (2014).
- Lebeis, S. L., et al. Salicylic acid modulates colonization of the root microbiome by specific bacterial taxa. Science. 349 (6250), 860-864 (2015).
- Ofek-Lalzar, M., et al. Niche and host-associated functional signatures of the root surface microbiome. Nature Commununications. 5, 4950(2014).
- Niu, B., Paulson, J. N., Zheng, X., Kolter, R. Simplified and representative bacterial community of maize roots. Proceedings of the National Academy of Science of the United States of America. 114 (12), E2450-E2459 (2017).
- Fitzpatrick, C. R., et al. Assembly and ecological function of the root microbiome across angiosperm plant species. Proceedings of the National Academy of Science of the United States of America. 115 (6), E1157-E1165 (2018).
- Ramirez, K. S., Lauber, C. L., Knight, R., Bradford, M. A., Fierer, N. Consistent effects of nitrogen fertilization on soil bacterial communities in contrasting systems. Ecology. 91 (12), 3463-3470 (2010).
- Paungfoo-Lonhienne, C., et al. Turning the table: Plants consume microbes as a source of nutrients. PLoS One. 5 (7), e11915(2010).
- Yeoh, Y. K., et al. The core root microbiome of sugarcanes cultivated under varying nitrogen fertilizer application. Environmental Microbiology. 18 (5), 1338-1351 (2016).
- Bulgarelli, D., Schlaeppi, K., Spaepen, S., Ver Loren van Themaat, E., Schulze-Lefert, P. Structure and functions of the bacterial microbiota of plants. Annual Review of Plant Biology. 64, 807-838 (2013).
- Peiffer, J. A., et al. Diversity and heritability of the maize rhizosphere microbiome under field conditions. Proceedings of the National Academy of Science of the United States of America. 110 (16), 6548-6553 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon