Method Article
Microscopia a luce correlativa (CLEM) per il monitoraggio e la proteina virale di Imaging strutture in cellule di Cryo-immobilizzata associate
In questo articolo
Riepilogo
Un metodo di microscopia luce correlativa (CLEM) viene applicato alle strutture intracellulari indotte da virus immagine tramite microscopia elettronica (EM) in cellule che sono già stati scelti da microscopia chiara (LM). LM ed EM sono combinati in un approccio di imaging ibrido per ottenere una visione integrata delle interazioni virus-ospite.
Abstract
Grazie alla sua alta risoluzione, la microscopia elettronica (EM) è uno strumento indispensabile per virologi. Tuttavia, una delle principali difficoltà quando si analizzano le cellule transfettate o infettate da virus tramite EM sono la minor efficienza di infezione o trasfezione, ostacolando l'esame di queste cellule. Per superare questa difficoltà, microscopia (LM) può essere eseguita prima per allocare la sottopopolazione delle cellule trasfettate o infetti. Così, approfittando dell'uso di proteine fluorescenti (FPs) fuso a proteine virali, LM è qui utilizzato per registrare le posizioni delle cellule "trasfettate con positivo", esprimendo un FP e che cresce su un supporto con un modello alfanumerico. Successivamente, le cellule sono ulteriormente trattate per EM tramite alta pressione congelamento (HPF), congelare sostituzione (FS) e l'incorporamento di resina. Il passo di congelamento ultrarapido garantisce membrana eccellente conservazione delle celle selezionate che possono poi essere analizzati a livello ultrastrutturale di microscopia elettronica a trasmissione (TEM). Qui, viene fornito un flusso di lavoro luce correlativa passo dopo passo la microscopia elettronica (CLEM), che descrive la preparazione del campione, imaging e correlazione in dettaglio. Il disegno sperimentale può essere anche applicato per risolvere molte questioni di biologia delle cellule.
Introduzione
L'idea di combinare due modalità di microscopia per ottenere una migliore immagine di un determinato processo biologico è piuttosto vecchio. Così, il primo studio sui virus usando "correlativa microscopia" è stato pubblicato nel 1960 come due pubblicazioni distinte1,2. In quello studio, gli autori hanno analizzato i cambiamenti nella morfologia del nucleo indotta da adenovirus mediante due tecniche di microscopia. Nella prima pubblicazione, osservazioni di microscopia elettronica (EM) che descrive i dettagli morfologici connessi con l'infezione dell'adenovirus sono stati segnalati1. In una seconda pubblicazione, le diverse strutture osservate da EM sono state correlate con immagini di microscopia (LM) di pattern di colorazione istochimica, per definire la natura delle strutture precedentemente osservato da EM2.
In questi primi studi, tuttavia, le loro osservazioni sono state eseguite utilizzando differenti cellule infettate preparate come esperimenti indipendenti. La "correlazione" era, infatti, inteso come la combinazione di informazioni provenienti da due modalità di imaging per capire un certo fenomeno, confrontando tutti i reperti che sono stati ottenuti con dosaggi diversi al fine di comprendere un dato biologico processo.
Al giorno d'oggi, la microscopia correlativa di termine, noto anche come correlativo luce e microscopia elettronica (CLEM), viene applicata a un numero crescente di metodi (rivisto in riferimenti3,4,5), con la comunanza che sia tecniche di imaging (LM ed EM) vengono effettuate sullo stesso campione. La combinazione di entrambi i metodi risulta, quindi, in un'analisi multi-modale, multi-scala e multi-dimensionale di quel campione3. I vantaggi sono che LM può fornire un'ampia panoramica di molte cellule diverse, che consentano l'identificazione di sottopopolazioni di cellule esprimenti una proteina o le proteine di interesse all'interno di una popolazione eterogenea delle cellule. EM supera il limite di risoluzione di LM, fornendo un'immagine ad alta risoluzione di un particolare evento intracellulare. Inoltre, EM consente la visualizzazione del contesto subcellulare non fluorescenti, compresi tutti gli organelli membrana associato, grandi complessi macromolecolari (ad es., ribosomi, centrioli, ecc.) ed elementi del citoscheletro, quindi fornendo ulteriori informazioni spaziali, il cosiddetto "spazio di riferimento"6e dare un contesto allo spot fluorescente rilevati da LM.
Negli ultimi anni, CLEM è diventato un potente strumento non solo per cella biologi5, ma anche per virologi (rivisto in riferimento7) disposti a comprendere le interazioni virus-cellula complesse che portano ad una propagazione di virus di successo. Così, come virus modificare le membrane cellulari e organelli a proprio vantaggio la comprensione è essenziale per sviluppare farmaci antivirali per sradicare il virus patogeni.
Qui, un metodo di CLEM è descritto che permette la rilevazione di LM delle cellule che esprimono proteine virali fuse ad una proteina fluorescente (FP). Queste cellule vengono successivamente cryo-immobilizzata e ulteriormente preparato per l'analisi ultrastrutturale tramite microscopia elettronica di trasmissione (TEM) per acquisire nuove conoscenze in come l'espressione di queste proteine ridisporre le membrane intracellulari (Figura 1). CLEM è stato eseguito con cellule chimicamente fissate nella maggior parte degli studi virologia pubblicati in data8,9,10,11,12,13,14 ,15,16,17,18,19. Questo è principalmente dovuto alla necessità di inattivare materiale infettivo per motivi di sicurezza biologica in biosicurezza livello-2 e -3 (BSL-2 e BSL-3) laboratori, dove cryo-immobilizzazione delle cellule non è solitamente possibile. Per quelle domande che richiedono la conservazione ottimale delle membrane cellulari, vetrificazione tramite alta pressione congelamento (HPF) è, tuttavia, altamente consigliata20. In questi casi, può essere applicato il protocollo di CLEM descritto qui. È interessante notare che, soprattutto quando si lavora con campioni infetti, HPF può essere effettuato su campioni che sono stati precedentemente inattivati chimicamente, ad esempio nei laboratori BSL-2 e BSL-3. La combinazione di fissazione chimica seguita da HPF è una possibilità di profitto almeno parzialmente i vantaggi della crioconservazione metodi21,22.
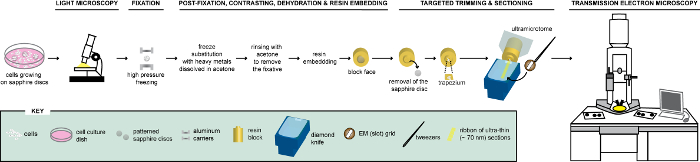
Figura 1 : Rappresentazione schematica del flusso di lavoro per l'analisi delle cellule via CLEM. Cellule che crescono su dischi zaffiro fantasia in primo luogo vengono analizzate da LM per localizzare le cellule che esprimono FPs prima della loro trasformazione per EM. Una volta individuato, cellule immediatamente corretti da HPF e FS per essere successivamente incorporato in resina. Al momento di polimerizzazione della resina, il supporto dove crescevano le cellule (= dischi zaffiro) devono essere rimossi dal blocco di resina. Il blocco contenente le cellule incorporate viene tagliato un piccolo trapezio da cui vengono sezionate le celle rimanenti, esprimendo FPs, con un coltello di diamante. Sezioni ultrasottili sono raccolti sulle griglie di slot e ulteriormente esaminati da TEM per ottenere informazioni ultrastrutturali di queste cellule. Questa figura è adattata e modificata con il permesso di riferimento29. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. preparazione di zaffiro fantasia dischi per colture cellulari
- Trasferire i dischi in zaffiro fantasia (Vedi Tabella materiali), con un modello alfanumerico inciso su una delle loro superfici, in un tubo di centrifugazione conico da 15 mL.
Nota: In alternativa, dischi zaffiro spessore mm 0,05 convenzionali senza modello alfanumerico possono essere utilizzati in cui viene creato un modello di riferimento di carbonio rivestimento li con una griglia di finder sulla parte superiore. A questo scopo, si dovrebbe pulire con etanolo prima il modello di carbonio viene applicato. - Lavarli accuratamente con etanolo. Loro diffusione fuori in una capsula di Petri con carta da filtro e lasciarli asciugare. Metterli su un vetrino separato da altro usando la pinzetta lunga sottile.
- Controllare con un microscopio invertito (ingrandimento 4x) se il pattern di coordinate è leggibile su ogni disco in zaffiro.
Nota: Se questo non è il caso, capovolgere i dischi zaffiro con l'aiuto delle pinzette. - Conservare i dischi in zaffiro in una capsula di Petri.
Nota: Il protocollo può essere messo in pausa qui.
2. cellula cultura con dischi in zaffiro fantasia
- Sterilizzare i dischi zaffiro fantasia con un reticolante ultravioletta (UV) per 5 min.
- Prendere i dischi in zaffiro per la biosicurezza di cultura cellulare. Sterilizzare le pinzette lunghe con etanolo nella cappa di biosicurezza.
- Aggiungere metà del terreno di coltura delle cellule (Vedi Tabella materiali) a una piastra di coltura delle cellule. Trasferire i dischi in zaffiro con attenzione con le pinzette lunghe nella piastra di coltura delle cellule.
- Semi 1 x 105 Huh7-Lunet le cellule, che esprimono la polimerasi T7 risospesa nell'altra metà del terreno di coltura cellulare, sulla piastra di coltura.
Nota: Calcolare il numero di cellule per essere seminato in modo tale che il punto finale dell'esperimento il confluency di cella dovrebbe essere non più di 20 – 30%. Densità cellulare alta ostacolerà la rilocazione delle cellule nelle fasi successive di EM. Inoltre, confluency di sopra potrebbe causare il distacco delle cellule dai dischi di zaffiro. - Controllare che i dischi di zaffiro rimangono nella parte inferiore della piastra di coltura e che le coordinate alfanumeriche sono ancora leggibili con il microscopio invertito.
Nota: Se il galleggiante di dischi nel medium di coltura delle cellule, utilizzare le pinzette lunghe per spingere i dischi verso il basso e alla fine di capovolgere indietro nell'orientamento corretto. - Transfect le cellule il giorno successivo come precedentemente segnalato in riferimenti21,23 seguendo le istruzioni del produttore dell'agente transfezione.
3. LM Imaging delle cellule che crescono su dischi zaffiro fantasia
- Trasferire i dischi in zaffiro al punto finale dell'esperimento su un supporto metallico di imaging contenente 30 – 50 µ l / posizione del medium senza indicatore di pH per ridurre la fluorescenza di fondo non specifici.
Nota: La piastra di metallo descritta qui (Figura 2) è stata progettata al workshop laboratorio europeo di biologia molecolare (EMBL). In assenza di questo piatto o uno simile, piastre di coltura cellulare con fondo di vetro potrebbero essere anche utilizzati invece per LM. È importante cambiare il mezzo di coltura cellulare normale per mezzo senza indicatore di pH. Si noti inoltre che i tempi di esposizione lunghi dovrebbero essere evitati perché potrebbero causare photobleaching, che conduce ad accumulazione di specie (ROS) reattive dell'ossigeno e danno fisiologico delle cellule24,25. - Le cellule con un microscopio a fluorescenza invertito widefield della battuta.
Nota: Quando si utilizza una proteina fluorescente verde (GFP)-proteina taggata, come mostrato nei risultati rappresentativi, 450-490 nm e filtri di eccitazione e di emissione di 500 – 550 nm, rispettivamente, deve essere utilizzata. - In primo luogo acquisire un'immagine di ingrandimento basso (10 – 20 X) delle cellule di allocare le cellule che esprimono FPs. Record le coordinate dei dischi zaffiro fantasia dove si trovano le celle di interesse mediante microscopia in contrasto (DIC) differenziale di interferenza.
Nota: Cuciture diverse aree potrebbe aiutare ad per avere una panoramica migliore della posizione della cella/celle di interesse. Nota che quello microscopia di contrasto di fase potrebbe essere eventualmente utilizzato anche qui. - Acquisire immagini ad alto ingrandimento (fluorescenza e DIC) (63 – 100 X, obiettivi di immersione in olio) delle stesse cellule/cellule di meglio discernere la localizzazione sottocellulare della proteina di interesse all'interno dello spazio intracellulare.
Nota: Immagini DIC forniscono informazioni contestuali. Questa informazione è spesso critica per correlare immagini LM con micrografie EM più tardi perché la correlazione finale è più precisa con "anatomici" della cella. Come alcuni dischi zaffiro potrebbero rompersi durante cryo-immobilizzazione, si consiglia di preparare triplici copie a condizione. Inoltre, alcune cellule si staccano dai dischi durante HPF e le fasi successive del presente protocollo, mentre l'ultrastruttura delle altre cellule potrebbe contenere manufatti in conseguenza di danni di congelamento. Pertanto, almeno due settori dei dischi dovrebbero essere imaged tramite LM per garantire che alla fine ci sarà abbastanza cellule per essere analizzati. Soprattutto, queste due aree dovrebbero essere sui lati opposti del disco. In questo modo il blocco di resina dove le cellule sono incorporate può essere tagliato in due metà e sezioni di queste aree possono essere ottenuti.

Figura 2 : Schema per l'imaging di LM di cellule che crescono fantasia dischi in zaffiro. (A) i dischi di zaffiro sono trasferiti con l'aiuto di pinzette bene una piastra di imaging. (B-C) Questo piatto è stato appositamente progettato al workshop EMBL di Heidelberg per adattarsi al microscopio ottico, dove i dischi zaffiro possono essere imaged con mezzo di coltura cellulare senza indicatore di pH. Si compone di una slitta in metallo, 75 x 25 mm x 1 mm, con quattro-cinque fori fresati in esso con una punta di 3 mm per adattarsi alle dimensioni dei dischi zaffiro. Inoltre, piccole scanalature sono stati fresati su ogni lato dei fori ad un angolo di 90°. Questi fori rendono più facile per accedere i dischi con il forcipe quando manipolarli. Per consentire condizioni ottimali di imaging vetrini coprioggetti n. 0 (0.085 a 0,13 mm di spessore) erano incollati sotto i fori con colla UV e posto sotto luce UV per indurire. Immagine ad alto ingrandimento (D) del disco zaffiro con motivi raffiguranti le coordinate alfanumeriche che sono incise sulla superficie. I dischi di zaffiro fantasia sono commercialmente disponibili (Vedi Tabella materiali). Clicca qui per visualizzare una versione più grande di questa figura.
4. Cryo-immobilizzazione di cellule che crescono su Zaffiro fantasia dischi tramite HPF
- Posizionare i vettori di alluminio "A" e "B" per HPF (Vedi Tabella materiali) in una capsula di Petri con carta da filtro imbevuta di 1-hexadecene. Immergere i dischi zaffiro fantasia contenente le cellule in una capsula di Petri con 1-hexadecene.
Nota: Spostare i dischi un po' per mondare il terreno di coltura cellulare. - Montare i dischi zaffiro tra una "A" e un elemento portante in alluminio "B" nel supporto HPF come segue (Figura 3B). Sul fondo, posizionare un elemento portante "B" con il lato piatto rivolto verso l'alto. Inserire i dischi di zaffiro su questo lato piatto con le cellule rivolto verso l'alto. Aggiungere un elemento portante di "A" con la sua profondità di 0,1 mm rivolto verso le celle.
Nota: Poiché i dischi di zaffiro fantasia sono più spessi che i dischi di zaffiro convenzionali, il vettore commercialmente disponibile "A" (progettato per essere utilizzato con dischi in zaffiro di 0.05 mm non-fantasia) doveva essere tagliato fino a 0,05 mm a questo lato di 0,2 mm profondità presso l'EMBL Workshop al fine di adattarsi nel supporto HPF (Figura 3A). A questo scopo, "Un" vettore viene inserito alla fine di un tubo di metallo affinché sua 0,2 mm di lato è esposto e rimane ancora mentre il taglio esso. Il taglio lato negativo è poi levigato affinché sia perfettamente piatto. In alternativa, un elemento portante di alluminio speciale può essere utilizzato in cima il disco in zaffiro fantasia (commercialmente disponibili, vedere Tabella materiali), essendo in questo caso l'HPF "sandwich" composto da solo il disco in zaffiro e questo vettore. - Chiudere il fermo della macchina HPF e congelare le cellule.
Nota: Questo protocollo è specifico per una determinata macchina di HPF. Altre macchine di HPF potrebbero essere usata in alternativa6,26. In ogni caso, si prega di essere consapevole dell'esistenza di macchine di nuova generazione da parte dei fornitori.
Attenzione: Usare occhiali e cuffie di protezione dell'udito. -
Posizionare il congelato "sandwich" in una scatola di polistirolo con azoto liquido per l'archiviazione temporanea.
Attenzione: Usare occhiali di protezione e mettersi in contatto con gli ufficiali di sicurezza locale per essere informato circa i potenziali pericoli durante la gestione dell'azoto liquido.- Per evitare confusione, posto ogni "sandwich" in un tubo del microcentrifuge separati 0,5 mL (dentro la scatola in polistirolo) contrassegnato con un numero.
Nota: Il protocollo può essere messo in pausa qui. Se freeze sostituzione (FS) non può essere eseguita immediatamente, i campioni possono essere conservati in cryo-tubi (con un foro perforato nella parte superiore) all'interno di scatole, che si svolgono in un rack in un azoto liquido criogenico dewar (Vedi Tabella materiali). Questo contenitore di azoto liquido dovrebbe trovarsi in una stanza con i sensori di gas ossigeno, per evitare il soffocamento in caso di una carenza di ossigeno.
Attenzione: Tutte le procedure descritte di seguito (punti 5 e 6) devono essere effettuate in una cappa di biosicurezza. Inoltre, la maggior parte dei reagenti utilizzati sono pericolosa. Prima del loro utilizzo, è obbligatorio leggere attentamente il Material Safety Data Sheets, fornite dai produttori, come pure di chiedere gli ufficiali di sicurezza circa le normative locali per garantire una manipolazione sicura. Per il corretto smaltimento di questi materiali utilizzati, nonché per l'uso corretto delle attrezzature descritte di seguito, è anche tenuto a consultare salute del locale Istituto e le procedure di sicurezza.
- Per evitare confusione, posto ogni "sandwich" in un tubo del microcentrifuge separati 0,5 mL (dentro la scatola in polistirolo) contrassegnato con un numero.
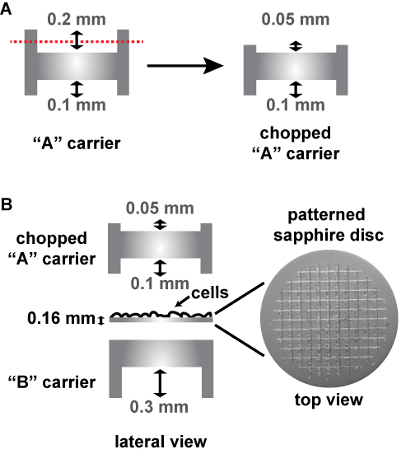
Figura 3 : Schema dell'Assemblea di zaffiro fantasia dischi tra due elementi portanti di alluminio per HPF. (A) Cut-away visualizzazioni di un vettore convenzionale "A", che deve essere tagliato il suo lato più profondo da 0,2 mm a 0,05 mm. (B) il disco in zaffiro spesse 0,16 mm con il modello alfanumerico inciso sulla superficie è assemblato in due elementi portanti di alluminio per HPF , come segue: il disco zaffiro è collocato sul lato piatto di un vettore "B" con le cellule rivolto verso l'alto. Un trito "Un" vettore con la sua profondità di 0,1 mm rivolto verso le cellule è messo in cima al vicino "HPF sandwich". Gli elementi portanti in alluminio e i dischi di zaffiro fantasia sono commercialmente disponibili (Vedi Tabella materiali). Clicca qui per visualizzare una versione più grande di questa figura.
5. FS di celle Cryo-fisse
- Riempire la macchina di FS (Vedi Tabella materiali) con azoto liquido. Impostare la temperatura a-90 ° C e attendere che la macchina raggiunga la temperatura.
- Preparare il supporto di FS contenente 0,2% tetrossido di osmio (OsO4) (v/v), 0,1% acetato di uranile (UA) (w/v) in acetone distillato di vetro mentre la macchina di FS si sta raffreddando.
Nota: Per preparare, (per esempio) 12 mL di terreno di FS, mescolare 600 µ l di 4% OsO4con 0,012 g di UA in un contenitore di vetro. Sciogliere questa miscela in un dispositivo di bagno ad ultrasuoni per 5 minuti aggiungere 11,4 mL di acetone distillato di vetro con una pipetta e mescolare bene. - Riempire le provette per microcentrifuga da 1,5 mL con 200-500 µ l di terreno di FS, chiudere il coperchio e trasferirli alla macchina FS. Aspettare 10 min per il mezzo di FS si raffreddi.
- Trasferire i congelati "panini" contenente i dischi zaffiro con cellule in azoto liquido con pinzette raffreddate lunghe per le provette contenenti il mezzo di FS.
Attenzione: Usare occhiali e guanti di cryo-protettivo.
Nota: L'HPF "panini" potrebbe essere smontato spontaneamente durante la loro movimentazione. In questo caso, assicurarsi che i dischi di zaffiri congelati vengono trasferiti nelle provette microcentrifuga. I vettori di alluminio possono essere scartati. - Eseguire il FS come segue fino al successivo giorno27: da-90 ° C a-80 ° C per 8 h; da-80 ° C a-50 ° C per 8 h; da-50 ° C a-20 ° C per 2 h; da-20 ° C a 0 ° C per 2 h.
6. resina incorporamento di Freeze sostituito campioni
Attenzione: Tutte le procedure descritte di seguito devono essere effettuate in una cappa di biosicurezza. Inoltre, la maggior parte dei reagenti utilizzati sono pericolosa. Prima del loro utilizzo, è obbligatorio leggere attentamente il Material Safety Data Sheets, fornite dai produttori, come pure di chiedere gli ufficiali di sicurezza circa le normative locali per garantire una manipolazione sicura. Per il corretto smaltimento di questi materiali utilizzati, nonché per l'uso corretto delle attrezzature descritte di seguito, è anche tenuto a consultare salute del locale Istituto e le procedure di sicurezza.
- Estrarre gli esemplari sostituiti dalla macchina FS e metterli sul ghiaccio per 20 min.
- Mantenere i campioni a temperatura ambiente per 20 min e lavarli accuratamente (3 volte, 10 min. ciascuno) con acetone distillato di vetro. Scartare i vettori di alluminio con le pinzette lunghe se sono ancora nei tubi del microcentrifuge dopo il FS.
- Si infiltrano le cellule (sui rimanenti dischi zaffiro) in una serie di resina epossidica quattro fasi utilizzando le incubazioni 1h in resina 25%, 50% e 75% in acetone distillato di vetro, seguita da incubazione overnight con 100% resina. Scambiare la resina 100% il giorno successivo per 1h.
- Con attenzione, inserire i dischi zaffiro flusso-attraverso gli anelli montati nei bagni di reagente riempiti con 100% resina. Questi anelli sono utilizzati come stampi di polimerizzazione per 10 dischi in zaffiro.
Nota: I dischi di zaffiro devono essere completamente spinto verso il basso con le coordinate verso l'alto in maniera leggibile. Ciò può essere verificato con l'aiuto di un binocolo collegato ad un aspiratore di fumi. In alternativa, potrebbe essere utilizzato un binocolo all'interno di una cappa di biosicurezza. Importante, ci sono i numeri (1-10) incisi sul fondo dei reagenti che aiutano a identificare i campioni. Inoltre, per l'identificazione dei campioni un tag di carta potrebbe essere incluse nel blocco della resina. - Polimerizzare i campioni in forno a 60 ° C per 48 h.
7. rimozione di dischi zaffiro fantasia dai blocchi di resina polimerizzata
- Rimuovere i blocchi di resina dura contenente le celle da forno.
Nota: Il protocollo può essere messo in pausa qui. Se non, lasciare raffreddare la blocchi fino a temperatura ambiente prima di tagliarli. - Tagliare l'incorporamento stampi con una lama di rasoio per avere accesso ai blocchi di resina. Rimuovere i blocchi di resina cilindrico con una pinzetta.
Nota: Se un tag di carta non è stato incluso resina bloccare prima della polimerizzazione, numero i blocchi con un pennarello indelebile e memorizzarli in provette per microcentrifuga da 1,5 mL. Il protocollo può essere messo in pausa qui. - Immergere la punta del blocco di resina che contiene il disco in zaffiro in una scatola di polistirolo contenenti azoto liquido finché non si arresta "spumeggiante". Quindi, immergere la punta del blocco di resina in ebollizione H2O.
- Ripetere i due passaggi precedenti come tante volte quanto necessario fino a quando il disco in zaffiro cade fuori del blocco di resina.
Attenzione: Usare occhiali e guanti di cryo-protettivo.
Nota: Se questo non funziona, è possibile utilizzare una lama di rasoio per rimuovere un po' di resina polimerizzata intorno ai dischi prima di riprovare. Il protocollo può essere messo in pausa qui.
8. mirato sgrossatura e il sezionamento ultrasottile per TEM
- Identificare l'area del viso di blocco di resina dove le celle di interesse sono presenti esaminando il volto di blocco con il binocolo di un ultramicrotomo.
Nota: L'impronta negativa del modello delle coordinate dai dischi zaffiro fantasia viene mantenuto sul viso di blocco. Ciò permette l'individuazione della zona dove le cellule di interesse si trovano per la rifilatura mirato. Le immagini di LM devono essere capovolta verticalmente per facilitare l'individuazione della posizione stessa del viso blocco. - Tagliare il monostrato cellulare incorporato contenente cellule di interesse per una piccola piramide piatta con la forma di un trapezio (~ 200 µm × 250 µm di media dimensione) con una lama di rasoio pulita.
- Ottenere 70 nm sezioni ultrasottili delle cellule incorporate con un coltello di diamante 35°.
- Inserire le sezioni sulle griglie di EM slot con l'aiuto di pinzette ultrafine.
Nota:: l'uso di mesh griglie dovrebbero essere evitati perché le sezioni potrebbero essere mascherate dalle barre della griglia. - Memorizzare le griglie in una casella della griglia.
Nota: Il protocollo può essere messo in pausa qui.
9. TEM analisi di sezioni ultrasottili
- Posizionare la griglia di slot nel supporto del microscopio elettronico a trasmissione. Acquisire immagini a basso ingrandimento (panoramica), dove è visibile il profilo di cellula completa della cellula di interesse.
Nota:: le cellule di interesse può essere trovate usando i profili di cella e le posizioni delle celle ad uno altro come riferimenti, confrontando le opinioni di DIC acquisita prima da LM. - Acquisire immagini ad alto ingrandimento della cella/celle di interesse, per cercare di trovare, a livello ultrastrutturale, caratteristiche che corrispondono ai segnali fluorescenti osservati in precedenza da LM.
Nota: Montage immagini possono essere ottenute a forte ingrandimento per avere una panoramica ad alta risoluzione di cella/celle di interesse. Inoltre, spesso è necessario acquisire immagini della stessa cella in sezioni consecutive seriale per poter ricostruire la cella in 3D prima di confrontare le immagini di fluorescenza.
Risultati
Utilizzando la procedura presentata qui (Figura 1), Huh7-Lunet cellule che esprimono stabilmente la T7 RNA polimerasi sono state seminate su dischi di zaffiri fantasia e successivamente transfected con un plasmide esprimente due domini del virus dell'epatite C (HCV) non strutturali della proteina NS5A, taggati con GFP (GFP-D1-pTM_AH) (Figura 4). Il giorno seguente, i dischi di zaffiro fantasia, dove le cellule crescevano, erano tolto i piatti di cultura cellulare ed imaged per mezzo di LM (Figura 4, Figura 2). Quelle cellule che esprimono la AH-D1-GFP sono stati identificati dalla presenza di fluorescenza verde nel citosol e registrati per salvare le loro posizioni all'interno del modello di coordinata dei dischi zaffiro (figure 5A, 5B). Immediatamente dopo il loro esame di LM, i dischi in zaffiro contenente le celle di interesse sono stati assemblati tra due elementi portanti di alluminio (Figura 3) e sottoposti a cryo-immobilizzazione di HPF. Le cellule sono state poi congelare sostituito e successivamente incorporati in resina epossidica.
Dopo la rimozione dei dischi zaffiro dai blocchi di resina polimerizzata, l'impronta del modello alfanumerico era visibile sulla faccia del blocco, che ha permesso di recuperare le aree dove si trovavano le celle di interesse. Il resto del blocco di resina è stato tagliato via per generare un piccolo trapezio che harboring le celle di interesse. Sezioni ultrasottili serie sono sono ottenute da questo trapezio, raccolti sulle griglie di EM slot e ulteriormente analizzate da TEM (Figura 5C). Nota che ottenere manualmente seriale sezioni consecutive anche se fattibile28 è laborioso e richiede personale ben addestrato e qualificato. È anche importante che le cellule hanno un basso confluency quando sottoposti a questo protocollo di CLEM (Figura 5A) al fine di garantire un rapido riconoscimento della stessa cella precedentemente identificato da LM. In caso contrario, avendo più alto confluency di cella potrebbe drammaticamente rallentare il successo individuazione delle cellule a livello di EM.
Analisi TEM di diverse sezioni ultrasottili della cella di interesse (figure 5, 5E), esprimendo l'AH-D1-GFP, ha rivelato la presenza nel suo spazio intracellulare di grandi accumulazioni di vescicole della morfologia variabile delimitato dalla che circonda il cytosol da un singolo strato lipidico (Figura 5E).
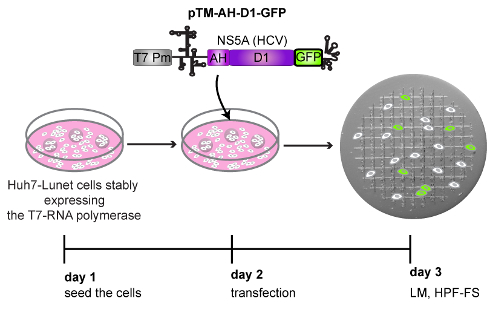
Figura 4 : Rappresentazione schematica del flusso di lavoro seguito per eseguire l'esempio di CLEM raffigurato Figura 5 . Huh7-Lunet cellule che esprimono stabilmente la T7 RNA polimerasi transfected con un plasmide DNA codifica l'elica amphipathic (AH) e il dominio 1 (D1) di proteina NS5A HCV, etichetta al suo C-terminale con GFP (pTM_AH-D1-GFP). L'espressione di questa parte della proteina NS5A è controllata trascrizionalmente da un promotore T7 (T7 Pm) e ribosomi da un elemento di IRES (luogo dell'entrata interna ribosoma) virus (EMCV) dell'encefalomiocardite, indicato da una struttura secondaria. Cellule di ventiquattro h post-transfezione (24 hpt) crescono modellato dischi in zaffiro ed esprimendo la AH-D1-GFP sono stati rilevati da LM e immediatamente sottoposti a HPF-FS. Clicca qui per visualizzare una versione più grande di questa figura.
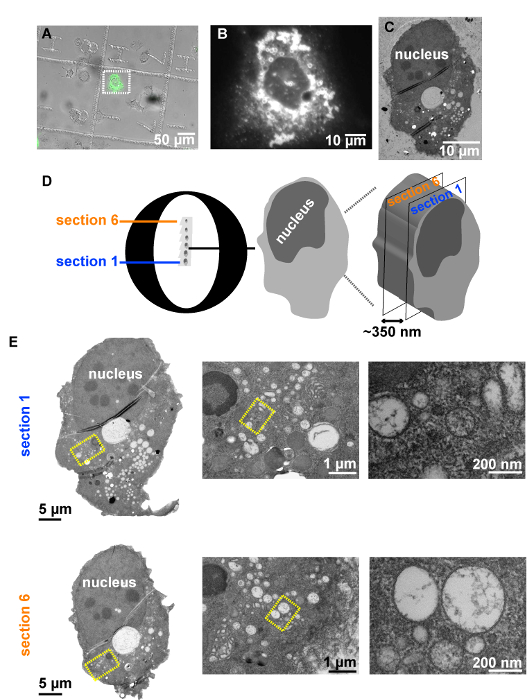
Figura 5 : Esempio di una routine di CLEM. (A) Huh7-Lunet cellule che esprimono stabilmente la polimerasi T7 analizzata da microscopia di fluorescenza per allocare cellule GFP-positive coltivate su dischi zaffiro fantasia, 24 ore dopo la trasfezione con il plasmide pTM_AH-D1-GFP. Immagine di ingrandimento superiore (B) di una singola cella selezionata all'interno del rettangolo tratteggiato bianco per essere analizzati da CLEM. (C) immagine TEM di una sezione ultrasottile della stessa cella dopo HPF, FS e l'incorporamento di resina. (D) rappresentazione schematica delle sezioni ultrasottili di serie 70-nm su una griglia di slot EM contenente la cella di interesse. (E) sulla sinistra: panoramiche TEM delle due sezioni della cellula di interesse. A destra: immagini TEM ad alto ingrandimento delle zone intracellulare selezionate in rettangoli gialli a sinistra, svelando un numero elevato di vescicole delimitate dal cytosol tramite una singola membrana. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
La metodologia di CLEM ha presentata qui a studiare l'impatto di un'espressione di proteina virale sulle membrane cellulari è stata utilizzata con successo prima per delucidare l'HCV associata replica strutture, principalmente doppia membrana vescicole (DMV)21, come pure per determinare i blocchi di edificio critici necessari per formare queste strutture indotta da HCV23. Si noti che nel nostro primo lavoro usando CLEM per studiare HCV replica21, una versione leggermente modificata del protocollo descritto qui è stata applicata. In quanto Studio, spessore dischi zaffiro convenzionale 0,05 mm, senza modello alfanumerico, sono stati utilizzati in cui un modello di riferimento è stato creato da carbonio rivestimento li con una griglia di finder sulla parte superiore (Vedi Tabella materiali). Questa versione del protocollo corrente può essere applicata alla fine, con il vantaggio che il "Un" vettore per HPF sono utilizzabili direttamente, senza la necessità di taglio basso come in Figura 3A. In alternativa, come indicato nel protocollo, una più spessa "Un" vettore può essere utilizzata (Vedi Tabella materiali) con dischi in zaffiro fantasia, senza la necessità di un elemento portante "B".
Interessante, questo protocollo può essere applicato non solo studiare campioni di BSL-1, come le cellule transfected con proteine virali come descritto qui e altrove19,23, ma anche di studiare le cellule infettate da virus. Anche se lavoro con agenti patogeni umani è solitamente limitato ai laboratori BSL-2 e BSL-3, in alcuni paesi è ancora possibile effettuare cryo-immobilizzazione in queste condizioni di biosicurezza. In quei BSL-2 e laboratori BSL-3 dove non è possibile, a causa di regolamenti locali o l'assenza di una macchina di HPF vetrificazione, cellule infettate da virus possono essere ancora preparate utilizzando questo metodo, se la fissazione chimica con aldeidi è effettuata in anticipo, vale a dire prima di lasciare le strutture BSL-2 o BSL-3. Inoltre, aldeidi devono essere raffreddato immediatamente dopo la fissazione per mantenere la fluorescenza, mentre il resto del protocollo è identico a quello descritto qui. Questa tecnica può essere considerata ridondante perché le cellule sono fissate due volte, chimicamente e tramite vetrificazione. Tuttavia, questo protocollo di fissazione doppia porta infatti a una molto migliore conservazione del DMV HCV-indotto in confronto il DMV trovata in cellule sottoposte a fissazione chimica solo21.
Per ulteriori modifiche e la risoluzione dei problemi il lettore si riferisce alle note in tutta la parte di protocollo di questo manoscritto. Queste note descrivono insidie da evitare, come pure delle alternative a superare presunte difficoltà che potrebbero sorgere durante l'esecuzione di questo metodo.
Il requisito principale per l'applicazione di questa tecnica è una macchina di HPF. Quando cryo-immobilizzare le cellule via HPF non è fattibile (a causa dell'assenza di una macchina di HPF) o non richiesto (quando la conservazione di membrana non deve necessariamente essere ottimale), le cellule possa essere chimicamente fisso e successivamente preparati e analizzati da EM8, 9,10,11,12,13,14,15,16,17,18 ,19. Questa opzione non richiede l'uso di dischi in zaffiro, ma piastre di coltura cellulare con i modelli di griglia per lo spostamento di celle o gruppi di cellule. Il vantaggio principale dell'uso di questi piatti è il loro diametro maggiore rispetto ai dischi in zaffiro, che permette la proiezione di grandi superfici. Così, l'applicazione del presente protocollo CLEM è stato applicato con successo per studiare l'effetto di un composto antivirale contro HCV15 o visualizzare le riorganizzazioni di membrana indotte dalle proteine non strutturali di norovirus19. Un altro problema che può limitare le prestazioni di questo metodo è l'assenza di un dispositivo commerciale di FS. In questo caso, sistemi di FS in casa base possono essere utilizzati invece. Anche se i dispositivi automatici di FS potrebbero ridurre la gestione degli incidenti, dispositivi fatti in casa vengono utilizzati con successo per esempio a laboratori di Paul Walther e30 di Kent McDonald .
Per quanto riguarda la fissazione chimica, il protocollo descritto qui garantisce una conservazione ottimale delle strutture intracellulari20. Pertanto, nel caso in cui i suddetti dispositivi HPF e FS sono disponibili, vetrificanti le celle di interesse sarebbe comodo.
Futuro delle alternative a questo approccio di CLEM includono la possibilità di utilizzare questo metodo non solo di acquisire informazioni 2D a livello ultrastrutturale, ma anche di acquisire informazioni 3D sull'architettura della membrana e dell'organello alterazioni causate da virus. I metodi di 3D-EM, compreso tomografia elettronica (ET) e microscopia analisi del fascio ionico focalizzato (FIB-SEM) (ampiamente descritta in29), potrebbero essere applicati anche alle cellule che sono state preparate seguendo questa corrente protocollo21, 31. Inoltre, informazioni 3D potrebbero essere ottenute anche a livello di LM, quando si utilizza un microscopio confocale, che permette l'acquisizione di z-stack. Infatti, questa opzione è altamente raccomandata quando una correlazione precisa tra DataSet LM ed EM è desiderato (Vedi ad esempio17). Le informazioni incluse negli stack di z 3D aiuta a migliorare la correlazione con le immagini TEM 2D. Così, in un tale scenario, il miglior montaggio immagini LM ed EM possa essere selezionato e poi sottoposti ad uno dei software disponibili, come il plugin di CE-CLEM di ICY (http://icy.bioimageanalysis.org/)32 o il plugin Landmark corrispondenze di correlazione Immagine di J (http://imagej.net/Landmark_Correspondences), con conseguente generazione di LM-EM immagini sovrapposte.
Quando l'informazione temporale è necessaria per comprendere la cinetica di un certo evento, time-lapse imaging consente di monitorare dinamiche delle cellule viventi in combinazione con EM. Durante l'evento di interesse, le cellule sono risolto immediatamente, generando una "istantanea bloccata" che possa essere successivamente analizzata con EM, fornendo informazioni dettagliate ultrastrutturali su quel particolare momento al momento dell'immobilizzazione. Al fine di ottenere quella "istantanea bloccata", dopo l'osservazione in tempo reale, le cellule possono essere chimicamente fisso33 o cryo-immobilizzata6. Poiché molti processi cellulari accadere più velocemente rispetto ai processi di diffusione di fissazione chimica, se possibile, congelamento ultra-rapido deve essere eseguita. Tuttavia, è importante tener conto che le macchine di HPF differiscono nella loro effettiva risoluzione34.
Inoltre, anche se questo protocollo è stato progettato per l'incorporamento delle cellule in una resina epossidica, le cellule possono essere anche incorporate in resine a bassa viscosità, come Lowicryls, LR bianco o oro LR. L'uso di questi mezzi incorporamento consente di preservare l'antigenicità35,36, come pure la fluorescenza37,38 e, pertanto, sono usati principalmente per Post-embedding su sezione immunolabeling39 , 40 e sulla sezione CLEM41,42,43,44, dove la LM è fatto dopo l'incorporamento. Entrambi gli approcci (immuno-EM e sulla sezione CLEM) devono essere fondamentali per quegli esperimenti in cui strutture non caratteristici si trovano facilmente via TEM e/o come controllo contro miscorrelation tra LM ed EM segnali. Allo stesso modo, etichettatura con gli anticorpi che possono essere visualizzati da entrambe modalità di imaging (LM ed EM) può essere effettuata45 al fine, ad esempio, identificare cellule transfected in LM (pre-embedding fase) ed ottenere una localizzazione molto più accurata della Segnale GFP via relativa etichettatura specifica di immuno-EM (fase post-embedding). Si deve tener conto, tuttavia, quel permeabilizzazione è condotta prima LM per consentire l'accesso degli anticorpi allo spazio intracellulare, che potrebbe comportare una sub-ottima conservazione dell'architettura delle cellule a livello di EM. Interessante, questo protocollo è anche adatto per esperimenti di multi-colore che può essere raggiunto con l'uso di altri tag fluorescente, diverso da GFP (come illustrato di seguito). In conclusione, ci sono molte possibilità putativo di adattare questo protocollo, il LM e/o sui lati EM, a seconda delle domande che vengono affrontate. Per una descrizione completa di altri protocolli alternativi si rinvia a5,46. Indipendentemente da come vengono combinate le modalità di microscopia, insieme il risultato è un guadagno di informazioni, che ci permette di capire meglio come i virus e le loro proteine interagiscono con i loro ospiti nella vita reale.
Il punto più critico all'interno di questo metodo è la raccolta di sezioni di serie dalle cellule di interesse. Come evidenziato nella sezione risultati rappresentativi, ciò richiede personale esperto, come pure un sacco di pazienza. Cosa importante, questo passaggio è essenziale per trovare le celle indietro a livello di EM per due motivi. In primo luogo, in questa sorta di protocollo di CLEM utilizzando pre-embedding LM, le coordinate sono visibili solo a livello di LM e sul viso di blocco di resina dopo l'incorporamento. Tuttavia, essi non sarà visibile sulle sezioni di TEM. Pertanto, rifilatura mirato sul fronte di blocco verso il basso per le regioni di interesse (ROI) deve essere compiuta con attenzione con una lama di rasoio per assicurare che le sezioni che vengono successivamente ottenute contengono le cellule che esprimono un dato FP. In secondo luogo, la "scansione" diverse sezioni è necessaria trovare la migliore sovrapposizione tra la LM e le acquisizioni di EM. Contrariamente ai metodi dove LM viene eseguita nella fase post-embedding, in questo caso la sovrapposizione di LM-EM non è così precisa. La precisione di sovrapposizione basso è a causa di differenze nella risoluzione assiale LM ed EM, ritiro durante l'elaborazione del campione di EM e la compressione durante il sezionamento42. Tuttavia, i metodi di monitoraggio efficiente, come l'uso di punti di riferimento, aiutano a trovare le cellule indietro. Questo include la posizione di una cella a altra, così come la forma delle cellule e del loro nucleo. A questo proposito, come spiegato nel protocollo, DIC immagini forniscono informazioni "anatomiche" delle cellule che sono fondamentali per migliorare la correlazione. In alternativa, i nuclei o altri organelli cellulari ben riconoscibile (ad esempio mitocondri o lipidica goccioline) possono essere macchiato prima LM e utilizzati come punti di riferimento.
Infine, è degno di menzione che sebbene questo manoscritto è incentrato sull'uso di questa tecnica per studi di virologia, la portata di questo disegno sperimentale può essere ampliata per affrontare questioni biologiche più generali.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Siamo molto grati ai membri del personale di microscopia elettronica Core Facilities (FECOM) all'EMBL (Heidelberg) e presso l'Università di Heidelberg. Inoltre vorremmo ringraziare Ulrike Herian, Stephanie Kallis e Andrea Hellwig (Università di Heidelberg), così come Eberhardt Schmidt e Renate Kunz (Università di Ulm) per assistenza tecnica. Lavoro di R.B. e il suo team (U.H. e I.R.-B.) è stato sostenuto dal Deutsche Forschungsgemeinschaft, SFB1129, TRR83, TP13 e TP11.
Materiali
| Name | Company | Catalog Number | Comments |
| UV Crosslinker | Stratagene | 400072 | Stratalinker 1800, 230 Vac, equipped with 254-nm UV light bulbs, 8 Watts/each. |
| Patterned sapphire discs | Engineering Office M. Wohlwend, Sennwald, Switzerland | 627 | They have a 0.3 mm diameter and are 0.16 mm thick, as well as an an etched alphanumeric pattern to allow for re-location of the cells. They can be only ordered per e-mail: martin-wohlwend@bluewin.ch. |
| Slim and long tweezers | Electron Microscopy Sciences, Hatfield, PA, USA | 72919-SS | "Style SS", for handling sapphire discs. |
| Cell culture medium | Thermo Fisher Scientific, Waltham, MA, USA | 41965-062 | Dulbecco's modified Eagle medium (DMEM) supplemented with 2 mM L-glutamine, nonessential amino acids, 100 units penicillin per ml, 100 µg streptomycin per ml and 10% fetal calf serum (DMEM complete, see below). Package containing 10 bottles, 500 mL/each. |
| L-glutamine | Thermo Fisher Scientific, Waltham, MA, USA | 25030-123 | Package containing 20 bottles of 100 mL/each. |
| Nonessential amino acids | Thermo Fisher Scientific, Waltham, MA, USA | 11140-068 | Package containing 20 bottles of 100 mL/each. |
| Penicillin/Streptomycin | Thermo Fisher Scientific, Waltham, MA, USA | 15140-163 | Package containing 20 bottles of 100 mL/each. |
| Fetal calf serum | Sigma Aldrich, St. Louis, MI, USA | F7524 | Bottle of 50 mL |
| Inverted microscope | Olympus Deutschland, Hamburg, Germany | CKX41 | It is an inverted microscope with trinocular head options and fluorescence upgrade capability. |
| Huh7-Lunet cells stably expressing the T7 RNA-polymerase | Avaliable at Prof. Dr. Ralf Bartenschlager laboratory | Not available | Contact Prof. Bartenschlager at: ralf.bartenschlager@med.uni-heidelberg.de. |
| Mirus TransIT-LT1 Transfection Reagent | Mirus Bio LLC, Madison, USA | MIR 2304 | Broad spectrum, low toxicity, DNA transfection reagent. Vial of 0.4 mL |
| Inverted widefield fluorescence microscope | Carl Zeiss Microscopy GmbH, Germany | Zeiss Observer.Z1 | Inverted fluorescence microscope for experiments involving living cell cultures. |
| 1-hexadecene | Merck, Darmstadt, Germany | 8220640100 | Bottle of 100 mL |
| "A" aluminium holders for high pressure frezing (HPF) | Engineering Office M. Wohlwend, Sennwald, Switzerland | 241 | This is the holder that has been used for this protocol and has 2 different depths of 0.1 and 0.2 mm. It can be only ordered per e-mail: martin-wohlwend@bluewin.ch. |
| "B" aluminium holders for HPF | Engineering Office M. Wohlwend, Sennwald, Switzerland | 242 | This is the holder that has been used for this protocol.It is 0.3 mm thick.They can be only ordered per e-mail: martin-wohlwend@bluewin.ch. |
| "A" aluminium holder for HPF (special for working with patterned sapphire discs) | Engineering Office M. Wohlwend, Sennwald, Switzerland | 737 | This is the holder than can be used alone with the patterned sapphire disc (as an alternative to the current protocol), without the need of a "B" holder or the edition of the "A" holder. It is 0.84 mm thick. It can be only ordered per e-mail: martin-wohlwend@bluewin.ch. |
| Sapphire discs | Engineering Office M. Wohlwend, Sennwald, Switzerland | 405 | These sapphires discs are the "conventional" one, with a 0.3 mm diameter and a thickness of 0.05 mm . They can be also used for CLEM as an alternative instead of patterned sapphire discs. |
| Finder grids | Electron Microscopy Sciences, Hatfield, Philadelphia, USA | LF135-Cu | These 135 mesh grids are used to create an alphanumeric pattern on "conventional" sapphire discs (described above) via carbon coating, so that they can be used for CLEM. 100 grids/vial. |
| HPF machine | ABRA Fluid AG, Widnau, Switzerland | HPM 010 | This HPF machine has been used for this protocol. |
| HPF machine | Leica Microsystems, Vienna, Austria | EMPACT 2 | "EMPACT 2", as an alternative to the use of the HPM machine that it has been used in this protocol (described above). |
| Cryo-tubes | Thermo Fisher Scientific, Waltham, MA, USA | Z763667-500EA | For long term-storage of cryo-immobilized samples in liquid nitrogen. Package containing 500 cryovials. |
| Liquid nitrogen dewar | Cole-Parmer GmbH, Wertheim, Germany | GZ-05094-60 | For long term-storage of cryo-immobilized samples in liquid nitrogen, equipped with cryo-racks with a capacity of 1600 cryo-tubes. |
| Automatic freeze substitution (AFS) machine | Leica Microsystems, Vienna, Austria | EM AFS2 | This machine performs freeze substitution and progressive lowering of temperature (PLT) techniques and allows low temperature embedding and polymerization of resins. |
| Osmium tetroxide (OsO4) | Electron Microscopy Sciences, Hatfield, PA, USA | 19150 | 4% aqueous solution, one box containing 10 x 2 mL ampules. |
| Uranyl-Acetate (UA) | Serva, Heidelberg, Germany | 77879.01 | Bottle containing 25 grs of (UA)-2 H2O. |
| Sonicator | Bandelin Electronic, Berlin, Germany | 329 | "Sonorex Super RK 31" is a high-power ultrasonic cleaning bath for aqueous cleaning solution that is used in this protocol to mix OsO4 and Ua when preparing the FS medium. |
| Glass-distilled acetone | Electron Microscopy Sciences, Hatfield, PA, USA | 10015 | Bottle of 100 mL |
| Stereomicroscope | Leica Microsystems, Vienna, Austria | Leica M80 | Routine stereomicroscope for daily inspections, equipped with a camera for capturing images. |
| Epoxy resin | Electron Microscopy Sciences, Hatfield, PA, USA | 14120 | Embed 812 is a kit containing several components that must be mix together in the proportions given by the manufacturers. |
| Flow through rings | Leica Microsystems, Vienna, Austria | 16707157 | Package containing 100 pieces. |
| Reagent baths | Leica Microsystems, Vienna, Austria | 16707154 | Package containing 100 pieces. |
| Ultramicrotome | Leica Microsystems, Vienna, Austria | Leica EM UC7 | Ultramicrotome for preparation of semi- and ultrathin sections at room temperature. |
| Ultra 35 ° diamond knife | Diatome Ltd., Nidau Switzerland | AGG339-735 | This knife have an edge length of 3 mm. |
| Ultra fine tweezers | Electron Microscopy Sciences, Hatfield, PA, USA | E78318 | "Style 3X", for handling EM grids. |
| EM slot grids | Electron Microscopy Sciences, Hatfield, PA, USA | G2010-Cu | 100 grids/vial. |
| EM grid storage box | Electron Microscopy Sciences, Hatfield, PA, USA | 71155 | It has capacity for 100 grids. |
Riferimenti
- Morgan, C., Godman, G. C., Breitenfeld, P. M., Rose, H. M. A correlative study by electron and light microscopy of the development of type 5 adenovirus. I. Electron microscopy. Journal of Experimental Medicine. 112, 373-382 (1960).
- Godman, G. C., Morgan, C., Breitenfeld, P. M., Rose, H. M. A correlative study by electron and light microscopy of the development of type 5 adenovirus. II. Light microscopy. Journal of Experimental Medicine. 112, 383-402 (1960).
- Caplan, J., Niethammer, M., Taylor, R. M., Czymmek, K. J. The power of correlative microscopy: multi-modal, multi-scale, multi-dimensional. Current Opinion in Structural Biology. 21, 686-693 (2011).
- de Boer, P., Hoogenboom, J. P., Giepmans, B. N. Correlated light and electron microscopy: ultrastructure lights up. Nature Methods. 12, 503-513 (2015).
- Müller-Reichert, T., Verkade, P. Correlative light and electron microscopy III, First edition. , Elsevier/Academic Press. Cambridge, MA. (2017).
- Brown, E., Mantell, J., Carter, D., Tilly, G., Verkade, P. Studying intracellular transport using high-pressure freezing and Correlative Light Electron Microscopy. Seminars in Cell and Developmental Biology. 20, 910-919 (2009).
- Bykov, Y. S., Cortese, M., Briggs, J. A., Bartenschlager, R. Correlative light and electron microscopy methods for the study of virus-cell interactions. FEBS Letters. , (2016).
- Spuul, P., et al. Assembly of alphavirus replication complexes from RNA and protein components in a novel trans-replication system in mammalian cells. Journal of Virology. 85, 4739-4751 (2011).
- Nagel, C. H., Dohner, K., Binz, A., Bauerfeind, R., Sodeik, B. Improper tagging of the non-essential small capsid protein VP26 impairs nuclear capsid egress of herpes simplex virus. PLoS One. 7, 44177(2012).
- Sharma, M., Kamil, J. P., Coughlin, M., Reim, N. I., Coen, D. M. Human cytomegalovirus UL50 and UL53 recruit viral protein kinase UL97, not protein kinase C, for disruption of nuclear lamina and nuclear egress in infected cells. Journal of Virology. 88, 249-262 (2014).
- Kallio, K., et al. Template RNA length determines the size of replication complex spherules for Semliki Forest virus. Journal of Virology. 87, 9125-9134 (2013).
- Martinez, M. G., Snapp, E. L., Perumal, G. S., Macaluso, F. P., Kielian, M. Imaging the alphavirus exit pathway. Journal of Virology. 88, 6922-6933 (2014).
- Lebrun, M., et al. Varicella-zoster virus induces the formation of dynamic nuclear capsid aggregates. Virology. 454-455, 311-327 (2014).
- Madela, K., et al. A simple procedure to analyze positions of interest in infectious cell cultures by correlative light and electron microscopy. Methods in Cell Biology. 124, 93-110 (2014).
- Berger, C., et al. Daclatasvir-like inhibitors of NS5A block early biogenesis of hepatitis C virus-induced membranous replication factories, independent of RNA replication. Gastroenterology. 147, 1094-1105 (2014).
- van der Schaar, H. M., et al. Illuminating the Sites of Enterovirus Replication in Living Cells by Using a Split-GFP-Tagged Viral Protein. mSphere. 1, (2016).
- Vale-Costa, S., et al. Influenza A virus ribonucleoproteins modulate host recycling by competing with Rab11 effectors. Journal of Cell Science. 129, 1697-1710 (2016).
- Wang, L., et al. Visualization of HIV T Cell Virological Synapses and Virus-Containing Compartments by Three-Dimensional Correlative Light and Electron Microscopy. Journal of Virology. 91, (2017).
- Doerflinger, S. Y., et al. Membrane alterations induced by nonstructural proteins of human norovirus. PLOS Pathogens. 13, 1006705(2017).
- Dahl, R., Staehelin, L. A. High-pressure freezing for the preservation of biological structure: theory and practice. Journal of Electron Microscopy Technique. 13, 165-174 (1989).
- Romero-Brey, I., et al. Three-dimensional architecture and biogenesis of membrane structures associated with hepatitis C virus replication. PLOS Pathogens. 8, 1003056(2012).
- Sosinsky, G. E., et al. The combination of chemical fixation procedures with high pressure freezing and freeze substitution preserves highly labile tissue ultrastructure for electron tomography applications. Journal of Structural Biology. 161, 359-371 (2008).
- Romero-Brey, I., et al. NS5A Domain 1 and Polyprotein Cleavage Kinetics Are Critical for Induction of Double-Membrane Vesicles Associated with Hepatitis C Virus Replication. MBio. 6, 00759(2015).
- Dixit, R., Cyr, R. Cell damage and reactive oxygen species production induced by fluorescence microscopy: effect on mitosis and guidelines for non-invasive fluorescence microscopy. The Plant Journal. 36, 280-290 (2003).
- Jou, M. J., Jou, S. B., Guo, M. J., Wu, H. Y., Peng, T. I. Mitochondrial reactive oxygen species generation and calcium increase induced by visible light in astrocytes. Annals of the New York Academy of Sciences. 1011, 45-56 (2004).
- McDonald, K. L., Morphew, M., Verkade, P., Muller-Reichert, T. Recent advances in high-pressure freezing: equipment- and specimen-loading methods. Methods in Molecular Biology. 369, 143-173 (2007).
- Walther, P., Ziegler, A. Freeze substitution of high-pressure frozen samples: the visibility of biological membranes is improved when the substitution medium contains water. Journal of Microscopy. 208, 3-10 (2002).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314, 1-340 (1986).
- Romero-Brey, I., Bartenschlager, R. Viral Infection at High Magnification: 3D Electron Microscopy Methods to Analyze the Architecture of Infected Cells. Viruses. 7, 6316-6345 (2015).
- McDonald, K. L., Webb, R. I. Freeze substitution in 3 hours or less. Journal of Microscopy. 243, 227-233 (2011).
- Villinger, C., Neusser, G., Kranz, C., Walther, P., Mertens, T. 3D Analysis of HCMV Induced-Nuclear Membrane Structures by FIB/SEM Tomography: Insight into an Unprecedented Membrane Morphology. Viruses. 7, 5686-5704 (2015).
- Paul-Gilloteaux, P., et al. eC-CLEM: flexible multidimensional registration software for correlative microscopies. Nature Methods. 14, 102-103 (2017).
- Polishchuk, R. S., Mironov, A. A. Correlative video light/electron microscopy. Current Protocols in Cell Biology Supplement. , Chapter 4, Unit 4 8(2001).
- McDonald, K. L. A review of high-pressure freezing preparation techniques for correlative light and electron microscopy of the same cells and tissues. Journal of Microscopy. 235, 273-281 (2009).
- Newman, G. R., Jasani, B., Williams, E. D. A simple post-embedding system for the rapid demonstration of tissue antigens under the electron microscope. The Histochemical Journal. 15, 543-555 (1983).
- Schwarz, H., Humbel, B. M. Influence of fixatives and embedding media on immunolabelling of freeze-substituted cells. Scanning Microscopy. 3, Supplement. Discussion 63-54 57-63 (1989).
- Luby-Phelps, K., Ning, G., Fogerty, J., Besharse, J. C. Visualization of identified GFP-expressing cells by light and electron microscopy. Journal of Histochemistry and Cytochemistry. 51, 271-274 (2003).
- Nixon, S. J., et al. A single method for cryofixation and correlative light, electron microscopy and tomography of zebrafish embryos. Traffic. 10, 131-136 (2009).
- McDonald, K. L. Rapid embedding methods into epoxy and LR White resins for morphological and immunological analysis of cryofixed biological specimens. Microscopy and Microanalysis. 20, 152-163 (2014).
- Webster, P., Schwarz, H., Griffiths, G. Preparation of cells and tissues for immuno EM. Methods in Cell Biology. 88, 45-58 (2008).
- Kukulski, W., et al. Correlated fluorescence and 3D electron microscopy with high sensitivity and spatial precision. Journal of Cell Biology. 192, 111-119 (2011).
- Peddie, C. J., et al. Correlative and integrated light and electron microscopy of in-resin GFP fluorescence, used to localise diacylglycerol in mammalian cells. Ultramicroscopy. 143, 3-14 (2014).
- Hampoelz, B., et al. Pre-assembled Nuclear Pores Insert into the Nuclear Envelope during Early Development. Cell. 166, 664-678 (2016).
- Lemercier, N., et al. Microtome-integrated microscope system for high sensitivity tracking of in-resin fluorescence in blocks and ultrathin sections for correlative microscopy. Scientific Reports. 7, 13583(2017).
- Takizawa, T., Powell, R. D., Hainfeld, J. F., Robinson, J. M. FluoroNanogold: an important probe for correlative microscopy. Journal of Biological Chemistry. 8, 129-142 (2015).
- Romero-Brey, I. 3D electron microscopy (EM) and correlative light electron microscopy (CLEM) methods to study virus-host interactions. Methods in Molecular Biology: Influenza Virus Methods & Protocols. Yamauchi, Y. , Springer. (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon