Method Article
Microscopia fotoacustica ultravioletta ad alta velocità per l'imaging istologico con colorazione virtuale assistita da Deep Learning
In questo articolo
Riepilogo
Viene dimostrato un microscopio fotoacustico ultravioletto ad alta velocità e open-top in grado di fornire immagini istologiche intraoperatorie per l'analisi del margine chirurgico, compresa la configurazione del sistema, l'allineamento ottico, la preparazione del campione e le procedure sperimentali.
Abstract
L'analisi del margine chirurgico (SMA), una procedura essenziale per confermare la completa escissione del tessuto canceroso nella chirurgia di resezione tumorale, richiede strumenti diagnostici intraoperatori per evitare ripetuti interventi chirurgici a causa di un margine chirurgico positivo. Recentemente, sfruttando l'elevato assorbimento ottico intrinseco di DNA/RNA a 266 nm di lunghezza d'onda, è stata sviluppata la microscopia fotoacustica ultravioletta (UV-PAM) per fornire immagini istologiche ad alta risoluzione senza etichettatura, mostrando grandi promesse come strumento intraoperatorio per la SMA. Per consentire lo sviluppo di UV-PAM per SMA, qui viene presentato un sistema UV-PAM ad alta velocità e open-top, che può essere utilizzato in modo simile alle microscopie ottiche convenzionali. Il sistema UV-PAM offre un'elevata risoluzione laterale di 1,2 μm e un'elevata velocità di imaging di 55 kHz A-line rate con scansione a specchio galvanometrica a un asse. Inoltre, per garantire che le immagini UV-PAM possano essere facilmente interpretate dai patologi senza ulteriore formazione, le immagini UV-PAM originali in scala di grigi sono praticamente macchiate da un algoritmo di apprendimento profondo per imitare le immagini standard macchiate di ematossilina ed eosina, consentendo un'analisi istologica senza allenamento. L'imaging della fetta di cervello del topo viene eseguito per dimostrare le elevate prestazioni del sistema UV-PAM open-top, illustrando il suo grande potenziale per le applicazioni SMA.
Introduzione
L'analisi del margine chirurgico (SMA), che richiede un esame dei campioni di tessuto al microscopio, è una procedura essenziale per determinare se tutte le cellule tumorali vengono rimosse dal corpo di un paziente in un intervento chirurgico di resezione1. Pertanto, un microscopio in grado di fornire rapidamente immagini istologiche è di vitale importanza per la SMA per evitare ripetuti interventi chirurgici causati da una rimozione incompleta delle cellule tumorali. Tuttavia, secondo l'attuale metodo gold standard basato sulla microscopia ottica a campo luminoso, il tessuto asportato deve essere fissato in formalina, incorporato nella paraffina, sezionato in fette sottili (4-7 μm) e quindi colorato da ematossilina ed eosina (H & E) prima dell'imaging, che richiede tempo (3-7 giorni) e laborioso 2,3 . Una sezione congelata è un'alternativa rapida per la SMA congelando, affettando e macchiando rapidamente il tessuto, che può fornire immagini istologiche in 20-30 minuti4. Tuttavia, le caratteristiche istologiche sono spesso distorte e richiedono una formazione abile, il che ostacola l'applicabilità della tecnica a più tipi di organi5.
Per la SMA sono state sviluppate tecniche di microscopia ottica in grado di fornire immagini cellulari senza o con pochi passaggi di elaborazione tissutale. Tuttavia, ognuno di loro soffre di problemi diversi. Ad esempio, la tomografia a coerenza ottica6 e la microscopia a riflettanza confocale7 soffrono di bassa specificità a causa del loro basso contrasto intrinseco di scattering. Sebbene la microscopia con eccitazione superficiale ultravioletta8 e microscopia a foglio luminoso9 possa fornire immagini ad alta risoluzione e ad alto contrasto per la SMA, la procedura di colorazione tossica e volatile di solito non può essere eseguita in una sala operatoria, il che prolunga i tempi di consegna. La microscopia multi-fotone10 e la microscopia Raman stimolata11 possono fornire informazioni dettagliate per la SMA. Tuttavia, l'alto costo dei laser ultraveloci richiesti che vengono utilizzati per generare effetti non lineari impedisce la loro ampia applicabilità.
Recentemente, sfruttando l'assorbimento ottico intrinseco, è stata sviluppata la microscopia fotoacustica ultravioletta senza etichetta (UV-PAM) per fornire immagini istologiche ad alta risoluzione12. In UV-PAM, l'energia fotonica della luce UV di eccitazione (ad esempio, 266 nm) viene prima assorbita dal DNA / RNA nei nuclei cellulari13 e quindi convertita in calore, inducendo l'emissione di onde acustiche attraverso l'espansione termico-elastica14. Rilevando le onde acustiche generate, è possibile ottenere immagini UV-PAM bidimensionali (2D) dei nuclei cellulari tramite la proiezione di ampiezza massima dei segnali acustici, fornendo informazioni istologiche per SMA. Per consentire le applicazioni cliniche di UV-PAM, è stato sviluppato un UV-PAM ad alta velocità basato sulla scansione a specchio galvanometrica per fornire immagini istologiche per un campione di biopsia cerebrale (5 mm x 5 mm) entro 18 minuti, mostrando un grande potenziale nelle applicazioni sensibili al tempo15. Per convalidare ulteriormente la possibilità di UV-PAM per l'imaging di tessuti spessi, è stato proposto un sistema UV-PAM in modalità riflessione con uno scanner impermeabile di sistemi microelettromeccanici a un asse, dimostrando con successo l'esame istopatologico intraoperatorio del colon umano e dei tessuti epatici16. Poiché l'immagine UV-PAM originale è in scala di grigi mentre l'immagine macchiata di H&E gold standard è nei colori rosa e viola, è difficile per i patologi interpretare direttamente le immagini UV-PAM. Per risolvere questo problema, è stato proposto un algoritmo di deep learning per trasferire immagini UV-PAM in scala di grigi in immagini virtuali macchiate di H & E in tempo quasi reale in modo che i patologi possano comprendere le immagini senza alcuna formazione aggiuntiva17.
Questo lavoro riporta un sistema UV-PAM ad alta velocità e open-top che può essere utilizzato in modo simile alle microscopie ottiche convenzionali, fornendo sia immagini istologiche in scala di grigi originali che immagini virtualmente macchiate assistite da un algoritmo di apprendimento profondo. Una fetta di cervello di topo fissata in formalina e incorporata in paraffina (FFPE) viene ripresa dal sistema UV-PAM per dimostrare la somiglianza tra le nostre immagini UV-PAM virtualmente macchiate e le immagini standard macchiate di H & E, mostrando il suo potenziale per le applicazioni SMA.
Protocollo
Tutti gli esperimenti sugli animali eseguiti in questo lavoro sono approvati dal Comitato etico animale presso l'Università della Scienza e della Tecnologia di Hong Kong.
1. Sistema UV-PAM open-top (Figura 1)
- Illuminazione ottica
- Utilizzare un laser a stato solido pompato a diodi Q-switch (lunghezza d'onda di 266 nm) come sorgente di eccitazione.
- Utilizzare due lenti convesse per espandere il raggio laser e posizionare un foro stenopeico vicino al punto focale della prima lente convessa per eseguire il filtraggio spaziale.
- Riflettere il raggio laser verso l'alto utilizzando uno specchio galvanometrico 1D (1D GM).
- Utilizzare una lente obiettiva per mettere a fuoco il raggio laser e passare attraverso il centro di un trasduttore ad ultrasuoni a forma di anello (UT) prima di essere strettamente focalizzato su un campione.
- Rilevamento ad ultrasuoni
- Utilizzare un UT focalizzato a forma di anello per rilevare le onde ultrasoniche. I diametri interno ed esterno dell'area attiva UT sono rispettivamente di 3 mm e 6 mm. Lunghezza focale: 6,3 mm; frequenza centrale: 40 MHz; -6 dB larghezza di banda: 84%.
- Fissare l'UT in un serbatoio dell'acqua realizzato in laboratorio con una finestra otticamente trasparente nella parte inferiore, coperta da un sottile coperchio di quarzo per consentire il passaggio della luce UV. Assicurarsi che l'area attiva dell'UT sia rivolta verso l'alto. Collegare il serbatoio dell'acqua a uno stadio manuale a due assi per controllare la posizione laterale dell'UT.
- Accendere il laser UV, regolare la posizione dell'UT per consentire al raggio laser di passare attraverso dal centro dell'UT. Spegnere il laser UV. Quindi, riempire il serbatoio dell'acqua con acqua deionizzata per immergere completamente l'UT.
- Collegare l'uscita dell'UT a due amplificatori (guadagno totale = 56 dB) e collegare l'uscita del secondo amplificatore a una scheda di acquisizione dati (DAQ).
- Collegare un portacampioni a uno stadio manuale dell'asse z collegato a stadi motorizzati xy. Il portacampioni ha un foro vuoto che consente il passaggio della luce UV. Attaccare quattro pezzi di nastro biadesivo che circondano il foro.
- Allineamento del sistema
- Attaccare del nastro nero a una diapositiva di vetro e posizionare la diapositiva di vetro per coprire il foro del portacampioni, con il nastro nero rivolto verso il basso. Premere il vetrino per assicurarsi che sia fissato sul supporto del campione. Quindi, abbassare il portacampioni per immergere il vetrino nell'acqua.
- Scollegare l'UT a forma di anello e gli amplificatori, collegare l'UT all'pulsatore/ricevitore e collegare l'uscita dell'impulso/ricevitore a un oscilloscopio. Azionare il pulsatore/ricevitore in modalità Pulse-Echo. Impostare l'ampiezza dell'impulso e il guadagno dell'pulsatore/ricevitore rispettivamente a 6 e 20 dB.
- Abilitare il pulsatore/ricevitore e regolare la posizione z del portacampioni per trovare la posizione del piano focale acustico in cui i segnali ultrasonici sono massimi.
- Cambiare il pulsatore/ricevitore in modalità di trasmissione e impostare il guadagno su 60 dB. Abilitare l'uscita laser. Regolare la posizione z dell'obiettivo per massimizzare i segnali PA misurati dall'oscilloscopio.
- Regolare la posizione laterale dell'UT a forma di anello per rendere il segnale PA generato simmetrico e massimo, il che rappresenta che i fuochi ottici e acustici sono coalizzati nel piano laterale. Quindi, regolare la posizione z del portacampioni per massimizzare i segnali PA.
- Ripetere i passaggi 1.4.3 e 1.4.4 per ottimizzare sia la simmetria che l'ampiezza dei segnali PA. Registra il ritardo temporale (cioè il tempo necessario alle onde PA per raggiungere l'UT) sull'oscilloscopio quando i segnali PA sono ottimizzati.
- Spostare il supporto del campione in diverse posizioni del nastro nero. Regolare la planarità del portacampioni in modo che i segnali PA generati da ciascuna posizione del nastro nero abbiano lo stesso ritardo temporale di quello misurato al punto 1.4.5.
- Spegnere il laser e collegare l'UT ai due amplificatori.
2. Preparazione del campione
- Fetta di cervello di topo FFPE
- Sacrificare un topo con un sovradosaggio di anestesia. Quindi, raccogliere il cervello del topo seguendo il protocollo descritto nel riferimento18.
- Fissare il cervello raccolto in formalina tamponata neutra al 10% per 24 ore.
- Elaborare il cervello fisso per disidratazione con alcol graduato, pulizia con xilene e incorporamento con paraffina come descritto nel riferimento19.
NOTA: Elaborare il campione in una cappa aspirante. - Affettare il cervello incorporato in fette sottili (5 μm di spessore) usando un microtomo. Posizionare le fette campione su vetrini di quarzo. Asciugare i vetrini in forno a 60 °C per 1 ora.
- Deparaffinizzare le sezioni utilizzando un agente di compensazione (vedi Tabella dei materiali), che rimuove la paraffina per evitare segnali di fondo elevati poiché la paraffina è altamente assorbente con l'eccitazione UV.
NOTA: Elaborare il campione in una cappa aspirante.
- Nuova fetta di cervello di topo
- Sacrificare un topo con un sovradosaggio di anestesia. Quindi, raccogliere il cervello del topo seguendo il protocollo indicato nel riferimento18.
- Lavare il cervello del topo con soluzione salina tamponata con fosfato (PBS) per rimuovere il sangue.
- Tagliare una fetta del campione di cervello (~ 5 mm di spessore) a mano, quindi lavare il campione con PBS per rimuovere il sangue sulla sezione trasversale.
3. Procedure sperimentali
- Posizionamento del campione
- Preparare un serbatoio campione realizzato in laboratorio con una membrana trasparente UV (polietilene, ∼ 10 μm di spessore). Aggiungere una goccia d'acqua alla membrana e quindi posizionare il campione biologico sul serbatoio del campione per coprire l'acqua. Nel caso del campione di fetta FFPE, utilizzare del nastro adesivo per fissare il vetrino sulla membrana.
- Posizionare il serbatoio del campione contenente il campione sul supporto del campione, assicurandosi di coprire il foro vuoto del portacampioni.
- Impostare il laser UV sulla modalità di trigger esterno. Utilizzando il nostro programma LabVIEW creato in laboratorio (vedere Tabella dei materiali), impostare i parametri di scansione come segue.
- Impostare la frequenza di ripetizione laser a 55 kHz; impostare il tipo di segnalee del driver GM su triangolare e l'intervallo di tensione di guida su 0,018 V (che rappresenta ±0,018 V; fattore di scala: 1 V/°). Impostare GMnum su 22 e dy su 2 in modo che dopo ogni 22 trigger laser, il GM finisca un quarto del periodo di scansione e il motore dell'asse y sposti una dimensione del passo di 0,3125 μm. Impostare dx su 192 in modo che quando il motore dell'asse y raggiunge la posizione preimpostata (ad esempio, 5 mm), il motore dell'asse x sposti una dimensione del passo di 30 μm.
NOTA: la dimensione del passo incrementale più piccola è impostata su 0,15625 μm per entrambi i motori dell'asse x e y. Pertanto, quando dy = 2, la dimensione del passo è uguale a 0,15625 x 2 = 0,3125 μm e quando dx = 192, la dimensione del passo è uguale a 0,15625 x 192 = 30 μm. Per scansionare l'intera area di 5 x 5 mm2, il motore dell'asse x si muoverà di 5000 μm / 30 μm = 167 volte, il che significa che 167 sotto-immagini verrebbero cucite per ottenere un'intera immagine. Vedere la Figura 2 per la traiettoria di scansione del sistema UV-PAM.
- Impostare la frequenza di ripetizione laser a 55 kHz; impostare il tipo di segnalee del driver GM su triangolare e l'intervallo di tensione di guida su 0,018 V (che rappresenta ±0,018 V; fattore di scala: 1 V/°). Impostare GMnum su 22 e dy su 2 in modo che dopo ogni 22 trigger laser, il GM finisca un quarto del periodo di scansione e il motore dell'asse y sposti una dimensione del passo di 0,3125 μm. Impostare dx su 192 in modo che quando il motore dell'asse y raggiunge la posizione preimpostata (ad esempio, 5 mm), il motore dell'asse x sposti una dimensione del passo di 30 μm.
- Avviare la scansione di prova su una piccola regione impostando il numero di passi mobili dei motori degli assi x e y (xn = 10 e yn = 1000). Regolare lo stadio manuale dell'asse z per posizionare il campione sul piano focale per ottenere il massimo dei segnali PA.
- Spostare i motori dell'asse x e y nel punto di partenza desiderato, impostare la regione di scansione impostando i valori xn e yn , quindi avviare il programma di acquisizione delle immagini (vedere Tabella dei materiali).
- Dopo l'acquisizione dell'immagine, spegnere il laser, rimuovere il portacampioni e conservare i campioni biologici.
- Per i tessuti biologici freschi, conservare i campioni in formalina tamponata neutra al 10%.
- Per le fette di cervello del topo FFPE, colorare con H & E seguendo il protocollo indicato nel riferimento20 per ottenere le corrispondenti immagini colorate di H & E.
- Utilizzare i segnali PA raccolti per ricostruire l'immagine di proiezione di ampiezza massima utilizzando un algoritmo di elaborazione delle immagini costruito in laboratorio (vedere Tabella dei materiali).
Risultati
La Figura 1 mostra lo schema del sistema UV-PAM ad alta velocità. In questa configurazione, i percorsi di eccitazione ottica e rilevamento ad ultrasuoni sono sullo stesso lato e sotto il campione, formando una modalità riflettente e un sistema open-top. Pertanto, è facile da usare e adatto per l'imaging di campioni spessi.
La Figura 2 mostra la traiettoria di scansione del sistema UV-PAM durante l'imaging. La sotto-immagine di ogni sezione (ad esempio, area di 5 mm x 30 μm) viene prima generata utilizzando un algoritmo di interpolazione sparsa, quindi un'intera immagine (ad esempio, un'area di 5 mm x 5 mm) viene ottenuta cucendo tutte le sotto-immagini utilizzando un algoritmo di elaborazione delle immagini personalizzato (vedi Tabella dei materiali).
La Figura 3A e la Figura 3B mostrano rispettivamente le immagini UV-PAM e H&E di una fetta di cervello di topo FFPE, entrambe con un campo visivo di 5 x 5 mm2. È possibile accedere all'algoritmo di elaborazione delle immagini da Github tramite il collegamento fornito nella Tabella dei materiali. Il tempo di acquisizione dell'immagine è stato inferiore a 18 minuti. La Figura 3C-F mostra le immagini ingrandite delle regioni contrassegnate nella Figura 3A, dove i singoli nuclei cellulari possono essere chiaramente risolti. Ancora più importante, i nuclei cellulari corrispondenti possono essere trovati nelle immagini standard colorate di H & E (Figura 3G-J), che mostrano l'elevata precisione dell'attuale sistema per l'imaging cellulare. Per trasferire l'immagine UV-PAM in scala di grigi su un'immagine virtuale macchiata da H&E, è stato applicato un algoritmo di deep learning17 (vedi Tabella dei materiali), che richiederebbe meno di 30 s per un'immagine con 8000 x 8000 pixel. Le corrispondenti immagini ingrandite sono mostrate nella Figura 3K-N. Le immagini UV-PAM praticamente macchiate forniscono quasi le stesse informazioni strutturali delle immagini colorate di H&E, mostrando la promessa per la traduzione clinica dell'attuale sistema UV-PAM.

Figura 1: Lo schema del sistema UV-PAM ad alta velocità e open-top. (A) La configurazione del sistema. (B) Fotografia del portacampioni e di un serbatoio dell'acqua collegato a uno stadio manuale a due assi. (C) Fotografia del trasduttore ad ultrasuoni a forma di anello fissato nel serbatoio dell'acqua e del serbatoio del campione che copre il foro del portacampioni. (D) Fotografia del serbatoio del campione con la membrana e il campione che verrebbero posizionati sul portacampioni. UV: ultravioletto; DAQ: Scheda di acquisizione dati; GM: Specchio galvanometrico. Fare clic qui per visualizzare una versione più grande di questa figura.
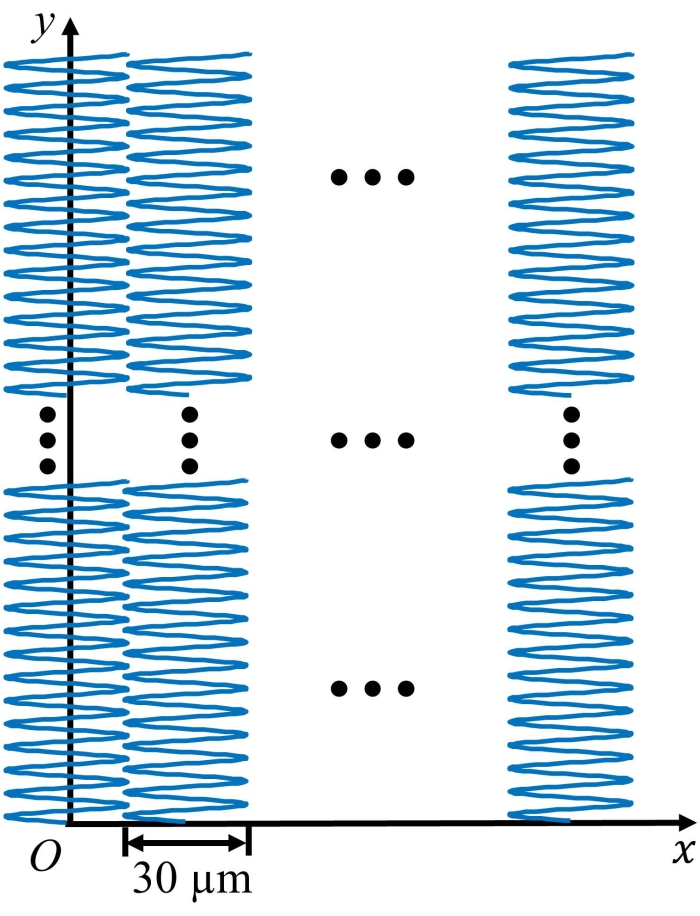
Figura 2: La traiettoria di scansione del sistema UV-PAM durante l'imaging. Fare clic qui per visualizzare una versione più grande di questa figura.
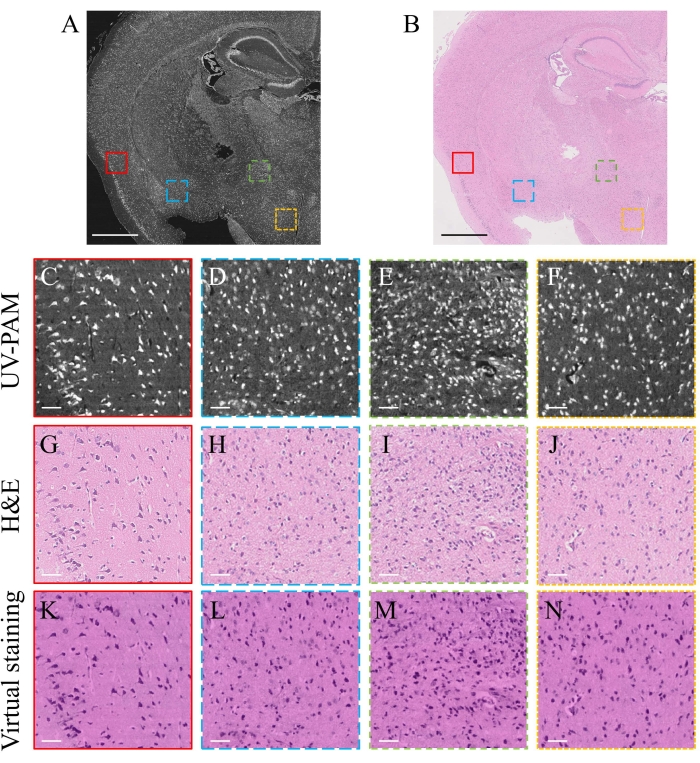
Figura 3: Risultati sperimentali dell'imaging UV-PAM con colorazione virtuale basata sul deep learning. (A) Immagine UV-PAM di una fetta di cervello di topo FFPE. (B) Immagine standard macchiata di H&E della stessa fetta. Barre di scala: 1 mm (C-F) Immagini ingrandite delle regioni contrassegnate in A. (G-J) Corrispondenti immagini colorate di H&E delle regioni contrassegnate in B. (K-N) Immagini corrispondenti virtualmente macchiate utilizzando un algoritmo di deep learning. Barre di scala: 50 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
In sintesi, è stato dimostrato un sistema UV-PAM ad alta velocità e open-top per l'imaging istologico. Vengono presentate le istruzioni dettagliate sulla configurazione del sistema, l'allineamento ottico, la preparazione del campione e le procedure sperimentali. Il programma di acquisizione delle immagini è accessibile da Github tramite il link fornito nella Tabella dei materiali. La risoluzione laterale del sistema attuale è ~ 1,2 μm che è stata misurata sperimentalmente in una recente pubblicazione21. Una fetta di cervello di topo è stata fotografata per dimostrare che il sistema attuale può ottenere un'immagine istologica entro 18 minuti per un'area di 5 x 5 mm2, che è una dimensione tipica della biopsia cerebrale22. Sebbene l'immagine originale sia in scala di grigi, con l'assistenza di uno strumento di colorazione virtuale digitale abilitato da un algoritmo di deep learning, il presente sistema può ulteriormente fornire immagini virtualmente macchiate quasi in tempo reale, garantendo un facile adattamento per i patologi per interpretare le immagini. Come tecnica di immagine senza etichetta, l'attuale sistema UV-PAM può anche fornire immagini istologiche per campioni di tessuto fresco non elaborati. Altri esempi (compresi campioni di tessuto fresco e sezionati congelati) sono stati dimostrati in una precedente pubblicazione17. I risultati sperimentali mostrano l'elevato potenziale dell'attuale sistema UV-PAM assistito da deep learning nelle applicazioni SMA.
Uno dei vantaggi del sistema UV-PAM è che il sistema è implementato in modalità di riflessione, consentendo l'imaging di tessuti spessi. Inoltre, il sistema UV-PAM open-top consente di posizionare il campione sulla finestra di scansione (la membrana del serbatoio del campione), che ha un funzionamento simile alle microscopie ottiche tradizionali. Pertanto, questo sistema è più user-friendly rispetto ad altri sistemi che richiedono che i campioni siano inseriti da due membrane11,14. Inoltre, utilizzando un GM 1D con un laser UV ad alta velocità di ripetizione, l'attuale sistema UV-PAM può raggiungere un'elevata velocità di imaging con un costo maggiore rispetto al sistema che utilizza l'eccitazione multifocale23.
Attualmente, la velocità di imaging è principalmente limitata dal tasso di ripetizione del budget laser e fotone. Con un laser che ha un alto tasso di ripetizione e un'elevata energia di impulso, il tempo di imaging può essere ulteriormente ridotto. Un'altra limitazione del sistema è che il campione può essere regolato solo approssimativamente sul piano focale dell'obiettivo trovando i segnali PA massimi, invece di visualizzare un'immagine in tempo reale per consentire agli utenti di visualizzare se il campione è a fuoco. Per visualizzare un'immagine quasi in tempo reale, è possibile applicare GM 2D.
Ci sono due passaggi critici nel protocollo: (a) il requisito confocale dei fuochi ottici e acustici dovrebbe essere ottimizzato per ottenere un'elevata sensibilità di rilevamento; b) l'intervallo di scansione del GM sull'asse x deve essere inferiore al punto focale acustico dell'UT a forma di anello per mantenere una sensibilità di rilevamento elevata simile (nella configurazione attuale, l'intervallo di scansione è di ~ 30 μm ±15 μm). Altrimenti, evidenti effetti di vignettatura attorno ai bordi si verificherebbero quando più immagini secondarie vengono cucite insieme per ottenere un'intera immagine.
Divulgazioni
T. T. W. W. e V. T. C. T. hanno un interesse finanziario in PhoMedics Limited, che, tuttavia, non ha sostenuto questo lavoro. Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Gli autori desiderano riconoscere il sostegno finanziario della Commissione per l'innovazione e la tecnologia di Hong Kong (ITS/036/19).
Materiali
| Name | Company | Catalog Number | Comments |
| Alcohol | Sigma Aldrich | PHR1373 | Sample dehydration |
| Amplifier | Mini Circuit | ZFL-500LN-BNC+ | Ultrasonic signal amplification |
| Controller | National Instruments | NI myRIO | System controller |
| Data acquisition card | Alazar Technologies | ATS9350 | Ultrasonic signal collection |
| Deep-learning algorithm | For transfering the grayscale UV-PAM image to a virtual H&E-stained image; https://github.com/TABLAB-HKUST/Deep-PAM | ||
| Formalin | Sigma Aldrich | R04586 | Sample fixation |
| H&E staining kit | Abcam | ab245880 | Sample staining |
| Histo-Clear II | National Diagnostics | HS-202 | Sample deparaffinization |
| Image acquisition program | National Instruments | LabVIEW | Lab-built program using LabVIEW; https://github.com/TABLAB-HKUST/LabVIEW-program-for-UV-PAM |
| Image processing algorithm | Mathworks | MATLAB | Lab-built algorithm using MATLAB; https://github.com/TABLAB-HKUST/ImageRec_GM-UVPAM |
| Kinematic platform mounts | Thorlabs | KM200B | Adjust the sample to be flat |
| Membrane | Glad | Cling wrap | Sandwiched in sample tank |
| Microscope objective lens | Thorlabs | LMU- 5X-NUV | Objective lens |
| Motorized stages | Physik Instrumente | L-509.10SD00 | Scanning stages |
| One-dimensional galvanometer mirror | Thorlabs | GVS411 | Fast scanning mirror |
| Oscilloscope | RIGOL Technologies | DS1102E | Ultrasonic signal readout |
| Phosphate-buffered saline | Sigma Aldrich | P3813 | Sample washing |
| Pinhole | Edmund Optics | #59–257 | Spatial filtering |
| Plano convex lens | Thorlabs | LA-4600-UV | Focusing lens |
| Plano convex lens | Thorlabs | LA-4663-UV | Collimating lens |
| Pulser/receiver | Imaginant | DPR300 | Pulse echo amplifier |
| Q-switch diode-pumped solid-state laser | Bright Solutions | WEDGE HF 266 nm | 266-nm laser |
| Ring-shaped ultrasonic transducer | University of Southern California | Ultrasonic signal detection | |
| Sample holder | Lab-made | Hold the sample tank | |
| Sample tank | Lab-made | Hold biological samples | |
| Single-axis Z-translational stage | Thorlabs | PT1 | Manual stage |
| Two-axis manual stage | Thorlabs | LX20 | Manual stage |
| Water tank | Lab-made | Ultrasonic signal transmission | |
| Xylene | Sigma Aldrich | XX0060 | Sample clearing |
Riferimenti
- Kunos, C., et al. Breast conservation surgery achieving ≥ 2 mm tumor-free margins results in decreased local-regional recurrence rates. The Breast Journal. 12, 28-36 (2006).
- Rosai, J. Why microscopy will remain a cornerstone of surgical pathology. Laboratory Investigation. 87 (5), 403-408 (2007).
- Yang, J., Caprioli, R. M. Matrix sublimation/recrystallization for imaging proteins by mass spectrometry at high spatial resolution. Analytical Chemistry. 83 (14), 5728-5734 (2011).
- Jaafar, H. Intra-operative frozen section consultation: concepts, applications and limitations. The Malaysian Journal of Medical Sciences: MJMS. 13 (1), 4(2006).
- Rastogi, V., et al. Artefacts: a diagnostic dilemma - a review. Journal of Clinical and Diagnostic Research. 7 (10), 2408-2413 (2013).
- Carrasco-Zevallos, O. M., et al. Review of intraoperative optical coherence tomography: technology and applications [Invited]. Biomedical Optics Express. 8 (3), 1607(2017).
- Gareau, D. S., Jeon, H., Nehal, K. S., Rajadhyaksha, M. Rapid screening of cancer margins in tissue with multimodal confocal microscopy. Journal of Surgical Research. 178 (2), 533-538 (2012).
- Fereidouni, F., et al. Microscopy with ultraviolet surface excitation for rapid slide-free histology. Nature Biomedical Engineering. 1 (12), 957-966 (2017).
- Tanaka, N., et al. Whole-tissue biopsy phenotyping of three-dimensional tumours reveals patterns of cancer heterogeneity. Nature Biomedical Engineering. 1 (10), 796-806 (2017).
- Jain, M., et al. Multiphoton microscopy: A potential intraoperative tool for the detection of carcinoma in situ in human bladder. Archives of Pathology and Laboratory Medicine. 139 (6), 796-804 (2015).
- Orringer, D. A., et al. Rapid intraoperative histology of unprocessed surgical specimens via fibre-laser-based stimulated Raman scattering microscopy. Nature Biomedical Engineering. 1 (2), 0027(2017).
- Wong, T. T. W., et al. Fast label-free multilayered histology-like imaging of human breast cancer by photoacoustic microscopy. Science Advances. 3 (5), 1602168(2017).
- Shen, C. -H. Detection and analysis of nucleic acids. Diagnostic Molecular Biology. , 167-185 (2019).
- Yao, J., Wang, L. V. Photoacoustic microscopy. Laser & Photonics Reviews. 7 (5), 1-36 (2012).
- Li, X., Kang, L., Zhang, Y., Wong, T. T. W. High-speed label-free ultraviolet photoacoustic microscopy for histology-like imaging of unprocessed biological tissues. Optics Letters. 45 (19), 5401(2020).
- Baik, J. W., et al. Intraoperative label-free photoacoustic histopathology of clinical specimens. Laser & Photonics Reviews. 15 (10), 2100124(2021).
- Kang, L., Li, X., Zhang, Y., Wong, T. T. W. Deep learning enables ultraviolet photoacoustic microscopy based histological imaging with near real-time virtual staining. Photoacoustics. 25, 100308(2022).
- Sterile Tissue Harvest | Protocol. , Available from: https://www.jove.com/science-education/10298/sterile-tissue-harvest (2022).
- Steps to Tissue Processing for Histopathology. , Available from: https://www.liecabiosystems.com/knowledge-pathway/an-introduction-to-specimen-processing/ (2022).
- An Intro to H&E Staining: Protocol, Best Practices, Steps & More. , Available from: https://www.liecabiosystems.com/knowledge-pathway/he-staining-overview-a-guide-to-best-practices/ (2022).
- Li, X., et al. Ultraviolet photoacoustic microscopy with tissue clearing for high-contrast histological imaging. Photoacoustics. 25, 100313(2022).
- Leinonen, V., et al. Assessment of β-amyloid in a frontal cortical brain biopsy specimen and by positron emission tomography with carbon 11-labeled pittsburgh compound B. Archives of Neurology. 65 (10), 1304-1309 (2008).
- Imai, T., et al. High-throughput ultraviolet photoacoustic microscopy with multifocal excitation. Journal of Biomedical Optics. 23 (03), (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon