È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging dal vivo dei primi progenitori cardiaci nell'embrione di topo
In questo articolo
Riepilogo
Presentiamo un protocollo dettagliato per la coltura e l'imaging di embrioni di topo che consente l'imaging 3D + tempo delle cellule progenitrici cardiache. Questo video-toolkit affronta le competenze chiave necessarie per immagini live di successo altrimenti difficili da acquisire da pubblicazioni di solo testo.
Abstract
I primi passi dello sviluppo del cuore implicano drastici cambiamenti nel comportamento e nella differenziazione cellulare. Mentre l'analisi degli embrioni fissi consente di studiare in dettaglio specifiche fasi di sviluppo in un'istantanea fissa, l'imaging dal vivo cattura eventi morfogenetici dinamici, come la migrazione cellulare, i cambiamenti di forma e la differenziazione, visualizzando l'embrione mentre si sviluppa. Questo integra l'analisi fissa e amplia la comprensione di come gli organi si sviluppano durante l'embriogenesi. Nonostante i suoi vantaggi, l'imaging dal vivo è raramente utilizzato nei modelli murini a causa delle sue sfide tecniche. I primi embrioni di topo sono sensibili se coltivati ex vivo e richiedono una manipolazione efficiente. Per facilitare un uso più ampio dell'imaging dal vivo nella ricerca sullo sviluppo del topo, questo articolo presenta un protocollo dettagliato per la microscopia viva a due fotoni che consente l'acquisizione a lungo termine negli embrioni di topo. Oltre al protocollo, vengono forniti suggerimenti sulla gestione degli embrioni e sull'ottimizzazione della coltura. Ciò aiuterà a comprendere gli eventi chiave nell'organogenesi precoce del topo, migliorando la comprensione della biologia dei progenitori cardiovascolari.
Introduzione
Il cuore si forma presto durante l'embriogenesi per iniziare a pompare sostanze nutritive all'intero embrione, mentre continua a svilupparsi1. Negli embrioni di topo, un giorno e mezzo dopo l'inizio della gastrulazione, un organo cardiaco rudimentale si assembla al polo anteriore 2,3. Allo stadio Early Streak (ES), i progenitori cardiaci nell'ingresso dell'epiblasto attraverso la striscia primitiva fino allo strato mesodermico nascente 4,5,6 e iniziano a migrare verso il polo anteriore, dove si differenziano per formare il tubo cardiaco primitivo. Durante questo processo, i primi progenitori cardiaci subiscono riarrangiamenti cellulari, trasformazioni di forma e differenziazione, oltre alla migrazione7 (Figura 1).
I primi progenitori cardiaci hanno attratto ricercatori per quasi un secolo grazie alla loro notevole capacità di differenziare e costruire un organo funzionale contemporaneamente. Negli ultimi due decenni, l'analisi clonale e i modelli di knockout condizionale hanno dimostrato che lo sviluppo precoce del cuore implica fonti cellulari distinte in un processo altamente dinamico 8,9,10. Tuttavia, la struttura 3D del tubo cardiaco primitivo e la natura dinamica della sua morfogenesi lo rendono difficile da studiare (Figura 1), e siamo lontani dal comprendere la sua piena complessità11.
Per studiare questi processi cellulari dinamici, i metodi di imaging dal vivo offrono ora un dettaglio senza precedenti 7,12,13,14. Nel modello murino, gli approcci dal vivo sono stati fondamentali per interrogare argomenti di sviluppo che sono difficili da affrontare con l'analisi statica 7,13,15. Mentre la coltura ex vivo a lungo termine e le configurazioni di microscopi robusti stanno avanzando rapidamente16,17, pochi ricercatori hanno l'esperienza per visualizzare con successo embrioni vivi. Sebbene le pubblicazioni cartacee forniscano dettagli tecnici sufficienti per riprodurre esperimenti di imaging dal vivo, alcune abilità e trucchi sono difficili da comprendere senza esempi visivi o assistenza peer-to-peer. Per accelerare questo processo di apprendimento e diffondere l'uso dell'imaging dal vivo tra i laboratori, abbiamo assemblato un protocollo video (Figura 2) che raccoglie le competenze necessarie per eseguire l'imaging dal vivo su embrioni di topo gastrulati.
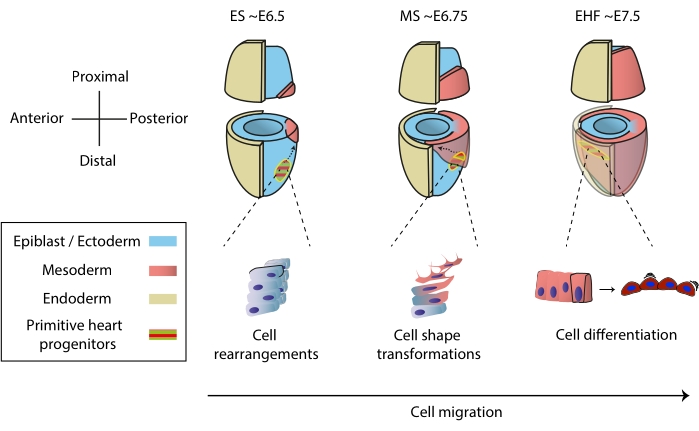
Figura 1: Differenziazione precoce delle cellule progenitrici cardiache nell'embrione di topo dall'inizio della gastrulazione allo stadio che precede la formazione primitiva del tubo cardiaco. Le cellule progenitrici cardiache entrano nel mesoderma subito dopo l'inizio della gastrulazione, migrando verso il lato opposto dell'embrione. La fase morfologica ed embrionale del giorno (E) sono scritte in cima ai diagrammi. Le frecce tratteggiate raffigurano la traiettoria di migrazione dei progenitori primitivi del tubo cardiaco durante la gastrulazione. Questa cifra è stata adattata da11. Abbreviazioni: ES = Early Streak; MS = Striscia media; EHF = Early Head Fold. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 2: Diagramma del flusso di lavoro per l'imaging in tempo reale dei primi progenitori cardiaci. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutte le procedure animali sono state approvate dal Comitato Etico per la Sperimentazione Animale CNIC, dalla Comunità di Madrid (Riferimento PROEX 220/15) e conformi alla Direttiva UE 2010/63UE e alla Raccomandazione 2007/526/CE relativa alla protezione degli animali utilizzati a fini sperimentali e ad altri scopi scientifici, applicata dalla legge spagnola ai sensi del Real Decreto 1201/2005.
Questo protocollo include l'uso di due maschi della linea di topi transgenici fluorescenti che riportano l'attività NOTCH Tg (CBF: H2BVenus,+)18. Vedere la tabella dei materiali per i dettagli su materiali, animali e attrezzature utilizzati in questo protocollo.
1. Personalizzazione degli strumenti e del supporto
- Tagliare pezzi di filo di tungsteno lunghi 1 cm e affilarli a 0,02-0,05 mm di diametro usando uno dei semplici metodi disponibili, ad esempio, immergendo la punta del filo in una soluzione satura di nitrito di sodio19.
- Preparare capillari di vetro a gomito.
- Posizionare il punto medio di un capillare di vetro standard da 1,0 mm su un accendino Bunsen per 3-6 s, tirando lentamente da entrambe le estremità fino a quando il capillare diventa sottile e flessibile. A quel punto, spezzare il capillare a metà e riscaldarlo brevemente per produrre una curva di 90° a 2 cm dalla punta.
- Ho visto un pezzo di polimetilmetacrilato che misura circa 7 mm di lunghezza, 4 mm di larghezza e 2 mm di spessore (Figura 3D).
NOTA: Le lastre di polimetilmetacrilato possono essere ottenute da apparecchiature di laboratorio riciclate o ordinate nuove. - Tenere il pezzo di metacrilato usando una morsa da banco. Quindi, praticare fori di dimensioni personalizzate trasversalmente attraverso l'intero pezzo (Figura 3D).
NOTA: In genere, gli embrioni di topo da E6,5 a E7,5 richiedono l'uso di trapani di diametro 0,2-0,5 mm.- Ruotare lentamente il trapano applicando una pressione bassa ma costante. Se un trapano si rompe all'interno del supporto, scartare il pezzo, poiché il metallo non può essere estratto.
- Utilizzare una lima fine per smussare i bordi del supporto e regolarne le dimensioni. Inoltre, creare una leggera pendenza sui bordi del supporto per facilitare il posizionamento dell'embrione (Figura 3D).
- Posizionare il supporto finito in un piatto con acqua distillata per risciacquarlo.
- Controllare il supporto sotto lo stereomicroscopio per vedere se i fori contengono polvere di perforazione. Se è presente polvere, utilizzare aghi di tungsteno per rimuoverlo.
- Per sterilizzare il supporto, posizionarlo in un tubo conico pieno di acqua distillata e farlo sonicare per 20 minuti con una potenza minima di 20 W.
2. Preparazione dei media
- Per inattivare il sistema del complemento a caldo, lasciare due aliquote da 500 μL di siero di ratto 20 commerciale o fatto in casa a 56 °C per30 minuti.
NOTA: Per un protocollo dettagliato su come preparare il siero per ratti, vedere21. - In una cappa di coltura cellulare, preparare due aliquote del mezzo per la coltura degli embrioni al microscopio. Per ciascuno, mescolare 490 μL di DMEM per l'imaging di cellule vive, 500 μL di siero di ratto inattivato e 10 μL di penicillina-streptomicina per ottenere una concentrazione finale di 50 μg/mL di penicillina e 50 μg/mL di streptomicina22.
NOTA: Il volume del terreno di coltura dipende dal tempo e dal numero di embrioni da coltivare. Come regola generale, la crescita ottimale richiede un minimo di 200 μL per embrione al giorno negli embrioni E7.0. - Utilizzando una siringa da 2 ml, filtrare il terreno attraverso un filtro a membrana da 0,22 μm di dimensioni dei pori in un nuovo tubo e posizionarlo con coperchio aperto in un incubatore per colture cellulari a 37 °C e al 7% di CO2 per 1 ora prima della coltura embrionale23.
- Preparare il mezzo di dissezione aggiungendo a 500 ml di DMEM integrato con flacone di L-glutammina quanto segue: 50 ml di siero fetale bovino, 10 ml di 25 mM di HEPES-NaOH (pH 7,2) e 10 ml di penicillina e streptomicina (50 mg / ml).
- Quindi, separarlo in aliquote da 50 ml, posizionare tre aliquote in un bagno a 37 °C e conservare il resto a 4 °C per un massimo di 3 mesi.
3. Dissezione embrionale
- Eutanasia di una topo gravida E6.75-E7.5.
- Estrarre l'utero e metterlo su una salvietta di carta asciutta e pulita. Quindi, tagliare l'utero, inserendo la punta delle forbici sottili dal lato mesometriale e facendo scorrere la lama lungo per esporre le decidue e trasferirle nel mezzo di dissezione. Per un protocollo dettagliato, vedere24.
- Sezionare gli embrioni, mantenendo intatto il cono ectoplacentare (Figura 3A,B). Per un metodo di dissezione dettagliato, fare riferimento a25.
- Quindi, staccare la membrana di Reichert, lasciandone parte sulla regione extraembrionale (Figura 3B).
NOTA: Le pinze sottili sono essenziali per questo passaggio. Gli utenti inesperti possono sezionare gli embrioni su piatti rivestiti di gel di agarosio al 2% per evitare di piegare le punte delle pinze contro la superficie del piatto (Figura 3C). - Per mantenere caldo il mezzo di dissezione, sostituirlo ogni 10 minuti. Inoltre, sezionare le decidue in gruppi di 3-4 mantenendo il resto in mezzo di dissezione posto in un bagno a 37 °C. Posizionare immediatamente gli embrioni sezionati in terreno di coltura.
- Selezionare gli embrioni con le caratteristiche di fluorescenza desiderate utilizzando uno stereomicroscopio a fluorescenza e posizionarli nel tubo contenente terreno di coltura nell'incubatore di coltura cellulare.
- Se gli embrioni sono agli stadi da E6.5 a E7.0, chiudere il coperchio e lasciare gli embrioni a recuperare per 2 ore prima dell'imaging.
NOTA: gli embrioni da E6.5 a E7.0 sono sensibili. Possono anche essere posizionati direttamente al microscopio, ma la precoltura consente di selezionare quelli che meglio recuperano dalla dissezione, aumentando le possibilità di successo.
4. Preparazione del microscopio
- Accendere tutti i componenti del microscopio, inclusi laser, computer e software. Accendere i termoregolatori e impostare a 37 °C 3 h in anticipo per garantire l'equilibrio.
- Se si utilizza un obiettivo di rilevamento ad immersione, pulirlo con una salvietta monouso priva di residui. Immergere la punta dell'obiettivo in acqua bidistillata utilizzando una capsula da 60 mm e lasciarla fino all'inizio dell'acquisizione.
- Accendere il controller CO2 e impostarlo su 7%.
- Posizionare una goccia di grasso siliconico ad alto vuoto su una teglia da 35 mm con fondo in vetro (diametro 14 mm), posizionare il supporto sopra la goccia e premere delicatamente per immobilizzarla.
- Sotto uno stereomicroscopio, aggiungere terreno di coltura per riempire la parte di vetro del piatto. Mentre lo fai, punta il flusso verso i fori del supporto per evitare la formazione di bolle d'aria in essi.
- Se le bolle rimangono, utilizzare il capillare di vetro preparato al punto 1.2, collegato a un tubo di silicone con un boccaglio, per aspirare delicatamente le bolle.
NOTA: L'estremità a gomito del capillare faciliterà l'accesso attraverso i fori.
5. Montaggio embrionale
- Trasferire 2-3 embrioni che sono stati lasciati recuperare nell'incubatrice al piatto con il titolare. Lasciare il resto nell'incubatore come backup.
- Impostare gli embrioni nei fori usando un paio di pinze e un ago di tungsteno affusolato. Utilizzare l'ago per agganciare l'embrione dal cono ectoplacentare e inserirlo in un foro di dimensioni corrispondenti. Per immobilizzare l'embrione, ruotare il cono di 90-120° in modo che la membrana del Reichter aderisca alle pareti del foro (Figura 3C). Per una descrizione alternativa, vedere26.
- Spostare con cautela il piatto contenente gli embrioni e il supporto sulla piastra del microscopio (Figura 3F,G).
NOTA: Se non viene spostato costantemente, l'embrione può uscire dal titolare. - Abbassare l'obiettivo fino a formare un menisco liquido all'interfaccia dell'obiettivo medio.
- Localizzare l'embrione utilizzando la luce trasmessa sotto il binocolo del microscopio e mettere a fuoco l'embrione.
- Montare la camera di incubazione e sigillarla attorno all'obiettivo usando del nastro adesivo (Figura 3F).
- Utilizzando un Live Capture nel software di controllo del microscopio, regolare i livelli di uscita laser e guadagno.
NOTA: Per l'acquisizione a due fotoni di proteine fluorescenti verdi (GFP) e reporter Tdtomato, in questo protocollo sono stati utilizzati il 30% di potenza laser in uscita a 980 nm e un guadagno del 70% su rivelatori non descan (Figura 3G). - Per evitare l'evaporazione, coprire il mezzo con olio di paraffina gocciolando lentamente sull'obiettivo.
NOTA: Questo è l'ultimo punto in cui è possibile montare embrioni aggiuntivi nel supporto. Una volta coperto con olio di paraffina, si dovrebbe pulire il supporto e sostituire il mezzo per farlo. Per metodi di montaggio alternativi vedere27.
6. Acquisizione di immagini
- Imposta la registrazione time-lapse. Impostare un intervallo di tempo di 5-10 minuti con uno spazio z di 3-5 μm tra le pile. Lasciare uno spazio vuoto sopra lo z-stack per anticipare la crescita dell'embrione, un minimo di 50 μm, aggiungendo 30 μm per ogni ora in cui l'acquisizione non sarà supervisionata.
- Se disponibile, abilitare l'opzione Salvataggio automatico per consentire la supervisione dell'acquisizione da un personal computer per consentire la regolazione delle dimensioni dello z-stack, se necessario, per adattarla all'area di interesse all'interno del fuoco.

Figura 3: Strumenti di imaging in tempo reale e configurazione . (A) Dissezione di embrioni di topo con un piatto rivestito di agarosio sotto lo stereomicroscopio. (B) Schema delle fasi per sezionare embrioni di topo per l'imaging dal vivo. (C) Posizionamento dell'embrione nel titolare. (D) Design e caratteristiche del titolare dell'embrione. (F) Camera dell'incubatore attorno all'obiettivo di immersione. (G) Schema della configurazione finale per l'acquisizione time-lapse. Barra di scala = 500 μm (A). Fare clic qui per visualizzare una versione ingrandita di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
Abbiamo usato il protocollo per visualizzare l'attivazione della segnalazione NOTCH nei primi progenitori cardiaci in procinto di differenziarsi in cellule endoteliali durante la morfogenesi primitiva del tubo cardiaco. Per questo, abbiamo incrociato topi selvatici C57BL / 6-N con topi Tg (CBF: H2BVenus, +)18 per ottenere embrioni che riportavano attività NOTCH attraverso la proteina fluorescente gialla Venere. A E7.5, la fluorescenza di Venere è presente in tutto l'ectoderma neurale, con alcuni...
Access restricted. Please log in or start a trial to view this content.
Discussione
I primi progenitori del cuore si organizzano in un tubo cardiaco primitivo che inizia a battere mentre si sta ancora formando. Capire come avviene questo processo è la chiave per individuare l'ampio spettro di difetti cardiaci congeniti a specifici eventi morfogenetici. Per questo, l'imaging dal vivo offre l'opportunità di studiare lo sviluppo embrionale normale e difettoso con una maggiore risoluzione temporale. Ciò è particolarmente utile per studiare le cellule progenitrici cardiache precoci mentre passano rapidam...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Gli autori riconoscono il Dr. Kenzo Ivanovitch per il precedente lavoro su questo metodo e il gruppo del Dr. Shigenori Nonaka (National Institutes of Natural Sciences, Giappone) per aver fornito le competenze iniziali sul montaggio degli embrioni. Questo studio è stato supportato da Grant PGC2018-096486-B-I00 del Ministerio de Ciencia e Innovación spagnolo e Grant H2020-MSCA-ITN-2016-722427 dal programma Horizon 2020 dell'UE a MT e Grant 1380918 dal programma operativo FEDER Andalucía 2014-2020 a JND. MS è stata sostenuta da una borsa di dottorato della Fondazione La Caixa (LCF / BQ / DE18 / 11670014) e dalla borsa di studio itinerante The Company of Biologists (DEVTF181145). Il CNIC è sostenuto dal Ministero della Scienza spagnolo e dalla Fondazione ProCNIC.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| #55 Forceps | Dumont | 11295-51 | |
| 35 mm Dish with glass coverslip bottom 14 mm Diameter | Mattek | P35G-1.5-14-C | |
| 35 mm vise table | Grandado | SKU 8798771617573 | |
| 50 mL tubes | BD Falcon | 352070 | |
| Distilled water | |||
| DMEM - Dulbecco's Modified Eagle Medium | Gibco | 11966025 | with L-Glutamine, without Glucose, without Na Pyruvate |
| Fetal Bovine Serum | Invitrogen | 10438-026 | |
| Fluorescent reporter transgenic mice (Tg(CBF:H2BVenus,+) | JAX | ||
| Fluorobrite DMEM | ThermoFisher | A1896701 | DMEM for live-cell imaging |
| High-vacuum silicone grease | Dow Corning | Z273554-1EA | |
| Holder for wires | Perlen Pressen | pwb1 | |
| LSM 780 Upright microscope | Zeiss | ||
| MaiTai Deepsee far red pulsed-laser tuned at 980 nm | Spectra-Physics | ||
| Non Descanned Detectors equipped with the filter sets cyan-yellow (BP450-500/BP520-560), green-red (BP500-520/BP570-610) and yellow-red (BP520-560/BP645-710) | Zeiss | ||
| Obj: 20x water dipping 1.0 NA, long working distance | Zeiss | ||
| P1000 and P200 pipettes | |||
| Paraffin Oil | Nidacon | VNI0049 | |
| Penicillin-streptomycin | Invitrogen | 15070-063 | (the final concentration should be 50 μg/mL penicillin and 50 μg/mL streptomycin) |
| Petri dishes 35 mm x 10 mm | BD Falcon | 351008 | |
| Pipette tips | |||
| Polymethyl methacrylate | Reused from old laboratory equipment | ||
| Rat Serum culture embryo, male rats SPRAGUE DAWLEY RjHan SD | Janvier Labs | 9979 | |
| Set of 160 mm fines | RS PRO | 541-6933 | |
| Standard 1.0 mm glass capillaries | Anima Lab | 1B100F-3 | |
| Sterile 0.22 μm syringe filter | Corning | 431218 | |
| Sterile 5 mL syringe | Fisher Scientific | 15809152 | |
| Tungsten needles | |||
| Ultrasonic homogeniser (sonicator) | Bandelin | BASO_17021 |
Riferimenti
- Tyser, R. C. V., et al. Calcium handling precedes cardiac differentiation to initiate the first heartbeat. eLife. 5, 17113(2016).
- Kelly, R. G., Buckingham, M. E., Moorman, A. F. Heart fields and cardiac morphogenesis. Cold Spring Harbor Perspectives in Medicine. 4 (10), 015750(2014).
- Evans, S. M., Yelon, D., Conlon, F. L., Kirby, M. L. Myocardial lineage development. Circulation Research. 107 (12), 1428-1444 (2010).
- Tam, P. P., Parameswaran, M., Kinder, S. J., Weinberger, R. P. The allocation of epiblast cells to the embryonic heart and other mesodermal lineages: the role of ingression and tissue movement during gastrulation. Development. 124 (9), Cambridge, England. 1631-1642 (1997).
- Kinder, S. J., Loebel, D. A. F., Tam, P. P. L. Allocation and early differentiation of cardiovascular progenitors in the mouse embryo. Trends in Cardiovascular Medicine. 11 (5), 177-184 (2001).
- Lawson, K. A. Fate mapping the mouse embryo. International Journal of Developmental Biology. 43 (7), 773-775 (1999).
- Ivanovitch, K., Temiño, S., Torres, M. Live imaging of heart tube development in mouse reveals alternating phases of cardiac differentiation and morphogenesis. eLife. 6, 30668(2017).
- Meilhac, S. M., Buckingham, M. E. The deployment of cell lineages that form the mammalian heart. Nature Reviews Cardiology. 15 (11), 705-724 (2018).
- Buckingham, M., Meilhac, S., Zaffran, S. Building the mammalian heart from two sources of myocardial cells. Nature Reviews Genetics. 6 (11), 826-835 (2005).
- Meilhac, S. M., Lescroart, F., Blanpain, C. D., Buckingham, M. E. Cardiac cell lineages that form the heart. Cold Spring Harbor Perspectives in Medicine. 4 (9), 013888(2014).
- Sendra, M., Domínguez, J. N., Torres, M., Ocaña, O. H. Dissecting the complexity of early heart progenitor cells. Journal of Cardiovascular Development and Disease. 9 (1), 5(2022).
- McDole, K., et al. In toto imaging and reconstruction of post-implantation mouse development at the single-cell level. Cell. 175 (3), 859-876 (2018).
- Saykali, B., et al. Distinct mesoderm migration phenotypes in extra-embryonic and embryonic regions of the early mouse embryo. eLife. 8, 42434(2019).
- Ichikawa, T., et al. Live imaging of whole mouse embryos during gastrulation: Migration analyses of epiblast and mesodermal cells. PLoS ONE. 8 (7), 64506(2013).
- Tyser, R. C. V., et al. Single-cell transcriptomic characterization of a gastrulating human embryo. Nature. 600 (7888), 285-289 (2021).
- Aguilera-Castrejon, A., et al. Ex utero mouse embryogenesis from pre-gastrulation to late organogenesis. Nature. 593 (7857), 119-124 (2021).
- Yue, Y., et al. in toto live imaging of cardiomyocyte behaviour during mouse ventricle chamber formation at single-cell resolution. Nature Cell Biology. 22 (3), 332-340 (2020).
- Nowotschin, S., Xenopoulos, P., Schrode, N., Hadjantonakis, A. K. A bright single-cell resolution live imaging reporter of Notch signaling in the mouse. BMC Developmental Biology. 13 (1), 15(2013).
- Cold Spring Harbor Protocols. Sharpened tungsten needles. Cold Spring Harbor Protocols. , (2012).
- Tam, P. P., Snow, M. H. The in vitro culture of primitive-streak-stage mouse embryos. Journal of Embryology and Experimental Morphology. 59, 131-143 (1980).
- Garcia, M. D., Udan, R. S., Hadjantonakis, A. K., Dickinson, M. E. Preparation of rat serum for culturing mouse embryos. Cold Spring Harbor Protocols. 2011 (4), 5593(2011).
- Tam, P. P. L. Postimplantation mouse development: Whole embryo culture and micro- manipulation. International Journal of Developmental Biology. 42 (7), 895-902 (1998).
- González Hinojosa, F. Optimización de propiedades fisicoquímicas y medios de cultivo para el cultivo del embrión de ratón ex vivo. Universidad de Jaén. Biología Experimental. , Available from: https://hdl.handle.net/10953.1/1400 (2021).
- Behringer, R., Gertsenstein, M., Vintersen Nagy, K., Nagy, A. Manipulating the mouse embryo: A laboratory manual, Fourth Edition. , Cold Harbor Laboratory Press. 814(2014).
- Shea, K., Geijsen, N. Dissection of 6.5 dpc mouse embryos. Journal of Visualized Experiments. (2), e160(2006).
- Nonaka, S. Modification of mouse nodal flow by applying artificial flow. Methods in Cell Biology. 91, 287-297 (2009).
- Garcia, M. D., Udan, R. S., Hadjantonakis, A. K., Dickinson, M. E. Time-lapse imaging of postimplantation mouse embryos. Cold Spring Harbor Protocols. 2011 (4), 5595(2011).
- Crainiciuc, G., et al. Behavioural immune landscapes of inflammation. Nature. 601 (7893), 415-421 (2022).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon