È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Research Article
Migliorare i risultati degli studenti con un'esperienza di ricerca universitaria basata su corsi di clonazione molecolare adattabile
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Un modulo di clonazione molecolare adattabile di Gibson Assembly è stato impiegato in un formato di esperienza di ricerca universitaria (CURE) basato su corsi per studenti di laboratori di biologia molecolare. La valutazione dei risultati di apprendimento degli studenti ha mostrato una migliore comprensione e fiducia nella clonazione molecolare dopo il completamento del CURE e nuovi plasmidi sono stati clonati per la ricerca sulla biosintesi di prodotti naturali.
Abstract
Il continuo progresso delle tecniche di biologia molecolare richiede che i curricula di biologia molecolare siano regolarmente perfezionati per preparare efficacemente gli studenti a entrare nel mondo del lavoro con competenze moderne. In particolare, l'emergere del Gibson Assembly, una tecnica di clonazione molecolare altamente personalizzabile e adattiva, ha fatto progredire il panorama della clonazione molecolare in numerosi ambienti di ricerca. Pertanto, abbiamo creato un modulo di clonazione Gibson Assembly per l'implementazione in un corso di laboratorio di biologia molecolare presso la California Polytechnic State University, San Luis Obispo e abbiamo valutato i risultati di apprendimento degli studenti dal modulo. Nel corso di tre iterazioni del corso, gli studenti hanno partecipato a un progetto indipendente basato su esperimenti che prevedeva la clonazione di tre librerie di plasmidi uniche per supportare progetti di ricerca sulla biosintesi di prodotti naturali. Agli studenti sono stati somministrati questionari pre e post per valutare la loro comprensione della clonazione molecolare e la loro fiducia nei termini e nelle tecniche di biologia molecolare. Le risposte degli studenti hanno mostrato un aumento significativo sia nell'apprendimento dei concetti di clonazione molecolare che nella fiducia auto-riferita con i termini e le tecniche di clonazione molecolare. Questo framework di moduli può essere generalizzato per insegnare a Gibson Assembly varie applicazioni, fornendo agli istruttori un toolkit per insegnare una tecnologia di clonazione adattabile ed emergente mentre avanzano i loro progetti di ricerca.
Introduzione
La formazione degli studenti universitari nei concetti fondamentali della biologia molecolare e nelle tecniche di laboratorio è fondamentale per il loro sviluppo scientifico e professionale poiché queste metodologie sono comuni in vari contesti di ricerca, tra cui il mondo accademico e l'industria. Pertanto, gli studenti di biologia (concentrazione di biologia molecolare e cellulare) e biochimica presso la California Polytechnic State University, San Luis Obispo (Cal Poly) sono tenuti a seguire un corso di laboratorio di biologia molecolare di divisione superiore per apprendere e applicare questi argomenti (CHEM / BIO 475). Un curriculum di base per questo corso è stato precedentemente sviluppato in cui gli studenti eseguono il clonaggio a base di topoisomerasi (TOPO) per assemblare un plasmide contenente actina preparato da un modello di DNA complementare di lievito (cDNA)1. Gli studenti progettano esperimenti basati su domande che imitano ipotesi di ricerca autentiche, aumentando la loro familiarità con le pratiche di laboratorio e l'apprendimento basato sull'indagine. Il continuo progresso nel campo della biologia molecolare richiede che i curricula corrispondenti si adattino per preparare gli studenti con competenze moderne per la forza lavoro. In particolare, l'uso dell'Assemblea di Gibson è diventato più predominante nella comunità scientifica; mentre il metodo è stato originariamente stabilito per sintetizzare cromosomi artificiali2, oltre 5.000 pubblicazioni al momento di questo rapporto hanno fatto riferimento al lavoro originale di Gibson et al. Gibson Assembly è unico rispetto alle metodologie di clonazione tradizionali: è altamente personalizzabile e può facilmente legare più frammenti di DNA lineare senza la necessità di siti di restrizione per produrre le giunzioni. Pertanto, abbiamo visto l'opportunità di rinnovare il curriculum CHEM/BIO 475 per incorporare le moderne tecniche di clonazione molecolare e migliorare il modello di corso basato sull'indagine.
È stato stabilito che le esperienze di ricerca degli studenti contribuiscono ad aumentare la comprensione concettuale, lo sviluppo delle competenze e la persistenza nella scienza3, ma non tutti gli studenti universitari hanno l'opportunità di partecipare direttamente a un laboratorio di ricerca. Per affrontare la sfida della limitata capacità degli studenti nei laboratori di ricerca, sono state sviluppate e impiegate esperienze di ricerca universitaria basate su corsi (CURE) per aumentare l'accessibilità della scienza attraverso una ricerca autentica in classe. Sebbene le CURE varino nella loro attuazione, sono state stabilite pratiche comuni che affrontano i cinque principi della ricerca scientifica. In un CURE ben progettato, gli studenti 1) utilizzeranno pratiche scientifiche, 2) collaboreranno a un progetto di ricerca, 3) tenteranno di fare nuove scoperte, 4) contribuiranno a un lavoro rilevante al di fuori della classe e 5) rivaluteranno e rivedranno ipotesi e metodi in caso di fallimento sperimentale4. Analogamente alle tradizionali esperienze di ricerca degli studenti in laboratorio, è stato dimostrato che le CURE rafforzano la fiducia degli studenti nella scienza, nelle competenze scientifiche, nella proprietà del progetto e nella persistenza in scienza, tecnologia, ingegneria e matematica (STEM)5. Mentre le CURE che coinvolgono il clonaggio molecolare sono state riportate in precedenza 6,7,8,9,10,11,12,13, non siamo a conoscenza di alcuna che enfatizzi l'adattabilità di Gibson Assembly per creare una libreria di autentica ricerca plasmidi.
Qui, riportiamo un'espansione dell'attuale curriculum CHEM/BIO 475 basato sull'indagine presso la California Polytechnic State University, San Luis Obispo con due importanti miglioramenti: l'esperienza pratica con Gibson Assembly e la partecipazione degli studenti a un CURE, che ha fornito costrutti plasmidi originali per progetti di ricerca finanziati dalla National Science Foundation (NSF-1708919 e NSF-2300890). Nel corso di tre implementazioni di questo curriculum, gli studenti hanno contribuito a due distinti progetti di ricerca incentrati sulla biosintesi di prodotti naturali di molecole bioattive prodotte da Actinomycetota. I prodotti naturali contengono spesso farmacofori con attività antibiotiche, antimicotiche e/o antitumorali, conferendo a queste piccole molecole importanza negli sforzi di scoperta di farmaci e potenziale di rilevanza clinica14. Questa ricerca richiede la creazione di librerie di plasmidi per consentire lo studio della funzione e del potenziale ingegneristico degli enzimi biosintetici batterici. In questo CURE, gli studenti hanno progettato ed eseguito esperimenti di Gibson Assembly per clonare le librerie di plasmidi uniche pertinenti a questi progetti di ricerca (Figura 1). Inoltre, il formato e il design del modulo si distinguono perché è facilmente adattabile per generare qualsiasi plasmide di interesse per altri progetti di ricerca.
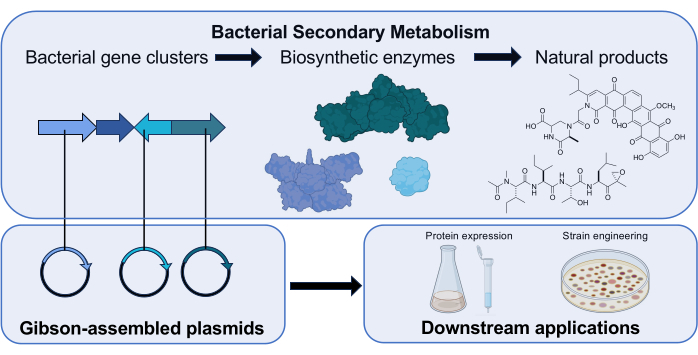
Figura 1: Panoramica del ruolo di Gibson Assembly nel nostro laboratorio di ricerca. Gli actinomycetota producono prodotti naturali a piccole molecole con bioattività clinicamente rilevanti utilizzando cluster genici che codificano enzimi biosintetici. Nella nostra ricerca, i plasmidi contenenti un gene biosintetico vengono assemblati tramite Gibson Assembly per indagini a valle sulla funzione dell'enzima codificato. Icone scientifiche da Biorender.com. Clicca qui per visualizzare una versione più grande di questa figura.
Panoramica del laboratorio
Il laboratorio di biologia molecolare è un corso di divisione superiore richiesto per le major di biochimica e biologia con una concentrazione di biologia molecolare e cellulare. Altri studenti che soddisfano i prerequisiti sono invitati a seguire il corso come elettivo di divisione superiore. Il corso è co-elencato tra il Dipartimento di Chimica e Biochimica e il Dipartimento di Biologia di Cal Poly. I docenti di entrambi i dipartimenti si alternano nell'insegnamento del corso ogni trimestre (due trimestri di Biologia, un quarto di Chimica e Biochimica).
La classe si riunisce in laboratorio due volte alla settimana per periodi di 170 minuti e una volta alla settimana per una lezione di 50 minuti. Le sezioni di laboratorio contengono un massimo di 16 studenti e ogni trimestre vengono offerte 2-3 sezioni di laboratorio. Il corso dura 10 settimane e l'esame finale viene somministrato durante l'ultimo incontro della settimana 10. Il tempo della lezione è dedicato alla discussione della teoria alla base di molte delle tecniche sperimentali condotte in laboratorio, nonché di argomenti attuali in biologia molecolare che non sono trattati in laboratorio. Il curriculum di base del laboratorio comprende il processo di clonazione del gene dell'actina dal lievito1, che richiede circa 7 settimane (13-14 incontri di laboratorio). Le tecniche includono il micropipettaggio, l'isolamento dell'RNA del lievito, l'amplificazione di un gene del lievito mediante la reazione a catena della polimerasi a trascrizione inversa (RT-PCR), il clonaggio topografico, lo screening blu-bianco, l'isolamento del plasmide e la verifica dell'inserto mediante restriction digest e PCR, l'analisi in silico dei cloni e l'analisi della sequenza del DNA. Il curriculum per le ultime 3 settimane del corso è a discrezione dell'istruttore ma prevede che gli studenti completino un "progetto indipendente" con risultati indefiniti.
Panoramica dell'esperimento
Uno degli obiettivi del nostro gruppo di ricerca sono le vie biosintetiche in Actinomycetota. Durante la progettazione del progetto indipendente, abbiamo immaginato che gli studenti creassero plasmidi attraverso Gibson Assembly da utilizzare nei nostri progetti di ricerca che studiano la biosintesi di prodotti naturali. Mentre le iterazioni del modulo del corso qui valutate erano specifiche per i plasmidi che consentono la manipolazione di percorsi biosintetici, il flusso di lavoro di Gibson Assembly è enormemente adattabile per altri progetti di clonaggio molecolare (Figura 2). Il flusso di lavoro è stato suddiviso in tre diversi esperimenti (A, B e C) che sono stati completati in due periodi di lezione e sei periodi di laboratorio (3 settimane in totale) (vedi File supplementare 1 e File supplementare 2). Gli esperimenti sono stati preceduti da fogli di lavoro per supportare la preparazione degli studenti e valutare la comprensione degli studenti (File supplementare 3, File supplementare 4 e File supplementare 5). Il flusso di lavoro viene presentato in un formato flessibile in base alle esigenze e agli interessi dell'insegnante.

Figura 2: Flusso di lavoro del modulo Gibson Assembly. Il Giorno 1 e il Giorno 4 sono periodi di lezione in cui gli studenti completano l'analisi di sequenze in silico e la progettazione sperimentale. I giorni 2-3 e 5-8 sono riunioni di laboratorio in cui vengono condotte le fasi per clonare nuovi plasmidi tramite Gibson Assembly, seguite dall'isolamento e dallo screening. Il diagramma di flusso pittorico è raggruppato in base ai tre esperimenti eseguiti dagli studenti (A, B e C). Istruzioni e protocolli più dettagliati sono disponibili nei manuali dell'istruttore e dello studente forniti rispettivamente come File supplementare 1 e File supplementare 2 . Icone scientifiche da Biorender.com. Clicca qui per visualizzare una versione più grande di questa figura.
Il modulo di progetto indipendente Gibson Assembly è stato testato per la prima volta nel trimestre primaverile del 2019 in CHEM/BIO 475. Nel 2020 e nel 2021, il corso è stato tenuto online a causa della pandemia di SARS-CoV-2. Quando l'istruzione in presenza è ripresa nella primavera del 2022 e del 2023, gli studenti del corso sono stati invitati a partecipare a uno studio che valutava i risultati dell'apprendimento di un progetto indipendente Gibson Assembly in cui sarebbero stati clonati plasmidi originali e rilevanti per la ricerca. Nel 2019, gli studenti hanno creato una libreria di plasmidi costituita da cassette geniche del genoma di Micromonospora echinospora ATCC 15837 che sono state clonate in pKC1132 (Figura 3). Questa libreria di plasmidi viene utilizzata nel nostro laboratorio di ricerca per inattivare geni di interesse in un presunto cluster di geni biosintetici per il prodotto naturale TLN-0522015. Per completare i nostri studi sull'inattivazione genica, nel 2022 gli studenti hanno clonato una piccola libreria di geni dal presunto cluster genico TLN-05220 in pUC19 (figura3); Il nostro gruppo di ricerca ha utilizzato questi plasmidi per il subclonaggio di geni in vettori di espressione, tra cui pET28b, per la sovraespressione e la purificazione delle proteine. Gli studenti della coorte 2023 hanno contribuito al lavoro in corso su un progetto di ingegneria biosintetica sull'epoxomicina sintetasi16. In team di 3-4 persone, gli studenti hanno clonato domini ingegnerizzati di moduli di peptide sintetasi17 non ribosomiale in vari vettori di espressione proteica per ottimizzare la sovraespressione e la purificazione di questi enzimi nel nostro laboratorio di ricerca (Figura 3). La ridondanza è stata integrata nel piano di clonazione per ciascuna coorte. Ad esempio, la coorte del 2019 conteneva 44 studenti e 15 plasmidi sono stati assegnati alla classe per la clonazione. Così, la clonazione di ciascun plasmide è stata tentata due o tre volte.
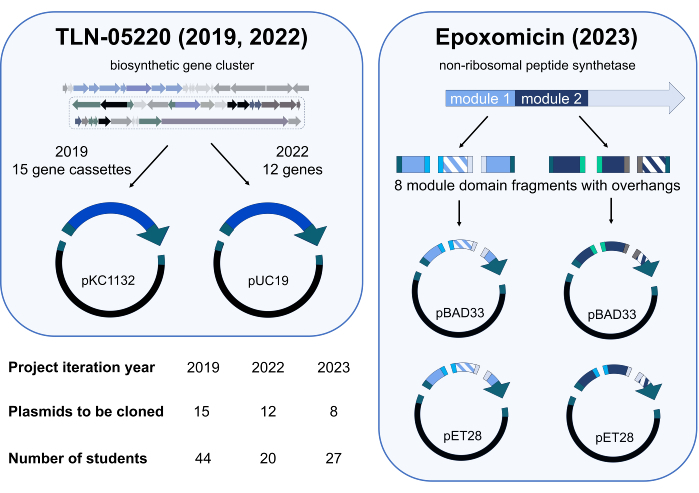
Figura 3: Riepilogo dei plasmidi clonati e degli studenti partecipanti durante le iterazioni di progetti indipendenti del 2019, 2022 e 2023. Il progetto Gibson Assembly è stato implementato tre volte. In ogni offerta, gli studenti partecipanti hanno clonato una diversa libreria di plasmidi da utilizzare in progetti di ricerca che esplorano percorsi biosintetici. I progetti del 2019 e del 2022 hanno sostenuto il nostro lavoro in corso sul prodotto naturale TLN-0522015 con due reazioni di assemblaggio Gibson in frammenti (una cassetta genica o un gene e un vettore). Il progetto del 2023 prevedeva lo scambio di domini all'interno dei moduli 1 e 2 di un enzima peptide sintetasi non ribosomiale (NRPS) coinvolto nella biosintesi dell'epoxomicina16. I frammenti tratteggiati rappresentano due diversi mutanti del dominio scambiato, mentre i colori solidi rappresentano i domini che non sono stati scambiati. In totale, sono stati generati otto diversi frammenti genici (quattro per il modulo 1 e quattro per il modulo 2) con sporgenze compatibili per il Gibson Assembly. Per ogni modulo, sono state assemblate due diverse combinazioni di tre frammenti genici con uno dei due diversi vettori (pBAD33 e pET28, quattro frammenti totali per assemblaggio), per generare otto plasmidi NRPS ingegnerizzati. Icone scientifiche da Biorender.com. Clicca qui per visualizzare una versione più grande di questa figura.
Panoramica della valutazione
Almeno 1 settimana prima dell'inizio del progetto indipendente Gibson Assembly, gli studenti delle coorti 2022 e 2023 sono stati invitati a partecipare a un'indagine sui risultati dell'apprendimento, progettata in modo simile a uno studio condotto in un corso di laboratorio di biochimica presso Cal Poly18. Gli studenti partecipanti hanno completato un pre-questionario a scelta multipla durante la riunione di laboratorio prima dell'inizio del progetto indipendente e un post-questionario a scelta multipla durante la riunione finale di laboratorio (cioè dopo aver completato il modulo Gibson Assembly). I questionari pre e post consistevano in 28 domande identiche con due domande aggiuntive nel post-questionario (30 in totale). Sono state scritte dieci domande di contenuto per valutare la conoscenza degli studenti degli enzimi e dei meccanismi coinvolti nel clonaggio molecolare (ad esempio, reazione a catena della polimerasi [PCR], assemblaggio di Gibson, trasformazione, screening blu-bianco). Le sette domande seguenti hanno chiesto agli studenti di autovalutare la loro familiarità con i termini di clonazione molecolare (ad esempio, DNA polimerasi, esonucleasi, ligasi). Le successive 10 domande hanno permesso agli studenti di autovalutare la loro capacità di eseguire tecniche di clonaggio molecolare (ad esempio, analisi della sequenza del DNA, reazioni di digestione di restrizione, elettroforesi su gel di agarosio). Gli studenti hanno anche riferito se si sentirebbero a proprio agio nel perseguire una carriera in biologia molecolare basata sulla loro conoscenza delle tecniche di clonazione molecolare. Due domande aggiuntive sono state incluse nel post-questionario per consentire agli studenti di autovalutare il loro impegno nell'apprendimento durante il corso e se il corso è stato un'esperienza di apprendimento preziosa (Supplemental File 6 e Supplemental File 7). Tutti i dati delle risposte al questionario pre e post questionario degli studenti del 2022 e del 2023 sono stati combinati per l'analisi e sono disponibili nella Tabella supplementare S1.
Protocollo
Gli studi che hanno coinvolto partecipanti umani sono stati esaminati e approvati da soggetti umani nel Research Institutional Review Board di Cal Poly (2022-113-CP (IRB)). I partecipanti hanno fornito il loro consenso informato scritto per partecipare a questo studio.
Il seguente protocollo delinea la preparazione dell'istruttore (passaggi 1.1-1.3), le azioni degli studenti per un modulo didattico a tre esperimenti che include la PCR per ottenere frammenti lineari (passaggi 2.1-2.7), l'assemblaggio, la trasformazione e la selezione di cloni di Gibson (passaggi 3.1-3.5), l'isolamento e lo screening dei plasmidi (passaggi 4.1-4.4) e la valutazione dei risultati di apprendimento (5.1-5.2). La preparazione dell'istruttore descrive un esempio rappresentativo di progettazione di primer e preparazione di una mappa plasmidica desiderata in silico. Tutte le sezioni del protocollo sono adattabili ad altri plasmidi desiderati. Ogni esperimento degli studenti è diviso in due incontri di laboratorio di 3 ore.
1. Preparazione dell'istruttore
- Progettazione di primer di Gibson Assembly utilizzando la procedura guidata di assemblaggio Benchling
- Determinare la fonte del modello di DNA per gli inserti genici, come il DNA genomico, il plasmide e il DNA sintetico.
NOTA: Per i geni che saranno amplificati da modelli di DNA genomico complessi, si consiglia di determinare prima i siti di legame unici per i primer all'interno del modello di DNA genomico che sono specifici per il gene o i geni di interesse, utilizzando il National Center for Biotechnology Information (NCBI) Basic Local Alignment Search Tool (BLAST)21, come dettagliato nel File supplementare 1. Per modelli semplici come plasmidi o DNA sintetico, procedi al passaggio successivo. - Recuperare le sequenze di DNA dell'inserto genico e del vettore (ad esempio, vedere la sequenza genica della polichetide idrossilasi e la sequenza pUC19 fornite come file GenBank nel file supplementare 8). Importa la sequenza di inserimento desiderata e la sequenza vettoriale desiderata in Benchling, ciascuna come un nuovo file di sequenza di DNA. Aprire ogni sequenza importata da includere nella reazione di assemblaggio Gibson.
NOTA: È possibile importare file di sequenze di DNA in diversi formati, tra cui GenBank e FASTA. Si può anche importare una sequenza direttamente da un database con un numero di accesso o incollare una sequenza nucleotidica copiata da un altro file o visualizzatore. - Nella parte inferiore dello schermo, individuare lo strumento Assembly Wizard . Fare clic su Creazione guidata assemblaggio, quindi selezionare Crea nuovo assemblaggio. Dalle opzioni fornite, selezionate Gibson e fate clic su Avvia per iniziare l'assemblaggio.
- Nella finestra della sequenza vettoriale , selezionare tutte le basi da includere nell'assieme dalla spina dorsale vettoriale. Iniziare la selezione in corrispondenza della posizione nucleotidica all'estremità 3' dell'inserto genico (ad esempio, la posizione 657 di pUC19) e selezionare tutti i nucleotidi rimanenti da includere (ad esempio, attraverso la posizione 656 per includere l'intera sequenza di pUC19). Una volta selezionato, fai clic sulla scheda Backbone nella parte inferiore dello schermo| Imposta frammento.
NOTA: Alterna tra la visualizzazione della mappa di sequenza e quella del plasmide del vettore per selezionare tutte le basi desiderate di un modello circolare. In alternativa, se si prevede di preparare il frammento vettoriale tramite digest di restrizione (invece della PCR) per l'assemblaggio di Gibson, linearizzare la sequenza della spina dorsale con un sito di taglio dell'enzima di restrizione (ad esempio, XbaI). A tale scopo, fare clic sul sito di taglio, tenere premuto Maiusc e fare nuovamente clic sul sito di taglio . Quindi fare clic su Imposta frammento per impostare la spina dorsale. Tenere presente che i primer specifici del vettore non verranno generati da Benchling se la spina dorsale viene linearizzata con un enzima di restrizione nella procedura guidata di assemblaggio. - Nella finestra della sequenza di inserimento , selezionare tutte le basi dell'inserto da includere nell'assieme. Una volta selezionato, fai clic sulla scheda Inserisci nella parte inferiore dello schermo | Imposta frammento.
- In caso di più inserimenti genici, fare clic sul pulsante + sul lato destro della procedura guidata di assemblaggio. Nella finestra della sequenza di inserimento , selezionare tutte le basi di inserimento da includere nell'assieme. Una volta selezionato, fai clic sulla scheda Inserisci nella parte inferiore dello schermo | Imposta frammento.
- Una volta impostati tutti i frammenti, rinominare l'assieme con il nome del plasmide desiderato e fare clic su Assembla a destra della procedura guidata di assemblaggio. Selezionate la posizione della cartella desiderata sia per la cartella Sequenza che per la cartella Primer. Fare clic su Crea per assemblare la sequenza plasmidico ricombinante.
- Aprire il plasmide assemblato e fare clic su Cronologia assemblaggi per visualizzare una mappa plasmidica di base e una panoramica delle sequenze da cui è stata derivata. Assicurarsi che le posizioni dei nucleotidi per ogni frammento corrispondano al design desiderato.
- Nella scheda Parametri assieme , visualizzare i nomi dei primer progettati per l'assieme. Si noti che ci saranno due primer progettati per ogni inserto (uno in avanti e uno in retro), e i nomi dei primer saranno derivati dai titoli dei file di sequenza del DNA. Assicurarsi inoltre che le temperature di fusione del primer e le temperature di ricottura suggerite siano compatibili in questa finestra (vedere File supplementare 8, Primer rappresentativi).
- Se le temperature di fusione o di ricottura del primer non sono ideali, regolare manualmente le sequenze di primer (ad esempio, rimuovendo i nucleotidi per ridurre la temperatura di fusione) all'interno dei file della sequenza di primer. In alternativa, fare clic su Riapri e regolare le impostazioni del primer facendo clic sul pulsante Impostazioni enzima/primer accanto al pulsante Assembla nella procedura guidata di assemblaggio. Quindi, ripetere la fase di assemblaggio e riesaminare le sequenze di primer.
- Se una libreria di plasmidi deve essere clonata con molti geni diversi clonati nella stessa spina dorsale vettoriale,
- Utilizzare la stessa coppia di primer per il vettore in ogni caso. Per fare ciò, rimuovere manualmente tutti i nucleotidi dai primer vettoriali che installerebbero le sporgenze specifiche dell'inserto. Questi nucleotidi si troverebbero all'estremità 5' dell'innesco. Dopo la rimozione, assicurarsi che le temperature di fusione del primer e le temperature di ricottura siano compatibili (vedere File supplementare 8, Primer rappresentativi).
- Assicurarsi che tutte le coppie di primer dell'inserto abbiano temperature di fusione e temperature di ricottura molto simili.
NOTA: Ciò ridurrà la necessità di ottimizzare la temperatura di ricottura di ogni coppia e consentirà di eseguire contemporaneamente il ciclo di tutte le reazioni PCR degli studenti utilizzando lo stesso programma di termociclatore.
- Una volta completata la progettazione del primer per i plasmidi desiderati, fare clic su Finalizza nella procedura guidata di assemblaggio. Ordina i primer da un'azienda di sintesi del DNA e recupera i campioni dei modelli di DNA per inserti e vettori.
- Testare le coppie di primer specifici per l'inserto e il vettore per ottenere temperature di ricottura ottimali utilizzando un esperimento di PCR in gradiente con i modelli di DNA. Testare inizialmente le temperature di ricottura consigliate da Benchling ± 3 °C.
- Una volta determinate le temperature di ricottura, creare aliquote di soluzioni di primer, modelli di DNA e reagenti necessari per le reazioni PCR per gli studenti.
- Determinare la fonte del modello di DNA per gli inserti genici, come il DNA genomico, il plasmide e il DNA sintetico.
- Prima degli esperimenti, fornisci agli studenti dei fogli di lavoro per supportare la preparazione e valutare la comprensione (Supplemental File 4, Supplemental 5 e Supplemental 6).
- Raccolta di dati sui risultati dell'apprendimento
NOTA: Se sei interessato a raccogliere i dati sui risultati dell'apprendimento dal modulo Gibson Assembly e a pubblicare i risultati, ottieni l'approvazione per la ricerca con soggetti umani secondo le politiche dell'istituto.- Invitare gli studenti a partecipare a un'indagine sui risultati dell'apprendimento almeno 1 settimana prima dell'inizio del progetto indipendente Gibson Assembly.
- Chiedi agli studenti partecipanti di completare il pre-questionario a scelta multipla (File supplementare 6) durante l'incontro di laboratorio prima dell'inizio del progetto indipendente.
2. Esperimento A: PCR per ottenere frammenti lineari
- Ottieni primer e soluzioni di DNA stampo per la PCR dall'istruttore.
- Pipettare reazioni da 25 μL tra cui DNA polimerasi MasterMix, DNA stamp, primer diretti e inversi e acqua priva di nucleasi secondo le istruzioni del manuale dello studente (vedere il file supplementare 2).
- Ciclica la reazione in un termociclatore secondo il manuale dello studente nel File supplementare 2. Assicurarsi che la temperatura di ricottura della reazione sia appropriata per gli inneschi e che il tempo di prolungamento sia appropriato per la lunghezza dell'amplicone desiderato. Per istruzioni dettagliate, fare riferimento a File supplementare 2 e File supplementare 3 .
- Conservare le reazioni PCR a 4 °C o -20 °C fino al successivo periodo di laboratorio.
- Analizzare 5 μL di ciascuna reazione tramite elettroforesi su gel di agarosio. Mentre il gel è in funzione, aggiungere 1 μL di enzima di restrizione DpnI a ciascuna reazione che ha utilizzato il DNA plasmidico come modello. Incubare questa miscela per 1 ora a 37 °C.
NOTA: Il DpnI degraderà qualsiasi stampo di DNA plasmidico circolare rimasto, rendendo meno probabile l'ottenimento di falsi positivi nella fase di trasformazione. - Eseguire l'imaging del gel per confermare che la PCR ha avuto successo e che è stata raggiunta l'amplificazione corretta. Purifica qualsiasi reazione PCR riuscita con un kit di purificazione PCR disponibile in commercio. Misurare la concentrazione (ng/μL) del prodotto PCR purificato utilizzando uno spettrofotometro a microvolumi da utilizzare nei successivi calcoli di assemblaggio Gibson.
- Prima della prossima riunione di classe, progetta la ricetta di reazione per l'assemblaggio di Gibson (vedi File supplementare 2 e File supplementare 4).
3. Esperimento dello studente B: Assemblaggio, trasformazione e selezione di cloni di Gibson
- Pipettare la reazione di assemblaggio Gibson secondo la ricetta progettata. Incubare la reazione a 50 °C per 15 minuti. Mentre le reazioni incubano, preparatevi alla trasformazione scongelando cellule di Escherichia coli chimicamente competenti su ghiaccio.
- Trasformare 2 μl del gruppo Gibson in celle chimicamente competenti tramite shock termico. Pipettare 100 μl delle cellule trasformate su due piastre di selezione e distribuire con perle sterilizzate. Preparare 10-4, 10-5 e 10-6 diluizioni delle cellule rimanenti su agar Luria-Bertani (LB). Piastra 100 μL di ogni diluizione seriale su LB (nessuna selezione) e spalmata con perle sterilizzate.
NOTA: Questo passaggio consente di calcolare l'efficienza di trasformazione. - Incubare le piastre per una notte a 37 °C. Conservare le piastre a 4 °C fino al periodo successivo della lezione.
- Conta le colonie su tutte le piastre e calcola l'efficienza della trasformazione come indicato nel File Supplementare 2. Riavvitare una piastra selettiva con quattro colonie da una delle due piastre selettive e incubare questa piastra a 37 °C per una notte.
NOTA: Questo serve come coltura di backup dei tuoi cloni positivi. - Usando un pennarello, seleziona, cerchia ed etichetta quattro colonie distinte sulla piastra di selezione con le iniziali dello studente e un numero (ad esempio, ABC1). Recuperare una nuova piastra LB-agar contenente antibiotico per la selezione, utilizzare un pennarello per dividere la piastra in quadranti (cioè quarti), quindi utilizzare ~1/2 di ciascuna colonia per ri-striare sul quadrante rispettivamente etichettato e l'altro 1/2 per inoculare una coltura LB liquida da 5 ml per ciascuna delle quattro colonie selezionate. Assicurati di etichettare le provette con la rispettiva identità della colonia (ad esempio, ABC1) e di aggiungere la corretta concentrazione di antibiotico per la selezione. Incubare le colture liquide in un incubatore vibrante a 37 °C per una notte e la piastra di agar in un incubatore statico a 37 °C; conservare le colture liquide e le piastre a 4 °C fino al periodo di lezione successivo.
4. Esperimento C: Isolamento e screening dei plasmidi
- Isolare il DNA plasmidico dalle colture liquide dell'Esperimento B utilizzando un kit di miniprep. Misurare la concentrazione di plasmide isolato (in ng/μL).
- Progettare un digest di restrizione o uno screening PCR per analizzare i plasmidi isolati (vedere le istruzioni dettagliate nel File supplementare 2 e riportare le aspettative nel File supplementare 5).
- Pipettare le reazioni di restrizione o le reazioni secondo le ricette progettate e incubare alle temperature e alle durate progettate in precedenza.
- Analizza i risultati tramite elettroforesi su gel di agarosio.
5. Valutazione
- Chiedi agli studenti partecipanti di completare il post-questionario a scelta multipla (File supplementare 7) la riunione di laboratorio dopo il completamento del modulo Gibson Assembly.
- Combina tutti i dati delle risposte pre e post questionario per l'analisi.
- Esaminare la significatività statistica dell'aumento del punteggio medio di ogni singola domanda di contenuto tra i questionari utilizzando i t-test accoppiati a due code e la dimensione dell'effetto d di Cohen.
- Valuta l'aumento della fiducia degli studenti per ogni singolo termine e tecnica per determinare l'entità del cambiamento e la sua significatività statistica.
- Determina l'impatto del background degli studenti e dei fattori accademici sull'apprendimento degli studenti utilizzando il guadagno di apprendimento normalizzato (NLG) e i t-test non accoppiati.
- Valutare l'atteggiamento degli studenti nei confronti dell'esperienza progettuale e delle carriere in biologia molecolare valutando i punteggi medi.
Risultati
Successo degli studenti nella clonazione
In ogni iterazione del modulo Gibson Assembly (2019, 2022 e 2023), agli studenti è stato chiesto di preparare un rapporto che riassumesse i loro risultati. Nel 2019, 36 studenti su 44 (81,8%) hanno riferito di aver clonato con successo i loro plasmidi sulla base dei risultati dello schermo che hanno progettato per l'esperimento C. Un totale di 14 studenti su 20 (70,0%) ha riportato successo nella clonazione dei costrutti asseg...
Discussione
Qui, proponiamo un progetto di laboratorio universitario regolabile in aula che insegna agli studenti la clonazione molecolare attraverso Gibson Assembly in un contesto di ricerca basato su corsi. In totale, 28 nuovi plasmidi sono stati clonati da studenti universitari in un ambiente scolastico. Il successo riportato dai singoli studenti variava dal 44,4% all'81,8% in tre coorti e il successo complessivo della clonazione è stato dell'80% (28 dei 35 plasmidi totali assegnati sono stati c...
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti o altri conflitti di interesse.
Riconoscimenti
Gli autori ringraziano Andrea Laubscher per il supporto tecnico e Michael Black, Sandi Clement e Javin Oza per le utili discussioni sull'implementazione del laboratorio didattico e sulla valutazione dei risultati dell'apprendimento. Gli autori ringraziano tutti gli studenti che hanno partecipato allo studio sui risultati di apprendimento nelle coorti 2019, 2022 e 2023, nonché gli studenti di ricerca Nathan Kuhn e Aayushi Adettiwar che hanno assistito alla preparazione dei reagenti per l'implementazione del laboratorio didattico. Gli autori riconoscono anche il sostegno finanziario del William and Linda Frost Fund, del Center for Applications in Biotechnology's Chevron Biotechnology Applied Research Endowment Grant e della National Science Foundation (NSF-1708919 e NSF-2300890).
Materiali
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
Riferimenti
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon