Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Research Article
Улучшение результатов студентов с помощью адаптируемого исследовательского опыта бакалавриата на основе курса молекулярного клонирования
* Эти авторы внесли равный вклад
В этой статье
Резюме
Адаптируемый модуль молекулярного клонирования Gibson Assembly был использован в формате студенческого исследовательского опыта (CURE) на основе курса для студентов лабораторных курсов молекулярной биологии. Оценка результатов обучения учащихся показала улучшение понимания и уверенности в молекулярном клонировании после завершения CURE, и новые плазмиды были клонированы для исследований биосинтеза природных продуктов.
Аннотация
Непрерывное развитие методов молекулярной биологии требует, чтобы учебные программы по молекулярной биологии регулярно совершенствовались для эффективной подготовки студентов к выходу на рынок труда с современными компетенциями. В частности, появление Gibson Assembly, гибкого и адаптивного метода молекулярного клонирования, продвинуло ландшафт молекулярного клонирования в многочисленных исследовательских средах. Таким образом, мы создали модуль клонирования Gibson Assembly для использования в лабораторном курсе молекулярной биологии в Калифорнийском политехническом государственном университете в Сан-Луис-Обиспо и оценили результаты обучения студентов с помощью этого модуля. В течение трех итераций курса студенты участвовали в независимом экспериментальном проекте, который включал клонирование трех уникальных библиотек плазмид для поддержки исследовательских проектов в области биосинтеза натуральных продуктов. Студентам были предоставлены анкеты до и после обучения, чтобы оценить их понимание молекулярного клонирования и их уверенность в терминах и методах молекулярной биологии. Ответы студентов показали значительное увеличение как в изучении концепций молекулярного клонирования, так и в самооценке уверенности в терминах и методах молекулярного клонирования. Эта модульная структура может быть обобщена для обучения Gibson Assembly различным приложениям, предоставляя инструкторам набор инструментов для обучения адаптируемой и новой технологии клонирования при продвижении их исследовательских проектов.
Введение
Обучение студентов фундаментальным концепциям молекулярной биологии и лабораторным методам имеет решающее значение для их научного и профессионального развития, поскольку эти методологии широко распространены в различных исследовательских средах, включая академические круги и промышленность. Таким образом, студенты специальностей «Биология» (специализация «Молекулярная и клеточная биология») и «Биохимия» в Калифорнийском политехническом государственном университете в Сан-Луис-Обиспо (Cal Poly) должны пройти лабораторный курс молекулярной биологии высшего дивизиона, чтобы изучить и применить эти темы (CHEM/BIO 475). Ранее была разработана базовая учебная программа для этого курса, в которой студенты выполняют клонирование на основе топоизомеразы (ТОПО) для сборки актинсодержащей плазмиды, которая получена из матрицы комплементарной ДНК (кДНК) дрожжей1. Учащиеся разрабатывают эксперименты на основе вопросов, которые имитируют подлинные исследовательские гипотезы, расширяя их знакомство с лабораторными практиками и обучением, основанным на исследованиях. Непрерывное развитие в области молекулярной биологии требует, чтобы соответствующие учебные программы были адаптированы для подготовки студентов с современными компетенциями для работы. В частности, использование Gibson Assembly стало более распространенным в научном сообществе; Несмотря нато, что метод был первоначально разработан для синтеза искусственных хромосом2, более 5000 публикаций на момент написания этого отчета ссылались на оригинальную работу Gibson et al. Сборка Gibson уникальна по сравнению с традиционными методологиями клонирования: она обладает широкими возможностями настройки и может легко лигировать несколько линейных фрагментов ДНК без необходимости использования сайтов рестрикции для создания соединений. Таким образом, мы увидели возможность пересмотреть учебную программу CHEM/BIO 475, включив в нее современные методы молекулярного клонирования и улучшив модель курса, основанную на исследованиях.
Былоустановлено, что студенческий исследовательский опыт способствует повышению концептуального понимания, развитию навыков и настойчивости в науке3, однако не все студенты бакалавриата имеют возможность непосредственно участвовать в исследовательской лаборатории. Для решения проблемы ограниченного вместимости студентов в исследовательских лабораториях были разработаны и использованы исследовательские программы бакалавриата на основе курсов (CURE) для повышения доступности науки за счет подлинных исследований в классе. Несмотря на то, что CURE различаются по своей реализации, были установлены общие практики, которые учитывают пять принципов научных исследований. В хорошо разработанном CURE студенты будут 1) использовать научные практики, 2) сотрудничать в исследовательском проекте, 3) пытаться сделать новые открытия, 4) вносить свой вклад в работу, актуальную за пределами аудитории, и 5) пересматривать и пересматривать гипотезы и методы вслучае неудачи эксперимента. Подобно традиционному исследовательскому опыту учащихся в лаборатории, было доказано, что CURE укрепляют уверенность учащихся в науке, научные навыки, ответственность за проект и настойчивость в науке, технологиях, инженерии и математике (STEM)5. В то время как ранее сообщалось о CURE, включающих молекулярное клонирование6,7,8,9,10,11,12,13, мы не знаем ни одного из них, которые подчеркивают способность Gibson Assembly создавать библиотеку подлинных исследовательских плазмид.
В этой статье мы сообщаем о расширении текущей учебной программы CHEM/BIO 475 в Калифорнийском политехническом государственном университете в Сан-Луис-Обиспо с двумя основными улучшениями: практическим опытом использования Gibson Assembly и участием студентов в CURE, которое предоставило оригинальные плазмидные конструкции для исследовательских проектов, финансируемых Национальным научным фондом (NSF-1708919 и NSF-2300890). В ходе трех реализаций этой учебной программы студенты внесли свой вклад в два отдельных исследовательских проекта, направленных на биосинтез биоактивных продуктов биоактивных молекул, производимых Actinomycetota. Натуральные продукты часто содержат фармакофоры с антибиотической, противогрибковой и/или противоопухолевой активностью, что придает этим малым молекулам важность в усилиях по разработке лекарств и потенциал для клинической значимости. Это исследование требует создания библиотек плазмид для изучения как функции, так и инженерного потенциала бактериальных биосинтетических ферментов. В рамках этого исследования студенты разработали и провели эксперименты по сборке Гибсона для клонирования уникальных плазмидных библиотек, относящихся к этим исследовательским проектам (рис. 1). Кроме того, формат и дизайн модуля отличаются тем, что он легко адаптируется для генерации любых плазмид, представляющих интерес для других исследовательских проектов.
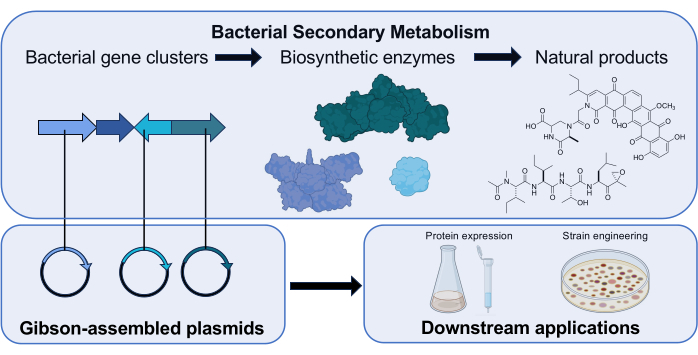
Рисунок 1: Обзор роли Gibson Assembly в нашей исследовательской лаборатории. Actinomycetota продуцируют низкомолекулярные натуральные продукты с клинически значимой биологической активностью, используя кластеры генов, кодирующие биосинтетические ферменты. В нашем исследовании плазмиды, содержащие ген биосинтеза, собираются с помощью Gibson Assembly для последующих исследований функции закодированного фермента. Иконы науки от Biorender.com. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Обзор лаборатории
Лаборатория молекулярной биологии - это курс высшего дивизиона, обязательный для специальностей биохимии и специальностей биологии со специализацией в молекулярной и клеточной биологии. Другие студенты, отвечающие предварительным требованиям, могут пройти курс в качестве факультатива для старших классов. Курс является совместным курсом факультета химии и биохимии и факультета биологии Калифорнийского политехнического университета. Преподаватели обоих факультетов по очереди преподают этот курс каждый семестр (две четверти биологии, одна четверть химии и биохимии).
Класс собирается в лаборатории два раза в неделю по 170 минут и один раз в неделю на 50-минутную лекцию. Лабораторные секции вмещают максимум 16 студентов, и каждый квартал предлагается 2–3 лабораторные секции. Курс длится 10 недель, а заключительный экзамен проводится во время последней встречи на 10-й неделе. Лекционное время посвящается обсуждению теории, лежащей в основе многих экспериментальных методов, проводимых в лаборатории, а также актуальных тем молекулярной биологии, которые не освещаются в лаборатории. Основная учебная программа лаборатории включает в себя процесс клонирования гена актина из дрожжей1, который занимает около 7 недель (13–14 лабораторных заседаний). Методы включают микропипетирование, выделение РНК дрожжей, амплификацию гена дрожжей с помощью полимеразной цепной реакции с обратной транскрипцией (ОТ-ПЦР), клонирование ТОПО, сине-белый скрининг, выделение плазмид и верификацию вкладыша с помощью рестрикционного расщепления и ПЦР, анализ клонов in silico и анализ последовательности ДНК. Учебный план на последние 3 недели курса остается на усмотрение преподавателя, но включает в себя выполнение студентами «самостоятельного проекта» с неопределенными результатами.
Обзор эксперимента
Одним из направлений нашей исследовательской группы являются пути биосинтеза у Actinomycetota. При разработке независимого проекта мы предполагали, что студенты будут создавать плазмиды с помощью Gibson Assembly для использования в наших исследовательских проектах по биосинтезу натуральных продуктов. В то время как итерации модуля курса, которые оцениваются здесь, были специфичны для плазмид, которые позволяют манипулировать биосинтетическими путями, рабочий процесс сборки Гибсона чрезвычайно легко адаптируется для других проектов молекулярного клонирования (рис. 2). Рабочий процесс был разделен на три различных эксперимента (А, В и В), которые были завершены в течение двух лекционных периодов и шести лабораторных периодов (всего 3 недели) (см. Дополнительный файл 1 и Дополнительный файл 2). Экспериментам предшествовали рабочие листы для поддержки подготовки учащихся и оценки их понимания (Дополнительный файл 3, Дополнительный файл 4 и Дополнительный файл 5). Рабочий процесс представлен в формате, который гибко соответствует потребностям и интересам преподавателя.

Рисунок 2: Рабочий процесс модуля Gibson Assembly. День 1 и день 4 — это лекции, на которых студенты выполняют анализ последовательностей in silico и планирование эксперимента. Дни 2–3 и 5–8 – это лабораторные совещания, на которых проводятся этапы клонирования новых плазмид с помощью Gibson Assembly, за которыми следует изоляция и скрининг. Графическая блок-схема сгруппирована по трем экспериментам, которые выполняют учащиеся (A, B и C). Более подробные инструкции и протоколы можно найти в руководствах для инструктора и студента, предоставленных в виде Дополнительного файла 1 и Дополнительного файла 2 соответственно. Иконы науки от Biorender.com. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Модуль независимого проекта Gibson Assembly был впервые опробован в весеннем квартале 2019 года в CHEM/BIO 475. В 2020 и 2021 годах курс преподавался онлайн из-за пандемии SARS-CoV-2. Когда весной 2022 и 2023 годов очное обучение возобновилось, студентам курса было предложено принять участие в исследовании, оценивающем результаты обучения в рамках независимого проекта Gibson Assembly, в рамках которого будут клонированы оригинальные, имеющие отношение к исследованиям плазмиды. В 2019 году студенты создали библиотеку плазмид, состоящую из генных кассет из генома Micromonospora echinospora ATCC 15837, которые были клонированы в pKC1132 (рис. 3). Эта библиотека плазмид используется в нашей исследовательской лаборатории для инактивации генов, представляющих интерес в предполагаемом биосинтетическом кластере генов для природного продукта TLN-0522015. В дополнение к нашим исследованиям по инактивации генов студенты в 2022 году клонировали небольшую библиотеку генов из предполагаемого кластера генов TLN-05220 в pUC19 (Figure 3); Наша исследовательская группа использовала эти плазмиды для субклонирования генов в экспрессирующие векторы, включая pET28b, для сверхэкспрессии и очистки белков. Студенты когорты 2023 года внесли свой вклад в текущую работу над проектом биосинтетической инженерии на эпоксомицинсинтетазе16. В группах из 3–4 человек студенты клонировали сконструированные домены модулей нерибосомной пептидсинтетазы17 в различные векторы экспрессии белков для оптимизации сверхэкспрессии и очистки этих ферментов в нашей исследовательской лаборатории (рис. 3). Избыточность была встроена в план клонирования для каждой когорты. Например, когорта 2019 года состояла из 44 студентов, и 15 плазмид были распределены по классу для клонирования. Таким образом, клонирование каждой плазмиды было предпринято два или три раза.
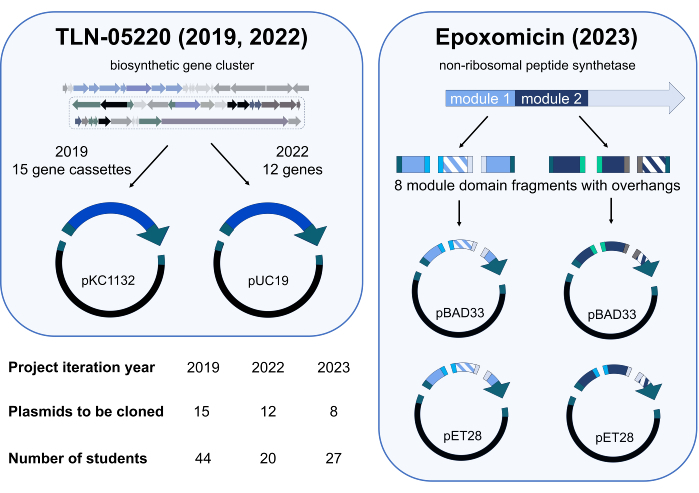
Рисунок 3: Краткая информация о клонировании плазмид и студентах-участниках в ходе независимых итераций проектов в 2019, 2022 и 2023 годах. Проект Gibson Assembly был развернут три раза. В каждом предложении студенты-участники клонировали различную библиотеку плазмид для использования в исследовательских проектах по изучению путей биосинтеза. Проекты в 2019 и 2022 годах поддержали нашу текущую работу над природным продуктом TLN-0522015 с двумя реакциями сборки Гибсона с двумя фрагментами (одна генная кассета или ген и вектор). Проект 2023 года включал в себя замену доменов в модулях 1 и 2 фермента нерибосомальной пептидсинтетазы (NRPS), который участвует в биосинтезе эпоксомицина16. Заштрихованные фрагменты представляют двух разных мутантов поменянного домена, а сплошные цвета представляют домены, которые не были поменяны местами. В общей сложности было получено восемь различных фрагментов генов (четыре для модуля 1 и четыре для модуля 2) с выступами, совместимыми для сборки Gibson. Для каждого модуля были собраны две различные комбинации трех фрагментов гена с одним из двух различных векторов (pBAD33 и pET28, всего четыре фрагмента на сборку) для создания восьми сконструированных NRPS-плазмид. Иконы науки от Biorender.com. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Обзор оценки
По крайней мере за 1 неделю до начала независимого проекта Gibson Assembly студентам из когорт 2022 и 2023 годов было предложено принять участие в исследовании результатов обучения, разработанном аналогично исследованию, проведенному в рамках обзора лабораторного курса биохимии в Cal Poly18. Студенты-участники заполнили предварительную анкету с несколькими вариантами ответов на лабораторной встрече до начала независимого проекта и последующую анкету с несколькими вариантами ответов во время заключительной лабораторной встречи (т.е. после того, как они завершили модуль Gibson Assembly). Анкеты до и после опроса состояли из 28 идентичных вопросов с двумя дополнительными вопросами в постанкете (всего 30). Десять вопросов по содержанию были написаны для оценки знаний студентов о ферментах и механизмах, участвующих в молекулярном клонировании (например, полимеразная цепная реакция [ПЦР], сборка Гибсона, трансформация, сине-белый скрининг). В следующих семи вопросах студентам было предложено самостоятельно оценить свое знакомство с терминами молекулярного клонирования (например, ДНК-полимераза, экзонуклеаза, лигаза). Следующие 10 вопросов позволили студентам самостоятельно оценить свои способности к выполнению методов молекулярного клонирования (например, анализ последовательности ДНК, реакции рестрикционного переваривания, электрофорез в агарозном геле). Студенты также сообщили, будет ли им комфортно продолжать карьеру в области молекулярной биологии, основываясь на своих знаниях о методах молекулярного клонирования. В постанкету были включены два дополнительных вопроса, чтобы студенты могли самостоятельно оценить свою приверженность обучению на курсе и то, был ли курс ценным опытом обучения (Дополнительный файл 6 и Дополнительный файл 7). Все данные ответов студентов на анкеты до и после опроса 2022 и 2023 годов были объединены для анализа и доступны в дополнительной таблице S1.
протокол
Исследования с участием людей были рассмотрены и одобрены людьми в Исследовательском институциональном наблюдательном совете Cal Poly (2022-113-CP (IRB)). Участники предоставили письменное информированное согласие на участие в данном исследовании.
В приведенном ниже протоколе описывается подготовка преподавателя (шаги 1.1–1.3), действия студентов в рамках учебного модуля из трех экспериментов, включающего ПЦР для получения линейных фрагментов (шаги 2.1–2.7), сборку, преобразование и отбор клонов Гибсона (шаги 3.1–3.5), выделение плазмид и скрининг (этапы 4.1–4.4) и оценку результатов обучения (5.1–5.2). Подготовка инструктора описывает один из типичных примеров дизайна праймера и подготовки желаемой плазмидной карты in silico. Все разделы протокола могут быть адаптированы для других желаемых плазмид. Каждый студенческий эксперимент разделен на две 3-часовые лабораторные встречи.
1. Подготовка инструктора
- Проектирование грунтовки Gibson Assembly с помощью мастера сборки Benchling
- Определите источник матрицы ДНК для генных вставок, таких как геномная ДНК, плазмида и синтетическая ДНК.
ЗАМЕТКА: Для генов, которые будут амплифицированы из сложных геномных матриц ДНК, рекомендуется сначала определить уникальные сайты связывания праймеров в матрице геномной ДНК, специфичные для интересующего гена (генов), используя инструмент поиска базового локального выравнивания (BLAST) Национального центра биотехнологической информации (NCBI)21, как подробно описано в Дополнительном файле 1. Для простых шаблонов, таких как плазмиды или синтетическая ДНК, перейдите к следующему шагу. - Извлечение последовательностей ДНК генной вставки (вставок) и вектора (например, см. последовательность гена поликетидгидроксилазы и последовательность pUC19, предоставленную в виде файлов GenBank в Дополнительном файле 8). Импортируйте требуемые последовательности вставки и требуемую векторную последовательность в Benchling, каждую в виде нового файла последовательности ДНК. Откройте каждую импортированную последовательность, которая будет включена в реакцию сборки Гибсона.
ЗАМЕТКА: Файлы последовательностей ДНК можно импортировать в нескольких форматах, включая GenBank и FASTA. Можно также импортировать последовательность непосредственно из базы данных с номером доступа или вставить нуклеотидную последовательность, скопированную из другого файла или просмотрщика. - В нижней части экрана найдите инструмент «Мастер сборки ». Нажмите «Мастер сборок», затем выберите «Создать новую сборку». Из предложенных вариантов выберите Gibson и нажмите кнопку «Пуск », чтобы начать сборку.
- В окне векторной последовательности выберите все основания, которые должны быть включены в сборку, из векторной основы. Начните отбор с нуклеотидной позиции на 3'- конце генной вставки (например, в позиции 657 pUC19) и выберите все оставшиеся нуклеотиды для включения (например, через позицию 656 для включения всей последовательности pUC19). После выбора перейдите на вкладку «Магистраль » в нижней части экрана| «Установить фрагмент».
ЗАМЕТКА: Переключайтесь между видами карты последовательности и плазмиды вектора, чтобы выбрать все нужные основания круглого шаблона. В качестве альтернативы, если вы планируете получить фрагмент вектора с помощью рестрикционного дайджеста (вместо ПЦР) для сборки Gibson, линеаризуйте последовательность основного скелета с помощью сайта среза фермента рестрикции (например, XbaI). Для этого щелкните место разреза, удерживайте клавишу Shift и щелкните место выреза еще раз. Затем нажмите Set Fragment , чтобы задать основу. Имейте в виду, что векторно-специфичные праймеры не будут генерироваться методом Benchling, если основная цепь линеаризована ферментом рестрикции в Assembly Wizard. - В окне последовательности вставок выберите все основания вставки, которые должны быть включены в сборку. После выбора нажмите на вкладку «Вставка » в нижней части экрана | Набор фрагментов.
- В случае нескольких генных вставок нажмите кнопку + в правой части мастера сборки. В окне последовательности вставок выберите все основания вставки, которые должны быть включены в сборку. После выбора нажмите на вкладку «Вставка » в нижней части экрана | Набор фрагментов.
- После того, как все фрагменты будут установлены, переименуйте сборку в нужное имя плазмида и нажмите кнопку Собрать справа от мастера сборки. Выберите нужное расположение папки для папки Sequence Folder и Primer Folder. Нажмите кнопку Создать , чтобы собрать рекомбинантную плазмидную последовательность.
- Откройте собранный плазмид и нажмите кнопку История сборки , чтобы просмотреть базовую карту плазмиды и обзор последовательностей, из которых она была получена. Убедитесь, что положения нуклеотидов для каждого фрагмента соответствуют желаемой конструкции.
- На вкладке "Параметры сборки " просмотрите названия грунтовок, предназначенных для сборки. Обратите внимание, что для каждой вставки будет разработано по два праймера (прямой и обратный), а названия праймеров будут производиться из названий файлов последовательностей ДНК. Также убедитесь, что температуры плавления грунтовки и рекомендуемые температуры отжига совместимы в этом окне (см . Дополнительный файл 8, Репрезентативные грунтовки).
- Если температуры плавления праймера или температуры отжига не идеальны, вручную отрегулируйте последовательности праймера (например, удаляя нуклеотиды для снижения температуры плавления) в файлах последовательностей праймера. В качестве альтернативы нажмите « Открыть повторно » и отрегулируйте параметры праймера, нажав кнопку «Настройки фермента/праймера» рядом с кнопкой «Собрать » в мастере сборки. Затем повторите шаг сборки и повторно изучите последовательности грунтовок.
- Если библиотека плазмид должна быть клонирована с большим количеством различных генов, клонированных в одну и ту же векторную основу,
- В каждом случае используйте одну и ту же пару праймеров для вектора. Для этого вручную удалите все нуклеотиды из векторных праймеров, которые установят выступы, характерные для вставки. Эти нуклеотиды будут обнаружены на 5'- конце праймера. После снятия убедитесь, что температуры плавления грунтовки и температуры отжига совместимы (см . Дополнительный файл 8, Репрезентативные грунтовки).
- Убедитесь, что все пары капсюлей пластин имеют очень схожие температуры плавления и отжига.
ЗАМЕТКА: Это снизит потребность в оптимизации температуры отжига для каждой пары и позволит одновременно повторять все студенческие ПЦР-реакции с использованием одной и той же программы термоамплификатора.
- После того, как проектирование грунтовки для желаемых плазмид будет завершено, нажмите кнопку «Завершить » в мастере сборки. Закажите праймеры в компании, занимающейся синтезом ДНК, и получите образцы матриц ДНК для вставок и векторов.
- Протестируйте вставные и векторно-специфичные пары праймеров для достижения оптимальных температур отжига с помощью эксперимента градиентной ПЦР с матричками ДНК. Первоначально испытайте рекомендованные Benchling температуры отжига ± 3 °C.
- После определения температур отжига создать аликвоты растворов праймеров, матрицы ДНК и необходимые реагенты для ПЦР-реакций для студентов.
- Определите источник матрицы ДНК для генных вставок, таких как геномная ДНК, плазмида и синтетическая ДНК.
- Перед началом экспериментов раздайте учащимся рабочие листы для поддержки подготовки и оценки понимания (Дополнительный файл 4, Дополнительный файл 5 и Дополнительный раздел 6).
- Сбор данных о результатах обучения
ЗАМЕТКА: Если вы заинтересованы в сборе данных о результатах обучения из модуля Gibson Assembly и публикации результатов, получите одобрение на проведение исследований с участием людей в соответствии с политикой учреждения.- Предложите учащимся принять участие в исследовании результатов обучения не менее чем за 1 неделю до начала независимого проекта Gibson Assembly.
- Попросите студентов-участников заполнить анкету с несколькими вариантами ответов (Дополнительный файл 6) на лабораторной встрече до начала самостоятельного проекта.
2. Студенческий эксперимент А: ПЦР для получения линейных фрагментов
- Получите праймеры и шаблонные растворы ДНК для ПЦР у инструктора.
- Пипетка 25 мкл реакции, включающие ДНК-полимеразу MasterMix, ДНК-матрицу, прямые и обратные праймеры и безнуклеазную воду в соответствии с инструкциями в руководстве для учащихся (см. Дополнительный файл 2).
- Проведите реакцию в термоамплификаторе в соответствии с руководством для учащихся в Дополнительном файле 2. Убедитесь, что температура отжига реакции соответствует длине грунтовки, а время продления соответствует длине желаемой ампликона. Подробные инструкции см. в Дополнительном файле 2 и Дополнительном файле 3 .
- Храните ПЦР-реакции при температуре 4 °C или −20 °C до следующего лабораторного периода.
- Проанализируйте 5 мкл каждой реакции с помощью электрофореза в агарозном геле. Пока гель работает, добавьте 1 мкл фермента рестрикции DpnI в каждую реакцию, в которой в качестве матрицы использовалась плазмидная ДНК. Выдерживайте эту смесь в течение 1 часа при температуре 37 °C.
ЗАМЕТКА: DpnI разрушает любую оставшуюся кольцевую матрицу плазмидной ДНК, что снижает вероятность получения ложноположительных результатов на этапе трансформации. - Визуализируйте гель, чтобы подтвердить, что ПЦР прошла успешно и была достигнута правильная амплификация. Очистите любые успешные ПЦР-реакции с помощью коммерчески доступного набора для очистки ПЦР. Измерьте концентрацию (нг/л) очищенного продукта ПЦР с помощью микрообъемного спектрофотометра для использования в последующих сборочных расчетах Gibson.
- Перед следующим занятием разработайте рецепт реакции для сборки Gibson (см. Дополнительный файл 2 и Дополнительный файл 4).
3. Студенческий эксперимент Б: Сборка Гибсона, трансформация и отбор клонов
- Пипеткой проведите реакцию сборки Гибсона в соответствии с разработанным рецептом. Инкубировать реакцию при 50 °C в течение 15 минут. Пока реакции инкубируются, подготовьтесь к трансформации путем размораживания химически компетентных клеток кишечной палочки на льду.
- Преобразуйте 2 мкл сборки Гибсона в химически компетентные ячейки с помощью теплового шока. Пипеткой нанесите 100 мкл трансформированных клеток на две селекционные пластины и распределите стерилизованными шариками. Приготовьте 10-4, 10-5 и 10-6 разведений оставшихся клеток на агаре Лурия-Бертани (LB). Планшет 100 мкл каждого серийного разведения на LB (без отбора) и намазать стерилизованными шариками.
ЗАМЕТКА: На этом шаге можно рассчитать эффективность преобразования. - Инкубируйте пластины в течение ночи при температуре 37 °C. Храните пластины при температуре 4 °C до следующего урока.
- Подсчитайте колонии на всех пластинах и рассчитайте эффективность преобразования в соответствии с инструкциями в Дополнительном файле 2. Повторно сгруппируйте одну селективную пластину с четырьмя колониями с каждой селективной пластины и инкубируйте эту пластину при температуре 37 °C в течение ночи.
ЗАМЕТКА: Это служит резервной культурой ваших позитивных клонов. - С помощью маркера выделите, обведите и обозначьте четыре отдельные колонии на выборочной пластине инициалами учащегося и номером (например, ABC1). Возьмите новую пластину с LB-агаром, содержащую антибиотик для выбора, используйте маркер, чтобы разделить пластину на квадранты (т.е. четверти), а затем используйте ~1/2 каждой колонии, чтобы повторно нанести полосу на соответственно помеченный квадрант, и другую 1/2, чтобы инокулировать 5 мл жидкой культуры LB для каждой из четырех выбранных колоний. Обязательно пометьте пробирки соответствующей колонией (например, ABC1) и добавьте правильную концентрацию антибиотика для выбора. Инкубировать жидкие культуры в встряхивающем инкубаторе при 37 °С в течение ночи и в агаровой тарелке в статическом инкубаторе при 37 °С; хранить жидкие культуры и планшеты при температуре 4 °C до следующего периода занятий.
4. Студенческий эксперимент В: Выделение плазмид и скрининг
- Выделите плазмидную ДНК из жидких культур из эксперимента B с помощью набора miniprep. Измерьте концентрацию выделенной плазмиды (в нг/мкл).
- Разработайте дайджест рестрикции или ПЦР-скрининг для анализа выделенных плазмид (см. подробные инструкции в Дополнительном файле 2 и сообщите об ожиданиях в Дополнительном файле 5).
- Пипетка для рестрикции переваривания или реакции в соответствии с разработанными рецептами и инкубация при температурах и продолжительности, рассчитанных ранее.
- Анализируйте результаты с помощью электрофореза в агарозном геле.
5. Оценка
- Предложите студентам-участникам заполнить анкету с несколькими вариантами ответов (Дополнительный файл 7) на лабораторной встрече после завершения модуля Gibson Assembly.
- Объедините все данные из ответов до и после анкеты для анализа.
- Изучите статистическую значимость увеличения среднего балла по каждому отдельному содержательному вопросу между анкетами с использованием двухстороннего парного t-критерия и величины d-эффекта Коэна.
- Оцените повышение уверенности учащихся для каждого отдельного семестра и методики, чтобы определить степень изменения и его статистическую значимость.
- Определите влияние студенческого происхождения и академических факторов на обучение учащихся с помощью нормализованного прироста знаний (NLG) и непарных t-критериев.
- Оцените отношение учащихся к проектному опыту и карьере в области молекулярной биологии, оценивая средние баллы.
Результаты
Успехи учащихся в клонировании
В каждой итерации модуля Gibson Assembly (2019, 2022 и 2023 гг.) студентам предлагалось подготовить отчет, обобщающий их выводы. В 2019 году 36 из 44 студентов (81,8%) сообщили, что они успешно клонировали свои плазмиды на основе результатов скринин...
Обсуждение
Здесь мы предлагаем корректируемый лабораторный проект в классе для студентов, который обучает студентов молекулярному клонированию с помощью Gibson Assembly в исследовательской среде, основанной на курсе. В общей сложности 28 новых плазмид были клонированы студентами бак?...
Раскрытие информации
Авторы заявляют, что у них нет конкурирующих финансовых интересов или других конфликтов интересов.
Благодарности
Авторы выражают благодарность Андреа Лаубшер за техническую поддержку, а также Майклу Блэку, Сэнди Клементу и Джавину Озе за полезные дискуссии о внедрении учебной лаборатории и оценке результатов обучения. Авторы выражают признательность всем студентам, принимавшим участие в исследовании результатов обучения в когортах 2019, 2022 и 2023 годов, а также студентам-исследователям Натану Куну и Ааюши Адеттивару, которые помогали в подготовке реагентов к внедрению учебной лаборатории. Авторы также выражают признательность за финансовую поддержку со стороны Фонда Уильяма и Линды Фрост, гранта Центра приложений в биотехнологии Chevron Biotechnology Applied Research Endowment Grant и Национального научного фонда (NSF-1708919 и NSF-2300890).
Материалы
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
Ссылки
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены