Method Article
Ingegnerizzazione non virale di cellule T umane primarie tramite integrazione mirata di giunzione terminale mediata da omologia di grandi modelli di DNA
In questo articolo
Riepilogo
Viene fornito un protocollo dettagliato per l'utilizzo della tecnologia CRISPR/Cas9 per ottenere un knock-in mirato altamente efficiente di grandi costrutti multicistronici in cellule T umane primarie attraverso la via di riparazione del DNA HMEJ (omologia-mediata da end joining). Le cellule T ingegnerizzate con questo protocollo adattabile alle cGMP mantengono un'eccellente espansione cellulare, citotossicità e produzione di citochine.
Abstract
Molte attuali terapie cellulari adottive si basano su vettori lenti- o retrovirali per ingegnerizzare le cellule T per l'espressione di un recettore chimerico dell'antigene (CAR) o di un recettore esogeno delle cellule T (TCR) per colpire uno specifico antigene associato al tumore. La dipendenza da vettori virali per la produzione di cellule T terapeutiche aumenta significativamente la tempistica, i costi e la complessità della produzione, limitando al contempo la traduzione di nuove terapie, in particolare in ambito accademico. Viene presentato un processo per l'ingegneria non virale efficiente delle cellule T utilizzando CRISPR/Cas9 e l'unione delle estremità mediata dall'omologia per ottenere un'integrazione mirata di un grande carico di DNA multicistronico. Questo approccio ha raggiunto frequenze di integrazione paragonabili a quelle dei vettori virali, producendo al contempo cellule T altamente funzionali in grado di esercitare una potente efficacia antitumorale sia in vitro che in vivo. In particolare, questo metodo è rapidamente adattabile alle attuali buone pratiche di fabbricazione (cGMP) e allo scale-up clinico, fornendo un'opzione a breve termine per la produzione di cellule T terapeutiche da utilizzare negli studi clinici.
Introduzione
Le cellule T sono un componente chiave del sistema immunitario adattativo, possedendo capacità citolitica diretta, la capacità di modulare la risposta immunitaria attraverso la produzione di citochine, la licenza delle cellule B e delle cellule dendritiche e la creazione della memoria immunologica1. Svolgono un ruolo fondamentale nello sviluppo immunitario, nell'omeostasi e nella sorveglianza, nella protezione dagli agenti patogeni e nella prevenzione e difesa dal cancro, nonché nell'allergia e nell'autoimmunità1. Le cellule T possiedono un'enorme diversità di recettori delle cellule T (TCR) che vengono generati attraverso la ricombinazione V(D)J, consentendo alle cellule T di riconoscere una vasta gamma di antigeni e di montare risposte immunitarie efficaci contro vari agenti patogeni 1,2. Le cellule T possono essere generalmente classificate in due categorie, le cellule T CD4, note anche come cellule T helper, che aiutano principalmente altre cellule immunitarie, come le cellule B, a coordinare la risposta immunitaria, e le cellule T CD8, o cellule T citotossiche, che uccidono direttamente le cellule infette o cancerose riconoscendo antigeni specifici presentati sulla loro superficie1.
Lo sviluppo dei recettori chimerici dell'antigene (CAR) ha portato a un massiccio aumento dell'interesse per l'ingegneria genomica delle cellule T per le immunoterapie. Le CAR sono proteine ingegnerizzate che fondono domini di legame dell'antigene derivati da anticorpi con domini di segnalazione delle cellule T, consentendo alle cellule T di identificare e colpire le cellule che esprimono l'epitopo specifico riconosciuto dalla porzione anticorpale della CAR3. Questi recettori sono stati utilizzati per una varietà di immunoterapie, tra cui le malattie infettive e l'autoimmunità, ma la tecnologia è più avanzata per le immunoterapie contro il cancro.
Le cellule CAR-T hanno avuto un enorme successo nel trattamento di leucemie e linfomi, ma hanno mostrato un'efficacia limitata per il trattamento dei tumori solidi 4,5. Ciò ha portato a un'ondata di ulteriori sviluppi volti a migliorare l'efficacia delle cellule CAR-T per le indicazioni dei tumori solidi. Sono stati sviluppati diversi approcci, tra cui l'armatura delle citochine, il knockout genico del checkpoint, i recettori negativi dominanti, i recettori delle chemochine, l'espressione di più CAR in una cellula, la modifica del CAR per migliorare la segnalazione intracellulare e l'integrazione in loci predeterminati, ad esempio il locus TRAC, per sfruttare i meccanismi di regolazione dell'ospite per prevenire l'esaurimento 6,7,8. Molti di questi approcci richiedono un carico genetico più ampio e/o un'integrazione sito-specifica. Approcci alternativi includono anche l'uso di TCR transgenici per consentire alle cellule T di colpire i neoantigeni intracellulari 9,10. Tuttavia, questo ha lo svantaggio significativo di richiedere che il TCR abbia specificità sia per l'epitopo del neoantigene che per la molecola HLA, limitando l'uso dell'eventuale prodotto terapeutico ai pazienti che esprimono l'HLA affine. Inoltre, molti tumori alterano o riducono l'espressione di HLA in risposta all'immunoterapia, riducendo notevolmente l'efficacia delle cellule T che esprimono TCR transgenici11.
La maggior parte delle terapie cellulari CAR-T o TCR-T negli studi clinici sono prodotte utilizzando vettori retrovirali, come lentivirus o gammaretrovirus, raggiungendo un'elevata frequenza di integrazione con carichi di dimensioni moderate. Tuttavia, i vettori virali soffrono di lunghi tempi di produzione a causa degli attuali requisiti delle buone pratiche di fabbricazione (cGMP) e di profili di integrazione non specifici che creano un rischio di mutagenesi inserzionale12,13. Inoltre, può essere difficile produrre retrovirus transgenico ad alti titoli se il carico supera i 5 kb14. Altri vettori, come quelli derivati dal virus adeno-associato ricombinante (rAAV), non si integrano naturalmente ma possono trasportare il modello del donatore di DNA verso il nucleo e possono essere utilizzati in combinazione con CRISPR/Cas9 per facilitare l'ingegneria genomica mediata dalla ricombinazione diretta dall'omologia (HDR). Tuttavia, questi virus hanno anche flussi di lavoro di produzione lunghi e complicati e sono limitati dalle dimensioni del carico (<4,7 kb) e dalla necessità di includere bracci di omologia lunghi (500-1000 bp)15,16,17,18.
L'ingegneria genomica non virale utilizzando trasposoni o una combinazione di nucleasi mirate e un modello di donatore di DNA è stata riportata nei linfociti umani primari 8,19,20. Tuttavia, questi approcci sono limitati dalla risposta tossica alle molecole di DNA nudo nel citoplasma in seguito al riconoscimento da parte di sensori di DNA citoplasmatico espressi nei linfociti21. Sono stati fatti tentativi per utilizzare inibitori di piccole molecole di queste vie di rilevamento del DNA durante la trasfezione, ma la ridondanza di queste vie può complicare il loro uso nei protocolli cGMP22. In particolare, i vettori trasposoni, come la Bella Addormentata, PiggyBac e Tc Buster, consentono l'integrazione di un grande carico genetico con alte efficienze, ma hanno un profilo di integrazione non specifico23,24. L'integrazione transgenica mirata e non virale utilizzando modelli di DNA plasmidico, lineare o a singolo filamento in combinazione con una nucleasi mirata per HDR è un'alternativa interessante, ma è stata limitata dalla scarsa efficienza, soprattutto con carichi genetici sempre più grandi, con un'efficienza inferiore al 10% riportata quando si utilizza un carico superiore a 1,5 kb 8,19.
Qui, presentiamo il protocollo passo-passo per l'inserimento non virale, di giunzione terminale mediata da omologia (HMEJ) di grandi carichi utili di DNA in cellule T umane primarie, come descritto in Webber, Johnson et al.25. HMEJ utilizza bracci di omologia corti da 48 bp affiancati da siti bersaglio di gRNA Cas9 per consentire un'integrazione mirata ad alta efficacia di un grande carico di DNA rispetto all'HDR tradizionale. Un metodo per ridurre la citotossicità del DNA plasmidico nelle cellule T primarie consiste nell'impiegare plasmidi con dorsali ridotte al minimo, come i minicerchi o i nanoplasmidi25. I minicerchi sono vettori plasmidici miniaturizzati prodotti mediante escissione dell'origine della replicazione e del gene di resistenza agli antibiotici attraverso la ricombinazione dopo l'amplificazione plasmidica; hanno dimostrato di migliorare l'ingegneria non virale delle cellule T e di ridurre la tossicità cellulare23,24. I nanoplasmidi hanno anche una dimensione complessiva ridotta ottenuta attraverso l'uso di un'origine minima di replicazione e di un marcatore di selezione non tradizionale26. In base alla nostra esperienza, le piattaforme vettoriali di minicerchi e nanoplasmidi offrono un miglioramento comparabile in termini di efficienza e una ridotta tossicità rispetto ai plasmidi tradizionali25.
Qui, presentiamo un protocollo dettagliato che sinergizza l'ottimizzazione temporale della somministrazione e della composizione dei reagenti, nonché l'utilizzo di HMEJ e CRISPR/Cas9 per ottenere un'elevata efficacia e l'ingegneria genomica sito-specifica di cellule T umane primarie con modelli di DNA multicistronico di grandi dimensioni (>6,3 kb) per l'uso in immunoterapie e una varietà di altre applicazioni25. Raggiungiamo una maggiore integrazione con HMEJ e bracci di omologia a 48 bp rispetto all'HR tradizionale utilizzando bracci di omologia da 1 kb, in particolare con carichi genetici >1,5 kb25,27. È importante sottolineare che le cellule T ingegnerizzate attraverso la riparazione HMEJ mantengono un'eccellente espansione cellulare, citotossicità e produzione di citochine, pur mantenendo un fenotipo25 non esaurito. Questo protocollo è facilmente adattabile agli standard cGMP ed è scalabile per un numero di cellule clinicamente rilevante, consentendo una rapida transizione all'uso futuro in una varietà di studi clinici25.
Protocollo
Tutti gli esperimenti sono stati eseguiti con precauzioni universali per i patogeni trasmessi per via ematica, con tecnica sterile/asettica, dispositivi di protezione individuale e apparecchiature di biosicurezza di livello 2 (BSL2). Tutti gli esperimenti qui descritti sono stati approvati dall'Institutional Biosafety Committee (IBC) dell'Università del Minnesota. I dettagli dei reagenti e delle attrezzature utilizzate in questo studio sono elencati nella Tabella dei materiali.
1. Preparazione dei terreni
- Preparare integratori per i terreni completi delle cellule T (MTC).
- Ricostituire l'IL-2 umana ricombinante a una concentrazione di 12000 UI/mL aggiungendo acido acetico 100 mM sterilizzato con filtro e mescolare bene mediante pipettaggio. Quindi, diluire ulteriormente a una concentrazione di 6000 UI/mL con albumina sierica bovina (BSA) sterilizzata con filtro allo 0,2% in 1x soluzione salina tamponata con fosfato (1x PBS) e mescolare bene mediante pipettaggio per ottenere una soluzione di conservazione prolungata.
- Ricostituire l'IL-7 umana ricombinante a una concentrazione di 200 ng/μL con acqua sterile e mescolare bene mediante pipettaggio. Quindi, diluire ulteriormente fino a una concentrazione di 100 ng/μL con BSA sterilizzato con filtro allo 0,2% in PBS e mescolare bene mediante pipettaggio per ottenere una soluzione di conservazione prolungata.
- Ricostituire l'IL-15 umana ricombinante a una concentrazione di 200 ng/μL con acqua sterile e mescolare bene mediante pipettaggio. Quindi, diluire ulteriormente fino a una concentrazione di 100 ng/μL con BSA sterilizzato con filtro allo 0,2% in PBS e mescolare bene mediante pipettaggio per ottenere una soluzione di conservazione prolungata.
NOTA: Conservare ciascuna citochina in piccole aliquote a una temperatura compresa tra -20 °C e -80 °C per un massimo di sei mesi ed evitare ripetuti cicli di congelamento/disgelo.
- Preparare i terreni TCM.
- Preparare il terreno basale aggiungendo il 2,6% di integratore per l'espansione delle cellule T e il 2,5% di sostituzione del siero delle cellule immunitarie al terreno basale di espansione delle cellule T. Per ulteriori informazioni, vedere la tabella dei materiali .
- Preparare la MTC aggiungendo l'1% di L-glutammina, l'1% di penicillina/streptomicina, 10 mM di N-acetil-L-cisteina, 300 UI/mL di IL-2 umana ricombinante, 5 ng/mL di IL-7 umana ricombinante e 5 ng/mL di IL-15 umana ricombinante ai terreni basali.
- Sterilizzare la MTC facendo passare i terreni attraverso un filtro da 0,22 μm in un flacone sterile.
- Mantenere la MTC a 4 °C per un massimo di 2 settimane.
- Preparare il terreno basale aggiungendo il 2,6% di integratore per l'espansione delle cellule T e il 2,5% di sostituzione del siero delle cellule immunitarie al terreno basale di espansione delle cellule T. Per ulteriori informazioni, vedere la tabella dei materiali .
- Preparare i supporti di ripristino.
- Preparare il terreno basale aggiungendo il 2,6% di integratore per l'espansione delle cellule T e il 2,5% di sostituzione del siero delle cellule immunitarie al terreno basale di espansione delle cellule T.
- Preparare i terreni di recupero aggiungendo l'1% di L-glutammina, 10 mM di N-acetil-L-cisteina, 300 UI/mL di IL-2 umana ricombinante, 5 ng/mL di IL-7 umana ricombinante, 5 ng/mL di IL-15 umana ricombinante ai terreni basali e 1 μg/mL di DNasi.
NOTA: Non aggiungere penicillina/streptomicina ai terreni di recupero, poiché può ridurre il recupero delle cellule post-elettroporazione. - Sterilizzare il terreno di recupero facendo passare il terreno attraverso un filtro da 0,22 μm in un flacone sterilizzato.
- Conservare i terreni di recupero a 4 °C per un massimo di 2 settimane.
- Preparare i terreni di recupero aggiungendo l'1% di L-glutammina, 10 mM di N-acetil-L-cisteina, 300 UI/mL di IL-2 umana ricombinante, 5 ng/mL di IL-7 umana ricombinante, 5 ng/mL di IL-15 umana ricombinante ai terreni basali e 1 μg/mL di DNasi.
- Preparare il terreno basale aggiungendo il 2,6% di integratore per l'espansione delle cellule T e il 2,5% di sostituzione del siero delle cellule immunitarie al terreno basale di espansione delle cellule T.
2. Selezione del sito e progettazione del modello
- Determinare una posizione di destinazione genomica per il knock-in.
NOTA: È possibile utilizzare una posizione di approdo sicuro, come AAVS1, se si desidera un impatto minimo sulla cella target. Un sito bersaglio che interrompe l'espressione genica può essere utilizzato per eliminare contemporaneamente il gene bersaglio e inserire il costrutto di interesse in un unico passaggio. Un sito bersaglio e un donatore progettati per integrarsi nel frame con un gene endogeno possono essere utilizzati per creare fusioni geniche o in combinazione con una sequenza di salto ribosomiale 2A per porre il costrutto sotto il controllo trascrizionale di un promotore endogeno (Figura 1). - Progettare il modello di carico in modo tale che la cassetta di espressione sia affiancata da bracci di omologia da 48 bp corrispondenti al sito di integrazione e affiancati da siti bersaglio di gRNA linearizzanti.
NOTA: Il gRNA linearizzante del plasmide può essere un gRNA28 universale (UgRNA) (GGGAGGCGUUCGGGCCACAG) progettato per colpire una sequenza (GGGAGGCGTTCGGGGCCACAG) non presente nel genoma umano o murino25, o la sequenza di gRNA bersaglio genomico, in modo tale che un singolo gRNA possa tagliare il sito di knock-in genomico e anche linearizzare il modello di carico (Figura 1). Sia i nanoplasmidi che i minicerchi possono essere utilizzati come vettori per il modello di carico. I plasmidi standard possono essere utilizzati al posto dei nanoplasmidi o dei minicerchi per alcune linee trasformate, ma si tradurranno in una riduzione del knock-in e dell'espansione cellulare nelle cellule primarie più sensibili come le cellule T.
3. Isolamento e attivazione delle cellule T
- Ottenere cellule T da un fornitore commerciale o isolare le cellule T dalle PBMC utilizzando la selezione immunomagnetica18.
- Determinare il numero di cellule T necessarie per l'esperimento, quindi scongelare, lavare e risospendere le cellule T a una concentrazione di 1 × 106 cellule per mL in MTC. Aggiungere 1 mL di questa miscela ai pozzetti di una piastra a 24 pozzetti.
- Agitare le perle di attivazione delle cellule T per risospendere le perle e aggiungere le perle in un rapporto di 2:1 perline: cella a ciascun pozzetto. Incubare queste cellule per 36 ore in un incubatore a 37 °C e 5% di CO2 prima di iniziare l'ingegneria delle cellule T.
NOTA: Un tempo di incubazione inferiore o superiore a 36 ore dopo l'attivazione ridurrà l'efficienza, la vitalità e l'espansione cellulare post-ingegnerizzazionedelle cellule T 25.
- Agitare le perle di attivazione delle cellule T per risospendere le perle e aggiungere le perle in un rapporto di 2:1 perline: cella a ciascun pozzetto. Incubare queste cellule per 36 ore in un incubatore a 37 °C e 5% di CO2 prima di iniziare l'ingegneria delle cellule T.
4. Ingegneria delle cellule T
- Determinare le condizioni sperimentali necessarie per completare l'esperimento, compresi i controlli sperimentali.
NOTA: È importante includere una condizione solo plasmidico come controllo dell'espressione episomiale. Una condizione di elettroporazione con il sito bersaglio chimicamente modificato gRNA28 e/o l'mRNA Cas9 in assenza di un donatore di plasmidi può anche essere utilizzata come controllo negativo. - Preparare una piastra di recupero.
- Per ogni condizione sperimentale, compresi i controlli sperimentali, aggiungere 300 μL di terreno di recupero a un pozzetto di una piastra di coltura tissutale a 24 pozzetti e riscaldare la piastra in un incubatore a 37 °C e CO2 al 5%.
NOTA: Vedere la Tabella 1 per i numeri di cella, il volume di recupero e le condizioni alternativi.
- Per ogni condizione sperimentale, compresi i controlli sperimentali, aggiungere 300 μL di terreno di recupero a un pozzetto di una piastra di coltura tissutale a 24 pozzetti e riscaldare la piastra in un incubatore a 37 °C e CO2 al 5%.
- Preparare le cellule T.
- Raccogliere la miscela di perle di attivazione stimolata tra cellule T e cellule T, contare le cellule vitali e risospendere le perle di attivazione tra cellule T e cellule T a una concentrazione di 1-5 × 106 cellule/mL in un mezzo di microcentrifuga da 1,5 mL.
- Posizionare la provetta da microcentrifuga in un magnete (vedere la Tabella dei materiali) e lasciarla incubare per 3 minuti.
NOTA: La rimozione del cordone è necessaria se si utilizzano perle di attivazione delle cellule T per attivare le cellule T. La rimozione del tallone potrebbe non essere necessaria se viene utilizzato un metodo alternativo di attivazione. - Dopo l'incubazione e senza rimuoverli dal magnete, trasferire i terreni e le cellule T in una nuova provetta. Questo tubo contiene cellule T stimolate con le perle di attivazione delle cellule T rimosse. Contare le cellule T e metterle in provette coniche da 14 mL o 50 mL. Rabboccare la provetta con 1× PBS e conservare le cellule in un incubatore a 37 °C e 5% di CO2 fino al momento della centrifugazione.
- Preparare la miscela di cellule T.
- Determinare la quantità di Master Mix Buffer (82% di soluzione di cella primaria e 18% di supplemento) necessaria moltiplicando il numero di condizioni di elettroporazione più uno (n + 1). (ad es. 6 condizioni di elettroporazione pianificate + 1 = 7,7 × 18 μL per condizione = 126 μL di Master Mix Buffer).
NOTA: Questi volumi sono specifici per il sistema di elettroporazione 4D utilizzato in questo studio (vedi Tabella dei materiali). Altri sistemi di elettroporazione richiedono volumi e concentrazioni diverse. Volume della soluzione della cella primaria = Volume totale del tampone Master Mix necessario × 0,82 e Volume del supplemento = Volume totale del Master Mix Buffer necessario x 0,18. (ad es. 0,82 × 126 μL = 103,32 μL di soluzione di cella primaria e 0,18 × 126 μL = 22,68 μL). - Mescolare la soluzione di cella primaria e l'integratore insieme per creare il tampone Master Mix.
- Centrifugare le cellule T in 1x PBS per 10 minuti a 200 × g (a temperatura ambiente) e aspirare il surnatante.
- Risospendere le cellule T nel Master Mix Buffer a una concentrazione di 1 × 106 cellule per 18 μL di Master Mix Buffer.
- Determinare la quantità di Master Mix Buffer (82% di soluzione di cella primaria e 18% di supplemento) necessaria moltiplicando il numero di condizioni di elettroporazione più uno (n + 1). (ad es. 6 condizioni di elettroporazione pianificate + 1 = 7,7 × 18 μL per condizione = 126 μL di Master Mix Buffer).
- Preparare i reagenti.
- Utilizzando le concentrazioni delle soluzioni stock, determinare il volume del plasmide HMEJ, il gRNA del sito target, il gRNA di linearizzazione del plasmide (UgRNA) e l'mRNA di Cas9 necessari per raggiungere la massa indicata per elettroporazione (Tabella 2).
- Aggiungere i reagenti a un pozzetto di una piastra a 96 pozzetti su ghiaccio per ogni condizione.
- Preparare la miscela per elettroporazione.
- Se necessario, aggiungere 18 μl di miscela di cellule T a ciascun pozzetto della piastra a 96 pozzetti.
- Se necessario, aggiungere un ulteriore tampone Master Mix per garantire che tutte le condizioni di elettroporazione abbiano un volume totale finale di 22 μL.
NOTA: Il volume di 22 μl include un volume in eccesso del 10% per tenere conto della potenziale perdita di pipettaggio.
- Eseguire l'elettroporazione.
- Accendere l'apparecchiatura e caricare il programma.
- Determinare il numero di cuvette necessarie e aprire il numero richiesto di cuvette. Segnare un'estremità della cuvetta e del tappo e rimuovere il tappo dalla cuvetta.
NOTA: Ci sono 16 pozzetti per cuvetta per reazioni da 20 μL. - Pipettare delicatamente 20 μl della miscela per elettroporazione dai pozzetti della piastra a 96 pozzetti su e giù 1-2 volte e caricare in cuvette.
- Richiudere la cuvetta utilizzando la marcatura effettuata al punto 4.7.2 per evitare di mettere il tappo all'indietro. Toccare la cuvetta 3-5 volte per rimuovere potenziali bolle d'aria, quindi posizionare l'attrezzatura e elettroporare la cuvetta.
- Rimuovere delicatamente la cuvetta dall'apparecchiatura, posizionarla nella cappa e lasciare riposare le celle per 15 minuti a temperatura ambiente.
- Dopo il riposo, estrarre 80 μl di terreno di recupero riscaldato dalla piastra di recupero e aggiungerli a ciascun campione (aggiungere sul lato della cuvetta, non direttamente sulle cellule), pipettare molto delicatamente su e giù una volta e trasferire il volume totale di 100 μl nella piastra di recupero.
NOTA: Le cellule sono molto fragili dopo l'elettroporazione. Una manipolazione delicata è di fondamentale importanza. - Incubare le cellule a 37 °C e 5% di CO2 per 30 minuti.
- Aggiungere la MTC per diluire le cellule a una concentrazione di 1 × 106 cellule/mL. Sostituire metà del terreno il giorno successivo e poi ogni 3-4 giorni con MTC con concentrazioni di citochine di 2× per tutta la durata dell'esperimento.
- Dopo aver diluito a 1 × 106 cellule/mL, restimolare le cellule aggiungendo perle di attivazione delle cellule T in un rapporto di 0,5:1: cellula a ciascun pozzetto.
NOTA: Le celle potrebbero espandersi rapidamente. Potrebbe essere necessario eseguire le modifiche del mezzo e spostare le cellule in pozzetti più grandi. - Il giorno 3, rimuovere le perle di attivazione delle cellule T.
- Dopo la durata della crescita desiderata, raccogliere le cellule.
NOTA: Se nella cuvetta sono presenti pozzetti inutilizzati, è possibile contrassegnare i pozzetti utilizzati e l'intera cuvetta può essere collocata in un conico da 50 ml e conservata a 4 °C per un uso futuro.
Risultati
Qui, un modello multicistronico di grandi dimensioni (>6,3 kb) chiamato costrutto "Giant Minicircle" è stato integrato nel locus TRAC nelle cellule T umane primarie utilizzando l'editing CRISPR/Cas9; un gRNA specifico per TRAC (TCTCTCAGCTGGTACACGGC), l'UgRNA e il nanoplasmide HMEJ (Figura 2A) con una condizione priva di gRNA specifico per TRAC, il gRNA universale e l'mRNA Cas9 utilizzati come controllo negativo. I campioni che includono il costrutto Giant Minicircle, il gRNA specifico per TRAC, il gRNA universale e l'mRNA Cas9 hanno avuto un tasso medio di knock-in del 23,35% quando hanno misurato l'espressione di GFP (23,5% ± 5,247), mentre il controllo negativo non ha mostrato alcuna espressione di GFP (Figura 2B). Non ci sono state differenze significative nell'espansione del fold (Figura 2C) e nella vitalità (Figura 2D) tra le condizioni sperimentali. Questi risultati dimostrano un knock-in ad alta efficienza di un modello molto grande, pur mantenendo un'eccellente vitalità ed espansione cellulare.

Figura 1: Rappresentazione schematica del progetto del costrutto HMEJ. Clicca qui per visualizzare una versione più grande di questa figura.
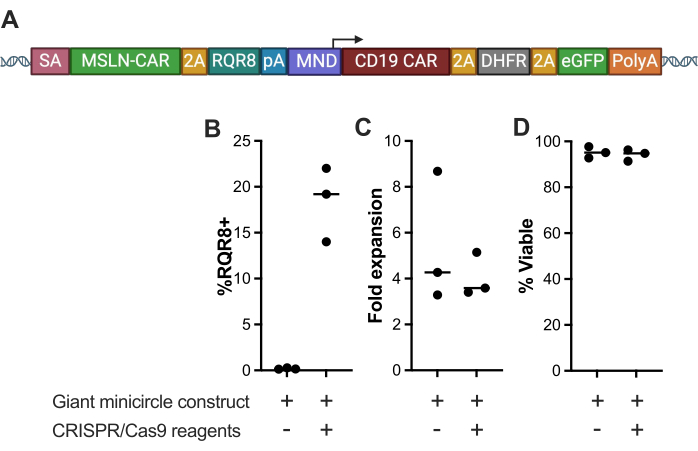
Figura 2: Caratterizzazione di cellule T elettroporate con il costrutto del minicerchio gigante. (A) Rappresentazione schematica del costrutto Giant Minicircle, un grande modello multicistronico (>6.3 kb) che codifica per un CAR anti-mesotelina e RQR8 sotto il promotore TRAC con un linker GSG, un CAR anti-CD19 e una muteina DHFR ed eGFP sotto il promotore MND. (B) percentuale di espressione di RQR8, (C) espansione del fold e (D) vitalità delle cellule T nove giorni dopo l'elettroporazione con il costrutto Giant Minicircle e i reagenti CRISPR-Cas9, rispetto a un controllo negativo elettroporato con il Giant Minicircle privo di reagenti CRISPR-Cas9. (***p < 0,001). Clicca qui per visualizzare una versione più grande di questa figura.
| Dimensione della piastra | Volume della cuvetta | Celle/Pozzetto | Volume dei supporti di ripristino | Volume di 1x TCM con NAC | Volume finale totale |
| Piastra a 24 pozzetti | 22/22 μL | 1-3 x 106 | 300 μl | 680 μl | 1 ml |
| Piastra a 6 pozzetti | 100/110 μL | 4-20 x 106 | 1 ml | 2,9 ml | 4 ml |
| Piastra G-Rex a 24 pozzetti | 20-110 μl | 1-20 x 106 | 400 μl | 5,6 ml | 6 ml |
Tabella 1: Concentrazioni cellulari per cuvette e piastre di recupero.
| Dimensione cuvetta | Plasmide modello | Sito bersaglio gRNA | GRNA di linearizzazione plasmidica | mRNA Cas9 |
| 20 μl | 1-2 μg (1 μg) | 1-3 μg (1 μg) | 1-3 μg (1 μg) | 1-3 μg (1,5 μg) |
| 100 μl | 5-10 μg (5 μg) | 5-15 μg (5 μg) | 5-15 μg (5 μg) | 5-15 μg (5 μg) |
Tabella 2: Quantità di reagenti CRISPR/Cas9 e modello di DNA necessari.
Discussione
Con la continua evoluzione della terapia cellulare adottiva (ACT), vi è una crescente domanda di metodi efficienti e non virali per ingegnerizzare le cellule immunitarie senza i costi elevati e la complessità associati ai vettori basati su virus. Un obiettivo chiave in questo settore è il raggiungimento di un'integrazione sito-specifica, che migliora la coerenza, la sicurezza e la funzione dei prodotti cellulari ingegnerizzati. Mentre studi recenti hanno dimostrato il successo dell'integrazione non virale di piccoli costrutti genetici, come i geni reporter e le singole sequenze CAR o TCR29, è necessario estendere questi metodi a cassette di espressione multigenica più grandi. Queste cassette più grandi sono necessarie per migliorare la funzione delle cellule immunitarie, come l'aggiunta di recettori per le chemochine o l'armatura delle citochine, migliorare la specificità (ad esempio, i sistemi di porte logiche) o aumentare la sicurezza con i kill switch. A tal fine, abbiamo cercato di sviluppare approcci non virali in grado di integrare in modo efficiente il carico genetico sito-specifico25.
Ci sono diversi passaggi critici in tutto il protocollo. Per ridurre al minimo la tossicità, è necessario un nanoplasmide o un minicerchio pulito e di alta qualità. In questo studio, i plasmidi preparati in commercio hanno ottenuto i risultati migliori. Anche il periodo di 36 ore tra l'attivazione delle cellule T e l'elettroporazione è fondamentale per ottenere risultati ideali. È anche importante ridurre al minimo la manipolazione e la manipolazione delle cellule subito dopo l'elettroporazione, mentre le cellule si riprendono. È anche importante aggiungere nuovamente nuove perle di attivazione delle cellule T alla coltura dopo l'elettroporazione per ottenere la migliore frequenza di integrazione del carico e l'espansione cellulare più ottimali.
Il protocollo, come descritto qui, utilizza un sistema di elettroporazione disponibile in commercio (vedi Tabella dei materiali). Anche altri sistemi di elettroporazione possono essere adatti, ma probabilmente richiederanno l'ottimizzazione delle condizioni di elettroporazione specifiche del dispositivo e possibilmente la gestione delle celle post-elettroporazione.
Ci sono diverse limitazioni all'ingegnerizzazione mediata da HMEJ delle cellule T umane. Il protocollo può funzionare con i plasmidi, ma i risultati ottimali richiedono l'uso di nanoplasmidi o minicerchi per la consegna delle merci, che non sono facili da produrre come i plasmidi standard. Inoltre, sebbene sia stata dimostrata l'integrazione di successo di una cassetta multicistronica molto grande, è probabile che ci sia un limite di carico superiore in cui la frequenza di integrazione genica inizierà a diminuire.
Questo studio presenta l'HMEJ come un metodo di ingegneria genomica non virale potente ed efficiente per la produzione di cellule T ingegnerizzate, in particolare nel contesto dell'immunoterapia del cancro. Superando i limiti dei vettori virali tradizionali, questo approccio offre un'alternativa economica, scalabile e più sicura per la generazione di cellule T geneticamente modificate con funzionalità avanzate. La capacità di integrare con precisione cassette multigeniche di grandi dimensioni apre nuove possibilità per l'ingegnerizzazione delle cellule immunitarie per il trattamento di un'ampia gamma di malattie, tra cui tumori, malattie infettive e malattie autoimmuni. Inoltre, la compatibilità del metodo HMEJ con i processi di produzione di livello clinico ne garantisce l'applicabilità pratica in contesti terapeutici del mondo reale, aprendo la strada a terapie cellulari più accessibili ed efficienti nel prossimo futuro.
Divulgazioni
B.R.W. e B.S.M. sono i principali ricercatori degli accordi di ricerca sponsorizzati finanziati da Intima Biosciences per supportare il lavoro in questo manoscritto. Sono stati depositati brevetti che coprono i metodi e gli approcci delineati in questo manoscritto.
Riconoscimenti
BRW riconosce i finanziamenti dell'Office of Discovery and Translation, le sovvenzioni NIH R21CA237789, R21AI163731, P01CA254849, P50CA136393, U54CA268069, R01AI146009, le sovvenzioni DOD HT9425-24-1-1005, HT9425-24-1-1002, HT9425-24-1-0231 e, il Children's Cancer Research Fund, il Fanconi Anemia Research Fund e il Randy Shaver Cancer and Community Fund. BSM riconosce i finanziamenti dell'Office of Discovery and Translation, le sovvenzioni NIH R01AI146009, R01AI161017, P01CA254849, P50CA136393, U24OD026641, U54CA232561, P30CA077598, U54 CA268069, le sovvenzioni DOD HT9425-24-1-1005, HT9425-24-1-1002, HT9425-24-1-0231 e il Children's Cancer Research Fund, il Fanconi Anemia Research Fund e il Randy Shaver Cancer and Community Fund.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetic Acid | Millipore Sigma | A6283 | |
| 1x DPBS, no calcium, no magnesium | Thermo Fisher Scientific | 14190144 | |
| 2.5% CTS Immune Cell Serum Replacement | Thermo Fisher Scientific | A2596101 | |
| Amaxa P3 Primary Cell 4D-Nucleofactor X Kit L | Lonza | V4XP-3024 | |
| Amaxa P3 Primary Cell 4D-Nucleofactor X Kit S | Lonza | V4XP-3032 | |
| Bovine Serum Albumin | Thermo Fisher Scientific | 15561020 | |
| Chemically Modified Guide RNAs | Integrated DNA Technologies | na | Custom design |
| CleanCap Cas9 mRNA | Trilink | L-7206 | |
| CTS OpTmizer T cell Expansion Media SFM +OpTmizer T cell Expansion Supplement | Thermo Fisher Scientific | A1048501 | |
| DNase I | Stem Cell Technologies | 07900 | |
| Dynabeads Human T-Activator CD3/CD28 | Thermo Fisher Scientific | 11141D | |
| DynaMag-2 | Thermo Fisher Scientific | 12321D | |
| Human IL15 | PeproTech | 200-15 | |
| Human IL2 | PeproTech | 200-02 | |
| Human IL7 | PeproTech | 200-07 | |
| L-Glutamine | Thermo Scientific | 25030081 | |
| Lonza 4D nucelofector Core | Lonza | AAF-1003B | |
| Lonza 4D nucelofector X Unit | Lonza | AAF-1003X | |
| Minicircle | System Biosciences | MN910A-1 | Custom design |
| N-Acetyl-L-cysteine | MiliporeSigma | A9165 | |
| Nanoplasmid | Aldevron | na | Custom design |
| Penicillin/Streptomycin | Thermo Fisher Scientific | 15-140-122 |
Riferimenti
- Sun, L., Su, Y., Jiao, A., Wang, X., Zhang, B. T cells in health and disease. Signal Transduct Target Ther. 8 (1), 235(2023).
- Takahama, Y. Journey through the thymus: stromal guides for T-cell development and selection. Nature reviews. Immunology. 6 (2), 127-135 (2006).
- Dotti, G., Gottschalk, S., Savoldo, B., Brenner, M. K. Design and development of therapies using chimeric antigen receptor-expressing T cells. Immunol Rev. 257 (1), 107-126 (2014).
- DeRenzo, C., Gottschalk, S. Genetic modification strategies to enhance CAR T cell persistence for patients with solid tumors. Front Immunol. 10, 218(2019).
- Liu, X., et al. A chimeric switch-receptor targeting PD1 augments the efficacy of second-generation CAR T cells in advanced solid tumors. Cancer Res. 76 (6), 1578-1590 (2016).
- Yeku, O. O., Purdon, T. J., Koneru, M., Spriggs, D., Brentjens, R. J. Armored CAR T cells enhance anti-tumor efficacy and overcome the tumor microenvironment. Sci Rep. 7 (1), 10541(2017).
- Schober, K., et al. Orthotopic replacement of T-cell receptor α- and β-chains with preservation of near-physiological T-cell function. Nat Biomed Eng. 3 (12), 974-984 (2019).
- Roth, T. L., et al. Reprogramming human T cell function and specificity with non-viral genome targeting. Nature. 559 (7714), 405-409 (2018).
- Chandran, S. S., Klebanoff, C. A. T cell receptor-based cancer immunotherapy: Emerging efficacy and pathways of resistance. Immunol Rev. 290 (1), 127-147 (2019).
- Rosenberg, S. A., Restifo, N. P. Adoptive cell transfer as personalized immunotherapy for human cancer. Science (New York, N.Y.). 348 (6230), 62-68 (2015).
- Campoli, M., Ferrone, S. HLA antigen and NK cell activating ligand expression in malignant cells: a story of loss or acquisition. Semin Immunopathol. 33 (4), 321-334 (2011).
- Walther, W., Stein, U. Viral vectors for gene transfer: a review of their use in the treatment of human diseases. Drugs. 60 (2), 249-271 (2000).
- Bulcha, J. T., Wang, Y., Ma, H., Tai, P. W. L., Gao, G. Viral vector platforms within the gene therapy landscape. Signal Transduct Target Ther. 6 (1), 53(2021).
- Kumar, M., Keller, B., Makalou, N., Sutton, R. E. Systematic determination of the packaging limit of lentiviral vectors. Hum Gene Ther. 12 (15), 1893-1905 (2001).
- Samulski, R. J., Muzyczka, N. AAV-mediated gene therapy for research and therapeutic purposes. Annu Rev Virol. 1 (1), 427-451 (2014).
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature reviews. Drug Discov. 18 (5), 358-378 (2019).
- Pomeroy, E. J., et al. a genetically engineered primary human natural killer cell platform for cancer immunotherapy. Mol Ther. 28 (1), 52-63 (2020).
- Johnson, M. J., Laoharawee, K., Lahr, W. S., Webber, B. R., Moriarity, B. S. Engineering of primary human B cells with CRISPR/Cas9 targeted nuclease. Sci Rep. 8 (1), 12144(2018).
- Nguyen, D. N., et al. Polymer-stabilized Cas9 nanoparticles and modified repair templates increase genome editing efficiency. Nat Biotechnol. 38 (1), 44-49 (2020).
- Lock, D., et al. Automated, scaled, transposon-based production of CAR T cells. J Immunother Cancer. 10 (9), e005189(2022).
- Semenova, N., et al. Multiple cytosolic DNA sensors bind plasmid DNA after transfection. Nucleic Acids Res. 47 (19), 10235-10246 (2019).
- Wimberger, S., et al. Simultaneous inhibition of DNA-PK and Polϴ improves integration efficiency and precision of genome editing. Nat Comm. 14 (1), 4761(2023).
- Monjezi, R., et al. Enhanced CAR T-cell engineering using non-viral Sleeping Beauty transposition from minicircle vectors. Leukemia. 31 (1), 186-194 (2017).
- Kay, M. A., He, C. -Y., Chen, Z. -Y. A robust system for production of minicircle DNA vectors. Nat Biotechnol. 28 (12), 1287-1289 (2010).
- Webber, B. R., et al. Cas9-induced targeted integration of large DNA payloads in primary human T cells via homology-mediated end-joining DNA repair. Nat Biomed Eng. 8, 1553-1570 (2024).
- Williams, J. A., Paez, P. A. Improving cell and gene therapy safety and performance using next-generation Nanoplasmid vectors. Mol Ther Nucleic Acids. 32, 494-503 (2023).
- Wierson, W. A., et al. Efficient targeted integration directed by short homology in zebrafish and mammalian cells. eLife. 9, 53968(2020).
- Hendel, A., et al. Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells. Nat Biotechnol. 33 (9), 985-989 (2015).
- Moretti, A., et al. The past, present, and future of non-viral CAR T cells. Front Immunol. 13, 867013(2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon