このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
の生産 C.エレガンストランスジーン galK選択可能なマーカー
要約
のために導入遺伝子を産生する能力線虫(Caenorhabditis elegans)ネイティブ調節要素のすべてが保持されるようにfosmidsによって運ばれるゲノムDNAを使うことが特に魅力的です。説明でrecombineering経由導入遺伝子の生産のためのシンプルで堅牢な手続きです。 galK選択可能なマーカー。
要約
トランスジェニック動物の作成 が広く、Cで利用されている興味またはタンデムアフィニティー精製の 世代の遺伝子の調節と発現パターンを研究するためにGFPの融合タンパク質の使用を含む線虫の研究(TAP)その精製を容易にするために特定の遺伝子のタグ付きバージョン。一般的に導入遺伝子はGFPレポーター遺伝子または目的のcDNAの上流にプロモーターを配置することで生成され、これはしばしば代表的発現パターンを生成します。しかし、このような3'非翻訳領域または別のプロモーターの制御要素として遺伝子調節の重要な要素は、このアプローチによって検出し損ねる可能性がある。さらに、単一のスプライスバリアントは、通常はこの方法で研究することができる。対照的に、フォスミドDNAクローンによって運ばの使用ワームのゲノムDNAは、おそらくほとんどの場合ではない本物の発現パターンとタイミングをキャプチャする大きな能力を許可する生体内で遺伝子の調節に関与するすべての要素が含まれています。フォスミドDNAを用いて導入遺伝子の生成を容易にするために、我々はE.を説明大腸菌は遺伝子の任意の場所にGFP、TAP -タグ、または興味の他の配列を挿入するrecombineering手順をベースに。手順は、高効率で目的の変更を取得、その結果recombineeringで正と負の選択の手順の両方のための選択マーカーとしてgalK遺伝子を使用しています。さらに、一般的に使用されるGFPとTAPの融合遺伝子に相同性ア ームに挟まれたgalKの遺伝子を含むプラスミドは、GFPまたはTAP融合タンパク質を生成する際に50%のオリゴのコストを減少する可能です。これらのプラスミドは、豊富なPCR産物の精製の必要性を排除R6Kの複製起点を使います。最後に、我々はまた、フォスミドを直接注入またはトランスジェニック動物を生成するためにワームに砲撃できるようにするフォスミドバックボーンへのUNC - 119マーカーを統合する手法を示す。このビデオでは、このメソッドを使用してrecombineeringを経由して導入遺伝子の生成に関わる手順を示しています。
プロトコル
概要
トランスジェニックC.の世代で使用されている多くの組換え遺伝子虫は、プロモーター配列と博士アンディ火1のラボで生成されたベクトルの一つにクローニングされたcDNAを、おそらく遺伝子で構成されています。これらの組換え遺伝子が頻繁にGFPレポーター遺伝子を製造または所望のパターンでcDNAを発現に関してで成功している一方で、これらの導入遺伝子は別のプロモーター、エンハンサーエレメント、および制御に重要な役割を果たす3'非翻訳領域(UTR)の要素を欠くこともできます生体 2 の遺伝子発現の。たとえば、両方のDAF - 12とFAH - 1遺伝子は、3,4,5を構築するプロモーターで失われた近位プロモーターの外にある重要なエンハンサー要素を持っている。さらに多くの導入遺伝子のコンストラクトは、適切なマイクロRNA遺伝子6,7,8で規制を防ぐUNC - 54 3'UTRを使用してください。その結果、ワームのゲノムDNAの大きなセグメントで発生する導入遺伝子は、プロモーター、スプライスバリアント、および3'UTRの制御要素のすべてを取得するための理想的でしょう。最近C.ゲノムDNAの約40 kbの領域からなると、ほぼすべてのゲノムをカバーする1本の線虫フォスミドライブラリーを構築されています。ワームのゲノムDNAの使用は、特定の遺伝子が2,8,9,10,11の本物の発現パターンとタイミングをキャプチャする大きな能力でこれらのフォスミドDNAクローンの結果によって運ばれる。
しかし、ゲノムDNAの大きな領域で作業するような標準的な分子生物学技術12を使用しての大きな困難として実用的な課題となっています。これらの制限を克服するために、技術はE.の相同組換えによりfosmidsまたは細菌人工染色体を変更する大腸菌が開発されており、12,13を recombineeringと呼ばれる。 RecombineeringはC.によって運ばれる遺伝子内の任意の位置にGFP、タンデムアフィニティー精製(TAP)、タグ、または興味の他の配列のシームレスな挿入が可能虫フォスミドクローン2,10,14。相同組換えは、特別に変更されたE.の標的部位と標的DNAに相同性の50 bpの領域に挟まれたPCR産物との間で発生する大腸菌株。
我々は最近、Cの変更のための2段階の手順を記載している目的の場所にgalKの遺伝子を挿入してから、所望の配列2にこの遺伝子を置換などのrecombineeringによる線虫の fosmids。 galK遺伝子は、それが選択的な増殖培地15の使用を経由するため、反対を選択することができるようにプロセスの両方のステップのための効果的な選択マーカーとして機能します。フォスミドの修正の最初の段階では、galK遺伝子が目的の位置に相同組換えを介して挿入され、そして正しく修正されたfosmidsは、炭素源2,15としてガラクトースを利用する能力のためのポジティブセレクションによって識別されます。第二段階では、galKの遺伝子は、所望の配列に置換され、正しく変更さfosmidsはgalK +細菌2,15を殺す有毒ガラクトース誘導体のdeoxygalactoseの使用を通じてgalKの遺伝子に対する負の選択によって識別されます。 galKの利点は、代わりに高効率2,15と、目的の変更を取得するには別個の各ステップの遺伝子、およびその結果を持っている他のマーカーの正と負の選択のステップで使用される単一の遺伝子の能力、です。
C.にこの技術の応用を促進する線虫研究は、我々は、使用可能なリソースにいくつかの変更を行いました。最初に、GFPとTAPタグは一般的にワームの導入遺伝子を生成するために使用されているので、galK遺伝子のソースとして使用するpMOD4 galK - GとpMOD4 galK - GTプラスミドにこれらのタグの各々に相同性の50 bpの領域に組み込まれ2。これらの地域は、やや高価なオリゴの2番目のセットを注文する必要がなくなりますフォスミド変更の両方の段階に使用されるオリゴの単一のセットを許可する。第二に、これらのプラスミドは、プラスミド、親がrecombineeringのために使用される細菌で複製することができるではない、とだけなどEC100 2などの特殊な株で複製することができるように、親プラスミドまたは大規模なPCR産物の精製を消化するための必要性を排除R6Kの複製起点を使う、16(表1と表2)。トランスジェニックC.を生成するの最後に、一般的な方法虫は、UNC - 119変異17の救助を経由してトランスジェニックワームの選択に続いて微粒子銃砲撃を使用することです。砲撃とfosmidsの互換性を確保するために、我々は、フォスミドバックボーン2〜上のUNC - 119マーカーを統合するために使用することができますpLoxP UNC - 119プラスミドを開発した。
I.オリゴ設計
recombineeringで所望の配列は、遺伝子内の任意の部位に挿入することができます。一般的なサイトでは、機能ドメイン、スプライスバリアント、またはプロテアーゼによるそのような切断などの翻訳後修飾によって5'末端または3'末端にあります。プラスミドは、開始コドンを含み、3'の終止コドン(図1)を欠いているとして、私たちの研究室で作成されたプラスミドpMOD4 GFPは、任意のサイトでFLAGタグGFPを挿入するために使用することができます。対照的に、TAPタグは、5'および3'精製18,19の間に使用TEV切断による融合のための特定のバージョンを持っています。
- 遺伝子内のタグの挿入部位を計画。挿入部位を検討する際に、代替プロモーター、機能ドメイン、別のスライス、および翻訳後修飾を考慮してください。異なる挿入部位はすべて、いずれか、または特定の遺伝子のいくつかのアイソフォームのタグ付けに使用することができる。挿入ポイントの上流と下流に50 bpの領域を識別します。
- オリゴ(表1)を設計。どちら100nMの規模 - 統合されたDNAの技術からゲル精製のオリゴまたはUltramerのオリゴは手続きのために使用することができます。
galKの recombineeringを実行するには、pMOD4 galK - GとpMOD4の両方に存在するgalKカセット 、に結合する目的の変更するサイトとこれらのプライマーの3'末端に隣接する領域に50塩基の相同性をgalKのプライマーを設計する必要があります。 galK - GT(図1)。フォワードオリゴは5'末端になります------- 50 bpの相同性------- CCTGTTGACAATTAATCATCGGCA - 3'および5' -------相補鎖上の50 bpの相同性として逆方向の - ------ TCAGCACTGTCCTGCTCCT - 3'。
GFPとgalKの recombineeringを実行するには、(図1)に変更される所望の部位に隣接する領域に50塩基の相同性をpMOD4 galK - GまたはpMOD4 galK - GTプライマーを設計する必要があります。フレーム内に融合タンパク質を保管してください。必要に応じてATGは破棄することができます。これらのプライマーの3'末端はgalKカセットに隣接するGFPの相同性の領域に結合する。注意:GFPの最初と最後のコドンを読み取り枠を示すために下線が引かれています。フォワードオリゴは5'末端になります------- 50 bpの相同性-------補完的に-3'3'と逆方向の5' ------- 50 bpの相同性をGATTACAAGGACGATGACGATAAGATGAG ATGを鎖------- CAA AGCTTGTGGGCTTTTGTATAG - 3'
galK C短期TAPのrecombineeringを実行するには、(図1)に変更される所望の部位に隣接する領域に50塩基の相同性をpMOD4 galK - GTプライマーを設計する必要があります。フレーム内に融合タンパク質を保管してください。これらのプライマーの3'末端はgalKカセットに隣接するTAPの相同性の領域に結合する。注:TAPの最初と最後のコドンを読み取り枠を示すために下線が引かれています。フォワードオリゴは5'末端になります------- 50 bpの相同性------ GAAAAGAGAAGATGGAAAAAG ATGを - -3'と逆方向の5' -------相補鎖上の50 bpの相同性- ------ GGT TGACTTCCCCGC -3"
galK N短期TAPのrecombineeringを実行するには、(図1)に変更される所望の部位に隣接する領域に50塩基の相同性をpMOD4 galK - GTプライマーを設計する必要があります。フレーム内に融合タンパク質を保管してください。これらのプライマーの3'末端はgalKカセットに隣接するTAPの相同性の領域に結合する。注:TAPの最初と最後のコドンを読み取り枠を示すために下線が引かれています。フォワードオリゴは5'末端になります------- 50 bpの相同性------ GCAGGCCTTGCGC ATGを - -3'と逆方向の5' -------相補鎖上の50 bpの相同性- ------ AAG TGCCCCGGAGGATGAGATTTTCT -3" - 後のステップでだけでなく、フォスミドの配列決定のためのPCR用のオリゴヌクレオチドに隣接のセットを生成する。これらは、上流と下流の挿入部位の約100 bpをバインドする標準的なPCRオリゴです。
II。 SW016細菌にフォスミド転送
C.からfosmids 虫フォスミドライブラリEPI300菌株(F - MCRAΔ(MRR - hsdRMS - mcrBC)φ80dlacZΔM15ΔlacX74recA1 endA1 araD139Δ(ARA、ロイシン)7697嘎魯galKλ- RPSL nupG trfAトナ )(シャーロットのバイオテクノロジーズ、マディソン、ウィスコンシン州で提供されています)これは、フォスミド式が精製(表2)中にDNAの収量を向上させるために、セルごとに単一のコピーを超えて上昇することができます。 recombineeringの場合は、フォスミドはSW106細菌株(MCRAΔ(MRR - hsdRMS - mcrBC)ΔlacX74deoR endA1 araD139Δ(ARA、ロイシン)7697 RPSL recA1 nupGφ80dlacZΔM15[λc1857(CRO - bioA)<>テトに転送する必要があります。リプレッサー、温度に敏感なλとアラビノース誘導性Creリコンビナーゼ(表2)15の制御下λred相同組換え遺伝子を運ぶ](CRO - bioA)<> ARAC -のpBADのCreΔgalK)(NCI -フレデリック)菌株。
- Wormbを使用してGeneServiceから目的の遺伝子(GOI)のフォスミドクローン(ケンブリッジ、英国)を注文ガイドとしてASE。クローンを選択したとき、我々は、シーケンスの中央でGOIを持っているものを選択してください。隣接遺伝子を除外するものが好ましいかもしれないが、見つけるのは難しいかもしれない。
- 文化37 12.5μg/ mLのクロラムフェニコールを含むLBのGOIのフォスミドクローン℃の
- フォスミド1.5mLの一晩培養し、EPICENTREフォスミド準備キット(シャーロットのバイオテクノロジーズ、マディソン、WI)を用いてミニプレップフォスミドDNAを成長する。我々は、前のステップでRiboshredderミックスを追加することを指示で説明されている代替プロトコルに従ってください。
- 分光光度計によるフォスミドDNA濃度を決定する。
- 32℃で12.5μg/ mLのクロラムフェニコールを含むLBブロスで14 mLのスナップキャップチューブにSW106 5 mlの一晩培養を成長させることによりエレクトロSW106細胞を準備します。
- 2 Lのフラスコにクロラムフェニコールを含むLB 100ml中に1mlを接種する。 OD 600 0.6から0.8へのSW106細菌を培養する。熱ショックしないでください。
- 5分間5000xgの遠心分離によってペレットは、穏やかにボルテックスによりペレットを再懸濁し、50 mlの氷冷した10%グリセロールを追加。一度この洗浄ステップを繰り返します。
- ペレットを遠心分離によりSW106と各上清のすべてが〜500μLを吸引する
- 穏やかにボルテックスでペレットを再懸濁します。 100μlの液体窒素やドライアイス上に分注し、-80℃で保存将来の使用のために凍結する。
- 0.1 cmのギャップのキュベットで1350ボルトでエッペンドルフ2510エレクトロを使用して、フォスミドDNAの〜50 ngで細菌をelectroporatingでエレクトロSW106細胞にフォスミドDNAを変換する。
- 32℃で1時間に1 mLのLBで細菌を回復する
- プレートのクロラムフェニコールを含むLBプレート上にアリコート(12.5μg/ mL)を、32℃で一晩。
- コロニーPCRによるGOIの存在を確認してください。 32℃で12.5μg/ mLのクロラムフェニコールを含むLBの5mLの一晩培養して成長する隣接するオリゴを用いた標準的なPCR反応に文化の0.5μLを追加し、PCRの前に細菌を溶解するために5分に最初の95 ° Cインキュベーションを増加させる。
- 長期保存のためにグリセロールストックを準備します。
III。 RecombineeringでgalK遺伝子の挿入
フォスミド変更の第一段階では、galK遺伝子は相同組換えによりフォスミドに挿入され、正しく変更さfosmidsは、唯一の炭素源(図2A)とガラクトースを含む最少培地での増殖により対象として選択されます。 SW106細菌は、最少培地でゆっくりと成長し、3〜5日は、コロニーを確認する必要があります。
- 0.2%ガラクトースを含むMOPS最少培地のプレートを準備します。 MOPS最少培地はTeknova株式会社(ホリスター、カリフォルニア州)(カタログ#M2106)から入手可能ですが、含まれるグルコースを使用しないでください。
0.2%ガラクトース(1 L)とMOPS最少培地
オートクレーブ870 mLの水に15グラムの寒天
55℃まで冷却し、追加します。100mLの 10X MOPS最少培地 5 mLの は0.2 mg / mlのD -ビオチン(濾過滅菌) 4.5mLの 10 mg / mlのL -ロイシン(1%、温水は、その後冷却し、滅菌濾過) 10mLの 20%のガラクトース(オートクレーブ) 1 mLの EtOH中で12.5 mg / mlのクロラムフェニコール 2.55 mLの 20パーセントのNH 4 Cl 10mLの 0.132 Mリン酸水素二カリウム - PCRは、上記の設計したプライマーを用いてpMOD4 galK - GまたはpMOD4 galK - GTのカセットを増幅する。我々は、Phusion(ニューイングランドバイオラブズ、イプスウィッチ、MA)またはGoTaq(プロメガ社、マディソン、WI)を使用している。
- ゲルとなるバンドを精製する。ゲルまたは光度計分光光度計により収量を定量化する。このPCR産物は、ステップ3.14の準備ができています。
- クロラムフェニコールと5 mlのLB(12.5μg/μLの)のフォスミドDNAを含むSW106細胞の一晩培養を接種する。 32℃で成長する
- 42に水浴を振っ設定° Cまでホルダーに滅菌を250mlのフラスコでウォームアップ。振とう水浴の使用は、高効率を得るために重要です。
- 2 LのフラスコにLBとクロラムフェニコールの100mlに一晩培養液1 mlを加え。 OD 0.6から0.8に成長する。これは、通常3-4時間かかります。
- 42℃で250mlのフラスコや熱ショックにSW106細胞の50 mlを° Cを正確に20分間。 100rpmで振とうしながら水浴中で非誘導コントロールとして32℃で残っている細菌にしておきます。
- 10分間氷上で誘導および非誘導細菌を冷却する。
- 5分間〜5000xgで2つの滅菌遠心管とペレットにサンプルを移す。
- 上清のすべてを捨て、穏やかにボルテックス(すなわち設定3-4)1 mlの氷冷した10%グリセロールでペレットを再懸濁します。
- 再懸ときは、別の49ミリリットルの氷冷10%グリセロールを追加し、そしてペレット〜5000でのサンプル5分間XGの。
- 再びステップ3.9、3.10、および3.11を繰り返します。
- 反転管ですべての上清を除き、残りの液体中のペレット(約500μLずつ)懸濁します。 100μLの試料へのアリコートを、ドライアイスで凍結、-80℃で保存します。これらは数週間から数カ月に適しています。 (私達は通常ここに停止し、次の日にエレクトロポレーションを行う)。
- 1350ボルトでエッペンドルフ2510エレクトロセットで0.1 cmのギャップのキュベットを用いてPCR産物の150 ngで誘導および非誘導SW106細胞をエレクトロポ。
- 14ミリリットルファルコンチューブに1 mLのLBで細菌を回復する。 4.5時間32℃でインキュベートする。
- ペレットは、15秒間、13200 rpmで遠心中の細菌。細菌はM9に再懸濁し、その後二度(レシピは下記参照)リッチな培地を除去洗浄する。
- M9培地(1 L)
- 6グラムのNa 2 HPO 4
- 3グラムKH 2 PO 4
- 1グラムのNH 4 Cl
- 0.5グラムのNaCl
- オートクレーブ
- 回目の洗浄後、上清を除去し、ペレットをMOPS最少培地にM9(100μL、10倍希釈液100μL、100μL1:100)で連続希釈液をメッキする前に、1 mLのM9に再懸濁する。
- 32℃インキュベーターで℃で3〜5日間インキュベートする。注:真陽性がゆっくり成長するように我慢して。
- ストリーク1%ガラクトースおよびクロラムフェニコール12.5μg/ mLとを添加したマッコンキー寒天インジケータープレート(BD#281810)上にいくつかのコロニーを。最後のステップの後に現れるコロニーのすべては、galKの +になるはずですが、どんなgalKを取り除くために-汚染物質、それは次のステップに進む前に、単一の、鮮やかなピンク色のコロニーを得ることが重要である。 galK -コロニーは無色/白になるとgalK + の細菌が32で一晩インキュベーションした後、発酵ガラクトースに起因するpHの変化に明るいピンク/赤のためになる℃、
- 単一コロニーを採取し、32で成長のために5 mlのLB +クロラムフェニコール一晩培養を接種℃に
- 隣接するオリゴを用いてPCRを介して適切な位置にgalK遺伝子の挿入を確認してください。標準的なPCR反応に文化の0.5μLを追加し、細菌を溶解するために5分に最初の95℃のインキュベーションを増加させる。 PCR産物をgalK遺伝子の存在のためにサイズにシフトアップする必要があります。
- ストレージのためにグリセロールストックを準備します。
IV。 Recombineeringでタグ配列を持つgalKの交換
この段階ではgalK遺伝子は 、目的のタグ配列に置き換えられ、正しく修正されたfosmidsは有毒ガラクトースアナログdeoxygalactose(DOG)(図2B)によってgalK遺伝子に対して選択によって対象として選択されます。
- 0.2%deoxygalactose(DOG)と0.2%グリセロールを含むMOPS最少培地のプレートを準備します。 MOPS最少培地はTeknova株式会社(ホリスター、カリフォルニア州)(カタログ#M2106)から入手可能ですが、含まれるグルコースを使用しないでください。
0.2%DOGとグリセロール(1L)とMOPS最少培地
オートクレーブ860 mLの水に15グラムの寒天
55℃まで冷却し、追加します。100mLの 10X MOPS最少培地 5 mLの は0.2 mg / mlのD -ビオチン(濾過滅菌) 4.5mLの 10 mg / mlのL -ロイシン(1%、温水は、その後冷却し、滅菌濾過) 10mLの deoxygalactose 20%(濾過滅菌) 10mLの 20%グリセロール(オートクレーブ) 1 mLの EtOH中で12.5 mg / mlのクロラムフェニコール 2.55 mLの 20パーセントのNH 4 Cl 10mLの 0.132 Mリン酸水素二カリウム - PCRは、最初のラウンドで使用したのと同じオリゴを使用したり、短いGFPまたはTAP -特異的オリゴ(ステップ1.2の内側の配列を使用してpMOD4 GFP、pBS1761(N短期TAP)、またはpBS1479(C短期TAP)からタグの断片を増幅)。複数のコンストラクトを作成している場合は、同じPCR産物は、すべての構造体のために使用できるため、より短いオリゴヌクレオチドを使用する場合に特に便利です。
- ゲルPCR産物を精製し、ゲルまたは分光光度計を介して濃度を測定する。
- 上記の手順3.4から3.13以下の挿入galK遺伝子とフォスミドを運ぶ誘導および非誘導有能SW106を生成します。
- 1350ボルトでエッペンドルフ2510エレクトロセットで0.1 cmのギャップのキュベットを用いてPCR産物の〜100 ngで誘導および非誘導SW106細胞をエレクトロポ。
- 4.5時間32℃シェーカーで14ミリリットルスナップキャップチューブとインキュベート1 mlのLBで回復する。
- 手順3.16と3.17のように細菌を洗浄し、希釈する。 0.2パーセント2 - デオキシ - ガラクトース(DOG)と0.2%グリセロールを含むMOPS最少培地プレート上にプレートの細菌。
- 三日間32℃でインキュベートする。
- 四コロニーを5 mLを作るために使用されます12.5μg/ mLのクロラムフェニコールを含むLBで一晩培養。これらは、カセットが挿入されたことを確認するために、上記のコロニーPCRに使用されます。我々は両方の短いGFPを使用/右を挿入し、右のサイトを示すために、特定のオリゴと隣接するオリゴをタップします。 GFPは、〜800 bpであるとgalKが 1.4キロバイトであるのに対し、TAPは〜550 bpである。
- グリセロールストックを準備します。
Cre - loxP組換えによってUNC - 119遺伝子のV.の追加
変更fosmidsとトランスジェニック動物を生成する一般的な方法は、微粒子銃砲撃を使用することです。このテクニックは、CにフォスミドDNAを導入するDNAでコーティングされた金粒子を使用していますエレガンス 。トランスジェニック動物は、通常、UNC - 119の遺伝子を持つUNC - 119変異体の救助を経由して識別されます。このステップでは、UNC - 119遺伝子は、プラスミドpLoxP UNC - 119(図2C)とCre - loxP組換えによってシスにフォスミドバックボーンに追加されます。
- ステップ2.5から2.9を使用して、ステップ4.9から変更されたフォスミドを運んで有能なSW106細菌を準備します。 42で誘導しない℃に
- 50 ngのとエレクトロ。 pLoxP UNC - 119 1350ボルトで設定エッペンドルフ2510エレクトロに0.1 cmのギャップのキュベットを使用してミニプレップから。
- 32℃で1時間、0.1%のアラビノースを含むLB培地で細菌を回復する
- 50μg/mLアンピシリン、12.5μg/ mlのクロラムフェニコールを含むLBプレート上にプレートのアリコート。 ℃で一晩32でインキュベートする。これは、フォスミドにpLoxP UNC - 119の統合を選択します。
- 50μg/mLアンピシリン、12.5μg/ mLのクロラムフェニコールを含むLB培地で一晩培養して成長する。 UNC - 119 F(5' - CAAATCCGTGACCTCGACAC - 3')とUNC - 119 R(5' - CACAGTTGTTTCTCGAATTTGG - 3')オリゴ(表1)とUNC - 119遺伝子の存在を確認するPCRのために0.5μLを使用してください。
- 最後のフォスミドのグリセロールストックを作る。
VI。大規模フォスミドの調製
砲撃に必要なフォスミドDNAのより多くの数量を得る容易にするために、このステップではフォスミドはEPI300細菌に転送されます。この株は、DNAの準備中に歩留まりを向上させるフォスミドコピー数を増加させる能力を持っています。
- 32℃でアンピシリンおよびクロラムフェニコールを含むLBのステップ5.5からの細菌の5mLの一晩培養して成長する培養液1.5 mLからフォスミドを分離するEPICENTREフォスミドプレップキットを使用してください。
- エレクトロ〜1350ボルトのエッペンドルフ2510エレクトロセットで0.1 cmのギャップのキュベットを使用してEPI300細菌に50 ngのを。 EPI300細菌は、シャーロットのバイオテクノロジー(マディソン、WI)から購入することができます。
- 37℃で1時間、LBで細菌を回復℃に50μg/ mlの12.5μg/ mlのクロラムフェニコールを含むLB寒天上にプレートのアリコート。
- 含まれている手順を使用して変更フォスミドを含むEPI300細菌を培養すると誘導する。 50 mLの誘導の文化は、フォスミドDNAを精製> 10μgを与える。 EPICENTREフォスミドプレップキットを使用してフォスミドを精製する。
VII。砲撃
- 10μgを使用してください。攻めへのフォスミドDNAとして記述さDP38ワーム株(D. Hochbaum、A.ファーガソン、およびA.フィッシャー、マスコミでJoveの、)の。
VIII。代表的な結果
負の選択のステップで、堅牢で> 90%の成功率をrecombineering経由fosmidsの変更は日常的に2を観測している。このプロトコルはまた、導入遺伝子の調製は、かなり急速になっている完了するために〜2週間かかります。プロトコルはまた、成功20を有する他の研究室で試みられている。
| オリゴ | シーケンス |
| C長期的なTAPのF | ATGGAAAAGAGAAGATGGAAAAAG |
| C短期TAP R | GGTTGACTTCCCCGC |
| FLAG - GFP F | ATGGATTACAAGGACGATGACGATAAGATGAG |
| FLAG - GFP R | CAAAGCTTGTGGGCTTTTGTATAG |
| N項のTAP F | ATGGCAGGCCTTGCGC |
| N項TAP R | AAGTGCCCCGGAGGATGAGATTTTCT |
| galK F | CCTGTTGACAATTAATCATCGGCA |
| galK R | TCAGCACTGTCCTGCTCCT |
| UNC - 119 F | CAAATCCGTGACCTCGACAC |
| UNC - 119 R | CACAGTTGTTTCTCGAATTTGG |
PCRに用いた表1。オリゴヌクレオチド。
| プラスミド | 元 | で入手可能 |
| Fosmidクローン | Geneservice(株) | Geneservice |
| pGalK | 15 | NCI -フレデリック |
| pMOD4 - RT - G | 2 | Addgene |
| pMOD4 - galK - G | ||
| pMOD4 - galK - GT | ||
| pLoxP - UNC - 119 | ||
| pMOD4 - GFP | ||
| 菌 | ||
| SW106 | 15 | NCI -フレデリック |
| EPI300 | シャーロットのバイオテクノロジー | EPICENTRE |
| EC100D PIR - 116 | ||
表2。ひずみとベクトルの可用性。
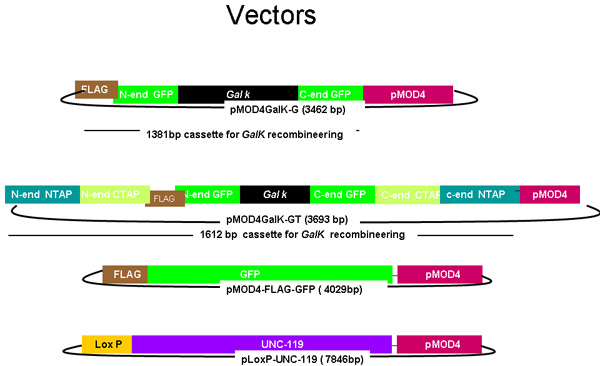
図1。
pMOD4 - galK - G、およびpMOD4 - galK - GT、pMOD4 GFP、pLoxP - UNC - 119の図
PMOD 4 galK - GTが galKで構成されている間pMOD4 - galk - Gプラスミドは、5'および3'FLAGの両端(ブラウン)- GFP(緑色)と同一の50ヌクレオチドの領域に挟まれたgalKカセット (黒)で構成されています5'および3'N末端とC末端のTAP(青とオレンジ、それぞれ)の両端に同一のFLAG - GFPの相同性領域および50ヌクレオチド領域の両方に挟まれたカセット。 pMOD4 - FLAG - GFPは、5'FLAGタグとpLoxP UNC - 119 loxP部位を含むプラスミドでUNC - 119のゲノム配列(紫)から構成されるとの完全なGFPカセットで構成されています。すべてのプラスミドは、SW106で複製することができないR6KベースpMOD4(赤)バックボーンを利用する。
図2。
galKの recombineeringプロセスの概要
図2A - 2C galKカセットを使用してrecombineeringに関連する手順と時間を示す別の図。これらは、図2dにマージされているのと同じ数字ですが、明快さと読みやすさのために別々に提供。興味のフォスミドは、最初のFLAG - GFPまたはTAPでこのカセットの交換に続いてFLAG - GFPまたはTAP(図2A)に相同性の50bpの領域に挟まれたgalKカセットの挿入を含む二段階の手順で変更されます(図2B)。後でトランスジェニック動物の生成に使用するUNC - 119マーカーは、フォスミド骨格上のloxPサイト(図2C)に挿入されます。
図2DはgalKの recombineering手順のマージされた図を示しています。
図2A - 2Cをマージから、各ステップに要する時間を含め、上記で説明したようにgalKの recombineeringの手順の概要。
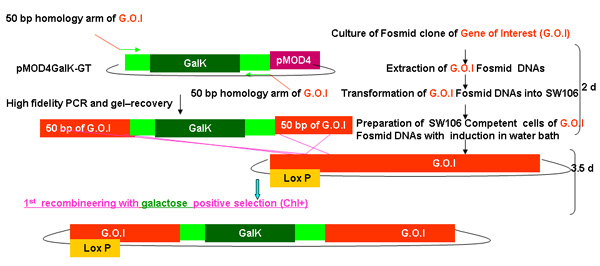
図2a。galk の recombineeringでgalk挿入 。
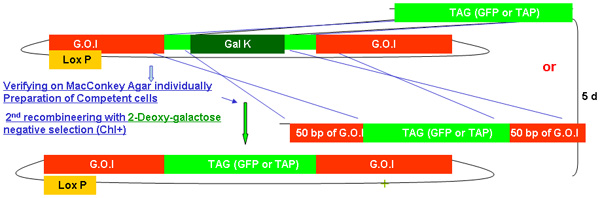
図2b。galk の recombineeringのTAG(GFP / TAP)の挿入。
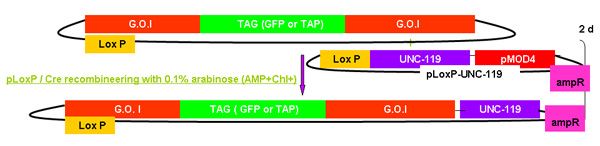
図2c。UNC - 119の追加。
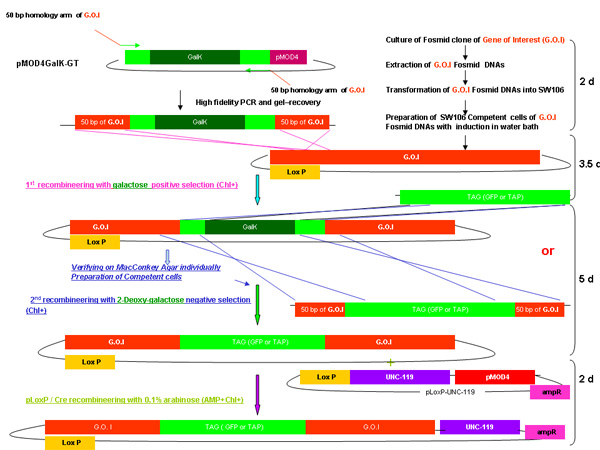
図2d。galk の recombineeringのマージされた概要。
ディスカッション
fosmidsから導入遺伝子の世代は、ネイティブなプロモーターエレメント、スプライシング変異体、および3'UTR調節要素のすべてを保持することの利点を提供しています。これは、ネイティブの発現パターン、または他のアプローチは5失敗した機能的な導入遺伝子の構造をより反映し、導入遺伝子の構築につながることができます。その結果導入遺伝子は、GFPまたはTAPのタグを含む?...
開示事項
謝辞
著者は、技術の開発の手助けのためにリンジーナッシュに感謝します。この作品は、ALF、ピッツバーグ大学からピッツバーグOAIC(AG024827)、およびシード資金の大学からのパイロットプロジェクトの助成にNIHの助成金AG028977によって賄われていた。
資料
| Name | Company | Catalog Number | Comments |
| FosmidMAX kit | Epicentre Biotechnologies | FMAX046 | |
| GoTaq | Promega Corp. | M7122 | |
| MOPS Media | TEKnova, Inc. | M2120 | |
| 0.132 M Potassium phosphate solution | TEKnova, Inc. | M2102 | |
| D-galactose | Sigma-Aldrich | G0750 | |
| 2-deoxygalactose | Sigma-Aldrich | D4407 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Leucine | Sigma-Aldrich | L8000 | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Phusion DNA polymerase | New England Biolabs | F-530S | |
| MacConkey agar base | BD Biosciences | 281810 | |
| Arabinose | Sigma-Aldrich | A3131 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S5136 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Sodium chloride | Sigma-Aldrich | S5886 | |
| Glycerol | Sigma-Aldrich | G2025 | |
| Bacto Agar | BD Biosciences | 214010 |
参考文献
- Mello, C., Fire, A. DNA transformation. Methods Cell Biol. 48, 451-482 (1995).
- Zhang, Y., Nash, L., Fisher, A. L. A simplified, robust, and streamlined procedure for the production of C. elegans transgenes via recombineering. BMC Dev Biol. 8, 119-119 (2008).
- Antebi, A., Yeh, W. H., Tait, D., Hedgecock, E. M., Riddle, D. L. daf-12 encodes a nuclear receptor that regulates the dauer diapause and developmental age in C. elegans. Genes and Development. 14, 1512-1527 (2000).
- Snow, M. I., Larsen, P. L. Structure and expression of daf-12: a nuclear hormone receptor with three isoforms that are involved in development and aging in Caenorhabditis elegans. Biochim. Biophys. Acta. 1494, 104-116 (2000).
- Fisher, A. L., Page, K. E., Lithgow, G. J., Nash, L. The Caenorhabditis elegans K10C2.4 Gene Encodes a Member of the Fumarylacetoacetate Hydrolase Family. A CAENORHABDITIS ELEGANS MODEL OF TYPE I TYROSINEMIA. J Biol.Chem. 283, 9127-9135 (2008).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75, 855-862 (1993).
- Lehrbach, N. J. LIN-28 and the poly(U) polymerase PUP-2 regulate let-7 microRNA processing in Caenorhabditis elegans. Nat Struct Mol Biol. 16, 1016-1020 (2009).
- Tursun, B., Cochella, L., Carrera, I., Hobert, O. A toolkit and robust pipeline for the generation of fosmid-based reporter genes in C. elegans. PLoS One. 4, e4625-e4625 (2009).
- Bamps, S., Hope, I. A. Large-scale gene expression pattern analysis, in situ, in Caenorhabditis elegans. Brief. Funct. Genomic. Proteomic. , (2008).
- Dolphin, C. T., Hope, I. A. Caenorhabditis elegans reporter fusion genes generated by seamless modification of large genomic DNA clones. Nucleic Acids Res. 34, e72-e72 (2006).
- Sarov, M. A recombineering pipeline for functional genomics applied to Caenorhabditis elegans. Nat. Methods. 3, 839-844 (2006).
- Yang, X. W., Model, P., Heintz, N. Homologous recombination based modification in Escherichia coli and germline transmission in transgenic mice of a bacterial artificial chromosome. Nat Biotechnol. 15, 859-865 (1997).
- Court, D. L., Sawitzke, J. A., Thomason, L. C. Genetic engineering using homologous recombination. Annu.Rev.Genet. 36, 361-388 (2002).
- Westenberg, M., Bamps, S., Soedling, H., Hope, I. A., Dolphin, C. T. Escherichia coli MW005: lambda Red-mediated recombineering and copy-number induction of oriV-equipped constructs in a single host. BMC Biotechnol. 10, 27-27 (2010).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36-e36 (2005).
- Penfold, R. J., Pemberton, J. M. An improved suicide vector for construction of chromosomal insertion mutations in bacteria. Gene. 118, 145-146 (1992).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Puig, O. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat.Biotechnol. 17, 1030-1032 (1999).
- Achilleos, A., Wehman, A. M., Nance, J. PAR-3 mediates the initial clustering and apical localization of junction and polarity proteins during C. elegans intestinal epithelial cell polarization. Development. 137, 1833-1842 (2010).
- Maduro, M., Pilgrim, D. Identification and cloning of unc-119, a gene expressed in the Caenorhabditis elegans nervous system. Genetics. 141, 977-988 (1995).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved