Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
La production de C. elegans Les transgènes via Recombineering avec le GalK Marqueurs de sélection
Dans cet article
Résumé
La capacité de produire des transgènes Caenorhabditis elegans En utilisant l'ADN génomique porté par fosmides est particulièrement intéressante car tous les éléments natifs réglementaires sont conservés. Décrite est une procédure simple et robuste pour la production de transgènes via recombineering avec le GalK Marqueur de sélection.
Résumé
La création d'animaux transgéniques est largement utilisée en C. la recherche elegans y compris l'utilisation de protéines de fusion GFP pour étudier la régulation et de profil d'expression de gènes d'intérêt ou de génération de purification d'affinité en tandem (TAP) taggés versions de gènes spécifiques pour faciliter leur purification. Typiquement transgènes sont générés en plaçant un promoteur en amont d'un gène rapporteur GFP ou de l'ADNc d'intérêt, et cela produit souvent un motif d'expression représentant. Toutefois, des éléments critiques de la régulation des gènes, tels que les éléments de contrôle dans la région 3 'non traduite ou promoteurs alternatifs, pourraient être manquées par cette approche. En outre seul un variant d'épissage unique peut être généralement étudiés par ce moyen. En revanche, l'utilisation de l'ADN génomique ver porté par des clones d'ADN fosmide inclut probablement la plupart sinon tous les éléments impliqués dans la régulation des gènes in vivo qui permet la plus grande capacité à capter le profil d'expression authentique et le calendrier. Afin de faciliter la génération de transgènes en utilisant l'ADN fosmide, nous décrivons un E. coli procédure basée sur recombineering d'insérer la GFP, un TAP-tag, ou d'autres séquences d'intérêt dans n'importe quel endroit dans le gène. La procédure utilise le gène galK comme marqueur de sélection pour les deux étapes de sélection positive et négative dans recombineering qui aboutit à l'obtention de la modification souhaitée avec une grande efficacité. En outre, des plasmides contenant le gène galK flanqué par des bras d'homologie à la GFP couramment utilisés et TAP gènes de fusion sont disponibles, qui réduisent le coût des oligos de 50% lors de la génération une protéine de fusion GFP ou TAP. Ces plasmides utiliser l'origine de réplication R6K, qui exclut la nécessité d'une purification complète de produits de PCR. Enfin, nous avons également démontrer une technique pour intégrer le marqueur unc-119 à la colonne vertébrale qui permet à l'fosmide fosmide d'être directement injectés ou bombardés en vers pour générer des animaux transgéniques. Cette vidéo montre les procédures impliquées dans la génération d'un transgène par le biais recombineering utilisant cette méthode.
Protocole
Aperçu
Beaucoup de transgènes utilisés dans la production de transgéniques C. elegans consistent en des séquences promoteur et peut-être un gène d'ADNc cloné dans l'un des vecteurs générés par le laboratoire du Dr Andy incendie 1. Bien que ces transgènes sont souvent couronnés de succès en ce qui concerne la production d'un gène rapporteur GFP ou exprimant un ADNc dans un motif désiré, ces transgènes peuvent pas les promoteurs de remplacement, des éléments activateurs, et 3 'non traduite région (UTR) des éléments qui jouent un rôle important dans le contrôle l'expression des gènes in vivo 2. Par exemple, à la fois les gènes daf-12 et Fah-1 ont d'importants éléments enhancer qui se situent en dehors du promoteur proximal qui ont été manqués dans les constructions promoteur ne 3,4,5. D'autres constructions transgéniques nombreuses utiliser le 3'UTR unc-54 qui empêche la régulation par les gènes de microARN appropriée 6,7,8. Par conséquent, générer des transgènes avec de larges segments d'ADN génomique ver serait idéal pour capturer tous les promoteurs, les variants d'épissage, et 3 'UTR des éléments de contrôle. Récemment, un C. elegans fosmide bibliothèque qui se compose de 40 ko ~ régions de l'ADN génomique et couvre la quasi-totalité du génome a été construite. L'utilisation de l'ADN génomique ver portés par ces clones d'ADN fosmide résultats dans la plus grande capacité à capter le profil d'expression authentique et le calendrier des gènes spécifiques 2,8,9,10,11.
Cependant, travailler avec de grandes régions de l'ADN génomique pose des problèmes pratiques tels que les grandes difficultés dans l'utilisation de techniques standard de biologie moléculaire 12. Pour surmonter ces limitations, les techniques de modifier ou de fosmides chromosomes bactériens artificiels par recombinaison homologue dans E. coli ont été développés et sont appelés recombineering 12,13. Recombineering permet l'insertion transparente de la GFP, une purification par affinité en tandem (TAP)-tag, ou d'autres séquences d'intérêt dans n'importe quel endroit dans le gène porté par le C. elegans fosmide clone 2,10,14. La recombinaison homologue se produit entre un produit de PCR flanqué de régions d'homologie 50 pb sur le site cible et l'ADN cible dans spécialement modifiés E. coli.
Nous avons récemment décrit une procédure en deux étapes pour la modification de C. fosmides elegans par recombineering qui implique l'insertion du gène galK à l'endroit désiré et ensuite remplacer ce gène avec la séquence désirée 2. Le gène galK sert de marqueur de sélection efficace pour les deux étapes dans le processus, car il peut être sélectionné pour et le contre par l'utilisation d'un milieu de croissance sélective 15. Dans la première étape de la modification fosmide, le gène galK est inséré par recombinaison homologue à l'endroit désiré, et le fosmides correctement modifié identifiés par sélection positive pour la capacité d'utiliser le galactose comme source de carbone 2,15. Dans la deuxième étape, le gène galK est remplacé par la séquence désirée, et le fosmides correctement modifiés sont identifiés par une sélection négative contre le gène galK par l'utilisation des dérivés toxiques deoxygalactose galactose qui tue les bactéries galK + 2,15. Un avantage de l'galK est la capacité d'un seul gène à être utilisé pour les étapes de sélection positive et négative, au lieu d'autres marqueurs, qui ont des gènes distincts pour chaque étape, et les résultats à obtenir la modification souhaitée avec un rendement élevé 2,15.
Pour faciliter l'application de cette technique à C. elegans recherche, nous avons apporté plusieurs modifications aux ressources disponibles. Premièrement, les balises GFP et TAP sont couramment utilisés pour générer des transgènes ver, de sorte que nous avons construit dans les régions d'homologie 50 pb à chacune de ces balises dans le pMOD4 galK-G et pMOD4 galK-GT plasmides qui servent comme source du gène galK 2. Ces régions permettent à un seul ensemble d'oligos à être utilisé pour les deux étapes de la modification fosmide qui économise de la nécessité de commander un deuxième ensemble d'oligos un peu cher. Deuxièmement, ces plasmides utiliser l'origine de réplication R6K ce qui exclut la nécessité de digérer le parent purification de plasmides ou étendue de produits de PCR comme le plasmide parent n'est pas capable de se répliquer dans les bactéries utilisées pour recombineering, et ne peut se reproduire dans des souches particulières telles que EC100 2 , 16 (tableau 1 et tableau 2). Enfin, une façon commune de générer transgénique C. elegans est par l'utilisation de bombardement biolistique suivie par la sélection pour les vers transgéniques via le sauvetage de la mutation unc-119 17. Pour rendre le fosmides compatible avec les bombardements, nous avons développé le pLoxP unc-119 plasmide qui peut être utilisé pour intégrer le marqueur unc-119 à la colonne vertébrale fosmide 2.
I. Oligo Conception
Avec recombineering les séquences souhaitées peut être inséré à n'importe quel site dans le gène. Des sites communs sont à l'extrémité 5 'ou 3' selon les domaines fonctionnels, les variants d'épissage, ou modifications post-traductionnelles telles que le clivage par les protéases. La GFP pMOD4 plasmide créé par notre laboratoire peut être utilisé pour insérer un FLAG-GFP marqués à tout site comme le plasmide comprend un codon initiateur et manque de codon stop de 3 '(figure 1). En revanche, l'étiquette TAP a des versions spécifiques pour les 5 'et 3' fusions dû au clivage TEV utilisé lors de la purification 18,19.
- Plan du site d'insertion dans le gène marqueur. Considérez promoteurs suppléant, domaines fonctionnels, alternative tranchage et modifications post-traductionnelles lors de l'examen du site d'insertion. Différents sites d'insertion peuvent être utilisés pour étiqueter toutes, une ou certaines isoformes d'un gène spécifique. Identifier les 50 régions pb en amont et en aval du point d'insertion.
- Conception des oligos (tableau 1). Soit une échelle de 100 nm - gel d'oligos purifiée ou oligos Ultramer des Technologies de l'ADN intégré peut être utilisé pour la procédure.
Pour effectuer recombineering galK, vous avez besoin pour concevoir des amorces galK avec 50 pb homologie à une région flanquant le site désiré d'être modifiée et l'extrémité 3 'de ces amorces se lient à la cassette galK, qui est présent dans les deux pMOD4 galK-G et pMOD4 galK-GT (figure 1). L'attaquant sera oligo 5'------- ------- 50 homologie pb CCTGTTGACAATTAATCATCGGCA-3 'et le reverse que 5'------- 50 homologie pb sur le brin complémentaire - ------ TCAGCACTGTCCTGCTCCT-3 '.
Pour effectuer recombineering galK avec la GFP, dont vous avez besoin pour concevoir pMOD4 galK-G ou pMOD4 galK-GT avec 50 amorces homologie pb à une zone flanquant le site désiré d'être modifiées (Figure 1). Soyez sûr de garder la protéine de fusion dans le cadre. L'ATG peut être écartée si désiré. L'extrémité 3 'de ces amorces se lient à des régions d'homologie GFP flanquant la cassette galK. Remarque: Les codons premier et le dernier de la GFP sont soulignés afin de démontrer le cadre de lecture. Les oligo avant seront 5'------- ------- 50 homologie pb ATG GATTACAAGGACGATGACGATAAGATGAG -3 '3' et le reverse 5'------- 50 homologie pb sur la complémentarité ------- brin CAA AGCTTGTGGGCTTTTGTATAG-3 '
Pour effectuer recombineering galK TAP C-term, vous avez besoin pour concevoir pMOD4 galK-GT avec 50 amorces homologie pb à une zone flanquant le site désiré d'être modifiées (Figure 1). Soyez sûr de garder la protéine de fusion dans le cadre. L'extrémité 3 'de ces amorces se lient à des régions d'homologie TAP flanquant la cassette galK. Remarque: Les codons premier et le dernier de TAP sont soulignés afin de démontrer le cadre de lecture. Les oligo avant seront 5'------- ------ 50 homologie pb ATG GAAAAGAGAAGATGGAAAAAG - -3 'et l'inverse une 5'------- 50 homologie pb sur le brin complémentaire - ------ GGT -3 TGACTTCCCCGC '
Pour effectuer recombineering galK TAP N-terme, vous avez besoin pour concevoir pMOD4 galK-GT avec 50 amorces homologie pb à une zone flanquant le site désiré d'être modifiées (Figure 1). Soyez sûr de garder la protéine de fusion dans le cadre. L'extrémité 3 'de ces amorces se lient à des régions d'homologie TAP flanquant la cassette galK. Remarque: Les codons premier et le dernier de TAP sont soulignés afin de démontrer le cadre de lecture. Les oligo avant seront 5'------- ------ 50 homologie pb ATG GCAGGCCTTGCGC - -3 'et l'inverse une 5'------- 50 homologie pb sur le brin complémentaire - ------ AAG -3 TGCCCCGGAGGATGAGATTTTCT ' - Générer un ensemble de flanquement oligos pour la PCR dans les étapes ultérieures ainsi que pour le séquençage de l'fosmide. Ce sont des standards oligos de PCR qui doit lier ~ 100 pb en amont et en aval du site d'insertion.
II. Transfert à l'fosmide SW016 bactéries
Le fosmides de la C. elegans fosmide bibliothèques sont fournies dans la souche bactérienne EPI300 (F-MCRA Δ (MRR-hsdRMS-mcrBC) φ80dlacZΔM15 ΔlacX74 recA1 endA1 araD139 Δ (ara, leu) 7697 galU galK λ-rpsL nupG trfA tona) (Biotechnologies Epicentre, Madison, WI ) qui permet l'expression fosmide être augmentée au-dessus d'un seul exemplaire par cellule pour améliorer les rendements ADN au cours de la purification (tableau 2). Pour recombineering, le fosmide devra être transféré à la souche bactérienne SW106 (MCRA Δ (MRR-hsdRMS-mcrBC) ΔlacX74 DeOr endA1 araD139 Δ (ara, leu) 7697 rpsL recA1 nupG φ80dlacZΔM15 [λc1857 (Cro-bioA) <> Têt ] (CRO-bioA) <> araC-pBAD Cre ΔgalK) (NCI-Frederick) souche qui porte les gènes λred recombinaison homologue sous le contrôle d'un λ sensibles à la température et un répresseur inductible arabinose recombinase Cre (Tableau 2) 15.
- Commander le clone du fosmide gène d'intérêt (IGE) du GeneService (Cambridge, UK) en utilisant Wormbase comme un guide. Lors de la sélection des clones, nous choisissons ceux qui ont les pouvoirs publics indiens dans le centre de la séquence. Celles qui excluent les gènes voisins pourrait être préférable, mais pourrait être difficile à trouver.
- Culture du clone fosmide des IGE dans LB contenant 12,5 pg / ml de chloramphénicol à 37 ° C.
- Cultiver une culture de 1,5 ml de nuit de la fosmide, et mini-prep de l'ADN en utilisant l'Epicentre fosmide fosmide de préparation de kit (Biotechnologies Epicentre, Madison, WI). Nous suivons le protocole alternatif décrit dans les instructions qui consiste à ajouter le mélange Riboshredder à une étape antérieure.
- Déterminer la concentration en ADN fosmide avec un spectrophotomètre.
- Préparer le SW106 électrocompétentes cellules en cultivant une culture de 5 ml de nuit de SW106 dans un composant logiciel enfichable 14 ml bouchon du tube avec du bouillon LB avec 12,5 pg / ml de chloramphénicol à 32 ° C.
- Inoculer 1 ml dans 100 ml de LB avec du chloramphénicol dans un ballon de 2 L. Cultivez SW106 bactéries à une DO 600 de 0,6 à 0.8. Ne PAS de choc thermique.
- Pellet par centrifugation à 5000xg pendant 5 minutes, resuspendre le culot en vortexant doux, et ajouter 50 ml glacée glycérol à 10%. Répétez cette étape de lavage une fois.
- Pellet l'SW106 par centrifugation et aspirer tout sauf ~ 500 pl de chaque surnageant
- Resuspendre les granulés par tourbillonnement doux. Gel des aliquots de 100 ul dans l'azote liquide ou sur la glace sèche, et conserver à -80 ° C pour une utilisation future.
- Transformez l'ADN fosmide en électrocompétentes cellules par électroporation SW106 les bactéries avec ~ 50 ng d'ADN en utilisant une fosmide Eppendorf 2510 à 1350 volts électroporateur de 0,1 cm cuvettes fossé.
- Récupérer les bactéries dans une LB ml pendant 1 heure à 32 ° C.
- Aliquotes plaque sur des plaques LB avec du chloramphénicol (12,5 ug / ml) et incuber à 32 ° C pendant la nuit.
- Vérifiez la présence du RBE par PCR sur colonie. Cultiver une culture de 5 ml nuit dans LB avec 12,5 pg / ml de chloramphénicol à 32 ° C. Ajouter 0,5 ul de la culture à une réaction PCR standard avec les oligos d'accompagnement, et d'augmenter l'initiale d'incubation à 95 ° C à 5 minutes pour lyser les bactéries avant la PCR.
- Préparer un stock de glycérol pour stockage à long terme.
III. Insertion du gène galK par Recombineering
Dans la première étape de la modification fosmide, le gène galK est inséré dans le fosmide par recombinaison homologue, et le fosmides correctement modifié sont sélectionnés par croissance sur un milieu minimal contenant du galactose comme seule source de carbone (figure 2A). Le SW106 bactéries se développent lentement sur le support minimal et 3-5 jours sont nécessaires pour voir des colonies.
- Préparer MOPS minimale plaques supports contenant du galactose 0,2%. Les médias MOPS minimale est disponible à partir Teknova Inc (Hollister, Californie) (catalogue # M2106) mais ne pas utiliser le glucose inclus.
Les médias MOPS minime avec 0,2% de galactose (1 L)
Autoclaver 15 grammes d'agar dans 870 ml d'eau
Laisser refroidir à 55 ° C et ajouter:100 ml MOPS 10X milieu minimal 5 ml 0,2 mg / ml de d-biotine (filtré stérile) 4,5 ml 10 mg / ml de L-leucine (1%, chauffée, puis refroidie et filtrée stérile) 10 ml Galactose à 20% (en autoclave) 1 ml 12,5 mg / ml de chloramphénicol dans EtOH 2,55 ml 20% de NH 4 Cl 10 ml 0,132 M de phosphate de potassium dibasique - PCR amplifient les pMOD4 galK-G ou pMOD4 galK-GT cassettes en utilisant les amorces conçues ci-dessus. Nous avons utilisé Phusion (New England Biolabs, Ipswich, MA) ou GoTaq (Promega, Madison, WI).
- Gel de purifier la bande qui en résulte. Quantifier le rendement par le gel ou spectrophotomètre Nanodrop. Ce produit de PCR est prêt pour l'étape 3.14.
- Inoculer une culture de nuit de SW106 cellules contenant l'ADN fosmide en 5 ml LB avec du chloramphénicol (12,5 pg / pl). Croître à 32 ° C.
- Réglez secouant bain d'eau à 42 ° C à l'échauffement avec un ballon de 250 ml stérile dans le support. L'utilisation d'un bain d'eau agité est essentielle pour obtenir un rendement élevé.
- Ajouter 1 ml de culture de la nuit à 100 ml de LB et chloramphénicol dans un ballon de 2 L. Cultivez à une DO de 0,6 à 0,8. Cela prend généralement 3-4 heures.
- Transférer 50 ml de cellules SW106 à la fiole de 250 ml et de choc thermique à 42 ° C pendant exactement 20 minutes. dans un bain-marie agitant à 100 rpm Laissez les bactéries restantes à 32 ° C que le contrôle non induites.
- Refroidir les bactéries induites et non induits sur la glace pendant 10 minutes.
- Transférer les échantillons à deux tubes à centrifuger stériles et de bouletage à ~ 5000xg pendant 5 minutes.
- Décanter tout le surnageant et resuspendre le culot dans 1 ml glacée glycérol à 10% par vortex douce (c'est à dire la mise 3-4).
- Lorsque resuspendues, ajouter un autre 49 ml glacée de glycérol 10%, et les échantillons à granulés ~ 5000xg pendant 5 minutes.
- Répétez l'étape 3.9, 3.10, et 3.11 à nouveau.
- Retirez tous surnageant en inversant les tubes, et remettre le culot dans le liquide restant (environ 500 ul chacun). Aliquoter en 100 échantillons uL, geler sur la glace sèche, et conserver à -80 ° C. Ils sont bons pour quelques semaines ou mois. (En général, nous arrêter ici et d'effectuer l'électroporation le lendemain).
- Électroporation l'induit et non induits SW106 cellules avec 150 ng de produit PCR en utilisant des cuvettes 0,1 cm écart dans un ensemble Eppendorf électroporateur 2510 à 1350 volts.
- Récupérer les bactéries dans une LB mL dans un tube de 14 ml Falcon. Incuber à 32 ° C pendant 4,5 heures.
- Pellet de la bactérie dans une microcentrifugeuse à 13200 rpm pendant 15 secondes. Les bactéries sont resuspendues dans M9 et ensuite lavé deux fois enlever tout milieu riche (voir ci-dessous pour la recette).
- Milieu M9 (1 L)
- 6g Na 2 HPO 4
- 3g KH 2 PO 4
- 1g NH 4 Cl
- 0.5g de NaCl
- AUTOCLAVE
- Après le second lavage, le surnageant est éliminé et le culot est resuspendu dans 1 ml M9 avant l'étalement des dilutions en série dans le M9 (100 ul, 100 ul d'une dilution de 1:10, et 100 uL 1:100) sur des supports MOPS minime.
- Incuber 3-5 jours à 32 ° C dans un incubateur. Remarque: Soyez patient comme vrais positifs se développent lentement.
- Streak quelques colonies sur des plaques d'agar MacConkey indicateur (BD # 281810) complémenté avec 1% de galactose et le chloramphénicol 12,5 pg / mL. Toutes les colonies apparues après la dernière étape devrait être + galK, mais afin de se débarrasser de tout galK - les contaminants, il est important d'obtenir seule, lumineux colonies roses avant de procéder à la deuxième étape. Le galK - colonies sera blanc / couleur et les bactéries + galK sera rouge vif / rose due à un changement de pH résultant de galactose fermenté après une nuit d'incubation à 32 ° C.
- Choisissez une seule colonie et inoculer une LB + 5 ml de chloramphénicol culture de la nuit à la croissance à 32 ° C.
- Confirmer l'insertion du gène galK au bon endroit par PCR utilisant les oligos accompagnement. Ajouter 0,5 ul de la culture à une réaction PCR standard et d'augmenter l'initiale d'incubation à 95 ° C à 5 minutes pour lyser les bactéries. Le produit de PCR doit être upshifted en taille en raison de la présence du gène galK.
- Préparer un stock de glycérol pour le stockage.
IV. Remplacement des galK avec des séquences Tag par Recombineering
Dans cette étape le gène galK est remplacée par les séquences tag désiré et le fosmides correctement modifiés sont choisis par la sélection contre le gène galK par les toxiques du galactose analogiques deoxygalactose (DOG) (figure 2B).
- Préparer MOPS minimale plaques support contenant deoxygalactose 0,2% (DOG) et le glycérol 0,2%. Les médias MOPS minimale est disponible à partir Teknova Inc (Hollister, Californie) (catalogue # M2106) mais ne pas utiliser le glucose inclus.
MOPS minimal avec les médias CHIEN 0,2% et de glycérol (1 L)
Autoclaver 15 grammes d'agar dans 860 ml d'eau
Laisser refroidir à 55 ° C et ajouter:100 ml MOPS 10X milieu minimal 5 ml 0,2 mg / ml de d-biotine (filtré stérile) 4,5 ml 10 mg / ml de L-leucine (1%, chauffée, puis refroidie et filtrée stérile) 10 ml Deoxygalactose 20% (filtré stérile) 10 ml 20% de glycérol (autoclave) 1 ml 12,5 mg / ml de chloramphénicol dans EtOH 2,55 ml 20% de NH 4 Cl 10 ml 0,132 M de phosphate de potassium dibasique - PCR d'amplifier les fragments tag de pMOD4 GFP, pBS1761 (N terme TAP), ou pBS1479 (C-term TAP) en utilisant les oligos même que celui utilisé lors du premier tour ou en utilisant la GFP ou courte TAP-spécifiques oligos (les séquences intérieures à l'étape 1.2 ). Si vous faites des constructions multiples, il est particulièrement utile d'utiliser les oligos courts que le même produit PCR peut être utilisée pour toutes les constructions.
- Gel purifier le produit de PCR et de mesurer la concentration par le biais de gel ou de la spectrophotométrie.
- Générer induits et non induits SW106 compétente effectuant l'fosmide avec le gène inséré galK étapes suivantes 3.4 à 3.13 ci-dessus.
- Électroporation l'induit et non induits SW106 cellules avec ~ 100 ng de produit PCR en utilisant des cuvettes 0,1 cm écart dans un ensemble Eppendorf électroporateur 2510 à 1350 volts.
- Récupérer dans une LB ml dans un composant logiciel enfichable 14 ml bouchon du tube et incuber dans un shaker à 32 ° C pendant 4,5 heures.
- Lavez et diluer les bactéries comme dans les étapes 3.16 et 3.17. Plaque bactéries sur des plaques MOPS milieu minimal contenant 0,2% de 2-désoxy-galactose (DOG) et le glycérol 0,2%.
- Incuber à 32 ° C pendant trois jours.
- Quatre colonies sont utilisées pour faire 5 mlcultures de la nuit dans LB avec 12,5 pg / ml de chloramphénicol. Ils sont utilisés pour la colonie PCR comme ci-dessus pour confirmer que la cassette a été insérée. Nous utilisons à la fois la plus courte GFP / TAP oligos spécifiques et les oligos accompagnement pour démontrer l'insert à droite et bon site. GFP est ~ 800 pb et TAP est ~ 550 pb alors galK est de 1,4 kb.
- Préparer un stock de glycérol.
Ajout V. unc-119 gènes par recombinaison cre-loxP
Un moyen commun de générer des animaux transgéniques avec les fosmides modifié par l'utilisation de bombardement biolistique. Cette technique utilise des particules d'or enrobées d'ADN à introduire l'ADN fosmide dans C. elegans. Les animaux transgéniques sont généralement identifiés par l'intermédiaire de sauvetage des mutants unc-119 avec un transgène unc-119. Dans cette étape, l'UNC-119 du gène est ajouté à la colonne vertébrale fosmide en cis par cre-loxP recombinaison avec le pLoxP unc-119 plasmide (figure 2C).
- Préparer compétents SW106 bactéries portant le fosmide modifiés à l'étape 4.9 en utilisant les étapes 2.5 à 2.9. NE PAS faire à 42 ° C.
- Électroporation avec 50 ng. pLoxP unc-119 à partir d'un mini-préparation en utilisant 0,1 cuvettes fossé cm dans une Eppendorf 2510 électroporateur fixé à 1350 volts.
- Récupérer les bactéries dans LB contenant 0,1% d'arabinose pendant 1 heure à 32 ° C.
- Aliquotes plaque sur des plaques LB contenant 50μg/mL ampicilline et 12,5 pg / ml de chloramphénicol. Incuber à 32 ° C pendant la nuit. qui sélectionne pour l'intégration des pLoxP UNC-119 dans le fosmide.
- Cultivez une culture de nuit dans LB contenant 50μg/mL ampicilline et 12,5 pg / ml de chloramphénicol. Utiliser 0,5 uL de PCR vérifier la présence du gène unc-119 avec l'UNC-119 F (5'-CAAATCCGTGACCTCGACAC-3 ') et UNC-119 R (5'-CACAGTTGTTTCTCGAATTTGG-3') oligos (tableau 1).
- Faire un stock de glycérol de l'fosmide finale.
VI. Grande Échelle Préparation fosmide
Afin de faciliter l'obtention des plus grandes quantités d'ADN nécessaires à la fosmide bombardement, dans cette étape de la fosmide est transféré à la EPI300 bactéries. Cette souche a la capacité d'augmenter le nombre de copies fosmide pour accroître les rendements lors de la préparation d'ADN.
- Cultiver une culture de 5 ml de nuit de la bactérie de l'étape 5.5 dans LB contenant de l'ampicilline et le chloramphénicol à 32 ° C. Utilisez Epicentre fosmide de préparation kit pour isoler l'fosmide de 1,5 mL de la culture.
- Électroporation ~ 50 ng dans le EPI300 bactéries utilisant 0,1 cm cuvettes écart dans un ensemble Eppendorf électroporateur 2510 à 1350 volts. Le EPI300 bactéries peuvent être achetés auprès Biotechnologies Epicentre (Madison, WI).
- Récupérer les bactéries dans LB pendant 1 heure à 37 ° C. Aliquotes plaque sur gélose LB contenant 50 pg / mL et 12,5 pg / ml de chloramphénicol.
- Grow et induire la EPI300 bactéries contenant le fosmide modifié en utilisant les instructions incluses. Une culture de 50 mL induite donnera> 10 pg d'ADN purifié fosmide. Purifier l'fosmide avec le kit de préparation fosmide Epicentre.
VII. Bombardement
- Utilisez 10 ug. de l'ADN fosmide pour bombarder la souche DP38 ver tel que décrit (D. Hochbaum, A. Ferguson et A. Fisher, Jupiter, sous presse).
VIII. Les résultats représentatifs
La modification de fosmides via recombineering robuste et les taux de réussite de> 90% dans l'étape de sélection négative sont régulièrement observés 2. Ce protocole prend aussi ~ 2 semaines pour compléter ce qui rend la préparation des transgènes assez rapide. Le protocole a également été jugé par d'autres laboratoires avec succès 20.
| Oligo | Séquence |
| F TAP C-term | ATGGAAAAGAGAAGATGGAAAAAG |
| C-term TAP R | GGTTGACTTCCCCGC |
| FLAG-GFP F | ATGGATTACAAGGACGATGACGATAAGATGAG |
| FLAG-GFP R | CAAAGCTTGTGGGCTTTTGTATAG |
| F TAP N terme | ATGGCAGGCCTTGCGC |
| N terme TAP R | AAGTGCCCCGGAGGATGAGATTTTCT |
| galK F | CCTGTTGACAATTAATCATCGGCA |
| galK R | TCAGCACTGTCCTGCTCCT |
| UNC-119 F | CAAATCCGTGACCTCGACAC |
| UNC-119 R | CACAGTTGTTTCTCGAATTTGG |
Tableau 1. Oligonucléotides utilisés pour la PCR.
| Les plasmides | Source | Disponible à l'adresse |
| Fosmide clone | Geneservice Ltd | Geneservice |
| pGalK | 15 | NCI-Frederick |
| pMOD4-RT-G | 2 | Addgene |
| pMOD4-galK-G | ||
| pMOD4-galK-GT | ||
| pLoxP-UNC-119 | ||
| pMOD4-GFP | ||
| Bactéries | ||
| SW106 | 15 | NCI-Frederick |
| EPI300 | Biotechnologies Epicentre | Epicentre |
| EC100D pir-116 | ||
Tableau 2. Strain et la disponibilité des vecteurs.
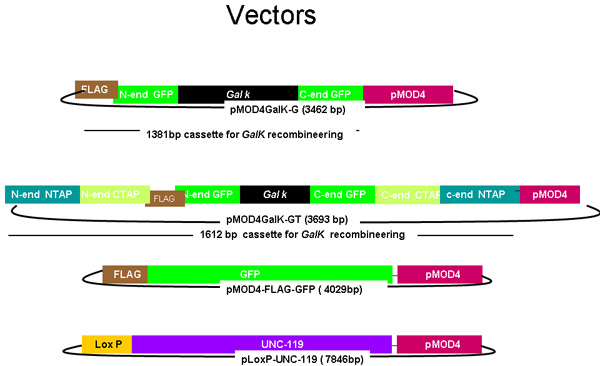
Figure 1.
Schéma de pMOD4-galK-G, et pMOD4-galK-GT, pMOD4 GFP, pLoxP-UNC-119
Le plasmide pMOD4-galK-G se compose de la cassette galK (noir) flanqué de 50 régions nucléotidique identique à la 5 'et 3' extrémités de FLAG (Brown)-GFP (green) tandis PMOD 4-galK-GT se compose des galK cassettes flanqué de deux régions d'homologie FLAG-GFP et 50 régions nucléotidique identique à l'extrémité 5 'et 3' de la N-terminal et C-terminale TAP (bleu et orange, respectivement). pMOD4-FLAG-GFP se compose de la cassette GFP complète avec une étiquette FLAG 5 'et pLoxP unc-119 se compose de l'UNC-119 séquences génomiques (violet) dans un plasmide contenant un site loxP. Tous les plasmides R6K utiliser la base de pMOD4 (rouge) épine dorsale qui est incapable de se reproduire dans SW106.
Figure 2.
Aperçu du processus recombineering galK
Figure 2A-2C chiffres distincts montrant les étapes et le temps impliqué dans recombineering utilisation de la cassette galK. Ce sont les mêmes chiffres qui sont fusionnés dans la figure 2d, mais présentés séparément pour la clarté et la facilité de lecture. Un fosmide d'intérêt est d'abord modifié dans une procédure en deux étapes impliquant l'insertion de la cassette galK flanqué de 50 pb régions d'homologie avec FLAG-GFP ou TAP (figure 2A) suivie par le remplacement de cette cassette par FLAG-GFP ou TAP ( Figure 2B). Plus tard, le marqueur unc-119 pour une utilisation dans la génération d'animaux transgéniques est inséré dans le site loxP sur l'épine dorsale fosmide (figure 2C).
Figure 2D montre une figure de la procédure a fusionné recombineering galK.
Aperçu de la procédure recombineering galK comme décrit dans le délai ci-dessus, y compris requis pour chaque étape de la fusion de la figure 2A à 2C.
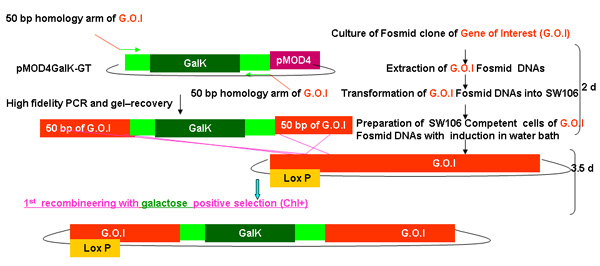
Figure 2a. L'insertion dans le galK recombineering galK.
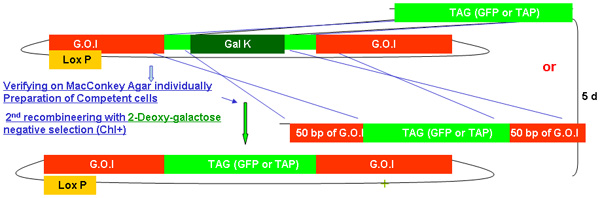
Figure 2b. Le TAG (GFP / TAP) d'insertion dans recombineering galK.
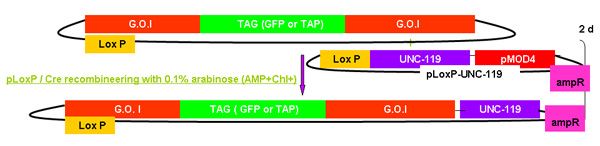
Figure 2c. L'ajout d'unc-119.
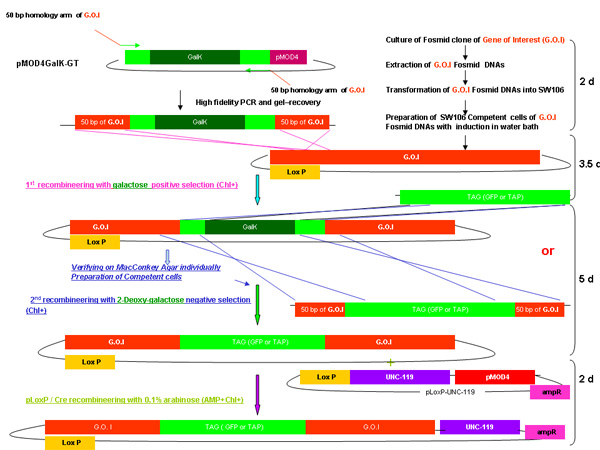
Figure 2d. L'aperçu fusionnée de recombineering galK.
Discussion
La génération des transgènes de fosmides offre l'avantage de conserver l'ensemble des éléments promoteur natif, les variants d'épissage, et 3 'UTR éléments réglementaires. Cela peut conduire à la construction d'un transgène qui est plus représentatif de la tendance d'expression maternelle, ou la construction d'un transgène fonctionnel lorsque d'autres approches échouent 5. Les transgènes qui en résulte peut mener une série de balises épitope incluant la GFP ou...
Déclarations de divulgation
Remerciements
Les auteurs tiennent à remercier Lindsey Nash pour les aider à développer la technique. Ce travail a été financé par le NIH octroi AG028977 à Alf, une subvention de projet pilote de l'Université de Pittsburgh OAIC (AG024827), et fonds d'amorçage de l'Université de Pittsburgh.
matériels
| Name | Company | Catalog Number | Comments |
| FosmidMAX kit | Epicentre Biotechnologies | FMAX046 | |
| GoTaq | Promega Corp. | M7122 | |
| MOPS Media | TEKnova, Inc. | M2120 | |
| 0.132 M Potassium phosphate solution | TEKnova, Inc. | M2102 | |
| D-galactose | Sigma-Aldrich | G0750 | |
| 2-deoxygalactose | Sigma-Aldrich | D4407 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Leucine | Sigma-Aldrich | L8000 | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Phusion DNA polymerase | New England Biolabs | F-530S | |
| MacConkey agar base | BD Biosciences | 281810 | |
| Arabinose | Sigma-Aldrich | A3131 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S5136 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Sodium chloride | Sigma-Aldrich | S5886 | |
| Glycerol | Sigma-Aldrich | G2025 | |
| Bacto Agar | BD Biosciences | 214010 |
Références
- Mello, C., Fire, A. DNA transformation. Methods Cell Biol. 48, 451-482 (1995).
- Zhang, Y., Nash, L., Fisher, A. L. A simplified, robust, and streamlined procedure for the production of C. elegans transgenes via recombineering. BMC Dev Biol. 8, 119-119 (2008).
- Antebi, A., Yeh, W. H., Tait, D., Hedgecock, E. M., Riddle, D. L. daf-12 encodes a nuclear receptor that regulates the dauer diapause and developmental age in C. elegans. Genes and Development. 14, 1512-1527 (2000).
- Snow, M. I., Larsen, P. L. Structure and expression of daf-12: a nuclear hormone receptor with three isoforms that are involved in development and aging in Caenorhabditis elegans. Biochim. Biophys. Acta. 1494, 104-116 (2000).
- Fisher, A. L., Page, K. E., Lithgow, G. J., Nash, L. The Caenorhabditis elegans K10C2.4 Gene Encodes a Member of the Fumarylacetoacetate Hydrolase Family. A CAENORHABDITIS ELEGANS MODEL OF TYPE I TYROSINEMIA. J Biol.Chem. 283, 9127-9135 (2008).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75, 855-862 (1993).
- Lehrbach, N. J. LIN-28 and the poly(U) polymerase PUP-2 regulate let-7 microRNA processing in Caenorhabditis elegans. Nat Struct Mol Biol. 16, 1016-1020 (2009).
- Tursun, B., Cochella, L., Carrera, I., Hobert, O. A toolkit and robust pipeline for the generation of fosmid-based reporter genes in C. elegans. PLoS One. 4, e4625-e4625 (2009).
- Bamps, S., Hope, I. A. Large-scale gene expression pattern analysis, in situ, in Caenorhabditis elegans. Brief. Funct. Genomic. Proteomic. , (2008).
- Dolphin, C. T., Hope, I. A. Caenorhabditis elegans reporter fusion genes generated by seamless modification of large genomic DNA clones. Nucleic Acids Res. 34, e72-e72 (2006).
- Sarov, M. A recombineering pipeline for functional genomics applied to Caenorhabditis elegans. Nat. Methods. 3, 839-844 (2006).
- Yang, X. W., Model, P., Heintz, N. Homologous recombination based modification in Escherichia coli and germline transmission in transgenic mice of a bacterial artificial chromosome. Nat Biotechnol. 15, 859-865 (1997).
- Court, D. L., Sawitzke, J. A., Thomason, L. C. Genetic engineering using homologous recombination. Annu.Rev.Genet. 36, 361-388 (2002).
- Westenberg, M., Bamps, S., Soedling, H., Hope, I. A., Dolphin, C. T. Escherichia coli MW005: lambda Red-mediated recombineering and copy-number induction of oriV-equipped constructs in a single host. BMC Biotechnol. 10, 27-27 (2010).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36-e36 (2005).
- Penfold, R. J., Pemberton, J. M. An improved suicide vector for construction of chromosomal insertion mutations in bacteria. Gene. 118, 145-146 (1992).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Puig, O. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat.Biotechnol. 17, 1030-1032 (1999).
- Achilleos, A., Wehman, A. M., Nance, J. PAR-3 mediates the initial clustering and apical localization of junction and polarity proteins during C. elegans intestinal epithelial cell polarization. Development. 137, 1833-1842 (2010).
- Maduro, M., Pilgrim, D. Identification and cloning of unc-119, a gene expressed in the Caenorhabditis elegans nervous system. Genetics. 141, 977-988 (1995).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon