JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
생산 C. 엘레간스 Transgenes galK 선택 마커
요약
대한 transgenes를 생산하는 능력 Caenorhabditis 엘레간스 기본 규제 요소가 모두 유지됩니다로서 fosmids에 의해 실시 게놈 DNA를 사용하는 것이 특히 매력적이다. transgenes의 생산을위한 간단하고 강력한 절차와 recombineering 통해이다 설명 galK 선택 마커.
초록
유전자 변형 동물의 생성은 널리 C로 활용 규정과이자 또는 탠덤 친화 정화의 세대 (TAP) 특정 유전자의 버전이 자신의 정화를 촉진하는 태그의 유전자의 표현 패턴을 연구하기 위해 GFP 융합 단백질의 사용을 포함 엘레간스 연구. 일반적으로 transgenes는 GFP의 리포터 유전자 또는 cDNA의 관심의 상류 발기인을 배치에 의해 생성되며, 이것은 종종 대표 표현식 패턴을 생산하고 있습니다. 그러나, 3 '번역되지 않은 지역이나 대안 발기인의 제어 요소와 같은 유전자 조절의 중요한 요소는,이 접근법에 의해보고 싶었어 수 있습니다. 또한 단 하나의 스플 라이스 변종은 일반적으로이 방법으로 공부하실 수 있습니다. 반면, fosmid의 DNA를 복제하여 실시 웜 게놈의 DNA의 사용 가능성이 포함 대부분의 진정한 표현의 패턴과 타이밍을 캡처하는 더 큰 능력을 허용하는 생체내의 유전자 조절에 관련된 모든 요소하지 않을 경우. fosmid DNA를 사용하여 transgenes의 생성을 촉진하기 위해, 우리는 E.을 설명 대장균의 유전자에 위치에 GFP, TAP 태그 또는 관심의 다른 시퀀스를 삽입하는 recombineering 절차를 기반으로. 절차는 높은 효율로 원하는 수정을 얻는 결과 recombineering에 긍정적이고 부정적인 선택 단계 모두 선택 마커로 galK 유전자를 사용합니다. 또한, 일반적으로 사용되는 GFP에 상동의 무기 둘러싸인과 융합 유전자를 TAP galK 유전자를 포함하는 plasmids는 GFP 또는 탭 융합 단백질을 생성할 때 50 % oligos의 비용을 줄일 수있는 사용할 수 있습니다. 이러한 plasmids은 광범위한 PCR 제품 정화의 필요성을 걸로 R6K 복제 원점을 사용합니다. 마지막으로, 우리는 또한 fosmid 직접 주입 또는 유전자 변형 동물을 생성하는 벌레로 폭격을 할 수있는 fosmid 백본에 UNC - 119 마커를 통합하는 방법을 보여줍니다. 이 동영상은이 방법을 사용 recombineering 통해 transgene을 생성에 관련된 절차를 보여줍니다.
프로토콜
개요
형질 C.의 생성에 사용되는 많은 transgenes 엘레간스는 모터의 시퀀스로 구성 아마도 cDNA 유전자 박사 앤디 화재 1의 실험실에 의해 생성된 벡터 중 하나에 복제. 이러한 transgenes 자주 GFP의 리포터 유전자를 생산하거나 원하는 패턴의 cDNA를 표현 관련하여 성공하는 동안 이러한 transgenes는 다른 발기인, 확장기 요소 및 컨트롤에서 중요한 역할을 3 '번역되지 않은 지역 (UTR) 요소를 부족 수 있습니다 생체내 2 유전자의 표현. 예를 들어, DAF - 12와 파 - 1 유전자 모두는 3,4,5을 구성 발기인에보고 있던 근위 발기인 이외의 거짓말 중요한 향상제 요소를합니다. 더 많은 transgene 구조에 적절한 microRNA 유전자 6,7,8에 의해 규제를 방지 UNC - 54 3'UTR을 사용합니다. 따라서 웜 게놈의 DNA의 큰 세그먼트와 transgenes을 생성하는 것은 발기인, 스플 라이스 변종, 3 'UTR 제어 요소를 모두 캡처를위한 이상적인 것입니다. 최근 C. 게놈 DNA의 ~ 40 이하 지역으로 구성되어 있으며 거의 게놈 전체를 커버 엘레간스 fosmid 라이브러리 구축되었습니다. 웜 게놈의 DNA의 사용은 특정 유전자 2,8,9,10,11의 진정한 표현의 패턴과 타이밍을 캡처하는 더 큰 능력이 fosmid의 DNA 클론 결과에 의해 실시.
그러나 게놈 DNA 큰 지역과 함께 작업하는 것은 이러한 표준 분자 생물학 기법 12 사용 큰 어려움으로 실천 과제를 포즈. 이러한 한계를 극복하기 위해, 기술 E.에 상동 재조합을 통해 fosmids 또는 박테리아 인공 염색체를 수정 대장균이 개발되어 있고 12,13을 recombineering 칭했다 있습니다. Recombineering는 C.에 의해 실시 유전자의 어떤 위치에 GFP, 탠덤 선호도 정화 (TAP) - 태그, 또는 관심의 다른 시퀀스의 원활한 삽입이 가능합니다 엘레간스는 클론 2,10,14을 fosmid. 상동 재조합 특별히 수정 E.의 대상 사이트 및 대상 DNA와 상동 50 BP 지역 둘러싸인 PCR 제품 사이에 발생 콜리 변종.
우리는 최근 C.의 수정을위한 2 단계 절차를 설명했습니다 원하는 위치에 galK의 유전자를 삽입하고 다음 원하는 순서 2와이 유전자를 대체 관련 recombineering하여 엘레간스의 fosmids. galK 유전자는이 선택적 성장 매체 15 사용을 통해과에 대해 선택할 수로 그 과정에서 두 단계에 대한 효과적인 선택 마커 역할을합니다. fosmid 수정의 첫 번째 단계에서 galK 유전자가 원하는 위치에 상동 재조합을 통해 삽입하고 올바르게 수정 fosmids은 탄소 소스 2,15으로 갈락토 오스를 활용할 수있는 능력에 대해 긍정적인 선택에 의해 식별됩니다. 두 번째 단계에서 galK 유전자가 원하는 순서로 대체됩니다, 그리고 정확하게 수정 fosmids는 galK + 세균 2,15 죽이는 독성 갈락토 오스 파생 deoxygalactose의 사용을 통해 galK 유전자에 대한 부정적인 선택을 통해 식별됩니다. galK의 장점은 대신 고효율 2,15과 원하는 수정을 얻기에서 별도의 각 단계에 유전자, 그리고 결과를 가지고 다른 마커의 긍정과 부정 선택 단계에 사용할 수있는 단일 유전자의 능력입니다.
C.이 기술의 응용을 촉진하기 엘레간스 연구, 우리는 사용 가능한 자원을 몇 가지 변경했습니다. 첫째, GFP 및 TAP 태그는 일반적으로 웜 transgenes를 생성하는 데 사용됩니다, 그래서 우리는 galK 유전자의 원천 역할을 pMOD4 galK - G와 pMOD4 galK - GT plasmids로이 태그의 각 상동 50 BP 지역에 지어진 2. 이들 지역은 다소 비싼 oligos의 두 번째 세트를 위해 필요를 저장 fosmid 수정 두 단계에 사용할 oligos의 단일 집합을 허용합니다. 둘째, 이러한 plasmids은 플라스미드 부모 recombineering에 사용되는 박테리아에서 복제 할 수 없습니다로서 부모 플라스미드 또는 광범위한 PCR 제품 정화를 소화 필요 걸로 R6K 복제 원점을 사용하고, 그러한 EC100이 특수 종자에 복제할 수 있습니다 , 16 (표 1 및 표 2). 형질 C. 생성의 마지막으로, 일반적인 방법 엘레간스은 UNC - 119 변이 17 구조를 통해 유전자 변형 벌레에 대한 선택에 의해 다음 biolistic 폭격의 사용을 통해이다. 폭격으로 fosmids 호환하기 위해서, 우리는 fosmid 백본 2에서 UNC - 119 마커를 통합하는 데 사용할 수있는 pLoxP UNC - 119 플라스미드를 개발했습니다.
I. Oligo 디자인
recombineering하여 원하는 시퀀스는 유전자 내의 모든 사이트에 삽입하실 수 있습니다. 일반적인 사이트는 5 '끝에 3'기능 도메인, 스플 라이스 변종, 또는 프로 테아제에 의해 그러한 절단과 같은 사후 translational 수정에 따라 끝 부분에 있습니다. 플라스미드는 개시 코돈을 포함하여 3 '정지 코돈 (그림 1) 부족으로 우리 연구실에서 만든 플라스미드 pMOD4 GFP는 어떤 사이트에서 깃발 태그 GFP를 삽입하는 데 사용할 수 있습니다. 반면, TAP 태그 5 '와 3'정화 18,19 중에 사용 TEV 절단으로 인한 fusions 특정 버전이 있습니다.
- 유전자 내의 태그 삽입의 사이트를 계획합니다. 삽입 사이트를 고려할 때 대체 발기인, 기능 도메인, 대안 깔끔히 및 사후 translational 수정을 고려하십시오. 다른 삽입 사이트는 모두, 하나, 또는 특정 유전자의 일부 isoforms에 태그를 사용할 수 있습니다. 삽입 지점의 상류와 하류 50 BP 영역을 식별합니다.
- oligos (표 1)을 디자인합니다. 100 NM 규모 중 하나는 - 통합 DNA 기술에서 젤 정화의 oligos 또는 Ultramer의 oligos는 프로 시저에 사용할 수 있습니다.
galK의 recombineering을 수행하기 위해서는 pMOD4 galK - G와 pMOD4 모두 존재 galK 카세트,에 바인딩 원하는 수정할 수 사이트로 이러한 primers의 3 '끝에 측면을 노릴 지역 50 BP의 상동과 galK의 primers를 디자인해야합니다 galK - GT (그림 1). 앞으로 oligo는 5' 것입니다 ------- 50 BP의 상동 ------- CCTGTTGACAATTAATCATCGGCA - 3 '와 5' ------- 보완 스트랜드 50 BP의 상동으로 반대 하나 - ------ TCAGCACTGTCCTGCTCCT - 3.
GFP와 galK의 recombineering을 수행하기 위해서는 (그림 1) 수정할 수있는 원하는 사이트를 측면 면적 50 BP의 상동과 pMOD4 galK - G 또는 pMOD4 galK - GT primers를 디자인해야합니다. 프레임의 융합 단백질을 유지해야합니다. 원하는 경우 ATG는 삭제할 수 있습니다. 이 primers의 3 '끝 galK 카세트 측면을 노릴 GFP의 상동의 지역에 바인딩합니다. 참고 : GFP의 처음이자 마지막 codons가 읽기 프레임을 증명하기 위해 밑줄 수 있습니다. 앞으로 oligo는 5' 것입니다 ------- 50 BP의 상동 ------- 보완에 -3 '세'와 반대로 한 5' ------- 50 BP의 상동을 GATTACAAGGACGATGACGATAAGATGAG ATG 스트랜드 ------- CAA AGCTTGTGGGCTTTTGTATAG - 3 '
galK C - 용어 탭 recombineering을 수행하기 위해서는 (그림 1) 수정할 수있는 원하는 사이트를 측면 면적 50 BP의 상동과 pMOD4 galK - GT primers를 디자인해야합니다. 프레임의 융합 단백질을 유지해야합니다. 이 primers의 3 '끝 galK 카세트 측면을 노릴 탭 상동의 지역에 바인딩합니다. 참고 : TAP의 처음이자 마지막 codons가 읽기 프레임을 증명하기 위해 밑줄 수 있습니다. 앞으로 oligo는 5' 것입니다 ------- 50 BP의 상동 ------ ATG GAAAAGAGAAGATGGAAAAAG - -3 '와 반대로 한 5' ------- 보완 스트랜드 50 BP의 상동 - ------ GGT TGACTTCCCCGC의 -3 '
galK N - 용어 탭 recombineering을 수행하기 위해서는 (그림 1) 수정할 수있는 원하는 사이트를 측면 면적 50 BP의 상동과 pMOD4 galK - GT primers를 디자인해야합니다. 프레임의 융합 단백질을 유지해야합니다. 이 primers의 3 '끝 galK 카세트 측면을 노릴 탭 상동의 지역에 바인딩합니다. 참고 : TAP의 처음이자 마지막 codons가 읽기 프레임을 증명하기 위해 밑줄 수 있습니다. 앞으로 oligo는 5' 것입니다 ------- 50 BP의 상동 ------ ATG GCAGGCCTTGCGC - -3 '와 반대로 한 5' ------- 보완 스트랜드 50 BP의 상동 - ------ AAG TGCCCCGGAGGATGAGATTTTCT의 -3 ' - 나중 단계에서뿐만 아니라 fosmid의 시퀀싱을위한 PCR에 대한 oligos 측면을 노릴 세트를 생성합니다. 이들은 상류와 하류 삽입 사이트의 ~ 100 BP를 바인딩해야 표준 PCR의 oligos입니다.
II. SW016 박테리아에 Fosmid 전송
C.에서 fosmids 엘레간스 fosmid 라이브러리는 EPI300 세균 변형 (F - mcrA Δ (mrr - hsdRMS - mcrBC) φ80dlacZΔM15 ΔlacX74 recA1 endA1 araD139 Δ (아라, 레우) 7697 galU galK λ - rpsL nupG trfA 토나) (Epicentre의 Biotechnologies, 매디슨, WI에서 제공하는 ) 이것은 fosmid 표현은 정화 (표 2) 중 DNA의 수율을 개선하기 위해 셀 당 하나의 사본 이상 증가 수 있습니다. recombineering 들어, fosmid는 SW106 박테리아 스트레인 (mcrA Δ (mrr - hsdRMS - mcrBC) ΔlacX74 deoR endA1 araD139 Δ (아라, 레우) 7697 rpsL recA1 nupG φ80dlacZΔM15 [λc1857 (CRO - bioA) <> Tet로 전송해야합니다 억제 온도에 민감한 λ와 arabinose inducible CRE recombinase (표 2) 15의 통제하에 λred 상동 재조합 유전자를 전달] (CRO - bioA) <> araC - PBAD Cre 호텔 ΔgalK) (NCI - 프레드릭) 변형.
- Wormb를 사용 GeneService에서 관심 진 (고이)의 fosmid 클론 (캠브리지, 영국)을 주문가이드로 ASE. 클론을 선택할 때, 우리는 순서의 중앙에 고이을 가지고 사람을 선택합니다. 인접 유전자를 제외 것들은 바람직있을 수 있지만 찾기 힘들 수 있습니다.
- 37 12.5 μg / ML chloramphenicol을 포함한 LB에 고이의 fosmid 클론 문화 ° C.
- fosmid의 1.5 ML 야간 문화, 그리고 Epicentre fosmid 준비 키트 (Epicentre의 Biotechnologies, 매디슨, WI)를 사용하여 미니 준비 fosmid DNA를 성장. 우리는 이전 단계에서 Riboshredder 믹스를 추가 관련 지침에 설명되어있는 다른 프로토콜을 따르십시오.
- 분광 광도계로 fosmid DNA 농도를 결정합니다.
- 32 ° C.에서 12.5 μg / ML chloramphenicol와 LB의 국물과 함께 14 ML 스냅 캡 튜브 SW106의 5 ML 야간 문화를 재배하여 electrocompetent SW106 세포 준비
- 2 L 플라스크에 chloramphenicol과 LB 100 ML에 한 ML의 예방. OD 600 0.6-0.8로 SW106 박테리아를 성장. 열 충격하지 마십시오.
- 5 분 5000xg에서 원심 분리하여 펠렛은 부드러운 vortexing하여 펠렛을 resuspend, 50 ML 얼음처럼 차가운 10 % 글리세롤을 추가합니다. 한때이 세척 단계를 반복합니다.
- 펠렛 원심 분리에 의해 SW106과 대기음을 제외한 모든 각 뜨는 500 μl를 ~
- 부드러운 vortexing하여 알약을 Resuspend. 100 μl 액체 질소 또는 드라이 아이스에 aliquots하고, -80 ° C에서 저장 나중에 사용하기 위해 고정.
- 0.1 cm 갑 cuvettes에 1,350 볼트에서 Eppendorf 2510 electroporator를 사용하여 DNA의 fosmid ~ 50 NG와 박테리아를 electroporating하여 electrocompetent SW106 세포에 fosmid DNA를 변환.
- 32 ° C. 1 시간 1 ML의 LB에서 박테리아를 복구
- 32 ° C 하룻밤에 chloramphenicol (12.5 μg / ML) 및 부화와 LB 플레이트에 플레이트 aliquots.
- 식민지 PCR로 고이의 존재를 확인합니다. 32 ° C.에서 12.5 μg / ML chloramphenicol와 LB의 5 ML 야간 문화를 성장 측면 oligos과 표준 PCR 반응에 문화의 0.5 μL를 추가하고, PCR하기 전에 박테리아를 lyse 5 분 초기 95 ° C 부화를 향상시킬 수 있습니다.
- 장기 저장을위한 글리세롤 주식을 준비합니다.
III. Recombineering로 galK 진 삽입
fosmid 수정의 첫 번째 단계에서 galK 유전자는 상동 재조합에 의해 fosmid에 삽입하고, 올바르게 수정 fosmids는 유일한 탄소 소스 (그림 2A)로 갈락토 오스를 포함한 최소한의 미디어에 성장에 의해 선택됩니다. SW106 박테리아는 최소한의 미디어에 천천히 성장하고 3-5일은 식민지를 참조해야합니다.
- 0.2 % 갈락토 오스를 포함한 맙스 최소한의 미디어 접시를 준비합니다. 맙스 최소한 미디어 Teknova 주식 회사 (홀리스터, CA) (카탈로그 # M2106)에서 구할 수 있지만 포함된 포도당을 사용하지 마십시오.
0.2 % 갈락토 오스 (1 패)로 처리한다 최소한의 미디어
압력솥 870 ML의 물에 15g의 한천
55 쿨 ° C 및 추가 :100 ML 10X 맙스 최소한의 미디어 5 ML 0.2 MG / ML D - 비오틴 (살균 필터) 4.5 ML 10 MG / ML L - 류신 (1 %, 온수, 그때 냉각 및 무균 여과) 10 ML 20 % 갈락토 오스 (autoclaved) 1 ML EtOH에서 12.5 MG / ML Chloramphenicol 2.55 ML 20% NH 4 Club 호텔 10 ML 0.132 M 이염의 인산 나트륨 - PCR은 pMOD4 galK - G 이상 설계된 primers를 사용하여 pMOD4 galK - GT의 카세트를 증폭. 우리는 Phusion (뉴 잉글랜드 Biolabs, 입스 위치, MA) 또는 GoTaq을 (Promega, 매디슨, WI) 사용하고 있습니다.
- 젤 결과 밴드를 정화. 젤 또는 Nanodrop 분광 광도계에 의해 수율을 수량. 이 PCR 제품은 3.14 단계에 대한 준비가되었습니다.
- chloramphenicol (12.5 μg / μL) 5 ML의 LB에 fosmid DNA를 포함하는 SW106 세포의 야간 문화를 예방. 32 ° C.에서 성장
- 42 물 목욕을 흔들어 설정 ° C로 홀더에 멸균 250 ML 플라스크와 함께 워밍업. 떨고 물을 욕조의 사용은 높은 효율을 얻을 수 중요합니다.
- 2 L 플라스크에 LB 및 chloramphenicol 100 ML에 밤새 문화 1 ML을 추가합니다. OD 0.6-0.8로 성장합니다. 이것은 일반적으로 3~4시간 걸립니다.
- 42 250 ML의 술병과 열 충격을 SW106 세포의 50 ML을 전송 ° C 정확히 20 분. 100 RPM에서 흔들림 waterbath에 uninduced 제어로 32 ° C에서 남아있는 박테리아를 둡니다.
- 10 분 얼음 유도하고 uninduced 박테리아를 쿨.
- 5 분 ~ 5000xg 두 멸균 원심 분리기 튜브 및 펠렛에 샘플을 전송합니다.
- 뜨는 모두를 붓고 부드러운 vortexing (즉, 설정 3-4)로 1 ML 얼음처럼 차가운 10 % 글리세롤의 펠렛을 resuspend.
- resuspended 때, 또 다른 49 ML 얼음처럼 차가운 10 % 글리세롤을 추가하고, 펠렛 ~ 5000의 샘플5 분 XG.
- 다시 단계 3.9, 3.10 및 3.11를 반복합니다.
- 튜브 반전하여 모든 표면에 뜨는를 제거하고, 나머지 액체의 펠렛을 (약 500 μL 각) resuspend. 100 μL 샘플로 나누어지는이 드라이 아이스에 고정하고, -80 ° C.에 저장 이러한 개월 주 좋다. (우리가 일반적으로 여기에 중단하고 다음날 electroporation을 수행합니다.)
- 1,350 볼트에서 Eppendorf 2510 electroporator 세트에 0.1 cm의 간격 cuvettes를 사용하여 PCR 제품 150 NG로 유도하고 uninduced SW106 세포를 Electroporate.
- 14 ML 팔콘 튜브 1 ML의 LB에있는 박테리아를 복구합니다. 4.5 시간 동안 32 ° C에서 알을 품다.
- 펠렛은 15 초 동안 13,200 rpm으로 microfuge에 박테리아. 박테리아는 M9의 resuspended 후 두 번있는 다양한 매체를 (제조법은 아래 참조) 제거 세탁 있습니다.
- M9 중간 (1 L)
- 6g 나 2 HPO 4
- 3g KH 2 PO 4
- 1g NH 4 Club 호텔
- 0.5g NaCl
- 압력솥
- 두 번째 세척 후, 뜨는이 제거되고 펠렛은 맙스 최소한의 미디어에 M9 (100 μL, 1시 10분 희석 100 μL, 100 μL 1:100)에서 시리얼 dilutions을 도금 전에 1 ML M9에 resuspended 있습니다.
- 32 ° 인큐베이터에서 C에서 3~5일 품어. 참고 : 진정한 긍정적이 서서히 증가로 환자하십시오.
- MacConkey 한천 표시기 플레이트에 리크 몇 식민지가 (BD # 281810) 1 % 갈락토 오스와 chloramphenicol 12.5 μg / ML과 보완. 마지막 단계 이후에 나타나는 식민지의 모든 galK +는해야하지만, 어떤 galK 제거하기 위해 - 오염 물질, 그것은 두 번째 단계로 진행하기 전에 하나, 밝은 분홍색 식민지를 얻는 것이 중요합니다. galK - 식민지 무색 / 화이트되며 galK +는 박테리아가 32에서 하룻밤 배양 후 발효 갈락토 오스로 인한 산도 변화에 밝은 핑크 / 레드 인해됩니다 ° C.
- 하나의 식민지 선택 및 32에 성장을위한 5 ML의 LB + chloramphenicol 야간 문화를 예방 ° C.
- 측면 oligos를 사용하여 PCR을 통해 적절한 위치에 galK 유전자의 삽입을 확인합니다. 표준 PCR 반응에 문화의 0.5 μL를 추가하고 박테리아를 lyse 5 분 초기 95 ° C 부화를 향상시킬 수 있습니다. PCR 제품은 galK 유전자의 존재로 인해 크기 upshifted해야합니다.
- 저장 글리세롤 주식을 준비합니다.
IV. Recombineering의 태그 시퀀스로 galK 교체
이 단계에서는 galK 유전자가 원하는 태그 시퀀스와 정확하게 수정 fosmids이 독성 갈락토 오스 아날로그 deoxygalactose (개) (그림 2B)로 galK 유전자에 대한 선택에 의해 선택에 의해 대체됩니다.
- 0.2 % deoxygalactose (개)와 0.2 % 글리세롤을 포함 맙스 최소한의 미디어 접시를 준비합니다. 맙스 최소한 미디어 Teknova 주식 회사 (홀리스터, CA) (카탈로그 # M2106)에서 구할 수 있지만 포함된 포도당을 사용하지 마십시오.
0.2 % 도크 및 글리세롤 (1 패)로 처리한다 최소한의 미디어
압력솥 860 ML의 물에 15g의 한천
55 쿨 ° C 및 추가 :100 ML 10X 맙스 최소한의 미디어 5 ML 0.2 MG / ML D - 비오틴 (살균 필터) 4.5 ML 10 MG / ML L - 류신 (1 %, 온수, 그때 냉각 및 무균 여과) 10 ML 20 % deoxygalactose (살균 필터) 10 ML 20 % 글리세롤 (autoclaved) 1 ML EtOH에서 12.5 MG / ML Chloramphenicol 2.55 ML 20% NH 4 Club 호텔 10 ML 0.132 M 이염의 인산 나트륨 - PCR은 첫 번째 라운드에서 사용하거나 짧은 GFP 또는 TAP 특정 oligos를 (단계 1.2의 내부 시퀀스를 사용하여 동일한 oligos를 사용 pMOD4 GFP, pBS1761 (N - 용어 TAP) 또는 pBS1479 (C - 용어 TAP)에서 태그 조각을 증폭 ). 여러 개의 구성을하고있다면, 그것은 동일한 PCR 제품이 구성의 모든 사용할 수로 짧은 oligos를 사용하는 특히 유용합니다.
- 젤 PCR 제품을 정화하고 젤 또는 분광 광도법을 통해 농도를 측정합니다.
- 유도 이상 다음과 같은 단계를 3.4-3.13를 삽입 galK 유전자와 fosmid을 운반 능력 SW106를 uninduced 생성합니다.
- 1,350 볼트에서 Eppendorf 2510 electroporator 세트에 0.1 cm의 간격 cuvettes를 사용하여 PCR 제품의 ~ 100 NG로 유도하고 uninduced SW106 세포를 Electroporate.
- 4.5 시간 동안 32 ° C 통에 14 ML 스냅 캡 튜브와 부화 1 ML의 LB에서 복구할 수 있습니다.
- 씻고 단계 3.16 및 3.17에서와 같이 박테리아를 희석. 플레이트 0.2 2 데옥시 - 갈락토 오스 % (개)와 0.2 % 글리세롤을 포함 맙스 최소한의 미디어 접시에 박테리아.
- 사흘 동안 32 ° C에서 알을 품다.
- 네 식민지 5 ML하기 위해 사용됩니다12.5 μg / ML의 chloramphenicol과 LB에서 하룻밤 문화. 카세트가 삽입되었는지 확인하기 위해 위와 같이 이들은 식민지 PCR에 사용됩니다. 우리는 모두 짧은 GFP를 사용하여 / 오른쪽 넣고 바로 사이트를 설명하는 특정 oligos과 측면 oligos를 누릅니다. GFP는 ~ 800 BP입니다 galK은 1.4 KB입니다 반면 TAP는 ~ 550 BP입니다.
- 글리세롤 주식을 준비합니다.
CRE - loxP 재조합에 의해 UNC - 119 유전자의 V. 애디션
수정된 fosmids와 유전자 변형 동물을 생성하는 일반적인 수단 biolistic 폭격의 사용을 통해이다. 이 기술은 C.에 fosmid DNA를 소개하는 DNA - 코팅 금 입자를 사용하여 엘레간스. 유전자 변형 동물은 일반적으로 UNC - 119의 transgene와 UNC - 119 돌연변이의 구조를 통해 식별됩니다. 이 단계에서, UNC - 119 유전자는 pLoxP와 CRE - loxP 재조합에 의해 CIS의 fosmid 백본에 추가됩니다 UNC - 119 플라스미드 (그림 2C).
- 단계 2.5-2.9를 사용 단계 4.9에서 수정된 fosmid을 운반 능력 SW106 박테리아를 준비합니다. 42 유도하지 마십시오 ° C.
- 50 NG와 Electroporate. pLoxP 1,350 볼트에서 설정 Eppendorf 2510 electroporator에 0.1 cm의 간격 cuvettes을 사용하여 미니 초등학교에서 UNC - 119.
- 32 ° C. 1 시간 동안 0.1 % arabinose를 포함 LB에 박테리아를 복구
- 50μg/mL 암피실린 및 12.5 μg / ML의 chloramphenicol을 포함한 LB 플레이트에 플레이트 aliquots. ° C 야간 32에서 품어. 어느 fosmid에 UNC - 119 pLoxP 통합을위한 선택합니다.
- 50μg/mL 암피실린 및 12.5 μg / ML chloramphenicol을 포함한 LB에 야간 문화를 성장. UNC - 119 F (5' - CAAATCCGTGACCTCGACAC - 3 ')와 UNC - 119 R (5' - CACAGTTGTTTCTCGAATTTGG - 3') oligos (표 1)와 UNC - 119 유전자의 존재를 확인 PCR을위한 0.5 μL를 사용합니다.
- 최종 fosmid의 글리세롤 주식을합니다.
VI. 대규모 Fosmid 준비
충격에 필요한 fosmid DNA의 큰 수량을 취득 촉진하기 위해,이 단계에서 fosmid은 EPI300 박테리아로 전송됩니다. 이 변형은 DNA 준비 중에 수율을 높일 수있는 fosmid 사본 번호를 높일 수있는 능력이있다.
- 32 ° C.에 암피실린 및 chloramphenicol을 포함한 LB 단계에서 5.5에서 박테리아의 5 ML 야간 문화를 성장 문화의 1.5 ML에서 fosmid을 분리 Epicentre fosmid 준비 키트를 사용하십시오.
- Electroporate ~ 1,350 볼트에서 Eppendorf 2510 electroporator 세트에 0.1 cm의 간격 cuvettes를 사용하여 EPI300 박테리아에 50 NG 있습니다. EPI300 박테리아는 Epicentre의 Biotechnologies (매디슨, WI)에서 구입하실 수 있습니다.
- 37 1 시간 LB에서 박테리아를 복구 ° C. 50 μg / ML 및 12.5 μg / ML의 chloramphenicol을 포함한 LB 한천의 플레이트 aliquots.
- 포함된 지침을 사용하여 수정된 fosmid 들어있는 EPI300 박테리아를 성장하고 유도. 50 ML 유도 문화 fosmid DNA를 정화의> 10 μg을 제공할 것입니다. Epicentre fosmid 준비 키트 fosmid 정화.
VII. 충격
- 10 μg을 사용합니다. 충격에 fosmid DNA의 DP38 웜 변종은 (D. Hochbaum, A. 퍼거슨, 그리고 A. 피셔, 조브, 언론)을 설명했다.
VIII. 대표 결과
부정적인 선택 단계에서 강력하고> 90 %의 성공 속도를 recombineering 통해 fosmids의 변경은 정기적으로 2 관찰하고 있습니다. 이 프로토콜은 또한 ~ 2 주 상당히 빠른 transgenes의 준비를하게하는 완료 걸립니다. 프로토콜은 또한 성공에 20의 다른 실험실에 의해 시도되었습니다.
| Oligo | 순서 |
| C - 장기 탭 F | ATGGAAAAGAGAAGATGGAAAAAG |
| C - TAP 용어 R | GGTTGACTTCCCCGC |
| 플래그 - GFP F | ATGGATTACAAGGACGATGACGATAAGATGAG |
| 플래그 - GFP R | CAAAGCTTGTGGGCTTTTGTATAG |
| N - 장기 탭 F | ATGGCAGGCCTTGCGC |
| N - 용어 TAP R | AAGTGCCCCGGAGGATGAGATTTTCT |
| galK F | CCTGTTGACAATTAATCATCGGCA |
| galK R | TCAGCACTGTCCTGCTCCT |
| UNC - 119 F | CAAATCCGTGACCTCGACAC |
| UNC - 119 R | CACAGTTGTTTCTCGAATTTGG |
PCR에 사용되는 표 1. Oligonucleotides.
| Plasmids | 출처 | 에서 구할 수 |
| Fosmid 클론 | Geneservice (주) | Geneservice |
| pGalK | 15 | NCI - 프레드릭 |
| pMOD4 - RT - G | 2 | Addgene |
| pMOD4 - galK - G | ||
| pMOD4 - galK - GT | ||
| pLoxP - UNC - 119 | ||
| pMOD4 - GFP | ||
| 박테리아 | ||
| SW106 | 15 | NCI - 프레드릭 |
| EPI300 | Epicentre의 Biotechnologies | Epicentre |
| EC100D PIR - 116 | ||
표 2. 스트레인 및 벡터 상황.
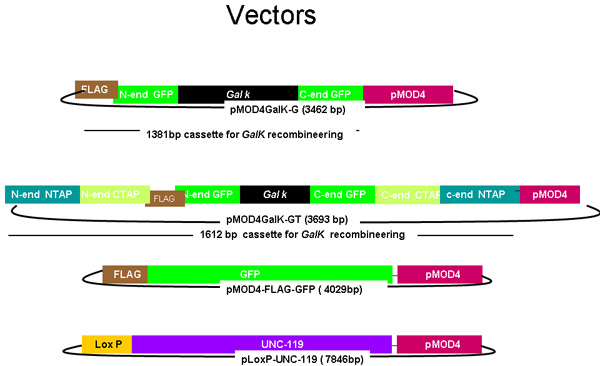
그림 1.
pMOD4 - galK - G 및 pMOD4 - galK - GT, pMOD4 GFP, pLoxP - UNC - 119 다이어그램
pMOD 4 galK - GT는 galK로 구성되어있는 반면 pMOD4 - galk - G 플라스미드는 5 '와 3'국기의 종료 (브라운) - GFP (녹색)과 동일 50 뉴클레오 티드 지역 둘러싸인 galK 카세트 (블랙)로 구성되어 있습니다 5 '및 3'N - 터미널과 C - 터미널 TAP (각각 파란색과 오렌지색)의 종료와 동일 플래그 - GFP의 상동 지역 50 뉴클레오 티드 지역 모두 둘러싸인 카세트. pMOD4 - 플래그 - GFP는 5 '플래그 태그와 pLoxP UNC - 119 loxP 사이트를 포함하는 플라스미드의 UNC - 119 게놈 시퀀스 (자주색)로 구성되어있는 전체 GFP 카세트로 구성되어 있습니다. 모든 plasmids은 SW106의 복제 수없는 R6K 기반 pMOD4 (적색) 백본을 활용한다.
그림 2.
galK의 recombineering 프로세스의 개요
galK 카세트를 사용하여 recombineering에 관련된 단계와 시간을 보여주는 그림 2A - 2C 구분 인물. 이들은 그림 2D에 합병지만, 선명도와 읽기의 용이성을 위해 별도로 제공되는 동일한 수치입니다. 관심 fosmid 먼저 (깃발 GFP 또는 TAP하여이 카세트의 교체 뒤에 깃발 GFP 또는 TAP (그림 2A)에 상동 50 BP 지역 둘러싸인 galK 카세트의 삽입과 관련된 두 단계 절차 수정 그림 2B). 나중에 유전자 변형 동물을 생성에 사용하기 위해 UNC - 119 마커는 fosmid 백본 (그림 2C)에 LoxP 사이트에 삽입됩니다.
그림 2D는 galK의 recombineering 절차의 병합 모습을 보여줍니다.
그림 2A - 2C를 병합에서 각 단계에 필요한 시간을 포함하여 상기에서 설명한 galK의 recombineering 절차의 개요.
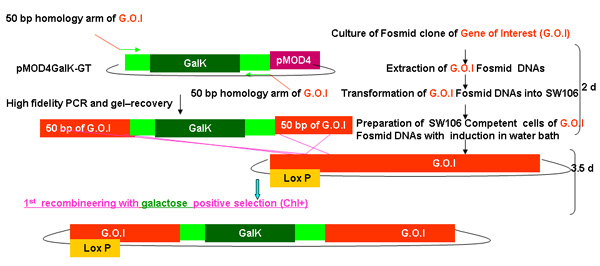
그림 2A. galk의 recombineering의 galk 삽입.
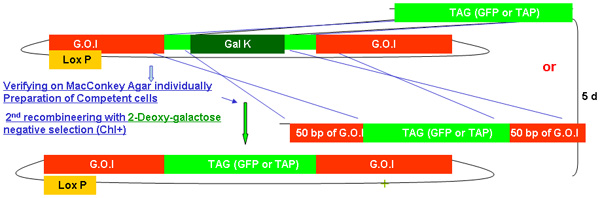
그림 2B. galk의 recombineering의 태그 (GFP / TAP) 삽입.
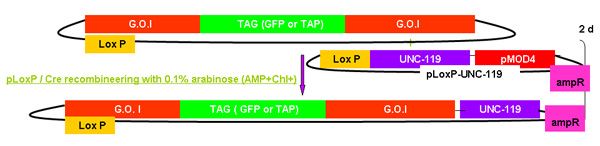
그림 2C. UNC - 119 또한.
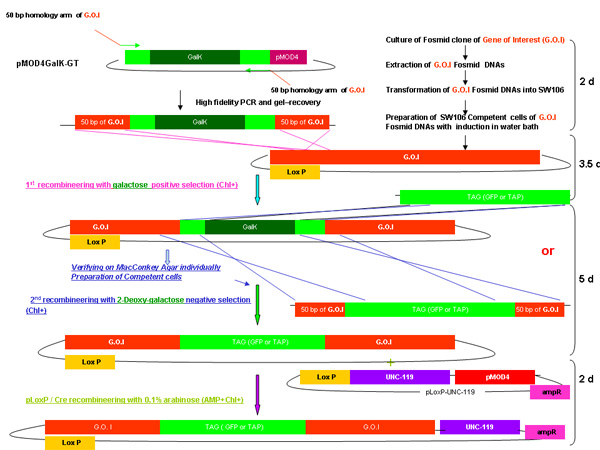
그림 2D. galk의 recombineering의 합병 개요.
토론
fosmids에서 transgenes의 생성은 기본 발기인 요소, 스플 라이스 변종, 3 'UTR 규제 요소를 모두 유지의 혜택을 제공합니다. 이것은 기본 표현식 패턴, 또는 다른 접근법 5 실패 기능성 transgene의 건설이 더 반영하는 transgene의 건설로 이어질 수 있습니다. 결과 transgenes는 GFP 또는 TAP 태그를 포함한 에피토프 태그의 다양한 휴대하실 수 있습니다.
transgenes의 건설은 모든 SW106...
공개
감사의 말
저자는 기술을 개발에 대한 도움 린지 내쉬 감사하고 싶습니다. 이 작품은 ALF, 피츠버그 대학에서 피츠버그 OAIC 대학 (AG024827) 및 씨앗 기금에서 파일럿 프로젝트 부여하는 NIH 부여 AG028977에 의해 투자되었다.
자료
| Name | Company | Catalog Number | Comments |
| FosmidMAX kit | Epicentre Biotechnologies | FMAX046 | |
| GoTaq | Promega Corp. | M7122 | |
| MOPS Media | TEKnova, Inc. | M2120 | |
| 0.132 M Potassium phosphate solution | TEKnova, Inc. | M2102 | |
| D-galactose | Sigma-Aldrich | G0750 | |
| 2-deoxygalactose | Sigma-Aldrich | D4407 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Leucine | Sigma-Aldrich | L8000 | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Phusion DNA polymerase | New England Biolabs | F-530S | |
| MacConkey agar base | BD Biosciences | 281810 | |
| Arabinose | Sigma-Aldrich | A3131 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S5136 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Sodium chloride | Sigma-Aldrich | S5886 | |
| Glycerol | Sigma-Aldrich | G2025 | |
| Bacto Agar | BD Biosciences | 214010 |
참고문헌
- Mello, C., Fire, A. DNA transformation. Methods Cell Biol. 48, 451-482 (1995).
- Zhang, Y., Nash, L., Fisher, A. L. A simplified, robust, and streamlined procedure for the production of C. elegans transgenes via recombineering. BMC Dev Biol. 8, 119-119 (2008).
- Antebi, A., Yeh, W. H., Tait, D., Hedgecock, E. M., Riddle, D. L. daf-12 encodes a nuclear receptor that regulates the dauer diapause and developmental age in C. elegans. Genes and Development. 14, 1512-1527 (2000).
- Snow, M. I., Larsen, P. L. Structure and expression of daf-12: a nuclear hormone receptor with three isoforms that are involved in development and aging in Caenorhabditis elegans. Biochim. Biophys. Acta. 1494, 104-116 (2000).
- Fisher, A. L., Page, K. E., Lithgow, G. J., Nash, L. The Caenorhabditis elegans K10C2.4 Gene Encodes a Member of the Fumarylacetoacetate Hydrolase Family. A CAENORHABDITIS ELEGANS MODEL OF TYPE I TYROSINEMIA. J Biol.Chem. 283, 9127-9135 (2008).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75, 855-862 (1993).
- Lehrbach, N. J. LIN-28 and the poly(U) polymerase PUP-2 regulate let-7 microRNA processing in Caenorhabditis elegans. Nat Struct Mol Biol. 16, 1016-1020 (2009).
- Tursun, B., Cochella, L., Carrera, I., Hobert, O. A toolkit and robust pipeline for the generation of fosmid-based reporter genes in C. elegans. PLoS One. 4, e4625-e4625 (2009).
- Bamps, S., Hope, I. A. Large-scale gene expression pattern analysis, in situ, in Caenorhabditis elegans. Brief. Funct. Genomic. Proteomic. , (2008).
- Dolphin, C. T., Hope, I. A. Caenorhabditis elegans reporter fusion genes generated by seamless modification of large genomic DNA clones. Nucleic Acids Res. 34, e72-e72 (2006).
- Sarov, M. A recombineering pipeline for functional genomics applied to Caenorhabditis elegans. Nat. Methods. 3, 839-844 (2006).
- Yang, X. W., Model, P., Heintz, N. Homologous recombination based modification in Escherichia coli and germline transmission in transgenic mice of a bacterial artificial chromosome. Nat Biotechnol. 15, 859-865 (1997).
- Court, D. L., Sawitzke, J. A., Thomason, L. C. Genetic engineering using homologous recombination. Annu.Rev.Genet. 36, 361-388 (2002).
- Westenberg, M., Bamps, S., Soedling, H., Hope, I. A., Dolphin, C. T. Escherichia coli MW005: lambda Red-mediated recombineering and copy-number induction of oriV-equipped constructs in a single host. BMC Biotechnol. 10, 27-27 (2010).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36-e36 (2005).
- Penfold, R. J., Pemberton, J. M. An improved suicide vector for construction of chromosomal insertion mutations in bacteria. Gene. 118, 145-146 (1992).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Puig, O. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat.Biotechnol. 17, 1030-1032 (1999).
- Achilleos, A., Wehman, A. M., Nance, J. PAR-3 mediates the initial clustering and apical localization of junction and polarity proteins during C. elegans intestinal epithelial cell polarization. Development. 137, 1833-1842 (2010).
- Maduro, M., Pilgrim, D. Identification and cloning of unc-119, a gene expressed in the Caenorhabditis elegans nervous system. Genetics. 141, 977-988 (1995).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유