このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
オーラルバイオフィルムの包括的、生化学的構造とトランスクリプトーム評価のための分析ツールボックスは、ミュータンス連鎖球菌によって仲介
要約
歯の表面に形成されたバイオフィルムは非常に複雑で、そのアーキテクチャを調節する定数先天性および外因性の環境上の課題、、生理学およびトランスクリプトームにさらされている。我々は、バイオフィルムの研究の他の領域に適合させることができる口腔バイオフィルムの組成、構造、組織および遺伝子発現を調べるためにツールボックスを開発しました。
要約
バイオフィルムは、可変密度と組成物1、2の細胞外マトリックスにenmeshed微生物細胞の高度に動的な、組織的かつ構造化されたコミュニティです。一般的には、バイオフィルムは、細胞クラスター(またはmicrocolonies)と複雑な細胞外マトリックスで発生microcolonies、のさらなる発展と安定の形成に続いて表面に初期微生物付着から発生する。バイオフィルムの行列の港のexopolysaccharides(EPS)、および歯科バイオフィルムの大部分もその例外ではありません。特に主にミュータンス連鎖球菌3によって仲介されるカリエス病、関連付けられているもの。 EPSは、主に基板3としてスクロースを用いてグルコシルトランスフェラーゼなどの細胞外酵素、によって微生物(S.ミュータンス 、重要な貢献者)によって合成される。
歯の表面に形成されたバイオフィルムの研究は、特に口腔内で発生する複雑な食事療法 - ホスト - 微生物の相互作用に関連する環境上の課題への一定の暴露に起因し挑戦しています。これらの複雑な相互作用に応答して、よりよい行列の構造組織と構成の動的な変化の理解、生理学およびトランスクリプトーム/バイオフィルム細胞のプロテオームプロファイルは、さらに病原性を調節する方法口腔バイオフィルムの現在の知識を進めるだろう。したがって、我々は、データ解析のためのカスタムメイドのソフトウェアで一般に入手可能と斬新なテクニックを組み合わせることで、構造的、生化学的および分子レベルでのバイオフィルムの解析を容易にするための分析ツールボックスを開発しました。標準的な分析(比色アッセイ、RT -定量PCRとマイクロアレイ)と小説蛍光技術は、(細菌およびEPSの同時ラベリングのための)口腔バイオフィルム研究の複雑な性質に対処するためのデータ解析のための特定のソフトウェアに統合されました。
ツールボックスは4つの異なるが、相互接続手順(図1)で構成される:1)バイオアッセイ、2)生データの入力、3)データ処理、および4)データ分析。我々は、ツールボックスの有用性と柔軟性を実証するためのin vitroバイオフィルムモデルと特定の実験条件で私たちを使用していました。バイオフィルムモデルは、単純な再現性と複数である単一の実験で複製、同時に4を行うことができます。また、それにより時間的な評価、様々な微生物種の包含5と異なる実験条件の影響の評価(例えば治療6、ノックアウト変異体の比較対親株5;炭水化物の可用性7)。ここで、我々はマイクロアレイデータマイニング/組織(MDV)と蛍光イメージング解析(DUOSTAT)のための新しいソフトウェア(I)を含むツールボックスの2つの特定のコンポーネントを、説明、および(ii) その場 EPS標識に 。我々はまた、ツールボックスは、バイオフィルムの分析、データの編成、統合と解釈を支援する方法を示す実験ケースを提供しています。
プロトコル
1。 STEP 1 - バイオアッセイ
生物膜法は、歯の代用物としてハイドロキシアパタイトのディスク(HA)を使用します(クラークソンクロマトグラフィー製品、(株)、南ウィリアムスポート、ペンシルバニア州、米国;表面積= 2.7 ± 0.2 cm 2)を唾液でコーティングされた(買収ペリクルの存在を模倣する)、置く垂直位置4、5、8インチ
- 生化学的ア ッセイ。
- バイオフィルムは、超音波処理9で均質化(I)であるか、(ⅱ)生化学的ア ッセイ8のためそのまま保持。均質化したバイオフィルムの懸濁液の総蛋白質、無機、および細胞外および細胞内の多糖類の構成/内容(モスらの詳細を参照してください。、オーラルバイオフィルム8の生理学を研究するためにプロトコルを)、(乾燥重量)バイオマスの決定に使用することができます。無傷のバイオフィルムは、標準的な解糖系のpH低下、酸を殺す、プロトンの透過性およびF - ATPase活性のアッセイを(モスらの詳細を参照してください。、オーラルバイオフィルム8の生理学を研究するためのプロトコル)を使用してそれらの生理学的反応を分析することができます。
- トランスクリプトーム解析。
- 標準的なcDNAマイクロアレイおよびRT -定量PCRのプロトコル(例えばhttp://pfgrc.jcvi.org/index.php/microarray/protocols.htmlは)特定の病原体(例えば、S.ミュータンス )のトランスクリプトーム応答を測定するために組み合わせて使用することができます。様々な発達段階6、7、10、11でさまざまな環境と治療上の課題に対するバイオフィルム内。 RNAの品質(原因バイオフィルム集団とEPS -マトリックスの存在の不均質な性質)、2)データマイニングと解釈を(のために1):ツールボックスでは、我々は歯のバイオフィルムのトランスクリプトーム解析のための重要な2つのメソッドが含まれ大規模なデータは)マイクロアレイによって生成された設定。
- RNAの分離。バイオフィルムから抽出したRNAの品質の問題に対処するためには、我々は、EPS -リッチマトリックス12(http://dx.doi.org/10.1016/jでenmeshed細菌細胞からのRNA単離および精製の ために特別に開発された最適化されたプロトコルを使用してください。ab.2007.03.021)。このプロトコルは、RT -定量PCRとマイクロアレイの両方を使用してトランスクリプトーム解析のための最適とみなされるRNAの完全性番号(RIN)8.5よりも高いが、用意されています(例については、6、11を参照)。
- 標準的なcDNAマイクロアレイおよびRT -定量PCRのプロトコル(例えばhttp://pfgrc.jcvi.org/index.php/microarray/protocols.htmlは)特定の病原体(例えば、S.ミュータンス )のトランスクリプトーム応答を測定するために組み合わせて使用することができます。様々な発達段階6、7、10、11でさまざまな環境と治療上の課題に対するバイオフィルム内。 RNAの品質(原因バイオフィルム集団とEPS -マトリックスの存在の不均質な性質)、2)データマイニングと解釈を(のために1):ツールボックスでは、我々は歯のバイオフィルムのトランスクリプトーム解析のための重要な2つのメソッドが含まれ大規模なデータは)マイクロアレイによって生成された設定。
- 蛍光イメージング。バイオフィルムの構造的な組織。
- 文学で利用可能な共焦点蛍光イメージングのプロトコルのほとんどは、微生物のラベルに焦点を当てています。バイオフィルムの主要な成分としてEPSの分析は、主にいくつかの例外を除い5、13、14と、蛍光イメージングを含む口腔バイオフィルムの研究で軽視されてきました。我々は、斬新な標識技術や再現性の視覚化と同時に、無傷のバイオフィルム内のEPSおよび細菌細胞の定量化のための特定のソフトウェアを開発しました。犯罪統計システム(http://www.imageanalysis.dkで入手可能)とDUOSTAT(http://www.imageanalysis.dkが)提供していますがアミラ5(ヴィサージイメージングGmbHは、ベルリン、ドイツ)は、バイオフィルムの三次元再構成のために使用されています定量分析。
- バイオフィルムのEPS -細菌の構造組織のイメージング。のAlexa Fluor ® 647標識デキストラン複合体は、EPS -行列を可視化するために使用されます。蛍光標識デキストランは、連鎖球菌グルコシルトランスフェラーゼ- GTFS(特にGtfBとGtfC)のためのプライマーとして機能し、同時にバイオフィルム開発11の経過とともに体外多糖マトリックスの合成中に組み込むことができます。
EPSラベル:- "バイオフィルムの準備"プロトコル8次の1時間のためのろ過滅菌した唾液でコーティングハイドロキシアパタイトディスク、。
- ピペットは2.8 24ウェルプレートの各ウェルの培養培地の添加、そして暗い部屋にもたらす。
- 培養液中にデキストランコンジュゲートのAlexa Fluorの1μMを追加、ピペッティングして培養液中に染料を混合し、ダウン(約10倍)。
- 唾液でコーティングされたHA(SHA)ディスク(1時間のインキュベーション後に)二度滅菌ABバッファでの浸漬洗浄及びデキストランコンジュゲートのAlexa Fluorを含む培地に置いてください。
- 37アルミ箔とインキュベートするとプレート° C、5%CO 2をカバーしています 。インキュベーション時間の持続時間は、各実験の設計に依存します。蛍光標識デキストランは、こので使用されている濃度で細菌細胞を染色していない11を言う。 このようなCalcofluor 14とのような他のEPS -染色技法、、組み合わせて使用することができます。
- 細菌の標識:
- バイオフィルム中の細菌成分(例えばSYTO 9)染色標準蛍光核酸を用いたバイオフィルム形成の終了時にラベルが付いていますが、他の蛍光技術に使用されるコースを取ることができます(特定の蛍光標識抗体を15またはGFP発現細胞16は 、例えば種)バイオフィルム内の1つまたは複数の細菌種を検出する。図2は、24時間古いS.の3D画像にEPS(赤)と細菌/ microcoloniesの同時標識(緑)を示しています。 SHAのディスクの表面に形成されたミュータンスは、バイオフィルム。
- 画像の取得:各バイオフィルムは、5から10までランダムに選択された位置にスキャンされ、そしてZシリーズは、標準プロトコルを使用して共焦点顕微鏡を走査型レーザーによるこれらの各位置での光学セクショニングによって生成されています。水をまたはX25(WD 2ミリメートルオリンパスLPlan N; NA 1.05)我々は、X10(;開口数0.45の距離3〜4ミリメートルを作業カールツァイス)を装備したオリンパスFV千二光子顕微鏡(オリンパス、東京、日本)を使用浸対物。励起波長は810 nmであり、およびAlexa Fluor 647(EPS)用のフィルタがHQ655/40M-2Pフィルタである間SYTO 9の発光波長フィルター(バクテリア)は、540分の495 OlyMPFC1フィルタです。保存して、生の画像を保存する。
- バイオフィルムのEPS -細菌の構造組織のイメージング。のAlexa Fluor ® 647標識デキストラン複合体は、EPS -行列を可視化するために使用されます。蛍光標識デキストランは、連鎖球菌グルコシルトランスフェラーゼ- GTFS(特にGtfBとGtfC)のためのプライマーとして機能し、同時にバイオフィルム開発11の経過とともに体外多糖マトリックスの合成中に組み込むことができます。
- 文学で利用可能な共焦点蛍光イメージングのプロトコルのほとんどは、微生物のラベルに焦点を当てています。バイオフィルムの主要な成分としてEPSの分析は、主にいくつかの例外を除い5、13、14と、蛍光イメージングを含む口腔バイオフィルムの研究で軽視されてきました。我々は、斬新な標識技術や再現性の視覚化と同時に、無傷のバイオフィルム内のEPSおよび細菌細胞の定量化のための特定のソフトウェアを開発しました。犯罪統計システム(http://www.imageanalysis.dkで入手可能)とDUOSTAT(http://www.imageanalysis.dkが)提供していますがアミラ5(ヴィサージイメージングGmbHは、ベルリン、ドイツ)は、バイオフィルムの三次元再構成のために使用されています定量分析。
2。 STEP 2 - RAWデータの入力
直接生データファイルへの生化学的およびRT -定量PCRアッセイ(RDF - MS Excelファイル)から入力生データ。マイクロアレイデータの場合、JCVI Spotfinder(http://pfgrc.jcvi.org/index.php/bioinformatics.html)または同様のソフトウェアにスキャンしたスライドのシングルチャンネル画像を読み込む。 JCVIの仕様に合わせてスポットのグリッドを作成してから、手動でグリッド内のすべてのスポットに合うように調整します。各スポットの強度値を測定し、"。MeVの"ファイルに保存し、"生のマイクロアレイのデータ"に格納。
3。 STEP 3 - データ処理
統計分析のためのRDFの生データを(生化学およびRT - QPCR)整理。 "データ処理されたファイル"( - MS ExcelファイルDPF)にそれらを転送します。マイクロアレイおよび蛍光イメージング分析のために、特定のソフトウェアは、(現在利用可能な、特注の)データを処理するために使用されます。
- マイクロアレイデータ:
- 特定のソフトウェアを使用してデータの正規化のステップに手順2("生のマイクロアレイのデータ"に保存されている)で生成された生データを提出する。 JCVIマイクロアレイデータ解析ソフトウェアMIDAS(http://www.tm4.org/midas.html)を使用してデータを正規化する。で、スライドの複製の分析に続いて、デフォルトの設定でLOWESSと標準偏差の正則化を使用してください。正規化されたデータを含むファイルを保管してください。この段階(生データファイルおよび正規化されたデータファイル準備完了)で、パブリックアクセスのデータベース(http://www.ncbi.nlm.nih.gov/geoでたとえばNCBI遺伝子発現オムニバス(GEO)のデータベース)での預金マイクロアレイのデータ、と将来の参照用にアクセッション番号を記録する。
- 蛍光イメージングデータ
- バイオフィルムの構造を定量化。
- バイオフィルムの各構成成分(EPSや細菌)に対応する定量的なデータは、犯罪統計システムとMATLAB 5.1でスクリプトとして記述された新開発DUOSTAT、によって計算されます。生の蛍光画像は、ソフトウェアにアップロードされ、以下のように分析されています:
- バイオマス、厚さ、層の分布と定量化とバイオフィルムの三次元構造を(http://www.imageanalysis.dkでマニュアルを参照してください)特徴付ける他のパラメータを計算するために犯罪統計システムを使用してください。
- このようなEPSや細菌の2つのバイオフィルム成分、(または別の微生物種及び/又は細胞外成分)の共局在を計算するためにDUOSTATを使用します。
- MATLAB 5.1のDUOSTATのパス、二バイオフィルムの構成要素の画像を含む開いている画像のフォルダを設定します。
- 相関および分析される2つのチャンネルを選択してください。二つの画像のスタックはまた、3次元すべてで同一のピクセルサイズ(X、Y、Z)、および各スタック内の画像の数が同じである必要があります。
- 男性によると、各チャネルのしきい値を設定する犯罪統計システムのUAL。ソフトウェアの操作インタフェース(; http://www.imageanalysis.dkでDUOSTATマニュアルビデオの記事を参照)の指示に従ってください。入力DPFに得られたデータを。図4.4の例を参照してください。
- 三次元バイオフィルムの再構成。
- バイオフィルムの三次元構造は、アミラを用いて可視化されています。ソフトウェアに"Raw画像のフォルダ"に保存されている蛍光画像をインポートし、そして(http://www.amira.comでアミラマニュアルを参照してバイオフィルム内の各コンポーネントの3次元レンダリングを作成するvoltexとアイソサーフェスレンダリングを使用してください/ドキュメント/マニュアルとリリース- notes.html)。図4.4の例を参照してください。
- バイオフィルムの各構成成分(EPSや細菌)に対応する定量的なデータは、犯罪統計システムとMATLAB 5.1でスクリプトとして記述された新開発DUOSTAT、によって計算されます。生の蛍光画像は、ソフトウェアにアップロードされ、以下のように分析されています:
- バイオフィルムの構造を定量化。
4。ステップ4 - データ分析
DPFの生化学的、RT -定量PCRと犯罪統計システム- DUOSTATアッセイから定量的なデータは統計分析のための準備が整いました。統計解析が実行された後、グラフおよび/またはテーブルが("代表的な結果"の項を参照)を構築することができます。
1。ソフトウェアマイクロアレイデータビジュアライザー(MDV)を用いたマイクロアレイデータの編成。
マイクロアレイと、複数の実験条件を使用するときの複雑さや大規模なデータの出力のために設定し、我々はマイクロアレイデータビジュアライザー(MDV)7(http://www.oralgen.lanl.gov/で入手可能)という名前のデータマイニングと組織のソフトウェアを設計。
クラス予測のための0.001のカットオフPの値とし、クラスの比較のためにBRB - ArrayTools(http://linus.nci.nih.gov/BRB-ArrayTools.html)を使用して統計分析を行った後、生成されたデータを提出することができます。 MDVに、として、次のとおりです。
- Excelを使用して、MDVタブ区切りのテキストファイルにBRBデータを変換します。遺伝子座のタグ番号に付属するすべての文字を削除して、たとえば:SMU.1423cは、"C"の文字を削除します。
- MDVを開きます。ファイルに移動し、"注釈ソースの選択"を選択します。 "フラットファイル"を選択してください。ダイアログボックスが表示されたら、遺伝子名、遺伝子オントロジー(GO)数、経路、および機能的分類のデータのための注釈が含まれるファイルを選択して[参照]ボタンを使用してください。
- ファイルに移動し、項目1からの各ファイルをインポートします。各ファイルには、分析のための実験条件を表します。分析のための複数の条件がある場合、例えば、二つの異なる治療法(治療1と2)と制御(車両が)項目4に進みます。
- このケースでは、可能な比較は以下のとおりです(A)処理1対コントロール(処理1のために示差的に発現された遺伝子のための)、(B)処理2と制御、および(C)処理1対処理2。作業仮説に応じて、遺伝子の異なるセットを選択することができます。
- 比較して異なって発現遺伝子のみを選択する(ユニークな遺伝子が唯一の治療法1によって影響を受ける)。この場合、セット関数のメニューから減算関数を使用します。から比較Bから遺伝子を減算し、結果を保存します。その後、得られたデータからCを引く。同じアクションはBの遺伝子のみ(のみ処理2の影響を受けたユニークな遺伝子)を得るために行うことができます。
- AとBの両方で検出された遺伝子のみを選択するには、比較およびBの結果を結合し、結果を保存するには、Set関数メニューから組合の機能を使用してください。その後、結果のデータからCを引く。
- これらのアクションは、より直感的である、と仮説駆動型のデータの編成を容易にMDVで特色にベン図を、使用して視覚化することができます。ベン図のデータ出力は(図3およびビデオの記事で画面のビューを参照してください)画面に表示されます。
- タブ区切りのテキストファイルにデータを保存するには、ファイルに移動し、エクスポート]を選択します。
- タブ区切りのテキストファイルをエクスポートします。それらは、Excelを使用して、そしてテーブルのMDVから出力されるデータを編成および/またはグラフィカル表示(図4.2、4.3、ビデオの記事を参照)に隠れた開きます。
5。駐在結果
ここでは、分析ツールボックスは、複数の変数や実験条件によるバイオフィルム研究の様々なアッセイを統合する方法の例を提供しています。
実験的なケース:
バイオフィルムの開発7時のデンプンとショ糖に応答して、トランスクリプトームストレプトコッカスミュータンスのダイナミクス。
背景:
ホスト唾液アミラーゼおよび連鎖球菌のグルコシルトランスフェラーゼによる食事の澱粉とショ糖の相互作用は、S.の形成と病原性を高めることができるインクリメによるバイオフィルム内のミュータンスexopolysaccharides合成、糖代謝とacidogenicity 11 asing。この複雑な宿主 - 病原体 - 食の相互作用は、虫歯の疾患に関連する病原性バイオフィルムの形成を調節することがあります。我々は、さらにS.方法を理解するための包括的な生化学的およびトランスクリプトーム解析を(全体のゲノムのプロファイリングを含む)を実施ミュータンスは、アミラーゼ7の存在下でバイオフィルムの発達の異なる段階におけるデンプンやショ糖に応答します。
分析ツールボックスは、種々の実験条件と時間の点で形成されたバイオフィルムの生化学的および分子アッセイの統合に私たちを支援するために使用されていました。分析ツールボックスを使って全体のデータ出力は、図4の順次的(4.1〜4.4)で表示されます。それはここに主な焦点は、ツールボックスのユーティリティではなく、データの解釈と議論を実証するためであることは注目に値する。
- 生化学とさらにマイクロアレイ解析のための実験条件を選択するためにRT - qPCRアッセイを組み合わせる。当初、我々はS.によってバイオフィルムの開発のための炭水化物の最適な濃度を選択するために6つの異なるショ糖と5種類のデンプンとスクロースの組み合わせをテスト私たちのin vitroモデルを用いてミュータンス 。に基づいて3つの特定の濃度を(図4.1で強調表示)の選択に私達を導きバイオアッセイから同時にデータの出力を検査する能力(上昇)不溶性exopolysaccharidesの量とバイオフィルムのgtfBの (拡張)、式(不溶性グルカン合成に関与する) 。
- マイクロアレイ解析。食餌性炭水化物の選択(3つ)の濃度はバイオフィルムを形成するために使用された、バイオフィルムの開発プロセスのさまざまな段階を反映した特定の時間ポイントで削除された。バイオフィルムは、(3つの実験群と4時間の点で分割)BRB -アレイツールとMDVで全ゲノムプロファイリング(cDNAマイクロアレイ)を組み合わせることにより、トランスクリプトーム解析を行った。
RNAを抽出し、バイオフィルム固有のプロトコル12を用いて精製し、分析ツール- Boxの4段階のプロセスを通じて解析をマイクロアレイに供した。図4.2は、MDVはショ糖とでんぷんの組み合わせがS.の強化された病原性(虫歯を発生させる潜在的な)に関連付けられた特定の転写応答を引き起こすことを私たちの作業仮説に基づいて関心のある遺伝子を選択するために役立った方法を示しています。バイオフィルム内のミュータンス 。 BRB - ArrayTools(図4.2A)によって生成された生データは、MDVは、ベン図(図4.2bのを)使用して処理した。本研究では、我々の仮説に関連する遺伝子のグループは、差動比較で表したものです(0.5%ショ糖+ 1%澱粉対1%ショ糖)とB(0.5%ショ糖+ 1%澱粉対0.5%ショ糖)、ではなく、比較C(0.5%、1%ショ糖対スクロース)(図4.2Aと4.2bの)の遺伝子。図4.2cに示すように、MDVは大幅に分析される遺伝子の総数を削減し、同時に直接の組み合わせでショ糖やでんぷんの影響に関連していない遺伝子のフィルタリングアウト。 MDVデータ出力は、澱粉の示差的に発現された遺伝子のそれぞれの機能分類(上下調節)によって編成されたデータをグラフィカルに表示するために変換されました+スクロースバイオフィルム(対スクロース成長したバイオフィルム)各時(図4.3)、4点の時間。 MDVのソフトウェアはユーザーフレンドリーと私たちのマイクロアレイ実験から大規模かつ複雑なデータセットのマイニング/組織を促進される。 - バイオフィルムイメージング付随して、バイオフィルムの構造組織が 犯罪統計システム- DUOSTAT -アミラは、ツールボックス(澱粉の三次元再構成および定量分析の例を参照してください+図4.4のショ糖で栽培されたバイオフィルムに含まれて使用して分析した;も参照してくださいビデオの記事)。形態、分布およびEPSとmicrocoloniesの構造的な関係は、アミラによる3Dサーフェスレンダリングを用いて可視化することができます。選択された領域のクローズアップビューは、マイクロでの細菌- EPS固有の構造的関係(図4.4)を示している、生成することができます。さらに、共焦点画像の同じセットが同時にバイオマス/微小コロニーの測定、細菌細胞の空間分布と共局在し、EPSのために犯罪統計システム- DUOSTATで処理することができます。例えば、ディスクの表面から液相へのEPSと細菌の垂直分布は犯罪統計システム- DUOSTAT用いた三次元共焦点バイオフィルムの画像の各光学部から算出した(図4.4のグラフを、。映像の記事を参照してください)。データは、バイオフィルムの深さにわたって細菌(緑線)よりEPS(赤線)の高い割合を示しています。さらに、細菌細胞のほとんどは、特にバイオフィルムの中央と外側の層では、EPS(青線)に関連付けられています。この観察ではデンプンやショ糖で育ったバイオフィルムがenmeshingているexopolymers、(で特に豊富であることを示しています細菌細胞の)ほとんどとの密接な接触で、そのような構造的な組織は、バイオフィルム13の安定性と凝集性を高めます。三次元レンダリングと共焦点画像の定量的な測定は(でんぷんのS.ミュータンス +スクロース栽培バイオフィルムによってGtfB型不溶性グルカンの合成を増加)生化学的および遺伝子発現データを補完するバイオフィルムの構造に関する追加情報を提供する(他の例は、5、6、11、13を参照)。
S.方法の包括的な分析を提供する私たちは別のバイオアッセイからのデータを、取得、整理、統合する支援分析ツール-ボックス、 ミュータンスは(。。; Klein ら 、2010 7 Klein らの詳細を参照して、2009 11)口腔内に見られる食事-宿主相互作用の結果として、複雑な環境変化に応答することができる。

バイオフィルム解析のための分析ツール- Boxの図1。フローチャート。

図2バイオフィルムのEPSと細菌の共焦点蛍光イメージング。 EPS(赤)と細菌/ SHAのディスクの表面に形成されたストレプトコッカスミュータンスのバイオフィルムの三次元レンダリングでmicrocolonies(緑)の同時可視化。

図3。マイクロアレイデータマイニングとマイクロアレイデータビジュアライザー(MDV)ソフトウェアを使用している組織。関心のある遺伝子を選択し、遺伝子名や機能クラスの注釈を加えてするベン図の機能を使用してください。

図4.1。S.のバイオフィルム形成の評価生化学的ア ッセイ(A)とRT -定量PCR(B)を用いてミュータンス 。 INS(不溶性exopolysaccharides)データはgtfB表現のパターンとよく相関し、バイオフィルムのバイオマスと。 0.5%のショ糖が最適なバイオフィルムの開発に必要な最小濃度は 、in vitro のモデルで私たちを使用していたのに対し、1%のショ糖は、最大INS形成のための濃度、SHAの表面上にgtfB発現とバイオフィルムの蓄積であった。S. 0.5%のスクロースの存在下で増殖したミュータンスの細胞は、+ 1%の澱粉は、最高のバイオマスが得られ、および強化されたgtfB式 (B2)と相関し、他のバイオフィルム、より多くのINSを発表した。これらの炭水化物濃度はさらにトランスクリプトーム解析のために選択した。
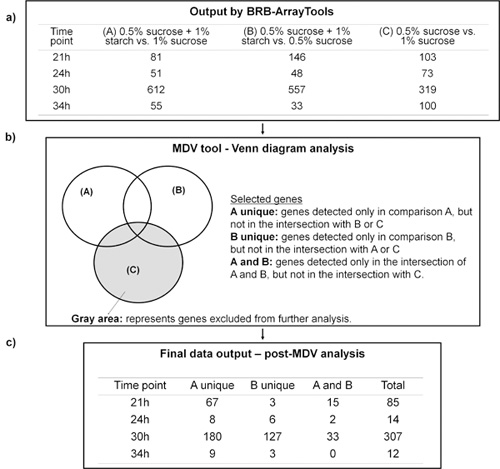
図4.2。MDVのソフトウェアと一緒にBRB -アレイツールを使ってマイクロアレイデータの解析。 a)の差、各比較(A、BまたはC)とBRB -アレイツールを使用して評価時点で表現として検出する遺伝子の数を表します。興味のある遺伝子を選択するベン図を用いてb)のマイクロアレイデータビジュアライザー(MDV)。 C)遺伝子は、MDVの分析に応じて選択する。

図4.3。S. ミュータンスの遺伝子が差澱粉で表現さ+機能的なクラスが主催する様々な時間点におけるスクロースバイオフィルム(対スクロースバイオフィルム)。遺伝子の注釈は、ロスアラモス国立研究所(www.oralgen.lanl.gov)によって、または同じWebサイトで入手可能な公表された文献によって提供される情報に基づいています。

図4.4。澱粉の三次元レンダリングと犯罪統計システム- DUOSTAT分析+スクロースバイオフィルム。
Access restricted. Please log in or start a trial to view this content.
ディスカッション
本発表では、我々は、分析ツールボックス(EPS /細菌イメージングおよびマイクロアレイデータマイニング/処理)、システムに統合された種々のアッセイの汎用性と有用性の二つの重要な構成要素を示した。明らかに、ツールボックスは、in vitroモデルで使用して、異なる実験条件に対応してバイオフィルム生化学、アーキテクチャおよび遺伝子発現のさまざまな側面の包括的?...
Access restricted. Please log in or start a trial to view this content.
開示事項
謝辞
著者らは、MDVの開発のための博士ゲイリー謝とハーバートリーに感謝します。我々はまた、博士に感謝します。シモーネドゥアルテ、ラミロ村田、在圭田、ジャクリーンのAbranches、およびツールボックスの分析コンポーネントの彼らの技術的および科学的貢献のためのさんステイシーグレゴワール。この研究は、歯科および顎顔面研究所の国立研究所からUSPHS研究助成金DE018023によって部分的にサポートされていました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Syto 9 | Invitrogen | S34854 | |
| Syto 60 | Invitrogen | S11342 | |
| Dextran conjugated alexa 647 | Invitrogen | D22914 | |
| Olympus FV1000 two-photon laser scanning microscope | Olympus Corporation |
参考文献
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- Leme, P. aes, Koo, A. F., Bellato, H., Bedi, C. M., G, J. A. C. ury The role of sucrose in cariogenic dental biofilm formation--new insight. J Dent Res. 85, 878-887 (2006).
- Koo, H., Schobel, B. D., Scott-Annem, K., Watson, G., Bowen, W. H., Cury, J. A., Rosalen, P. L., Park, Y. K. Apigenin and tt-farnesol with fluoride effects on S. mutans biofilms and dental caries. J Dent Res. 84, 1016-1020 (2005).
- Koo, H., Xiao, J., Klein, M. I., Jeon, J. G. Exopolysaccharides produced by Streptococcus mutans glucosyltransferases modulate the establishment of microcolonies within multispecies biofilms. J Bacteriol. 192, 3024-3032 (2010).
- Jeon, J. G., Klein, M. I., Xiao, J., Gregoire, S., Rosalen, P. L., Koo, H. Influences of naturally occurring agents in combination with fluoride on gene expression and structural organization of Streptococcus mutans in biofilms. BMC Microbiol. 9, 228-228 (2009).
- Klein, M. I., DeBaz, L., Agidi, S., Lee, H., Xie, G., Lin, A. H. M., Hamaker, B. R., Lemos, J. A., Koo, H. Dynamics of Streptococcus mutans transcriptome in response to starch and sucrose during biofilm development. PLoS ONE. , 0013478-0013478 (2010).
- Lemos, J. A., Abranches, J., Koo, H., Marquis, R. E., Burne, R. A. Protocols to Study the Physiology of Oral Biofilms. Methods Mol Biol. 666, 87-102 (2010).
- Koo, H., Hayacibara, M. F., Schobel, B. D., Cury, J. A., Rosalen, P. L., Park, Y. K., Vacca-Smith, A. M., Bowen, W. H. Inhibition of Streptococcus mutans biofilm accumulation and polysaccharide production by apigenin and tt-farnesol. J Antimicrob Chemother. 52, 782-789 (2003).
- Koo, H., Seils, J., Abranches, J., Burne, R. A., Bowen, W. H., Quivey, R. G. Influence of apigenin on gtf gene expression in Streptococcus mutans UA159. Antimicrob. Agents Chemother. 50, 542-546 (2006).
- Klein, M. I., Duarte, S., Xiao, J., Mitra, S., Foster, T. H., Koo, H. Structural and molecular basis of the role of starch and sucrose in Streptococcus mutans biofilm development. Appl Environ Microbiol. 75, 837-841 (2009).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Anal Biochem. 365, 208-214 (2007).
- Xiao, J., Koo, H. Structural organization and dynamics of exopolysaccharide matrix and microcolonies formation by Streptococcus mutans in biofilms. J Appl Microbiol. 108, 2103-2113 (2010).
- Thurnheer, T., Gmür, R., Shapiro, S., Guggenheim, B. Mass transport of macromolecules within an in vitro model of supragingival plaque. Appl Environ Microbiol. 69, 1702-1709 (2003).
- Chalmers, N. I., Palmer, R. J. J. r, Du-Thumm, L., Sullivan, R., Shi, W., Kolenbrander, P. E. Use of quantum dot luminescent probes to achieve single-cell resolution of human oral bacteria in biofilms. Appl Environ Microbiol. 73, 630-636 (2007).
- Deng, D. M., Hoogenkamp, M. A., Ten Cate, J. M. >, Crielaard, W. Novel metabolic activity indicator in Streptococcus mutans biofilms. J Microbiol Methods. 77, 67-71 (2009).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved