Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein analytisches Werkzeug-Box für umfassende biochemische, strukturelle und Transkriptom Evaluation of Oral Biofilme von Mutans Streptokokken Mediated
In diesem Artikel
Zusammenfassung
Biofilme auf der Zahnoberfläche gebildet sind sehr komplex und in denen konstante angeborenen und exogene ökologischen Herausforderungen, die ihre Architektur modulieren, Physiologie und Transkriptom. Wir entwickelten eine Toolbox, um die Zusammensetzung, die strukturelle Organisation und Genexpression von oralen Biofilmen, die auf andere Bereiche der Biofilm-Forschung angepasst werden können zu prüfen.
Zusammenfassung
Biofilme sind hochdynamische, organisiert und strukturiert Gemeinden von mikrobiellen Zellen in eine extrazelluläre Matrix der variablen Dichte und Zusammensetzung 1, 2 verstrickt. In der Regel bilden sich Biofilme von der ersten mikrobiellen Anlage auf einer Fläche durch die Bildung von Zellverbänden (oder Mikrokolonien) und die weitere Entwicklung und Stabilisierung der Mikrokolonien, die in einer komplexen extrazellulären Matrix auftreten gefolgt. Die Mehrheit der Biofilm Matrizen Hafen Exopolysaccharide (EPS) und zahnärztliche Biofilme sind keine Ausnahme, vor allem diejenigen mit Karies Krankheit, die meist durch Streptococcus mutans 3 sind vermittelten verbunden. Die EPS werden von Mikroorganismen (S. mutans, einen wesentlichen Beitrag) durch extrazelluläre Enzyme, wie Glucosyltransferasen Verwendung von Saccharose in erster Linie als Substrat 3 synthetisiert.
Studien von Biofilmen auf der Zahnoberfläche gebildet sind besonders anspruchsvoll aufgrund ihrer ständigen Belastung durch Umwelt-Herausforderungen mit komplexen Diät-host-mikrobielle auftretenden Wechselwirkungen in der Mundhöhle verbunden. Ein besseres Verständnis der dynamischen Veränderungen der strukturellen Organisation und Zusammensetzung der Matrix, Physiologie und Transkriptom / Proteom-Profil von Biofilm-Zellen als Antwort auf diese komplexen Wechselwirkungen würden weiter vorantreiben dem aktuellen Wissenstand, wie orale Biofilme modulieren Pathogenität. Deshalb haben wir ein analytisches Werkzeug-Box Biofilm Analyse bei strukturellen, biochemischen und molekularen Ebene zu erleichtern durch die Kombination von allgemein verfügbaren und neuen Techniken mit maßgeschneiderter Software für die Datenanalyse entwickelt. Standard-analytische (kolorimetrische Assays, RT-qPCR und Microarrays) und neuartige Fluoreszenz-Verfahren (für die gleichzeitige Kennzeichnung von Bakterien und EPS) wurden mit spezieller Software für die Datenanalyse, die komplexe Natur der oralen Biofilm-Forschung-Adresse integriert.
Das Tool-Box besteht aus 4 verschiedenen, aber miteinander verbundenen Schritten (Abbildung 1) besteht aus: 1) Bioassays, 2) Raw Data Input, 3) Data Processing, und 4) Data Analysis. Wir nutzten unsere in vitro Biofilm-Modell und spezifischen experimentellen Bedingungen, den Nutzen und die Flexibilität der Tool-Box zu demonstrieren. Der Biofilm-Modell ist einfach, reproduzierbar und mehrere Replikate von einem einzigen Experiment kann gleichzeitig 4 erfolgen, 5. Darüber hinaus ermöglicht es zeitliche Auswertung, Einbindung verschiedener Mikroben-Spezies 5 und Beurteilung der Auswirkungen von unterschiedlichen experimentellen Bedingungen (z. B. Behandlungen 6; Vergleich von Knockout-Mutanten vs Elternstamm 5; Kohlenhydrate Verfügbarkeit 7). Hier beschreiben wir zwei spezifische Komponenten der Tool-Box, einschließlich (i) neue Software für Microarray Data Mining / Organisation (MDV) und Fluoreszenz-Imaging-Analyse (DUOSTAT), und (ii) in situ EPS-Kennzeichnung. Wir bieten auch eine experimentelle Fall zeigt, wie das Tool-Box kann mit Biofilmen Analyse, Daten-Organisation, Integration und Interpretation zu unterstützen.
Protokoll
1. SCHRITT 1 - Bioassays
Der Biofilm-Verfahren nutzt Scheiben aus Hydroxylapatit (HA) als Surrogat-Zahn (Clarkson Chromatography Products, Inc., South Williamsport, PA, USA, und die Fläche = 2,7 ± 0,2 cm 2) beschichtet mit Speichel (Nachahmen der Anwesenheit von erworbenen Häutchen), platziert in eine vertikale Position 4, 5, 8.
- Biochemische Assays.
- Die Biofilme sind entweder (i) homogenisiert durch Beschallung 9 oder (ii) intakt gehalten für die biochemische Assays 8. Die homogenisierte Biofilmen Suspension kann zur Bestimmung der Biomasse verwendet werden (dry-Gewicht), Gesamt-Protein, anorganische und extra-und intrazellulären Polysaccharide Zusammensetzung / content (siehe Einzelheiten in Lemos et al., Protokolle der Physiologie des oralen Biofilmen 8 Studie). Die intakte Biofilme können für ihre physiologischen Reaktionen unter Verwendung von Standard glykolytischen pH-Drop-, Säure-killing, Proton Durchlässigkeit und F-ATPase-Aktivität Assays (siehe Details in Lemos et al., Protokolle der Physiologie des oralen Biofilmen 8 Studie) untersucht werden.
- Transkriptom-Analysen.
- Standard-cDNA-Microarray-und RT-qPCR-Protokolle (zB http://pfgrc.jcvi.org/index.php/microarray/protocols.html) kann in Kombination zur Bestimmung der Transkriptom-Reaktion von spezifischen Krankheitserregern (zB S. mutans) verwendet werden innerhalb von Biofilmen zu verschiedenen Umwelt-und therapeutische Herausforderungen in verschiedenen Entwicklungsstadien 6, 7, 10, 11. In der Tool-Box, haben wir zwei Methoden, die entscheidend für die Transkriptom-Analyse von dentalen Biofilmen sind: 1) RNA-Qualität (wegen der heterogenen Natur der Biofilm Bevölkerung und Anwesenheit von EPS-Matrix), und 2) Data Mining und Interpretation (durch großen Datenmengen erzeugt durch Microarray).
- RNA-Isolierung. Um die Frage der Qualität von RNA aus Biofilmen extrahiert Adresse verwenden wir ein optimiertes Protokoll speziell für die RNA-Isolierung und Aufreinigung von Bakterienzellen in EPS-reiche Matrix 12 (http://dx.doi.org/10.1016/j verstrickt entwickelt . ab.2007.03.021). Dieses Protokoll bietet RNA Integrity Number (RIN) größer ist als 8,5, was als optimal für die Transkriptom-Analyse mit beiden RT-qPCR und Microarrays (Beispiele siehe 6, 11).
- Standard-cDNA-Microarray-und RT-qPCR-Protokolle (zB http://pfgrc.jcvi.org/index.php/microarray/protocols.html) kann in Kombination zur Bestimmung der Transkriptom-Reaktion von spezifischen Krankheitserregern (zB S. mutans) verwendet werden innerhalb von Biofilmen zu verschiedenen Umwelt-und therapeutische Herausforderungen in verschiedenen Entwicklungsstadien 6, 7, 10, 11. In der Tool-Box, haben wir zwei Methoden, die entscheidend für die Transkriptom-Analyse von dentalen Biofilmen sind: 1) RNA-Qualität (wegen der heterogenen Natur der Biofilm Bevölkerung und Anwesenheit von EPS-Matrix), und 2) Data Mining und Interpretation (durch großen Datenmengen erzeugt durch Microarray).
- Fluoreszenz-Imaging. Strukturelle Organisation von Biofilmen.
- Die meisten der konfokalen Fluoreszenzmikroskopie Protokolle in der Literatur konzentriert sich auf die mikrobielle Kennzeichnung. Die Analyse der EPS als ein wichtiger Bestandteil des Biofilms wurde weitgehend in der oralen Biofilm-Forschung mit Fluoreszenz-Bildgebung vernachlässigt, mit wenigen Ausnahmen 5, 13, 14. Wir haben eine neue Kennzeichnung Technik und spezielle Software für reproduzierbare Visualisierung und Quantifizierung von EPS-und Bakterienzellen gleichzeitig innerhalb intakter Biofilm entwickelt. Amira 5 (Visage Imaging GmbH, Berlin, Deutschland) ist für die 3D-Rekonstruktion des Biofilmen verwendet, während COMSTAT (abrufbar unter http://www.imageanalysis.dk) und DUOSTAT (http://www.imageanalysis.dk) bieten die quantitative Analyse.
- Imaging der strukturellen Organisation der EPS-Bakterien in Biofilmen. Die Alexa Fluor 647-markiertem Dextran-Konjugat wird verwendet, um die EPS-Matrix zu visualisieren. Die Fluoreszenz-markierten Dextran dient als Grundierung für Streptokokken Glucosyltransferasen-Gtfs (insbesondere GtfB und GtfC), und kann gleichzeitig während der Exopolysaccharid Matrixsynthese im Laufe der Biofilmbildung 11 aufgenommen werden.
EPS Etikettierung:- Coat Hydroxyapatit-Discs mit Filter-sterilisiert Speichel für 1h, nach dem "Biofilm Preparation"-Protokoll 8.
- Pipette 2,8 ml Kulturmedium in jede Vertiefung von 24-Well-Platten und euch an einem dunklen Raum.
- Add 1 uM von Dextran konjugiert Alexa Fluor in Kulturmedium mischen Farbstoff in Kulturmedium durch Auf-und Abpipettieren (ca. 10 mal).
- Dip-waschen der Speichel-beschichteten HA (SHA)-Discs (nach 1 h Inkubation) zweimal in steriler AB-Puffer und legen Sie sie in das Medium mit Dextran konjugiert Alexa Fluor.
- Die Platte mit Alu-Folie und bei 37 ° C, 5% CO 2. Die Dauer der Inkubationszeit hängt von der jeweiligen experimentellen Designs. Die fluoreszenzmarkierten Dextran keine Flecken die bakteriellen Zellen in Konzentrationen in diesem als genutztsagen 11. Andere EPS-Färbetechniken, wie mit Calcofluor 14 können in Kombination verwendet werden.
- Bakterien Etikettierung:
- Bakterien in Biofilmen Komponenten werden am Ende der Biofilmbildung mit Standard-Fluoreszenz-Nukleinsäure-Färbung (zB SYTO 9) bezeichnet, andere Fluoreszenz-Techniken (zB Spezies-spezifisch fluoreszenzmarkierten Antikörpern 15 oder GFP-exprimierenden Zellen 16) kann natürlich verwendet werden erfassen ein oder mehrere Bakterienarten in den Biofilmen. Abbildung 2 zeigt die gleichzeitige Kennzeichnung von EPS (rot) und Bakterien / Mikrokolonien (grün) in ein 3D-Bild eines 24-h alten S. mutans Biofilm auf der Oberfläche eines SHA-Disc gebildet.
- Bilder Akquisition: Jeder Biofilm ist bei 5 bis 10 zufällig ausgewählten Positionen gescannt und z-Serie sind durch optische Schnitte an jeder dieser Positionen durch Laser-Scanning-konfokale Mikroskopie unter Verwendung von Standard-Protokollen erzeugt. Wir verwenden eine Olympus FV 1000 Zwei-Photonen-Mikroskop (Olympus, Tokyo, Japan) mit x10 ausgestattet (Carl Zeiss, numerische Apertur 0,45; Arbeitsabstand 3-4 mm) oder x25 (Olympus LPlan N; NA 1,05; WD 2 mm) Wasser Immersionsobjektive. Die Anregungswellenlänge beträgt 810 nm und Emissionswellenlänge Filter für SYTO 9 (Bakterien) 495/540 OlyMPFC1 Filter, während die Filter für Alexa Fluor 647 (EPS) ist HQ655/40M-2P Filter. Speichern und Speichern von RAW-Bildern.
- Imaging der strukturellen Organisation der EPS-Bakterien in Biofilmen. Die Alexa Fluor 647-markiertem Dextran-Konjugat wird verwendet, um die EPS-Matrix zu visualisieren. Die Fluoreszenz-markierten Dextran dient als Grundierung für Streptokokken Glucosyltransferasen-Gtfs (insbesondere GtfB und GtfC), und kann gleichzeitig während der Exopolysaccharid Matrixsynthese im Laufe der Biofilmbildung 11 aufgenommen werden.
- Die meisten der konfokalen Fluoreszenzmikroskopie Protokolle in der Literatur konzentriert sich auf die mikrobielle Kennzeichnung. Die Analyse der EPS als ein wichtiger Bestandteil des Biofilms wurde weitgehend in der oralen Biofilm-Forschung mit Fluoreszenz-Bildgebung vernachlässigt, mit wenigen Ausnahmen 5, 13, 14. Wir haben eine neue Kennzeichnung Technik und spezielle Software für reproduzierbare Visualisierung und Quantifizierung von EPS-und Bakterienzellen gleichzeitig innerhalb intakter Biofilm entwickelt. Amira 5 (Visage Imaging GmbH, Berlin, Deutschland) ist für die 3D-Rekonstruktion des Biofilmen verwendet, während COMSTAT (abrufbar unter http://www.imageanalysis.dk) und DUOSTAT (http://www.imageanalysis.dk) bieten die quantitative Analyse.
2. SCHRITT 2 - RAW DATA INPUT
Eingang Rohdaten aus biochemischen und RT-qPCR-Assays direkt in Raw Data File (RDF-MS Excel-Datei). Für Microarrays Daten, laden Single-Channel-Bilder von gescannten Dias in JCVI Spotfinder (http://pfgrc.jcvi.org/index.php/bioinformatics.html) oder ähnliche Software. Erstellen Sie einen Ort Gitter nach JCVI Spezifikationen und dann manuell anpassen, um alle Spots innerhalb des Gitters passt. Messen Sie die Helligkeitswerte jedes Spots und speichern in ". MeV"-Dateien gespeichert und in "Raw Microarray-Daten".
3. SCHRITT 3 - DATENVERARBEITUNG
Planen Sie den Rohdaten (biochemische und RT-qPCR) in der RDF für die statistische Analyse. Übertragen sie in "Daten verarbeitet File" (DPF - MS Excel-Datei). Für Microarray-und Fluoreszenz-Imaging-Analyse werden spezifische Software (derzeit verfügbar und maßgeschneidert) verwendet werden, um die Daten zu verarbeiten.
- Microarray Daten:
- Reichen Sie die Rohdaten in Schritt 2 (gespeichert in "Raw Microarrays data"), um Daten Normalisierung Schritt mit spezieller Software generiert. Normalisieren von Daten mit dem JCVI Microarray-Daten-Analyse-Software MIDAS (http://www.tm4.org/midas.html). Verwenden Sie LOWESS und Standardabweichung Regularisierung mit Standardeinstellungen, gefolgt von in-Bild-Analyse zu replizieren. Speichern Sie die Dateien mit den normalisierten Daten. In diesem Stadium (Rohdaten und normalisierte Daten-Dateien bereit), Kaution Microarray-Daten in eine öffentlich zugängliche Datenbank (zB NCBI Gene Expression Omnibus (GEO) Datenbank http://www.ncbi.nlm.nih.gov/geo) und notieren Sie die Zugangsnummer für die Zukunft.
- Fluoreszenz-Imaging-Daten
- Biofilm-Struktur Quantifizierung.
- Die quantitativen Daten, die der jeweiligen Bauteil (EPS und Bakterien) von Biofilmen wird durch COMSTAT und der neu entwickelte DUOSTAT, die als Skript in Matlab 5.1 geschrieben wurden, berechnet. Die rohen Fluoreszenzbilder sind in der Software hochgeladen und wie folgt analysiert:
- Verwenden Sie COMSTAT auf Biomasse, Dicke, Schicht für Vertrieb und sonstige Parameter, die Quantifizierung und Charakterisierung der dreidimensionalen Struktur von Biofilmen (siehe Handbuch auf http://www.imageanalysis.dk) zu berechnen.
- Verwenden Sie DUOSTAT die Co-Lokalisation von zwei Biofilm-Komponenten wie EPS und Bakterien (oder verschiedene mikrobielle Spezies und / oder extrazelluläre Komponenten) zu berechnen:
- Richten Sie den Pfad der DUOSTAT in MATLAB 5.1, und öffnen Bild Ordner, die die Bilder der beiden Biofilm Komponenten umfassen.
- Wählen Sie die zwei Kanäle, korreliert und analysiert werden. Die beiden Bildstapel muss auch identisch Pixelgrößen in allen drei Dimensionen (x, y, z), und die gleiche Anzahl von Bildern in jedem Stapel.
- Richten Sie Schwellenwerte für jeden Kanal nach dem MannUAL von COMSTAT. Folgen Sie den Anweisungen in der Bedienung der Software-Schnittstelle (siehe Video Artikel; DUOSTAT Handbuch unter http://www.imageanalysis.dk). Geben Sie die Daten in DPF erhalten. Siehe Beispiel in Abbildung 4.4.
- Dreidimensionale Rekonstruktion Biofilmen.
- Die dreidimensionale Architektur des Biofilmen wird visualisiert mit Amira. Importieren Sie die Fluoreszenzbilder in "Raw Images Folder" in Software gespeichert sind, und verwenden Sie VOLTEX und Iso-Oberflächen-Rendering zu 3-D Renderings der einzelnen Komponenten in den Biofilmen zu erstellen (siehe Amira Handbuch unter http://www.amira.com / documentation / manuals-and-release-notes.html). Siehe Beispiel auf Abbildung 4.4.
- Die quantitativen Daten, die der jeweiligen Bauteil (EPS und Bakterien) von Biofilmen wird durch COMSTAT und der neu entwickelte DUOSTAT, die als Skript in Matlab 5.1 geschrieben wurden, berechnet. Die rohen Fluoreszenzbilder sind in der Software hochgeladen und wie folgt analysiert:
- Biofilm-Struktur Quantifizierung.
4. SCHRITT 4 - Datenanalyse
Die quantitativen Daten aus biochemischen, RT-qPCR und COMSTAT-DUOSTAT Assays in den DPF sind bereit für die statistische Analyse. Nach der statistischen Analyse durchgeführt wird, kann Grafiken und / oder Tabellen aufgebaut (siehe "Repräsentative Ergebnisse") werden.
1. Microarray-Daten-Organisation mit der Software Microarray-Daten Visualizer (MDV).
Aufgrund der Komplexität und der Ausgang der großen Datenmengen bei der Verwendung von Microarrays und mehreren experimentellen Bedingungen haben wir ein Data-Mining-und Organisations-Software namens Microarray-Daten Visualizer (MDV) 7 (erhältlich bei http://www.oralgen.lanl.gov/) .
Nach Durchführung der statistischen Analyse mit BRB-ArrayTools (http://linus.nci.nih.gov/BRB-ArrayTools.html) mit einem Cutoff-P-Wert von 0,001 für die Klasse Vorhersage und für die Klasse gegenüber, können die Daten generiert eingereicht werden zum MDV, wie folgt:
- Mit Excel, konvertieren Sie die BRB-Daten in MDV tabulatorgetrennte Textdateien. Entfernen Sie alle Buchstaben, die mit dem Gen Locus-Tag-Nummer zu kommen, zum Beispiel: SMU.1423c, löschen Sie den Buchstaben "c".
- Öffnen MDV. Gehen Sie zu Datei, und wählen Sie "Select Annotation Source". Wählen Sie "Flat File". Wenn das Dialogfeld präsentiert, verwenden Sie die Browse-Tasten, um die Dateien, die Anmerkungen für die Gen-Namen, Gene Ontology (GO)-Nummer, Pfad und funktionelle Klassifizierung Daten enthalten wählen.
- Gehen Sie zu Datei, und importieren Sie die einzelnen Dateien aus Punkt 1. Jede der Dateien stellt die experimentellen Bedingungen für die Analyse. Wenn es mehrere Bedingungen werden zur Analyse, zum Beispiel zwei verschiedene Behandlungen (Behandlung 1 und 2) und Kontrolle (Fahrzeug) gehen zu Punkt 4.
- In diesem Fall sind die möglichen Vergleiche: (A) Behandlung 1 vs Kontrolle (für Gene differentiell exprimiert aufgrund der Behandlung 1), (B) Behandlung 2 vs Kontrolle und (C) Behandlung 1 vs Behandlung 2. Abhängig von der Arbeitshypothese, können andere Gruppe von Genen ausgewählt werden.
- Wählen Sie nur Gene differentiell exprimiert im Vergleich A (unique Gene durch die Behandlung 1 nur betroffen). In diesem Fall, verwenden Sie die Subtrahieren Funktion aus dem Set Functions-Menü. Subtrahieren Sie die Gene aus dem Vergleich B von A, und speichern Sie das Ergebnis. Dann subtrahieren C aus den erhaltenen Daten. Die gleichen Aktionen kann getan werden, um nur die Gene in B (unique Gene durch die Behandlung 2 nur betroffen) zu erhalten.
- Um nur die Gene in den beiden A-und B erkannt, die Union-Funktion aus dem Set Functions-Menü, das Ergebnis für den Vergleich A und B zu kombinieren, und speichern Sie das Ergebnis. Dann subtrahieren C der Ergebnisdaten.
- Diese Aktionen können visualisiert mit Venn-Diagramm, in MDV, die mehr intuitiv erscheinen, und erleichtert die Hypothese-driven Datenorganisation. Das Venn-Diagramm der Datenausgabe wird im Display angezeigt werden (siehe die Bildschirmansicht in Abbildung 3 und Video-Artikel).
- Zum Speichern der Daten auf tab-getrennte Textdateien, gehen Sie auf Datei und wählen Sie Exportieren.
- Exportieren Sie die Tab-begrenzte Textdateien. Öffnen Sie sie mit Hilfe von Excel und organisieren die Datenausgabe von MDV in Tabellen und / oder verdeckte, um eine grafische Darstellung (siehe Abbildungen 4.2, 4.3 und Video-Artikel).
5. Repräsentative Ergebnisse
Hier bieten wir ein Beispiel, wie die Analyse-Tool-Box integriert die verschiedenen Assays eines Biofilms Studie mit mehreren Variablen und experimentellen Bedingungen.
Experimentelle Fall:
Dynamics of Streptococcus mutans Transkriptom in Reaktion auf Stärke und Saccharose während Biofilm Entwicklung 7.
Hintergrund:
Die Wechselwirkungen von Nahrung Stärke und Saccharose mit Host-Speichel-Amylase und Streptokokken Glucosyltransferasen könnte die Bildung und Virulenz von S. mutans in Biofilmen durch InkrementeAsing Exopolysaccharide Synthese, Zuckerstoffwechsel und acidogenicity 11. Diese komplexe Wirt-Pathogen-Interaktion Ernährung modulieren kann die Bildung pathogener Biofilmen im Zusammenhang mit Karies Krankheit. Wir haben eine umfassende biochemische und Transkriptom-Analyse (einschließlich ganzer genomischen Profiling) zu verstehen, wie S. mutans reagiert auf Stärke und Saccharose in verschiedenen Stadien der Biofilmbildung in Gegenwart von Amylase 7.
Das Analyse-Tool-Box verwendet wurde, um uns bei der Integration der biochemischen und molekularen Untersuchungen von Biofilmen unter verschiedenen experimentellen Bedingungen und Zeitpunkten gebildet zu unterstützen. Die gesamten Daten-Ausgang mit dem Analyse-Tool-Box ist in einem sequentiell Weise in Abbildung 4 (4,1-4,4) vorgestellt. Es ist bemerkenswert, dass der Schwerpunkt hier, um die Nützlichkeit der Tool-Box, anstatt die Interpretation der Daten und die Diskussion zeigen wird.
- Die Kombination der biochemischen und RT-qPCR-Assays auf die experimentellen Bedingungen für die weitere Analyse-Microarrays zu wählen. Zunächst testeten wir 6 verschiedene Saccharose und 5 verschiedene Stärke und Saccharose-Kombinationen, um die optimale Konzentration der Kohlenhydrate für Biofilme Entwicklung von S. wählen mutans mit unseren in-vitro-Modell. Die Möglichkeit, gleichzeitig die Daten untersuchen Ausgabe des Bioassays führte uns in die Auswahl von drei bestimmten Konzentrationen (hervorgehoben in Abbildung 4.1), basierend auf (erhöhte) Menge an unlöslichen Exopolysaccharide und (enhanced) Ausdruck GtfB (verantwortlich für unlösliche Glucan-Synthese) in den Biofilmen .
- Microarray-Analyse. Die ausgewählte (drei) Konzentrationen der Kohlenhydrate aus der Nahrung wurden verwendet, um Biofilme bilden, die zu bestimmten Zeitpunkten widerspiegelt die verschiedenen Stufen von Biofilmen Entwicklungsprozess entfernt wurden. Die Biofilme (unterteilt in 3 Versuchsgruppen und 4 Zeit-Punkte) wurden Transkriptom-Analyse durch die Kombination gesamten genomischen Profiling (cDNA-Microarrays) mit BRB-Array-Tools und MDV unterzogen.
Die RNA wurde extrahiert und gereinigt mit Biofilm-spezifisches Protokoll 12, und dann einer Analyse durch die 4 Stufen-Verfahren des Analytical Tool-Box-Mikroarray. Abbildung 4.2 zeigt, wie die MDV dazu beigetragen, die Gene von Interesse nach unserer Arbeitshypothese, dass Saccharose und Stärke Kombination spezifische transkriptionelle Antwort mit erhöhter Virulenz (kariogenen Potenzial) von S. zugeordneten Trigger wählen mutans in Biofilmen. Die Rohdaten von BRB-ArrayTools (Abbildung 4.2a) generiert wurde von MDV mit dem Venn-Diagramm (Abb. 4.2b) verarbeitet. In dieser Studie sind die Gruppe von Genen im Zusammenhang mit unserer Hypothese die unterschiedlich im Vergleich zum Ausdruck A (0,5% Saccharose + 1% Stärke vs 1% Saccharose) und B (0,5% Saccharose + 1% Stärke vs 0,5% Saccharose), aber nicht die Gene im Vergleich C (0,5% Saccharose vs 1% Saccharose) (Abbildung 4.2a und 4.2b). Wie in Abbildung 4.2c gezeigt, der MDV stark reduziert die Gesamtzahl der Gene zu analysieren und gleichzeitig die herausgefilterten die Gene nicht direkt auf den Einfluss von Saccharose und Stärke in Kombination verwandt. Die MDV Datenausgabe wurde auch eine grafische Darstellung zeigt die Daten von funktionellen Klasse der einzelnen Gene differentiell exprimiert (up-und down-regulierten) in der Stärke organisiert umgewandelt + Saccharose-Biofilmen (vs. Saccharose-grown Biofilme) an jedem der 4 Zeitpunkten (Abbildung 4,3). Das MDV-Software ist benutzerfreundlich und erleichtert den Bergbau / Organisation der große und komplexe Daten aus unserer Microarray-Experimenten setzt. - . Biofilm Bildgebung Begleitend die strukturelle Organisation der Biofilme wurde mittels COMSTAT-DUOSTAT-Amira enthalten in der Tool-Box (siehe ein Beispiel für die 3D-Rekonstruktion und quantitative Analyse der Stärke + Saccharose-grown Biofilm in Abbildung 4.4, siehe auch Video-Artikel). Die Morphologie, Verteilung und strukturelle Verwandtschaft von EPS und Mikrokolonien visualisiert werden können mit 3D-Oberflächen-Rendering von Amira. Close-up Blick auf ausgewählten Bereich erzeugt werden, was zeigt, bestimmte Bakterien-EPS strukturellen Beziehungen auf der Mikroebene (Abb. 4.4). Darüber hinaus kann die gleiche Menge von konfokalen Bildern von COMSTAT-DUOSTAT für Biomasse / Mikrokolonie Messungen, räumliche Verteilung und Kolokalisation von Bakterienzellen und EPS gleichzeitig verarbeitet werden. Zum Beispiel wurde die vertikale Verteilung von EPS und Bakterien aus Disc-Oberfläche zur Fluid-Phase von jedem optischen Schnitt der dreidimensionalen konfokalen Biofilm Bilder mit COMSTAT-DUOSTAT berechnet (Grafik in Abbildung 4.4;. Siehe Video Artikel). Die Daten zeigen einen höheren Anteil an EPS (rote Linie) als Bakterien (grüne Linie) in den Biofilmen Tiefe. Außerdem sind die meisten der Bakterienzellen mit EPS (blaue Linie) verbunden, vor allem in den mittleren und äußeren Schichten des Biofilms. Diese Beobachtung deutet darauf hin, dass Biofilme in Stärke und Saccharose gewachsen besonders reich an exopolymers sind, sind die Kiemen (in engen Kontakt mit) die meisten der Bakterienzellen, wie strukturelle Organisation verbessert die Stabilität und den Zusammenhalt der Biofilme 13. Die dreidimensionalen Darstellungen und quantitative Messungen der konfokalen Bilder liefern zusätzliche Informationen über die Struktur des Biofilms, der die biochemische und Genexpressionsdaten ergänzt (erhöhte GtfB-type unlöslich-Glucan-Synthese durch S. mutans in Stärke + Saccharose-grown Biofilme) (für weitere Beispiele 5 zu sehen, 6, 11, 13).
Das Analyse-Tool-Box unterstützt uns zu erhalten, zu organisieren und die Integration der Daten aus verschiedenen Bioassays, die eine umfassende Analyse, wie S. vorgesehen mutans kann bis zu komplexen Veränderungen der Umwelt als Folge der Diät-Wirt-Interaktionen in der Mundhöhle gefunden zu reagieren (siehe Details in Klein et al, 2009 11;.. Klein et al, 2010 7).

Abbildung 1. Flow-Chart des Analytical Tool-Box für Biofilm-Analyse.

Abbildung 2. Konfokale Fluoreszenz-Imaging von EPS und Bakterien in Biofilmen. Gleichzeitige Darstellung von EPS (rot) und Bakterien / Mikrokolonien (grün) in die dreidimensionale Darstellung von Streptococcus mutans Biofilm auf SHA Disc-Oberfläche gebildet.

Abbildung 3. Microarray Data Mining und Organisation mittels Microarray-Daten Visualizer (MDV) Software. Verwendung von Venn-Diagramm-Funktion zu wählen aus Genen von Interesse, und gefolgt von der Zugabe von Gen-Namen und-Funktionsklasse Annotation.

Abbildung 4.1. Evaluation der Biofilmbildung von S. mutans mittels biochemischer Assays (A) und RT-qPCR (B). Die INS (unlöslich Exopolysaccharide) Daten korrelieren gut mit dem Muster der GtfB Ausdruck, und mit der Biomasse der Biofilme. Saccharose bei 1% war die Konzentration für eine maximale INS Bildung, GtfB Ausdruck und Biofilm Akkumulation auf der SHA Oberfläche, während 0,5% Saccharose die minimale Konzentration für eine optimale Entwicklung benötigt Biofilm mit war unsere in-vitro-Modell. S. mutans-Zellen in Anwesenheit von 0,5% Saccharose gewachsen + 1% Stärke ergab die höchste Biomasse und präsentiert mehr INS als andere Biofilmen, die mit erweiterten GtfB Ausdruck (B2) korreliert. Diese Kohlenhydratkonzentrationen wurden für weitere Transkriptom-Analyse ausgewählt.
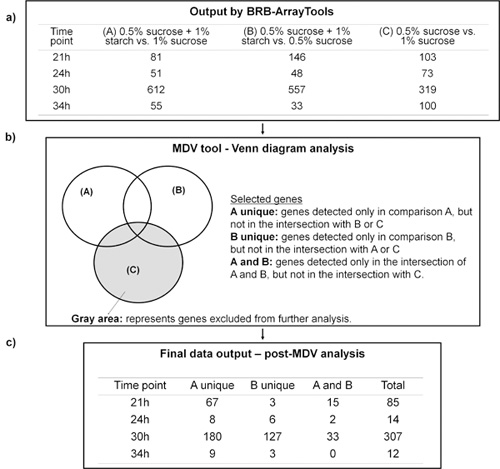
Abbildung 4.2. Microarray Datenanalyse mit BRB-Array-Tools in Verbindung mit MDV-Software. a) Stellen Sie die Anzahl der Gene als differentiell in jedem Vergleich (A, B oder C) und Zeitpunkt evaluiert mit BRB-Array-Tools zum Ausdruck erkannt. b) Microarray-Daten Visualizer (MDV) mit Hilfe des Venn-Diagramm, um Gene von Interesse auszuwählen. c) Gene ausgewählt nach MDV-Analyse.

Abbildung 4.3. S. mutans Gene differentiell in Stärke ausgedrückt + Saccharose-Biofilmen (vs. Saccharose-Biofilme) an verschiedenen Zeitpunkten durch funktionelle Klasse organisiert. Gene Anmerkungen beruhen auf den Angaben der Los Alamos National Laboratory (www.oralgen.lanl.gov) oder durch veröffentlichte Literatur zur gleichen Webseite bereitgestellt werden.

Abbildung 4.4. Dreidimensionale Darstellung und COMSTAT-DUOSTAT Analyse der Stärke + Saccharose-Biofilm.
Access restricted. Please log in or start a trial to view this content.
Diskussion
In dieser Präsentation haben wir gezeigt, zwei kritische Komponenten des Analytical Tool-Box (EPS / Bakterien Bildgebung und Microarray Data Mining / Verarbeitung), die Vielseitigkeit und Brauchbarkeit der verschiedenen Tests in das System integriert. Offensichtlich ist die Tool-Box das umfassende (Vergleich) und die gleichzeitige Analyse der verschiedenen Aspekte der Biofilme Biochemie, Architektur und Genexpression als Reaktion auf die verschiedenen experimentellen Bedingungen mit Hilfe eines in-vitro-Modell...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Danksagungen
Die Autoren bedanken sich bei Dr. Gary Xie und Herbert Lee für die Entwicklung des MDV danken. Wir danken auch Drs. Simone Duarte, Ramiro Murata, Jae-Gyu Jeon, Jacqueline Abranches und Frau Stacy Gregoire für ihre technischen und wissenschaftlichen Beitrag für die analytische Komponenten der Tool-Box. Diese Studie wurde zum Teil durch USPHS Forschungsstipendium DE018023 vom National Institute of Dental und kraniofaziale Forschung gefördert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Syto 9 | Invitrogen | S34854 | |
| Syto 60 | Invitrogen | S11342 | |
| Dextran conjugated alexa 647 | Invitrogen | D22914 | |
| Olympus FV1000 two-photon laser scanning microscope | Olympus Corporation |
Referenzen
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- Leme, P. aes, Koo, A. F., Bellato, H., Bedi, C. M., G, J. A. C. ury The role of sucrose in cariogenic dental biofilm formation--new insight. J Dent Res. 85, 878-887 (2006).
- Koo, H., Schobel, B. D., Scott-Annem, K., Watson, G., Bowen, W. H., Cury, J. A., Rosalen, P. L., Park, Y. K. Apigenin and tt-farnesol with fluoride effects on S. mutans biofilms and dental caries. J Dent Res. 84, 1016-1020 (2005).
- Koo, H., Xiao, J., Klein, M. I., Jeon, J. G. Exopolysaccharides produced by Streptococcus mutans glucosyltransferases modulate the establishment of microcolonies within multispecies biofilms. J Bacteriol. 192, 3024-3032 (2010).
- Jeon, J. G., Klein, M. I., Xiao, J., Gregoire, S., Rosalen, P. L., Koo, H. Influences of naturally occurring agents in combination with fluoride on gene expression and structural organization of Streptococcus mutans in biofilms. BMC Microbiol. 9, 228-228 (2009).
- Klein, M. I., DeBaz, L., Agidi, S., Lee, H., Xie, G., Lin, A. H. M., Hamaker, B. R., Lemos, J. A., Koo, H. Dynamics of Streptococcus mutans transcriptome in response to starch and sucrose during biofilm development. PLoS ONE. , 0013478-0013478 (2010).
- Lemos, J. A., Abranches, J., Koo, H., Marquis, R. E., Burne, R. A. Protocols to Study the Physiology of Oral Biofilms. Methods Mol Biol. 666, 87-102 (2010).
- Koo, H., Hayacibara, M. F., Schobel, B. D., Cury, J. A., Rosalen, P. L., Park, Y. K., Vacca-Smith, A. M., Bowen, W. H. Inhibition of Streptococcus mutans biofilm accumulation and polysaccharide production by apigenin and tt-farnesol. J Antimicrob Chemother. 52, 782-789 (2003).
- Koo, H., Seils, J., Abranches, J., Burne, R. A., Bowen, W. H., Quivey, R. G. Influence of apigenin on gtf gene expression in Streptococcus mutans UA159. Antimicrob. Agents Chemother. 50, 542-546 (2006).
- Klein, M. I., Duarte, S., Xiao, J., Mitra, S., Foster, T. H., Koo, H. Structural and molecular basis of the role of starch and sucrose in Streptococcus mutans biofilm development. Appl Environ Microbiol. 75, 837-841 (2009).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Anal Biochem. 365, 208-214 (2007).
- Xiao, J., Koo, H. Structural organization and dynamics of exopolysaccharide matrix and microcolonies formation by Streptococcus mutans in biofilms. J Appl Microbiol. 108, 2103-2113 (2010).
- Thurnheer, T., Gmür, R., Shapiro, S., Guggenheim, B. Mass transport of macromolecules within an in vitro model of supragingival plaque. Appl Environ Microbiol. 69, 1702-1709 (2003).
- Chalmers, N. I., Palmer, R. J. J. r, Du-Thumm, L., Sullivan, R., Shi, W., Kolenbrander, P. E. Use of quantum dot luminescent probes to achieve single-cell resolution of human oral bacteria in biofilms. Appl Environ Microbiol. 73, 630-636 (2007).
- Deng, D. M., Hoogenkamp, M. A., Ten Cate, J. M. >, Crielaard, W. Novel metabolic activity indicator in Streptococcus mutans biofilms. J Microbiol Methods. 77, 67-71 (2009).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten