Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Аналитический инструмент коробки комплексных биохимические, структурные и транскриптома Оценка устного Биопленки посредничестве Mutans стрептококков
В этой статье
Резюме
Биопленки образуются на поверхности зубов, являются очень сложными и подвергаются постоянной врожденной и экзогенных экологических проблем, которые модулируют их архитектуре, физиологии и транскриптом. Мы разработали инструментарий для изучения состава, структурной организации и экспрессии генов устных биопленок, которые могут быть адаптированы к другим областям биопленки исследований.
Аннотация
Biofilms are highly dynamic, organized and structured communities of microbial cells enmeshed in an extracellular matrix of variable density and composition 1, 2. In general, biofilms develop from initial microbial attachment on a surface followed by formation of cell clusters (or microcolonies) and further development and stabilization of the microcolonies, which occur in a complex extracellular matrix. The majority of biofilm matrices harbor exopolysaccharides (EPS), and dental biofilms are no exception; especially those associated with caries disease, which are mostly mediated by mutans streptococci 3. The EPS are synthesized by microorganisms (S. mutans, a key contributor) by means of extracellular enzymes, such as glucosyltransferases using sucrose primarily as substrate 3.
Studies of biofilms formed on tooth surfaces are particularly challenging owing to their constant exposure to environmental challenges associated with complex diet-host-microbial interactions occurring in the oral cavity. Better understanding of the dynamic changes of the structural organization and composition of the matrix, physiology and transcriptome/proteome profile of biofilm-cells in response to these complex interactions would further advance the current knowledge of how oral biofilms modulate pathogenicity. Therefore, we have developed an analytical tool-box to facilitate biofilm analysis at structural, biochemical and molecular levels by combining commonly available and novel techniques with custom-made software for data analysis. Standard analytical (colorimetric assays, RT-qPCR and microarrays) and novel fluorescence techniques (for simultaneous labeling of bacteria and EPS) were integrated with specific software for data analysis to address the complex nature of oral biofilm research.
The tool-box is comprised of 4 distinct but interconnected steps (Figure 1): 1) Bioassays, 2) Raw Data Input, 3) Data Processing, and 4) Data Analysis. We used our in vitro biofilm model and specific experimental conditions to demonstrate the usefulness and flexibility of the tool-box. The biofilm model is simple, reproducible and multiple replicates of a single experiment can be done simultaneously 4, 5. Moreover, it allows temporal evaluation, inclusion of various microbial species 5 and assessment of the effects of distinct experimental conditions (e.g. treatments 6; comparison of knockout mutants vs. parental strain 5; carbohydrates availability 7). Here, we describe two specific components of the tool-box, including (i) new software for microarray data mining/organization (MDV) and fluorescence imaging analysis (DUOSTAT), and (ii) in situ EPS-labeling. We also provide an experimental case showing how the tool-box can assist with biofilms analysis, data organization, integration and interpretation.
протокол
1. ШАГ 1 - биопробы
Биопленки метод использует диски гидроксиапатита (ГА) в качестве суррогатной зуба (Кларксон хроматографии Products, Inc, Южной Уильямспорт, Пенсильвания, США; площадь поверхности = 2,7 ± 0,2 см 2) покрытый слюной (имитируя наличие приобрел пленку), размещаемых в вертикальном положении, 4, 5, 8.
- Биохимические анализы.
- Биопленки либо (я) гомогенизированные ультразвуком 9 или (II) сохранены для биохимических анализов 8. Гомогенизированные подвески биопленки могут быть использованы для определения биомассы (сухой вес), общий белок, неорганических и внеклеточных и внутриклеточных полисахаридов Состав / содержание (подробности см. в Лемос и соавт., Протоколы по изучению физиологии Устные Биопленки 8). Нетронутым биопленки могут быть проанализированы для своих физиологических реакций с использованием стандартных гликолитических рН-капля, кислотно-убийства, протонной проницаемости и F-АТФазы анализов деятельности (подробности см. в Лемос и соавт., Протоколы по изучению физиологии Устные Биопленки 8).
- Транскриптома анализа.
- Стандартный кДНК микрочипов и РТ-КПЦР протоколов (например http://pfgrc.jcvi.org/index.php/microarray/protocols.html) может быть использован в комбинации для определения транскриптома-ответ конкретных патогенных микроорганизмов (например, С. mutans) в пределах биопленки на различные экологические и терапевтических задач на различных стадиях развития 6, 7, 10, 11. В ящик для инструмента, мы включили два метода, которые имеют решающее значение для транскриптом анализ зубов биопленки: 1) качество РНК (из-за гетерогенный характер биопленки населения и наличие EPS-матрица), и 2) анализ данных и интерпретация (из-за большой набор данных, порожденных микрочипов).
- РНК изоляции. Чтобы решить вопрос качества РНК извлекается из биопленки, мы используем оптимизированный протокол, разработанный специально для РНК выделения и очистки от бактериальных клеток запуталась в EPS богатых матрицы 12 (http://dx.doi.org/10.1016/j . ab.2007.03.021). Этот протокол обеспечивает целостность РНК номер (РИН) выше, чем 8.5, которая считается оптимальной для транскриптом анализа с использованием как RT-КПЦР и Microarrays (примеры см. 6, 11).
- Стандартный кДНК микрочипов и РТ-КПЦР протоколов (например http://pfgrc.jcvi.org/index.php/microarray/protocols.html) может быть использован в комбинации для определения транскриптома-ответ конкретных патогенных микроорганизмов (например, С. mutans) в пределах биопленки на различные экологические и терапевтических задач на различных стадиях развития 6, 7, 10, 11. В ящик для инструмента, мы включили два метода, которые имеют решающее значение для транскриптом анализ зубов биопленки: 1) качество РНК (из-за гетерогенный характер биопленки населения и наличие EPS-матрица), и 2) анализ данных и интерпретация (из-за большой набор данных, порожденных микрочипов).
- Флуоресценции. Структурная организация биопленки.
- Большинство протоколов конфокальной флуоресцентной визуализации имеющиеся в литературе сосредоточена на микробную маркировки. Анализ EPS в качестве основного компонента биопленки в значительной степени пренебрегали в устной исследования биопленки с участием флуоресценции, за редким исключением 5, 13, 14. Мы разработали методику новой маркировки и специальное программное обеспечение для воспроизводимых визуализации и количественной оценки EPS и бактериальные клетки одновременно в пределах нетронутыми биопленки. Амира 5 (Visage изображений GmbH, Берлин, Германия) используется для 3D-реконструкцию биопленок, в то время КОМСТАТ (продается в http://www.imageanalysis.dk) и DUOSTAT (http://www.imageanalysis.dk) обеспечивают количественного анализа.
- Изображений структурной организации EPS-бактерий в биопленки. Alexa Fluor 647-меченого декстрана сопряженных используется для визуализации EPS-матрицы. Меченных флуоресцентными декстран служит грунт для стрептококковой glucosyltransferases-Gtfs (в частности, GtfB и GtfC), и могут быть одновременно включены в экзополисахарида синтеза матрицы в течение биопленки развития 11.
EPS маркировке:- Пальто гидроксиапатита диски с фильтром стерилизовать слюны в течение 1 ч, после "биопленки Препарат" протокол 8.
- Пипетка 2,8 мл культуральной среды в каждую лунку 24 луночного, и довести до темной комнате.
- Добавить 1 мкМ декстрана сопряженных Alexa Fluor в культуральной среде, смешать краску в культуральной среде с помощью пипетки вверх и вниз (примерно в 10 раз).
- Dip-мытье слюной покрытием HA (SHA) диски (после 1 ч инкубации) два раза в стерильном буфере AB и поместите их в среде, содержащей декстран сопряженных Alexa Fluor.
- Обложка пластинки с алюминиевой фольгой и инкубировать при температуре 37 ° C, 5% СО 2. Продолжительность инкубационного периода зависит от каждого эксперимента. Флуоресцентно меченого декстрана не окрашивает бактериальных клеток в концентрациях, используемых в этом качествеговорят 11. Другие ЭПС-окрашивание методы, такие как с Calcofluor 14, могут быть использованы в комбинации.
- Бактерии маркировке:
- Бактерии компонентов в биопленки помечены в конце биопленки с использованием стандартных флуоресцентных нуклеиновой кислоты пятна (например, SYTO 9); другие флуоресценции методов (например, конкретные виды люминесцентных меченых антител 15 или GFP-клеток, экспрессирующих 16) можно, конечно, используется для обнаружения одного или нескольких видов бактерий в биопленки. На рисунке 2 показан одновременной маркировки EPS (красный) и бактерии / микроколоний (зеленый) в 3D-изображение 24-х старых С. mutans биопленки образуются на поверхности диска ССЗ.
- Изображения приобретения: Каждый биопленки сканируется на 5 до 10 случайно выбранных позиций, а серии Z генерируются оптического секционирования на каждой из этих позиций лазерной сканирующей конфокальной микроскопии с использованием стандартных протоколов. Мы используем Olympus FV 1000 двухфотонного микроскопа (Olympus, Токио, Япония), оснащенных x10 (Carl Zeiss, числовая апертура 0,45; рабочее расстояние 3-4 мм) или x25 (Olympus LPlan N; Н. А. 1,05; WD 2 мм) воды погружение целей. Возбуждения длина волны 810 нм, а длина волны излучения фильтр для SYTO 9 (бактерий) 495/540 OlyMPFC1 фильтр, а фильтр для Alexa Fluor 647 (EPS) является HQ655/40M-2P фильтр. Сохранить и хранения необработанных изображений.
- Изображений структурной организации EPS-бактерий в биопленки. Alexa Fluor 647-меченого декстрана сопряженных используется для визуализации EPS-матрицы. Меченных флуоресцентными декстран служит грунт для стрептококковой glucosyltransferases-Gtfs (в частности, GtfB и GtfC), и могут быть одновременно включены в экзополисахарида синтеза матрицы в течение биопленки развития 11.
- Большинство протоколов конфокальной флуоресцентной визуализации имеющиеся в литературе сосредоточена на микробную маркировки. Анализ EPS в качестве основного компонента биопленки в значительной степени пренебрегали в устной исследования биопленки с участием флуоресценции, за редким исключением 5, 13, 14. Мы разработали методику новой маркировки и специальное программное обеспечение для воспроизводимых визуализации и количественной оценки EPS и бактериальные клетки одновременно в пределах нетронутыми биопленки. Амира 5 (Visage изображений GmbH, Берлин, Германия) используется для 3D-реконструкцию биопленок, в то время КОМСТАТ (продается в http://www.imageanalysis.dk) и DUOSTAT (http://www.imageanalysis.dk) обеспечивают количественного анализа.
2. ШАГ 2 - RAW ДАННЫХ
Входной необработанные данные из биохимических и РТ-КПЦР анализов непосредственно в сырье файла данных (RDF-файл MS Excel). Для микрочипов данных, нагрузка одноканального изображения отсканированных слайдов в JCVI Spotfinder (http://pfgrc.jcvi.org/index.php/bioinformatics.html) или аналогичное программное обеспечение. Создать месте сетки по JCVI спецификаций, а затем вручную настроить, чтобы соответствовать все места в сетке. Измерьте интенсивность значения каждого пятна и сохранить в ". МэВ" файлы и хранятся в "Исходные данные Microarray".
3. ШАГ 3 - ОБРАБОТКИ ДАННЫХ
Организация сырые данные (биохимические и РТ-КПЦР) в RDF для статистического анализа. Передача их в "данных, обрабатываемых файлов" (DPF - MS Excel файл). Для анализа изображений микрочипов и флуоресценция, специальное программное обеспечение (в настоящее время в наличии и на заказ) используются для обработки данных.
- Microarray данных:
- Отправить сырые данные, полученные на шаге 2 (хранится в "Исходные данные Microarrays") для нормализации данных шаг, используя специальное программное обеспечение. Нормализация данных с помощью микрочипов JCVI анализа данных программное обеспечение MIDAS (http://www.tm4.org/midas.html). Используйте LOWESS и стандартные отклонения регуляризации с настройками по умолчанию, а затем в слайд повторить анализ. Хранить файлы, содержащие нормированных данных. На этом этапе (исходных файлов данных и файлов данных нормированы в состоянии готовности), депозитные микрочипов данных в базу данных доступа (например, NCBI экспрессии генов Omnibus (GEO) базы данных в http://www.ncbi.nlm.nih.gov/geo), и записать регистрационный номер, для дальнейшего использования.
- Флуоресценции данных
- Биопленка структуры количественной оценке.
- Количественные данные, соответствующие каждой структурной составляющей (EPS и бактерий) биопленок рассчитывается путем КОМСТАТ и недавно разработанный DUOSTAT, которые были написаны как скрипт в MATLAB 5.1. Сырья флуоресцентные изображения, загруженные в программном обеспечении и проанализировать следующим образом:
- Используйте КОМСТАТ для расчета биомассы, толщина слоя распределения и других параметров, которые характеризуют количество и трехмерную структуру биопленки (см. руководство по http://www.imageanalysis.dk).
- Используйте DUOSTAT для расчета со-локализация двух биопленки компонентов, таких как EPS и бактерий (или разных видов микроорганизмов и / или компонентов внеклеточного):
- Настройка пути DUOSTAT в MATLAB 5.1 и открывать папки изображений, которые включают изображения двух компонентов биопленки.
- Выберите два канала, который будет коррелировать и анализировать. Две стопки изображения также должны иметь одинаковые размеры пикселя во всех трех измерениях (х, у, г), и то же количество изображений в каждом стеке.
- Настройка пороговых значений для каждого канала в соответствии с человекаUAL из КОМСТАТ. Следуйте инструкциям в интерфейсе работы программного обеспечения (см. видео статьи; DUOSTAT руководство в http://www.imageanalysis.dk). Входные данные, полученные в DPF. См. пример на рисунке 4.4.
- Трехмерная реконструкция биопленки.
- Трехмерной архитектуры биопленки визуализируется использованием Амира. Импорт флуоресценции изображений, хранящихся в "Raw Папка Images" в программное обеспечение, а также использовать Voltex и изо-поверхности рендеринга для создания 3-D визуализации каждого из компонентов в биопленки (см. Амира руководство в http://www.amira.com / документации / пособий и отпустить-notes.html). См. пример на рисунке 4.4.
- Количественные данные, соответствующие каждой структурной составляющей (EPS и бактерий) биопленок рассчитывается путем КОМСТАТ и недавно разработанный DUOSTAT, которые были написаны как скрипт в MATLAB 5.1. Сырья флуоресцентные изображения, загруженные в программном обеспечении и проанализировать следующим образом:
- Биопленка структуры количественной оценке.
4. ШАГ 4 - АНАЛИЗ ДАННЫХ
Количественные данные биохимических, RT-КПЦР и КОМСТАТ-DUOSTAT анализы в DPF готовы для статистического анализа. После статистического анализа выполняется, графики и / или таблицы может быть построена (см. "Результаты представитель" раздел).
1. Microarray организации данных с помощью программного обеспечения Microarray данных Visualizer (МДВ).
Из-за сложности и вывода больших массивов данных при использовании микрочипов и нескольких экспериментальных условиях, мы разработали программное обеспечение интеллектуального анализа данных и организации под названием Microarray данных Visualizer (МДВ) 7 (доступны на http://www.oralgen.lanl.gov/) .
После проведения статистического анализа с использованием BRB-ArrayTools (http://linus.nci.nih.gov/BRB-ArrayTools.html) со значением отсечки Р 0,001 для класса прогнозирования и для класса сравнения, данные, полученные могут быть представлены для MDV, следующим образом:
- Использование Excel, конвертировать данные в BRB MDV с разделителями табуляции текстовых файлов. Удалить все письма, которые приходят с Номер тэга Локус генов, например: SMU.1423c, удалить букву "с".
- Открытое MDV. Переход к файлу, и выберите "Select Аннотация Источник". Выберите «плоский файл». Сталкиваясь с диалогового окна, используйте кнопки Обзор, чтобы выбрать файлы, которые содержат аннотации для генной имя, Джин Онтология (ГО) номер, путь, и функциональной классификации данных.
- Переход к файлу, а импорт каждого из файлов из пункта 1. Каждый из файлов представляет экспериментальные условия для проведения анализа. Если Есть несколько условий для анализа, например, две различные процедуры (лечение 1 и 2) и контроля (транспортного средства) перейти к пункту 4.
- В этом случае, возможно сравнения: (А) лечение 1 по сравнению с контрольной (для генов, дифференциально экспрессирующихся в результате лечения 1); (Б) лечение 2 против контроля, и (C) лечение 1 vs лечения 2. В зависимости от рабочей гипотезы, различный набор генов могут быть выбраны.
- Выберите только гены разному экспрессируются в сравнении (уникальные гены, пострадавшим от лечения 1 только). В этом случае воспользуйтесь функцией вычитания из меню функций множества. Вычтите из сравнения генов B от и сохранить результат. Затем вычитаем С от полученных данных. Те же действия можно сделать, чтобы получить только гены B (уникальные гены, пострадавшим от лечения 2 только).
- Чтобы выделить только гены обнаружены в А и Б, используйте Союза функцию из меню Функции множества объединить результаты для сравнений и В, и сохранить результат. Затем вычитаем C из результата запроса.
- Эти действия могут быть визуализированы использованием диаграммы Венна, Лучшее в MDV, которая является более интуитивным, а также облегчает гипотеза управляемой организации данных. Диаграммы Венна выходных данных будут отображаться на экране (см. экран на рисунке 3 и видео статьи).
- Чтобы сохранить данные с разделителями табуляции текстовых файлов, перейдите в меню Файл и выберите Экспорт.
- Экспорт с разделителями табуляции текстовых файлов. Открытые их с помощью Excel, а также организовать выход данных от MDV в виде таблиц и / или скрытого для графического дисплея (рис. 4.2, 4.3 и видео статьи).
5. ПРЕДСТАВИТЕЛЬ РЕЗУЛЬТАТЫ
Здесь мы приведем пример того, как аналитический инструмент коробки интегрирует различные анализы биопленки исследования с нескольких переменных и условий эксперимента.
Экспериментальные случае:
Динамика mutans Streptococcus транскриптома в ответ на крахмала и сахарозы в течение биопленки развития 7.
Справочная информация:
Взаимодействий диетического крахмала и сахарозы с принимающими слюнных амилазы и стрептококковой glucosyltransferases может повысить образование и вирулентности S. mutans в пределах биопленки на приращенияasing экзополисахариды синтеза, обмена веществ и сахара acidogenicity 11. Этот комплекс хозяин-патоген-диета взаимодействие может модулировать образование патогенных биопленки, связанные с стоматологических заболеваний кариесом. Мы провели всесторонний биохимических и transcriptomic анализа (в том числе целый профилирования геномной) в целях дальнейшего понять, как С. mutans реагирует на крахмала и сахарозы в различных стадиях развития биопленки в присутствии амилазы 7.
Аналитический инструмент, коробки были использованы, чтобы помочь нам в интеграции биохимические и молекулярные анализы биопленки образуются при различных экспериментальных условиях и временных точках. Общий объем производства данных с использованием аналитических инструментов окна представлен в последовательно образом на рисунке 4 (4,1 до 4,4). Стоит отметить, что основное внимание здесь, чтобы продемонстрировать полезность набора инструментальных средств, а не интерпретации данных и обсуждения.
- Сочетание биохимических и РТ-КПЦР анализы, чтобы выбрать условия эксперимента для дальнейшего анализа микрочипов. Первоначально, мы протестировали 6 различных сахарозы и 5 различных крахмала и сахарозы комбинации, чтобы выбрать оптимальные концентрации углеводов для развития биопленки С. mutans используя нашу модель в пробирке. Способность исследовать одновременно вывода данных из биопробы мы руководствовались при выборе трех конкретных концентраций (выделена на рисунке 4.1) на основе (повышенный) количество нерастворимого экзополисахариды и (расширенный) выражение gtfB (отвечает за синтез нерастворимого глюкана) в биопленки .
- Microarray анализа. Выбран (три) концентрации пищевые углеводы были использованы для формирования биопленок, которые были удалены в определенных временных точках отражающих различные этапы процесса развития биопленки. Биопленок (разделено на 3 экспериментальные группы и 4 временных точках) были подвергнуты анализу транскриптом, объединив весь профилирование генома (кДНК микрочипы), с BRB-Array Инструменты и MDV.
РНК выделяли и очищали с использованием биопленки конкретного протокола 12, а затем подвергается микрочипов анализа через 4 шаг процесса аналитического инструмента-Box. Рисунок 4.2 иллюстрирует, как MDV помогли выбрать гены интерес в соответствии с нашей рабочей гипотезы, что сахароза и крахмал сочетание вызывает специфический транскрипционный ответ связан с повышенной вирулентностью (кариесогенных потенциал) С. mutans в биопленки. Сырые данные, полученные в BRB-ArrayTools (рис. 4.2а) было обработано MDV использованием диаграммы Венна (рис. 4.2b). В данном исследовании группа генов, связанных с нашей гипотезой те разному экспрессируются в сравнении (0,5% сахарозы + 1% крахмала по сравнению с 1% сахарозы) и B (0,5% сахарозы + 1% крахмала по сравнению с 0,5% сахарозы), но не генами в сравнении C (0,5% по сравнению с сахарозой 1% сахарозы) (рис. 4.2а и 4.2b). Как показано на рисунке 4.2c, MDV значительно уменьшается общее число генов, которые будут проанализированы и в то же время отфильтрованные гены непосредственно не связаны с влиянием сахароза и крахмал в комбинации. Выход MDV данные были также преобразованы в графический дисплей, отображающий данные организованного функционального класса каждого из генов, дифференциально экспрессирующихся (вверх и вниз регулируемой) в крахмал + сахароза-биопленок (по сравнению с сахарозой выращенных биопленки), на каждом из 4-х временных точек (рис. 4.3). Программное обеспечение MDV удобна для пользователя и способствовала добыча / организация больших и сложных наборов данных из наших экспериментов микрочипов. - . Биопленка изображений Одновременно структурной организации биопленки была проанализирована с помощью КОМСТАТ-DUOSTAT-Амира, включенных в ящик для инструментов (см. пример 3D-реконструкции и количественного анализа крахмала + сахароза выращенных биопленки на рисунке 4.4, см. также видео статьи). Морфология, распределение и структурные отношения EPS и микроколоний могут быть визуализированы с использованием 3D рендеринг поверхности Амира. Крупный план вид выбранной области могут быть созданы, что свидетельствует специфических бактерий-EPS структурных связей на микроуровне (рис. 4.4). Кроме того, тот же набор конфокальной изображения могут быть обработаны КОМСТАТ-DUOSTAT для биомассы / микроколония измерений, пространственное распределение и колокализации клетки бактерий и EPS одновременно. Например, вертикальное распределение EPS и бактерий из поверхности диска, чтобы жидкость фазы рассчитывалась по каждой из оптических разделе трехмерных изображений конфокальной биопленки использованием КОМСТАТ-DUOSTAT (график на рисунке 4.4;. См. видео статьи). Данные показывают более высокие пропорции EPS (красная линия), чем бактерии (зеленая линия) по всей глубине биопленки. Кроме того, большинство бактериальных клеток связаны с EPS (синяя линия), особенно в средних и наружных слоев биопленки. Это наблюдение показывает, что биопленки выращенных в крахмала и сахарозы особенно богаты exopolymers, которые хватая (в тесном контакте с-) большая часть бактериальных клеток; такой структурной организации повышает стабильность и сплоченность биопленки 13. Трехмерной визуализации и количественного измерения конфокальных изображений предоставляют дополнительную информацию о структуре биопленки, которая дополняет и биохимических генных данных выражения (увеличение GtfB типа нерастворимых-глюкан синтеза С. mutans в крахмале + сахароза выращенных биопленки) (Дополнительные примеры см. 5, 6, 11, 13).
Аналитический инструмент-Box помогал нам получить, систематизировать и интегрировать данные из различных биопробы, в котором содержится всеобъемлющий анализ того, как С. mutans могут реагировать на сложные изменения в окружающей среде в результате диеты хозяина взаимодействия найдены в полости рта (подробности см. в Кляйн и др., 2009 11,.. Кляйн и др., 2010 7).

Рисунок 1. Блок-схема аналитического инструмента-Box биопленочной анализа.

Рисунок 2. Конфокальной флуоресценции ЭПС и бактерий в биопленки. Одновременная визуализация EPS (красный) и бактерии / микроколоний (зеленый) в трехмерном оказание стрептококками биопленки образуются на поверхности диска ССЗ.

Рисунок 3. Microarray Data Mining и организация Использование Microarray данных Visualizer (MDV) программного обеспечения. Использование функции диаграммы Венна, чтобы выбрать из генов интересов, а также с последующим добавлением имени гена и функциональная аннотация класса.

Рисунок 4.1. Оценка образование биопленки С. mutans с использованием биохимических анализов (А) и RT-КПЦР (B). INS (нерастворимые экзополисахариды) данные хорошо коррелируют с динамикой gtfB выражении, так и с биомассой биопленки. Сахароза на 1% концентрации для максимального формирование INS, gtfB выражения и накопления биопленки на поверхности SHA в то время как 0,5% сахарозы была минимальной концентрации, необходимые для оптимального развития биопленки используя нашу модель в пробирке. С. mutans клеток, выращенных в присутствии 0,5% сахарозы + 1% крахмала, привел к самой высокой биомассой, и представил более INS, чем другие биопленки, что коррелировало с повышенной gtfB выражение (B2). Эти углеводы концентрации были отобраны для дальнейшего транскриптом анализа.
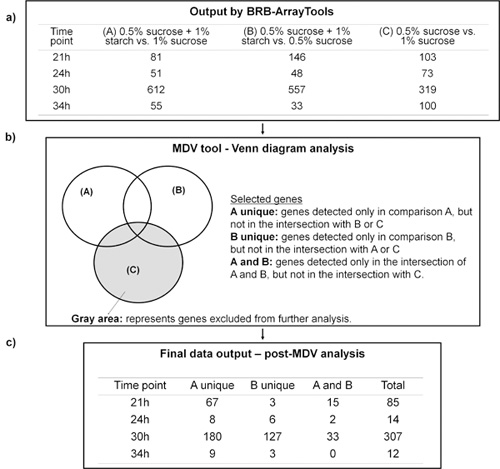
Рисунок 4.2. Microarray данные анализа с использованием BRB-Array инструменты в сочетании с программным обеспечением MDV. а) представлять число генов определяется как разному экспрессируются в каждой сравнения (A, B или C) и момент времени оцениваются с помощью BRB-Array Tools. б) Microarray данных Visualizer (МДВ) с помощью диаграммы Венна для выбора генов, представляющих интерес. в) Гены выбраны в соответствии с анализом MDV.

Рисунок 4.3. С. mutans генов, дифференциально экспрессирующихся в крахмале + сахароза-биопленок (по сравнению с сахарозой, биопленки), на различных временных точках организована функционального класса. Гена аннотации основаны на информации, предоставленной в Лос-Аламосской национальной лаборатории (www.oralgen.lanl.gov) или по литературе доступны на том же сайте.

Рисунок 4.4. Трехмерная визуализация и КОМСТАТ-DUOSTAT анализа крахмала + сахароза-биопленки.
Access restricted. Please log in or start a trial to view this content.
Обсуждение
В этой презентации мы показали два важных компонентов аналитического инструмента-Box (EPS / бактерий визуализации и анализа данных микрочипов / обработки), универсальность и полезность различных анализов интегрированы в систему. Очевидно, ящик для инструментов способствовало всеобъемлю...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Благодарности
Авторы хотели бы поблагодарить д-ра Гэри Се и Герберта Ли за развитие MDV. Мы также благодарим д-ра. Симоне Дуарте, Рамиро Murata, Дже-Гю Чон, Жаклин Abranches, и г-жа Грегуар Стейси за их техническим и научным вкладом в аналитические компоненты набора инструментальных средств. Это исследование было поддержано частично USPHS Исследовательский грант DE018023 из Национального института стоматологических и черепно-лицевых исследований.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Syto 9 | Invitrogen | S34854 | |
| Syto 60 | Invitrogen | S11342 | |
| Dextran conjugated alexa 647 | Invitrogen | D22914 | |
| Olympus FV1000 two-photon laser scanning microscope | Olympus Corporation |
Ссылки
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- Leme, P. aes, Koo, A. F., Bellato, H., Bedi, C. M., G, J. A. C. ury The role of sucrose in cariogenic dental biofilm formation--new insight. J Dent Res. 85, 878-887 (2006).
- Koo, H., Schobel, B. D., Scott-Annem, K., Watson, G., Bowen, W. H., Cury, J. A., Rosalen, P. L., Park, Y. K. Apigenin and tt-farnesol with fluoride effects on S. mutans biofilms and dental caries. J Dent Res. 84, 1016-1020 (2005).
- Koo, H., Xiao, J., Klein, M. I., Jeon, J. G. Exopolysaccharides produced by Streptococcus mutans glucosyltransferases modulate the establishment of microcolonies within multispecies biofilms. J Bacteriol. 192, 3024-3032 (2010).
- Jeon, J. G., Klein, M. I., Xiao, J., Gregoire, S., Rosalen, P. L., Koo, H. Influences of naturally occurring agents in combination with fluoride on gene expression and structural organization of Streptococcus mutans in biofilms. BMC Microbiol. 9, 228-228 (2009).
- Klein, M. I., DeBaz, L., Agidi, S., Lee, H., Xie, G., Lin, A. H. M., Hamaker, B. R., Lemos, J. A., Koo, H. Dynamics of Streptococcus mutans transcriptome in response to starch and sucrose during biofilm development. PLoS ONE. , 0013478-0013478 (2010).
- Lemos, J. A., Abranches, J., Koo, H., Marquis, R. E., Burne, R. A. Protocols to Study the Physiology of Oral Biofilms. Methods Mol Biol. 666, 87-102 (2010).
- Koo, H., Hayacibara, M. F., Schobel, B. D., Cury, J. A., Rosalen, P. L., Park, Y. K., Vacca-Smith, A. M., Bowen, W. H. Inhibition of Streptococcus mutans biofilm accumulation and polysaccharide production by apigenin and tt-farnesol. J Antimicrob Chemother. 52, 782-789 (2003).
- Koo, H., Seils, J., Abranches, J., Burne, R. A., Bowen, W. H., Quivey, R. G. Influence of apigenin on gtf gene expression in Streptococcus mutans UA159. Antimicrob. Agents Chemother. 50, 542-546 (2006).
- Klein, M. I., Duarte, S., Xiao, J., Mitra, S., Foster, T. H., Koo, H. Structural and molecular basis of the role of starch and sucrose in Streptococcus mutans biofilm development. Appl Environ Microbiol. 75, 837-841 (2009).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Anal Biochem. 365, 208-214 (2007).
- Xiao, J., Koo, H. Structural organization and dynamics of exopolysaccharide matrix and microcolonies formation by Streptococcus mutans in biofilms. J Appl Microbiol. 108, 2103-2113 (2010).
- Thurnheer, T., Gmür, R., Shapiro, S., Guggenheim, B. Mass transport of macromolecules within an in vitro model of supragingival plaque. Appl Environ Microbiol. 69, 1702-1709 (2003).
- Chalmers, N. I., Palmer, R. J. J. r, Du-Thumm, L., Sullivan, R., Shi, W., Kolenbrander, P. E. Use of quantum dot luminescent probes to achieve single-cell resolution of human oral bacteria in biofilms. Appl Environ Microbiol. 73, 630-636 (2007).
- Deng, D. M., Hoogenkamp, M. A., Ten Cate, J. M. >, Crielaard, W. Novel metabolic activity indicator in Streptococcus mutans biofilms. J Microbiol Methods. 77, 67-71 (2009).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены