Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une analyse boîte à outils de l'évaluation biochimique, structurel et transcriptomique globale des biofilms buccaux médiée par Streptococcus mutans
Dans cet article
Résumé
Les biofilms formés sur les surfaces dentaires sont très complexes et constamment exposés à innée et exogènes aux défis environnementaux, qui modulent leur architecture, la physiologie et du transcriptome. Nous avons développé une boîte à outils pour examiner la composition, l'organisation structurelle et l'expression des gènes des biofilms buccaux, qui peut être adapté à d'autres domaines de la recherche biofilm.
Résumé
Les biofilms sont très dynamiques, les communautés organisées et structurées de cellules microbiennes empêtré dans une matrice extracellulaire de densité variable et de la composition 1, 2. En général, les biofilms se développent à partir d'attachement microbienne initiale sur une surface suivie de la formation des amas de cellules (ou microcolonies) et le développement et la stabilisation des microcolonies, qui se produisent dans une matrice complexe extracellulaire. La majorité des matrices exopolysaccharides biofilm port (EPS), et les biofilms dentaires ne font pas exception, surtout ceux associés à la maladie carieuse, qui sont principalement médiée par streptocoques mutants 3. Les EPS sont synthétisées par des microorganismes (S. mutans, un contributeur clé) au moyen d'enzymes extracellulaires, tels que le saccharose glucosyltransférases utilisant principalement comme substrat 3.
Les études sur les biofilms formés sur les surfaces dentaires sont particulièrement difficiles en raison de leur exposition constante aux défis environnementaux complexes associés à l'alimentation hôte microbienne interactions se produisant dans la cavité buccale. Une meilleure compréhension de l'évolution dynamique de l'organisation structurelle et de la composition de la matrice, la physiologie et du transcriptome / protéome le profil du biofilm-cellules en réponse à ces interactions complexes permettrait d'améliorer les connaissances actuelles sur la manière dont les biofilms buccaux modulent la pathogénicité. Par conséquent, nous avons développé une boîte à outils analytiques pour faciliter l'analyse de biofilm au niveau structurel, biochimique et moléculaire en combinant des techniques couramment disponibles et roman avec logiciel personnalisé pour l'analyse des données. Standard d'analyse (essais colorimétriques, RT-qPCR et les microarrays) et de nouvelles techniques de fluorescence (pour l'étiquetage simultanée des bactéries et des EPS) ont été intégrés à un logiciel spécifique pour l'analyse de données pour répondre à la nature complexe de la recherche biofilm orale.
La boîte à outils se compose de quatre étapes distinctes mais reliées entre elles (figure 1): 1) essais biologiques, 2) premières données d'entrée, 3) le traitement des données, et 4) l'analyse des données. Nous avons utilisé notre modèle in vitro de biofilm et les conditions expérimentales spécifiques pour démontrer l'utilité et la souplesse de la boîte à outils. Le modèle de biofilm est simple, reproductible et plusieurs répliques d'une expérience unique qui peut être fait simultanément 4, 5. En outre, elle permet une évaluation temporelle, l'inclusion d'espèces microbiennes différentes 5 et l'évaluation des effets des différentes conditions expérimentales (par exemple traitements 6; comparaison des mutants KO vs souche parentale 5, la disponibilité des glucides 7). Ici, nous décrivons deux composantes spécifiques de la boîte à outils, y compris (i) un nouveau logiciel pour l'exploration de données biopuces / organisation (MDV) et à l'analyse d'imagerie de fluorescence (DUOSTAT), et (ii) in situ EPS-étiquetage. Nous fournissons également un cas expérimental montrant comment la boîte à outils peuvent aider à l'analyse des biofilms, organisation des données, l'intégration et l'interprétation.
Protocole
1. ETAPE 1 - ESSAIS BIOLOGIQUES
La méthode utilise des disques de biofilm hydroxyapatite (HA) comme substitut dent (Clarkson produits de chromatographie, Inc, South Williamsport, PA, USA; surface = 2,7 ± 0,2 cm 2) revêtement avec de la salive (mimant la présence de pellicule acquise), placé dans une position verticale 4, 5, 8.
- Dosages biochimiques.
- Les biofilms sont (i) soit homogénéisé par sonication 9 ou (ii) conservés intacts pour les dosages biochimiques 8. La suspension homogénéisée biofilms peuvent être utilisées pour la détermination de la biomasse (poids sec), protéines totales, inorganiques, et extracellulaires et intracellulaires des polysaccharides de composition / contenu (voir détails dans Lemos et al. Protocoles pour étudier la physiologie des biofilms buccaux 8). Les biofilms intacts peuvent être analysés pour leurs réponses physiologiques à l'aide standard de la glycolyse pH-goutte de l'acide-tuant la perméabilité de protons, et des essais d'activité F-ATPase (voir détails dans Lemos et al. Protocoles pour étudier la physiologie des biofilms buccaux 8).
- Analyses du transcriptome.
- Standard ADNc microarray et RT-qPCR protocoles (http://pfgrc.jcvi.org/index.php/microarray/protocols.html par exemple) peuvent être utilisés en combinaison pour la détermination du transcriptome-réponse des agents pathogènes spécifiques (par exemple S. mutans) au sein de biofilms à divers défis environnementaux et thérapeutiques à différents stades de développement 6, 7, 10, 11. Dans la boîte à outils, nous avons inclus deux méthodes qui sont critiques pour l'analyse du transcriptome de biofilms dentaires: 1) la qualité de l'ARN (en raison de la nature hétérogène des populations et la présence de biofilm EPS-matrice), et 2) l'exploration des données et l'interprétation (en raison de vaste ensemble de données généré par micropuce).
- Isolement de l'ARN. Pour répondre à la question de la qualité de l'ARN extrait de biofilms, nous utilisons un protocole optimisé spécialement développé pour l'isolement et la purification d'ARN à partir de cellules bactériennes empêtré dans EPS matrice riche 12 (http://dx.doi.org/10.1016/j . ab.2007.03.021). Ce protocole prévoit Nombre intégrité de l'ARN (RIN) supérieur à 8,5, ce qui est considéré comme optimal pour l'analyse du transcriptome à l'aide deux RT-qPCR et de puces à ADN (pour des exemples, voir 6, 11).
- Standard ADNc microarray et RT-qPCR protocoles (http://pfgrc.jcvi.org/index.php/microarray/protocols.html par exemple) peuvent être utilisés en combinaison pour la détermination du transcriptome-réponse des agents pathogènes spécifiques (par exemple S. mutans) au sein de biofilms à divers défis environnementaux et thérapeutiques à différents stades de développement 6, 7, 10, 11. Dans la boîte à outils, nous avons inclus deux méthodes qui sont critiques pour l'analyse du transcriptome de biofilms dentaires: 1) la qualité de l'ARN (en raison de la nature hétérogène des populations et la présence de biofilm EPS-matrice), et 2) l'exploration des données et l'interprétation (en raison de vaste ensemble de données généré par micropuce).
- Imagerie par fluorescence. L'organisation structurelle des biofilms.
- La plupart des protocoles d'imagerie confocale de fluorescence disponibles dans la littérature se concentre sur l'étiquetage microbienne. L'analyse de l'EPS comme un élément majeur du biofilm a été largement négligée dans la recherche biofilm orale impliquant l'imagerie de fluorescence, à quelques exceptions près 5, 13, 14. Nous avons développé une technique de marquage roman et un logiciel spécifique pour la visualisation reproductibles et la quantification des EPS et des cellules bactériennes simultanément au sein de biofilms intacte. Amira 5 (Visage Imaging GmbH, Berlin, Allemagne) est utilisé pour la reconstruction 3D des biofilms, tandis COMSTAT (disponible à http://www.imageanalysis.dk) et DUOSTAT (http://www.imageanalysis.dk) fournissent les l'analyse quantitative.
- L'imagerie de l'organisation structurelle de l'EPS-bactéries dans les biofilms. Le Fluor Alexa 647-étiquetés dextran conjugué est utilisé pour visualiser les EPS-matrice. Le marqués par fluorescence dextrane sert d'amorce pour la streptococcique glucosyltransférases-gtfs (en particulier GtfB et GtfC), et peut être simultanément incorporé lors de la synthèse de matrice exopolysaccharide au cours du biofilm développement 11.
EPS étiquetage:- Les disques d'hydroxyapatite Manteau avec de la salive, stérilisée par filtration pour 1h, suite à la "Préparation biofilm" protocole n ° 8.
- Pipeter 2,8 ml de milieu de culture dans chaque puits de plaques de 24 puits, et amener à une pièce sombre.
- Ajouter 1 uM de dextran conjugué Alexa Fluor dans le milieu de culture, mélange de colorant dans un milieu de culture par aspiration et refoulement (environ 10 fois).
- Trempez-wash de la salive à revêtement HA (SHA) disques (après 1 h d'incubation) deux fois dans AB tampon stérile et les placer dans le milieu contenant le dextran conjugué Alexa Fluor.
- Recouvrir la plaque avec du papier aluminium et laisser incuber à 37 ° C, 5% de CO 2. La durée du temps d'incubation dépend de chaque modèle expérimental. Le marquage fluorescent dextran ne tache pas les cellules bactériennes à des concentrations utilisées dans ce domaine commedisent 11. Autres EPS techniques de coloration, comme avec calcofluor 14, peuvent être utilisés en combinaison.
- L'étiquetage des bactéries:
- Composants bactéries dans les biofilms sont marquées à la fin de la formation de biofilms en utilisant la norme acides nucléiques fluorescentes tache (par exemple SYTO 9); d'autres techniques de fluorescence (par exemple, des espèces spécifiques marqués par fluorescence des anticorps 15 ou GFP cellules exprimant 16) peut être utilisé pour des cours détecter un ou plusieurs espèces bactériennes dans les biofilms. La figure 2 montre l'étiquetage simultané des EPS (rouge) et les bactéries / microcolonies (vert) dans une image 3D d'un S. 24 h anciens biofilm mutans formée sur la surface d'un disque SHA.
- L'acquisition des images: Chaque biofilm est scannée de 5 à 10 postes choisis au hasard, et Z Series sont générés par sectionnement optique à chacune de ces positions par microscopie confocale à balayage laser à l'aide de protocoles standard. Nous utilisons un Olympus FV 1000 microscope à deux photons (Olympus, Tokyo, Japon) équipée x10 (Carl Zeiss; ouverture numérique 0,45; travail mm de distance 3-4) ou x25 (Olympus LPlan N; NA 1,05; WD 2 mm) d'eau objectifs à immersion. La longueur d'onde d'excitation est 810 nm, longueur d'émission et de filtre pour SYTO 9 (bactéries) est 495/540 OlyMPFC1 filtre, tandis que le filtre pour Alexa Fluor 647 (EPS) est HQ655/40M-2P filtre. Enregistrer et stocker des images brutes.
- L'imagerie de l'organisation structurelle de l'EPS-bactéries dans les biofilms. Le Fluor Alexa 647-étiquetés dextran conjugué est utilisé pour visualiser les EPS-matrice. Le marqués par fluorescence dextrane sert d'amorce pour la streptococcique glucosyltransférases-gtfs (en particulier GtfB et GtfC), et peut être simultanément incorporé lors de la synthèse de matrice exopolysaccharide au cours du biofilm développement 11.
- La plupart des protocoles d'imagerie confocale de fluorescence disponibles dans la littérature se concentre sur l'étiquetage microbienne. L'analyse de l'EPS comme un élément majeur du biofilm a été largement négligée dans la recherche biofilm orale impliquant l'imagerie de fluorescence, à quelques exceptions près 5, 13, 14. Nous avons développé une technique de marquage roman et un logiciel spécifique pour la visualisation reproductibles et la quantification des EPS et des cellules bactériennes simultanément au sein de biofilms intacte. Amira 5 (Visage Imaging GmbH, Berlin, Allemagne) est utilisé pour la reconstruction 3D des biofilms, tandis COMSTAT (disponible à http://www.imageanalysis.dk) et DUOSTAT (http://www.imageanalysis.dk) fournissent les l'analyse quantitative.
2. ETAPE 2 - RAW ENTREE DES DONNEES
Les données d'entrée des premières analyses biochimiques et RT-qPCR directement dans le fichier de données brutes (RDF-fichier MS Excel). Pour les données biopuces, charge à canal unique de diapositives des images numérisées dans JCVI Spotfinder (http://pfgrc.jcvi.org/index.php/bioinformatics.html) ou un logiciel similaire. Créer une grille place selon les spécifications JCVI puis régler manuellement pour s'adapter à tous points dans la grille. Mesurer les valeurs d'intensité de chaque spot et enregistrer en «. MeV" fichiers et stockées en "données de biopuces Raw".
3. ETAPE 3 - TRAITEMENT DES DONNÉES
Organiser les données brutes (biochimiques et RT-qPCR) dans le RDF pour l'analyse statistique. Les transférer dans "Fichier de données traitées" (DPF - fichier MS Excel). Pour l'analyse de l'imagerie de fluorescence et de microréseaux, des logiciels spécifiques (actuellement disponibles et sur mesure) sont utilisés pour traiter les données.
- Données de biopuces:
- Soumettre les données brutes obtenues à l'étape 2 (stockés dans des «données Microarrays Raw») à l'étape de normalisation des données en utilisant un logiciel spécifique. Normaliser les données en utilisant les données de microréseaux JCVI logiciel d'analyse de MIDAS (http://www.tm4.org/midas.html). Utilisez LOWESS et la régularisation déviation standard avec les paramètres par défaut, suivie par l'analyse de reproduire en diapositive. Stockez les fichiers contenant les données normalisées. A ce stade (fichiers de données brutes et normalisées fichiers de données prêt), dépôt de données de biopuces dans une base de données d'accès public (par exemple l'expression des gènes NCBI Omnibus (GEO) base de données à http://www.ncbi.nlm.nih.gov/geo), et enregistrer le numéro d'accession pour la future référence.
- Données d'imagerie de fluorescence
- Quantification de la structure du biofilm.
- Les données quantitatives correspondant à chaque composante structurelle (EPS et les bactéries) des biofilms est calculé en COMSTAT et le DUOSTAT nouvellement développé, qui ont été écrit comme un script dans MATLAB 5.1. Les images brutes de fluorescence sont téléchargés dans le logiciel et analysées comme suit:
- Utilisez COMSTAT pour calculer la biomasse, de l'épaisseur, la distribution et la couche d'autres paramètres qui quantifier et de caractériser la structure tridimensionnelle des biofilms (voir le manuel au http://www.imageanalysis.dk).
- Utilisez DUOSTAT pour calculer la co-localisation des deux composantes du biofilm, tels que EPS et des bactéries (ou d'espèces microbiennes différentes et / ou de composants extracellulaires):
- Mettre en place le chemin de la DUOSTAT dans MATLAB 5.1, et les dossiers image ouverte qui incluent les images des deux composantes du biofilm.
- Choisissez les deux canaux qui seront corrélées et analysées. Les deux piles d'images doit également avoir des tailles de pixels identiques dans les trois dimensions (x, y, z), et le même nombre d'images dans chaque pile.
- Mettre en place des seuils pour chaque canal, selon l'hommeUAL des COMSTAT. Suivez les instructions dans l'interface de fonctionnement du logiciel (voir l'article de la vidéo; manuelle DUOSTAT au http://www.imageanalysis.dk). Saisissez les données obtenues dans le DPF. Voir l'exemple à la figure 4.4.
- Reconstruction tridimensionnelle des biofilms.
- L'architecture en trois dimensions des biofilms est visualisée à l'aide d'Amira. Importez les images de fluorescence enregistrée dans "Dossier images brutes" dans le logiciel, et d'utiliser le rendu Voltex et l'iso-surface pour créer des rendus en 3-D de chacun des composants dans les biofilms (voir Amira manuel à http://www.amira.com / documentation / manuels et relâchez-notes.html). Voir par exemple sur la figure 4.4.
- Les données quantitatives correspondant à chaque composante structurelle (EPS et les bactéries) des biofilms est calculé en COMSTAT et le DUOSTAT nouvellement développé, qui ont été écrit comme un script dans MATLAB 5.1. Les images brutes de fluorescence sont téléchargés dans le logiciel et analysées comme suit:
- Quantification de la structure du biofilm.
4. ETAPE 4 - ANALYSE DE DONNÉES
Les données quantitatives provenant biochimiques, RT-qPCR et COMSTAT-DUOSTAT dosages dans le DPF sont prêtes pour l'analyse statistique. Après l'analyse statistique est effectuée, de graphiques et / ou tables peuvent être construites (voir «Les résultats représentatifs" section).
1. Microarray organisation des données en utilisant le logiciel Microarray données Visualizer (MDV).
En raison de la complexité et la sortie de grands ensembles de données lors de l'utilisation des puces et de multiples conditions expérimentales, nous avons conçu un logiciel de data mining et de l'organisation nommée Microarray données Visualizer (MDV) 7 (disponible à http://www.oralgen.lanl.gov/) .
Après avoir effectué l'analyse statistique en utilisant BRB-ArrayTools (http://linus.nci.nih.gov/BRB-ArrayTools.html) avec une valeur seuil P de 0,001 pour la prédiction de classe et de classe à titre de comparaison, les données générées peuvent être soumises au MDV, comme suit:
- Utilisation d'Excel, convertir les données dans des fichiers BRB MDV texte délimité par des tabulations. Retirez toutes les lettres qui viennent avec le numéro de Tag locus du gène, par exemple: SMU.1423c, supprimer la lettre "c".
- Ouvrez MDV. Allez dans Fichier, puis sélectionnez «Sélectionner la source d'annotation". Choisissez "fichier plat". Lorsqu'on leur a présenté la boîte de dialogue, utilisez les boutons Parcourir pour choisir les fichiers qui contiennent des annotations pour le nom du gène, Gene Ontology (GO) Numéro, voie, et les données de classification fonctionnelle.
- Allez dans Fichier, et l'importation de chacun des fichiers à partir du point 1. Chacun des fichiers représente les conditions expérimentales pour l'analyse. S'il ya plusieurs conditions pour l'analyse, par exemple deux traitements distincts (traitement 1 et 2) et de contrôle (véhicule) passez au point 4.
- Dans ce cas, les comparaisons possibles sont: (a) un traitement de contrôle 1 vs (pour les gènes exprimés différentiellement due au traitement 1), (B) le traitement 2 vs contrôle, et (C) le traitement 1 traitement vs 2. Selon l'hypothèse de travail, autre série de gènes peuvent être sélectionnés.
- Sélectionnez seulement des gènes différentiellement exprimés en comparaison A. (Gènes uniques affectés par le traitement 1 seulement) Dans ce cas, utilisez la fonction Soustraire Réglez les fonctions du menu. Soustraire les gènes de B de comparaison de A, et sauvegarder le résultat. Ensuite, soustrayez C à partir des données obtenues. Les mêmes actions peut être fait pour obtenir uniquement les gènes dans B (gènes uniques affectés par le traitement 2 seulement).
- Pour ne sélectionner que les gènes détectés dans les deux A et B, utilisez la fonction 'Union à partir du menu Set Fonctions de combiner les résultats des comparaisons A et B, et sauvegarder le résultat. Puis soustrayez C à partir des données résultat.
- Ces actions peuvent être visualisées en utilisant le diagramme de Venn, en vedette dans MDV, qui est plus intuitif et facilite fondée sur une hypothèse d'organisation des données. Le diagramme de Venn données de sortie sera affichée dans l'écran (voir la vue d'écran dans la figure 3 et l'article vidéo).
- Pour enregistrer les données dans des fichiers texte délimité par des tabulations, allez dans Fichier et choisissez Exporter.
- Exporter les fichiers texte délimité par des tabulations. Ouvrez les utiliser Excel, et d'organiser les données en sortie du MDV dans les tableaux et / ou clandestines pour un affichage graphique (voir les figures 4.2, 4.3 et l'article vidéo).
5. Les résultats représentatifs
Ici, nous fournissent un exemple de comment la boîte à outils analytiques intègre les divers dosages d'une étude de biofilms avec des variables multiples et des conditions expérimentales.
Cas expérimental:
Dynamique de Streptococcus mutans transcriptome en réponse à l'amidon et le saccharose lors de biofilm développement 7.
Contexte:
Les interactions de l'amidon alimentaire et du saccharose par l'amylase salivaire et l'hôte glucosyltransférases streptococciques pourrait améliorer la formation et la virulence de S. mutans au sein de biofilms par incrémentsasing la synthèse des exopolysaccharides, le métabolisme du sucre et de acidogenicity 11. Ce complexe hôte-pathogène-alimentation interaction peut moduler la formation de biofilms pathogènes liés à la maladie de la carie dentaire. Nous avons effectué une analyse complète biochimique et transcriptomique (y compris le profilage génomique entier) de mieux comprendre comment S. mutans répond à l'amidon et du sucrose à des stades distincts du développement du biofilm, en présence d'amylase 7.
La boîte à outils analytiques a été utilisée pour nous aider à l'intégration des dosages biochimiques et moléculaires de biofilms formés dans diverses conditions expérimentales et les points de temps. La production globale de données utilisant la boîte à outils analytiques sont présentées de manière séquentielle dans la figure 4 (4.1 à 4.4). Il est à noter que l'objectif majeur est ici de démontrer l'utilité de la boîte à outils plutôt que de l'interprétation des données et la discussion.
- En combinant les tests biochimiques et RT-qPCR pour sélectionner les conditions expérimentales pour l'analyse des microréseaux d'autres. Initialement, nous avons testé six sucrose distinctes et 5 d'amidon et de combinaisons de saccharose pour sélectionner les concentrations optimales des hydrates de carbone pour le développement des biofilms de S. mutans en utilisant notre modèle in vitro. La capacité d'examiner simultanément les données issues des essais biologiques nous a guidés dans la sélection de trois concentrations spécifiques (mises en évidence dans la figure 4.1) basée sur (élevée) le montant des exopolysaccharides insolubles et (amélioré) l'expression de gtfB (responsable de la synthèse glucane insoluble) dans les biofilms .
- L'analyse des microréseaux. Les sélectionnés (trois) des concentrations de l'glucides alimentaires ont été utilisés pour former des biofilms, qui ont été retirés au moment précis de points reflétant les différentes étapes du processus de développement des biofilms. Les biofilms (divisé en 3 groupes expérimentaux et de 4 points de temps) ont été soumis à l'analyse du transcriptome en combinant le profilage génomique entier (cDNA microarrays) avec BRB-Array Outils et MDV.
L'ARN a été extrait et purifié à l'aide de biofilms protocole spécifique 12, et ensuite soumis à l'analyse microarray à travers les 4 étapes de processus de la Boîte à outils d'analyse. La figure 4.2 illustre comment le MDV ont aidé à sélectionner les gènes d'intérêt selon notre hypothèse de travail que la combinaison de saccharose et l'amidon des déclencheurs spécifiques réponse transcriptionnelle associée à la virulence accrue (potentiel cariogène) de S. mutans au sein des biofilms. Les données brutes générées par BRB-ArrayTools (figure 4.2a) a été générée par MDV en utilisant le diagramme de Venn (figure 4.2b). Dans cette étude, le groupe de gènes liés à notre hypothèse sont ceux exprimés de façon différentielle par rapport A (0,5% de saccharose + 1% d'amidon vs 1% de saccharose) et B (0,5% de saccharose + amidon 1% vs 0,5% de saccharose), mais pas les gènes en comparaison C (0,5% de saccharose vs 1% de saccharose) (figure 4.2a et 4.2b). Comme le montre la figure 4.2c, le MDV considérablement réduit le nombre total de gènes à analyser et dans le même temps filtrée-out des gènes ne sont pas directement liées à l'influence de saccharose et d'amidon en combinaison. La sortie des données MDV a également été converti en un affichage graphique montrant les données organisées par la classe fonctionnelle de chacun des gènes différentiellement exprimés (haut et bas-réglementées) dans l'amidon + saccharose biofilms (vs saccharose grandi biofilms), à chaque des 4 points de temps (figure 4.3). Le logiciel MDV est convivial et animé l'extraction / l'organisation des données importantes et complexes ensembles de nos expériences de biopuces. - . Biofilm imagerie Parallèlement, l'organisation structurelle des biofilms a été analysée à l'aide COMSTAT-DUOSTAT-Amira inclus dans la boîte à outils (voir un exemple de la reconstruction 3D et l'analyse quantitative de l'amidon + saccharose grandi biofilm à la figure 4.4, voir également Article vidéo). La morphologie, la distribution et la relation structurelle des EPS et des microcolonies peuvent être visualisées en utilisant le rendu de surface 3D par Amira. Close-up vue sur la zone sélectionnée peut être généré, ce qui illustre spécifiques de bactéries EPS relations structurelles à l'échelle microscopique (Figure 4.4). Par ailleurs, le même ensemble d'images confocale peut être traitée par COMSTAT-DUOSTAT pour la biomasse / microcolonie mesures, la distribution spatiale et colocalisation des cellules des bactéries et des EPS en même temps. Par exemple, la distribution verticale des EPS et les bactéries de la surface du disque à la phase liquide a été calculée à partir de chaque section optique des images en trois dimensions biofilm confocale utilisant COMSTAT-DUOSTAT (graphique de la figure 4.4;. Voir article vidéo). Les données montrent des proportions plus élevées de l'EPS (ligne rouge) que les bactéries (ligne verte) à travers la profondeur des biofilms. Par ailleurs, la plupart des cellules bactériennes sont associées à des EPS (ligne bleue), en particulier dans les couches moyennes et externes du biofilm. Cette observation indique que les biofilms cultivés dans l'amidon et le saccharose sont particulièrement riches en exopolymères, qui sont emmailler (en gros contact avec la plupart) des cellules bactériennes; telle organisation structurelle améliore la stabilité et la cohésion du biofilm 13. Les rendus en trois dimensions et des mesures quantitatives des images confocales fournir des informations supplémentaires sur la structure du biofilm, ce qui complète les données d'expression génique et biochimiques (augmentation GtfB type-glucane insoluble synthèse par S. mutans en amidon + saccharose grandi biofilms) (pour des exemples supplémentaires, voir 5, 6, 11, 13).
L'analyse Tool-Box nous a aidé à obtenir, d'organiser et d'intégrer les données provenant des essais biologiques différents, ce qui a fourni une analyse exhaustive de la façon dont S. mutans peut répondre à des changements complexes de l'environnement en raison des interactions hôte-alimentation dans la cavité buccale (voir détails dans Klein et al, 2009 11;.. Klein et al, 2010 7).

Figure 1. Organigramme de l'analytique Boîte à outils pour l'analyse de biofilms.

Figure 2. Imagerie confocale de fluorescence d'EPS et de bactéries dans les biofilms. Visualisation simultanée de l'EPS (rouge) et les bactéries / microcolonies (vert) dans le rendu en trois dimensions de Streptococcus mutans biofilm formé sur la surface du disque SHA.

Figure 3. Microarray Data Mining et organisation à l'aide de données de microréseaux Visualizer (MDV) du logiciel. Utiliser la fonction diagramme de Venn pour sélectionner des gènes d'intérêt, et suivi par l'ajout du nom du gène et l'annotation de classe fonctionnelle.

Figure 4.1. Evaluation de la formation de biofilm par S. mutans en utilisant des analyses biochimiques (A) et RT-qPCR (B). Les données de l'INS (exopolysaccharides insolubles) corrèlent bien avec le profil d'expression gtfB, et avec la biomasse des biofilms. Saccharose à 1% est la concentration maximale pour la formation de l'INS, l'expression et l'accumulation gtfB biofilm à la surface tandis que les SHA sucrose 0,5% a été la concentration minimale requise pour le développement du biofilm optimale en utilisant notre modèle in vitro. S. cellules mutans cultivées en présence de saccharose 0,5% d'amidon + 1% était des plus de biomasse, et a présenté plus de l'INS que des autres films, qui sont corrélés avec l'expression accrue gtfB (B2). Ces concentrations de glucides ont été sélectionnés pour l'analyse du transcriptome supplémentaires.
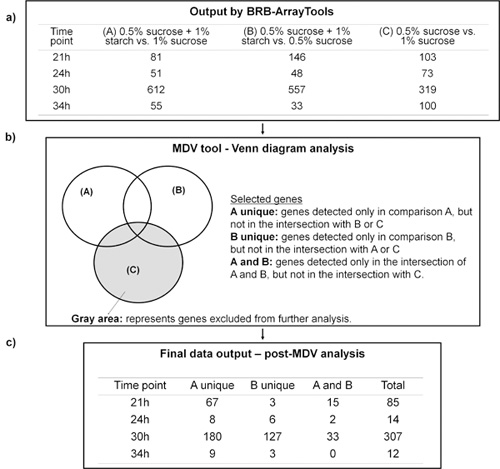
Figure 4.2. Biopuces d'analyse de données en utilisant BRB-Array Outils en conjonction avec le logiciel de MDV. a) Représente le nombre de gènes différentiellement exprimés détecté comme dans chaque comparaison (A, B ou C) et le point de temps évaluées à l'aide BRB-Array Outils. b) Les données biopuces Visualizer (MDV) en utilisant le diagramme de Venn pour sélectionner des gènes d'intérêt. c) des gènes sélectionnés selon l'analyse de MDV.

Figure 4.3. S. mutans gènes différentiellement exprimés en amidon + saccharose biofilms (vs saccharose biofilms), à différents points temporels organisé par la classe fonctionnelle. Annotations des gènes sont basées sur des informations fournies par le Laboratoire national de Los Alamos (www.oralgen.lanl.gov) ou par la littérature publiée disponible sur le site même.

Figure 4.4. Tridimensionnelle rendu et COMSTAT-DUOSTAT analyse de l'amidon + saccharose biofilm.
Access restricted. Please log in or start a trial to view this content.
Discussion
Dans cette présentation, nous avons démontré deux composantes essentielles de l'analyse Tool-Box (EPS / bactéries d'imagerie et de données de microréseaux minière / traitement), la polyvalence et l'utilité des différents essais intégrés dans le système. De toute évidence, la boîte à outils a facilité l'analyse complète (comparatif) et simultanée des différents aspects de la biochimie des biofilms, l'architecture et l'expression des gènes en réponse à différentes conditions ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Remerciements
Les auteurs tiennent à remercier le Dr Gary Xie et Herbert Lee pour le développement de la MDV. Nous remercions également les docteurs. Simone Duarte, Ramiro Murata, Jae-gyu Jeon, Abranches Jacqueline, et Mme Stacy Grégoire pour leur contribution technique et scientifique pour les composants analytiques de la boîte à outils. Cette étude a été soutenue en partie par USPHS subventions de recherche DE018023 de l'Institut national de recherche dentaire et craniofaciale.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Syto 9 | Invitrogen | S34854 | |
| Syto 60 | Invitrogen | S11342 | |
| Dextran conjugated alexa 647 | Invitrogen | D22914 | |
| Olympus FV1000 two-photon laser scanning microscope | Olympus Corporation |
Références
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- Leme, P. aes, Koo, A. F., Bellato, H., Bedi, C. M., G, J. A. C. ury The role of sucrose in cariogenic dental biofilm formation--new insight. J Dent Res. 85, 878-887 (2006).
- Koo, H., Schobel, B. D., Scott-Annem, K., Watson, G., Bowen, W. H., Cury, J. A., Rosalen, P. L., Park, Y. K. Apigenin and tt-farnesol with fluoride effects on S. mutans biofilms and dental caries. J Dent Res. 84, 1016-1020 (2005).
- Koo, H., Xiao, J., Klein, M. I., Jeon, J. G. Exopolysaccharides produced by Streptococcus mutans glucosyltransferases modulate the establishment of microcolonies within multispecies biofilms. J Bacteriol. 192, 3024-3032 (2010).
- Jeon, J. G., Klein, M. I., Xiao, J., Gregoire, S., Rosalen, P. L., Koo, H. Influences of naturally occurring agents in combination with fluoride on gene expression and structural organization of Streptococcus mutans in biofilms. BMC Microbiol. 9, 228-228 (2009).
- Klein, M. I., DeBaz, L., Agidi, S., Lee, H., Xie, G., Lin, A. H. M., Hamaker, B. R., Lemos, J. A., Koo, H. Dynamics of Streptococcus mutans transcriptome in response to starch and sucrose during biofilm development. PLoS ONE. , 0013478-0013478 (2010).
- Lemos, J. A., Abranches, J., Koo, H., Marquis, R. E., Burne, R. A. Protocols to Study the Physiology of Oral Biofilms. Methods Mol Biol. 666, 87-102 (2010).
- Koo, H., Hayacibara, M. F., Schobel, B. D., Cury, J. A., Rosalen, P. L., Park, Y. K., Vacca-Smith, A. M., Bowen, W. H. Inhibition of Streptococcus mutans biofilm accumulation and polysaccharide production by apigenin and tt-farnesol. J Antimicrob Chemother. 52, 782-789 (2003).
- Koo, H., Seils, J., Abranches, J., Burne, R. A., Bowen, W. H., Quivey, R. G. Influence of apigenin on gtf gene expression in Streptococcus mutans UA159. Antimicrob. Agents Chemother. 50, 542-546 (2006).
- Klein, M. I., Duarte, S., Xiao, J., Mitra, S., Foster, T. H., Koo, H. Structural and molecular basis of the role of starch and sucrose in Streptococcus mutans biofilm development. Appl Environ Microbiol. 75, 837-841 (2009).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Anal Biochem. 365, 208-214 (2007).
- Xiao, J., Koo, H. Structural organization and dynamics of exopolysaccharide matrix and microcolonies formation by Streptococcus mutans in biofilms. J Appl Microbiol. 108, 2103-2113 (2010).
- Thurnheer, T., Gmür, R., Shapiro, S., Guggenheim, B. Mass transport of macromolecules within an in vitro model of supragingival plaque. Appl Environ Microbiol. 69, 1702-1709 (2003).
- Chalmers, N. I., Palmer, R. J. J. r, Du-Thumm, L., Sullivan, R., Shi, W., Kolenbrander, P. E. Use of quantum dot luminescent probes to achieve single-cell resolution of human oral bacteria in biofilms. Appl Environ Microbiol. 73, 630-636 (2007).
- Deng, D. M., Hoogenkamp, M. A., Ten Cate, J. M. >, Crielaard, W. Novel metabolic activity indicator in Streptococcus mutans biofilms. J Microbiol Methods. 77, 67-71 (2009).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon